Method Article
Mesure de la migration et de la formation du biofilm de diverses bactéries
Dans cet article
Résumé
Nous présentons ici une méthode pratique pour l’isolement et l’identification des micro-organismes au sein de l’hôte. De cette façon, les propriétés physicochimiques des micro-organismes et les modes de vie possibles chez l’hôte sont clairement décrits.
Résumé
Comme les microbes qui se développent dans le corps de l’hôte ont principalement des capacités d’adaptation qui facilitent leur survie, les méthodes de classification et d’identification de leur nature seraient bénéfiques pour faciliter leur caractérisation. À l’heure actuelle, la plupart des études ne se concentrent que sur une méthode de caractérisation spécifique ; Cependant, l’isolement et l’identification des microorganismes de l’hôte sont un processus continu qui nécessite généralement plusieurs méthodes de caractérisation combinatoire. Dans cet article, nous décrivons des méthodes permettant d’identifier la capacité de formation de biofilms microbiens, l’état de respiration microbienne et leur comportement en chimiotaxie. Les méthodes sont utilisées pour identifier cinq microbes, dont trois ont été isolés dans le tissu osseux de rats Sprague-Dawley (SD) (Corynebacterium stationis, Staphylococcus cohnii subsp. urealyticus et Enterococcus faecalis) et deux de l’American Type Culture Collection (ATCC)-Staphylococcus aureus ATCC 25923 et Enterococcus faecalis V583. Les microbes isolés du tissu osseux du rat SD comprennent les microbes à Gram positif. Ces microbes se sont adaptés pour prospérer dans des environnements stressants et limitant les nutriments au sein de la matrice osseuse. Cet article vise à fournir aux lecteurs le savoir-faire spécifique nécessaire pour déterminer la nature et le comportement des microbes isolés en laboratoire.
Introduction
L’hôte mammifère représenté par le corps humain contient un grand nombre de micro-organismes. Ces micro-organismes sont largement distribués dans la bouche, le tube digestif, l’intestin et le sang de l’hôte et ont des effets différents sur la santé de l’hôte. La cavité buccale est l’hôte d’une pléthore de microbes qui peuvent moduler la susceptibilité de l’hôte aux infections. Des microbes tels que les streptocoques (p. ex., S. mitis/oralis, S. pseudopneumoniae et S. infantis) et Prevotella spp. colonisent la cavité buccale, formant un biofilm multi-espèces à la surface de la langue, provoquant une mauvaise haleine et fonctionnant comme un réservoir microbien pour les infections microbiennes. Ces agents pathogènes peuvent infecter l’os de la mâchoire en infiltrant les ligaments parodontaux qui maintiennent la racine de la dent dans l’os de la mâchoire1. La caractérisation de ces microbes isolés du corps de l’hôte est souvent un processus fastidieux, en particulier lorsque les microbes présentent des traits individuels nécessitant un traitement et des conditions de croissance spécifiques. Les microbes, tels que les pathogènes Helicobacter pylori, Clostridium difficile et Fusobacterium nucleatum, se développent dans l’environnement hostile de l’intestin, avec des besoins spécifiques en oxygène, en nutriments et en croissance, ce qui présente un défi dans les processus de caractérisation, en particulier dans l’étude de la pathogénicité de ces microbes. Par conséquent, une méthode standardisée d’isolement et d’étude de ces micro-organismes est nécessaire pour que les scientifiques et les praticiens puissent développer de nouveaux traitements médicaux.
Ce protocole utilise des microbes qui se développent dans la matrice osseuse des rats. Traditionnellement, à l’exception du système osseux représenté par la mâchoire, où la présence de dents rend la matrice osseuse plus sensible aux infections que les autres os1, on croit généralement que l’os sain de l’hôte est un environnement stérile. Cependant, des études ont montré que les micro-organismes pénètrent dans la circulation systémique à travers la paroi intestinale, affectant finalement la minéralisation osseuse2. À titre de preuve de concept, nous utilisons le protocole décrit pour caractériser les propriétés biochimiques d’isolats microbiens provenant du fémur et du tibia de rats SD sains (Corynebacterium stationis, Staphylococcus cohnii subsp. et Enterococcus faecalis). Ces isolats microbiens ont été sélectionnés car l’os est un environnement fermé et hypoxique, et la caractérisation de ces microbes à partir de l’os peut être une tâche difficile. Divers articles ont détaillé les processus utilisés dans l’étude de ces microbes ; Cependant, il existe peu de protocoles complets permettant d’identifier les microorganismes isolés par l’hôte.
Pour établir les conditions de culture appropriées, les besoins en oxygène du microbe doivent être compris via l’utilisation d’un milieu de thioglycolate fluide (FTM). Les microbes ayant des besoins différents en oxygène forment des couches stratifiées dans le liquide FTM clair3. Sur la base du profil de stratification, les besoins en oxygène du microbe sont ensuite utilisés pour étudier la croissance des cellules. Les microbes qui se développent à la surface du liquide FTM sont des aérobies obligatoires, tandis que les microbes qui se développent au fond sont des anaérobies obligatoires. Les microbes qui se développent en suspension dans le liquide FTM sont des anaérobies facultatifs ou aérotolérants. Le taux de croissance microbienne est établi en observant la phase de croissance exponentielle des cellules microbiennes. Le profil de croissance est ensuite comparé à la formation du biofilm du microbe. Les biofilms sont en grande partie composés de plusieurs espèces qui affectent directement et indirectement la santé des autres. Au cours de ce processus, les interactions bénéfiques entre les communautés microbiennes sélectionnent l’attachement, fournissant une structure spatiale qui favorise l’évolution des interactions réciproques actives. Par exemple, la co-culture de Paenibacillus amylolyticus et de Xanthomonas retroflexus présente des interactions symbiotiques facultatives dans un environnement statique, favorisant la croissance rapide du biofilm13. Les microbes s’adaptent aux tissus de l’hôte via la formation de biofilms pour une localisation durable, se protégeant contre les environnements difficiles et échappant au système immunitaire de l’hôte 4,5,6,7. Les biofilms sont généralement des structures denses et multicouches qui aident les micro-organismes à résister aux stimuli sous-critiques externes ; par exemple, E. faecalis dans la pulpe dentaire augmente sa résistance aux antibiotiques en augmentant la formation de biofilm lorsqu’il est confronté à des sous-concentrations de tétracycline et de vancomycine8.
La chimiotaxie permet aux micro-organismes de se déplacer en fonction de gradients chimiques, et les voies de signalisation sont largement distribuées dans une variété de bactéries pathogènes. Certains micro-organismes pathogènes migrent vers des sites spécifiques, sous la direction de signaux chimiques, pour provoquer des maladies infectieuses14. Par exemple, Xanthomonas spp. expriment 19 chimiorécepteurs et 11 flagellines dans l’hôte, déclenchant la liaison bactérienne et, finalement, l’ulcération15. Il existe également des protéines spécifiques dans les bactéries (protéines de liaison à la pectine) qui guident la migration spécifique des bactéries vers les nutriments, ce qui peut conduire à une meilleure croissance16. Ceci est également essentiel pour les bactéries qui peuvent exister dans des environnements pauvres en nutriments. Les cellules microbiennes s’appuient souvent sur la motilité chimiotactique pour se concentrer sur un environnement de croissance propice tout en échappant aux autres cellules prédatrices et aux toxines qui nuisent à la viabilité cellulaire. En nous appuyant sur des approches de gélose molle précédemment établies pour déterminer la chimiotaxie, nous développons une méthode diffusible pour générer un gradient de chimioattractant afin de tester la chimiotaxie microbienne.
Cet article décrit les méthodes de détermination des conditions de croissance, de la formation du biofilm et du tropisme chimique des bactéries, en utilisant Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 et Enterococcus faecalis V583 comme exemples (voir Figure 1). L’optimisation des conditions de croissance microbienne utilise la FTM pour déterminer les besoins en oxygène du microbe, tandis que la formation du biofilm utilise des surfaces en verre comme support solide, et la masse du biofilm est contre-colorée avec du violet cristallin. Le tropisme chimique du microbe repose sur son comportement chimiotactique, où, grâce à l’impression 3D (figure supplémentaire S1), une méthode standardisée est utilisée pour générer un réservoir chimique pour le chimioattractant dans une matrice de gélose molle (figure supplémentaire S2).
Protocole
REMARQUE : Voir le tableau des matériaux pour plus de détails sur tous les matériaux et équipements utilisés dans ce protocole. Utilisez des techniques aseptiques pour éviter la contamination.
1. Récupération bactérienne pour obtenir une seule colonie
- Préparez des plaques de gélose solide.
- Préparez un milieu de bouillon formidable (TB) contenant de l’agar, chaque litre de liquide contenant 11,8 g de tryptone, 23,6 g d’extrait de levure, 9,4 g de K2HPO4, 2,2 g de KH2PO4 et 15 g d’agar, pH = 7.
- Autoclavez la solution à 121 °C pendant 20 min à l’aide d’un bouchon perméable à l’air ou d’un film d’étanchéité de bouteille avec une bouche d’aération.
- Avant que le liquide ne refroidisse et ne se solidifie, verser le bouillon de tuberculose contenant de la gélose dans des boîtes de Pétri en plastique de 10 cm à un volume de 25 ml par assiette.
REMARQUE : Le bouillon doit être versé dans la plaque avant qu’il ne se solidifie à des températures inférieures à 60 °C. Utilisez des techniques aseptiques pour prévenir la contamination.
- Inoculation bactérienne à la plaque
- Prélever les souches cibles stockées à −80 °C et les décongeler à température ambiante dans une enceinte de sécurité biologique.
- À l’aide d’une pipette de 10 μL, prélever 10 μL de suspension bactérienne et l’étaler sur la plaque de tuberculose par stries.
- Scellez la plaque pour éviter toute contamination. Placez la plaque dans un incubateur biochimique à 37 °C. Vérifiez visuellement la croissance de la colonie après ~24 h. Choisissez des colonies uniques et amplifiez-les par PCR pour confirmer qu’elles ne contiennent qu’un seul type de bactérie.
2. Culture liquide bactérienne jusqu’à la phase de croissance logarithmique
- Préparez le milieu de culture liquide.
- Reportez-vous à la préparation des plaques de gélose solide à l’étape 1.1.1. pour préparer un milieu liquide TB, dont 1 L contient 11,8 g de tryptone, 23,6 g d’extrait de levure, 9,4 g de K2HPO4 et 2,2 g de KH2PO4, pH ~7.
REMARQUE : N’ajoutez pas d’agar. Les bactéries se développent plus rapidement dans un environnement de nutriments liquides. - Autoclavez la solution à 121 °C pendant 20 min à l’aide d’un bouchon perméable à l’air ou d’un film d’étanchéité de bouteille avec une bouche d’aération.
- Stocker le milieu de culture liquide à 4 °C.
- Reportez-vous à la préparation des plaques de gélose solide à l’étape 1.1.1. pour préparer un milieu liquide TB, dont 1 L contient 11,8 g de tryptone, 23,6 g d’extrait de levure, 9,4 g de K2HPO4 et 2,2 g de KH2PO4, pH ~7.
- Inoculation et culture bactériennes
- Dans l’enceinte de sécurité biologique, prélever une seule colonie à l’aide d’une boucle d’inoculation ou d’une pipette de 10 μL et inoculer les bactéries dans le milieu liquide de TB préparé dans un erlenmeyer.
- Fermez l’erlenmeyer avec de la gaze et placez-le dans un agitateur (200 tr/min) à 37 °C pour cultiver les bactéries.
REMARQUE : Le milieu liquide devient progressivement trouble sur 5 h ; Le temps nécessaire pour devenir trouble varie selon les bactéries.
3. Expérience FTM et détermination de la courbe de croissance
- Préparation du milieu fluide à base de thioglycolate (FTM)
- Préparez un milieu de thiolglycolate (TM), dont chaque litre contient 15 g de tryptone, 5 g d’extrait de levure, 5 g de glucose, 0,5 g de L-cystéine, 0,5 g de thioglycolate de sodium, 2,5 g de chlorure de sodium, 0,001 g de résazurine et 0,75 g d’agar.
REMARQUE : Le milieu solide disponible dans le commerce peut facilement être reconstitué avec de l’eau et stérilisé (étape 3.1.2.). - Pesez 29,3 g de milieu solide au thioglycolate, ajoutez 1 L d’eau distillée, chauffez et remuez jusqu’à dissolution complète et jusqu’à ce que la solution devienne rose.
- Ajouter TM (19 ml) dans un tube à essai avec un bouchon d’étanchéité. Après le scellement, autoclavez la solution à 121 °C pendant 20 min à l’aide d’un bouchon perméable à l’air ou d’un film d’étanchéité de bouteille avec une bouche d’aération.
REMARQUE : Après chauffage, la solution devient jaune clair.
- Préparez un milieu de thiolglycolate (TM), dont chaque litre contient 15 g de tryptone, 5 g d’extrait de levure, 5 g de glucose, 0,5 g de L-cystéine, 0,5 g de thioglycolate de sodium, 2,5 g de chlorure de sodium, 0,001 g de résazurine et 0,75 g d’agar.
- Inoculation, culture et observation de bactéries
- Préparez la suspension bactérienne pour qu’elle atteigne une DO de 0,6 à 0,8 dans un milieu liquide pour la tuberculose (voir l’étape 2.2.).
- Ouvrez le bouchon d’étanchéité du tube à essai dans des conditions aseptiques dans l’enceinte de sécurité biologique, ajoutez dès que possible 1 ml de la culture bactérienne dans le tube à essai contenant du FTM et refermez immédiatement le tube à essai.
- Agitez doucement le tube pour vous assurer que les bactéries sont réparties uniformément dans le liquide et placez le tube à essai à 37 °C pour une culture statique.
- Après 48 h d’incubation, observez la croissance de différentes bactéries dans le FTM dans divers tubes à essai et prenez des photos.
- Détermination de la courbe de croissance dans des conditions aérobies
- Préparez la culture bactérienne initiale avec une valeur de DO appropriée (voir étape 2.2.) et cultivez les bactéries jusqu’à ce qu’elles atteignent la phase de croissance logarithmique.
REMARQUE : Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 et Enterococcus faecalis V583 utilisés dans ce protocole ont mis ~5 h pour atteindre la phase logarithmique. - Ajouter 200 μL de la culture bactérienne (de l’étape 3.3.1) dans un milieu liquide TB dans chaque puits d’une plaque de 96 puits en trois exemplaires. À l’aide d’un lecteur de microplaques, déterminez la valeur de DO à 600 nm et diluez les différentes cultures bactériennes de sorte que la différence de DO entre les différents puits contenant des bactéries et le milieu liquide de la tuberculose (à blanc) soit comprise entre 0,01 et 0,02.
- Prenez une nouvelle plaque à 96 puits, ajoutez un milieu de tuberculose, transférez les différentes cultures bactériennes diluées de l’étape 3.3.2., en trois exemplaires, sur la plaque et placez-la sur un établi ultra-propre.
- Scellez la plaque à 96 puits avec du ruban adhésif d’étanchéité, placez-la dans le lecteur de microplaques et réglez le programme comme suit :
- Réglez la température à 37 °C ± 0,5.
- Réglez la boucle cinétique sur un total de 60 cycles, chacun d’une durée de 30 min.
- Réglez l’absorbance à 600 nm.
- Réglez l’agitation sur 1 600 s par cycle.
- Exécutez le programme et arrêtez-le lorsque la courbe de croissance se stabilise.
- Préparez la culture bactérienne initiale avec une valeur de DO appropriée (voir étape 2.2.) et cultivez les bactéries jusqu’à ce qu’elles atteignent la phase de croissance logarithmique.
- Détermination de la courbe de croissance dans des conditions anaérobies
- Suivez l’étape 3.3. mais conservez la plaque à 96 puits avec le milieu TB et les suspensions bactériennes dans un incubateur anaérobie avant de sceller la plaque avec du ruban d’étanchéité. Pompez et remplissez la boîte anaérobie avec de l’azote pour réduire la concentration d’oxygène à <3 %. Scellez la plaque à 96 puits avec du ruban adhésif et laissez-la dans la boîte anaérobie.
4. Test de capacité de formation de biofilm
- Cultivez les bactéries jusqu’à la phase de croissance logarithmique en milieu liquide.
- Se référer à la section 2 pour inoculer la bactérie dans le milieu liquide de la tuberculose.
- Cultivez les bactéries pendant la nuit dans un agitateur à 37 °C, en veillant à ce que la DO600 du milieu de culture bactérienne soit comprise entre 0,6 et 0,8.
- Divisez la culture bactérienne et cultivez dans différentes conditions.
- Lorsque la DO600 de la culture bactérienne est comprise entre 0,6 et 0,8, ajoutez 10 mL de cette suspension bactérienne dans un tube à essai en verre.
- Divisez chaque culture bactérienne en deux groupes : les conditions aérobies et anaérobies, et effectuez toutes les opérations en trois exemplaires.
- Pour le groupe aérobie, enveloppez le tube à essai contenant la suspension bactérienne avec du papier d’aluminium et incubez-le dans un bain-marie à 37 °C à température constante.
- Pour le groupe anaérobie, enveloppez le tube contenant la suspension bactérienne avec du papier d’aluminium et transférez-le dans un incubateur anaérobie. Retirez la feuille d’étain dans l’incubateur anaérobie et réduisez la concentration d’oxygène dans l’incubateur à <3 % en la pompant et en la remplissant d’azote. Fermez l’éprouvette à l’aide d’un bouchon en verre dépoli, retirez-la après l’étanchéité et incuberez-la dans un bain-marie à 37 °C.
- Toutes les 6 h, prélever trois tubes à essai de chacun des deux groupes (aérobie/anaérobie) pour observer la croissance du biofilm au fond, et colorer après l’étape 4.3.
- Jetez la suspension bactérienne et séchez le biofilm.
- Sortez les tubes à essai à différents moments et aspirez soigneusement la couche supérieure de la suspension bactérienne. Observez le biofilm blanc adhérer au fond du tube à essai.
REMARQUE : Veillez à ne pas perturber le biofilm lors de l’aspiration de la suspension bactérienne. - Ajoutez soigneusement 2 ml de solution saline tamponnée au phosphate (PBS) le long de la paroi du tube à essai, secouez-le doucement pour éliminer les bactéries planctoniques adsorbées dans le biofilm et aspirez soigneusement le tampon PBS.
- Mettez le tube à essai avec le biofilm adsorbé sur le fond dans le four pour qu’il sèche pendant 30 min.
- Sortez les tubes à essai à différents moments et aspirez soigneusement la couche supérieure de la suspension bactérienne. Observez le biofilm blanc adhérer au fond du tube à essai.
- Coloration
- Sortez les tubes à essai, ajoutez 2 ml de solution de coloration violette cristalline à 0,1 % dans chaque tube à essai et colorez les biofilms pendant 30 minutes à température ambiante.
- À l’aide d’une pipette, aspirez soigneusement la solution de colorant violet cristallin. Ajoutez soigneusement de l’eau distillée le long de la paroi du tube à essai pour éliminer la solution de colorant violet cristallin restante, en répétant 3x-5x jusqu’à ce que l’eau distillée ajoutée devienne presque incolore. Observez le biofilm violet au fond du tube à essai.
- Séchez à nouveau le tube à essai.
- Dissoudre le biofilm avec de l’éthanol à 95 % et mesurer la DO600 de la solution de biofilm.
- Sortez le tube à essai séché, ajoutez 10 ml d’éthanol à 95 % dans chaque tube à essai et secouez soigneusement pour vous assurer que le violet cristallin a été complètement dissous dans l’éthanol.
- Avec un volume de 200 μL par puits, ajoutez du violet de cristal dans de l’éthanol à une plaque de 96 puits et mesurez son absorbance à une longueur d’onde de 600 nm.
REMARQUE : Selon la loi de Beer-Lambert, l’absorbance est linéairement liée à la concentration de la solution, de sorte que la valeur de OD600 peut refléter la concentration de violet cristallin, qui reflète la quantité de biofilm dans la suspension bactérienne à différents moments.
5. Test de chimiotaxie bactérienne
- Préparer des plaques de gélose eutrophe différentielle.
- Reportez-vous à l’étape 1 pour préparer un milieu MH demi-dose contenant 0,5 % d’agar, dont chaque litre contient 11,8 g de tryptone, 23,6 g d’extrait de levure, 9,4 g de K2HPO4, 2,2 g de KH2PO4 et 5 g d’agar, pH ~ 7.
- Reportez-vous à l’étape 1 pour préparer 5 milieux TB contenant 0,5 % d’agar, dont chaque litre contient 59 g de tryptone, 118 g d’extrait de levure, 47 g de K2HPO4, 11 g de KH2PO4 et 5 g d’agar, pH ~ 7.
- Autoclavez la solution à 121 °C pendant 20 min à l’aide d’un bouchon perméable à l’air ou d’un film d’étanchéité de bouteille avec une bouche d’aération.
- Avant que le liquide ne soit refroidi et solidifié, versez 25 ml de bouillon MH demi-dose (contenant 0,5 % d’agar) par assiette, couvrez l’assiette avec un couvercle imprimé en 3D et placez-la dans une enceinte de sécurité biologique. Après refroidissement, retirez le couvercle pour observer le puits cylindrique (1,4 cm de diamètre) dans le milieu semi-solide.
REMARQUE : Le couvercle imprimé en 3D avec un matériau en résine ne peut pas être stérilisé à l’aide de vapeur à haute température. Pour tuer les bactéries de surface, il peut être pulvérisé avec de l’éthanol à 95 %, puis irradié avec une lampe UV pendant 30 min dans une enceinte de sécurité biologique. Lorsque vous retirez le couvercle, appliquez une force dans le sens vertical et soulevez-le avec précaution pour éviter d’endommager la gélose semi-solide. - Avant que le bouillon 5x TB (contenant 0,5 % d’agar) ne se solidifie, utilisez une pipette pour ajouter le bouillon 5x TB dans le puits de la plaque semi-solide jusqu’à ce que le bouillon affleure la surface du milieu.
- Refroidir la plaque dans une enceinte de sécurité biologique pour obtenir une plaque de gélose semi-solide contenant des corps eutrophes. Veillez à éviter la contamination de la plaque par des micro-organismes environnementaux.
REMARQUE : Le milieu semi-solide est encore un peu fluide et, par conséquent, doit être complètement refroidi avant d’être stocké à l’envers.
- Inoculation bactérienne, culture et photographie
- Placez la plaque de gélose avec le milieu de culture semi-solide dans une enceinte de sécurité biologique et laissez-la refroidir pendant ~30 min sous irradiation UV pour éviter la contamination bactérienne.
- Utilisez une pipette de 10 μL pour prélever 4 μL de la suspension bactérienne cultivée à une DO de ~0,6-0,8, insérez la pointe de la pipette au centre des boîtes de Pétri en plastique et retirez la suspension bactérienne à la pipette.
- Attendez que la suspension bactérienne soit absorbée dans le milieu pendant 1 min. Lorsque la suspension bactérienne a été complètement absorbée, divisez la plaque en deux groupes (aérobie/anaérobie) en trois exemplaires.
- Scellez le groupe aérobie avec du ruban adhésif et placez ces plaques à l’envers dans un incubateur biochimique à 37 °C pour la culture statique. Scellez le groupe anaérobie dans une boîte anaérobie comme décrit à l’étape 4.2, en inversant les plaques à 37 °C pour la croissance.
- Vérifiez la propagation et la migration des bactéries toutes les 12 h (à 0 h, 12 h, 24 h, 36 h et 48 h) et utilisez le système d’imagerie pour prendre des images de la plaque.
- Utilisez le logiciel ImageJ pour calculer les distances de migration bactérienne.
- Importez le fichier image acquis à l’aide du système d’imagerie.
- Réglez la barre d’échelle et appliquez-la à toutes les images.
- Lorsque vous prenez des images, ajoutez une règle, créez un segment de ligne, sélectionnez un segment de ligne droite (par exemple, 1 cm, puis réglez la longueur de ce segment de ligne sur 1 cm) et marquez la longueur du tableau.
- Ouvrez la fenêtre Définir l’échelle ; cliquez sur Analyser | Réglez l’échelle.
- Saisissez la longueur réelle dans Distance connue et Unité de longueur.
- Vérifiez Global.
- Entrez l’adresse du dossier de l’image d’origine et l’adresse du fichier de sortie.
- À l’aide de l’outil de ligne droite , sélectionnez les communautés une par une et ajustez la tolérance jusqu’à ce que la ligne d’outil droite s’adapte à la limite de la colonie bactérienne s’étendant des deux côtés.
- Mesurez la distance de migration de la colonie du centre aux deux extrémités séparément et dérivez la longueur en ligne droite en cliquant sur Analyser | Mesurer.
- Répétez les étapes 5.3.3 et 5.3.4 jusqu’à ce que toutes les communautés soient mesurées et enregistrez les résultats dans un fichier .csv pour une analyse plus approfondie.
Résultats
Ce travail décrit les approches adoptées pour caractériser les microbes isolés du microbiome hôte (Figure 1). À titre de preuve de concept, trois microbes ont été isolés de l’hôte du rat SD (C. stationis, S. cohnii et E. faecalis), et deux micro-organismes acquis commercialement (S. aureus ATCC 25923 et E. faecalis V583) ont été testés à l’aide de ce protocole. Pour établir les besoins en oxygène de chaque microbe à l’aide de la FTM, nous avons ajouté deux organismes témoins, un anaérobie obligatoire (Fusobacterium nucleatum ATCC 25586) et un anaérobie facultatif (Pseudomonas aeruginosa PAO1). Les résultats ont montré que les cinq microbes testés étaient des anaérobies facultatifs (figure 2A), ce qui correspond aux profils précédemment rapportés de ces microbes11. Pour élucider leurs besoins en oxygène, les microbes ont été cultivés dans des conditions aérobies et anaérobies. Tous les microbes testés ont présenté une courbe de croissance sigmoïdale dans des conditions aérobies et anaérobies ; cependant, la densité microbienne, telle qu’indiquée par la DOde 600 nm, a montré que les cinq microbes ont une préférence plus élevée pour les environnements riches en oxygène (Figure 2B,C). Ces observations sont cohérentes avec les capacités microbiennes à s’infiltrer et à infecter les os du rat, qui sont généralement considérés comme hypoxiques.
On a constaté que les cinq isolats bactériens formaient naturellement des biofilms, où la masse de biofilm produite était plus élevée dans des conditions anaérobies. En comparant la courbe de croissance, on a constaté que les biofilms d’E. faeclis et de S. aureus ATCC 25923 se formaient activement pendant la phase de croissance stationnaire dans des environnements aérobies et anaérobies (figure 3). Cette observation suggère que la formation du biofilm dépend de l’état de croissance des cellules, éventuellement régulé par des molécules de signalisation intracellulaires qui déclenchent la formation du biofilm11. Cette dépendance de la formation du biofilm sur les conditions de croissance est cohérente avec les conditions de nutriments et de croissance présentées par l’environnement de la matrice osseuse de l’hôte qui manque d’oxygène et de nutriments.
Les études sur la pathogenèse des microbes sont souvent liées à la capacité du microbe à migrer vers des sites considérés comme propices à la croissance. Il existe de nombreuses études sur la chimiotaxie microbienne, mais il n’existe pas d’approche standardisée pour l’étudier. De plus, l’utilisation de produits chimiques non diffusibles a été considérée comme un facteur limitant dans l’étude de ce phénomène. Ici, nous avons développé un couvercle en résine produit par impression 3D qui standardise la distance entre le réservoir chimique et le point d’inoculation (figure supplémentaire S1 et figure supplémentaire S2). Nous avons étudié la migration des microbes vers un environnement riche en nutriments, où 5 fois la tuberculose a été utilisée comme stimulant chimique. Les microbes ont été surveillés pendant 48 h dans des conditions aérobies et anaérobies, où les cinq microbes ont montré différents degrés de migration de tropisme (Figure 4A).
ImageJ a été utilisé pour mesurer la distance de migration des bactéries (Figure 4B) ; La migration vers le réservoir semblait être plus rapide que la migration dans la direction opposée. Le rapport entre la distance de migration vers le réservoir et celle dans la direction opposée montre l’expansion rapide de la colonie microbienne due à la chimiotaxie microbienne (Figure 5B). Dans cette étude, des bactéries telles que S. aureus ATCC 25923 ont migré plus rapidement que d’autres microbes tels que C. stationis. Il est intéressant de noter que le profil de migration des cultures chimiotactiques dans des conditions aérobies et anaérobies a montré que certaines bactéries, telles que E. faecalis, présentaient une variation dans les schémas de migration dans des conditions aérobies et anaérobies, où une expansion rapide a été observée dans des conditions anaérobies au fil du temps. De manière confondante, la souche de laboratoire E. faecalis V583 a favorisé un environnement aérobie pour la chimiotaxie. Ainsi, cela suggère que E. faecalis isolé de différentes sources a des besoins de croissance différents adaptés à l’environnement de croissance.

Figure 1 : Schéma du processus d’opération. (A) Les bactéries sont inoculées sur des plaques, puis dans un milieu liquide et cultivées jusqu’à la phase de croissance logarithmique. (B) Détermination de la capacité des bactéries à former des biofilms dans différentes conditions (aérobie/anaérobie). Trois échantillons ont été sélectionnés pour chaque point temporel (6 h) et répétés en parallèle. (C) Le protocole pour l’expérience du tropisme chimique des bactéries dans différentes conditions (aérobie/anaérobie). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Conditions de croissance microbienne. (A) Le profil de croissance des bactéries cultivées dans un milieu liquide à base de thioglycollate (De gauche à droite : I : Fusobacterium nucleatum ATCC 25586, II : Corynebacterium stationis, III : Staphylacoccus cohnii, IV : Enterococcus faecalis, V : Staphylacoccus aureus ATCC 25923, VI : Enterococcus faecalis V583 et VII : Pseudomonas aeruginosa PAO1). (B) Courbe de croissance de micro-organismes isolés de l’hôte dans des conditions aérobies. (C) Courbe de croissance de micro-organismes isolés de l’hôte dans des conditions anaérobies. Veuillez cliquer ici pour voir une version agrandie de cette figure.
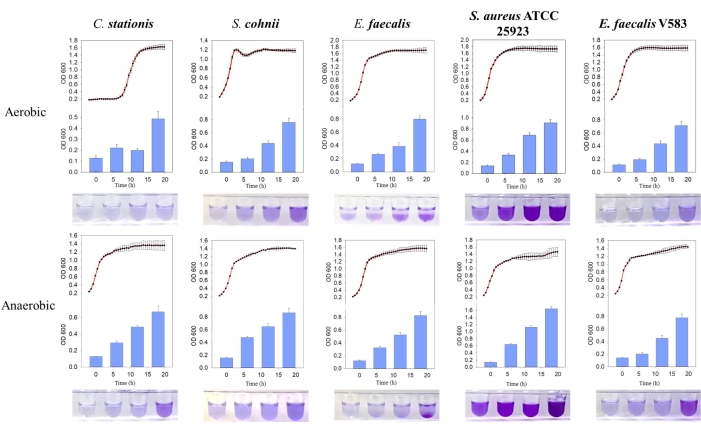
Figure 3 : Comparaison des courbes de croissance et de la formation du biofilm de différentes bactéries dans différentes conditions. De gauche à droite, Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 et Enterococcus faecalis V583. La courbe rouge est la courbe de croissance, et l’histogramme bleu est la quantité de biofilm produite. Veuillez cliquer ici pour voir une version agrandie de cette figure.
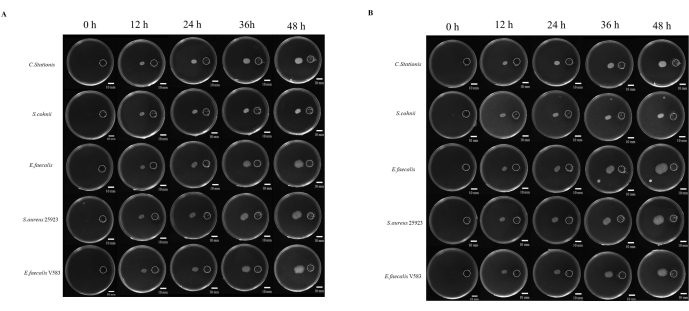
Figure 4 : Croissance microbienne vers l’eutrophisation. (A) Conditions aérobies, (B) Conditions anaérobies. Cinq types de bactéries (de haut en bas : Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 et Enterococcus faecalis V583) ont tendance à se développer jusqu’à l’eutrophisation sur la plaque nutritive différentielle. Barres d’échelle = 10 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Chimiotaxie de microbes dans différentes conditions (aérobie/anaérobie). (A) Les résultats de la migration du tropisme chimique de cinq bactéries sur une plaque contenant de la gélose riche en nutriments ; Le groupe de gauche représente les conditions aérobies et le groupe de droite les conditions anaérobies. Chaque expérience a été répétée en trois exemplaires en parallèle. (B) Analyse des données du tropisme chimique. La partie supérieure représente les conditions aérobies et la partie inférieure les conditions anaérobies. De gauche à droite, Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 et Enterococcus faecalis V583. Les colonnes rouge et bleue sont les distances de migration à partir du corps nutritif des bactéries qui migrent vers le corps nutritif et s’en éloignent. La ligne est la différence entre les deux valeurs (Rouge - Bleu). Barres d’échelle = 10 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Utilisation de couvercles imprimés en 3D pour faire la différence entre les nutriments. (A) Utiliser un logiciel pour préparer des rendus de moules d’impression 3D. (B) Moule à couverture plate imprimé avec 8 000 résine. (C) Utilisez la méthode ci-dessus pour obtenir les plaques de gélose de différence de nutriments. (D) Production de plaques de gélose de différence nutritive. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S2 : Construction de plaques de gélose par différence de nutriments et mesure de la distance de migration bactérienne à l’aide d’ImageJ. (A) La construction des plaques de gélose par différence de nutriments utilise un couvercle imprimé en 3D (contenant une saillie cylindrique) pour recouvrir la plaque. Une fois la gélose refroidie, un puits cylindrique est formé (le cercle rouge), qui est rempli de gélose 5x TB. Celle-ci est ensuite refroidie pour obtenir des plaques de gélose de différence de nutriments. (B) Utilisez ImageJ pour mesurer la distance de migration des bactéries dans différentes directions. Définir la barre d’échelle (Analyser | Définir l’échelle) et exporter la distance de migration à l’aide de l’option Droit (Analyser | mesurer). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S3 : Schémas de croissance de cinq bactéries sur gélose moyenne minimale M9-5x TB. Le groupe de gauche est pour les conditions aérobies et le groupe de droite pour les conditions anaérobies. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S4 : Expériences de contrôle sur les courbes de croissance ATCC 25586 de Fusobacterium nucleatum et la chimiotaxie de bactéries anaérobies. (A) Détermination des courbes de croissance de F. nucleatum ATCC 25586 dans des conditions aérobies et anaérobies. (B) Expériences de chimiotaxie de F. nucleatum ATCC 25586 sur des plaques de gélose demi-dose MH-5x TB dans différentes conditions d’oxygène. Élargissement partiel : Croissance de F. nucleatum ATCC 25586 sur la plaque dans des conditions anaérobies après 48 h (amplification partielle). Veuillez cliquer ici pour télécharger ce fichier.
| Composé | Concentration (g/L) |
| Na2HPO4 | 6.78 |
| KH2PO4 | 3 |
| NaCl | 0.5 |
| NH4Cl | 1 |
| MgSO4.7H 2O | 0.493 |
| CaCl2 | 0.011 |
| Glucose | 4 |
Tableau 1 : Composants et contenu des supports M9 Minimal.
Discussion
Nous avons isolé et identifié cinq espèces de bactéries par des méthodes séquentielles. La croissance des bactéries a des besoins minimaux en nutriments : le milieu minimal - un milieu contenant uniquement des sels inorganiques, une source de carbone et de l’eau. Bien que les bactéries du groupe expérimental aient été trouvées sur des plaques solides MH, nous avons utilisé un milieu MH à demi-dose pour vérifier la chimiotaxie des bactéries et avons obtenu de bons résultats. Cependant, nous avons également effectué des expériences de contrôle en utilisant un minimum de milieu. Le milieu de base M9 a été utilisé dans l’expérience (voir le tableau 1 pour sa composition et son contenu), et la gélose 5x TB a été utilisée pour le bloc de gélose nutritive. Les résultats expérimentaux sont présentés dans la figure supplémentaire S3. Bien que ces bactéries (à l’exception d’E. faecalis) se développent lentement et se déplacent lentement dans le milieu M9, il existe une tendance généralement similaire (vers la croissance sur gélose nutritive), comme observé dans ces expériences. Cependant, l’E. faecalis V583 n’a pas poussé dans le milieu minimal M9, peut-être en raison du manque de composants nécessaires à la croissance de l’E. faecalis dans le milieu M9. Par conséquent, nous recommandons d’utiliser un milieu MH à demi-dose comme substrat déficient en nutriments pour simuler la croissance et la migration bactériennes dans des conditions déficientes en nutriments. Cela facilite également l’utilisation du logiciel ImageJ pour mesurer la distance sur laquelle les bactéries ont tendance à migrer.
Les biofilms sont généralement définis comme des communautés microbiennes associées à la surface et constituent une stratégie de survie microbienne importante. Des organismes tels que Pseudomonas aeruginosa, Haemophilus influenzae, Streptococcus pneumoniae et Staphylococcus aureus peuvent être trouvés chez des hôtes asymptomatiques, et la découverte de micro-organismes adhérents in vivo est souvent appelée colonisation et non infection. Un problème très important est l’absence d’un modèle idéal de colonisation animale12. Les microbes présentent des attaches initiales aux surfaces chargées avant la formation du biofilm, où des matériaux tels que le verre et le polychlorure de vinyle (PVC) sont jugés appropriés pour les études liées au biofilm. Des études utilisant des plaques de PVC à 96 puits ont été utilisées pour étudier les biofilms microbiens9, dans lesquels les biofilms monospécifiques ou multispécifiques peuvent donner des biofilms faiblement ou densément emballés10. En tant que telle, l’utilisation de la plaque de PVC à 96 puits est considérée comme mal adaptée aux biofilms mal emballés, car la matrice du biofilm peut facilement être lavée pendant le processus de coloration du biofilm. Par conséquent, nous nous sommes appuyés sur des tubes à essai en verre pour étudier la formation de biofilms microbiens (Figure 3).
L’une des principales limites de cette méthode est qu’elle ne fonctionne pas avec les micro-organismes qui se développent dans des conditions spécifiques, et que toutes les bactéries ne peuvent pas migrer. De plus, bien que les biofilms soient un moyen efficace d’aider les micro-organismes à se défendre contre les environnements extérieurs défavorables, tous les micro-organismes hôtes ne sont pas capables de produire des biofilms. Par exemple, la souche ATCC 25586 de F. nucleatum , mentionnée précédemment, qui est associée à une symbiose orale ou intestinale humaine associée au développement du cancer du côlon17, ne se développe spécifiquement que dans des environnements anaérobies mais ne produit pas de biofilms. Nous l’avons utilisé dans des expériences FTM pour vérifier l’efficacité du média FT. Le milieu FT s’est avéré efficace pour fournir aux bactéries des concentrations dégradées d’oxygène.
À titre de comparaison, nous avons testé la courbe de croissance et le tropisme chimique de la bactérie anaérobie F. nucleatum ATCC 25586 (figure supplémentaire S4). Dans les expériences sur la courbe de croissance (figure supplémentaire S4A), la souche ATCC 25586 de F. nucleatum ne pouvait pas croître dans des conditions aérobies et a montré une agrégation bactérienne au fond des puits dans des conditions anaérobies. De même, dans les expériences de chimiotaxie (figure supplémentaire S4B), F. nucleatum ATCC 25586 ne s’est pas développé dans des conditions aérobies, et un faible nombre de bactéries a été trouvé après l’inoculation in situ dans des conditions anaérobies pendant 48 heures. De même, comme certaines bactéries ne peuvent pas rechercher activement des conditions de croissance appropriées en migrant, elles attendent des conditions de croissance appropriées en entrant dans un état de dormance à long terme18. Il convient de noter que la culture de bactéries anaérobies strictes nécessite un milieu spécial, tel que le milieu de fermentation ET (les plaques semi-solides doivent être complétées par 0,5 % d’agar), qui est également différent de l’isolement et de l’identification conventionnels. La méthode proposée est une méthode générale applicable à la caractérisation de la plupart des microorganismes hôtes, la technique ayant été démontrée pour cinq microbes, dont trois microbes isolés d’os murins et deux microbes de laboratoire. Ce protocole permettrait d’identifier correctement les isolats de microbiote dans les études futures.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Le développement de cette technique a été soutenu par les fonds du Fonds de recherche pour les jeunes scientifiques internationaux de la Fondation nationale des sciences naturelles de Chine (22050410270), du Fonds spécial de Shenzhen pour l’innovation et l’entrepreneuriat des talents de haut niveau à l’étranger (KQTD20170810111314625) et du programme d’équipe de recherche innovante et entrepreneuriale du Guangdong (2019ZT08Y191). Nous tenons à exprimer notre sincère gratitude à Mlle Chen Xinyi pour son aide dans la relecture du document et la gestion du laboratoire.
matériels
| Name | Company | Catalog Number | Comments |
| Chemical/Solution | |||
| 1% crystal violet dye solution | Solarbio | G1062 | 100 mL |
| Agar | Sigma-Aldrich | V900500 | Used to obtain semi-solid plates, 20 g |
| Centrifuge tube | Corning | 430790 | 15 mL |
| Fluid thioglycollate medium | Kinghunt | K0001 | 29.3 g |
| Mueller Hinton II Broth medium | Solarbio | NO.11865 | 100 g |
| Petri dishes | Bkman | B-SLPYM90-15 | Plastic Petri dishes, circular, 90 mm x 15 mm |
| Potassium Chloride | VETEC | WXBC4493V | 0.2 g |
| Potassium Dihydrogen Phosphate | aladdin | 04-11-7758 | 0.24 g |
| Sodium chloride | Macklin | S805275 | 8.0 g |
| Sodium phosphate dibasic | aladdin | 7558-79-4 | 1.44 g |
| Terrific Broth medium | Solarbio | LA2520 | 200 g |
| Kits/ Equipment | |||
| Anaerobic incubator | Longyue | ||
| Biochemical incubator | Blue pard | LRH-70 | |
| Microplate reader | Spark | ||
| Tanon 5200multi imaging system | Tanon | 5200CE | |
| Thermostatic water bath | Jinghong | DK-S28 |
Références
- Bernardi, S., et al. Combining culture and culture-independent methods reveals new microbial composition of halitosis patients' tongue biofilm. MicrobiologyOpen. 9 (2), 958 (2020).
- Tellez, G., Latorre, J. D., Kuttappan, V. A., et al. Rye affects bacterial translocation, intestinal viscosity, microbiota composition and bone mineralization in Turkey poults. PLOS One. 10, (2015).
- Brewer, J. H. Clear liquid mediums for the "aerobic" cultivation of anaerobes. Journal of the American Medical Association. 115 (8), 598 (1940).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Liu, C., et al. The regulation of bacterial biofilm formation by cAMP-CRP: a mini-review. Frontiers in Microbiology. 11, 802 (2020).
- Kumar, A., et al. Biofilms: survival and defense strategy for pathogens. International Journal of Medical Microbiology. , (2017).
- Penesyan, A., et al. Secondary effects of antibiotics on microbial biofilms. Frontiers in Microbiology. 11, 2109 (2020).
- Bernardi, S., et al. Subinhibitory antibiotic concentrations enhance biofilm formation of clinical Enterococcus faecalis isolates. Antibiotics. 10 (7), 874 (2021).
- Tsukatani, T., Fumihiko, S., Rieko, K. A rapid and simple measurement method for biofilm formation inhibitory activity using 96-pin microtiter plate lids. World Journal of Microbiology and Biotechnology. 36 (12), 1-9 (2020).
- Hartmann, R., et al. Emergence of three-dimensional order and structure in growing biofilms. Nature Physics. 15, 251-256 (2019).
- Liu, Z., et al. Advances in bacterial biofilm management for maintaining microbiome homeostasis. Biotechnology Journal. 15 (10), 1900320 (2020).
- Hall-Stoodley, L., Stoodley, P. Evolving concepts in biofilm infections. Cellular Microbiology. 11 (7), 1034-1043 (2009).
- Røder, H. L., et al. Enhanced bacterial mutualism through an evolved biofilm phenotype. The ISME Journal. 12 (11), 2608-2618 (2018).
- Matilla, M. A., Tino, K. The effect of bacterial chemotaxis on host infection and pathogenicity. FEMS Microbiology Reviews. 42 (1), 052 (2018).
- Moreira, L. M., et al. Chemotactic signal transduction and phosphate metabolism as adaptive strategies during citrus canker induction by Xanthomonas citri. Functional & Integrative Genomics. 15 (2), 197-210 (2015).
- Konishi, H., et al. Bacterial chemotaxis towards polysaccharide pectin by pectin-binding protein. Scientific Reports. 10 (1), 1-12 (2020).
- Kostic, A. D., et al. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment. Cell Host & Microbe. 14 (2), 207-215 (2013).
- Kochan, T. J., et al. Updates to Clostridium difficile spore germination. Journal of Bacteriology. 200 (16), 00218 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon