Method Article
Messung der Migration und Biofilmbildung verschiedener Bakterien
In diesem Artikel
Zusammenfassung
Hier stellen wir eine praktische Methode zur Isolierung und Identifizierung von Mikroorganismen im Wirt vor. Auf diese Weise werden die physikalisch-chemischen Eigenschaften von Mikroorganismen und mögliche Lebensweisen im Wirt anschaulich beschrieben.
Zusammenfassung
Da Mikroben, die im Wirtskörper gedeihen, in erster Linie über Anpassungsfähigkeiten verfügen, die ihr Überleben erleichtern, wären Methoden zur Klassifizierung und Identifizierung ihrer Natur von Vorteil, um ihre Charakterisierung zu erleichtern. Derzeit konzentrieren sich die meisten Studien nur auf eine bestimmte Charakterisierungsmethode; Die Isolierung und Identifizierung von Mikroorganismen aus dem Wirt ist jedoch ein kontinuierlicher Prozess und erfordert in der Regel mehrere kombinatorische Charakterisierungsmethoden. Darin beschreiben wir Methoden zur Identifizierung der mikrobiellen Biofilmbildungsfähigkeit, des mikrobiellen Atmungszustands und ihres chemotaxis Verhaltens. Die Methoden werden zur Identifizierung von fünf Mikroben verwendet, von denen drei aus dem Knochengewebe von Sprague-Dawley (SD)-Ratten (Corynebacterium stationis, Staphylococcus cohnii subsp. urealyticus und Enterococcus faecalis) und zwei aus der American Type Culture Collection (ATCC)-Staphylococcus aureus ATCC 25923 und Enterococcus faecalis isoliert wurden V583. Zu den Mikroben, die aus dem Knochengewebe der SD-Ratte isoliert wurden, gehören die grampositiven Mikroben. Diese Mikroben haben sich so angepasst, dass sie unter stressigen und nährstoffbegrenzenden Umgebungen innerhalb der Knochenmatrix gedeihen. Dieser Artikel zielt darauf ab, den Lesern das spezifische Know-how zur Bestimmung der Art und des Verhaltens der isolierten Mikroben in einer Laborumgebung zu vermitteln.
Einleitung
Der Säugetierwirt, der durch den menschlichen Körper repräsentiert wird, enthält eine große Anzahl von Mikroorganismen. Diese Mikroorganismen sind im Mund, im Verdauungstrakt, im Darm und im Blut des Wirts weit verbreitet und haben unterschiedliche Auswirkungen auf die Gesundheit des Wirts. Die Mundhöhle ist Wirt einer Vielzahl von Mikroben, die die Anfälligkeit des Wirts für Infektionen modulieren können. Mikroben wie Streptokokken (z . B. S. mitis/oralis, S. pseudopneumoniae und S. infantis) und Prevotella spp. besiedeln die Mundhöhle und bilden einen Multispezies-Biofilm auf der Zungenoberfläche, der Mundgeruch verursacht und als mikrobielles Reservoir für mikrobielle Infektionen fungiert. Diese Erreger können den Kieferknochen infizieren, indem sie die parodontalen Bänder infiltrieren, die die Zahnwurzel im Kieferknochen halten1. Die Charakterisierung dieser aus dem Wirtskörper isolierten Mikroben ist oft ein langwieriger Prozess, insbesondere wenn die Mikroben individuelle Merkmale aufweisen, die eine spezifische Behandlung und Wachstumsbedingungen erfordern. Mikroben wie die pathogenen Helicobacter pylori, Clostridium difficile und Fusobacterium nucleatum gedeihen in der rauen Umgebung des Darms mit spezifischen Sauerstoff-, Nährstoff- und Wachstumsanforderungen, was eine Herausforderung bei den Charakterisierungsprozessen darstellt, insbesondere bei der Untersuchung der Pathogenität dieser Mikroben. Daher ist eine standardisierte Methode zur Isolierung und Untersuchung dieser Mikroorganismen erforderlich, damit Wissenschaftler und Mediziner neue medizinische Behandlungen entwickeln können.
Dieses Protokoll verwendet Mikroben, die in der Knochenmatrix von Ratten gedeihen. Traditionell wird mit Ausnahme des knöchernen Systems, das durch den Kiefer repräsentiert wird und bei dem das Vorhandensein von Zähnen die Knochenmatrix anfälliger für Infektionen macht als andere Knochen1, allgemein angenommen, dass der gesunde Knochen des Wirts eine sterile Umgebung ist. Studien haben jedoch gezeigt, dass Mikroorganismen durch die Darmwand in den systemischen Kreislauf gelangen und letztendlich die Knochenmineralisierung beeinträchtigen2. Als Proof-of-Concept verwenden wir das beschriebene Protokoll, um die biochemischen Eigenschaften von mikrobiellen Isolaten aus dem Femur und der Tibia gesunder SD-Ratten (Corynebacterium stationis, Staphylococcus cohnii subsp. und Enterococcus faecalis) zu charakterisieren. Diese mikrobiellen Isolate wurden ausgewählt, da der Knochen eine geschlossene und hypoxische Umgebung ist und die Charakterisierung dieser Mikroben aus dem Knochen eine schwierige Aufgabe sein kann. In verschiedenen Artikeln wurden die Prozesse, die bei der Untersuchung dieser Mikroben verwendet werden, detailliert beschrieben. Es gibt jedoch nur wenige, die ein vollständiges Protokoll zur Identifizierung wirtsisolierter Mikroorganismen bieten.
Bei der Festlegung der richtigen Kulturbedingungen muss der Sauerstoffbedarf der Mikrobe durch die Verwendung von flüssigem Thioglycollatmedium (FTM) verstanden werden. Mikroben mit unterschiedlichem Sauerstoffbedarf bilden in der klaren FTM-Flüssigkeit3 geschichtete Schichten. Anhand des Schichtungsprofils wird dann der Sauerstoffbedarf der Mikrobe genutzt, um das Wachstum der Zellen zu untersuchen. Mikroben, die auf der Oberfläche der FTM-Flüssigkeit gedeihen, sind obligate Aerobier, während Mikroben, die am Boden wachsen, obligate Anaerobier sind. Mikroben, die als Suspension in der FTM-Flüssigkeit wachsen, sind entweder fakultative oder aerotolerante Anaerobier. Die mikrobielle Wachstumsrate wird durch die Beobachtung der exponentiellen Wachstumsphase der mikrobiellen Zellen ermittelt. Das Wachstumsprofil wird dann mit der Biofilmbildung der Mikrobe verglichen. Biofilme bestehen größtenteils aus mehreren Arten, die sich direkt und indirekt gegenseitig auf die Gesundheit beeinflussen. Während dieses Prozesses selektieren vorteilhafte Interaktionen zwischen mikrobiellen Gemeinschaften für die Bindung, wodurch eine räumliche Struktur entsteht, die die Evolution aktiver wechselseitiger Interaktionen begünstigt. Zum Beispiel zeigen die Co-Kulturen von Paenibacillus amylolyticus und Xanthomonas retroflexus fakultativ symbiotische Interaktionen in einer statischen Umgebung, die ein schnelles Biofilmwachstum fördern13. Mikroben passen sich über die Bildung von Biofilmen an das Wirtsgewebe an, um eine nachhaltige Lokalisierung zu gewährleisten, sich vor rauen Umgebungen zu schützen und dem Immunsystem des Wirts zu entgehen 4,5,6,7. Biofilme sind in der Regel dichte und mehrschichtige Strukturen, die Mikroorganismen helfen, externen unterkritischen Reizen zu widerstehen. zum Beispiel erhöht E. faecalis in der Zahnpulpa seine Resistenz gegen Antibiotika, indem es die Biofilmbildung erhöht, wenn es mit Unterkonzentrationen von Tetracyclin und Vancomycinkonfrontiert wird 8.
Chemotaxis ermöglicht es Mikroorganismen, sich entsprechend chemischer Gradienten zu bewegen, und Signalwege sind in einer Vielzahl von pathogenen Bakterien weit verbreitet. Einige pathogene Mikroorganismen wandern unter Anleitung chemischer Signale an bestimmte Stellen, um Infektionskrankheiten zu verursachen14. Zum Beispiel Xanthomonas spp. exprimieren 19 Chemorezeptor- und 11 Flagellin-Proteine im Wirt, was die bakterielle Bindung und schließlich die Ulzeration auslöst15. Es gibt auch spezifische Proteine in den Bakterien (Pektin-bindende Proteine), die die spezifische Migration von Bakterien zu Nährstoffen steuern, was zu einem besseren Wachstum führen kann16. Dies ist auch für Bakterien von entscheidender Bedeutung, die in nährstoffarmen Umgebungen vorkommen können. Mikrobielle Zellen verlassen sich oft auf die chemotaktische Motilität, um sich in eine förderliche Wachstumsumgebung zu ziehen und gleichzeitig anderen räuberischen Zellen und Toxinen auszuweichen, die die Lebensfähigkeit der Zellen beeinträchtigen. Aufbauend auf bereits etablierten Soft-Agar-Ansätzen zur Bestimmung der Chemotaxis entwickeln wir eine diffusionsfähige Methode zur Erzeugung eines Chemoattraktstoffgradienten zur Überprüfung der mikrobiellen Chemotaxis.
In dieser Arbeit werden die Methoden zur Bestimmung der Wachstumsbedingungen, der Biofilmbildung und des chemischen Tropismus von Bakterien am Beispiel von Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 und Enterococcus faecalis V583 beschrieben (siehe Abbildung 1). Bei der Optimierung der mikrobiellen Wachstumsbedingungen wird FTM verwendet, um den Sauerstoffbedarf der Mikrobe zu bestimmen, während bei der Biofilmbildung Glasoberflächen als feste Unterlage verwendet werden und die Biofilmmasse mit Kristallviolett gegengefärbt wird. Der chemische Tropismus der Mikrobe beruht auf ihrem chemotaktischen Verhalten, bei dem durch 3D-Druck (Ergänzende Abbildung S1) eine standardisierte Methode verwendet wird, um ein chemisches Reservoir für den Chemolockstoff in einer weichen Agar-Matrix zu erzeugen (Ergänzende Abbildung S2).
Protokoll
HINWEIS: In der Materialtabelle finden Sie Einzelheiten zu allen Materialien und Geräten, die in diesem Protokoll verwendet werden. Verwenden Sie aseptische Techniken, um eine Kontamination zu vermeiden.
1. Bakterielle Erholung, um eine einzige Kolonie zu erhalten
- Bereiten Sie feste Agarplatten vor.
- Bereiten Sie Terrific Broth (TB)-Medium mit Agar zu, wobei jeder Liter Flüssigkeit 11,8 g Trypton, 23,6 g Hefeextrakt, 9,4 g K2HPO4, 2,2 g KH2PO4 und 15 g Agar enthält, pH = 7.
- Autoklavieren Sie die Lösung 20 Minuten lang bei 121 °C mit einer luftdurchlässigen Kappe oder Flaschenversiegelungsfolie mit Entlüftungsöffnung.
- Bevor die Flüssigkeit abkühlt und erstarrt, gießen Sie die agarhaltige TB-Brühe in 10 cm große Petrischalen aus Kunststoff mit einem Volumen von 25 mL pro Platte.
HINWEIS: Die Brühe sollte in die Platte gegossen werden, bevor sie bei Temperaturen unter 60 °C fest wird. Verwenden Sie aseptische Techniken, um eine Kontamination zu verhindern.
- Bakterielle Inokulation auf die Platte
- Nehmen Sie die bei -80 °C gelagerten Zielstämme heraus und tauen Sie sie bei Raumtemperatur in einer biologischen Sicherheitswerkbank auf.
- Nehmen Sie mit einer 10-μl-Pipette 10 μl Bakteriensuspension auf und verteilen Sie sie durch Streifen auf der TB-Platte.
- Versiegeln Sie die Platte, um eine Kontamination zu vermeiden. Stellen Sie die Platte in einen biochemischen Inkubator bei 37 °C. Überprüfen Sie das Koloniewachstum visuell nach ~24 h. Wählen Sie einzelne Kolonien aus und amplifizieren Sie sie durch PCR, um zu bestätigen, dass sie nur eine Art von Bakterien enthalten.
2. Bakterielle Flüssigkultur bis zur logarithmischen Wachstumsphase
- Flüssiges Kulturmedium vorbereiten.
- Siehe die Herstellung von festen Agarplatten in Schritt 1.1.1. zur Herstellung von TB-Flüssigmedium, von dem 1 l 11,8 g Trypton, 23,6 g Hefeextrakt, 9,4 g K2HPO4 und 2,2 g KH2PO4 enthält, pH ~7.
HINWEIS: Fügen Sie kein Agar hinzu. Bakterien wachsen schneller in einer Umgebung mit flüssigen Nährstoffen. - Autoklavieren Sie die Lösung 20 Minuten lang bei 121 °C mit einer luftdurchlässigen Kappe oder Flaschenversiegelungsfolie mit Entlüftungsöffnung.
- Lagern Sie das Flüssigkulturmedium bei 4 °C.
- Siehe die Herstellung von festen Agarplatten in Schritt 1.1.1. zur Herstellung von TB-Flüssigmedium, von dem 1 l 11,8 g Trypton, 23,6 g Hefeextrakt, 9,4 g K2HPO4 und 2,2 g KH2PO4 enthält, pH ~7.
- Bakterielle Inokulation und Kultivierung
- In der biologischen Sicherheitswerkbank wird mit einer Impfschlaufe oder einer 10 μL-Pipette eine einzelne Kolonie entnommen und die Bakterien in dem vorbereiteten TB-Flüssigmedium in einem Erlenmeyerkolben inokuliert.
- Verschließen Sie den Erlenmeyerkolben mit Gaze und stellen Sie ihn in einen Schüttler (200 U/min) bei 37 °C, um die Bakterien zu kultivieren.
HINWEIS: Das flüssige Medium trübt sich im Laufe von 5 h allmählich; Die Zeit, die benötigt wird, um trüb zu werden, variiert je nach Bakterium.
3. FTM-Experiment und Bestimmung der Wachstumskurve
- Zubereitung von flüssigem Thioglycollat-Medium (FTM)
- Bereiten Sie Thiolglycollat-Medium (TM) vor, dessen jeder Liter 15 g Trypton, 5 g Hefeextrakt, 5 g Glukose, 0,5 g L-Cystein, 0,5 g Natriumthioglykolat, 2,5 g Natriumchlorid, 0,001 g Resazurin und 0,75 g Agar enthält.
HINWEIS: Handelsübliche feste Medien können leicht mit Wasser rekonstituiert und sterilisiert werden (Schritt 3.1.2.). - Wiegen Sie 29,3 g festes Thioglykollat-Medium, fügen Sie 1 l destilliertes Wasser hinzu, erhitzen Sie es und rühren Sie, bis es sich vollständig aufgelöst hat und die Lösung rosa wird.
- Geben Sie TM (19 mL) in ein Reagenzglas mit einem Verschlussstopfen. Nach dem Versiegeln die Lösung 20 Minuten lang bei 121 °C mit einem luftdurchlässigen Verschluss oder einer Flaschenversiegelungsfolie mit Entlüftungsöffnung autoklavieren.
HINWEIS: Nach dem Erhitzen wird die Lösung hellgelb.
- Bereiten Sie Thiolglycollat-Medium (TM) vor, dessen jeder Liter 15 g Trypton, 5 g Hefeextrakt, 5 g Glukose, 0,5 g L-Cystein, 0,5 g Natriumthioglykolat, 2,5 g Natriumchlorid, 0,001 g Resazurin und 0,75 g Agar enthält.
- Inokulation, Kultivierung und Beobachtung von Bakterien
- Bereiten Sie die Bakteriensuspension so vor, dass sie in flüssigem TB-Medium auf OD 0,6-0,8 heranwächst (siehe Schritt 2.2.).
- Öffnen Sie den Reagenzglas-Verschlussstopfen unter aseptischen Bedingungen in der biologischen Sicherheitswerkbank, geben Sie so bald wie möglich 1 mL der Bakterienkultur in das Reagenzglas, das FTM enthält, und verschließen Sie das Reagenzglas sofort wieder.
- Schütteln Sie das Röhrchen vorsichtig, um sicherzustellen, dass die Bakterien gleichmäßig in der Flüssigkeit verteilt sind, und stellen Sie das Reagenzglas für die statische Kultur auf 37 °C auf.
- Beobachten Sie nach 48 Stunden Inkubation das Wachstum verschiedener Bakterien in FTM in verschiedenen Reagenzgläsern und machen Sie Fotos.
- Bestimmung der Wachstumskurve unter aeroben Bedingungen
- Bereiten Sie die erste Bakterienkultur mit einem geeigneten OD-Wert vor (siehe Schritt 2.2.) und kultivieren Sie die Bakterien, bis sie die logarithmische Wachstumsphase erreichen.
HINWEIS: Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 und Enterococcus faecalis V583, die in diesem Protokoll verwendet wurden, brauchten ~5 Stunden, um die logarithmische Phase zu erreichen. - 200 μl der Bakterienkultur (aus Schritt 3.3.1) in TB-Flüssigmedium in jede Vertiefung einer 96-Well-Platte in dreifacher Ausfertigung geben. Verwenden Sie einen Mikroplatten-Reader, um den OD-Wert bei 600 nm zu bestimmen, und verdünnen Sie die verschiedenen Bakterienkulturen so, dass die OD-Differenz zwischen den verschiedenen bakterienhaltigen Wells und dem TB-Flüssigmedium (Blank) zwischen 0,01 und 0,02 liegt.
- Nehmen Sie eine neue 96-Well-Platte, fügen Sie TB-Medium hinzu, übertragen Sie die verschiedenen verdünnten Bakterienkulturen aus Schritt 3.3.2. in dreifacher Ausfertigung auf die Platte und stellen Sie sie auf eine ultrasaubere Werkbank.
- Versiegeln Sie die 96-Well-Platte mit Dichtungsband, legen Sie sie in den Mikroplatten-Reader und stellen Sie das Programm wie folgt ein:
- Stellen Sie die Temperatur auf 37 °C ± 0,5 ein.
- Stellen Sie den Kinetic Loop auf insgesamt 60 Zyklen ein, jeweils für 30 Minuten.
- Stellen Sie die Extinktion auf 600 nm ein.
- Stellen Sie die Erschütterung auf 1.600 s in einem Zyklus ein.
- Führen Sie das Programm aus und stoppen Sie es, wenn die Wachstumskurve ein Plateau erreicht.
- Bereiten Sie die erste Bakterienkultur mit einem geeigneten OD-Wert vor (siehe Schritt 2.2.) und kultivieren Sie die Bakterien, bis sie die logarithmische Wachstumsphase erreichen.
- Bestimmung der Wachstumskurve unter anaeroben Bedingungen
- Folgen Sie Schritt 3.3. Bewahren Sie jedoch die 96-Well-Platte mit dem TB-Medium und den Bakteriensuspensionen in einem anaeroben Inkubator auf, bevor Sie die Platte mit Dichtungsband verschließen. Pumpen und füllen Sie die anaerobe Box mit Stickstoff, um die Sauerstoffkonzentration auf <3% zu reduzieren. Verschließen Sie die 96-Well-Platte mit Dichtband und lassen Sie sie in der anaeroben Box.
4. Test der Fähigkeit zur Biofilmbildung
- Kultivieren Sie die Bakterien in einer logarithmischen Wachstumsphase in flüssigem Medium.
- Siehe Abschnitt 2 zur Inokulation der Bakterien im TB-Flüssigmedium.
- Kultivieren Sie die Bakterien über Nacht in einem Schüttler bei 37 °C und stellen Sie sicher, dass der OD600 des Bakterienkulturmediums zwischen 0,6 und 0,8 liegt.
- Teilen Sie die Bakterienkultur und kultivieren Sie sie unter verschiedenen Bedingungen.
- Wenn der OD600 der Bakterienkultur zwischen 0,6 und 0,8 liegt, geben Sie 10 ml dieser Bakteriensuspension in ein Reagenzglas aus Glas.
- Unterteilen Sie jede Bakterienkultur in zwei Gruppen: aerobe und anaerobe Bedingungen, und führen Sie alle Vorgänge in dreifacher Ausführung durch.
- Für die aerobe Gruppe wickeln Sie das Reagenzglas mit der Bakteriensuspension in Alufolie ein und inkubieren es in einem 37 °C warmen Wasserbad bei konstanter Temperatur.
- Für die anaerobe Gruppe wickeln Sie das Röhrchen mit der Bakteriensuspension mit Alufolie ein und überführen es in einen anaeroben Inkubator. Entfernen Sie die Alufolie im anaeroben Inkubator und reduzieren Sie die Sauerstoffkonzentration im Inkubator auf <3%, indem Sie ihn pumpen und mit Stickstoff befüllen. Verschließen Sie das Reagenzglas mit einem Milchglasstopfen, nehmen Sie es nach dem Versiegeln heraus und inkubieren Sie es in einem Wasserbad bei 37 °C.
- Alle 6 Stunden drei Reagenzgläser aus jeder der beiden Gruppen (aerob/anaerob) entnehmen, um das Wachstum des Biofilms am Boden zu beobachten, und gemäß Schritt 4.3 färben.
- Entsorgen Sie die Bakteriensuspension und trocknen Sie den Biofilm.
- Nehmen Sie die Reagenzgläser zu unterschiedlichen Zeitpunkten heraus und aspirieren Sie vorsichtig die obere Schicht der Bakteriensuspension. Beobachten Sie, wie der weiße Biofilm am Boden des Reagenzglases haftet.
HINWEIS: Achten Sie darauf, den Biofilm beim Ansaugen der Bakteriensuspension nicht zu stören. - Geben Sie vorsichtig 2 ml phosphatgepufferte Kochsalzlösung (PBS) entlang der Wand des Reagenzglases, schütteln Sie es vorsichtig, um die im Biofilm adsorbierten Planktonbakterien wegzuspülen, und aspirieren Sie den PBS-Puffer vorsichtig.
- Legen Sie das Reagenzglas mit dem am Boden adsorbierten Biofilm in den Ofen, um es 30 Minuten lang zu trocknen.
- Nehmen Sie die Reagenzgläser zu unterschiedlichen Zeitpunkten heraus und aspirieren Sie vorsichtig die obere Schicht der Bakteriensuspension. Beobachten Sie, wie der weiße Biofilm am Boden des Reagenzglases haftet.
- Färbung
- Nehmen Sie die Reagenzgläser heraus, geben Sie 2 ml 0,1%ige Kristallviolett-Färbelösung in jedes Reagenzglas und färben Sie die Biofilme 30 Minuten lang bei Raumtemperatur.
- Verwenden Sie eine Pipette, um die kristallviolette Farbstofflösung vorsichtig zu aspirieren. Fügen Sie vorsichtig destilliertes Wasser entlang der Wand des Reagenzglases hinzu, um die restliche kristallviolette Farbstofflösung abzuwaschen, und wiederholen Sie dies 3x-5x, bis das hinzugefügte destillierte Wasser fast farblos wurde. Beobachte den violetten Biofilm am Boden des Reagenzglases.
- Trocknen Sie das Reagenzglas erneut.
- Lösen Sie den Biofilm mit 95% Ethanol auf und messen Sie den OD600 der Biofilmlösung.
- Nehmen Sie das getrocknete Reagenzglas heraus, geben Sie 10 ml 95%iges Ethanol in jedes Reagenzglas und schütteln Sie es gründlich, um sicherzustellen, dass das Kristallviolett vollständig im Ethanol aufgelöst wurde.
- Bei einem Volumen von 200 μl pro Vertiefung wird Kristallviolett in Ethanol auf eine 96-Well-Platte gegeben und seine Absorption bei einer Wellenlänge von 600 nm gemessen.
HINWEIS: Nach dem Beer-Lambertschen Gesetz ist die Absorption linear mit der Konzentration der Lösung verbunden, so dass der Wert von OD600 die Konzentration von Kristallviolett widerspiegeln kann, die die Menge an Biofilm in der Bakteriensuspension zu verschiedenen Zeiten widerspiegelt.
5. Bakterieller Chemotaxis-Test
- Bereiten Sie differenzielle eutrophe Agarplatten vor.
- Beziehen Sie sich auf Schritt 1, um ein MH-Medium mit halber Dosis herzustellen, das 0,5 % Agar enthält, von dem jeder Liter 11,8 g Trypton, 23,6 g Hefeextrakt, 9,4 g K2HPO4, 2,2 g KH2PO4 und 5 g Agar enthält, pH ~7.
- In Schritt 1 finden Sie Informationen zur Herstellung von 5x TB-Medium mit 0,5 % Agar, von dem jeder Liter 59 g Trypton, 118 g Hefeextrakt, 47 g K2HPO4, 11 g KH2PO4 und 5 g Agar mit einem pH-Wert von ~ 7 enthält.
- Autoklavieren Sie die Lösung 20 Minuten lang bei 121 °C mit einer luftdurchlässigen Kappe oder Flaschenversiegelungsfolie mit Entlüftungsöffnung.
- Bevor die Flüssigkeit abgekühlt und verfestigt ist, gießen Sie 25 ml halbe Dosis MH-Bouillon (mit 0,5 % Agar) pro Platte, decken Sie die Platte mit einem 3D-gedruckten Deckel ab und stellen Sie sie in eine biologische Sicherheitswerkstatt. Entfernen Sie nach dem Abkühlen die Abdeckung, um die zylindrische Vertiefung (1,4 cm Durchmesser) im halbfesten Medium zu beobachten.
HINWEIS: Der 3D-gedruckte Deckel mit Harzmaterial kann nicht mit Hochtemperaturdampf sterilisiert werden. Um die Oberflächenbakterien abzutöten, kann es mit 95%igem Ethanol besprüht und anschließend mit einer UV-Lampe für 30 min in einer biologischen Sicherheitswerkbank bestrahlt werden. Wenn Sie den Deckel abnehmen, üben Sie Kraft in vertikaler Richtung aus und heben Sie ihn vorsichtig an, um eine Beschädigung des halbfesten Agars zu vermeiden. - Bevor die 5x TB Bouillon (mit 0,5% Agar) erstarrt, geben Sie mit einer Pipette die 5x TB Bouillon in die Vertiefung in der halbfesten Platte, bis die Brühe bündig mit der Oberfläche des Mediums abschließt.
- Kühlen Sie die Platte in einer biologischen Sicherheitswerkbank ab, um eine halbfeste Agarplatte mit eutrophen Körpern zu erhalten. Achten Sie darauf, eine Kontamination der Platte durch Mikroorganismen aus der Umwelt zu vermeiden.
HINWEIS: Das halbfeste Medium ist noch etwas flüssig und muss daher vollständig abgekühlt werden, bevor es auf dem Kopf stehend gelagert wird.
- Bakterielle Impfung, Kultivierung und Fotografie
- Legen Sie die Agarplatte mit dem halbfesten Nährmedium in eine biologische Sicherheitswerkbank und kühlen Sie sie für ~30 min unter UV-Bestrahlung ab, um eine bakterielle Kontamination zu verhindern.
- Verwenden Sie eine 10-μl-Pipette, um 4 μl der Bakteriensuspension aufzunehmen, die mit einem Außendurchmesser von ~0,6-0,8 kultiviert wurde, führen Sie die Pipettenspitze in die Mitte der Kunststoff-Petrischalen ein und pipettieren Sie die Bakteriensuspension heraus.
- Warten Sie, bis die Bakteriensuspension 1 Minute lang in das Medium aufgenommen wurde. Wenn die Bakteriensuspension vollständig absorbiert wurde, teilen Sie die Platte in dreifacher Ausfertigung in zwei Gruppen (aerob/anaerob).
- Versiegeln Sie die aerobe Gruppe mit Dichtungsband und legen Sie diese Platten kopfüber in einen biochemischen Inkubator bei 37 °C für die statische Kultur. Die anaerobe Gruppe wird wie in Schritt 4.2 beschrieben in einer anaeroben Box verschlossen und die Platten zum Wachstum bei 37 °C umgedreht.
- Überprüfen Sie die Ausbreitung und Migration von Bakterien alle 12 Stunden (bei 0 Stunden, 12 Stunden, 24 Stunden, 36 Stunden und 48 Stunden) und verwenden Sie das Bildgebungssystem, um Bilder der Platte aufzunehmen.
- Verwenden Sie die ImageJ-Software, um die Migrationsabstände von Bakterien zu berechnen.
- Importieren Sie die Bilddatei, die mit dem Imaging-System erfasst wurde.
- Legen Sie die Maßstabsleiste fest und wenden Sie sie auf alle Bilder an.
- Fügen Sie beim Aufnehmen von Bildern ein Lineal hinzu, erstellen Sie ein Liniensegment, wählen Sie ein geradliniges Segment aus (z. B. 1 cm und legen Sie dann die Länge dieses Liniensegments auf 1 cm fest) und markieren Sie die Länge des Bretts.
- Öffnen Sie das Fenster "Maßstab festlegen ". Klicken Sie auf Analysieren | Festlegen der Skalierung.
- Geben Sie die tatsächliche Länge in Bekannte Entfernung und Längeneinheit ein.
- Aktivieren Sie Global.
- Geben Sie die Ordneradresse des Originalbildes und die Adresse der Ausgabedatei ein.
- Verwenden Sie das Werkzeug "Gerade Linie ", um die Gemeinschaften nacheinander auszuwählen, und passen Sie die Toleranz an, bis die gerade Werkzeuglinie an die Grenze der Bakterienkolonie passt, die sich zu beiden Seiten erstreckt.
- Messen Sie den Migrationsabstand der Kolonie von der Mitte zu beiden Enden separat und leiten Sie die geradlinige Länge ab, indem Sie auf Analysieren | Messen.
- Wiederholen Sie die Schritte 5.3.3 bis 5.3.4, bis alle Communitys gemessen wurden, und speichern Sie die Ergebnisse zur weiteren Analyse in einer .csv Datei.
Ergebnisse
Diese Arbeit beschreibt die Ansätze zur Charakterisierung der isolierten Mikroben aus dem Wirtsmikrobiom (Abbildung 1). Als Proof-of-Concept wurden drei Mikroben aus dem SD-Wirt der Ratte isoliert (C. stationis, S. cohnii und E. faecalis), und zwei kommerziell erworbene Mikroorganismen (S. aureus ATCC 25923 und E. faecalis V583) wurden unter Verwendung dieses Protokolls getestet. Um den Sauerstoffbedarf einzelner Mikroben mittels FTM zu ermitteln, fügten wir zwei Kontrollorganismen hinzu, einen obligaten Anaeroben (Fusobacterium nucleatum ATCC 25586) und einen fakultativen Anaerobier (Pseudomonas aeruginosa PAO1). Die Ergebnisse zeigten, dass es sich bei den fünf getesteten Mikroben um fakultative Anaerobier handelte (Abbildung 2A), was mit den zuvor berichteten Profilen dieser Mikroben übereinstimmte11. Um ihren Sauerstoffbedarf zu ermitteln, wurden die Mikroben sowohl unter aeroben als auch unter anaeroben Bedingungen kultiviert. Alle getesteten Mikroben zeigten sowohl unter aeroben als auch unter anaeroben Bedingungen eine sigmoidale Wachstumskurve; Die mikrobielle Dichte, wie sie durch den OD600nm angezeigt wird, zeigte jedoch, dass alle fünf Mikroben eine höhere Präferenz für sauerstoffreiche Umgebungen haben (Abbildung 2B,C). Diese Beobachtungen stimmen mit den mikrobiellen Fähigkeiten überein, die Rattenknochen zu infiltrieren und zu infizieren, die allgemein als hypoxisch gelten.
Es wurde festgestellt, dass die fünf Bakterienisolate auf natürliche Weise Biofilme bilden, wobei die erzeugte Biofilmmasse unter anaeroben Bedingungen höher war. Durch den Vergleich der Wachstumskurve wurde festgestellt, dass die Biofilme von E. faecalis und S. aureus ATCC 25923 während der stationären Wachstumsphase sowohl in aeroben als auch in anaeroben Umgebungen aktiv gebildet werden (Abbildung 3). Diese Beobachtung deutet darauf hin, dass die Biofilmbildung vom Wachstumszustand der Zellen abhängt, der möglicherweise durch intrazelluläre Signalmoleküle reguliert wird, die die Biofilmbildung auslösen11. Diese Abhängigkeit der Biofilmbildung von den Wachstumsbedingungen stimmt mit den Nährstoff- und Wachstumsbedingungen überein, die in der sauerstoff- und nährstoffarmen Umgebung der Knochenmatrix des Wirts herrschen.
Studien über die Pathogenese von Mikroben werden oft mit der Fähigkeit der Mikrobe in Verbindung gebracht, an Orte zu wandern, die als förderlich für das Wachstum gelten. Es gibt viele Studien zur mikrobiellen Chemotaxis, aber es gibt keinen standardisierten Ansatz, um sie zu untersuchen. Darüber hinaus wurde die Verwendung von nicht-diffusiblen Chemikalien als limitierender Faktor bei der Untersuchung dieses Phänomens angesehen. Hier haben wir eine per 3D-Druck hergestellte Harzabdeckung entwickelt, die den Abstand zwischen dem chemischen Reservoir und dem Impfpunkt standardisiert (Ergänzende Abbildung S1 und Ergänzende Abbildung S2). Wir untersuchten die Migration der Mikroben in eine nährstoffreiche Umgebung, in der 5x TB als chemisches Stimulans verwendet wurde. Die Mikroben wurden 48 Stunden lang unter aeroben und anaeroben Bedingungen überwacht, wobei alle fünf Mikroben unterschiedliche Grade der Tropismusmigration zeigten (Abbildung 4A).
ImageJ wurde verwendet, um die Migrationsdistanz von Bakterien zu messen (Abbildung 4B); Die Migration in Richtung des Stausees schien schneller zu sein als die Migration in die entgegengesetzte Richtung. Das Verhältnis der Migrationsdistanz zum Reservoir zu der in die entgegengesetzte Richtung zeigt die rasche Ausdehnung der mikrobiellen Kolonie aufgrund der mikrobiellen Chemotaxis (Abbildung 5B). In dieser Studie wanderten Bakterien wie S. aureus ATCC 25923 schneller als andere Mikroben wie C. stationis. Interessanterweise zeigte das Migrationsprofil chemotaktischer Kulturen unter aeroben und anaeroben Bedingungen, dass einige Bakterien, wie z.B. E. faecalis, Variationen in den Migrationsmustern unter aeroben und anaeroben Bedingungen zeigten, wobei eine schnelle Expansion unter anaeroben Bedingungen im Laufe der Zeit beobachtet wurde. Verwirrenderweise begünstigte der Laborstamm E. faecalis V583 eine aerobe Umgebung für die Chemotaxis. Dies deutet darauf hin, dass E. faecalis , die aus verschiedenen Quellen isoliert wurden, unterschiedliche Wachstumsanforderungen haben, die an die Wachstumsumgebung angepasst sind.

Abbildung 1: Schematische Darstellung des Betriebsablaufs. (A) Bakterien werden auf Platten und dann in flüssiges Medium geimpft und bis zur logarithmischen Wachstumsphase gezüchtet. (B) Bestimmung der Fähigkeit von Bakterien, Biofilme unter verschiedenen Bedingungen (aerob/anaerob) zu bilden. Für jeden Zeitpunkt (6 h) wurden drei Proben ausgewählt und parallel wiederholt. (C) Das Protokoll für das chemische Tropismus-Experiment von Bakterien unter verschiedenen Bedingungen (aerob/anaerob). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Mikrobielle Wachstumsbedingungen. (A) Das Wachstumsprofil von Bakterien, die in flüssigem Thioglycollat-Medium kultiviert wurden (von links nach rechts: I: Fusobacterium nucleatum ATCC 25586, II: Corynebacterium stationis, III: Staphylacoccus cohnii, IV: Enterococcus faecalis, V: Staphylacoccus aureus ATCC 25923, VI: Enterococcus faecalis V583 und VII: Pseudomonas aeruginosa PAO1). (B) Wachstumskurve von Mikroorganismen, die unter aeroben Bedingungen aus dem Wirt isoliert wurden. (C) Wachstumskurve von Mikroorganismen, die unter anaeroben Bedingungen aus dem Wirt isoliert wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
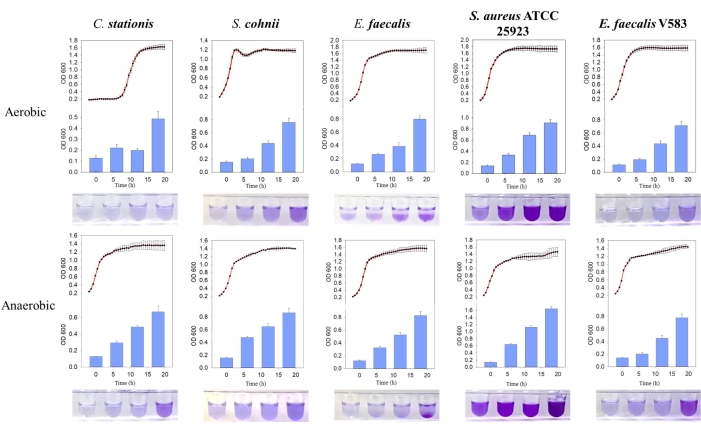
Abbildung 3: Vergleich der Wachstumskurven und der Biofilmbildung verschiedener Bakterien unter verschiedenen Bedingungen. Von links nach rechts sind Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 und Enterococcus faecalis V583. Die rote Kurve ist die Wachstumskurve und das blaue Histogramm ist die Menge des produzierten Biofilms. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
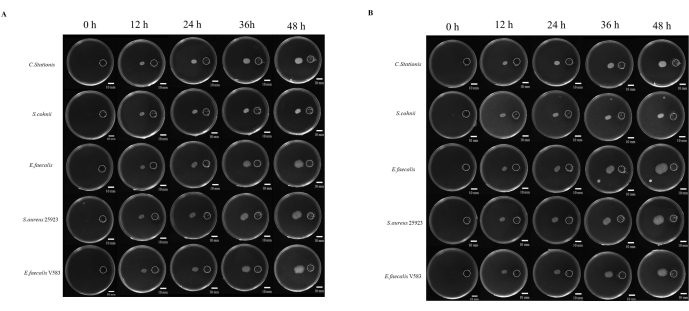
Abbildung 4: Mikrobielles Wachstum in Richtung Eutrophierung. (A) Aerobe Bedingungen, (B) anaerobe Bedingungen. Fünf Arten von Bakterien (von oben nach unten sind Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 und Enterococcus faecalis V583) neigen dazu, auf der differentiellen Nährstoffplatte in Richtung Eutrophierung zu wachsen. Maßstabsleisten = 10 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Chemotaxis von Mikroben unter verschiedenen Bedingungen (aerob/anaerob). (A) Die Ergebnisse der Migration von fünf Bakterien durch chemischen Tropismus auf einer Platte, die nährstoffreichen Agar enthält; Die linke Gruppe stellt die aeroben Bedingungen und die rechte Gruppe die anaeroben Bedingungen dar. Jedes Experiment wurde in dreifacher Ausfertigung parallel wiederholt. (B) Datenanalyse des chemischen Tropismus. Der obere Teil stellt die aeroben Bedingungen und der untere Teil die anaeroben Bedingungen dar. Von links nach rechts sind Corynebacterium stationis, Staphylacoccus cohnii, Enterococcus faecalis, Staphylacoccus aureus ATCC 25923 und Enterococcus faecalis V583. Die roten und blauen Säulen stellen die Migrationswege vom Nährstoffkörper von Bakterien dar, die zum Nährstoffkörper hin und von ihm weg wandern. Die Linie ist die Differenz zwischen den beiden Werten (Rot - Blau). Maßstabsleisten = 10 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Verwendung von 3D-gedruckten Deckeln zur Herstellung von Agarplatten mit Nährstoffunterschied. (A) Verwenden Sie Software, um 3D-Druck-Form-Renderings vorzubereiten. (B) Flache Deckform, bedruckt mit 8.000er Harz. (C) Verwenden Sie die obige Methode, um die Nährstoffdifferenz-Agarplatten zu erhalten. (D) Herstellung der Agarplatten für den Nährstoffunterschied. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S2: Konstruktion von Nährstoffdifferenz-Agarplatten und die Messung der bakteriellen Migrationsdistanz mit ImageJ. (A) Bei der Konstruktion von Agarplatten für Nährstoffdifferenzen wird ein 3D-gedruckter Deckel (mit einem zylindrischen Vorsprung) verwendet, um die Platte abzudecken. Nach dem Abkühlen des Agars bildet sich eine zylindrische Vertiefung (der rote Kreis), die mit 5x TB Agar gefüllt wird. Diese wird dann abgekühlt, um Agarplatten mit Nährstoffunterschied zu erhalten. (B) Verwenden Sie ImageJ, um den Migrationsabstand von Bakterien in verschiedene Richtungen zu messen. Festlegen der Maßstabsleiste (Analysieren | Festlegen des Maßstabs) und Exportieren der Migrationsentfernung mit der Option Gerade (Analysieren | Messen). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S3: Wachstumsmuster von fünf Bakterien auf M9 minimalem Medium - 5x TB Medium Agar. Die linke Gruppe ist für aerobe Bedingungen und die rechte Gruppe für anaerobe Bedingungen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S4: Kontrollexperimente an Fusobacterium nucleatum ATCC 25586 Wachstumskurven und Chemotaxis von anaeroben Bakterien. (A) Bestimmung der Wachstumskurven von F. nucleatum ATCC 25586 unter aeroben und anaeroben Bedingungen. (B) Chemotaxis-Experimente mit F. nucleatum ATCC 25586 auf MH-5x TB-Agarplatten mit halber Dosis unter verschiedenen Sauerstoffbedingungen. Teilweise Vergrößerung: Wachstum von F. nucleatum ATCC 25586 auf der Platte unter anaeroben Bedingungen nach 48 h (partielle Vergrößerung). Bitte klicken Sie hier, um diese Datei herunterzuladen.
| Verbindung | Konzentration (g/L) |
| Na2HPO4 | 6.78 |
| KH2PO4 | 3 |
| NaCl | 0.5 |
| NH4Cl | 1 |
| MgSO4,7H 2O | 0.493 |
| CaCl2 | 0.011 |
| Traubenzucker | 4 |
Tabelle 1: Komponenten und Inhalt des M9 Minimal-Mediums.
Diskussion
Wir haben fünf Bakterienarten isoliert und mit sequentiellen Methoden identifiziert. Das Wachstum von Bakterien hat einen minimalen Nährstoffbedarf: das minimale Medium - ein Medium, das nur anorganische Salze, eine Kohlenstoffquelle und Wasser enthält. Obwohl die Bakterien in der Versuchsgruppe auf MH-Festplatten gefunden wurden, verwendeten wir MH-Medium mit halber Dosis, um die Chemotaxis der Bakterien zu verifizieren, und erzielten gute Ergebnisse. Wir führten jedoch auch Kontrollexperimente mit minimalem Medium durch. Im Experiment wurde das Basismedium M9 verwendet (siehe Tabelle 1 für seine Zusammensetzung und seinen Gehalt), und für den Nähragarblock wurde weiterhin 5x TB-Agar verwendet. Die experimentellen Ergebnisse sind in der ergänzenden Abbildung S3 dargestellt. Obwohl diese Bakterien (außer E. faecalis) langsam wachsen und sich im M9-Medium langsam bewegen, gibt es im Allgemeinen einen ähnlichen Trend (zum Wachstum auf Nähragar), wie in diesen Experimenten beobachtet. E. faecalis V583 wuchs jedoch nicht auf M9-Minimummedium, möglicherweise aufgrund des Mangels an Komponenten, die für das Wachstum von E. faecalis in M9-Medium erforderlich waren. Daher empfehlen wir die Verwendung von MH-Medium mit halber Dosis als nährstoffarmes Substrat, um das Wachstum und die Migration von Bakterien unter nährstoffarmen Bedingungen zu simulieren. Dies erleichtert auch die Verwendung der ImageJ-Software, um die Entfernung zu messen, über die Bakterien zur Wanderung neigen.
Biofilme werden im Allgemeinen als oberflächenassoziierte mikrobielle Gemeinschaften definiert und sind eine wichtige mikrobielle Überlebensstrategie. Organismen wie Pseudomonas aeruginosa, Haemophilus influenzae, Streptococcus pneumoniae und Staphylococcus aureus können in asymptomatischen Wirten gefunden werden, und die Entdeckung von anhaftenden Mikroorganismen in vivo wird oft als Besiedlung und nicht als Infektion bezeichnet. Ein sehr wichtiges Problem ist das Fehlen eines idealen Tierbesiedlungsmodells12. Mikroben zeigen erste Anhaftungen an geladene Oberflächen, bevor sich Biofilme bilden, wobei Materialien wie Glas und Polyvinylchlorid (PVC) als geeignet für Biofilm-bezogene Studien erachtet werden. Studien mit PVC-96-Well-Platten wurden verwendet, um die mikrobiellen Biofilme9 zu untersuchen, wobei Einzel- oder Mehrspezies-Biofilme zu locker oder dicht gepackten Biofilmenführen können 10. Daher gilt die Verwendung der PVC-96-Well-Platte als ungeeignet für lose gepackte Biofilme, da die Biofilmmatrix während des Biofilmfärbeprozesses leicht abgewaschen werden kann. Daher haben wir uns auf Reagenzgläser aus Glas verlassen, um die Bildung mikrobieller Biofilme zu untersuchen (Abbildung 3).
Eine wesentliche Einschränkung dieser Methode besteht darin, dass sie nicht mit Mikroorganismen funktioniert, die unter bestimmten Bedingungen wachsen, und nicht alle Bakterien migrieren können. Darüber hinaus sind Biofilme zwar ein wirksames Mittel, um Mikroorganismen bei der Abwehr widriger äußerer Umwelteinflüsse zu unterstützen, aber nicht alle Wirtsmikroorganismen sind in der Lage, Biofilme zu bilden. Zum Beispiel wächst das bereits erwähnte F. nucleatum ATCC 25586, das mit einer menschlichen Mund- oder Darmsymbiose assoziiert ist, die mit der Entwicklung von Dickdarmkrebs assoziiert ist17, nur spezifisch in anaeroben Umgebungen, produziert aber keine Biofilme. Wir haben es in FTM-Experimenten verwendet, um die Wirksamkeit des FT-Mediums zu überprüfen. Das FT-Medium hat sich als wirksam bei der Versorgung von Bakterien mit Gradientenkonzentrationen von Sauerstoff erwiesen.
Zum Vergleich haben wir die Wachstumskurve und den chemischen Tropismus des anaeroben Bakteriums F. nucleatum ATCC 25586 getestet (Ergänzende Abbildung S4). In den Experimenten zur Wachstumskurve (Ergänzende Abbildung S4A) konnte F. nucleatum ATCC 25586 unter aeroben Bedingungen nicht wachsen und zeigte unter anaeroben Bedingungen eine bakterielle Aggregation am Boden der Vertiefungen. Auch in den Chemotaxis-Experimenten (Ergänzende Abbildung S4B) wuchs F. nucleatum ATCC 25586 nicht unter aeroben Bedingungen, und eine geringe Anzahl von Bakterien wurde nach einer in situ Inokulation unter anaeroben Bedingungen für 48 h gefunden. Da einige Bakterien nicht aktiv nach geeigneten Wachstumsbedingungen suchen können, indem sie migrieren, warten sie auf geeignete Wachstumsbedingungen, indem sie in einen langfristigen Ruhezustand eintreten18. Zu beachten ist, dass für die Kultivierung streng anaerober Bakterien ein spezielles Medium, wie z.B. ET-Fermentationsmedium (halbfeste Platten müssen mit 0,5% Agar ergänzt werden) benötigt wird, das sich ebenfalls von der herkömmlichen Isolierung und Identifizierung unterscheidet. Bei dieser vorgeschlagenen Methode handelt es sich um eine allgemeine Methode, die zur Charakterisierung der meisten Wirtsmikroorganismen anwendbar ist, wobei die Technik für fünf Mikroben demonstriert wird, darunter drei Mikroben, die aus Mausknochen isoliert wurden, und zwei Mikroben in Laborqualität. Dieses Protokoll würde die korrekte Identifizierung von Mikrobiota-Isolaten in zukünftigen Studien unterstützen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Entwicklung dieser Technik wurde durch die Mittel des Forschungsfonds für internationale junge Wissenschaftler der National Natural Science Foundation of China (22050410270), des Shenzhen Special Fund for Innovation and Entrepreneurship of Overseas High-level Talents Peacock Team (KQTD20170810111314625) und des Guangdong Innovative and Entrepreneurial Research Team Program (2019ZT08Y191) unterstützt. Wir möchten Frau Chen Xinyi unseren aufrichtigen Dank für ihre Unterstützung beim Korrekturlesen des Dokuments und bei der Laborleitung aussprechen.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemical/Solution | |||
| 1% crystal violet dye solution | Solarbio | G1062 | 100 mL |
| Agar | Sigma-Aldrich | V900500 | Used to obtain semi-solid plates, 20 g |
| Centrifuge tube | Corning | 430790 | 15 mL |
| Fluid thioglycollate medium | Kinghunt | K0001 | 29.3 g |
| Mueller Hinton II Broth medium | Solarbio | NO.11865 | 100 g |
| Petri dishes | Bkman | B-SLPYM90-15 | Plastic Petri dishes, circular, 90 mm x 15 mm |
| Potassium Chloride | VETEC | WXBC4493V | 0.2 g |
| Potassium Dihydrogen Phosphate | aladdin | 04-11-7758 | 0.24 g |
| Sodium chloride | Macklin | S805275 | 8.0 g |
| Sodium phosphate dibasic | aladdin | 7558-79-4 | 1.44 g |
| Terrific Broth medium | Solarbio | LA2520 | 200 g |
| Kits/ Equipment | |||
| Anaerobic incubator | Longyue | ||
| Biochemical incubator | Blue pard | LRH-70 | |
| Microplate reader | Spark | ||
| Tanon 5200multi imaging system | Tanon | 5200CE | |
| Thermostatic water bath | Jinghong | DK-S28 |
Referenzen
- Bernardi, S., et al. Combining culture and culture-independent methods reveals new microbial composition of halitosis patients' tongue biofilm. MicrobiologyOpen. 9 (2), 958 (2020).
- Tellez, G., Latorre, J. D., Kuttappan, V. A., et al. Rye affects bacterial translocation, intestinal viscosity, microbiota composition and bone mineralization in Turkey poults. PLOS One. 10, (2015).
- Brewer, J. H. Clear liquid mediums for the "aerobic" cultivation of anaerobes. Journal of the American Medical Association. 115 (8), 598 (1940).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Liu, C., et al. The regulation of bacterial biofilm formation by cAMP-CRP: a mini-review. Frontiers in Microbiology. 11, 802 (2020).
- Kumar, A., et al. Biofilms: survival and defense strategy for pathogens. International Journal of Medical Microbiology. , (2017).
- Penesyan, A., et al. Secondary effects of antibiotics on microbial biofilms. Frontiers in Microbiology. 11, 2109 (2020).
- Bernardi, S., et al. Subinhibitory antibiotic concentrations enhance biofilm formation of clinical Enterococcus faecalis isolates. Antibiotics. 10 (7), 874 (2021).
- Tsukatani, T., Fumihiko, S., Rieko, K. A rapid and simple measurement method for biofilm formation inhibitory activity using 96-pin microtiter plate lids. World Journal of Microbiology and Biotechnology. 36 (12), 1-9 (2020).
- Hartmann, R., et al. Emergence of three-dimensional order and structure in growing biofilms. Nature Physics. 15, 251-256 (2019).
- Liu, Z., et al. Advances in bacterial biofilm management for maintaining microbiome homeostasis. Biotechnology Journal. 15 (10), 1900320 (2020).
- Hall-Stoodley, L., Stoodley, P. Evolving concepts in biofilm infections. Cellular Microbiology. 11 (7), 1034-1043 (2009).
- Røder, H. L., et al. Enhanced bacterial mutualism through an evolved biofilm phenotype. The ISME Journal. 12 (11), 2608-2618 (2018).
- Matilla, M. A., Tino, K. The effect of bacterial chemotaxis on host infection and pathogenicity. FEMS Microbiology Reviews. 42 (1), 052 (2018).
- Moreira, L. M., et al. Chemotactic signal transduction and phosphate metabolism as adaptive strategies during citrus canker induction by Xanthomonas citri. Functional & Integrative Genomics. 15 (2), 197-210 (2015).
- Konishi, H., et al. Bacterial chemotaxis towards polysaccharide pectin by pectin-binding protein. Scientific Reports. 10 (1), 1-12 (2020).
- Kostic, A. D., et al. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment. Cell Host & Microbe. 14 (2), 207-215 (2013).
- Kochan, T. J., et al. Updates to Clostridium difficile spore germination. Journal of Bacteriology. 200 (16), 00218 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten