Method Article
Detecção de infecção por Helicobacter pylori e resistência a antibióticos por meio da análise quantitativa da reação em cadeia da polimerase nas fezes
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
O protocolo emprega uma amostragem de fezes não invasiva combinada com uma reação em cadeia da polimerase quantitativa para oferecer um método de diagnóstico conveniente e rápido para a infecção por Helicobacter pylori e sua resistência à claritromicina e quinolonas.
Resumo
O Helicobacter pylori (H. pylori) é amplamente prevalente em todo o mundo, com aproximadamente 50% da população global com histórico de infecção por H. pylori. Na China, a taxa de infecção varia de 40% a 70%. O H. pylori está associado principalmente a doenças gastrointestinais, como gastrite crônica, úlceras gástricas e úlceras duodenais. Atualmente, o tratamento clínico para a infecção por H. pylori envolve terapia tripla ou quádrupla. No entanto, o uso extensivo de antibióticos levou ao desenvolvimento de resistência a antibióticos em H. pylori. Portanto, detectar tanto o H. pylori quanto sua resistência a antibióticos é crucial para orientar o tratamento clínico.
Os métodos diagnósticos para H. pylori incluem teste respiratório de ureia (UBT), teste de antígeno, teste sorológico de anticorpos, endoscopia, teste rápido de urease (RUT) e cultura bacteriana. Embora os três primeiros métodos sejam não invasivos, eles não permitem a recuperação bacteriana e, portanto, não podem ser usados para testes de resistência. Os três últimos métodos são invasivos, caros, exigem alto conhecimento técnico e podem causar danos aos pacientes.
Portanto, um método rápido e não invasivo para detecção simultânea de infecção por H. pylori e resistência a antibióticos é de extrema importância clínica para a erradicação efetiva de H. pylori. Este artigo tem como objetivo apresentar um protocolo específico que combina reação em cadeia da polimerase quantitativa (qPCR) com a tecnologia de sonda fluorescente TaqMan para detectar rapidamente a infecção por H. pylori e a resistência a antibióticos. Este método fornece uma maneira conveniente, rápida e não invasiva de diagnosticar a infecção e resistência ao H. pylori , ao contrário da cultura bacteriana tradicional e de outras técnicas. A qPCR é usada para identificar a infecção e detectar mutações nos genes 23S rRNA e gyrA , que estão ligados à resistência à claritromicina e quinolonas, respectivamente. Em comparação com as técnicas de cultura convencionais, essa abordagem oferece um método não invasivo, econômico e eficiente em termos de tempo para detectar a infecção por Helicobacter pylori e determinar sua resistência a antibióticos.
Introdução
O Helicobacter pylori é uma bactéria gram-negativa, em forma de espiral, que pode infectar persistentemente o epitélio gástrico humano1. Em 1994, a Organização Mundial da Saúde classificou o H. pylori como cancerígeno do Grupo 1 para câncer gástrico, com ~ 3% dos indivíduos infectados desenvolvendo a doença2. Uma revisão sistemática publicada recentemente indicou que uma tendência geral de aumento da taxa de resistência a antibióticos do H. pylori foi observada na última década, que atingiu níveis alarmantes em todo o mundo, especialmente a resistência à claritromicina3. Na China, os dados da pesquisa mais recente mostraram que a prevalência agregada de H. pylori entre os chineses urbanos é de 27,08%; as taxas de resistência à claritromicina e levofloxacina foram de 50,83% e 47,17%4, superiores às dos Estados Unidos (levofloxacino 37,6%, claritromicina 31,5%)5e Europa (levofloxacino 15,8% e claritromicina 21,4%)6. Consequentemente, o diagnóstico e o tratamento precoces e precisos são cruciais. No entanto, o aumento da resistência a antibióticos do H. pylori reduz consideravelmente a eficácia do tratamento, destacando a necessidade urgente de pesquisas contínuas sobre seu diagnóstico e tratamento.
Os métodos diagnósticos por H. pylori são categorizados em técnicas invasivas e não invasivas7. Os métodos invasivos incluem exame histopatológico, teste rápido de urease (RUT) e cultura bacteriana8. O exame histopatológico depende do processamento e da observação microscópica de amostras de biópsia, com precisão limitada pelo preparo histológico e pela experiência patológica9. O RUT detecta o H. pylori por meio da atividade da urease, que hidrolisa a uréia para produzir amônia, resultando em um deslocamento alcalino no reagente, indicando a presença da bactéria9. É simples e econômico. A cultura bacteriana, considerada o "padrão ouro", geralmente tem uma taxa de sucesso abaixo de 50% devido às características fisiológicas únicas do H. pylori10.
Os métodos não invasivos incluem o teste respiratório da ureia 13C/14C (UBT), o diagnóstico sorológico e o teste do antígeno fecal (SAT)11. O teste respiratório opera com base em um princípio semelhante ao do RUT, detectando CO2 marcado isotopicamente no ar exalado para confirmar a infecção12. O diagnóstico sorológico detecta anticorpos, que podem persistir após a erradicação bacteriana, dificultando a avaliação da eficácia do tratamento13. O teste de antígeno fecal detecta antígenos específicos de H. pylori em amostras fecais, fornecendo um método não invasivo confiável com menor taxa de falsos positivos e boa acurácia para o diagnóstico de infecções ativas14.
Cada método diagnóstico tem suas vantagens e limitações15. Além da cultura bacteriana, outros métodos lutam para detectar a resistência aos antibióticos, enquanto a cultura é prejudicada por baixas taxas de sucesso16. Recentemente, métodos de diagnóstico molecular, como PCR quantitativo em tempo real (qPCR), têm sido amplamente aplicados à detecção microbiana17. A qPCR pode detectar com precisão o H. pylori e analisar mutações genéticas de resistência, oferecendo uma visão mais abrangente da infecção18.
As sondas fluorescentes são projetadas para se ligar especificamente a uma sequência alvo no DNA durante o processo de qPCR. Essas sondas são normalmente marcadas com um corante fluorescente, que emite um sinal quando a sonda se liga ao seu alvo e é clivada pela polimerase durante a amplificação. Isso fornece uma medição em tempo real do processo de PCR. As sondas fluorescentes são conhecidas por sua alta especificidade devido ao design exclusivo da sonda, que garante que apenas a sequência de DNA alvo seja detectada, reduzindo as chances de ligação não específica e melhorando a especificidade do ensaio. Isso torna a tecnologia de sonda fluorescente especialmente útil para detectar alvos de baixa abundância, como o DNA de Helicobacter pylori em amostras de fezes. Embora o SYBR Green seja um corante de ligação ao DNA comumente usado em PCR, ele pode se ligar a qualquer DNA de fita dupla, o que pode resultar em amplificação não específica e falsos positivos.
A cultura de Helicobacter pylori a partir de amostras clínicas não fornece dados diretos sobre resistência a antibióticos. Em contraste, a qPCR baseada em sonda fluorescente é mais rápida, mais conveniente e pode ser adaptada para detecção simultânea de H. pylori e marcadores de resistência a antibióticos. Este método não apenas fornece resultados mais rápidos do que os métodos tradicionais de cultura, mas também permite a detecção simultânea de genes de resistência a antibióticos, aumentando muito a eficiência e a conveniência da detecção. Este método fornece uma maneira conveniente, rápida e não invasiva de diagnosticar a infecção e resistência ao H. pylori , ao contrário da cultura bacteriana tradicional e de outras técnicas.
Existem duas questões cruciais que precisam ser consideradas para a resistência a antibióticos de H. pylori : i) a consistência do genótipo e fenótipo, ii) a multirresistência. Um estudo multicêntrico em larga escala na China constatou que os padrões de dupla resistência para claritromicina/levofloxacino foram de 26.1%. Os fenótipos e genótipos de H pylori resistentes à claritromicina e levofloxacino apresentaram concordância satisfatória (coeficiente kappa = 0,810 e 0,782, respectivamente)19. Portanto, é viável usar o método qPCR para detectar infecção por H. pylori e resistência a drogas. Os métodos de teste de antígeno fecal (SAT) e qPCR são cada vez mais usados devido à conveniência da amostragem. A sensibilidade, especificidade e acurácia dos métodos de detecção de H. pylori são as seguintes: qPCR > UBT > SAT > RUT> IgG > cultura20. Todos os métodos não invasivos são adequados para triagem primária. Como a qPCR também pode detectar genes de resistência a medicamentos, a qPCR e a cultura serão mais adequadas para orientar o tratamento.
Outras abordagens inovadoras, como algoritmos de inteligência artificial (IA) combinados com imagens endoscópicas21, microfluídica digital22 e RPA-CRISPR / Cas12a23, teoricamente mostram boa eficiência diagnóstica e perspectivas de aplicação promissoras. No entanto, a qPCR é atualmente a técnica de biologia molecular mais sofisticada com potencial para fornecer uma solução superior para o diagnóstico e tratamento de infecções por H. pylori .
Protocolo
Este estudo segue as diretrizes éticas estabelecidas pelo Comitê de Ética do Hospital Popular da Província de Guangdong, Southern Medical University, Guangzhou, China (Aprovação nº: KY2024-445-01). Informações detalhadas sobre os materiais utilizados nesta pesquisa (reagentes, produtos químicos, equipamentos e softwares) podem ser encontradas na Tabela de Materiais.
1. Seleção dos participantes
- Escolha participantes na faixa etária de 18 a 60 anos.
- Escolha participantes que possam ser submetidos à triagem de infecção por Helicobacter pylori , como pacientes que apresentem sintomas como gastrite, úlceras pépticas ou dispepsia, bem como indivíduos assintomáticos.
- Exclua do estudo pacientes que usaram antibióticos, agentes contendo bismuto (como cápsulas de citrato de bismuto de potássio, cápsulas de pectina de bismuto coloidal ou comprimidos compostos de bismuto-alumínio) ou medicamentos chineses antimicrobianos no último mês, ou aqueles que usaram inibidores da bomba de prótons ou antagonistas do receptor H2 (como omeprazol, pantoprazol, rabeprazol, cimetidina ou nizatidina) nas últimas 2 semanas. Exclua também do estudo as mulheres durante o período menstrual.
2. Coleta de amostras fecais
NOTA: Forneça a seção 2 como instruções aos participantes.
- Confirmar se a embalagem exterior do kit de recolha de amostras não está danificada e dentro do prazo de validade. Remova os materiais do kit.
- Coloque um papel coletor fecal branco no vaso sanitário e defece (para evitar contaminação com urina).
- Usando uma espátula, colete uma porção de fezes aproximadamente do tamanho de uma fava (~ 5 g) e transfira-a para o recipiente de amostra.
- Coloque a espátula de amostragem, juntamente com a amostra fecal, no tubo de coleta, garantindo que o nível do líquido suba para perto da linha de amostragem indicada no tubo.
- Aperte firmemente a tampa do tubo. Inverta o tubo várias vezes para garantir a mistura completa da amostra com a solução conservante.
- Armazenar a amostra sob refrigeração até a entrega ao laboratório clínico para a seguinte identificação de H. pylori e avaliação dos perfis de resistência a antibióticos via qPCR.
3. Extração de ácido nucleico
NOTA: Conclua todos os procedimentos dentro de um gabinete de biossegurança para evitar contaminação.
- Inverter o tubo de conservação da amostra e misturar bem. Transfira 1,0 mL da solução da amostra fecal para um tubo de microcentrífuga.
- Depois de misturar os tubos da centrífuga, colocá-los em um banho de metal a 80 °C por 10 min, com mistura intermitente por 30 s no quinto minuto. Depois que os tubos esfriarem à temperatura ambiente, centrifugue a 10.000 × g por 5 min e colete o sobrenadante para processamento posterior.
- Inverta a placa de 96 poços várias vezes para ressuspender as esferas magnéticas. Remova cuidadosamente a vedação da folha de alumínio, tomando cuidado para evitar tremores para evitar derramamento.
- Adicione 200 μL da amostra preparada (da etapa 3.2) a cada poço da placa de 96 poços contendo tampão de lise, garantindo que cada poço corresponda a uma única amostra. Coloque a placa de 96 poços no compartimento de amostra designado do instrumento de extração de ácido nucleico para extração automatizada.
- Armazene quaisquer amostras restantes e as amostras de ácido nucleico extraídas a -20 °C para preservação a longo prazo e uso futuro.
4. qPCR Detecção de ácidos nucléicos Helicobacter pylori e mutações de resistência à claritromicina e quinolonas
- Retire os reagentes da área de armazenamento de reagentes e descongele-os à temperatura ambiente.
NOTA: Este kit foi usado para a detecção qualitativa do ácido nucleico H . pylori , incluindo mutações no gene 23S rRNA (A2143G, A2143C, A2144G) e no gene gyrA (261A, 261G, 260T, 271A, 271T, 272G). Os primers, as sondas, a enzima Taq, a enzima UNG e os dNTPs estavam disponíveis no kit. Os genes-alvo de H. pylori e β-actina humana foram incluídos no controle de qualidade positivo. O geneda β-actina humana foi escolhido como controle de qualidade interno. - Com base no número de amostras a serem testadas, use N + 2 cópias do reagente liofilizado, onde N representa o número de amostras a serem testadas e 2 representa os CCs negativos e positivos.
- Centrifugue brevemente para garantir que o pó liofilizado assente no fundo dos tubos. Abrir a tampa do reagente liofilizado (tendo o cuidado de evitar qualquer derrame do pó) e adicionar 25 μL por alvéolo dos ácidos nucleicos extraídos das amostras a ensaiar, bem como os controlos positivo e negativo. Feche bem os tubos.
- Vortex os reagentes de PCR por 8-10 s, depois centrifugue brevemente por 3-5 s para evitar a formação de bolhas.
- Coloque a placa qPCR de 96 poços na máquina qPCR. Defina o programa de ciclagem da seguinte forma: primeiro, incube a mistura de reação (contendo primers, sondas, Taq polimerase, enzima UNG, dNTPs, etc.) a 42 ° C e 95 ° C (ambas as etapas em um ciclo) por 2 min; em seguida, cicle 10x com 10 s a 95 °C e 45 s a 65 °C; finalmente, ciclo 35x com 10 s a 95 ° C e 45 s a 58 ° C para desnaturação, recozimento e extensão.
- Defina os parâmetros de detecção do sinal de fluorescência da seguinte forma: Marcação fluorescente ROX de genes conservados de H. pylori ; Marcação fluorescente FAM de genes de resistência à claritromicina de H. pylori ; Marcação fluorescente HEX de genes de resistência a quinolonas de H. pylori ; e marcação fluorescente CY5 do gene da β-actina humana foram usados como controle interno do kit. Coletar dados a 58 °C. Após a conclusão da reação, certifique-se de que os dados sejam salvos para análise futura.
- Analise os dados usando um software específico para qPCR, pois o instrumento seleciona automaticamente o limite da linha de base. Defina todos os critérios diagnósticos, incluindo a presença de infecção ou resistência ao Helicobacter pylori , para um valor de TC ≤30; confirmam que eles exibem uma curva característica em forma de S.
Resultados
Aplicação de qPCR para detecção de infecção por Helicobacter pylori e resistência a antibióticos em amostras fecais
Projetamos primers e sondas com base em mutações pontuais nos genes conservados de Helicobacter pylori, bem como no gene 23S rRNA e no gene gyrA . Esses primers e sondas foram marcados com diferentes corantes fluorescentes e depois usados para detecção de qPCR. Os resultados do controle de qualidade para os experimentos de qPCR estavam dentro da faixa recomendada, indicando resultados de detecção confiáveis (Figura 1). Neste estudo, foram selecionadas cinco amostras representativas (S1-S5) para caracterizar a confiabilidade do protocolo experimental (Figura 2 e Tabela 1). S1 é uma amostra sem infecção por H. pylori , enquanto S2-S5 são amostras com diferentes perfis de resistência a medicamentos.
Para amostras positivas para infecção por Helicobacter pylori , a amostra S2 apresentou valores de TC dentro da faixa de detecção apenas para Helicobacter pylori, indicando que essa amostra é positiva para Helicobacter pylori e é sensível tanto à claritromicina quanto às quinolonas, permitindo o tratamento com qualquer um dos medicamentos. A amostra S3 apresentou valores de TC dentro da faixa de detecção tanto para infecção por Helicobacter pylori quanto para resistência à claritromicina, sem valores de TC detectados para resistência a quinolonas, sugerindo que a amostra S3 é de um paciente resistente à claritromicina. Da mesma forma, a amostra S4 apresentou valores de TC dentro da faixa de detecção de infecção por Helicobacter pylori e resistência a quinolonas, sem valores de TC detectados para resistência à claritromicina, indicando resistência às quinolonas e recomendando claritromicina para tratamento. Finalmente, a amostra S5 teve valores de TC detectáveis em todos os testes, indicando que a amostra é positiva para Helicobacter pylori e exibe dupla resistência à claritromicina e quinolonas; portanto, opções alternativas de tratamento devem ser consideradas pelos médicos. Em comparação com outros métodos de detecção de Helicobacter pylori , essa abordagem não apenas facilita a aquisição de amostras, mas também permite a detecção simultânea de Helicobacter pylori e seu perfil de resistência, fornecendo resultados confiáveis para orientar as decisões de tratamento apropriadas.
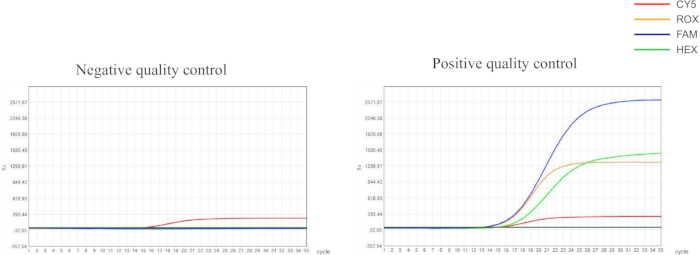
Figura 1: Resultados do controle de qualidade dos controles negativos e positivos. Os controlos negativos devem apresentar uma curva de crescimento em forma de S para o canal de detecção de CY5 com um valor de CT ≤ 30,00; os sinais de fluorescência para os canais FAM, HEX e ROX não devem apresentar aumentos significativos, com valores de TC > 30,00 ou nenhum sinal notável; os controles positivos devem apresentar curvas em forma de S em todos os canais de detecção com valores de TC ≤ 30,00. Clique aqui para ver uma versão maior desta figura.

Figura 2: Detecção de Helicobacter pylori e sua resistência a antibióticos em amostras fecais por qPCR. Cada cor representa um sinal fluorescente e diferentes sinais fluorescentes representam genes diferentes. Canais de fluorescência: marcação fluorescente ROX (laranja) de genes conservados de H. pylori ; Marcação fluorescente FAM (azul) de genes de resistência à claritromicina de H. pylori ; Marcação fluorescente HEX (verde) de genes de resistência a quinolonas de H. pylori ; Marcação fluorescente CY5 (vermelho) do gene de controle interno. Fenótipos de amostra: S1 é uma amostra negativa para infecção por H. pylori (nenhum sinal acima do limite); S2 é uma amostra positiva para H. pylori sem resistência a antibióticos (sinal ROX acima do limiar); S3 é uma amostra positiva para H. pylori com resistência à claritromicina (sinal FAM acima do limiar); S4 é uma amostra positiva para H. pylori com resistência a quinolonas (sinal HEX acima do limiar); S5 é uma amostra positiva para H. pylori resistente à claritromicina e quinolonas (sinal FAM e HEX acima do limiar). Clique aqui para ver uma versão maior desta figura.
| Amostra | H.Pylori (ROX) | Claritromicina (FAM) | Quinolonas (HEX) | CY5 | |||
| +/- | CT | +/- | CT | +/- | CT | CT | |
| S1 | - | - | - | - | - | - | 9.96 |
| S2 | + | 23.08 | - | - | - | - | 12.66 |
| S3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| S4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| negativo | - | - | - | - | - | - | 14.73 |
| Positivo | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
Tabela 1: Resultados da detecção de infecção por H. pylori e qPCR para resistência à claritromicina e quinolona. A tabela fornece os resultados qualitativos da infecção por H. pylori . A detecção de mutações no gene 23S rRNA indica resistência à claritromicina, enquanto as mutações no gene gyrA indicam resistência à quinolona. Símbolos: +/−, resultado qualitativo; +, resultado positivo; −, resultado negativo.
Discussão
Nos últimos anos, os métodos de detecção molecular têm sido amplamente aplicados no campo da microbiologia, alterando significativamente o manejo clínico de diversas doenças infecciosas. Esses métodos operam no nível genético, permitindo não apenas a confirmação da presença bacteriana, mas também a tipagem genética e o teste de resistência a antibióticos. A PCR quantitativa de fluorescência em tempo real (qPCR) é cada vez mais favorecida devido ao seu curto tempo de processamento, alta sensibilidade e precisão e baixo risco de contaminação cruzada. Tornou-se amplamente utilizado em laboratórios clínicos para o diagnóstico de infecções por Helicobacter pylori .
A combinação de soluções de preservação de coleta de amostras fecais com métodos de detecção de genes permite a triagem rápida e eficiente de H. pylori e outros testes de resistência a antibióticos por meio da análise de mutações no gene rRNA da claritromicina 23S e no gene gyrA da quinolona. Este método pode avaliar a sensibilidade e resistência do H. pylori aos medicamentos. Os métodos anteriores de detecção de antígenos fecais têm limitações notáveis, como precisão e estabilidade reduzidas devido a baixas concentrações de antígenos e degradação de antígenos em amostras. Além disso, os métodos tradicionais de detecção de antígenos podem não ter sensibilidade e especificidade suficientes em amostras complexas, afetando a confiabilidade dos resultados.
Para resolver esses problemas, propomos um método que combina amostras fecais com a tecnologia quantitativa de reação em cadeia da polimerase (qPCR). Essa abordagem aumenta significativamente a sensibilidade e a precisão da detecção, tornando os testes fecais mais confiáveis e eficientes e fornecendo resultados de maior qualidade para uso clínico.
Os padrões de controle de qualidade são cruciais para a detecção de qPCR. Nosso controle de qualidade inclui a calibração periódica do instrumento, a verificação do desempenho do sistema de detecção, a avaliação da eficiência da extração de ácidos nucleicos e o controle de qualidade de cada lote de experimentos. Primeiro, nossos instrumentos de amostragem são calibrados a cada 6 meses, e o equipamento de extração e o equipamento de amplificação são calibrados anualmente. Além disso, a calibração é feita para cada lote de reagentes. Após a calibração, podemos validar nosso desempenho para o sistema de detecção, incluindo o instrumento de PCR quantitativo de fluorescência Macrostone 96S, os kits de detecção de H. pylori e mutação de resistência, os reagentes de extração, bem como os tubos de amostragem, pipetas e pontas correspondentes. A taxa de conformidade, o limite de detecção, a reação cruzada, a capacidade anti-interferência e a precisão atendem às nossas necessidades. As concentrações de produtos de DNA foram determinadas por um testador de concentração de DNA para atender aos critérios de aceitação para a eficiência de extração de ácido nucleico. A concentração de DNA estava acima de 10 ng/μL. O A260 / A280 estava entre 1,6 e 2,0. As amostras não qualificadas devem ser extraídas novamente. Os resultados só são válidos quando o controle de qualidade diário está sob controle. Para H. pylori e testes de resistência a antibióticos (claritromicina e quinolonas), os critérios de controle de qualidade são os seguintes: os controles negativos devem exibir uma curva de crescimento em forma de S para o canal de detecção CY5 com um valor de CT ≤ 30,00; os sinais de fluorescência para os canais FAM, HEX e ROX não devem apresentar aumentos significativos, com valores de TC >30,00 ou nenhum sinal notável; os controles positivos devem apresentar curvas em forma de S em todos os canais de detecção com valores de TC ≤ 30,00. Estas condições devem ser satisfeitas no âmbito da mesma experiência; caso contrário, o teste será considerado inválido e deverá ser testado novamente. Observe que, devido a restrições proprietárias comerciais, as informações de sequência de primer e sonda não podem ser fornecidas neste artigo.
Em um estudo anterior, os pesquisadores empregaram uma metodologia idêntica à utilizada neste artigo, demonstrando que o teste de RT-PCR para infecção por H. pylori em amostras fecais exibiu desempenho diagnóstico excepcional24. Especificamente, o teste alcançou uma sensibilidade de 99,1%, especificidade de 100% e precisão diagnóstica de 99,1%, o que se alinhou estreitamente com os resultados das amostras de biópsia gástrica (93,9%) (Kappa = 0,929, p < 0,001). Este acordo notável ressaltou que o teste RT-PCR pode ser considerado um método não invasivo altamente confiável.
Além disso, o estudo também avaliou a capacidade do RT-PCR fecal na detecção de resistência a antibióticos em H. pylori, com foco particular na resistência à claritromicina e levofloxacina. Para a resistência à claritromicina, foram identificados 43 casos (37,3%) em amostras fecais, com sensibilidade de 79,6%, especificidade de 98,4% e acurácia diagnóstica de 78,0% (Kappa = 0,788, p < 0,001). Da mesma forma, para a resistência à levofloxacina, foram detectados 37 casos (32,1%), resultando em sensibilidade de 86,3%, especificidade de 91,1% e acurácia diagnóstica de 74,4% (Kappa = 0,739, p < 0,001). Esses achados foram altamente consistentes com os derivados de amostras de biópsia gástrica (resistência à claritromicina em 54 casos, resistência à levofloxacina em 36 casos), indicando que o teste de RT-PCR fecal é eficaz na detecção de resistência a antibióticos, possuindo alta especificidade e sensibilidade aceitável para ambos os antibióticos. A validação dessa metodologia no estudo anterior reforça ainda mais sua confiabilidade e aplicabilidade no contexto deste artigo.
Embora o protocolo de detecção fecal baseado em qPCR represente um avanço notável no campo do diagnóstico não invasivo para H. pylori, várias limitações exigem consideração meticulosa. Primeiro, o uso de amostras fecais torna o método afetado pela qualidade da amostra. Falsos negativos podem resultar de baixas cargas bacterianas nas fezes, particularmente em portadores assintomáticos ou pacientes com infecções em estágio inicial, onde a excreção de H. pylori no trato gastrointestinal é intermitente25. Além disso, ao coletar amostras fecais, a quantidade de bactérias pode variar dependendo de onde a amostra é coletada. Isso contrasta marcadamente com métodos invasivos, como a cultura de biópsia gástrica, que coletam amostras diretamente da mucosa gástrica e, portanto, são menos influenciados por flutuações na carga bacteriana. Em segundo lugar, embora a abordagem de amplificação direcionada do protocolo seja específica, ela pode falhar em detectar mutações de resistência raras ou emergentes que não são incorporadas ao design do primer, potencialmente levando a perfis de resistência incompletos26.
Pesquisas futuras devem se concentrar em avanços metodológicos para aprimorar os recursos de detecção de mutações, incluindo a adoção de PCR digital (dPCR) para melhorar a identificação de mutações de resistência de baixa abundância27. Os painéis de sonda requerem expansão estratégica para abranger polimorfismos clinicamente relevantes. A realização de estudos multicêntricos para avaliar comparativamente as metodologias de diagnóstico - incluindo UBT, teste de PCR da mucosa gástrica ou fecal e detecção de antígeno - fornece dados abrangentes de desempenho diagnóstico para a prática clínica, avançando assim na padronização dos protocolos de teste de PCR nas fezes. Essas melhorias combinadas fortaleceriam a utilidade da qPCR fecal como uma ferramenta de diagnóstico não invasiva, embora ainda sejam importantes para orientar o tratamento preciso com antibióticos.
Divulgações
Os autores declaram que não têm interesses conflitantes.
Agradecimentos
Este estudo foi financiado pela Fundação de Pesquisa para Talentos Avançados do Hospital Popular da Província de Guangdong [Grant No. KY012023293]. Este trabalho foi apoiado pela Jiangsu Mole Bioscience Co. Os financiadores não tiveram nenhum papel no desenho do estudo, na coleta e análise de dados, na decisão de publicar ou na preparação do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
Referências
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados