Method Article
대변 정량적 중합효소 연쇄 반응 분석을 통한 헬리코박터 파일로리 감염 및 항생제 내성 검출
* 이 저자들은 동등하게 기여했습니다
요약
이 프로토콜은 정량적 중합효소 연쇄 반응과 결합된 비침습적 대변 샘플링을 사용하여 헬리코박터 파일로리 감염 및 클라리스로마이신 및 퀴놀론에 대한 내성에 대한 편리하고 신속한 진단 방법을 제공합니다.
초록
헬리코박터 파일로리(H. pylori)는 전 세계적으로 널리 퍼져 있으며, 전 세계 인구의 약 50%가 헬리코박터 파일로리 감염 병력을 가지고 있습니다 . 중국에서는 감염률이 40%에서 70% 사이입니다. 헬리코박터 파일로리균은 주로 만성 위염, 위궤양, 십이지장 궤양과 같은 위장 질환과 관련이 있습니다. 현재 헬리코박터 파일로리 감염에 대한 임상 치료에는 삼중 또는 사중 요법이 포함됩니다. 그러나 항생제의 광범위한 사용으로 인해 헬리코박터 파일로리균에서 항생제 내성이 생겼습니다. 따라서 헬리코박터 파일로리균과 항생제 내성을 모두 검출하는 것은 임상 치료를 안내하는 데 매우 중요합니다.
헬리코박터 파일로리균의 진단 방법에는 요소 호흡 검사(UBT), 항원 검사, 혈청학적 항체 검사, 내시경 검사, 신속 우레아제 검사(RUT) 및 박테리아 배양이 포함됩니다. 처음 세 가지 방법은 비침습적이지만 박테리아 회수를 허용하지 않으므로 내성 테스트에 사용할 수 없습니다. 후자의 세 가지 방법은 침습적이고, 비용이 많이 들고, 높은 기술적 전문성이 필요하며, 환자에게 해를 끼칠 수 있습니다.
따라서 H. pylori 감염과 항생제 내성을 동시에 검출할 수 있는 비침습적이고 신속한 방법은 H. pylori의 효과적인 박멸을 위해 임상적으로 가장 중요합니다. 본 논문은 정량적 중합효소연쇄반응(qPCR)과 TaqMan 형광 프로브 기술을 결합하여 헬리코박터 파일로리 감염 및 항생제 내성을 신속하게 검출하는 특정 프로토콜을 소개하는 것을 목표로 합니다. 이 방법은 기존의 박테리아 배양 및 기타 기술과 달리 H. pylori 감염 및 내성을 진단하는 편리하고 신속하며 비침습적인 방법을 제공합니다. qPCR은 감염을 확인하고 23S rRNA 및 gyrA 유전자의 돌연변이를 감지하는 데 사용되며, 이는 각각 clarithromycin 및 quinolone에 대한 내성과 관련이 있습니다. 기존 배양 기술과 비교했을 때, 이 접근법은 헬리코박터 파일로리 감염을 검출하고 항생제 내성을 측정하기 위한 비침습적이고 비용 효율적이며 시간 효율적인 방법을 제공합니다.
서문
헬리코박터 파일로리(Helicobacter pylori )는 인간의 위 상피를 지속적으로 감염시킬 수 있는 그람 음성의 나선형 박테리아입니다1. 1994년 세계보건기구(WHO)는 헬리코박터 파일로리 균을 위암의 1군 발암물질로 분류했으며, 감염된 개인의 ~3%가 궁극적으로 이 질병을 발병시켰습니다2. 최근 발표된 체계적 문헌고찰에 따르면 지난 10년 동안 헬리코박터 파일로리 균의 항생제 내성 증가 추세가 관찰되었으며, 이는 전 세계적으로 특히 클래리스로마이신 내성 3에 심각한 수준에 도달했다. 중국에서 최신 설문 조사 데이터에 따르면 중국 도시 사람들 사이에서 H. pylori 의 총 유병률은 27.08%입니다. 클래리스로마이신과 레보플록사신의 내성률은 각각 50.83%와 47.17%4로 미국(레보플록사신 37.6%, 클래리스로마이신 31.5%)5및 유럽(레보플록사신 15.8%, 클라리스로마이신 21.4%)6보다 높았습니다. 따라서 조기에 정확한 진단과 치료가 중요합니다. 그러나 헬리코박터 파일로리 균의 항생제 내성이 증가함에 따라 치료 효과가 현저히 감소하고 있어 진단 및 치료에 대한 지속적인 연구가 시급히 필요하다는 점이 부각되고 있습니다.
헬리코박터 파일로리균 진단법은 침습적 기법과 비침습적 기법으로 분류된다7. 침습적 방법에는 조직병리학적 검사, 신속 우레아제 검사(RUT), 세균 배양 등이 있다8. 조직병리학적 검사는 생검 샘플의 처리와 현미경 관찰에 의존하며, 조직학적 준비와 병리학 전문 지식에 의해 정확성이 제한된다9. RUT는 우레아제의 활성을 통해 H. pylori를 검출하여 요소를 가수분해하여 암모니아를 생성하여 시약의 알칼리성 변화를 일으켜 박테리아9의 존재를 나타냅니다. 간단하고 비용 효율적입니다. "황금 표준"으로 간주되는 박테리아 배양은 H. pylori10의 고유한 생리학적 특성으로 인해 성공률이 50% 미만인 경우가 많습니다.
비침습적 방법에는 13C/14C 요소 호흡 검사(UBT), 혈청학적 진단 및 대변 항원 검사(SAT)가 포함됩니다11. 호흡 검사는 RUT와 유사한 원리로 작동하며, 내쉬는 공기에서 동위원소로 표지된 CO2 를 검출하여 감염을 확인합니다12. 혈청학적 진단은 박테리아 박멸 후에도 지속될 수 있는 항체를 검출하여 치료 효과를 평가하기 어렵게 만든다13. 대변 항원 검사는 대변 샘플에서 헬리코박터 파일로리 특이 항원을 검출하여 위양성 비율이 낮고 활성 감염을 진단하는 정확도가 높은 신뢰할 수 있는 비침습적 방법을 제공합니다14.
각 진단 방법에는 장점과 한계가 있다15. 박테리아 배양 외에도 다른 방법은 항생제 내성을 검출하는 데 어려움을 겪고 있으며, 배양은 낮은 성공률로 인해 어려움을 겪고 있습니다16. 최근에는 qPCR(Real-Time Quantitative PCR)과 같은 분자 진단 방법이 미생물 검출에 널리 적용되고 있습니다17. qPCR은 헬리코박터 파일로리(H. pylori )를 정확하게 검출하고 저항성 유전자 돌연변이를 분석하여 감염에 대한 보다 포괄적인 관점을 제공할 수 있습니다18.
형광 프로브는 qPCR 프로세스 중에 DNA의 타겟 염기서열에 특이적으로 결합하도록 설계되었습니다. 이러한 프로브는 일반적으로 형광 염료로 표지되며, 이 염료는 프로브가 표적에 결합할 때 신호를 방출하고 증폭 중에 중합효소에 의해 절단됩니다. 이를 통해 PCR 프로세스를 실시간으로 측정할 수 있습니다. 형광 프로브는 프로브의 독특한 설계로 인해 특이성이 높은 것으로 알려져 있으며, 이는 표적 DNA 염기서열만 검출하여 비특이적 결합 가능성을 줄이고 분석 특이성을 개선합니다. 따라서 형광 프로브 기술은 대변 샘플에서 헬리코박터 파일로리 DNA와 같은 저농도 표적을 검출하는 데 특히 유용합니다. SYBR Green은 PCR에서 일반적으로 사용되는 DNA 결합 염료이지만 모든 이중 가닥 DNA에 결합할 수 있으며, 이로 인해 비특이적 증폭 및 위양성이 발생할 수 있습니다.
임상 샘플에서 헬리코박터 파일로리균 을 배양하는 것은 항생제 내성 데이터를 직접 제공하지 않습니다. 반면, 형광 프로브 기반 qPCR은 더 빠르고 편리하며 헬리코박터 파일로리 균과 항생제 내성 마커를 동시에 검출할 수 있습니다. 이 방법은 기존 배양 방법보다 더 빠른 결과를 제공할 뿐만 아니라 항생제 내성 유전자를 동시에 검출할 수 있어 검출 효율성과 편의성을 크게 향상시킵니다. 이 방법은 기존의 박테리아 배양 및 기타 기술과 달리 H. pylori 감염 및 내성을 진단하는 편리하고 신속하며 비침습적인 방법을 제공합니다.
헬리코박터 파일로리 항생제 내성에 대해 고려해야 할 두 가지 중요한 문제가 있습니다: i) 유전자형과 표현형의 일관성, ii) 다제내성. 중국에서 실시된 대규모 다기관 연구에서는 클라리스로마이신/레보플록사신의 이중 내성 패턴이 26.1%인 것으로 나타났습니다. Clarithromycin 및 levofloxacin-resistant H pylori 표현형 및 유전자형은 만족스러운 일치를 보였다(카파 계수 = 각각 0.810 및 0.782)19. 따라서 qPCR 방법을 사용하여 H. pylori 감염 및 약물 내성을 검출하는 것이 가능합니다. 대변 항원 검사(SAT) 및 qPCR 방법은 샘플링의 편의성으로 인해 점점 더 많이 사용되고 있습니다. 헬리코박터 파일로리 검출 방법의 민감도, 특이도 및 정확도는 다음과 같습니다: qPCR > UBT > SAT > RUT> CagA IgG > 배양20. 모든 비침습적 방법은 1차 스크리닝에 적합합니다. qPCR은 약물 내성 유전자를 추가로 검출할 수 있기 때문에 qPCR과 배양이 치료 유도에 더 적합할 것입니다.
내시경 이미지(endoscopic images) 21, 디지털 미세유체역학(digital microfluidics)22 및 RPA-CRISPR/Cas12a23과 결합된 인공 지능(AI) 알고리즘과 같은 다른 혁신적인 접근 방식은 이론적으로 우수한 진단 효율성과 유망한 응용 분야 전망을 보여줍니다. 그러나 qPCR은 현재 헬리코박터 파일로리 감염의 진단 및 치료를 위한 우수한 솔루션을 제공할 수 있는 잠재력을 가진 가장 정교한 분자 생물학 기술입니다.
프로토콜
본 연구는 중국 광저우시 남방의과대학교 광동성 인민병원 윤리위원회가 제정한 윤리지침(승인번호: KY2024-445-01)을 준수한다. 이 연구에 사용된 재료(시약, 화학 물질, 장비 및 소프트웨어)에 대한 자세한 정보는 재료 표에서 확인할 수 있습니다.
1. 참가자 선정
- 18세에서 60세 사이의 참가자를 선택하십시오.
- 위염, 소화성 궤양 또는 소화 불량과 같은 증상을 보이는 환자 및 무증상 개인과 같이 헬리코박터 파일로리 감염 검사를 받을 수 있는 참가자를 선택하십시오.
- 지난 한 달 이내에 항생제, 비스무트 함유제(예: 칼륨 비스무트 시트레이트 캡슐, 콜로이드 비스무트 펙틴 캡슐 또는 비스무트-알루미늄 복합 정제) 또는 항균 한약을 사용한 환자 또는 지난 2주 이내에 양성자 펌프 억제제 또는 H2 수용체 길항제(예: 오메프라졸, 판토프라졸, 라베프라졸, 시메티딘 또는 니자티딘)를 사용한 환자. 또한 생리 기간 동안의 여성은 연구에서 제외합니다.
2. 배설물 시료 채취
참고: 참가자에게 지침으로 섹션 2를 제공합니다.
- 샘플링 키트의 외부 포장이 손상되지 않았는지, 유통 기한이 지나지 않았는지 확인하십시오. 키트에서 재료를 제거합니다.
- 변기에 흰색 배설물 수집 용지를 놓고 배변합니다(소변으로 오염되지 않도록 하기 위해).
- 주걱을 사용하여 대략 누에콩 크기(~5g)의 대변 부분을 채취하여 샘플 용기에 옮깁니다.
- 대변 샘플과 함께 샘플링 주걱을 수집 튜브에 넣고 액체 레벨이 튜브에 표시된 샘플링 라인 근처까지 올라가도록 합니다.
- 튜브 캡을 단단히 조입니다. 샘플과 방부제 용액이 완전히 혼합되도록 튜브를 여러 번 뒤집습니다.
- qPCR을 통해 H. pylori 를 식별하고 항생제 내성 프로파일을 평가하기 위해 임상 실험실로 배송될 때까지 샘플을 냉장 보관하십시오.
3. 핵산 추출
참고: 오염을 방지하기 위해 생물 안전 캐비닛 내에서 모든 절차를 완료하십시오.
- 표본 보존 튜브를 뒤집어 철저히 혼합합니다. 대변 검체 용액 1.0mL를 마이크로 원심분리기 튜브에 옮깁니다.
- 원심분리기 튜브를 혼합한 후 80°C의 금속 수조에 10분 동안 넣고 5분에 30초 동안 간헐적으로 혼합합니다. 튜브가 실온으로 냉각되면 10,000× g 에서 5분 동안 원심분리하고 추가 처리를 위해 상층액을 수집합니다.
- 96웰 플레이트를 여러 번 뒤집어 마그네틱 비드를 다시 매달아 놓습니다. 알루미늄 호일 씰을 조심스럽게 제거하고 엎지르지 않도록 흔들리지 않도록 주의하십시오.
- 준비된 시료 200μL(단계 3.2)를 용해 완충액이 포함된 96웰 플레이트의 각 웰에 추가하여 각 웰이 단일 샘플에 해당하도록 합니다. 자동 추출을 위해 96웰 플레이트를 핵산 추출 기기의 지정된 샘플 격실에 놓습니다.
- 남아 있는 모든 검체와 추출된 핵산 샘플은 장기 보존 및 향후 사용을 위해 -20°C에 보관하십시오.
4. qPCR: Helicobacter pylori 핵산 및 clarithromycin 및 quinolone에 대한 내성 돌연변이 검출
- 시약 보관 구역에서 시약을 꺼내 실온에서 해동합니다.
참고: 이 키트는 23S rRNA 유전자(A2143G, A2143C, A2144G) 및 gyrA 유전자(261A, 261G, 260T, 271A, 271T, 272G)의 돌연변이를 포함하여 H. pylori 핵산의 정성적 검출에 사용되었습니다. 프라이머, 프로브, Taq 효소, UNG 효소 및 dNTP를 키트에서 사용할 수 있었습니다. H. pylori와 인간 β-actin의 표적 유전자는 양성 품질 관리에 포함되었습니다. 인간β-actin 유전자는 내부 품질 관리로 선택되었습니다. - 테스트할 샘플 수에 따라 동결건조 시약의 N + 2 사본을 사용하며, 여기서 N은 테스트할 샘플의 수를 나타내고 2는 음성 및 양성 QC를 나타냅니다.
- 동결건조된 분말이 튜브 바닥에 가라앉도록 잠시 원심분리기를 사용합니다. 동결건조된 시약의 뚜껑을 열고(분말이 유출되지 않도록 주의) 테스트할 샘플에서 추출된 핵산의 웰당 25μL와 양성 및 음성 대조군을 추가합니다. 튜브를 단단히 닫으십시오.
- PCR 시약을 8-10초 동안 소용돌이친 다음 기포 형성을 피하기 위해 3-5초 동안 간단히 원심분리합니다.
- 96웰 qPCR 플레이트를 qPCR 기계에 놓습니다. 사이클링 프로그램을 다음과 같이 설정합니다: 먼저, 반응 혼합물(프라이머, 프로브, Taq 중합효소, UNG 효소, dNTP 등 포함)을 42°C 및 95°C(한 사이클의 두 단계)에서 2분 동안 배양합니다. 그런 다음 95 ° C에서 10 초, 65 ° C에서 45 초로 10 회 순환합니다. 마지막으로, 변성, 어닐링 및 확장을 위해 95°C에서 10초, 58°C에서 45초로 35X를 순환합니다.
- 형광 신호 검출 매개변수를 다음과 같이 설정합니다: H. pylori 보존 유전자의 ROX 형광 라벨링; H. pylori clarithromycin 저항성 유전자의 FAM 형광 라벨링; H. pylori quinolone 저항성 유전자의 HEX 형광 표지; 인간 β-액틴 유전자의 CY5 형광 표지는 키트의 내부 대조군으로 사용되었습니다. 58°C에서 데이터를 수집합니다. 반응이 완료되면 향후 분석을 위해 데이터가 저장되었는지 확인합니다.
- 기기가 기준선 임계값을 자동으로 선택하므로 qPCR 전용 소프트웨어를 사용하여 데이터를 분석합니다. 헬리코박터 파일로리 감염 또는 내성의 존재를 포함한 모든 진단 기준을 CT 값 ≤30으로 설정합니다. 특징적인 S자형 곡선을 나타내는지 확인합니다.
결과
대변 샘플에서 헬리코박터 파일로리 감염 및 항생제 내성 검출을 위한 qPCR의 적용
우리는 Helicobacter pylori의 보존된 유전자와 23S rRNA 유전자 및 gyrA 유전자의 점 돌연변이를 기반으로 프라이머와 프로브를 설계했습니다. 이러한 프라이머와 프로브는 서로 다른 형광 염료로 라벨링된 다음 qPCR 검출에 사용되었습니다. qPCR 실험에 대한 품질 관리 결과는 권장 범위 내에 있었으며, 이는 신뢰할 수 있는 검출 결과를 나타냅니다(그림 1). 본 연구에서는 실험 프로토콜의 신뢰성을 특성화하기 위해 5개의 대표 샘플(S1-S5)을 선택했습니다(그림 2 및 표 1). S1은 헬리코박터 파일로리 감염이 없는 검체이고, S2-S5는 약물 내성 프로파일이 다른 검체입니다.
헬리코박터 파일로리 감염에 대해 양성인 샘플의 경우, 샘플 S2는 헬리코박터 파일로리균에 대해서만 검출 범위 내의 CT 값을 보였으며, 이는 이 샘플이 헬리코박터 파일로리균에 대해 양성이며 클라리스로마이신과 퀴놀론에 모두 민감하여 두 약물 중 하나로 치료할 수 있음을 나타냅니다. 샘플 S3는 헬리코박터 파일로리 감염과 클라리스로마이신 내성 모두에 대한 검출 범위 내의 CT 값을 보여주었으며, 퀴놀론 내성에 대해서는 CT 값이 검출되지 않았는데, 이는 샘플 S3가 클라리스로마이신에 내성이 있는 환자의 것임을 시사합니다. 유사하게, 샘플 S4는 헬리코박터 파일로리 감염 및 퀴놀론 내성에 대한 검출 범위 내의 CT 값을 가졌지만, 클라리스로마이신 내성에 대한 CT 값은 검출되지 않았으며, 이는 퀴놀론에 대한 내성을 나타내며 치료를 위해 클라리스로마이신을 권장하는 것이었습니다. 마지막으로, 샘플 S5는 모든 테스트에서 검출 가능한 CT 값을 가졌으며, 이는 샘플이 헬리코박터 파일로리균에 대해 양성이며 클라리스로마이신과 퀴놀론에 대해 이중 내성을 나타낸다는 것을 나타냅니다. 따라서 임상의는 대체 치료 옵션을 고려해야 합니다. 다른 헬리코박터 파일로리 검출 방법과 비교했을 때, 이 접근법은 샘플 수집을 용이하게 할 뿐만 아니라 헬리코박터 파일로리균과 그 내성 프로파일을 동시에 검출할 수 있어 적절한 치료 결정을 안내하는 신뢰할 수 있는 결과를 제공합니다.
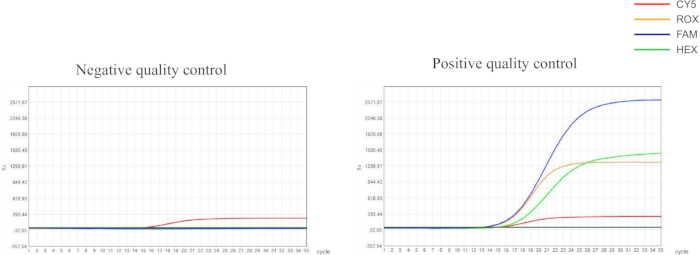
그림 1: negative 및 positive control의 품질 관리 결과. 음성 대조군은 CT 값이 30.00인 CY5 검출 채널에 대한 S자형 성장 곡선≤ 표시해야 합니다. FAM, HEX 및 ROX 채널에 대한 형광 신호는 CT 값이 30.00 >이거나 눈에 띄는 신호가 없는 경우 크게 증가해서는 안 됩니다. 양성 대조군은 모든 검출 채널에서 CT 값이 30.00≤ S자형 곡선을 나타내야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: qPCR에 의한 대변 샘플에서 헬리코박터 파일로리균 및 항생제 내성 검출.각 색상은 형광 신호를 나타내고 다른 형광 신호는 다른 유전자를 나타냅니다. 형광 채널: H. pylori 보존 유전자의 ROX 형광 라벨링(주황색); H. pylori clarithromycin 저항성 유전자의 FAM 형광 표지(파란색); H. pylori 퀴놀론 내성 유전자의 HEX 형광 표지(녹색); 내부 대조 유전자의 CY5 형광 표지(빨간색). 샘플 표현형: S1은 H. pylori 감염에 대한 음성 샘플입니다(임계값을 초과하는 신호 없음). S2는 항생제 내성이 없는 H. 파일로리 양성 샘플입니다(ROX 신호가 임계값 이상). S3는 clarithromycin에 대한 내성이 있는 H. pylori 양성 샘플입니다(임계값 이상의 FAM 신호). S4는 퀴놀론에 대한 내성이 있는 H. pylori 양성 샘플입니다(HEX 신호가 임계값을 초과함). S5는 clarithromycin 및 quinolone(임계값 이상의 FAM 및 HEX 신호)에 모두 내성이 있는 H. pylori 양성 샘플입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 견본 | H.파일로리(ROX) | 클라리스로마이신(FAM) | 퀴놀론(HEX) | 싸이5 | |||
| +/- | 코네티컷 | +/- | 코네티컷 | +/- | 코네티컷 | 코네티컷 | |
| 시즌 1 | - | - | - | - | - | - | 9.96 |
| 시즌 2 | + | 23.08 | - | - | - | - | 12.66 |
| 시즌3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| 시즌 4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| 시즌 5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| 네게티브 | - | - | - | - | - | - | 14.73 |
| 플러스 | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
표 1: 클라리스로마이신 및 퀴놀론 내성에 대한 H. pylori 감염 검출 및 qPCR의 결과.이 표는 헬리코박터 파일로리 감염의 정성적 결과를 제공합니다. 23S rRNA 유전자 돌연변이의 검출은 클래리스로마이신 내성을 나타내고, gyrA 유전자 돌연변이는 퀴놀론 내성을 나타냅니다. 기호: +/-, 정성적 결과; +, 긍정적 인 결과; −, 음의 결과.
토론
최근 몇 년 동안 분자 검출 방법이 미생물학 분야에 광범위하게 적용되어 여러 감염병의 임상 관리를 크게 변화시켰습니다. 이러한 방법은 유전자 수준에서 작동하여 박테리아 존재를 확인할 수 있을 뿐만 아니라 유전자 유형 분석 및 항생제 내성 검사를 수행할 수 있습니다. 실시간 형광 정량 PCR(qPCR)은 처리 시간이 짧고 감도와 정확도가 높으며 교차 오염 위험이 낮기 때문에 점점 더 선호되고 있습니다. 헬리코박터 파일로리 감염 진단을 위해 임상 실험실에서 널리 사용되고 있습니다.
분변 시료 채취 보존 솔루션과 유전자 검출 방법을 결합하면 H. pylori 를 빠르고 효율적으로 스크리닝하고 clarithromycin 23S rRNA 유전자 및 quinolone gyrA 유전자의 돌연변이 분석을 통해 추가 항생제 내성 검사를 수행할 수 있습니다. 이 방법은 H. pylori 민감도와 약물에 대한 내성을 평가할 수 있습니다. 이전의 분변 항원 검출 방법은 낮은 항원 농도 및 샘플의 항원 분해로 인한 정확도 및 안정성 저하와 같은 눈에 띄는 한계가 있었습니다. 또한 기존의 항원 검출 방법은 복잡한 샘플에서 충분한 감도와 특이도가 부족하여 결과의 신뢰성에 영향을 미칠 수 있습니다.
이러한 문제를 해결하기 위해 분변 샘플과 정량적 중합효소연쇄반응(qPCR) 기술을 결합한 방법을 제안합니다. 이 접근 방식은 검출 감도와 정확도를 크게 향상시켜 대변 검사를 보다 신뢰할 수 있고 효율적으로 만들고 임상용으로 더 높은 품질의 결과를 제공합니다.
품질 관리 표준은 qPCR 검출에 매우 중요합니다. 당사의 품질 관리에는 기기의 주기적 교정, 검출 시스템의 성능 검증, 핵산 추출 효율 평가 및 각 실험 배치의 품질 관리가 포함됩니다. 첫째, 당사의 샘플링 기기는 6개월마다 교정되며, 추출 장비 및 증폭 장비는 매년 교정됩니다. 또한 시약의 각 배치에 대해 보정이 수행됩니다. 보정 후 Macrostone 96S 형광 정량 PCR 기기, H. pylori 및 저항성 돌연변이 검출 키트, 추출 시약, 해당 샘플링 튜브, 피펫 및 팁을 포함한 검출 시스템에 대한 성능을 검증할 수 있습니다. 적합성 비율, 검출 한계, 교차 반응, 간섭 방지 기능 및 정밀도는 우리의 요구를 충족합니다. DNA 산물의 농도는 핵산 추출 효율에 대한 허용 기준을 충족하기 위해 DNA 농도 테스터에 의해 결정되었습니다. DNA 농도는 10ng/μL 이상이었습니다. A260/A280은 1.6에서 2.0 사이였습니다. 자격이 없는 샘플은 다시 추출해야 합니다. 결과는 일일 품질 관리가 통제되고 있을 때만 유효합니다. 헬리코박터 파일로리균 및 항생제 내성 검사(클라리스로마이신 및 퀴놀론)의 경우 품질 관리 기준은 다음과 같습니다: 음성 대조군은 CT 값이 30.00≤ CY5 검출 채널에 대해 S자형 성장 곡선을 표시해야 합니다. FAM, HEX 및 ROX 채널에 대한 형광 신호는 CT 값이 >30.00이거나 눈에 띄는 신호가 없는 경우 크게 증가해서는 안 됩니다. 양성 대조군은 모든 검출 채널에서 CT 값이 30.00≤ S자형 곡선을 나타내야 합니다. 이러한 조건은 동일한 실험 내에서 충족되어야 합니다. 그렇지 않으면 테스트가 유효하지 않은 것으로 간주되어 다시 테스트해야 합니다. 상업적 소유권 제약으로 인해 primer 및 probe sequence 정보는 이 문서에서 제공할 수 없습니다.
이전 연구에서 연구자들은 본 논문에 사용된 것과 동일한 방법론을 사용하여 대변 샘플에서 H. pylori 감염에 대한 RT-PCR 검사가 탁월한 진단 성능을 나타냈음을 입증했습니다24. 구체적으로, 이 검사는 민감도 99.1%, 특이도 100%, 진단 정확도 99.1%를 달성했는데, 이는 위 생검 샘플(93.9%)의 결과와 밀접하게 일치했다(Kappa = 0.929, p < 0.001). 이 놀라운 계약은 RT-PCR 테스트가 매우 신뢰할 수 있는 비침습적 방법으로 간주될 수 있음을 강조했습니다.
또한, 이 연구는 특히 클라리스로마이신(clarithromycin)과 레보플록사신(levofloxacin) 내성에 초점을 맞춰 H. pylori에서 항생제 내성을 검출하는 분변 RT-PCR의 능력을 평가했습니다. 클라리스로마이신 내성의 경우, 분변 샘플에서 43건(37.3%)이 확인되었으며, 민감도는 79.6%, 특이도는 98.4%, 진단 정확도는 78.0%였다(Kappa = 0.788, p < 0.001). 마찬가지로, 레보플록사신 내성에 대해서는 37례(32.1%)가 검출되어 민감도 86.3%, 특이도 91.1%, 진단 정확도 74.4%(Kappa = 0.739, p < 0.001)를 보였다. 이러한 결과는 위 생검 샘플에서 파생된 결과(54건의 클라리스로마이신 내성, 36건의 레보플록사신 내성)에서 파생된 결과와 매우 일치했으며, 분변 RT-PCR 검사가 항생제 내성을 검출하는 데 효과적이며 두 항생제에 대해 높은 특이성과 허용 가능한 민감도를 가지고 있음을 나타냅니다. 이전 연구에서 이 방법론에 대한 검증은 이 논문의 맥락에서 신뢰성과 적용 가능성을 더욱 뒷받침합니다.
qPCR을 기반으로 하는 대변 검출 프로토콜은 헬리코박터 파일로리균에 대한 비침습적 진단 분야에서 주목할 만한 발전을 이루었지만, 몇 가지 제한 사항으로 인해 세심한 고려가 필요합니다. 첫째, 배설물 샘플을 사용하면 샘플의 품질에 영향을 받는 방법이 됩니다. 위음성은 대변의 낮은 박테리아 부하로 인해 발생할 수 있으며, 특히 무증상 보균자 또는 초기 감염 환자의 경우 위장관으로 H . pylori 가 간헐적으로 배출됩니다25. 또한 배설물 샘플을 채취할 때 샘플을 채취하는 위치에 따라 박테리아의 양이 달라질 수 있습니다. 이는 위 점막을 직접 샘플링하여 박테리아 부하의 변동에 영향을 덜 받는 위 생검 배양과 같은 침습적 방법과 현저한 대조를 이룹니다. 둘째, 프로토콜의 표적 증폭 접근법이 특이적이기는 하지만, 프라이머 설계에 통합되지 않은 희귀하거나 새로운 내성 돌연변이를 검출하지 못할 수 있으며, 이로 인해 잠재적으로 불완전한 내성 프로파일이 생성될 수 있습니다26.
향후 연구는 저농도 내성 돌연변이의 식별을 개선하기 위한 디지털 PCR(dPCR)의 채택을 포함하여 돌연변이 검출 기능을 향상시키기 위한 방법론적 발전에 초점을 맞춰야 합니다27. 프로브 패널은 임상적으로 관련된 다형성을 포괄하기 위해 전략적 확장이 필요합니다. UBT, 위 점막 또는 분변 PCR 검사, 항원 검출을 포함한 진단 방법론을 비교하기 위한 다기관 연구를 수행함으로써 임상 실습을 위한 포괄적인 진단 성능 데이터를 제공하여 대변 PCR 검사 프로토콜의 표준화를 앞당깁니다. 이러한 개선이 결합되면 비침습적 진단 도구로서 분변 qPCR의 유용성이 강화되는 동시에 정확한 항생제 치료를 안내하는 데 여전히 중요합니다.
공개
저자들은 자신들이 서로 상충하는 이해관계가 없다고 선언한다.
감사의 말
이 연구는 광둥성 인민병원 선진인재연구재단(Research Foundation for Advanced Talents of Guangdong Provincial People's Hospital)의 지원을 받았다[보조금 번호. KY012023293]입니다. 이 작업은 Jiangsu Mole Bioscience Co.의 지원을 받았습니다. 자금 제공자는 연구 설계, 데이터 수집 및 분석, 출판 결정 또는 원고 준비에 아무런 역할도 하지 않았습니다.
자료
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
참고문헌
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유