Method Article
Nachweis einer Helicobacter pylori-Infektion und Antibiotikaresistenz mittels Stuhl-Quantitativer Polymerase-Kettenreaktionsanalyse
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das Protokoll verwendet eine nicht-invasive Stuhlprobenahme in Kombination mit einer quantitativen Polymerase-Kettenreaktion, um eine bequeme und schnelle Diagnosemethode für eine Helicobacter-pylori-Infektion und ihre Resistenz gegen Clarithromycin und Chinolone zu bieten.
Zusammenfassung
Helicobacter pylori (H. pylori) ist weltweit weit verbreitet, wobei etwa 50 % der Weltbevölkerung eine Vorgeschichte von H. pylori-Infektionen haben . In China liegt die Infektionsrate zwischen 40 und 70 Prozent. H. pylori wird hauptsächlich mit Magen-Darm-Erkrankungen wie chronischer Gastritis, Magengeschwüren und Zwölffingerdarmgeschwüren in Verbindung gebracht. Derzeit umfasst die klinische Behandlung einer H. pylori-Infektion entweder eine Dreifach- oder eine Vierfachtherapie. Der umfangreiche Einsatz von Antibiotika hat jedoch zur Entwicklung von Antibiotikaresistenzen bei H. pylori geführt. Daher ist der Nachweis sowohl von H. pylori als auch seiner Antibiotikaresistenz entscheidend für die klinische Behandlung.
Zu den diagnostischen Methoden für H. pylori gehören der Harnstoff-Atemtest (UBT), der Antigentest, der serologische Antikörpertest, die Endoskopie, der Urease-Schnelltest (RUT) und die Bakterienkultur. Die ersten drei Methoden sind zwar nicht-invasiv, ermöglichen jedoch keine bakterielle Erholung und können daher nicht für Resistenztests verwendet werden. Die drei letztgenannten Methoden sind invasiv, teuer, erfordern ein hohes technisches Know-how und können den Patienten schaden.
Daher ist eine nicht-invasive, schnelle Methode zum gleichzeitigen Nachweis einer H. pylori-Infektion und einer Antibiotikaresistenz von größter klinischer Bedeutung für die effektive Eradikation von H. pylori. Ziel dieser Arbeit ist es, ein spezifisches Protokoll vorzustellen, das die quantitative Polymerase-Kettenreaktion (qPCR) mit der TaqMan-Fluoreszenzsondentechnologie kombiniert, um eine H. pylori-Infektion und Antibiotikaresistenz schnell nachzuweisen. Diese Methode bietet im Gegensatz zu herkömmlichen Bakterienkulturen und anderen Techniken eine bequeme, schnelle und nicht-invasive Methode zur Diagnose von H . pylori-Infektionen und -Resistenzen. Die qPCR wird verwendet, um die Infektion zu identifizieren und Mutationen in den Genen 23S rRNA und gyrA nachzuweisen, die mit einer Resistenz gegen Clarithromycin bzw. Chinolone verbunden sind. Im Vergleich zu herkömmlichen Kulturtechniken bietet dieser Ansatz eine nicht-invasive, kostengünstige und zeiteffiziente Methode zum Nachweis einer Helicobacter pylori-Infektion und zur Bestimmung ihrer Antibiotikaresistenz.
Einleitung
Helicobacter pylori ist ein gramnegatives, spiralförmiges Bakterium, das das menschliche Magenepithel persistent infizieren kann1. Im Jahr 1994 stufte die Weltgesundheitsorganisation H. pylori als Karzinogen der Gruppe 1 für Magenkrebs ein, wobei ~3% der infizierten Personen letztendlich die Krankheitentwickelten 2. Eine kürzlich veröffentlichte systematische Übersichtsarbeit hat gezeigt, dass in den letzten zehn Jahren ein allgemeiner Trend zu einer erhöhten Antibiotikaresistenzrate von H. pylori beobachtet wurde, die weltweit ein alarmierendes Niveau erreicht hat, insbesondere die Clarithromycin-Resistenz3. In China zeigten die neuesten Umfragedaten, dass die Gesamtprävalenz von H. pylori unter städtischen Chinesen 27,08 % beträgt; Die Resistenzraten für Clarithromycin und Levofloxacin lagen bei 50,83 % und 47,17 %4 und sind damit höher als in den Vereinigten Staaten (Levofloxacin 37,6 %, Clarithromycin 31,5 %)5und Europa (Levofloxacin 15,8 % und Clarithromycin 21,4 %)6. Daher ist eine frühzeitige und genaue Diagnose und Behandlung von entscheidender Bedeutung. Die zunehmende Antibiotikaresistenz von H. pylori verringert jedoch die Wirksamkeit der Behandlung erheblich, was den dringenden Bedarf an kontinuierlicher Forschung zu Diagnose und Behandlung unterstreicht.
Die diagnostischen Methoden von H. pylori werden in invasive und nicht-invasive Techniken eingeteilt7. Zu den invasiven Methoden gehören die histopathologische Untersuchung, der Urease-Schnelltest (RUT) und die Bakterienkultur8. Die histopathologische Untersuchung beruht auf der Verarbeitung und mikroskopischen Beobachtung von Biopsieproben, wobei die Genauigkeit durch histologische Vorbereitung und pathologische Expertise begrenzt ist9. RUT weist H. pylori durch die Aktivität von Urease nach, die Harnstoff zu Ammoniak hydrolysiert, was zu einer alkalischen Verschiebung des Reagenzes führt, was auf das Vorhandensein des Bakteriumshinweist 9. Es ist einfach und kostengünstig. Bakterienkulturen, die als "Goldstandard" gelten, haben aufgrund der einzigartigen physiologischen Eigenschaften von H. pylori10 oft eine Erfolgsquote von unter 50 %.
Zu den nicht-invasiven Methoden gehören der 13C/14C-Harnstoff-Atemtest (UBT), die serologische Diagnostik und der Stuhlantigen-Test (SAT)11. Der Atemtest funktioniert nach einem ähnlichen Prinzip wie der RUT und weist isotopenmarkiertes CO2 in der Ausatemluft nach, um eine Infektion zu bestätigen12. In der serologischen Diagnose werden Antikörper nachgewiesen, die nach der bakteriellen Eradikation bestehen bleiben können, was die Beurteilung der Wirksamkeit der Behandlung erschwert13. Stuhlantigentests weisen H. pylori-spezifische Antigene in Stuhlproben nach und bieten eine zuverlässige nicht-invasive Methode mit einer geringeren Rate falsch positiver Ergebnisse und einer guten Genauigkeit bei der Diagnose aktiver Infektionen14.
Jedes Diagnoseverfahren hat seine Vor- und Nachteile15. Abgesehen von der Bakterienkultur haben andere Methoden Schwierigkeiten, Antibiotikaresistenzen nachzuweisen, während die Kultur durch niedrige Erfolgsraten behindert wird16. In jüngster Zeit werden molekulardiagnostische Methoden wie die quantitative Echtzeit-PCR (qPCR) in großem Umfang für den mikrobiellen Nachweis eingesetzt17. Die qPCR kann H. pylori genau nachweisen und Resistenzgenmutationen analysieren, was einen umfassenderen Überblick über die Infektion bietet18.
Fluoreszierende Sonden sind so konzipiert, dass sie während des qPCR-Prozesses spezifisch an eine Zielsequenz in der DNA binden. Diese Sonden sind in der Regel mit einem Fluoreszenzfarbstoff markiert, der ein Signal aussendet, wenn die Sonde an ihr Ziel bindet und während der Amplifikation von der Polymerase gespalten wird. Dies ermöglicht eine Echtzeit-Messung des PCR-Prozesses. Fluoreszierende Sonden sind bekannt für ihre hohe Spezifität aufgrund des einzigartigen Designs der Sonde, das sicherstellt, dass nur die Ziel-DNA-Sequenz nachgewiesen wird, wodurch die Wahrscheinlichkeit einer unspezifischen Bindung verringert und die Assay-Spezifität verbessert wird. Dies macht die Fluoreszenzsondentechnologie besonders nützlich für den Nachweis von Zielen mit geringer Abundanz, wie z. B. Helicobacter pylori-DNA in Stuhlproben. Obwohl SYBR Green ein häufig verwendeter DNA-bindender Farbstoff in der PCR ist, kann er an jede doppelsträngige DNA binden, was zu unspezifischer Amplifikation und falsch positiven Ergebnissen führen kann.
Die Kultivierung von Helicobacter pylori aus klinischen Proben liefert keine direkten Daten zur Antibiotikaresistenz. Im Gegensatz dazu ist die auf Fluoreszenzsonden basierende qPCR schneller, bequemer und kann für den gleichzeitigen Nachweis von H. pylori und Antibiotikaresistenzmarkern angepasst werden. Diese Methode liefert nicht nur schnellere Ergebnisse als herkömmliche Kultivierungsmethoden, sondern ermöglicht auch den gleichzeitigen Nachweis von Antibiotikaresistenzgenen, was die Nachweiseffizienz und den Komfort erheblich verbessert. Diese Methode bietet im Gegensatz zu herkömmlichen Bakterienkulturen und anderen Techniken eine bequeme, schnelle und nicht-invasive Methode zur Diagnose von H . pylori-Infektionen und -Resistenzen.
Es gibt zwei entscheidende Aspekte, die bei der Antibiotikaresistenz von H. pylori berücksichtigt werden müssen: i) die Konsistenz von Genotyp und Phänotyp, ii) die Multiresistenz. Eine groß angelegte multizentrische Studie in China ergab, dass die dualen Resistenzmuster für Clarithromycin/Levofloxacin 26,1 % betrugen. Clarithromycin- und Levofloxacin-resistente H-pylori-Phänotypen und -Genotypen zeigten eine zufriedenstellende Übereinstimmung (Kappa-Koeffizient = 0,810 bzw. 0,782)19. Daher ist es möglich, die qPCR-Methode zum Nachweis einer H. pylori-Infektion und Arzneimittelresistenz zu verwenden. Stuhlantigen-Test (SAT) und qPCR-Methoden werden aufgrund der Bequemlichkeit der Probenahme zunehmend eingesetzt. Die Sensitivität, Spezifität und Genauigkeit der Methoden zum Nachweis von H. pylori sind wie folgt: qPCR > UBT > SAT > RUT> CagA IgG > Kultur20. Alle nicht-invasiven Methoden sind für das Primärscreening geeignet. Da qPCR zusätzlich Arzneimittelresistenzgene nachweisen kann, sind qPCR und Kultur besser geeignet, um die Behandlung zu steuern.
Andere innovative Ansätze, wie z.B. Algorithmen der künstlichen Intelligenz (KI) in Kombination mit endoskopischen Bildern21, digitale Mikrofluidik22 und RPA-CRISPR/Cas12a23, zeigen theoretisch eine gute diagnostische Effizienz und vielversprechende Anwendungsperspektiven. Die qPCR ist jedoch derzeit die ausgefeilteste molekularbiologische Technik mit dem Potenzial, eine überlegene Lösung für die Diagnose und Behandlung von H. pylori-Infektionen zu bieten.
Protokoll
Diese Studie hält sich an die ethischen Richtlinien, die von der Ethikkommission des Volkskrankenhauses der Provinz Guangdong der Southern Medical University, Guangzhou, China, festgelegt wurden (Zulassungsnummer: KY2024-445-01). Detaillierte Informationen zu den in dieser Forschung verwendeten Materialien (Reagenzien, Chemikalien, Geräte und Software) finden Sie in der Materialtabelle.
1. Auswahl der Teilnehmer
- Wählen Sie Teilnehmer im Alter von 18 bis 60 Jahren.
- Wählen Sie Teilnehmer aus, die sich einem Helicobacter-pylori-Infektionsscreening unterziehen können, wie z. B. Patienten mit Symptomen wie Gastritis, Magengeschwüren oder Dyspepsie, sowie asymptomatische Personen.
- Schließen Sie Patienten aus der Studie aus, die innerhalb des letzten Monats Antibiotika, Wismut-haltige Mittel (wie Kalium-Wimutcitrat-Kapseln, kolloidale Wismut-Pektin-Kapseln oder Wismut-Aluminium-Verbindungstabletten) oder antimikrobielle chinesische Arzneimittel eingenommen haben, oder Patienten, die innerhalb der letzten 2 Wochen Protonenpumpenhemmer oder H2-Rezeptorantagonisten (wie Omeprazol, Pantoprazol, Rabeprazol, Cimetidin oder Nizatidin) eingenommen haben. Schließen Sie auch Frauen während ihrer Menstruation von der Studie aus.
2. Entnahme von Kotproben
HINWEIS: Geben Sie den Teilnehmern Abschnitt 2 als Anweisungen.
- Vergewissern Sie sich, dass die Außenverpackung des Probenahmekits unbeschädigt ist und das Verfallsdatum abgelaufen ist. Entfernen Sie die Materialien aus dem Kit.
- Legen Sie ein weißes Kotauffangpapier in die Toilettenschüssel und entleeren Sie den Stuhl (um eine Kontamination mit Urin zu vermeiden).
- Mit einem Spatel eine Portion Kot in der Größe einer Saubohne (~5 g) auffangen und in den Probenbehälter umfüllen.
- Legen Sie den Probenahmespatel zusammen mit der Kotprobe in das Entnahmeröhrchen und stellen Sie sicher, dass der Flüssigkeitsstand in die Nähe der auf dem Röhrchen angegebenen Probenahmelinie ansteigt.
- Ziehen Sie die Tubenkappe fest an. Drehen Sie das Röhrchen mehrmals um, um eine gründliche Durchmischung der Probe mit der Konservierungslösung zu gewährleisten.
- Lagern Sie die Probe bis zur Übergabe an das klinische Labor im Kühlschrank, um die anschließende Identifizierung von H. pylori und die Bewertung von Antibiotikaresistenzprofilen mittels qPCR durchzuführen.
3. Nukleinsäure-Extraktion
HINWEIS: Führen Sie alle Verfahren in einer Biosicherheitswerkbank durch, um eine Kontamination zu vermeiden.
- Drehen Sie das Konservierungsröhrchen um und mischen Sie es gründlich. Übertragen Sie 1,0 mL der Stuhlprobenlösung in ein Mikrozentrifugenröhrchen.
- Nach dem Mischen werden die Zentrifugenröhrchen 10 Minuten lang bei 80 °C in ein Metallbad gelegt, wobei in der fünften Minute 30 s lang intermittierend gemischt wird. Sobald die Röhrchen auf Raumtemperatur abgekühlt sind, zentrifugieren Sie sie 5 Minuten lang bei 10.000 × g und sammeln den Überstand für die weitere Verarbeitung.
- Drehen Sie die 96-Well-Platte mehrmals um, um die magnetischen Beads wieder zu resuspendieren. Entfernen Sie vorsichtig die Versiegelung der Aluminiumfolie und achten Sie darauf, dass sie nicht wackelt, um ein Verschütten zu verhindern.
- 200 μl der vorbereiteten Probe (aus Schritt 3.2) in jede Vertiefung der 96-Well-Platte mit Lysepuffer geben, wobei darauf zu achten ist, dass jede Vertiefung einer einzelnen Probe entspricht. Setzen Sie die 96-Well-Platte zur automatischen Extraktion in das dafür vorgesehene Probenfach des Nukleinsäure-Extraktionsgeräts ein.
- Lagern Sie alle verbleibenden Proben und die extrahierten Nukleinsäureproben bei -20 °C für die Langzeitkonservierung und zukünftige Verwendung.
4. qPCR: Nachweis von Helicobacter pylori-Nukleinsäuren und Resistenzmutationen gegen Clarithromycin und Chinolone
- Entnehmen Sie die Reagenzien aus dem Reagenzienlager und tauen Sie sie bei Raumtemperatur auf.
HINWEIS: Dieses Kit wurde für den qualitativen Nachweis von H. pylori-Nukleinsäuren verwendet, einschließlich Mutationen im 23S rRNA-Gen (A2143G, A2143C, A2144G) und im gyrA-Gen (261A, 261G, 260T, 271A, 271T, 272G). Die Primer, die Sonden, das Taq-Enzym, das UNG-Enzym und die dNTPs waren im Kit verfügbar. Die Zielgene von H. pylori und humanem β-Aktin wurden in die positive Qualitätskontrolle einbezogen. Als interne Qualitätskontrolle wurde das humane β-Aktin-Gen gewählt. - Basierend auf der Anzahl der zu testenden Proben verwenden Sie N + 2 Kopien des lyophilisierten Reagenzes, wobei N für die Anzahl der zu testenden Proben und 2 für die negativen und positiven QCs steht.
- Kurz zentrifugieren, um sicherzustellen, dass sich das lyophilisierte Pulver am Boden der Röhrchen absetzt. Öffnen Sie den Deckel des lyophilisierten Reagenzes (achten Sie darauf, dass das Pulver nicht verschüttet wird) und fügen Sie 25 μl der extrahierten Nukleinsäuren aus den zu testenden Proben sowie der Positiv- und Negativkontrolle pro Vertiefung hinzu. Verschließen Sie die Tuben fest.
- Die PCR-Reagenzien 8-10 s lang vortexen, dann 3-5 s kurz zentrifugieren, um Blasenbildung zu vermeiden.
- Setzen Sie die 96-Well-qPCR-Platte auf das qPCR-Gerät ein. Stellen Sie das Zyklusprogramm wie folgt ein: Inkubieren Sie zuerst das Reaktionsgemisch (mit Primern, Sonden, Taq-Polymerase, UNG-Enzym, dNTPs usw.) bei 42 °C und 95 °C (beide Schritte in einem Zyklus) für 2 Minuten; dann 10x mit 10 s bei 95 °C und 45 s bei 65 °C durchdrehen; Zum Schluss wird der 35-fache Zyklus mit 10 s bei 95 °C und 45 s bei 58 °C zum Denaturieren, Glühen und Verlängern durchgeführt.
- Stellen Sie die Parameter für die Detektion von Fluoreszenzsignalen wie folgt ein: ROX-Fluoreszenzmarkierung von H. pylori-konservierten Genen; FAM-Fluoreszenzmarkierung von H. pylori-Clarithromycin-Resistenzgenen; HEX-Fluoreszenzmarkierung von H. pylori-Chinolon-Resistenzgenen; und die CY5-Fluoreszenzmarkierung des menschlichen β-Aktin-Gens wurden als interne Kontrolle des Kits verwendet. Erfassen Sie Daten bei 58 °C. Stellen Sie nach Abschluss der Reaktion sicher, dass die Daten für zukünftige Analysen gespeichert werden.
- Analysieren Sie die Daten mit qPCR-spezifischer Software, da das Gerät automatisch den Ausgangsschwellenwert auswählt. Alle diagnostischen Kriterien, einschließlich des Vorhandenseins einer Helicobacter-pylori-Infektion oder -Resistenz, werden auf einen CT-Wert ≤30 festgelegt. bestätigen, dass sie eine charakteristische S-förmige Kurve aufweisen.
Ergebnisse
Anwendung der qPCR zum Nachweis von Helicobacter pylori-Infektionen und Antibiotikaresistenzen in Stuhlproben
Wir haben Primer und Sonden entwickelt, die auf Punktmutationen in den konservierten Genen von Helicobacter pylori sowie im 23S rRNA-Gen und im gyrA-Gen basieren. Diese Primer und Sonden wurden mit verschiedenen Fluoreszenzfarbstoffen markiert und dann für den qPCR-Nachweis verwendet. Die Ergebnisse der Qualitätskontrolle für die qPCR-Experimente lagen im empfohlenen Bereich, was auf zuverlässige Nachweisergebnisse hinweist (Abbildung 1). In dieser Studie wählten wir fünf repräsentative Stichproben (S1-S5) aus, um die Zuverlässigkeit des experimentellen Protokolls zu charakterisieren (Abbildung 2 und Tabelle 1). S1 ist eine Probe ohne H. pylori-Infektion , während S2-S5 Proben mit unterschiedlichen Arzneimittelresistenzprofilen sind.
Bei Proben, die positiv auf eine Helicobacter pylori-Infektion getestet wurden, zeigte Probe S2 CT-Werte innerhalb des Nachweisbereichs nur für Helicobacter pylori, was darauf hindeutet, dass diese Probe positiv für Helicobacter pylori ist und sowohl auf Clarithromycin als auch auf Chinolone reagiert, was eine Behandlung mit beiden Arzneimitteln ermöglicht. Probe S3 zeigte CT-Werte innerhalb des Nachweisbereichs sowohl für eine Helicobacter pylori-Infektion als auch für eine Clarithromycin-Resistenz, wobei keine CT-Werte für Chinolon-Resistenz festgestellt wurden, was darauf hindeutet, dass Probe S3 von einem Patienten stammt, der gegen Clarithromycin resistent ist. In ähnlicher Weise wies Probe S4 CT-Werte innerhalb des Nachweisbereichs für Helicobacter pylori-Infektion und Chinolon-Resistenz auf, wobei keine CT-Werte für Clarithromycin-Resistenz festgestellt wurden, was auf eine Resistenz gegen Chinolone hinweist und Clarithromycin zur Behandlung empfiehlt. Schließlich wies Probe S5 in allen Tests nachweisbare CT-Werte auf, was darauf hindeutet, dass die Probe positiv für Helicobacter pylori ist und eine duale Resistenz sowohl gegen Clarithromycin als auch gegen Chinolone aufweist; Daher sollten alternative Behandlungsmöglichkeiten von Klinikern in Betracht gezogen werden. Im Vergleich zu anderen Helicobacter pylori-Nachweismethoden erleichtert dieser Ansatz nicht nur die Probenentnahme, sondern ermöglicht auch den gleichzeitigen Nachweis von Helicobacter pylori und seines Resistenzprofils, was zuverlässige Ergebnisse liefert, die geeignete Behandlungsentscheidungen treffen.
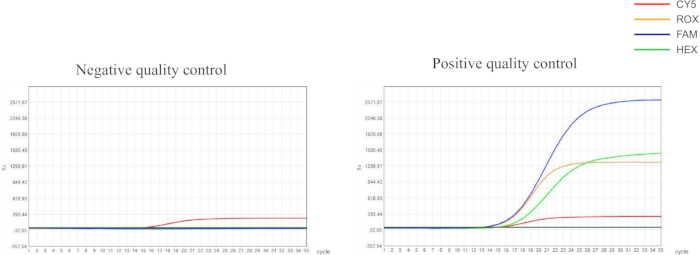
Abbildung 1: Ergebnisse der Qualitätskontrolle von Negativ- und Positivkontrollen. Negativkontrollen sollten eine S-förmige Wachstumskurve für den Nachweiskanal CY5 mit einem CT-Wert ≤ 30,00 anzeigen; Die Fluoreszenzsignale für FAM-, HEX- und ROX-Kanäle sollten keine signifikanten Erhöhungen aufweisen, wobei die CT-Werte > 30,00 oder kein nennenswertes Signal vorhanden sind. Positivkontrollen sollten in allen Detektionskanälen S-förmige Kurven mit CT-Werten ≤ 30,00 aufweisen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Nachweis von Helicobacter pylori und seiner Antibiotikaresistenz in Stuhlproben mittels qPCR. Jede Farbe steht für ein Fluoreszenzsignal, und unterschiedliche Fluoreszenzsignale stehen für unterschiedliche Gene. Fluoreszenzkanäle: ROX-Fluoreszenzmarkierung (orange) von H. pylori-konservierten Genen; FAM-Fluoreszenzmarkierung (blau) von H. pylori-Clarithromycin-Resistenzgenen ; HEX-Fluoreszenzmarkierung (grün) von H. pylori-Chinolon-Resistenzgenen ; CY5-Fluoreszenzmarkierung (rot) des internen Kontrollgens. Probenphänotypen: S1 ist eine negative Probe für eine H. pylori-Infektion (kein Signal über dem Schwellenwert); S2 ist eine H. pylori-positive Probe ohne Antibiotikaresistenz (ROX-Signal über dem Schwellenwert); S3 ist eine H. pylori-positive Probe mit Resistenz gegen Clarithromycin (FAM-Signal über dem Schwellenwert); S4 ist eine H. pylori-positive Probe mit Resistenz gegen Chinolone (HEX-Signal über dem Schwellenwert); S5 ist eine H. pylori-positive Probe, die sowohl gegen Clarithromycin als auch gegen Chinolone resistent ist (FAM- und HEX-Signal über dem Schwellenwert). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Probe | H.Pylori (ROX) | Clarithromycin (FAM) | Chinolone (HEX) | CY5 | |||
| +/- | CT | +/- | CT | +/- | CT | CT | |
| Nr. S1 | - | - | - | - | - | - | 9.96 |
| S2 | + | 23.08 | - | - | - | - | 12.66 |
| S3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| S4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| Negetiv | - | - | - | - | - | - | 14.73 |
| Positiv | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
Tabelle 1: Ergebnisse des Nachweises einer H. pylori-Infektion und der qPCR für Clarithromycin- und Chinolon-Resistenz. Die Tabelle enthält die qualitativen Ergebnisse der H. pylori-Infektion. Der Nachweis von Mutationen des 23S rRNA-Gens deutet auf eine Clarithromycin-Resistenz hin, während Mutationen des gyrA-Gens auf eine Chinolon-Resistenz hinweisen. Symbole: +/−, qualitatives Ergebnis; +, positives Ergebnis; −, negatives Ergebnis.
Diskussion
In den letzten Jahren wurden molekulare Nachweismethoden in großem Umfang auf dem Gebiet der Mikrobiologie eingesetzt, wodurch sich die klinische Behandlung mehrerer Infektionskrankheiten erheblich verändert hat. Diese Methoden arbeiten auf genetischer Ebene und ermöglichen nicht nur die Bestätigung des Vorhandenseins von Bakterien, sondern auch die Gentypisierung und Antibiotikaresistenztests. Die quantitative Real-Time-Fluoreszenz-PCR (qPCR) wird aufgrund ihrer kurzen Verarbeitungszeit, ihrer hohen Empfindlichkeit und Genauigkeit sowie ihres geringen Risikos einer Kreuzkontamination zunehmend bevorzugt. Es wird in klinischen Laboratorien häufig zur Diagnose von Helicobacter-pylori-Infektionen eingesetzt.
Die Kombination von Lösungen zur Konservierung von Stuhlproben mit Gennachweismethoden ermöglicht ein schnelles und effizientes Screening von H. pylori und weitere Antibiotikaresistenztests durch die Analyse von Mutationen im Clarithromycin-23S-rRNA-Gen und im Chinolon-gyrA-Gen. Mit dieser Methode kann die Empfindlichkeit und Resistenz von H. pylori gegenüber Arzneimitteln beurteilt werden. Bisherige Methoden zum Nachweis von fäkalen Antigenen weisen bemerkenswerte Einschränkungen auf, wie z. B. eine verringerte Genauigkeit und Stabilität aufgrund niedriger Antigenkonzentrationen und des Antigenabbaus in Proben. Darüber hinaus kann es bei herkömmlichen Antigennachweismethoden zu einer ausreichenden Sensitivität und Spezifität in komplexen Proben kommen, was die Zuverlässigkeit der Ergebnisse beeinträchtigt.
Um diese Probleme zu lösen, schlagen wir eine Methode vor, die Stuhlproben mit der quantitativen Polymerase-Kettenreaktion (qPCR)-Technologie kombiniert. Dieser Ansatz verbessert die Nachweisempfindlichkeit und -genauigkeit erheblich, macht Stuhltests zuverlässiger und effizienter und liefert qualitativ hochwertigere Ergebnisse für den klinischen Einsatz.
Qualitätskontrollstandards sind für den qPCR-Nachweis von entscheidender Bedeutung. Unsere Qualitätskontrolle umfasst die periodische Kalibrierung des Instruments, die Leistungsüberprüfung des Nachweissystems, die Bewertung der Effizienz der Nukleinsäureextraktion und die Qualitätskontrolle jeder Versuchscharge. Zunächst werden unsere Probenahmegeräte alle 6 Monate kalibriert, und die Extraktionsgeräte und Amplifikationsgeräte werden jährlich kalibriert. Zusätzlich wird für jede Charge von Reagenzien eine Kalibrierung durchgeführt. Nach der Kalibrierung können wir unsere Leistung für das Detektionssystem validieren, einschließlich des quantitativen Fluoreszenz-PCR-Instruments Macrostone 96S, der H . pylori - und Resistenzmutations-Detektionskits, der Extraktionsreagenzien sowie der entsprechenden Probenahmeröhrchen, Pipetten und Spitzen. Die Konformitätsrate, die Nachweisgrenze, die Kreuzreaktion, die Entstörungsfähigkeit und die Präzision erfüllen unsere Anforderungen. Die Konzentrationen der DNA-Produkte wurden von einem DNA-Konzentrationstester bestimmt, um die Akzeptanzkriterien für die Effizienz der Nukleinsäureextraktion zu erfüllen. Die DNA-Konzentration lag bei über 10 ng/μL. Die A260/A280 lag zwischen 1,6 und 2,0. Nicht qualifizierte Proben müssen erneut entnommen werden. Die Ergebnisse sind nur gültig, wenn die tägliche Qualitätskontrolle unter Kontrolle ist. Für H . pylori und Antibiotikaresistenztests (Clarithromycin und Chinolone) gelten folgende Qualitätskontrollkriterien: Negativkontrollen sollten eine S-förmige Wachstumskurve für den Nachweiskanal CY5 mit einem CT-Wert ≤ 30,00 aufweisen; Fluoreszenzsignale für FAM-, HEX- und ROX-Kanäle sollten keine signifikanten Erhöhungen aufweisen, mit CT-Werten >30,00 oder keinem nennenswerten Signal; Positivkontrollen sollten in allen Detektionskanälen S-förmige Kurven mit CT-Werten ≤ 30,00 aufweisen. Diese Bedingungen müssen innerhalb desselben Versuchs erfüllt sein; Andernfalls wird der Test als ungültig betrachtet und muss erneut getestet werden. Beachten Sie, dass aufgrund kommerzieller proprietärer Einschränkungen in diesem Artikel keine Informationen zu Primer- und Sondensequenzen bereitgestellt werden können.
In einer früheren Studie verwendeten die Forscher eine Methodik, die mit der in dieser Arbeit verwendeten identisch ist, und zeigten, dass RT-PCR-Tests auf H . pylori-Infektionen in Stuhlproben eine außergewöhnliche diagnostische Leistung zeigten24. Konkret erreichte der Test eine Sensitivität von 99,1 %, eine Spezifität von 100 % und eine diagnostische Genauigkeit von 99,1 %, was eng mit den Ergebnissen von Magenbiopsieproben (93,9 %) übereinstimmte (Kappa = 0,929, p < 0,001). Diese bemerkenswerte Übereinstimmung unterstreicht, dass die RT-PCR-Testung als eine äußerst zuverlässige nicht-invasive Methode angesehen werden kann.
Darüber hinaus untersuchte die Studie auch die Fähigkeit der fäkalen RT-PCR zum Nachweis von Antibiotikaresistenz bei H. pylori, wobei der Schwerpunkt auf Clarithromycin- und Levofloxacin-Resistenz lag. Für die Clarithromycin-Resistenz wurden 43 Fälle (37,3%) in Stuhlproben mit einer Sensitivität von 79,6%, einer Spezifität von 98,4% und einer diagnostischen Genauigkeit von 78,0% identifiziert (Kappa = 0,788, p < 0,001). In ähnlicher Weise wurden für die Levofloxacin-Resistenz 37 Fälle (32,1 %) nachgewiesen, was zu einer Sensitivität von 86,3 %, einer Spezifität von 91,1 % und einer diagnostischen Genauigkeit von 74,4 % führte (Kappa = 0,739, p < 0,001). Diese Befunde stimmten weitgehend mit denen aus Magenbiopsieproben überein (Clarithromycin-Resistenz in 54 Fällen, Levofloxacin-Resistenz in 36 Fällen), was darauf hindeutet, dass fäkale RT-PCR-Tests beim Nachweis von Antibiotikaresistenzen wirksam sind und eine hohe Spezifität und akzeptable Sensitivität für beide Antibiotika aufweisen. Die Validierung dieser Methodik in der vorangegangenen Studie unterstützt ihre Zuverlässigkeit und Anwendbarkeit im Rahmen dieser Arbeit weiter.
Während das auf qPCR basierende Stuhlnachweisprotokoll einen bemerkenswerten Fortschritt auf dem Gebiet der nicht-invasiven Diagnostik für H. pylori darstellt, erfordern einige Einschränkungen eine sorgfältige Abwägung. Erstens wirkt sich die Verwendung von Stuhlproben auf die Probenqualität aus. Falsch negative Ergebnisse können aus einer geringen bakteriellen Belastung des Stuhls resultieren, insbesondere bei asymptomatischen Trägern oder Patienten mit Infektionen im Frühstadium, bei denen die Ausscheidung von H. pylori in den Gastrointestinaltrakt intermittierend ist25. Darüber hinaus kann bei der Entnahme von Stuhlproben die Menge an Bakterien variieren, je nachdem, wo die Probe entnommen wird. Dies steht im deutlichen Gegensatz zu invasiven Methoden, wie z.B. der Magenbiopsiekultur, bei der direkt die Magenschleimhaut entnommen wird und somit weniger von Schwankungen der Bakterienlast beeinflusst wird. Zweitens kann der gezielte Amplifikationsansatz des Protokolls, obwohl er spezifisch ist, seltene oder neu auftretende Resistenzmutationen, die nicht in das Primer-Design integriert sind, möglicherweise nicht erkennen, was möglicherweise zu unvollständigen Resistenzprofilen führt26.
Zukünftige Forschung sollte sich auf methodische Fortschritte konzentrieren, um die Fähigkeiten zum Nachweis von Mutationen zu verbessern, einschließlich der Einführung der digitalen PCR (dPCR) zur verbesserten Identifizierung von Resistenzmutationen mit geringer Abundanz27. Sonden-Panels müssen strategisch erweitert werden, um klinisch relevante Polymorphismen einzubeziehen. Die Durchführung multizentrischer Studien zur vergleichenden Bewertung diagnostischer Methoden – einschließlich UBT, PCR-Tests der Magenschleimhaut oder des Stuhls und Antigennachweise – liefert umfassende diagnostische Leistungsdaten für die klinische Praxis und treibt so die Standardisierung von PCR-Testprotokollen im Stuhl voran. Diese kombinierten Verbesserungen würden die Nützlichkeit der fäkalen qPCR als nicht-invasives Diagnoseinstrument stärken und gleichzeitig weiterhin wichtig für eine präzise Antibiotikabehandlung sein.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden Interessen haben.
Danksagungen
Diese Studie wurde von der Research Foundation for Advanced Talents des Guangdong Provincial People's Hospital finanziert [Grant No. KY012023293]. Diese Arbeit wurde von Jiangsu Mole Bioscience Co. unterstützt. Die Geldgeber spielten keine Rolle beim Studiendesign, bei der Datenerhebung und -analyse, bei der Entscheidung über die Veröffentlichung oder bei der Erstellung des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
Referenzen
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten