Method Article
Detección de infección por Helicobacter pylori y resistencia a antibióticos mediante análisis cuantitativo de la reacción en cadena de la polimerasa en heces
* Estos autores han contribuido por igual
En este artículo
Resumen
El protocolo emplea una toma de muestras de heces no invasiva combinada con una reacción en cadena de la polimerasa cuantitativa para ofrecer un método de diagnóstico conveniente y rápido para la infección por Helicobacter pylori y su resistencia a la claritromicina y las quinolonas.
Resumen
Helicobacter pylori (H. pylori) es ampliamente prevalente en todo el mundo, con aproximadamente el 50% de la población mundial con antecedentes de infección por H. pylori. En China, la tasa de infección oscila entre el 40% y el 70%. H. pylori se asocia principalmente con enfermedades gastrointestinales como gastritis crónica, úlceras gástricas y úlceras duodenales. En la actualidad, el tratamiento clínico para la infección por H. pylori consiste en una terapia triple o cuádruple. Sin embargo, el uso extensivo de antibióticos ha llevado al desarrollo de resistencia a los antibióticos en H. pylori. Por lo tanto, la detección tanto de H. pylori como de su resistencia a los antibióticos es crucial para guiar el tratamiento clínico.
Los métodos de diagnóstico para H. pylori incluyen la prueba de aliento con urea (UBT), la prueba de antígenos, la prueba de anticuerpos serológicos, la endoscopia, la prueba rápida de ureasa (RUT) y el cultivo bacteriano. Si bien los tres primeros métodos no son invasivos, no permiten la recuperación bacteriana y, por lo tanto, no se pueden usar para pruebas de resistencia. Los tres últimos métodos son invasivos, costosos, requieren una gran experiencia técnica y pueden causar daño a los pacientes.
Por lo tanto, un método rápido y no invasivo para la detección simultánea de la infección por H. pylori y la resistencia a los antibióticos es de suma importancia clínica para la erradicación efectiva de H. pylori. Este artículo tiene como objetivo presentar un protocolo específico que combina la reacción en cadena de la polimerasa cuantitativa (qPCR) con la tecnología de sonda fluorescente TaqMan para detectar rápidamente la infección por H. pylori y la resistencia a los antibióticos. Este método proporciona una forma conveniente, rápida y no invasiva de diagnosticar la infección y la resistencia a H. pylori , a diferencia del cultivo bacteriano tradicional y otras técnicas. La qPCR se utiliza para identificar la infección y detectar mutaciones en los genes 23S rRNA y gyrA , que están relacionados con la resistencia a la claritromicina y las quinolonas, respectivamente. En comparación con las técnicas de cultivo convencionales, este enfoque ofrece un método no invasivo, rentable y eficiente en el tiempo para detectar la infección por Helicobacter pylori y determinar su resistencia a los antibióticos.
Introducción
Helicobacter pylori es una bacteria gramnegativa en forma de espiral que puede infectar de forma persistente el epitelio gástrico humano1. En 1994, la Organización Mundial de la Salud clasificó a H. pylori como un carcinógeno del Grupo 1 para el cáncer gástrico, con ~ 3% de los individuos infectados que finalmente desarrollaron la enfermedad2. Una revisión sistemática recientemente publicada ha indicado que en la última década se observó una tendencia general de aumento de la tasa de resistencia a antibióticos de H. pylori , que ha alcanzado niveles alarmantes en todo el mundo, especialmente la resistencia a la claritromicina3. En China, los datos de la última encuesta mostraron que la prevalencia agregada de H. pylori entre los chinos urbanos es del 27,08%; las tasas de resistencia para claritromicina y levofloxacino fueron del 50,83% y 47,17%4, que son superiores a las de Estados Unidos (levofloxacino 37,6%, claritromicina 31,5%)5y Europa (levofloxacino 15,8% y claritromicina 21,4%)6. En consecuencia, el diagnóstico y el tratamiento tempranos y precisos son cruciales. Sin embargo, el aumento de la resistencia a los antibióticos de H. pylori reduce considerablemente la eficacia del tratamiento, lo que pone de manifiesto la necesidad urgente de realizar investigaciones continuas sobre su diagnóstico y tratamiento.
Los métodos diagnósticos de H. pylori se clasifican en técnicas invasivas y no invasivas7. Los métodos invasivos incluyen el examen histopatológico, la prueba rápida de ureasa (RUT) y el cultivo bacteriano8. El examen histopatológico se basa en el procesamiento y la observación microscópica de las muestras de biopsia, con una precisión limitada por la preparación histológica y la experiencia en patología9. RUT detecta H. pylori a través de la actividad de la ureasa, que hidroliza la urea para producir amoníaco, lo que resulta en un cambio alcalino en el reactivo, lo que indica la presencia de la bacteria9. Es simple y rentable. El cultivo bacteriano, considerado el "estándar de oro", a menudo tiene una tasa de éxito inferior al 50% debido a las características fisiológicas únicas de H. pylori10.
Los métodos no invasivos incluyen la prueba de aliento con urea 13C/14C (UBT), el diagnóstico serológico y la prueba de antígeno en heces (SAT)11. La prueba de aliento funciona según un principio similar al del RUT, detectando CO2 marcado isotópicamente en el aire exhalado para confirmar la infección12. El diagnóstico serológico detecta anticuerpos, que pueden persistir después de la erradicación bacteriana, lo que dificulta la evaluación de la efectividad del tratamiento13. Las pruebas de antígenos en heces detectan antígenos específicos de H. pylori en muestras fecales, proporcionando un método no invasivo confiable con una tasa más baja de falsos positivos y una buena precisión para el diagnóstico de infecciones activas14.
Cada método diagnóstico tiene sus ventajas y limitaciones15. Aparte del cultivo bacteriano, otros métodos tienen dificultades para detectar la resistencia a los antibióticos, mientras que el cultivo se ve obstaculizado por las bajas tasas de éxito16. Recientemente, los métodos de diagnóstico molecular, como la PCR cuantitativa en tiempo real (qPCR), se han aplicado ampliamente a la detección microbiana17. La qPCR puede detectar con precisión H. pylori y analizar las mutaciones de los genes de resistencia, ofreciendo una visión más completa de la infección18.
Las sondas fluorescentes están diseñadas para unirse específicamente a una secuencia objetivo en el ADN durante el proceso de qPCR. Estas sondas generalmente se marcan con un tinte fluorescente, que emite una señal cuando la sonda se une a su objetivo y es escindida por la polimerasa durante la amplificación. Esto proporciona una medición en tiempo real del proceso de PCR. Las sondas fluorescentes son conocidas por su alta especificidad debido al diseño único de la sonda, que garantiza que solo se detecte la secuencia de ADN objetivo, lo que reduce las posibilidades de unión no específica y mejora la especificidad del ensayo. Esto hace que la tecnología de sonda fluorescente sea especialmente útil para detectar objetivos de baja abundancia, como el ADN de Helicobacter pylori en muestras de heces. Si bien SYBR Green es un colorante de unión al ADN comúnmente utilizado en PCR, puede unirse a cualquier ADN bicatenario, lo que puede resultar en una amplificación inespecífica y falsos positivos.
El cultivo de Helicobacter pylori a partir de muestras clínicas no proporciona datos directos sobre la resistencia a los antibióticos. Por el contrario, la qPCR basada en sonda fluorescente es más rápida, más cómoda y puede adaptarse para la detección simultánea de H. pylori y marcadores de resistencia a los antibióticos. Este método no solo ofrece resultados más rápidos que los métodos de cultivo tradicionales, sino que también permite la detección simultánea de genes de resistencia a los antibióticos, lo que mejora en gran medida la eficiencia y la comodidad de la detección. Este método proporciona una forma conveniente, rápida y no invasiva de diagnosticar la infección y la resistencia a H. pylori , a diferencia del cultivo bacteriano tradicional y otras técnicas.
Hay dos cuestiones cruciales que deben tenerse en cuenta para la resistencia a los antibióticos de H. pylori : i) la consistencia del genotipo y el fenotipo, ii) la resistencia a múltiples fármacos. Un estudio multicéntrico a gran escala en China encontró que los patrones de doble resistencia para claritromicina/levofloxacino eran del 26,1%. Los fenotipos y genotipos de H pylori resistentes a claritromicina y levofloxacino mostraron una concordancia satisfactoria (coeficiente kappa = 0,810 y 0,782, respectivamente)19. Por lo tanto, es factible utilizar el método qPCR para detectar la infección por H. pylori y la resistencia a los medicamentos. Los métodos de prueba de antígeno en heces (SAT) y qPCR se utilizan cada vez más debido a la conveniencia del muestreo. La sensibilidad, especificidad y precisión de los métodos para la detección de H. pylori son las siguientes: qPCR > UBT > SAT > RUT> IgG > cultivo20. Todos los métodos no invasivos son adecuados para el cribado primario. Dado que la qPCR también puede detectar genes de resistencia a los fármacos, la qPCR y el cultivo serán más adecuados para guiar el tratamiento.
Otros enfoques innovadores, como los algoritmos de inteligencia artificial (IA) combinados con imágenes endoscópicas21, la microfluídica digital22 y RPA-CRISPR/Cas12a23, muestran teóricamente una buena eficiencia diagnóstica y perspectivas de aplicación prometedoras. Sin embargo, la qPCR es actualmente la técnica de biología molecular más sofisticada con el potencial de proporcionar una solución superior para el diagnóstico y tratamiento de las infecciones por H. pylori .
Protocolo
Este estudio se adhiere a las directrices éticas establecidas por el Comité de Ética del Hospital Popular Provincial de Guangdong, Universidad Médica del Sur, Guangzhou, China (Aprobación No: KY2024-445-01). La información detallada sobre los materiales utilizados en esta investigación (reactivos, productos químicos, equipos y software) se puede encontrar en la Tabla de Materiales.
1. Selección de participantes
- Elija participantes en el rango de edad de 18 a 60 años.
- Elija participantes que puedan someterse a un cribado de detección de infección por Helicobacter pylori , como pacientes que presenten síntomas como gastritis, úlceras pépticas o dispepsia, así como personas asintomáticas.
- Excluir del estudio a los pacientes que hayan usado antibióticos, agentes que contengan bismuto (como cápsulas de citrato de bismuto de potasio, cápsulas de pectina de bismuto coloidal o tabletas de compuesto de bismuto y aluminio) o medicamentos antimicrobianos chinos en el último mes, o aquellos que hayan usado inhibidores de la bomba de protones o antagonistas de los receptores H2 (como omeprazol, pantoprazol, rabeprazol, cimetidina o nizatidina) en las últimas 2 semanas. También excluya del estudio a las mujeres durante su período menstrual.
2. Recogida de muestras fecales
NOTA: Proporcione la sección 2 como instrucciones a los participantes.
- Confirme que el embalaje exterior del kit de muestreo no esté dañado y dentro de la fecha de caducidad. Retira los materiales del kit.
- Coloque un papel blanco de recolección de heces en la taza del inodoro y defeque (para evitar la contaminación con orina).
- Con una espátula, recoja una porción de heces aproximadamente del tamaño de una haba (~5 g) y transfiérala al recipiente de la muestra.
- Coloque la espátula de muestreo, junto con la muestra fecal, en el tubo de recolección, asegurándose de que el nivel del líquido suba hasta cerca de la línea de muestreo indicada en el tubo.
- Apriete firmemente la tapa del tubo. Invierta el tubo varias veces para asegurar una mezcla completa de la muestra con la solución conservante.
- Almacene la muestra en refrigeración hasta su entrega al laboratorio clínico para la posterior identificación de H. pylori y la evaluación de los perfiles de resistencia a los antibióticos mediante qPCR.
3. Extracción de ácidos nucleicos
NOTA: Complete todos los procedimientos dentro de una cabina de bioseguridad para evitar la contaminación.
- Invierta el tubo de conservación de muestras y mezcle bien. Transfiera 1.0 mL de la solución de la muestra fecal a un tubo de microcentrífuga.
- Después de mezclar los tubos de centrífuga, colóquelos en un baño de metal a 80 °C durante 10 minutos, con mezcla intermitente durante 30 s al quinto minuto. Una vez que los tubos se hayan enfriado a temperatura ambiente, centrifugar a 10.000 × g durante 5 minutos y recoger el sobrenadante para su posterior procesamiento.
- Invierta la placa de 96 pocillos varias veces para volver a suspender las perlas magnéticas. Retire con cuidado el sello de papel de aluminio, teniendo cuidado de evitar sacudidas para evitar derrames.
- Añada 200 μL de la muestra preparada (del paso 3.2) a cada pocillo de la placa de 96 pocillos que contiene tampón de lisis, asegurándose de que cada pocillo corresponda a una sola muestra. Coloque la placa de 96 pocillos en el compartimento de muestra designado del instrumento de extracción de ácido nucleico para la extracción automatizada.
- Almacene las muestras restantes y las muestras de ácido nucleico extraídas a -20 °C para su conservación a largo plazo y su uso futuro.
4. qPCR Detección de ácidos nucleicos de Helicobacter pylori y mutaciones de resistencia a claritromicina y quinolonas
- Tome los reactivos del área de almacenamiento de reactivos y descongélelos a temperatura ambiente.
NOTA: Este kit se utilizó para la detección cualitativa del ácido nucleico de H. pylori, incluidas las mutaciones en el gen 23S rRNA (A2143G, A2143C, A2144G) y el gen gyrA (261A, 261G, 260T, 271A, 271T, 272G). Los cebadores, las sondas, la enzima Taq, la enzima UNG y los dNTP estaban disponibles en el kit. Los genes diana de H. pylori y β-actina humana se incluyeron en el control de calidad positivo. Se eligió el gen humano β-actina como control de calidad interno. - En función del número de muestras que se van a analizar, utilice N + 2 copias del reactivo liofilizado, donde N representa el número de muestras que se van a analizar y 2 representa los controles de calidad negativos y positivos.
- Centrifugar brevemente para asegurarse de que el polvo liofilizado se deposite en el fondo de los tubos. Abra la tapa del reactivo liofilizado (teniendo cuidado de evitar cualquier derrame del polvo) y agregue 25 μL por pocillo de los ácidos nucleicos extraídos de las muestras a analizar, así como los controles positivos y negativos. Cierre bien los tubos.
- Agite los reactivos de PCR durante 8-10 s, luego centrifugue brevemente durante 3-5 s para evitar la formación de burbujas.
- Coloque la placa qPCR de 96 pocillos en la máquina qPCR. Configure el programa de ciclo de la siguiente manera: primero, incube la mezcla de reacción (que contiene cebadores, sondas, polimerasa Taq, enzima UNG, dNTP, etc.) a 42 °C y 95 °C (ambos pasos en un ciclo) durante 2 minutos; luego, ciclo 10x con 10 s a 95 °C y 45 s a 65 °C; finalmente, ciclo 35x con 10 s a 95 °C y 45 s a 58 °C para desnaturalización, recocido y extensión.
- Establezca los parámetros de detección de la señal de fluorescencia de la siguiente manera: marcaje fluorescente ROX de genes conservados de H. pylori ; Marcaje fluorescente FAM de genes de resistencia a claritromicina de H. pylori ; Marcaje fluorescente HEX de genes de resistencia a quinolonas de H. pylori ; y el marcaje fluorescente CY5 del gen humano β-actina se utilizaron como control interno del kit. Recogida de datos a 58 °C. Una vez finalizada la reacción, asegúrese de que los datos se guarden para futuros análisis.
- Analice los datos utilizando un software específico de qPCR, ya que el instrumento selecciona automáticamente el umbral de referencia. Establecer todos los criterios diagnósticos, incluida la presencia de infección o resistencia a Helicobacter pylori , a un valor de CT ≤30; confirman que exhiben una curva característica en forma de S.
Resultados
Aplicación de qPCR para la detección de infección por Helicobacter pylori y resistencia a antibióticos en muestras fecales
Diseñamos cebadores y sondas basados en mutaciones puntuales en los genes conservados de Helicobacter pylori, así como en el gen 23S rRNA y el gen gyrA . Estos cebadores y sondas se marcaron con diferentes tintes fluorescentes y luego se utilizaron para la detección de qPCR. Los resultados del control de calidad para los experimentos de qPCR estuvieron dentro del rango recomendado, lo que indica resultados de detección confiables (Figura 1). En este estudio, seleccionamos cinco muestras representativas (S1-S5) para caracterizar la confiabilidad del protocolo experimental (Figura 2 y Tabla 1). S1 es una muestra sin infección por H. pylori , mientras que S2-S5 son muestras con diferentes perfiles de resistencia a fármacos.
Para las muestras positivas para la infección por Helicobacter pylori , la muestra S2 mostró valores de CT dentro del rango de detección solo para Helicobacter pylori, lo que indica que esta muestra es positiva para Helicobacter pylori y es sensible tanto a la claritromicina como a las quinolonas, lo que permite el tratamiento con cualquiera de los fármacos. La muestra S3 mostró valores de CT dentro del rango de detección tanto para la infección por Helicobacter pylori como para la resistencia a la claritromicina, sin que se detectaran valores de CT para la resistencia a las quinolonas, lo que sugiere que la muestra S3 es de un paciente resistente a la claritromicina. De manera similar, la muestra S4 presentó valores de CT dentro del rango de detección para infección por Helicobacter pylori y resistencia a quinolonas, sin que se detectaran valores de CT para resistencia a claritromicina, lo que indica resistencia a quinolonas y recomienda claritromicina para el tratamiento. Finalmente, la muestra S5 tuvo valores de CT detectables en todas las pruebas, lo que indica que la muestra es positiva para Helicobacter pylori y exhibe doble resistencia tanto a la claritromicina como a las quinolonas; Por lo tanto, los médicos deben considerar opciones de tratamiento alternativas. En comparación con otros métodos de detección de Helicobacter pylori , este enfoque no solo facilita la adquisición de muestras, sino que también permite la detección simultánea de Helicobacter pylori y su perfil de resistencia, proporcionando resultados fiables para guiar las decisiones de tratamiento adecuadas.
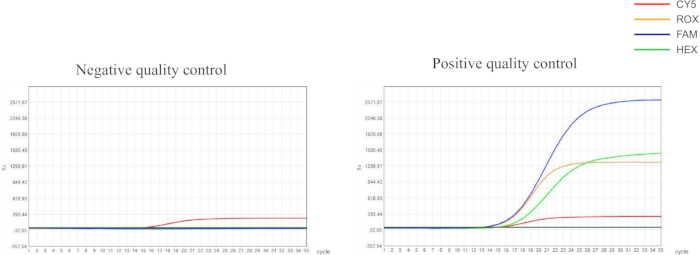
Figura 1: Resultados del control de calidad de los controles negativos y positivos. Los controles negativos deben mostrar una curva de crecimiento en forma de S para el canal de detección CY5 con un valor de CT ≤ 30,00; las señales de fluorescencia para los canales FAM, HEX y ROX no deben mostrar aumentos significativos, con valores de CT > 30.00 o ninguna señal notable; Los controles positivos deben presentar curvas en forma de S en todos los canales de detección con valores de CT ≤ 30,00. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Detección de Helicobacter pylori y su resistencia a antibióticos en muestras fecales por qPCR. Cada color representa una señal fluorescente, y diferentes señales fluorescentes representan diferentes genes. Canales de fluorescencia: marcaje fluorescente ROX (naranja) de genes conservados de H. pylori ; Marcaje fluorescente FAM (azul) de genes de resistencia a claritromicina de H. pylori ; Marcaje fluorescente HEX (verde) de genes de resistencia a quinolonas de H. pylori ; Marcaje fluorescente CY5 (rojo) del gen de control interno. Fenotipos de la muestra: S1 es una muestra negativa para la infección por H. pylori (sin señal por encima del umbral); S2 es una muestra positiva para H. pylori sin resistencia a antibióticos (señal ROX por encima del umbral); S3 es una muestra positiva para H. pylori con resistencia a claritromicina (señal FAM por encima del umbral); S4 es una muestra positiva para H. pylori con resistencia a quinolonas (señal HEX por encima del umbral); S5 es una muestra positiva para H. pylori resistente tanto a la claritromicina como a las quinolonas (señal FAM y HEX por encima del umbral). Haga clic aquí para ver una versión más grande de esta figura.
| Muestra | H.Pylori (ROX) | Claritromicina (FAM) | Quinolonas (HEX) | CY5 | |||
| +/- | CT | +/- | CT | +/- | CT | CT | |
| S1 | - | - | - | - | - | - | 9.96 |
| S2 | + | 23.08 | - | - | - | - | 12.66 |
| S3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| S4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| negetiva | - | - | - | - | - | - | 14.73 |
| Positivo | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
Tabla 1: Resultados de la detección de infección por H. pylori y qPCR para resistencia a claritromicina y quinolona. La tabla proporciona los resultados cualitativos de la infección por H. pylori. La detección de mutaciones en el gen 23S rRNA indica resistencia a la claritromicina, mientras que las mutaciones en el gen gyrA indican resistencia a las quinolonas. Símbolos: +/−, resultado cualitativo; +, resultado positivo; −, resultado negativo.
Discusión
En los últimos años, los métodos de detección molecular se han aplicado ampliamente en el campo de la microbiología, alterando significativamente el manejo clínico de varias enfermedades infecciosas. Estos métodos operan a nivel genético, lo que permite no solo la confirmación de la presencia bacteriana, sino también la tipificación de genes y las pruebas de resistencia a los antibióticos. La PCR cuantitativa de fluorescencia en tiempo real (qPCR) se ve cada vez más favorecida debido a su corto tiempo de procesamiento, alta sensibilidad y precisión, y bajo riesgo de contaminación cruzada. Se ha vuelto ampliamente utilizado en los laboratorios clínicos para el diagnóstico de infecciones por Helicobacter pylori .
La combinación de soluciones de conservación de muestras fecales con métodos de detección de genes permite el cribado rápido y eficiente de H . pylori y otras pruebas de resistencia a los antibióticos mediante el análisis de mutaciones en el gen de ARNr de claritromicina 23S y el gen gyrA de quinolona. Este método puede evaluar la sensibilidad y la resistencia de H. pylori a los fármacos. Los métodos anteriores de detección de antígenos fecales tienen limitaciones notables, como la reducción de la precisión y la estabilidad debido a las bajas concentraciones de antígenos y la degradación de antígenos en las muestras. Además, los métodos tradicionales de detección de antígenos pueden carecer de suficiente sensibilidad y especificidad en muestras complejas, lo que afecta a la fiabilidad de los resultados.
Para abordar estos problemas, proponemos un método que combina muestras fecales con la tecnología de reacción en cadena de la polimerasa cuantitativa (qPCR). Este enfoque mejora significativamente la sensibilidad y la precisión de la detección, lo que hace que las pruebas fecales sean más fiables y eficientes, y proporciona resultados de mayor calidad para uso clínico.
Los estándares de control de calidad son cruciales para la detección de qPCR. Nuestro control de calidad incluye la calibración periódica del instrumento, la verificación del rendimiento del sistema de detección, la evaluación de la eficiencia de la extracción de ácidos nucleicos y el control de calidad de cada lote de experimentos. En primer lugar, nuestros instrumentos de muestreo se calibran cada 6 meses, y el equipo de extracción y el equipo de amplificación se calibran anualmente. Además, la calibración se realiza para cada lote de reactivos. Después de la calibración, podemos validar nuestro rendimiento para el sistema de detección, incluido el instrumento de PCR cuantitativa de fluorescencia Macrostone 96S, los kits de detección de H. pylori y mutaciones de resistencia, los reactivos de extracción, así como los tubos de muestreo, pipetas y puntas correspondientes. La tasa de conformidad, el límite de detección, la reacción cruzada, la capacidad antiinterferente y la precisión satisfacen nuestras necesidades. Las concentraciones de productos de ADN se determinaron mediante un probador de concentración de ADN para cumplir con los criterios de aceptación de la eficiencia de extracción de ácidos nucleicos. La concentración de ADN fue superior a 10 ng/μL. El A260/A280 estaba entre 1,6 y 2,0. Las muestras no calificadas deben volver a extraerse. Los resultados solo son válidos cuando el control de calidad diario está bajo control. Para las pruebas de resistencia a H. pylori y antibióticos (claritromicina y quinolonas), los criterios de control de calidad son los siguientes: los testigos negativos deben mostrar una curva de crecimiento en forma de S para el canal de detección CY5 con un valor de CT ≤ 30,00; las señales de fluorescencia para los canales FAM, HEX y ROX no deben mostrar aumentos significativos, con valores de CT >30.00 o ninguna señal notable; Los controles positivos deben presentar curvas en forma de S en todos los canales de detección con valores de CT ≤ 30,00. Estas condiciones deben cumplirse dentro del mismo experimento; De lo contrario, la prueba se considerará inválida y deberá volver a probarse. Tenga en cuenta que, debido a las restricciones de propiedad comercial, no se puede proporcionar información sobre la secuencia del cebador y la sonda en este documento.
En un estudio anterior, los investigadores emplearon una metodología idéntica a la utilizada en este trabajo, demostrando que las pruebas de RT-PCR para la infección por H. pylori en muestras fecales mostraron un rendimiento diagnóstico excepcional24. En concreto, la prueba alcanzó una sensibilidad del 99,1%, una especificidad del 100% y una precisión diagnóstica del 99,1%, lo que coincidió estrechamente con los resultados de las muestras de biopsia gástrica (93,9%) (Kappa = 0,929, p < 0,001). Este notable acuerdo puso de manifiesto que las pruebas RT-PCR pueden considerarse un método no invasivo muy fiable.
Además, el estudio también evaluó la capacidad de la RT-PCR fecal para detectar la resistencia a los antibióticos en H. pylori, centrándose especialmente en la resistencia a la claritromicina y la levofloxacina. Para la resistencia a claritromicina se identificaron 43 casos (37,3%) en muestras fecales, con una sensibilidad del 79,6%, una especificidad del 98,4% y una precisión diagnóstica del 78,0% (Kappa = 0,788, p < 0,001). Del mismo modo, para la resistencia a levofloxacino, se detectaron 37 casos (32,1%), con una sensibilidad del 86,3%, una especificidad del 91,1% y una precisión diagnóstica del 74,4% (Kappa = 0,739, p < 0,001). Estos hallazgos fueron altamente consistentes con los derivados de las muestras de biopsia gástrica (resistencia a claritromicina en 54 casos, resistencia a levofloxacino en 36 casos), lo que indica que la RT-PCR fecal es efectiva en la detección de resistencia a antibióticos, poseyendo una alta especificidad y una sensibilidad aceptable para ambos antibióticos. La validación de esta metodología en el estudio previo apoya aún más su fiabilidad y aplicabilidad en el contexto de este trabajo.
Si bien el protocolo de detección fecal basado en qPCR representa un avance notable en el campo del diagnóstico no invasivo para H. pylori, varias limitaciones requieren una consideración meticulosa. En primer lugar, el uso de muestras fecales hace que el método se vea afectado por la calidad de la muestra. Los falsos negativos pueden ser el resultado de bajas cargas bacterianas en las heces, particularmente en portadores asintomáticos o pacientes con infecciones en etapa temprana, donde la diseminación de H. pylori en el tracto gastrointestinal es intermitente25. Además, al tomar muestras fecales, la cantidad de bacterias puede variar según el lugar donde se tome la muestra. Esto contrasta marcadamente con los métodos invasivos, como el cultivo de biopsia gástrica, que toman muestras directamente de la mucosa gástrica y, por lo tanto, están menos influenciados por las fluctuaciones de la carga bacteriana. En segundo lugar, aunque el enfoque de amplificación dirigida del protocolo es específico, es posible que no detecte mutaciones de resistencia raras o emergentes que no estén incorporadas en el diseño del cebador, lo que podría conducir a perfiles de resistencia incompletos26.
La investigación futura debe centrarse en los avances metodológicos para mejorar las capacidades de detección de mutaciones, incluida la adopción de la PCR digital (dPCR) para mejorar la identificación de mutaciones de resistencia de baja abundancia27. Los paneles de sonda requieren una expansión estratégica para abarcar polimorfismos clínicamente relevantes. La realización de estudios multicéntricos para evaluar comparativamente las metodologías diagnósticas, incluidas las pruebas de UBT, PCR de la mucosa gástrica o fecal y la detección de antígenos, proporciona datos completos de rendimiento diagnóstico para la práctica clínica, avanzando así en la estandarización de los protocolos de pruebas de PCR en heces. Estas mejoras combinadas reforzarían la utilidad de la qPCR fecal como herramienta de diagnóstico no invasiva, sin dejar de ser importante para guiar un tratamiento antibiótico preciso.
Divulgaciones
Los autores declaran que no tienen intereses contrapuestos.
Agradecimientos
Este estudio fue financiado por la Fundación de Investigación para Talentos Avanzados del Hospital Popular Provincial de Guangdong [Subvención No. KY012023293]. Este trabajo fue apoyado por Jiangsu Mole Bioscience Co. Los financiadores no tuvieron ningún papel en el diseño del estudio, la recopilación y el análisis de datos, la decisión de publicar o la preparación del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
Referencias
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados