Method Article
איתור זיהום הליקובקטר פילורי ועמידות לאנטיביוטיקה באמצעות ניתוח תגובת שרשרת פולימראז כמותי בצואה
* These authors contributed equally
In This Article
Summary
הפרוטוקול משתמש בדגימת צואה לא פולשנית בשילוב עם תגובת שרשרת פולימראז כמותית כדי להציע שיטת אבחון נוחה ומהירה לזיהום הליקובקטר פילורי ועמידותו בפני קלריתרומיצין וקינולונים.
Abstract
הליקובקטר פילורי (H. pylori) נפוץ מאוד ברחבי העולם, כאשר לכ-50% מאוכלוסיית העולם יש היסטוריה של זיהום בהליקובקטר פילורי. בסין, שיעור ההדבקה נע בין 40% ל-70%. הליקובקטר פילורי קשור בעיקר למחלות במערכת העיכול כמו דלקת קיבה כרונית, כיבי קיבה וכיבים בתריסריון. נכון לעכשיו, הטיפול הקליני בזיהום הליקובקטר פילורי כולל טיפול משולש או מרובע. עם זאת, השימוש הנרחב באנטיביוטיקה הוביל להתפתחות עמידות לאנטיביוטיקה בהליקובקטר פילורי. לכן, זיהוי הן של הליקובקטר פילורי והן של עמידותו לאנטיביוטיקה הוא חיוני להנחיית הטיפול הקליני.
שיטות האבחון להליקובקטר פילורי כוללות בדיקת נשימה אוריאה (UBT), בדיקת אנטיגן, בדיקת נוגדנים סרולוגיים, אנדוסקופיה, בדיקת אוראז מהירה (RUT) ותרבית חיידקים. בעוד ששלוש השיטות הראשונות אינן פולשניות, הן אינן מאפשרות התאוששות חיידקים ולכן לא ניתן להשתמש בהן לבדיקת עמידות. שלוש השיטות האחרונות הן פולשניות, יקרות, דורשות מומחיות טכנית גבוהה ועלולות לגרום נזק לחולים.
לכן, שיטה לא פולשנית ומהירה לזיהוי בו-זמני של זיהום בהליקובקטר פילורי ועמידות לאנטיביוטיקה היא בעלת חשיבות קלינית עליונה למיגור יעיל של הליקובקטר פילורי. מאמר זה נועד להציג פרוטוקול ספציפי המשלב תגובת שרשרת פולימראז כמותית (qPCR) עם טכנולוגיית בדיקה פלואורסצנטית של TaqMan כדי לזהות במהירות זיהום בהליקובקטר פילורי ועמידות לאנטיביוטיקה. שיטה זו מספקת דרך נוחה, מהירה ולא פולשנית לאבחון זיהום ועמידות להליקובקטר פילורי , בניגוד לתרבית חיידקים מסורתית וטכניקות אחרות. qPCR משמש לזיהוי הזיהום וזיהוי מוטציות בגנים 23S rRNA ו-gyrA , הקשורים לעמידות לקלריתרומיצין וקווינולונים, בהתאמה. בהשוואה לטכניקות תרבית קונבנציונליות, גישה זו מציעה שיטה לא פולשנית, חסכונית וחסכונית בזמן לאיתור זיהום הליקובקטר פילורי וקביעת עמידותו לאנטיביוטיקה.
Introduction
הליקובקטר פילורי הוא חיידק גרם שלילי בצורת ספירלה שיכול להדביק באופן מתמשך את אפיתל הקיבה האנושי1. בשנת 1994, ארגון הבריאות העולמי סיווג את הליקובקטר פילורי כמסרטן מקבוצה 1 לסרטן הקיבה, כאשר ~3% מהאנשים שנדבקו פיתחו בסופו של דבר את המחלה2. סקירה שיטתית שפורסמה לאחרונה הצביעה על מגמה כללית של עלייה בשיעור העמידות לאנטיביוטיקה של הליקובקטר פילורי בעשור האחרון, שהגיעה לרמות מדאיגות ברחבי העולם, במיוחד עמידות לקלריתרומיצין3. בסין, נתוני הסקר האחרונים הראו כי השכיחות המצטברת של הליקובקטר פילורי בקרב סינים עירוניים היא 27.08%; שיעורי העמידות לקלריתרומיצין ולבופלוקסצין היו 50.83% ו-47.17%4, שהם גבוהים יותר מארה"ב (לבופלוקסצין 37.6%, קלריתרומיצין 31.5%)5ואירופה (לבופלוקסצין 15.8% וקלריתרומיצין 21.4%)6. כתוצאה מכך, אבחון וטיפול מוקדמים ומדויקים הם קריטיים. עם זאת, העמידות הגוברת לאנטיביוטיקה של הליקובקטר פילורי מפחיתה במידה ניכרת את יעילות הטיפול, מה שמדגיש את הצורך הדחוף במחקר מתמשך לאבחון ולטיפול בה.
שיטות האבחון של הליקובקטר פילורי מסווגות לטכניקות פולשניות ולא פולשניות7. שיטות פולשניות כוללות בדיקה היסטופתולוגית, בדיקת אוראז מהירה (RUT) ותרבית חיידקים8. בדיקה היסטופתולוגית מסתמכת על עיבוד ותצפית מיקרוסקופית של דגימות ביופסיה, עם דיוק מוגבל על ידי הכנה היסטולוגית ומומחיות בפתולוגיה9. RUT מזהה הליקובקטר פילורי באמצעות פעילות של אוראז, אשר מבצע הידרוליזה של אוריאה לייצור אמוניה, וכתוצאה מכך שינוי אלקליין במגיב, ובכך מצביע על נוכחות החיידק9. זה פשוט וחסכוני. לתרבית חיידקים, הנחשבת ל"תקן הזהב", יש לעתים קרובות שיעור הצלחה מתחת ל-50% בשל המאפיינים הפיזיולוגיים הייחודיים של הליקובקטר פילורי10.
שיטות לא פולשניות כוללות בדיקת נשימה אוריאה 13C/14C (UBT), אבחון סרולוגי ובדיקת אנטיגן צואה (SAT)11. בדיקת הנשימה פועלת על פי עיקרון דומה לזה של RUT, ומזהה CO2 מסומן איזוטופית באוויר נשוף כדי לאשר זיהום12. אבחון סרולוגי מזהה נוגדנים, שעלולים להימשך לאחר מיגור חיידקים, מה שמקשה על הערכת יעילות הטיפול13. בדיקת אנטיגן צואה מזהה אנטיגנים ספציפיים להליקובקטר פילורי בדגימות צואה, ומספקת שיטה אמינה ולא פולשנית עם שיעור נמוך יותר של תוצאות חיוביות כוזבות ודיוק טוב לאבחון זיהומים פעילים14.
לכל שיטת אבחון יש את היתרונות והמגבלות שלה15. מלבד תרבית חיידקים, שיטות אחרות מתקשות באיתור עמידות לאנטיביוטיקה, בעוד שתרבית נפגעת על ידי שיעורי הצלחה נמוכים16. לאחרונה, שיטות אבחון מולקולאריות כגון PCR כמותי בזמן אמת (qPCR) יושמו באופן נרחב לזיהוי מיקרוביאלי17. qPCR יכול לזהות במדויק את הליקובקטר פילורי ולנתח מוטציות בגנים עמידים, ומציע מבט מקיף יותר על הזיהום18.
בדיקות פלואורסצנטיות נועדו להיקשר במיוחד לרצף מטרה ב-DNA במהלך תהליך ה-qPCR. בדיקות אלה מסומנות בדרך כלל בצבע פלואורסצנטי, הפולט אות כאשר הגשושית נקשרת למטרה שלה ונבקעת על ידי הפולימראז במהלך ההגברה. זה מספק מדידה בזמן אמת של תהליך ה-PCR. בדיקות פלואורסצנטיות ידועות בספציפיות הגבוהה שלהן בשל העיצוב הייחודי של הגשושית, המבטיח שרק רצף ה-DNA של המטרה מזוהה, מה שמפחית את הסיכוי לקשירה לא ספציפית ומשפר את ספציפיות הבדיקה. זה הופך את טכנולוגיית הבדיקה הפלואורסצנטית לשימושית במיוחד לאיתור מטרות בשפע נמוך, כגון DNA הליקובקטר פילורי בדגימות צואה. בעוד ש-SYBR Green הוא צבע קושר DNA נפוץ ב-PCR, הוא יכול להיקשר לכל DNA דו-גדילי, מה שעלול לגרום להגברה לא ספציפית ולתוצאות חיוביות כוזבות.
תרבית הליקובקטר פילורי מדגימות קליניות אינה מספקת נתוני עמידות לאנטיביוטיקה ישירות. לעומת זאת, qPCR מבוסס בדיקה פלואורסצנטית הוא מהיר יותר, נוח יותר וניתן להתאים אותו לזיהוי בו-זמני של הליקובקטר פילורי וסמני עמידות לאנטיביוטיקה. שיטה זו לא רק מספקת תוצאות מהירות יותר משיטות תרבות מסורתיות, אלא גם מאפשרת זיהוי בו זמנית של גנים עמידים לאנטיביוטיקה, מה שמשפר מאוד את יעילות הזיהוי והנוחות. שיטה זו מספקת דרך נוחה, מהירה ולא פולשנית לאבחון זיהום ועמידות להליקובקטר פילורי , בניגוד לתרבית חיידקים מסורתית וטכניקות אחרות.
ישנן שתי סוגיות מכריעות שיש לקחת בחשבון עבור עמידות לאנטיביוטיקה של הליקובקטר פילורי : 1) העקביות של הגנוטיפ והפנוטיפ, 2) העמידות לתרופות מרובות. מחקר רב-מרכזי רחב היקף בסין מצא כי דפוסי העמידות הכפולה לקלריתרומיצין/לבופלוקסצין היו 26.1%. פנוטיפים וגנוטיפים של H pylori עמידים לקלריתרומיצין ולבופלוקסאצין הראו הסכמה משביעת רצון (מקדם קאפה = 0.810 ו-0.782, בהתאמה)19. לכן, ניתן להשתמש בשיטת qPCR לאיתור זיהום בהליקובקטר פילורי ועמידות לתרופות. שיטות בדיקת אנטיגן צואה (SAT) ו-qPCR נמצאות בשימוש יותר ויותר בשל נוחות הדגימה. הרגישות, הספציפיות והדיוק של השיטות לזיהוי הליקובקטר פילורי הם כדלקמן: qPCR > UBT > SAT > RUT> CagA IgG > תרבית20. כל השיטות הלא פולשניות מתאימות לבדיקה ראשונית. מכיוון ש-qPCR יכול לזהות בנוסף גנים עמידים לתרופות, qPCR ותרבית יתאימו יותר להנחיית הטיפול.
גישות חדשניות אחרות, כגון אלגוריתמים של בינה מלאכותית (AI) בשילוב עם תמונות אנדוסקופיות21, מיקרופלואידיקה דיגיטלית22 ו-RPA-CRISPR/Cas12a23, מראות תיאורטית יעילות אבחון טובה וסיכויי יישום מבטיחים. עם זאת, qPCR היא כיום הטכניקה המתוחכמת ביותר לביולוגיה מולקולרית עם פוטנציאל לספק פתרון מעולה לאבחון וטיפול בזיהומי הליקובקטר פילורי .
Protocol
מחקר זה מקפיד על ההנחיות האתיות שנקבעו על ידי ועדת האתיקה של בית החולים העממי המחוזי גואנגדונג, האוניברסיטה הרפואית הדרומית, גואנגג'ואו, סין (מס' אישור: KY2024-445-01). מידע מפורט לגבי החומרים המשמשים במחקר זה (ריאגנטים, כימיקלים, ציוד ותוכנה) ניתן למצוא בטבלת החומרים.
1. בחירת משתתפים
- בחר משתתפים בטווח הגילאים 18 עד 60 שנים.
- בחר משתתפים שעשויים לעבור בדיקת זיהום הליקובקטר פילורי , כגון חולים המציגים תסמינים כמו דלקת קיבה, כיבים פפטיים או דיספפסיה, כמו גם אנשים ללא תסמינים.
- להוציא מהמחקר מטופלים שהשתמשו באנטיביוטיקה, חומרים המכילים ביסמוט (כגון כמוסות אשלגן ביסמוט ציטראט, כמוסות פקטין ביסמוט קולואידיות, או טבליות תרכובת ביסמוט-אלומיניום), או תרופות סיניות אנטי-מיקרוביאליות במהלך החודש האחרון, או כאלה שהשתמשו במעכבי משאבת פרוטון או אנטגוניסטים לקולטן H2 (כגון אומפרזול, פנטופרזול, רבפרזול, סימטידין או ניזטידין) במהלך השבועיים האחרונים. כמו כן, לא לכלול נשים במהלך המחזור החודשי שלהן מהמחקר.
2. איסוף דגימות צואה
הערה: ספק את סעיף 2 כהוראות למשתתפים.
- ודא שהאריזה החיצונית של ערכת הדגימה אינה פגומה ובתוך תאריך התפוגה. הסר את החומרים מהערכה.
- מניחים נייר איסוף צואה לבן בקערת האסלה ועושים את צרכיהם (כדי למנוע זיהום בשתן).
- בעזרת מרית, אוספים חלק של צואה בערך בגודל של שעועית רחבה (~5 גרם) ומעבירים אותה למיכל הדגימה.
- הנח את מרית הדגימה, יחד עם דגימת הצואה, לתוך צינור האיסוף, וודא שמפלס הנוזל עולה קרוב לקו הדגימה המצוין על הצינור.
- הדק היטב את מכסה הצינור. הפוך את הצינור מספר פעמים כדי להבטיח ערבוב יסודי של הדגימה עם תמיסת השימור.
- אחסן את הדגימה בקירור עד למסירה למעבדה הקלינית לזיהוי הבא של הליקובקטר פילורי והערכת פרופילי עמידות לאנטיביוטיקה באמצעות qPCR.
3. מיצוי חומצות גרעין
הערה: השלם את כל ההליכים בתוך ארון בטיחות ביולוגית כדי למנוע זיהום.
- הפוך את צינור שימור הדגימה וערבב היטב. העבירו 1.0 מ"ל מתמיסת דגימת הצואה לתוך צינור מיקרו-צנטריפוגה.
- לאחר ערבוב צינורות הצנטריפוגה, הניחו אותם באמבט מתכת בחום של 80 מעלות צלזיוס למשך 10 דקות, עם ערבוב לסירוגין למשך 30 שניות בדקה החמישית. לאחר שהצינורות התקררו לטמפרטורת החדר, צנטריפוגה ב-10,000 × גרם למשך 5 דקות ואספו את הסופרנטנט להמשך עיבוד.
- הפוך את צלחת 96 הבארות מספר פעמים כדי להשעות מחדש את החרוזים המגנטיים. הסר בזהירות את אטם נייר האלומיניום, הקפד להימנע מטלטול כדי למנוע שפיכה.
- הוסף 200 מיקרוליטר מהדגימה המוכנה (משלב 3.2) לכל באר של צלחת 96 הבארות המכילה מאגר ליזה, וודא שכל באר מתאימה לדגימה אחת. הנח את צלחת 96 הבארות בתא הדגימה המיועד של מכשיר מיצוי חומצות הגרעין למיצוי אוטומטי.
- אחסן את כל הדגימות שנותרו ואת דגימות חומצות הגרעין שחולצו ב-20 מעלות צלזיוס לשימור ארוך טווח ולשימוש עתידי.
4. qPCR איתור חומצות גרעין הליקובקטר פילורי ומוטציות עמידות לקלריתרומיצין וקינולונים
- קח את הריאגנטים מאזור אחסון הריאגנטים והפשיר אותם בטמפרטורת החדר.
הערה: ערכה זו שימשה לזיהוי איכותי של חומצת גרעין הליקובקטר פילורי , כולל מוטציות בגן 23S rRNA (A2143G, A2143C, A2144G) ובגן gyrA (261A, 261G, 260T, 271A, 271T, 272G). הפריימרים, הבדיקות, האנזים Taq, האנזים UNG וה-dNTPs היו זמינים בערכה. גני המטרה של הליקובקטר פילורי ו-β-אקטין אנושי נכללו בבקרת האיכות החיובית. הגןהאנושי β-אקטין נבחר כבקרת האיכות הפנימית. - בהתבסס על מספר הדגימות שייבדקו, השתמש בעותקים של N + 2 של המגיב הליופילי, כאשר N מייצג את מספר הדגימות שיש לבדוק ו-2 מייצג את ה-QCs השליליים והחיוביים.
- צנטריפוגה בקצרה כדי להבטיח שהאבקה הליופילית תתייצב בתחתית הצינורות. פתח את המכסה של המגיב הליופילי (הקפד למנוע שפיכה של האבקה) והוסף 25 מיקרוליטר לבאר של חומצות הגרעין המופקות מהדגימות לבדיקה, כמו גם את הבקרות החיוביות והשליליות. סגור את הצינורות בחוזקה.
- מערבולת את ריאגנטים ה-PCR למשך 8-10 שניות, ואז צנטריפוגה קצרה למשך 3-5 שניות כדי למנוע היווצרות בועות.
- הנח את לוחית ה-qPCR בעלת 96 הבארות על מכונת ה-qPCR. הגדר את תוכנית הרכיבה באופן הבא: ראשית, דגרו את תערובת התגובה (המכילה פריימרים, בדיקות, Taq polymerase, אנזים UNG, dNTPs וכו') ב-42 מעלות צלזיוס ו-95 מעלות צלזיוס (שני השלבים במחזור אחד) למשך 2 דקות; לאחר מכן, מחזור 10x עם 10 שניות ב-95 מעלות צלזיוס ו-45 שניות ב-65 מעלות צלזיוס; לבסוף, מחזור פי 35 עם 10 שניות ב-95 מעלות צלזיוס ו-45 שניות ב-58 מעלות צלזיוס לדנטורציה, חישול והארכה.
- הגדר את פרמטרי זיהוי אותות הקרינה כדלקמן: תיוג פלואורסצנטי ROX של גנים משומרים של הליקובקטר פילורי ; תיוג פלואורסצנטי FAM של גנים עמידים ל - H. pylori clarithromycin; תיוג פלואורסצנטי HEX של גנים עמידים להליקובקטר פילורי קווינולון; ותיוג פלואורסצנטי CY5 של גן β-אקטין אנושי שימשו כבקרה הפנימית של הערכה. אסוף נתונים בטמפרטורה של 58 מעלות צלזיוס. עם השלמת התגובה, ודא שהנתונים נשמרים לניתוח עתידי.
- נתח את הנתונים באמצעות תוכנה ספציפית ל-qPCR, מכיוון שהמכשיר בוחר אוטומטית את סף הבסיס. הגדר את כל קריטריוני האבחון, כולל נוכחות של זיהום או עמידות להליקובקטר פילורי , לערך CT ≤30; מאשרים שהם מציגים עקומה אופיינית בצורת S.
תוצאות
יישום qPCR לזיהוי זיהום הליקובקטר פילורי ועמידות לאנטיביוטיקה בדגימות צואה
עיצבנו פריימרים ובדיקות המבוססים על מוטציות נקודתיות בגנים השמורים של הליקובקטר פילורי, כמו גם בגן 23S rRNA ובגן gyrA . פריימרים ובדיקות אלה סומנו בצבעי פלורסנט שונים ולאחר מכן שימשו לזיהוי qPCR. תוצאות בקרת האיכות עבור ניסויי ה-qPCR היו בטווח המומלץ, מה שמצביע על תוצאות זיהוי אמינות (איור 1). במחקר זה, בחרנו חמש דגימות מייצגות (S1-S5) כדי לאפיין את המהימנות של פרוטוקול הניסוי (איור 2 וטבלה 1). S1 היא דגימה ללא זיהום בהליקובקטר פילורי , בעוד ש-S2-S5 הן דגימות עם פרופילי עמידות שונים לתרופות.
עבור דגימות חיוביות לזיהום הליקובקטר פילורי , דגימה S2 הראתה ערכי CT בטווח הזיהוי רק עבור הליקובקטר פילורי, מה שמצביע על כך שדגימה זו חיובית להליקובקטר פילורי ורגישה הן לקלריתרומיצין והן לקווינולונים, מה שמאפשר טיפול בכל אחת מהתרופות. דגימה S3 הראתה ערכי CT בטווח הזיהוי הן עבור זיהום הליקובקטר פילורי והן עבור עמידות לקלריתרומיצין, ללא ערכי CT שזוהו עבור עמידות לקווינולון, מה שמרמז על כך שהדגימה S3 היא מחולה עמיד לקלריתרומיצין. באופן דומה, לדגימה S4 היו ערכי CT בטווח הזיהוי של זיהום הליקובקטר פילורי ועמידות לקווינולון, כאשר לא זוהו ערכי CT לעמידות לקלריתרומיצין, מה שמעיד על עמידות לקווינולונים והמלצה על קלריתרומיצין לטיפול. לבסוף, לדגימה S5 היו ערכי CT ניתנים לזיהוי בכל הבדיקות, מה שמצביע על כך שהדגימה חיובית להליקובקטר פילורי ומציגה עמידות כפולה הן לקלריתרומיצין והן לקווינולונים; לפיכך, יש לשקול אפשרויות טיפול חלופיות על ידי רופאים. בהשוואה לשיטות אחרות לזיהוי הליקובקטר פילורי , גישה זו לא רק מקלה על רכישת דגימה אלא גם מאפשרת זיהוי בו זמנית של הליקובקטר פילורי ופרופיל העמידות שלו, ומספקת תוצאות אמינות להנחיית החלטות טיפול מתאימות.
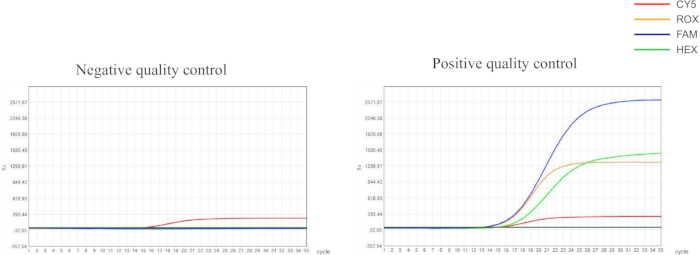
איור 1: תוצאות בקרת איכות של בקרות שליליות וחיוביות. פקדים שליליים צריכים להציג עקומת צמיחה בצורת S עבור ערוץ הזיהוי CY5 עם ערך CT ≤ 30.00; אותות פלואורסצנטיים עבור ערוצי FAM, HEX ו-ROX לא אמורים להראות עליות משמעותיות, עם ערכי CT > 30.00 או ללא אות בולט; בקרות חיוביות צריכות להציג עקומות בצורת S בכל ערוצי הזיהוי עם ערכי CT ≤ 30.00. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2: זיהוי הליקובקטר פילורי ועמידותו לאנטיביוטיקה בדגימות צואה על ידי qPCR. כל צבע מייצג אות פלואורסצנטי, ואותות פלואורסצנטיים שונים מייצגים גנים שונים. תעלות פלואורסצנטיות: תיוג פלואורסצנטי ROX (כתום) של גנים משומרים של הליקובקטר פילורי ; תיוג פלואורסצנטי FAM (כחול) של גנים עמידים ל - H. pylori clarithromycin; תיוג פלואורסצנטי HEX (ירוק) של גנים עמידים להליקובקטר פילורי קווינולון; תיוג פלואורסצנטי CY5 (אדום) של גן הבקרה הפנימי. פנוטיפים לדוגמה: S1 הוא דגימה שלילית לזיהום בהליקובקטר פילורי (אין אות מעל הסף); S2 היא דגימה חיובית להליקובקטר פילורי ללא עמידות לאנטיביוטיקה (אות ROX מעל הסף); S3 הוא דגימה חיובית להליקובקטר פילורי עם עמידות לקלריתרומיצין (אות FAM מעל הסף); S4 היא דגימה חיובית להליקובקטר פילורי עם עמידות לקווינולונים (אות HEX מעל הסף); S5 הוא דגימה חיובית להליקובקטר פילורי עמיד הן לקלריתרומיצין והן לקווינולונים (אות FAM ו-HEX מעל הסף). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| לדוגמה | הליקובקטר פילורי (ROX) | קלריתרומיצין (FAM) | קווינולונים (HEX) | CY5 | |||
| +/- | סי.טי | +/- | סי.טי | +/- | סי.טי | סי.טי | |
| עונה 1 | - | - | - | - | - | - | 9.96 |
| S2 | + | 23.08 | - | - | - | - | 12.66 |
| עונה 3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| עונה 4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| שלילי | - | - | - | - | - | - | 14.73 |
| חיובי | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
טבלה 1: תוצאות זיהוי זיהום בהליקובקטר פילורי ו-qPCR לעמידות לקלריתרומיצין וקווינולון. הטבלה מספקת את התוצאות האיכותיות של זיהום הליקובקטר פילורי. זיהוי מוטציות בגן 23S rRNA מצביע על עמידות לקלריתרומיצין, בעוד שמוטציות בגן gyrA מצביעות על עמידות לקווינולון. סמלים: +/-, תוצאה איכותית; +, תוצאה חיובית; - תוצאה שלילית.
Discussion
בשנים האחרונות, שיטות זיהוי מולקולאריות יושמו באופן נרחב בתחום המיקרוביולוגיה, ושינו באופן משמעותי את הניהול הקליני של מספר מחלות זיהומיות. שיטות אלו פועלות ברמה הגנטית, ומאפשרות לא רק אישור נוכחות חיידקים אלא גם בדיקת סוג גנים ועמידות לאנטיביוטיקה. PCR כמותי פלואורסצנטי בזמן אמת (qPCR) מועדף יותר ויותר בשל זמן העיבוד הקצר שלו, הרגישות והדיוק הגבוהים והסיכון הנמוך לזיהום צולב. הוא הפך לשימוש נרחב במעבדות קליניות לאבחון זיהומי הליקובקטר פילורי .
שילוב פתרונות שימור איסוף דגימות צואה עם שיטות זיהוי גנים מאפשר סריקה מהירה ויעילה של הליקובקטר פילורי ובדיקות עמידות נוספות לאנטיביוטיקה באמצעות ניתוח מוטציות בגן קלריתרומיצין 23S rRNA ובגן quinolone gyrA . שיטה זו יכולה להעריך את הרגישות והעמידות של הליקובקטר פילורי לתרופות. לשיטות קודמות לזיהוי אנטיגן צואתי יש מגבלות בולטות, כגון דיוק ויציבות מופחתים עקב ריכוזי אנטיגן נמוכים ופירוק אנטיגן בדגימות. בנוסף, שיטות זיהוי אנטיגן מסורתיות עשויות להיות חסרות רגישות וספציפיות מספיקות בדגימות מורכבות, מה שמשפיע על אמינות התוצאות.
כדי לטפל בבעיות אלה, אנו מציעים שיטה המשלבת דגימות צואה עם טכנולוגיית תגובת שרשרת פולימראז כמותית (qPCR). גישה זו משפרת משמעותית את רגישות הזיהוי והדיוק, הופכת את בדיקת הצואה לאמינה ויעילה יותר, ומספקת תוצאות באיכות גבוהה יותר לשימוש קליני.
תקני בקרת איכות הם חיוניים לזיהוי qPCR. בקרת האיכות שלנו כוללת כיול תקופתי של המכשיר, אימות הביצועים של מערכת הזיהוי, הערכת יעילות מיצוי חומצות הגרעין ובקרת האיכות של כל אצווה של ניסויים. ראשית, מכשירי הדגימה שלנו מכוילים כל 6 חודשים, וציוד החילוץ וציוד ההגברה מכוילים מדי שנה. בנוסף, הכיול נעשה עבור כל אצווה של ריאגנטים. לאחר הכיול, אנו יכולים לאמת את הביצועים שלנו עבור מערכת הזיהוי, כולל מכשיר ה-PCR הכמותי הפלואורסצנטי של Macrostone 96S, ערכות זיהוי הליקובקטר פילורי ומוטציות התנגדות, ריאגנטים למיצויים, כמו גם צינורות הדגימה, הפיפטות והטיפים המתאימים. קצב ההתאמה, מגבלת הזיהוי, התגובה הצולבת, יכולת נגד הפרעות והדיוק עונים על הצרכים שלנו. ריכוזי מוצרי ה-DNA נקבעו על ידי בודק ריכוז DNA כדי לעמוד בקריטריוני הקבלה ליעילות מיצוי חומצות הגרעין. ריכוז ה-DNA היה מעל 10 ננוגרם/מיקרוליטר. ה-A260/A280 היה בין 1.6 ל-2.0. יש לחלץ מחדש דגימות לא מוסמכות. התוצאות תקפות רק כאשר בקרת האיכות היומית נמצאת תחת שליטה. עבור הליקובקטר פילורי ובדיקת עמידות לאנטיביוטיקה (קלריתרומיצין וקווינולונים), קריטריוני בקרת האיכות הם כדלקמן: בקרות שליליות צריכות להציג עקומת גדילה בצורת S עבור ערוץ הזיהוי CY5 עם ערך CT ≤ 30.00; אותות פלואורסצנטיים עבור ערוצי FAM, HEX ו-ROX לא אמורים להראות עליות משמעותיות, עם ערכי CT >30.00 או ללא אות בולט; בקרות חיוביות צריכות להציג עקומות בצורת S בכל ערוצי הזיהוי עם ערכי CT ≤ 30.00. תנאים אלה חייבים להתקיים באותו ניסוי; אחרת, המבחן ייחשב פסול ויש להיבדק שוב. שים לב שעקב אילוצים קנייניים מסחריים, לא ניתן לספק מידע על פריימר ורצף בדיקה במאמר זה.
במחקר קודם, החוקרים השתמשו במתודולוגיה זהה לזו ששימשה במאמר זה, והראו כי בדיקת RT-PCR לזיהום הליקובקטר פילורי בדגימות צואה הציגה ביצועי אבחון יוצאי דופן24. באופן ספציפי, הבדיקה השיגה רגישות של 99.1%, סגוליות של 100% ודיוק אבחנתי של 99.1%, אשר התיישרו באופן הדוק עם תוצאות מדגימות ביופסיה של הקיבה (93.9%) (Kappa = 0.929, p < 0.001). הסכמה יוצאת דופן זו הדגישה כי ניתן להתייחס לבדיקת RT-PCR כשיטה לא פולשנית אמינה ביותר.
יתר על כן, המחקר גם העריך את היכולת של RT-PCR צואתי באיתור עמידות לאנטיביוטיקה בהליקובקטר פילורי, תוך התמקדות במיוחד בעמידות לקלריתרומיצין ולבופלוקסצין. עבור עמידות לקלריתרומיצין, זוהו 43 מקרים (37.3%) בדגימות צואה, עם רגישות של 79.6%, סגוליות של 98.4% ודיוק אבחנתי של 78.0% (Kappa = 0.788, p < 0.001). באופן דומה, עבור עמידות ללבופלוקסצין, זוהו 37 מקרים (32.1%), הניבו רגישות של 86.3%, סגוליות של 91.1% ודיוק אבחנתי של 74.4% (קאפה = 0.739, עמ ' < 0.001). ממצאים אלה היו עקביים מאוד עם אלה שנלקחו מדגימות ביופסיה של הקיבה (עמידות לקלריתרומיצין ב-54 מקרים, עמידות ללבופלוקסצין ב-36 מקרים), מה שמצביע על כך שבדיקת RT-PCR בצואה יעילה באיתור עמידות לאנטיביוטיקה, בעלת סגוליות גבוהה ורגישות מקובלת לשתי האנטיביוטיות. התיקוף של מתודולוגיה זו במחקר הקודם תומך עוד יותר במהימנותה וביישומה בהקשר של מאמר זה.
בעוד שפרוטוקול זיהוי הצואה המבוסס על qPCR מייצג התקדמות ניכרת בתחום האבחון הלא פולשני של הליקובקטר פילורי, מספר מגבלות מחייבות התייחסות קפדנית. ראשית, שימוש בדגימות צואה גורם לשיטה להיות מושפעת מאיכות הדגימה. שליליות כוזבות עלולות לנבוע מעומסי חיידקים נמוכים בצואה, במיוחד אצל נשאים אסימפטומטיים או חולים עם זיהומים בשלב מוקדם, כאשר שפיכת הליקובקטר פילורי למערכת העיכול היא לסירוגין25. יתר על כן, בעת לקיחת דגימות צואה, כמות החיידקים יכולה להשתנות בהתאם למקום שבו נלקחת הדגימה. זאת בניגוד ניכר לשיטות פולשניות, כגון תרבית ביופסיית קיבה, הדוגמות ישירות את רירית הקיבה ולכן מושפעות פחות מתנודות בעומס החיידקים. שנית, למרות שגישת ההגברה הממוקדת של הפרוטוקול היא ספציפית, היא עלולה להיכשל בזיהוי מוטציות עמידות נדירות או מתפתחות שאינן משולבות בתכנון הפריימר, מה שעלול להוביל לפרופילי התנגדות לא שלמים26.
מחקר עתידי צריך להתמקד בהתקדמות מתודולוגית לשיפור יכולות זיהוי המוטציות, כולל אימוץ PCR דיגיטלי (dPCR) לזיהוי משופר של מוטציות עמידות בשפע נמוך27. לוחות בדיקה דורשים הרחבה אסטרטגית כדי להקיף פולימורפיזמים רלוונטיים מבחינה קלינית. ביצוע מחקרים רב-מרכזיים להערכה השוואתית של מתודולוגיות אבחון - כולל UBT, בדיקת PCR ברירית הקיבה או בצואה וזיהוי אנטיגן - מספק נתוני ביצועי אבחון מקיפים לפרקטיקה קלינית, ובכך מקדם את הסטנדרטיזציה של פרוטוקולי בדיקת PCR בצואה. שיפורים משולבים אלה יחזקו את התועלת של qPCR צואתי ככלי אבחון לא פולשני, תוך שהם עדיין חשובים להנחיית טיפול אנטיביוטי מדויק.
Disclosures
המחברים מצהירים שאין להם אינטרסים מתחרים.
Acknowledgements
מחקר זה מומן על ידי קרן המחקר לכישרונות מתקדמים של בית החולים העממי של מחוז גואנגדונג [מענק מס. KY012023293]. עבודה זו נתמכה על ידי Jiangsu Mole Bioscience Co. למממנים לא היה כל תפקיד בתכנון המחקר, באיסוף הנתונים ובניתוחם, בהחלטה לפרסם או בהכנת כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
References
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved