Method Article
通过粪便定量聚合酶链反应分析检测 幽门螺杆菌 感染和抗生素耐药性
* 这些作者具有相同的贡献
摘要
该方案采用无创粪便采样结合定量聚合酶链反应,为 幽门螺杆菌 感染及其对克拉霉素和喹诺酮类药物的耐药性提供了一种方便快速的诊断方法。
摘要
幽门螺杆菌 (H. pylori) 在世界范围内广泛流行,全球约 50% 的人口有幽门螺杆菌感染史。在中国,感染率从 40% 到 70% 不等。幽门螺杆菌主要与胃肠道疾病有关,例如慢性胃炎、胃溃疡和十二指肠溃疡。目前,幽门螺杆菌感染的临床治疗包括三联或四联疗法。然而,抗生素的广泛使用导致幽门螺杆菌产生抗生素耐药性。因此,检测幽门螺杆菌及其抗生素耐药性对于指导临床治疗至关重要。
幽门螺杆 菌 的诊断方法包括尿素呼气试验 (UBT)、抗原检测、血清学抗体检测、内窥镜检查、快速尿素酶试验 (RUT) 和细菌培养。虽然前三种方法是非侵入性的,但它们不允许细菌回收,因此不能用于耐药性测试。后三种方法侵入性大、价格昂贵、需要高技术专长,并且可能对患者造成伤害。
因此,一种同时检测 幽门螺杆菌 感染和抗生素耐药性的无创快速方法对于有效根除 幽门螺杆菌具有最重要的临床意义。本文旨在介绍一种将定量聚合酶链反应 (qPCR) 与 TaqMan 荧光探针技术相结合以快速检测 幽门螺杆菌 感染和抗生素耐药性的特定方案。与传统的细菌培养和其他技术不同,这种方法提供了一种方便、快速和非侵入性的方法来诊断 幽门螺杆菌 感染和耐药性。qPCR 用于鉴定感染并检测 23S rRNA 和 gyrA 基因的突变,它们分别与对克拉霉素和喹诺酮类药物的耐药性有关。与传统培养技术相比,这种方法为检测 幽门螺杆菌 感染和确定其抗生素耐药性提供了一种无创、经济高效且省时的方法。
引言
幽 门螺杆菌是一种革兰氏阴性螺旋形细菌,可持续感染人胃上皮1。1994 年,世界卫生组织将 幽门螺杆菌 归类为胃癌的 1 类致癌物,~3% 的感染者最终会患上这种疾病2。最近发表的一项系统评价表明,在过去十年中观察到幽 门螺杆菌 抗生素耐药率增加的总体趋势,在全球范围内已达到令人震惊的水平,尤其是克拉霉素耐药性3。在中国,最新的调查数据显示, 幽门螺杆菌 在中国城市人群中的总患病率为 27.08%;克拉霉素和左氧氟沙星的耐药率分别为 50.83% 和 47.17%4,高于美国(左氧氟沙星 37.6%、克拉霉素 31.5%)5和欧洲(左氧氟沙星 15.8% 和克拉霉素 21.4%)6。因此,早期和准确的诊断和治疗至关重要。然而, 幽门螺杆菌 日益增长的抗生素耐药性大大降低了治疗效果,凸显了对其诊断和治疗进行持续研究的迫切需求。
幽门螺杆菌诊断方法分为侵入性和非侵入性技术7。侵入性方法包括组织病理学检查、快速尿素酶试验 (RUT) 和细菌培养8。组织病理学检查依赖于活检样本的处理和显微镜观察,准确性受组织学准备和病理学专业知识的限制9。RUT 通过脲酶的活性检测幽门螺杆菌,脲酶水解尿素产生氨,导致试剂发生碱性变化,从而表明细菌的存在9。它简单且具有成本效益。细菌培养被认为是“金标准”,由于幽门螺杆菌10 独特的生理特性,成功率通常低于 50%。
非侵入性方法包括 13C/14C 尿素呼气试验 (UBT)、血清学诊断和粪便抗原检测 (SAT)11。呼气试验的工作原理类似于 RUT,检测呼出空气中同位素标记的 CO2 以确认感染12。血清学诊断可检测到抗体,这些抗体在细菌根除后可能会持续存在,因此难以评估治疗效果13。粪便抗原检测可检测粪便样本中的 幽门螺杆菌特异性抗原,提供了一种可靠的非侵入性方法,假阳性率较低,诊断活动性感染的准确性高14。
每种诊断方法都有其优点和局限性15。除了细菌培养外,其他方法难以检测抗生素耐药性,而培养则因成功率低而受到阻碍16。最近,实时定量 PCR (qPCR) 等分子诊断方法已广泛应用于微生物检测17。qPCR 可以准确检测 幽门螺杆菌 并分析耐药基因突变,提供更全面的感染视图18。
荧光探针设计用于在 qPCR 过程中特异性结合 DNA 中的靶序列。这些探针通常用荧光染料标记,当探针与其靶标结合并在扩增过程中被聚合酶裂解时,荧光染料会发出信号。这提供了 PCR 过程的实时测量。荧光探针因其独特的探针设计而以其高特异性而闻名,可确保仅检测靶 DNA 序列,从而减少非特异性结合的机会并提高检测特异性。这使得荧光探针技术特别适用于检测低丰度靶标,例如粪便样本中的 幽门螺杆菌 DNA。虽然 SYBR Green 是 PCR 中常用的 DNA 结合染料,但它可以与任何双链 DNA 结合,这可能导致非特异性扩增和假阳性。
从临床样本中培养 幽门 螺杆菌并不能直接提供抗生素耐药性数据。相比之下,基于荧光探针的 qPCR 更快、更方便,并且可以适应同时检测 幽门螺杆菌 和抗生素耐药性标志物。该方法不仅比传统培养方法更快地获得结果,而且可以同时检测抗生素耐药基因,大大提高了检测效率和便利性。与传统的细菌培养和其他技术不同,这种方法提供了一种方便、快速和非侵入性的方法来诊断 幽门螺杆菌 感染和耐药性。
幽门螺杆菌抗生素耐药性需要考虑两个关键问题:i) 基因型和表型的一致性,ii) 多药耐药性。中国的一项大规模多中心研究发现,克拉霉素/左氧氟沙星的双重耐药模式为 26.1%。克拉霉素和左氧氟沙星耐药的幽门螺杆菌表型和基因型显示出令人满意的一致性(kappa 系数分别为 = 0.810 和 0.782)19。因此,使用 qPCR 方法检测幽门螺杆菌感染和耐药是可行的。由于采样的便利性,粪便抗原检测 (SAT) 和 qPCR 方法被广泛使用。幽门螺杆菌检测方法的灵敏度、特异性和准确性如下:qPCR > UBT > SAT > RUT>CagA IgG >培养20。所有非侵入性方法都适用于初步筛查。由于 qPCR 还可以检测耐药基因,因此 qPCR 和培养将更适合指导治疗。
其他创新方法,如人工智能 (AI) 算法结合内窥镜图像21、数字微流控22 和 RPA-CRISPR/Cas12a23,理论上显示出良好的诊断效率和广阔的应用前景。然而,qPCR 是目前最复杂的分子生物学技术,有可能为幽 门螺杆菌 感染的诊断和治疗提供卓越的解决方案。
研究方案
本研究遵循中国广州南方医科大学广东省人民医院伦理委员会制定的伦理指南(批准号:KY2024-445-01)。有关本研究中使用的材料(试剂、化学品、设备和软件)的详细信息,请参阅 材料表。
1. 参与者选择
- 选择 18 至 60 岁年龄段的参与者。
- 选择 可能接受幽 门螺杆菌感染筛查的参与者,例如出现胃炎、消化性溃疡或消化不良等症状的患者,以及无症状的个体。
- 将过去一个月内使用过抗生素、含铋剂(如柠檬酸铋钾胶囊、胶体铋果胶胶囊或铋铝复方片)或抗菌中药的患者排除在研究之外,或在过去 2 周内使用过质子泵抑制剂或 H2 受体拮抗剂(如奥美拉唑、泮托拉唑、雷贝拉唑、西咪替丁或尼扎替丁)的患者。还将月经期间的女性排除在研究之外。
2. 粪便样本的采集
注意:提供第 2 部分作为参与者的说明。
- 确认采样套件的外包装未损坏且在有效期内。从套件中取出材料。
- 将白色粪便收集纸放入马桶中并排便(以避免尿液污染)。
- 使用抹刀收集一部分大约蚕豆大小 (~5 g) 的粪便,并将其转移到样品容器中。
- 将采样刮刀和粪便样本一起放入收集管中,确保液位上升到管上指示的采样线附近。
- 拧紧管帽。将试管倒置数次,以确保样品与防腐剂溶液充分混合。
- 将样品冷藏保存,直到运送到临床实验室进行以下幽门螺杆 菌鉴定并通过 qPCR 评估抗生素耐药性概况。
3. 核酸提取
注:在生物安全柜内完成所有程序,以避免污染。
- 倒置标本保存管并充分混合。将 1.0 mL 粪便标本溶液转移到微量离心管中。
- 混合离心管后,将它们置于 80 °C 的金属浴中 10 分钟,第 5 分钟间歇混合 30 秒。一旦试管冷却至室温,以 10,000 × g 离心 5 分钟并收集上清液用于进一步处理。
- 多次倒置 96 孔板以重悬磁珠。小心地取下铝箔密封,注意避免晃动以防止溢出。
- 将 200 μL 制备的样品(来自步骤 3.2)添加到含有裂解缓冲液的 96 孔板的每个孔中,确保每个孔对应一个样品。将 96 孔板放入核酸提取仪的指定样品室中进行自动提取。
- 将任何剩余的标本和提取的核酸样品储存在 -20 °C 下,以备长期保存和将来使用。
4. 幽 门螺杆菌 核酸和克拉霉素和喹诺酮类药物耐药突变的 qPCR 检测
- 从试剂储存区取出试剂,并在室温下解冻。
注:该试剂盒用于定性检测 幽门螺杆菌 核酸,包括 23S rRNA 基因(A2143G、A2143C、A2144G)和 gyrA 基因(261A、261G、260T、271A、271T、272G)的突变。试剂盒中提供引物、探针、Taq 酶、UNG 酶和 dNTP。幽门 螺杆菌 和人 β-肌动蛋白的靶基因被纳入阳性质量控制。选择人β-actin 基因作为内部质量控制。 - 根据要检测的样品数量,使用 N + 2 份冻干试剂,其中 N 代表要检测的样品数量,2 代表阴性和阳性 QC。
- 短暂离心以确保冻干粉末沉淀在试管底部。打开冻干试剂的盖子(注意避免粉末溢出),每孔添加 25 μL 从待测样品中提取的核酸,以及阳性和阴性对照。拧紧管子。
- 将 PCR 试剂涡旋 8-10 秒,然后短暂离心 3-5 秒以避免形成气泡。
- 将 96 孔 qPCR 板放在 qPCR 机上。循环程序设置如下:首先,将反应混合物(包含引物、探针、Taq 聚合酶、UNG 酶、dNTP 等)在 42 °C 和 95 °C 下孵育 2 分钟(一个循环中的两个步骤);然后,循环 10 次,在 95 °C 下循环 10 秒,在 65 °C 下循环 45 秒;最后,循环 35 倍,在 95 °C 下循环 10 秒,在 58 °C 下循环 45 秒,以进行变性、退火和延伸。
- 设置荧光信号检测参数如下: 幽门螺杆菌 保守基因的 ROX 荧光标记; 幽门螺杆菌 克拉霉素耐药基因的 FAM 荧光标记; 幽门螺杆菌 喹诺酮类药物耐药基因的 HEX 荧光标记;和人 β-肌动蛋白基因的 CY5 荧光标记用作试剂盒的内参。在 58 °C 下收集数据。 反应完成后,请确保保存数据以备将来分析。
- 使用 qPCR 专用软件分析数据,因为仪器会自动选择基线阈值。将所有诊断标准(包括幽门 螺杆菌 感染或耐药性的存在)设置为 CT 值 ≤30;确认它们表现出特征性的 S 形曲线。
结果
qPCR 检测粪便样本中幽门螺杆菌感染和抗生素耐药性的应用
我们根据 幽门螺杆菌保守基因以及 23S rRNA 基因和 gyrA 基因中的点突变设计了引物和探针。这些引物和探针用不同的荧光染料标记,然后用于 qPCR 检测。qPCR 实验的质量控制结果在推荐范围内,表明检测结果可靠(图 1)。在这项研究中,我们选择了五个代表性样品 (S1-S5) 来表征实验方案的可靠性 (图 2 和 表 1)。S1 是没有 幽门螺杆菌 感染的样本,而 S2-S5 是具有不同耐药性特征的样本。
对于 幽门螺杆 菌感染阳性的样品,样品 S2 显示 CT 值仅在幽 门螺杆菌的检测范围内,表明该样品对幽门螺杆 菌 呈阳性,并且对克拉霉素和喹诺酮类药物都敏感,可以使用任何一种药物治疗。样本 S3 显示 CT 值在幽门螺杆 菌 感染和克拉霉素耐药的检测范围内,未检测到喹诺酮类耐药的 CT 值,表明样本 S3 来自对克拉霉素耐药的患者。同样,样本 S4 的 CT 值在 幽门螺杆菌 感染和喹诺酮类药物耐药的检测范围内,未检测到克拉霉素耐药的 CT 值,表明对喹诺酮类药物耐药并推荐克拉霉素治疗。最后,样品 S5 在所有测试中均具有可检测的 CT 值,表明样品对幽门 螺杆菌 呈阳性,并且对克拉霉素和喹诺酮类药物表现出双重耐药性;因此,临床医生应考虑替代治疗方案。与其他幽门 螺杆 菌检测方法相比,这种方法不仅有助于样品采集,还可以同时检测 幽门螺杆 菌及其耐药性,提供可靠的结果来指导适当的治疗决策。
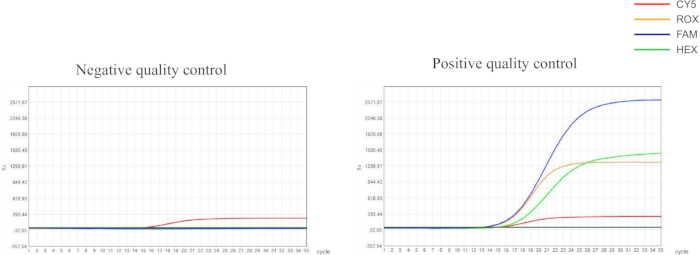
图 1:阴性对照和阳性对照的质量控制结果。 阴性对照应显示 CY5 检测通道的 S 形生长曲线,CT 值为 30.00≤;FAM、HEX 和 ROX 通道的荧光信号不应显示显著增加,CT 值> 30.00 或没有显著信号;阳性对照应在 CT 值为 30.00 的所有检测通道中呈现 S 形曲线≤。 请单击此处查看此图的较大版本。

图 2:通过 qPCR 检测粪便样本中的幽门螺杆菌及其抗生素耐药性。 每种颜色代表一个荧光信号,不同的荧光信号代表不同的基因。荧光通道:幽门螺杆菌保守基因的 ROX 荧光标记(橙色);幽门螺杆菌克拉霉素抗性基因的 FAM 荧光标记(蓝色);幽门螺杆菌喹诺酮类药物耐药基因的 HEX 荧光标记(绿色);内部对照基因的 CY5 荧光标记 (红色)。样本表型:S1 是幽门螺杆菌感染的阴性样本(无高于阈值的信号);S2 是幽门螺杆菌阳性样本,无抗生素耐药性 (ROX 信号高于阈值);S3 是幽门螺杆菌阳性样品,对克拉霉素具有抗性 (FAM 信号高于阈值);S4 是幽门螺杆菌阳性样品,对喹诺酮类药物具有抵抗力 (HEX 信号高于阈值);S5 是一种幽门螺杆菌阳性样品,对克拉霉素和喹诺酮类药物均耐药 (FAM 和 HEX 信号高于阈值)。请单击此处查看此图的较大版本。
| 样本 | 幽门螺杆菌 (ROX) | 克拉霉素 (FAM) | 喹诺酮类药物 (HEX) | CY5 系列 | |||
| +/- | 电脑断层扫描 | +/- | 电脑断层扫描 | +/- | 电脑断层扫描 | 电脑断层扫描 | |
| 第 1 季 | - | - | - | - | - | - | 9.96 |
| 第 2 季 | + | 23.08 | - | - | - | - | 12.66 |
| 第 3 页 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| 第 4 季 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| 第 5 季 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| 消极的 | - | - | - | - | - | - | 14.73 |
| 阳性 | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
表 1: 幽门螺杆菌 感染检测和克拉霉素和喹诺酮类药物耐药的 qPCR 结果。 该表提供了 幽门螺杆 菌感染的定性结果。检测到 23S rRNA 基因突变表明克拉霉素耐药,而 gyrA 基因突变表明喹诺酮类药物耐药。符号:+/−,定性结果;+,阳性结果;−,则结果为阴性。
讨论
近年来,分子检测方法在微生物学领域得到了广泛的应用,显著改变了几种传染病的临床管理。这些方法在基因水平上运行,不仅可以确认细菌的存在,还可以进行基因分型和抗生素耐药性测试。实时荧光定量 PCR (qPCR) 因其处理时间短、灵敏度高、准确性高、交叉污染风险低而受到越来越青睐。它已广泛用于临床实验室诊断 幽门螺杆菌 感染。
将粪便样本采集保存溶液与基因检测方法相结合,可以通过分析克拉霉素 23S rRNA 基因和喹诺酮类 gyrA 基因的突变,快速有效地筛选幽门螺杆菌,并进一步检测抗生素耐药性。该方法可以评估幽门螺杆菌的敏感性和对药物的耐药性。以前的粪便抗原检测方法存在明显的局限性,例如由于样品中的抗原浓度低和抗原降解而导致准确性和稳定性降低。此外,传统的抗原检测方法在复杂样本中可能缺乏足够的灵敏度和特异性,从而影响结果的可靠性。
为了解决这些问题,我们提出了一种将粪便样本与定量聚合酶链反应 (qPCR) 技术相结合的方法。这种方法显著提高了检测灵敏度和准确性,使粪便检测更加可靠和高效,并为临床应用提供更高质量的结果。
质控标准品对于 qPCR 检测至关重要。我们的质量控制包括仪器的定期校准、检测系统的性能验证、核酸提取效率的评价以及每批实验的质量控制。首先,我们的采样仪器每 6 个月校准一次,提取设备和扩增设备每年校准一次。此外,对每批试剂进行校准。校准后,我们可以验证检测系统的性能,包括 Macrostone 96S 荧光定量 PCR 仪器、 幽门螺杆菌 和耐药突变检测试剂盒、提取试剂以及相应的采样管、移液器和吸头。合格率、检出限、交叉反应、抗干扰能力、精度满足我们的需求。通过 DNA 浓度测试仪测定 DNA 产物的浓度,以满足核酸提取效率的可接受标准。DNA 浓度高于 10 ng/μL。A260/A280 在 1.6 到 2.0 之间。不合格的样品必须重新提取。只有在日常质量控制得到控制时,结果才有效。对于 幽门螺杆菌 和抗生素耐药性检测(克拉霉素和喹诺酮类),质量控制标准如下:阴性对照应显示 CY5 检测通道的 S 形生长曲线,CT 值为 30.00≤;FAM、HEX 和 ROX 通道的荧光信号不应显示显著增加,CT 值为 >30.00 或没有显著信号;阳性对照应在 CT 值为 30.00 的所有检测通道中呈现 S 形曲线≤。这些条件必须在同一实验中满足;否则,该测试将被视为无效,必须重新测试。请注意,由于商业专有限制,本文无法提供引物和探针序列信息。
在之前的一项研究中,研究人员采用了与本文中使用的方法相同的方法,证明粪便样本中幽门螺杆菌感染的 RT-PCR 检测表现出卓越的诊断性能24。具体来说,该测试实现了 99.1% 的敏感性、100% 的特异性和 99.1% 的诊断准确性,这与胃活检样本的结果 (93.9%) 非常一致 (Kappa = 0.929,p < 0.001)。 这一非凡的协议强调了 RT-PCR 检测可以被视为一种高度可靠的非侵入性方法。
此外,该研究还评估了粪便 RT-PCR 检测幽门螺杆菌抗生素耐药性的能力,特别关注克拉霉素和左氧氟沙星耐药性。对于克拉霉素耐药,在粪便样本中鉴定出 43 例 (37.3%),敏感性为 79.6%,特异性为 98.4%,诊断准确性为 78.0% (Kappa = 0.788,p < 0.001)。 同样,对于左氧氟沙星耐药性,检测到 37 例 (32.1%),敏感性为 86.3%,特异性为 91.1%,诊断准确性为 74.4% (Kappa = 0.739,p < 0.001)。 这些发现与胃活检样本得出的结果高度一致 (54 例克拉霉素耐药,36 例左氧氟沙星耐药),表明粪便 RT-PCR 检测可有效检测抗生素耐药性,对两种抗生素均具有高特异性和可接受的敏感性。在之前的研究中对该方法的验证进一步支持了其在本文中的可靠性和适用性。
虽然基于 qPCR 的粪便检测方案代表了 幽门螺杆菌无创诊断领域的显着进步,但有几个限制需要仔细考虑。首先,使用粪便样品会使方法受到样品质量的影响。假阴性可能是由于粪便中的细菌载量低造成的,特别是在无症状携带者或早期感染患者中, 幽门螺杆菌 脱落到胃肠道是间歇性的25。此外,在采集粪便样本时,细菌的数量可能会因样本采集地点而异。这与侵入性方法(例如胃活检培养)形成鲜明对比,后者直接对胃粘膜进行采样,因此受细菌载量波动的影响较小。其次,尽管该方案的靶向扩增方法是特异性的,但它可能无法检测到未纳入引物设计的罕见或新出现的耐药突变,这可能导致耐药谱不完整26。
未来的研究应侧重于方法学进步,以提高突变检测能力,包括采用数字 PCR (dPCR) 来改进对低丰度耐药突变的鉴定27。探针板需要战略扩展以包含临床相关的多态性。开展多中心研究以比较评估诊断方法(包括 UBT、胃粘膜或粪便 PCR 检测和抗原检测)为临床实践提供了全面的诊断性能数据,从而推进粪便 PCR 检测方案的标准化。这些综合改进将加强粪便 qPCR 作为非侵入性诊断工具的有用性,同时对于指导精确的抗生素治疗仍然很重要。
披露声明
作者声明他们没有利益争夺。
致谢
本研究由广东省人民医院先进人才研究基金资助 [Grant No.KY012023293]。这项工作得到了江苏莫尔生物科技有限公司的支持。资助者在研究设计、数据收集和分析、发表决定或手稿准备方面没有任何作用。
材料
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
参考文献
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。