Method Article
便定量ポリメラーゼ連鎖反応解析による ヘリコバクター・ピロリ 菌感染と抗生物質耐性の検出
* これらの著者は同等に貢献しました
要約
このプロトコルは、定量的ポリメラーゼ連鎖反応と組み合わせた非侵襲的な便サンプリングを採用して、 ヘリコバクターピロリ 感染とそのクラリスロマイシンおよびキノロンに対する耐性の便利で迅速な診断方法を提供します。
要約
ヘリコバクター・ピロリ菌(H. pylori)は世界中で広く蔓延しており、世界人口の約50%がピロリ菌に感染した経験があります。中国では、感染率は40%から70%の範囲です。ピロリ菌は、主に慢性胃炎、胃潰瘍、十二指腸潰瘍などの胃腸疾患に関連しています。現在、ピロリ菌感染症の臨床治療には、三重または四重療法が含まれます。しかし、抗生物質の広範な使用により、ピロリ菌の抗生物質耐性が発症しました。したがって、ピロリ菌とその抗生物質耐性の両方を検出することは、臨床治療を導くために重要です。
ピロリ菌の診断方法には、尿素呼気検査(UBT)、抗原検査、血清学的抗体検査、内視鏡検査、迅速ウレアーゼ検査(RUT)、および細菌培養が含まれます。最初の3つの方法は非侵襲的ですが、細菌の回復を許さないため、耐性試験には使用できません。後者の3つの方法は、侵襲的で高価であり、高度な技術的専門知識を必要とし、患者に害を及ぼす可能性があります。
したがって、 ピロリ菌 感染と抗生物質耐性を同時に検出するための非侵襲的で迅速な方法は、 ピロリ菌の効果的な根絶にとって臨床的に最も重要です。この論文は、定量的ポリメラーゼ連鎖反応(qPCR)と TaqMan 蛍光プローブ技術を組み合わせて、 ピロリ菌 感染と抗生物質耐性を迅速に検出する特定のプロトコールを紹介することを目的としています。この方法は、従来の細菌培養や他の技術とは異なり、 ピロリ菌 の感染と耐性を診断するための便利で迅速、かつ非侵襲的な方法を提供します。qPCRは、クラリスロマイシンとキノロンに対する耐性にそれぞれ関連する23S rRNA遺伝子と gyrA 遺伝子の感染を特定し、変異を検出するために使用されます。従来の培養技術と比較して、このアプローチは、 ヘリコバクター・ピロリ 菌感染を検出し、その抗生物質耐性を決定するための非侵襲的で費用対効果が高く、時間効率の良い方法を提供します。
概要
ヘリコバクター・ピロリ は、グラム陰性のらせん状の細菌で、ヒトの胃上皮1に持続的に感染する可能性があります。1994年、世界保健機関(WHO)はピ ロリ菌 を胃がんのグループ1発がん物質に分類し、最終的に感染者の~3%が発症しました2。最近発表されたシステマティックレビューでは、過去10年間に ピロリ菌 の抗生物質耐性率の増加の一般的な傾向が観察され、特にクラリスロマイシン耐性が世界的に驚くべきレベルに達していることが示されました3。中国では、最新の調査データによると、中国の都市部の人々の間で のピロリ菌 の総有病率は27.08%です。クラリスロマイシンおよびレボフロキサシンの耐性率は50.83%および47.17%4であり、米国(レボフロキサシン37.6%、クラリスロマイシン31.5%)5およびヨーロッパ(レボフロキサシン15.8%およびクラリスロマイシン21.4%)6よりも高くなっています。そのため、早期かつ正確な診断と治療が重要です。しかし、 ピロリ菌 の抗生物質耐性の増加は治療効果を大幅に低下させ、その診断と治療に関する継続的な研究が緊急に必要であることを強調しています。
ピロリ菌の診断方法は、侵襲的技術と非侵襲的技術に分類されます7。侵襲的な方法には、病理組織学的検査、迅速ウレアーゼ試験(RUT)、および細菌培養8が含まれます。病理組織学的検査は、生検サンプルの処理と顕微鏡観察に依存しており、組織学的調製と病理学の専門知識によって精度が制限されます9。RUTは、尿素を加水分解してアンモニアを生成するウレアーゼの活性を通じてピロリ菌を検出し、試薬にアルカリ性シフトをもたらすため、細菌9の存在を示します。シンプルで費用対効果が高いです。「ゴールドスタンダード」とされる細菌培養は、ピロリ菌10の特有の生理学的特性により、成功率が50%を下回ることがよくあります。
非侵襲的な方法には、13C/14C尿素呼気検査(UBT)、血清学的診断、および便抗原検査(SAT)11が含まれる。呼気検査はRUTと同様の原理で作動し、呼気中の同位体標識されたCO2 を検出し、感染を確認する12。血清学的診断では、細菌の根絶後も持続する可能性のある抗体が検出されるため、治療効果の評価が困難になります13。便抗原検査は、糞便サンプル中の ピロリ菌特異的抗原を検出し、偽陽性率が低く、活動性感染症の診断精度が高い信頼性の高い非侵襲的方法を提供します14。
各診断方法には、それぞれの利点と制限があります15。細菌培養以外にも、抗生物質耐性の検出に苦労している方法があり、培養の成功率の低さが阻害されている16。近年、リアルタイム定量PCR(qPCR)などの分子診断法が微生物検出に広く適用されている17。qPCRは 、ピロリ菌 を正確に検出し、耐性遺伝子の突然変異を解析することができ、感染のより包括的なビューを提供します18。
蛍光プローブは、qPCRプロセス中にDNAの標的配列に特異的に結合するように設計されています。これらのプローブは通常、蛍光色素で標識されており、プローブが標的に結合するとシグナルを発し、増幅中にポリメラーゼによって切断されます。これにより、PCRプロセスのリアルタイム測定が可能になります。蛍光プローブは、プローブのユニークな設計により、標的DNA配列のみが検出され、非特異的結合の可能性を減らし、アッセイ特異性を向上させるため、高い特異性で知られています。これにより、蛍光プローブ技術は、糞便サンプル中の ヘリコバクターピロリ DNAなどの微量ターゲットの検出に特に有用です。SYBR GreenはPCRで一般的に使用されるDNA結合色素ですが、任意の二本鎖DNAに結合できるため、非特異的な増幅や偽陽性が発生する可能性があります。
臨床サンプルから ヘリコバクター・ピロリ 菌を培養しても、抗生物質耐性のデータを直接得られるわけではありません。対照的に、蛍光プローブベースのqPCRは、より迅速で便利であり、 ピロリ菌 と抗生物質耐性マーカーの同時検出に適応できます。この方法は、従来の培養方法よりも迅速な結果が得られるだけでなく、抗生物質耐性遺伝子の同時検出も可能にし、検出効率と利便性を大幅に向上させます。この方法は、従来の細菌培養や他の技術とは異なり、 ピロリ菌 の感染と耐性を診断するための便利で迅速、かつ非侵襲的な方法を提供します。
ピロリ菌の抗生物質耐性については、i)遺伝子型と表現型の一貫性、ii)多剤耐性という2つの重要な問題を考慮する必要があります。中国での大規模な多施設研究では、クラリスロマイシン/レボフロキサシンの二重耐性パターンは26.1%であることがわかりました。クラリスロマイシン耐性およびレボフロキサシン耐性Hピロリの表現型および遺伝子型は、満足のいく一致を示した(κ係数=それぞれ0.810および0.782)19。したがって、qPCR法を使用してピロリ菌の感染と薬剤耐性を検出することは可能です。糞便抗原検査(SAT)やqPCR法は、サンプリングの利便性からますます利用されるようになっています。H. pylori 検出法の感度、特異性、および精度は、qPCR > UBT > SAT > RUT>CagA IgG >培養20 です。すべての非侵襲的方法は、一次スクリーニングに適しています。qPCRはさらに薬剤耐性遺伝子を検出できるため、qPCRと培養は治療の指導により適しています。
人工知能(AI)アルゴリズムと内視鏡画像21、デジタルマイクロ流体工学22、RPA-CRISPR/Cas12a23を組み合わせたその他の革新的なアプローチは、理論的には良好な診断効率と有望な応用の見通しを示しています。しかし、qPCRは現在、最も洗練された分子生物学的手法であり、ピロリ菌感染症の診断と治療に優れたソリューションを提供できる可能性があります。
プロトコル
この研究は、中国広州市南医科大学広東省人民病院の倫理委員会によって確立された倫理ガイドラインに準拠しています (承認番号: KY2024-445-01)。本研究で使用した材料(試薬、化学薬品、装置、ソフトウェア)の詳細については、 材料表をご覧ください。
1. 参加者の選考
- 18歳から60歳までの参加者をお選びください。
- 胃炎、消化性潰瘍、消化不良などの症状を呈する患者や無症状の個人など、 ヘリコバクターピロリ 感染スクリーニングを受ける可能性のある参加者を選択してください。
- 抗生物質、ビスマス含有薬剤 (クエン酸ビスマスカリウムカプセル、コロイド状ビスマスペクチンカプセル、ビスマス-アルミニウム化合物錠剤など)、または抗菌漢方薬を使用した患者、またはプロトンポンプ阻害剤またはH2受容体拮抗薬を使用した患者 過去 2 週間以内にオメプラゾール、パントプラゾール、ラベプラゾール、シメチジン、ニザチジンなど)。また、月経期間中の女性も研究から除外します。
2.糞便サンプルの収集
注:セクション2を参加者への指示として提供します。
- Sampリングキットの外装に損傷がなく、有効期限内であることを確認してください。キットから材料を取り出します。
- 便器に白い糞便収集紙を置き、排便します(尿による汚染を避けるため)。
- へらを使用して、ソラマメとほぼ同じサイズ(~5 g)の糞便の一部を収集し、サンプル容器に移します。
- サンプリングスパチュラを糞便サンプルと一緒に収集チューブに入れ、液面がチューブに示されているサンプリングラインの近くまで上昇することを確認します。
- チューブキャップをしっかりと締めます。チューブを数回反転させて、サンプルが防腐剤溶液と完全に混合されるようにします。
- サンプルは、 ピロリ菌 の次の同定とqPCRによる抗生物質耐性プロファイルの評価のために臨床検査室に配送されるまで冷蔵下で保管します。
3. 核酸抽出
注意: 汚染を避けるために、バイオセーフティキャビネット内ですべての手順を完了してください。
- 試料保存チューブを反転させ、十分に混合します。糞便検体溶液1.0mLを微量遠心チューブに移します。
- 遠心分離管を混合した後、80°Cの金属浴に10分間入れ、5分後に30秒間断続的に混合します。チューブが室温まで冷えたら、10,000 × g で5分間遠心分離し、上清を回収してさらに処理します。
- 96ウェルプレートを複数回反転させて、磁気ビーズを再懸濁します。アルミホイルシールは、こぼれないように揺れないように注意しながら、慎重に取り外します。
- 調製したサンプル(ステップ3.2)200 μLを、溶解バッファーを含む96ウェルプレートの各ウェルに加え、各ウェルが1つのサンプルに対応するようにします。96ウェルプレートを核酸抽出装置の指定サンプルコンパートメントに入れて、自動抽出を行います。
- 残った検体と抽出した核酸サンプルは、長期保存および将来の使用のために-20°Cで保管してください。
4. ヘリコバクター・ピロリ 核酸のqPCR検出とクラリスロマイシンおよびキノロンに対する耐性変異
- 試薬保管場所から試薬を取り出し、室温で解凍してください。
注:このキットは、23S rRNA遺伝子(A2143G、A2143C、A2144G)およびgyrA遺伝子(261A、261G、260T、271A、271T、272G)の突然変異を含む、H.ピロリ核酸の定性検出に使用されました。プライマー、プローブ、Taq酵素、UNG酵素、およびdNTPはキットに含まれていました。H.ピロリ菌とヒトβ-アクチンの標的遺伝子は、ポジティブ品質管理に含まれていました。ヒトβ-アクチン遺伝子を内部品質管理として選択しました。 - 試験するサンプルの数に基づいて、凍結乾燥試薬のN + 2コピーを使用します(Nは試験するサンプルの数、2は陰性および陽性のQCを表します)。
- 凍結乾燥粉末がチューブの底に沈殿するように、短時間遠心分離します。凍結乾燥試薬の蓋を開け(粉末がこぼれないように注意)、試験するサンプルから抽出した核酸のウェルあたり25μL、および陽性および陰性コントロールを加えます。チューブをしっかりと閉じます。
- PCR試薬を8〜10秒間ボルテックスし、次に気泡の形成を避けるために3〜5秒間短時間遠心分離します。
- 96ウェルqPCRプレートをqPCRマシンに置きます。サイクリングプログラムを次のように設定します:まず、反応混合物(プライマー、プローブ、Taqポリメラーゼ、UNG酵素、dNTPなどを含む)を42°Cおよび95°Cで2分間インキュベートします(両方のステップを1サイクルで)。次に、95°Cで10秒、65°Cで45秒で10xサイクルします。最後に、95°Cで10秒、58°Cで45秒で35倍サイクルを行い、変性、アニーリング、伸長を行います。
- 蛍光シグナル検出パラメータを次のように設定します: H.ピロリ 保存遺伝子のROX蛍光標識。 H. pylori clarithromycin 耐性遺伝子の FAM 蛍光標識; H.ピロリ キノロン耐性遺伝子のHEX蛍光標識;ヒト β-アクチン遺伝子のCY5蛍光標識をキットの内部コントロールとして使用しました。58°Cでデータを収集します。 反応が完了したら、データが将来の分析のために保存されていることを確認してください。
- qPCR専用のソフトウェアを使用してデータを解析すると、装置が自動的にベースライン閾値を選択します。 ヘリコバクター・ピロリ 菌の感染または耐性の存在を含むすべての診断基準をCT値≤30に設定します。それらが特徴的なS字型の曲線を示すことを確認します。
結果
糞便中のヘリコバクター・ピロリ感染と抗生物質耐性の検出のためのqPCRの応用
私たちは、 ヘリコバクター・ピロリ菌の保存遺伝子、23S rRNA遺伝子、 gyrA 遺伝子の点変異に基づいてプライマーとプローブを設計しました。これらのプライマーとプローブは、異なる蛍光色素で標識され、その後、qPCR検出に使用されました。qPCR 実験の品質管理結果は推奨範囲内にあり、信頼性の高い検出結果を示しました(図 1)。この研究では、実験プロトコルの信頼性を特徴付けるために、5つの代表的なサンプル(S1-S5)を選択しました(図2 および 表1)。S1は ピロリ菌 に感染していないサンプルですが、S2-S5は薬剤耐性プロファイルが異なるサンプルです。
ヘリコバクター・ピロリ菌感染症陽性の検体については、検体S2はヘリコバクター・ピロリ菌のみの検出範囲内のCT値を示し、この検体はヘリコバクター・ピロリ菌陽性であり、クラリスロマイシンとキノロンの両方に感受性があり、どちらの薬剤でも治療可能であることを示しています。サンプルS3は、ヘリコバクター・ピロリ菌感染とクラリスロマイシン耐性の両方について検出範囲内のCT値を示し、キノロン耐性についてはCT値は検出されなかったことから、サンプルS3はクラリスロマイシンに耐性のある患者からのものであることが示唆されました。同様に、サンプルS4のCT値はヘリコバクターピロリ感染とキノロン耐性の検出範囲内にあり、クラリスロマイシン耐性のCT値は検出されず、キノロンに対する耐性を示し、治療にクラリスロマイシンを推奨しました。最後に、サンプルS5はすべてのテストで検出可能なCT値を示し、サンプルがヘリコバクターピロリに対して陽性であり、クラリスロマイシンとキノロンの両方に対して二重の耐性を示すことを示しています。したがって、臨床医は代替治療の選択肢を検討すべきである。他のヘリコバクター・ピロリ検出法と比較して、このアプローチはサンプル取得を容易にするだけでなく、ヘリコバクター・ピロリとその耐性プロファイルを同時に検出できるため、適切な処理決定を導くための信頼性の高い結果が得られます。
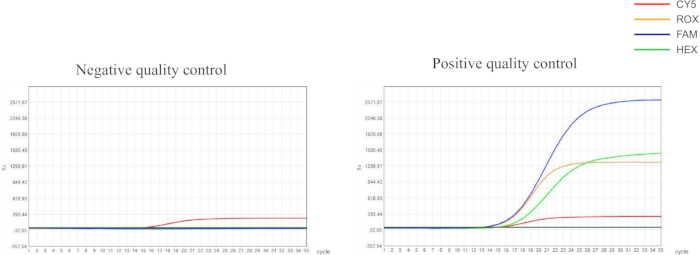
図1:ネガティブコントロールとポジティブコントロールの品質管理結果。 ネガティブコントロールは、CT値が30.00≤CY5検出チャネルのS字型成長曲線を表示する必要があります。FAM、HEX、およびROXチャンネルの蛍光シグナルは、CT値が30.00>か、顕著なシグナルがないため、有意な増加を示さないようにする必要があります。ポジティブコントロールは、CT値が30.00≤すべての検出チャネルでS字型の曲線を示す必要があります。 この図の拡大版を表示するには、ここをクリックしてください。

図2: qPCRによる糞便サンプル中のヘリコバクターピロリとその抗生物質耐性の検出。各色は蛍光シグナルを表し、異なる蛍光シグナルは異なる遺伝子を表します。蛍光チャンネル:ピロリ菌保存遺伝子のROX蛍光標識(オレンジ色)。H. pylori clarithromycin 耐性遺伝子の FAM 蛍光標識 (青);H.ピロリキノロン耐性遺伝子のHEX蛍光標識(緑)。内部制御遺伝子のCY5蛍光標識(赤)。サンプル表現型:S1はピロリ菌感染の陰性サンプルです(閾値を超えるシグナルはありません)。S2 は、抗生物質耐性 (ROX シグナルが閾値を超える) のないピロリ菌陽性サンプルです。S3 は、クラリスロマイシンに対する耐性 (閾値を超える FAM シグナル) を持つピロリ菌陽性のサンプルです。S4は、キノロン系薬剤(閾値を超えるHEXシグナル)に対する耐性を持つピロリ菌陽性のサンプルです。S5は、クラリスロマイシンとキノロンの両方(閾値を超えるFAMおよびHEXシグナル)に耐性のあるピロリ菌陽性サンプルです。この図の拡大版を表示するには、ここをクリックしてください。
| 見本 | H.ピロリ(ROX) | クラリスロマイシン(FAM) | キノロン(HEX) | CY5 | |||
| +/- | CT検査 | +/- | CT検査 | +/- | CT検査 | CT検査 | |
| S1の | - | - | - | - | - | - | 9.96 |
| S2の | + | 23.08 | - | - | - | - | 12.66 |
| S3の | + | 19.58 | + | 22.41 | - | - | 11.4 |
| S4の | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5の | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| ネゲティブ | - | - | - | - | - | - | 14.73 |
| + | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
表1: クラリスロマイシンおよびキノロン耐性に対するピロリ菌感染検出およびqPCRの結果。この表は、ピロリ菌感染の定性的結果を示しています。23S rRNA遺伝子変異の検出はクラリスロマイシン耐性を示し、gyrA遺伝子変異の検出はキノロン耐性を示します。記号:+/-、定性的結果;+、肯定的な結果。−、陰性結果。
ディスカッション
近年、分子検出法は微生物学の分野で広く適用されており、いくつかの感染症の臨床管理を大きく変えています。これらの方法は遺伝子レベルで機能するため、細菌の存在を確認するだけでなく、遺伝子タイピングや抗生物質耐性試験も可能になります。リアルタイム蛍光定量PCR(qPCR)は、処理時間が短く、感度と精度が高く、クロスコンタミネーションのリスクが低いため、ますます好まれています。 ヘリコバクター・ピロリ 感染症の診断のために臨床検査室で広く使用されています。
糞便サンプル採取保存溶液と遺伝子検出法を組み合わせることで、 ピロリ菌 の迅速かつ効率的なスクリーニングが可能になり、クラリスロマイシン23S rRNA遺伝子とキノロン gyrA 遺伝子の変異の解析によるさらなる抗生物質耐性試験が可能になります。この方法では、 ピロリ菌 の感受性と薬剤に対する耐性を評価できます。従来の糞便抗原検出法では、抗原濃度が低いことや検体中の抗原分解により精度や安定性が低下するなど、顕著な制約がありました。さらに、従来の抗原検出法では、複雑なサンプルに対して十分な感度と特異性が欠けており、結果の信頼性に影響を及ぼす可能性があります。
これらの課題に対して、糞便サンプルと定量的ポリメラーゼ連鎖反応(qPCR)技術を組み合わせた方法を提案します。このアプローチにより、検出感度と精度が大幅に向上し、糞便検査の信頼性と効率が向上し、臨床使用のためのより高品質な結果が得られます。
品質管理基準は、qPCR検出に不可欠です。当社の品質管理には、機器の定期的な校正、検出システムの性能検証、核酸抽出効率の評価、および各実験バッチの品質管理が含まれます。まず、サンプリング機器は6か月ごとに校正され、抽出装置と増幅装置は毎年校正されます。さらに、試薬のバッチごとにキャリブレーションが行われます。キャリブレーション後、Macrostone 96S 蛍光定量 PCR 装置、 ピロリ菌 および耐性変異検出キット、抽出試薬、対応するサンプリングチューブ、ピペット、チップなどの検出システムの性能を検証できます。適合率、検出限界、交差反応、干渉防止能力、および精度は、当社のニーズを満たしています。DNA産物の濃度は、核酸抽出効率の許容基準を満たすために、DNA濃度テスターによって決定されました。DNA濃度は10 ng/μLを超えていました。A260/A280は1.6から2.0の間でした。不適格なサンプルは再抽出する必要があります。結果は、日々の品質管理が管理されている場合にのみ有効です。 ピロリ菌 および抗生物質耐性試験(クラリスロマイシンおよびキノロン)の場合、品質管理基準は次のとおりです:ネガティブコントロールは、CT値が30.00≤CY5検出チャネルのS字型成長曲線を表示する必要があります。FAM、HEX、およびROXチャネルの蛍光シグナルは、CT値が>30.00であるか、顕著なシグナルがない場合、有意な増加を示さないようにする必要があります。ポジティブコントロールは、CT値が30.00≤すべての検出チャネルでS字型の曲線を示す必要があります。これらの条件は、同じ実験内で満たす必要があります。それ以外の場合、テストは無効と見なされ、再テストする必要があります。商業的な所有権の制約により、プライマーおよびプローブ配列の情報は本論文で提供できないことに注意してください。
以前の研究では、研究者はこの論文で利用されたものと同じ方法論を採用し、糞便サンプル中の ピロリ菌 感染のRT-PCR検査が優れた診断性能を示したことを実証しました24。具体的には、感度99.1%、特異度100%、診断精度99.1%を達成し、胃生検体の結果(93.9%)と密接に一致しました(Kappa = 0.929、 p < 0.001)。この注目すべき合意は、RT-PCR検査が信頼性の高い非侵襲的方法と見なすことができることを強調しました。
さらに、この研究では、特にクラリスロマイシンとレボフロキサシンの耐性に焦点を当てて、 ピロリ菌の抗生物質耐性を検出する糞便RT-PCRの能力も評価しました。クラリスロマイシン耐性については、糞便サンプル中に43例(37.3%)が同定され、感度は79.6%、特異度は98.4%、診断精度は78.0%でした(Kappa = 0.788、 p < 0.001)。同様に、レボフロキサシン耐性についても、37例(32.1%)が検出され、感度は86.3%、特異度は91.1%、診断精度は74.4%(Kappa = 0.739、 p < 0.001)であった。これらの所見は、胃生検体から得られた知見(クラリスロマイシン耐性54例、レボフロキサシン耐性36例)と非常に一致しており、糞便RT-PCR検査が抗生物質耐性の検出に有効であり、両方の抗生物質に対して高い特異性と許容感度を有することを示しました。先行研究におけるこの方法論の検証は、本論文の文脈におけるその信頼性と適用性をさらに裏付けている。
qPCRに基づく糞便検出プロトコルは、 ピロリ菌の非侵襲的診断の分野における顕著な進歩を表していますが、いくつかの制限により細心の注意を払う必要があります。まず、糞便サンプルを使用すると、サンプルの品質に影響されます。偽陰性は、糞便中の細菌量が少ないために生じる可能性があり、特に無症候性キャリアまたは初期感染患者では、胃腸管への ピロリ菌 の排出が断続的です25。また、糞便サンプルを採取する場合、サンプルを採取する場所によって細菌の量が異なることがあります。これは、胃粘膜を直接サンプリングするため、細菌負荷の変動による影響が少ない胃生検培養などの侵襲的な方法とは著しく対照的です。第二に、プロトコルの標的増幅アプローチは特異的であるが、プライマーデザインに組み込まれていないまれなまたは新たな耐性変異を検出できない可能性があり、不完全な耐性プロファイル26につながる可能性がある。
今後の研究では、低存在量耐性変異の同定を改善するためのデジタルPCR(dPCR)の採用など、突然変異検出能力を強化するための方法論の進歩に焦点を当てるべきである27。プローブパネルは、臨床的に関連する多型を網羅するための戦略的な拡張が必要です。UBT、胃粘膜または糞便PCR検査、抗原検出などの診断方法論を比較評価するための多施設研究を実施することで、臨床診療のための包括的な診断性能データが提供され、便PCR検査プロトコルの標準化が進んでいます。これらの改善を組み合わせることで、糞便qPCRの非侵襲的診断ツールとしての有用性が強化されると同時に、正確な抗菌薬治療を導くためにも重要である。
開示事項
著者は、競合する利益がないことを宣言します。
謝辞
この研究は、広東省人民病院の先端才能研究基金会によって資金提供されました[助成金番号]。KY012023293]。この研究は、Jiangsu Mole Bioscience Co.の支援を受けました。資金提供者は、研究デザイン、データ収集と分析、出版の決定、または原稿の準備に関与していませんでした。
資料
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
参考文献
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved