Method Article
Rilevamento dell'infezione da Helicobacter pylori e della resistenza agli antibiotici tramite l'analisi quantitativa della reazione a catena della polimerasi delle feci
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Il protocollo impiega un campionamento non invasivo delle feci combinato con una reazione a catena quantitativa della polimerasi per offrire un metodo diagnostico conveniente e rapido per l'infezione da Helicobacter pylori e la sua resistenza alla claritromicina e ai chinoloni.
Abstract
L'Helicobacter pylori (H. pylori) è ampiamente diffuso in tutto il mondo, con circa il 50% della popolazione mondiale che ha una storia di infezione da H. pylori. In Cina, il tasso di infezione varia dal 40% al 70%. L'H. pylori è principalmente associato a malattie gastrointestinali come la gastrite cronica, le ulcere gastriche e le ulcere duodenali. Attualmente, il trattamento clinico per l'infezione da H. pylori prevede una tripla o quadrupla terapia. Tuttavia, l'uso estensivo di antibiotici ha portato allo sviluppo di resistenza agli antibiotici in H. pylori. Pertanto, rilevare sia l'H. pylori che la sua resistenza agli antibiotici è fondamentale per guidare il trattamento clinico.
I metodi diagnostici per H. pylori includono il test del respiro dell'urea (UBT), il test dell'antigene, il test sierologico degli anticorpi, l'endoscopia, il test rapido dell'ureasi (RUT) e la coltura batterica. Sebbene i primi tre metodi non siano invasivi, non consentono il recupero batterico e, quindi, non possono essere utilizzati per i test di resistenza. Gli ultimi tre metodi sono invasivi, costosi, richiedono un'elevata competenza tecnica e possono causare danni ai pazienti.
Pertanto, un metodo rapido e non invasivo per il rilevamento simultaneo dell'infezione da H. pylori e della resistenza agli antibiotici è della massima importanza clinica per l'eradicazione efficace di H. pylori. Questo articolo mira a introdurre un protocollo specifico che combina la reazione a catena della polimerasi quantitativa (qPCR) con la tecnologia della sonda fluorescente TaqMan per rilevare rapidamente l'infezione da H. pylori e la resistenza agli antibiotici. Questo metodo fornisce un modo comodo, rapido e non invasivo per diagnosticare l'infezione e la resistenza da H. pylori , a differenza della coltura batterica tradizionale e di altre tecniche. La qPCR viene utilizzata per identificare l'infezione e rilevare mutazioni nei geni 23S rRNA e gyrA , che sono legati alla resistenza rispettivamente alla claritromicina e ai chinoloni. Rispetto alle tecniche di coltura convenzionali, questo approccio offre un metodo non invasivo, economico ed efficiente in termini di tempo per rilevare l'infezione da Helicobacter pylori e determinarne la resistenza agli antibiotici.
Introduzione
L'Helicobacter pylori è un batterio gram-negativo, a forma di spirale, che può infettare in modo persistente l'epitelio gastrico umano1. Nel 1994, l'Organizzazione Mondiale della Sanità ha classificato H. pylori come cancerogeno di gruppo 1 per il cancro gastrico, con ~3% degli individui infetti che alla fine hanno sviluppato la malattia2. Una revisione sistematica pubblicata di recente ha indicato che nell'ultimo decennio è stata osservata una tendenza generale all'aumento del tasso di resistenza agli antibiotici di H. pylori , che ha raggiunto livelli allarmanti in tutto il mondo, in particolare la resistenza alla claritromicina3. In Cina, gli ultimi dati dell'indagine hanno mostrato che la prevalenza aggregata di H. pylori tra i cinesi urbani è del 27,08%; i tassi di resistenza per claritromicina e levofloxacina sono stati del 50,83% e del 47,17%4, che sono superiori a quelli degli Stati Uniti (levofloxacina 37,6%, claritromicina 31,5%)5e dell'Europa (levofloxacina 15,8% e claritromicina 21,4%)6. Di conseguenza, la diagnosi e il trattamento precoci e accurati sono fondamentali. Tuttavia, la crescente resistenza agli antibiotici di H. pylori riduce considerevolmente l'efficacia del trattamento, evidenziando l'urgente necessità di una ricerca continua sulla sua diagnosi e trattamento.
I metodi diagnostici di H. pylori sono classificati in tecniche invasive e non invasive7. I metodi invasivi includono l'esame istopatologico, il test rapido dell'ureasi (RUT) e la coltura batterica8. L'esame istopatologico si basa sull'elaborazione e l'osservazione microscopica di campioni bioptici, con un'accuratezza limitata dalla preparazione istologica e dall'esperienza patologica9. RUT rileva H. pylori attraverso l'attività dell'ureasi, che idrolizza l'urea per produrre ammoniaca, determinando uno spostamento alcalino nel reagente, indicando così la presenza del batterio9. È semplice ed economico. La coltura batterica, considerata il "gold standard", ha spesso un tasso di successo inferiore al 50% a causa delle caratteristiche fisiologiche uniche di H. pylori10.
I metodi non invasivi includono il test del respiro dell'urea 13C/14C (UBT), la diagnosi sierologica e il test dell'antigene fecale (SAT)11. Il test del respiro funziona secondo un principio simile a quello del RUT, rilevando la CO2 marcata isotopicamente nell'aria espirata per confermare l'infezione12. La diagnosi sierologica rileva gli anticorpi, che possono persistere dopo l'eradicazione batterica, rendendo difficile valutare l'efficacia del trattamento13. Il test dell'antigene fecale rileva gli antigeni specifici di H. pylori nei campioni fecali, fornendo un metodo affidabile e non invasivo con un tasso inferiore di falsi positivi e una buona precisione per la diagnosi di infezioni attive14.
Ogni metodo diagnostico ha i suoi vantaggi e limiti15. Oltre alla coltura batterica, altri metodi faticano a rilevare la resistenza agli antibiotici, mentre la coltura è ostacolata da bassi tassi di successo16. Recentemente, i metodi diagnostici molecolari come la PCR quantitativa in tempo reale (qPCR) sono stati ampiamente applicati alla rilevazione microbica17. La qPCR è in grado di rilevare con precisione l'H. pylori e di analizzare le mutazioni dei geni di resistenza, offrendo una visione più completa dell'infezione18.
Le sonde fluorescenti sono progettate per legarsi in modo specifico a una sequenza target nel DNA durante il processo di qPCR. Queste sonde sono tipicamente marcate con un colorante fluorescente, che emette un segnale quando la sonda si lega al suo bersaglio e viene scissa dalla polimerasi durante l'amplificazione. Ciò fornisce una misurazione in tempo reale del processo PCR. Le sonde fluorescenti sono note per la loro elevata specificità grazie al design unico della sonda, che garantisce che venga rilevata solo la sequenza di DNA bersaglio, riducendo le possibilità di legame aspecifico e migliorando la specificità del saggio. Ciò rende la tecnologia delle sonde fluorescenti particolarmente utile per rilevare bersagli a bassa abbondanza, come il DNA di Helicobacter pylori , in campioni di feci. Sebbene SYBR Green sia un colorante legante il DNA comunemente usato nella PCR, può legarsi a qualsiasi DNA a doppio filamento, il che può causare un'amplificazione non specifica e falsi positivi.
La coltura di Helicobacter pylori da campioni clinici non fornisce direttamente dati sulla resistenza agli antibiotici. Al contrario, la qPCR basata su sonda fluorescente è più veloce, più conveniente e può essere adattata per il rilevamento simultaneo di marcatori di H. pylori e resistenza agli antibiotici. Questo metodo non solo fornisce risultati più rapidi rispetto ai metodi di coltura tradizionali, ma consente anche il rilevamento simultaneo dei geni di resistenza agli antibiotici, migliorando notevolmente l'efficienza e la convenienza del rilevamento. Questo metodo fornisce un modo comodo, rapido e non invasivo per diagnosticare l'infezione e la resistenza da H. pylori , a differenza della coltura batterica tradizionale e di altre tecniche.
Ci sono due questioni cruciali che devono essere considerate per l'antibiotico-resistenza all'H. pylori : i) la consistenza del genotipo e del fenotipo, ii) la multifarmaco-resistenza. Uno studio multicentrico su larga scala in Cina ha rilevato che i modelli di doppia resistenza per claritromicina/levofloxacina erano del 26,1%. I fenotipi e i genotipi dell'H pylori resistenti alla claritromicina e alla levofloxacina hanno mostrato un accordo soddisfacente (coefficiente kappa = 0,810 e 0,782, rispettivamente)19. Pertanto, è possibile utilizzare il metodo qPCR per rilevare l'infezione da H. pylori e la resistenza ai farmaci. I metodi del test dell'antigene delle feci (SAT) e qPCR sono sempre più utilizzati a causa della comodità del campionamento. La sensibilità, la specificità e l'accuratezza dei metodi per la rilevazione di H. pylori sono le seguenti: qPCR > UBT > SAT > RUT> CagA IgG > coltura20. Tutti i metodi non invasivi sono adatti per lo screening primario. Poiché la qPCR può inoltre rilevare i geni di resistenza ai farmaci, la qPCR e la coltura saranno più adatte per guidare il trattamento.
Altri approcci innovativi, come gli algoritmi di intelligenza artificiale (AI) combinati con immagini endoscopiche21, la microfluidica digitale22 e RPA-CRISPR/Cas12a23, mostrano teoricamente una buona efficienza diagnostica e prospettive applicative promettenti. Tuttavia, la qPCR è attualmente la tecnica di biologia molecolare più sofisticata con il potenziale per fornire una soluzione superiore per la diagnosi e il trattamento delle infezioni da H. pylori .
Protocollo
Questo studio aderisce alle linee guida etiche stabilite dal Comitato etico dell'Ospedale del popolo provinciale del Guangdong, Southern Medical University, Guangzhou, Cina (Approvazione n.: KY2024-445-01). Informazioni dettagliate sui materiali utilizzati in questa ricerca (reagenti, sostanze chimiche, attrezzature e software) sono disponibili nella Tabella dei materiali.
1. Selezione dei partecipanti
- Scegli i partecipanti nella fascia di età compresa tra i 18 e i 60 anni.
- Scegli i partecipanti che possono sottoporsi a screening per l'infezione da Helicobacter pylori , come i pazienti che presentano sintomi come gastrite, ulcere peptiche o dispepsia, nonché gli individui asintomatici.
- Escludere dallo studio i pazienti che hanno utilizzato antibiotici, agenti contenenti bismuto (come capsule di citrato di bismuto di potassio, capsule di pectina di bismuto colloidale o compresse composte di bismuto e alluminio) o farmaci cinesi antimicrobici nell'ultimo mese, o coloro che hanno utilizzato inibitori della pompa protonica o antagonisti del recettore H2 (come omeprazolo, pantoprazolo, rabeprazolo, cimetidina o nizatidina) nelle ultime 2 settimane. Escludere anche le donne durante il periodo mestruale dallo studio.
2. Raccolta di campioni fecali
NOTA: Fornire la sezione 2 come istruzioni ai partecipanti.
- Verificare che l'imballaggio esterno del kit di campionamento sia integro ed entro la data di scadenza. Rimuovere i materiali dal kit.
- Metti una carta bianca per la raccolta delle feci nella tazza del water e defeca (per evitare la contaminazione con l'urina).
- Usando una spatola, raccogliere una porzione di feci delle dimensioni di una fava (~5 g) e trasferirla nel contenitore del campione.
- Posizionare la spatola di campionamento, insieme al campione fecale, nella provetta di raccolta, assicurandosi che il livello del liquido salga vicino alla linea di campionamento indicata sulla provetta.
- Serrare saldamente il tappo del tubo. Capovolgere la provetta più volte per garantire una miscelazione completa del campione con la soluzione conservante.
- Conservare il campione in refrigerazione fino alla consegna al laboratorio clinico per la successiva identificazione di H. pylori e la valutazione dei profili di resistenza agli antibiotici tramite qPCR.
3. Estrazione dell'acido nucleico
NOTA: Completare tutte le procedure all'interno di una cabina di biosicurezza per evitare la contaminazione.
- Capovolgere la provetta di conservazione dei campioni e mescolare accuratamente. Trasferire 1,0 mL della soluzione del campione fecale in una provetta per microcentrifuga.
- Dopo aver mescolato le provette da centrifuga, metterle in un bagno metallico a 80 °C per 10 minuti, con miscelazione intermittente per 30 s al quinto minuto. Una volta che le provette si sono raffreddate a temperatura ambiente, centrifugare a 10.000 × g per 5 minuti e raccogliere il surnatante per l'ulteriore lavorazione.
- Capovolgere più volte la piastra a 96 pozzetti per risospendere le microsfere magnetiche. Rimuovere con cautela la guarnizione in foglio di alluminio, facendo attenzione a non scuotere per evitare fuoriuscite.
- Aggiungere 200 μl del campione preparato (dal passaggio 3.2) a ciascun pozzetto della piastra da 96 pozzetti contenente il tampone di lisi, assicurandosi che ogni pozzetto corrisponda a un singolo campione. Posizionare la piastra a 96 pozzetti nell'apposito scomparto del campione dello strumento di estrazione dell'acido nucleico per l'estrazione automatizzata.
- Conservare i campioni rimanenti e i campioni di acido nucleico estratti a -20 °C per la conservazione a lungo termine e l'uso futuro.
4. qPCR Rilevazione degli acidi nucleici Helicobacter pylori e mutazioni di resistenza alla claritromicina e ai chinoloni
- Prelevare i reagenti dall'area di stoccaggio dei reagenti e scongelarli a temperatura ambiente.
NOTA: Questo kit è stato utilizzato per la rilevazione qualitativa dell'acido nucleico di H. pylori , comprese le mutazioni nel gene 23S rRNA (A2143G, A2143C, A2144G) e nel gene gyrA (261A, 261G, 260T, 271A, 271T, 272G). I primer, le sonde, l'enzima Taq, l'enzima UNG e i dNTP erano disponibili nel kit. I geni bersaglio di H. pylori e della β-actina umana sono stati inclusi nel controllo di qualità positivo. Il geneumano della β-actina è stato scelto come controllo di qualità interno. - In base al numero di campioni da testare, utilizzare N + 2 copie del reagente liofilizzato, dove N rappresenta il numero di campioni da testare e 2 rappresenta i QC negativi e positivi.
- Centrifugare brevemente per assicurarsi che la polvere liofilizzata si depositi sul fondo delle provette. Aprire il coperchio del reagente liofilizzato (facendo attenzione ad evitare qualsiasi fuoriuscita della polvere) e aggiungere 25 μL per pozzetto degli acidi nucleici estratti dai campioni da testare, nonché i controlli positivi e negativi. Chiudere bene i tubi.
- Agitare i reagenti PCR per 8-10 s, quindi centrifugare brevemente per 3-5 s per evitare la formazione di bolle.
- Posizionare la piastra qPCR a 96 pozzetti sulla macchina qPCR. Impostare il programma ciclico come segue: in primo luogo, incubare la miscela di reazione (contenente primer, sonde, Taq polimerasi, enzima UNG, dNTP, ecc.) a 42 °C e 95 °C (entrambe le fasi in un ciclo) per 2 minuti; quindi, ciclo 10x con 10 s a 95 °C e 45 s a 65 °C; infine, ciclo 35x con 10 s a 95 °C e 45 s a 58 °C per la denaturazione, la ricottura e l'estensione.
- Impostare i parametri di rilevamento del segnale di fluorescenza come segue: marcatura fluorescente ROX di geni conservati da H. pylori ; marcatura fluorescente FAM dei geni di resistenza alla claritromicina di H. pylori ; marcatura fluorescente HEX di geni di resistenza ai chinoloni di H. pylori ; e la marcatura fluorescente CY5 del gene della β-actina umana sono stati utilizzati come controllo interno del kit. Raccogli dati a 58 °C. Al termine della reazione, assicurarsi che i dati vengano salvati per analisi future.
- Analizza i dati utilizzando un software specifico per qPCR, poiché lo strumento seleziona automaticamente la soglia di base. Impostare tutti i criteri diagnostici, inclusa la presenza di infezione o resistenza da Helicobacter pylori , su un valore CT ≤30; confermare che presentano una caratteristica curva a forma di S.
Risultati
Applicazione della qPCR per il rilevamento dell'infezione da Helicobacter pylori e della resistenza agli antibiotici in campioni fecali
Abbiamo progettato primer e sonde basati su mutazioni puntiformi nei geni conservati di Helicobacter pylori, così come nel gene 23S rRNA e nel gene gyrA . Questi primer e sonde sono stati marcati con diversi coloranti fluorescenti e quindi utilizzati per il rilevamento qPCR. I risultati del controllo di qualità per gli esperimenti qPCR rientravano nell'intervallo raccomandato, indicando risultati di rilevamento affidabili (Figura 1). In questo studio, abbiamo selezionato cinque campioni rappresentativi (S1-S5) per caratterizzare l'affidabilità del protocollo sperimentale (Figura 2 e Tabella 1). S1 è un campione senza infezione da H. pylori , mentre S2-S5 sono campioni con diversi profili di resistenza ai farmaci.
Per i campioni positivi per l'infezione da Helicobacter pylori , il campione S2 ha mostrato valori CT all'interno dell'intervallo di rilevamento solo per Helicobacter pylori, indicando che questo campione è positivo per Helicobacter pylori ed è sensibile sia alla claritromicina che ai chinoloni, consentendo il trattamento con entrambi i farmaci. Il campione S3 ha mostrato valori CT all'interno dell'intervallo di rilevamento sia per l'infezione da Helicobacter pylori che per la resistenza alla claritromicina, senza valori CT rilevati per la resistenza ai chinoloni, suggerendo che il campione S3 proviene da un paziente resistente alla claritromicina. Allo stesso modo, il campione S4 presentava valori CT all'interno dell'intervallo di rilevamento per l'infezione da Helicobacter pylori e la resistenza ai chinoloni, senza valori CT rilevati per la resistenza alla claritromicina, indicando resistenza ai chinoloni e raccomandando la claritromicina per il trattamento. Infine, il campione S5 presentava valori CT rilevabili in tutti i test, indicando che il campione è positivo per Helicobacter pylori e mostra una doppia resistenza sia alla claritromicina che ai chinoloni; Pertanto, i medici dovrebbero prendere in considerazione opzioni di trattamento alternative. Rispetto ad altri metodi di rilevamento dell'Helicobacter pylori , questo approccio non solo facilita l'acquisizione del campione, ma consente anche il rilevamento simultaneo dell'Helicobacter pylori e del suo profilo di resistenza, fornendo risultati affidabili per guidare le decisioni terapeutiche appropriate.
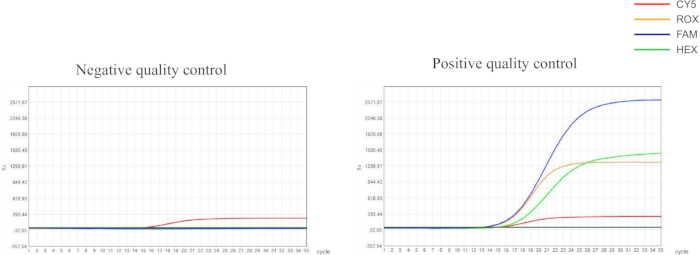
Figura 1: Risultati del controllo di qualità dei controlli negativi e positivi. I controlli negativi devono visualizzare una curva di crescita a forma di S per il canale di rilevamento CY5 con un valore CT ≤ 30,00; i segnali di fluorescenza per i canali FAM, HEX e ROX non dovrebbero mostrare aumenti significativi, con valori CT > 30,00 o nessun segnale notevole; i controlli positivi devono presentare curve a forma di S in tutti i canali di rilevamento con valori CT ≤ 30,00. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Rilevamento dell'Helicobacter pylori e della sua resistenza agli antibiotici in campioni fecali mediante qPCR. Ogni colore rappresenta un segnale fluorescente e diversi segnali fluorescenti rappresentano geni diversi. Canali di fluorescenza: marcatura fluorescente ROX (arancione) di geni conservati di H. pylori ; Marcatura fluorescente FAM (blu) dei geni di resistenza alla claritromicina di H. pylori ; marcatura fluorescente HEX (verde) dei geni di resistenza ai chinoloni di H. pylori ; Marcatura fluorescente CY5 (rosso) del gene di controllo interno. Fenotipi del campione: S1 è un campione negativo per l'infezione da H. pylori (nessun segnale sopra la soglia); S2 è un campione positivo per H. pylori senza resistenza agli antibiotici (segnale ROX superiore alla soglia); S3 è un campione positivo per H. pylori con resistenza alla claritromicina (segnale FAM superiore alla soglia); S4 è un campione positivo per H. pylori con resistenza ai chinoloni (segnale HEX sopra la soglia); S5 è un campione positivo per H. pylori resistente sia alla claritromicina che ai chinoloni (segnale FAM e HEX sopra la soglia). Clicca qui per visualizzare una versione più grande di questa figura.
| Campione | H.Pylori (ROX) | Claritromicina (FAM) | Chinoloni (HEX) | CY5 | |||
| +/- | CT | +/- | CT | +/- | CT | CT | |
| S1 | - | - | - | - | - | - | 9.96 |
| S2 | + | 23.08 | - | - | - | - | 12.66 |
| S3 | + | 19.58 | + | 22.41 | - | - | 11.4 |
| S4 | + | 20.65 | - | - | + | 26.06 | 14.59 |
| S5 | + | 19.82 | + | 22.49 | + | 21.65 | 9.48 |
| negetivo | - | - | - | - | - | - | 14.73 |
| Positivo | + | 12.7 | + | 14.47 | + | 13.07 | 14.78 |
Tabella 1: Risultati del rilevamento dell'infezione da H. pylori e della qPCR per la resistenza alla claritromicina e ai chinoloni. La tabella fornisce i risultati qualitativi dell'infezione da H. pylori . Il rilevamento delle mutazioni del gene 23S rRNA indica la resistenza alla claritromicina, mentre le mutazioni del gene gyrA indicano la resistenza ai chinoloni. Simboli: +/−, risultato qualitativo; +, risultato positivo; −, risultato negativo.
Discussione
Negli ultimi anni, i metodi di rilevamento molecolare sono stati ampiamente applicati nel campo della microbiologia, alterando significativamente la gestione clinica di diverse malattie infettive. Questi metodi operano a livello genetico, consentendo non solo la conferma della presenza batterica, ma anche la tipizzazione genetica e i test di resistenza agli antibiotici. La PCR quantitativa a fluorescenza in tempo reale (qPCR) è sempre più apprezzata grazie al suo breve tempo di elaborazione, all'elevata sensibilità e precisione e al basso rischio di contaminazione incrociata. È diventato ampiamente utilizzato nei laboratori clinici per la diagnosi delle infezioni da Helicobacter pylori .
La combinazione di soluzioni di conservazione per la raccolta di campioni fecali con metodi di rilevamento genico consente uno screening rapido ed efficiente di H. pylori e ulteriori test di resistenza agli antibiotici attraverso l'analisi delle mutazioni nel gene rRNA della claritromicina 23S e nel gene chinolone gyrA . Questo metodo può valutare la sensibilità e la resistenza di H. pylori ai farmaci. I precedenti metodi di rilevamento dell'antigene fecale presentano notevoli limitazioni, come la riduzione dell'accuratezza e della stabilità a causa delle basse concentrazioni di antigene e della degradazione dell'antigene nei campioni. Inoltre, i metodi tradizionali di rilevamento dell'antigene possono non avere una sensibilità e una specificità sufficienti in campioni complessi, compromettendo l'affidabilità dei risultati.
Per affrontare questi problemi, proponiamo un metodo che combina campioni fecali con la tecnologia di reazione a catena della polimerasi quantitativa (qPCR). Questo approccio migliora significativamente la sensibilità e l'accuratezza del rilevamento, rendendo i test fecali più affidabili ed efficienti e fornendo risultati di qualità superiore per l'uso clinico.
Gli standard di controllo della qualità sono fondamentali per il rilevamento della qPCR. Il nostro controllo di qualità include la calibrazione periodica dello strumento, la verifica delle prestazioni del sistema di rilevamento, la valutazione dell'efficienza di estrazione dell'acido nucleico e il controllo di qualità di ogni lotto di esperimenti. Innanzitutto, i nostri strumenti di campionamento vengono calibrati ogni 6 mesi e le apparecchiature di estrazione e amplificazione vengono calibrate annualmente. Inoltre, la calibrazione viene eseguita per ogni lotto di reagenti. Dopo la calibrazione, possiamo convalidare le nostre prestazioni per il sistema di rilevamento, compreso lo strumento di PCR quantitativa a fluorescenza Macrostone 96S, i kit di rilevamento delle mutazioni di H. pylori e di resistenza, i reagenti di estrazione, nonché le relative provette, pipette e puntali di campionamento. Il tasso di conformità, il limite di rilevamento, la reazione incrociata, la capacità anti-interferenza e la precisione soddisfano le nostre esigenze. Le concentrazioni dei prodotti a base di DNA sono state determinate da un tester di concentrazione del DNA per soddisfare i criteri di accettazione per l'efficienza di estrazione degli acidi nucleici. La concentrazione di DNA era superiore a 10 ng/μL. L'A260/A280 era compreso tra 1,6 e 2,0. I campioni non qualificati devono essere nuovamente estratti. I risultati sono validi solo quando il controllo qualità quotidiano è sotto controllo. Per i test di H. pylori e di resistenza agli antibiotici (claritromicina e chinoloni), i criteri di controllo della qualità sono i seguenti: i controlli negativi devono mostrare una curva di crescita a forma di S per il canale di rilevamento CY5 con un valore CT ≤ 30,00; i segnali di fluorescenza per i canali FAM, HEX e ROX non dovrebbero mostrare aumenti significativi, con valori CT >30,00 o nessun segnale notevole; i controlli positivi devono presentare curve a forma di S in tutti i canali di rilevamento con valori CT ≤ 30,00. Queste condizioni devono essere soddisfatte all'interno dello stesso esperimento; In caso contrario, il test sarà considerato non valido e dovrà essere ripetuto. Si noti che, a causa di vincoli proprietari commerciali, non è possibile fornire informazioni sul primer e sulla sequenza della sonda in questo documento.
In uno studio precedente, i ricercatori hanno utilizzato una metodologia identica a quella utilizzata in questo articolo, dimostrando che il test RT-PCR per l'infezione da H. pylori in campioni fecali ha mostrato prestazioni diagnostiche eccezionali24. In particolare, il test ha raggiunto una sensibilità del 99,1%, una specificità del 100% e un'accuratezza diagnostica del 99,1%, che si è allineata strettamente con i risultati dei campioni di biopsia gastrica (93,9%) (Kappa = 0,929, p < 0,001). Questo notevole accordo ha sottolineato che il test RT-PCR può essere considerato un metodo non invasivo altamente affidabile.
Inoltre, lo studio ha anche valutato la capacità della RT-PCR fecale nel rilevare la resistenza agli antibiotici in H. pylori, concentrandosi in particolare sulla resistenza alla claritromicina e alla levofloxacina. Per la resistenza alla claritromicina, sono stati identificati 43 casi (37,3%) in campioni fecali, con una sensibilità del 79,6%, una specificità del 98,4% e un'accuratezza diagnostica del 78,0% (Kappa = 0,788, p < 0,001). Allo stesso modo, per la resistenza alla levofloxacina, sono stati rilevati 37 casi (32,1%), con una sensibilità dell'86,3%, una specificità del 91,1% e un'accuratezza diagnostica del 74,4% (Kappa = 0,739, p < 0,001). Questi risultati erano altamente coerenti con quelli derivati da campioni di biopsia gastrica (resistenza alla claritromicina in 54 casi, resistenza alla levofloxacina in 36 casi), indicando che il test RT-PCR fecale è efficace nel rilevare la resistenza agli antibiotici, possedendo un'elevata specificità e una sensibilità accettabile per entrambi gli antibiotici. La convalida di questa metodologia nello studio precedente supporta ulteriormente la sua affidabilità e applicabilità nel contesto di questo articolo.
Sebbene il protocollo di rilevamento fecale basato sulla qPCR rappresenti un notevole progresso nel campo della diagnostica non invasiva per H. pylori, diverse limitazioni richiedono una considerazione meticolosa. Innanzitutto, l'utilizzo di campioni fecali rende il metodo influenzato dalla qualità del campione. I falsi negativi possono derivare da basse cariche batteriche nelle feci, in particolare nei portatori asintomatici o nei pazienti con infezioni in fase iniziale, dove la diffusione di H. pylori nel tratto gastrointestinale è intermittente25. Inoltre, quando si prelevano campioni fecali, la quantità di batteri può variare a seconda di dove viene prelevato il campione. Ciò contrasta notevolmente con i metodi invasivi, come la coltura bioptica gastrica, che campionano direttamente la mucosa gastrica e sono quindi meno influenzati dalle fluttuazioni della carica batterica. In secondo luogo, sebbene l'approccio di amplificazione mirata del protocollo sia specifico, potrebbe non riuscire a rilevare mutazioni di resistenza rare o emergenti che non sono incorporate nel design del primer, portando potenzialmente a profili di resistenza incompleti26.
La ricerca futura dovrebbe concentrarsi sui progressi metodologici per migliorare le capacità di rilevamento delle mutazioni, compresa l'adozione della PCR digitale (dPCR) per una migliore identificazione delle mutazioni di resistenza a bassa abbondanza27. I pannelli di sonde richiedono un'espansione strategica per comprendere polimorfismi clinicamente rilevanti. La conduzione di studi multicentrici per valutare comparativamente le metodologie diagnostiche, tra cui UBT, test PCR della mucosa gastrica o fecale e rilevamento dell'antigene, fornisce dati completi sulle prestazioni diagnostiche per la pratica clinica, facendo così progredire la standardizzazione dei protocolli di test PCR delle feci. Questi miglioramenti combinati rafforzerebbero l'utilità della qPCR fecale come strumento diagnostico non invasivo, pur essendo importante per guidare un trattamento antibiotico preciso.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Questo studio è stato finanziato dalla Research Foundation for Advanced Talents del Guangdong Provincial People's Hospital [Grant No. KY012023293]. Questo lavoro è stato supportato da Jiangsu Mole Bioscience Co. I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio, nella raccolta e nell'analisi dei dati, nella decisione di pubblicare o nella preparazione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| Disposable fecal collection and storage tube | Mole | Collect fecal specimens | |
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube | |
| Helicobacter pylori nucleic acid, clarithromycin, and quinolone resistance mutation detection kit | Mole | Detection of Helicobacter pylori infection and antibiotic resistance; freeze-dried H. pylori reagent (containing primers, probes, Taq polymerase, UNG enzyme, dNTPs, etc.), a positive control for H. pylori (containing Helicobacter pylori and human β-actin target genes), and a negative control for H. pylori (containing human β-actin target genes) | |
| Mole 96M automated nucleic acid extractor | Mole | For DNA extraction | |
| Nucleic acid extraction kit | Mole | To extract nucleic acid; contains lysis buffer (guanidine salt, tris hydroxymethyl aminomethane, Tween-20, sodium chloride), Wash Buffer 1 (sodium chloride), Wash Buffer 2 (tris hydroxymethyl aminomethane, Tween-20), Wash Buffer 3 (magnetic beads, Tween-20), Wash Buffer 4 (nuclease-free water), and Elution Buffer (tris hydroxymethyl aminomethane), along with a magnetic rack | |
| SLAN Fully automatic medical PCR analysis system | HONGSHI | Data Analysis | |
| SLAN-96S Real-Time PCR machine | HONGSHI | Fluorescent quantitative PCR amplification | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
Riferimenti
- Yuan, C., et al. The global prevalence of and factors associated with Helicobacter pylori infection in children: a systematic review and meta-analysis. Lancet Child Adolesc Health. 6 (3), 185-194 (2022).
- Watari, J., et al. Helicobacter pylori associated chronic gastritis, clinical syndromes, precancerous lesions, and pathogenesis of gastric cancer development. World J Gastroenterol. 20 (18), 5461-5473 (2014).
- Yu, Y., et al. Global primary antibiotic resistance rate of Helicobacter pylori in recent 10 years: A systematic review and meta-analysis. Helicobacter. 29 (3), e13103 (2024).
- Wang, L., et al. cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Ho, J. J. C., Navarro, M., Sawyer, K., Elfanagely, Y., Moss, S. F. Helicobacter pylori antibiotic resistance in the United States between 2011 and 2021: A systematic review and meta-analysis. American J Gastroenterol. 117 (8), 1221-1230 (2022).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Guevara, B., Cogdill, A. G. Helicobacter pylori: A review of current diagnostic and management strategies. Dig Dis Sci. 65 (7), 1917-1931 (2020).
- Rotimi, O., Cairns, A., Gray, S., Moayyedi, P., Dixon, M. F. Histological identification of Helicobacter pylori: Comparison of staining methods. J Clin Pathol. 53 (10), 756-759 (2000).
- Lim, L. L., Ho, K. Y., Ho, B., Salto-Tellez, M. Effect of biopsies on sensitivity and specificity of ultra-rapid urease test for detection of Helicobacter pylori infection: A prospective evaluation. World J Gastroenterol. 10 (13), 1907-1910 (2004).
- Hortelano, I., Moreno, Y., Vesga, F. J., Ferrús, M. A. Evaluation of different culture media for detection and quantification of H. pylori in environmental and clinical samples. Int Microbiol. 23 (4), 481-487 (2020).
- Wang, Y. K., et al. Diagnosis of Helicobacter pylori infection: Current options and developments. World J Gastroenterol. 21 (40), 11221-11235 (2015).
- Gisbert, J. P., Pajares, J. M. Review article: 13C-Urea breath test in the diagnosis of Helicobacter pylori infection -- a critical review. Aliment Pharmacol Ther. 20 (10), 1001-1017 (2004).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: A review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Gisbert, J. P., Pajares, J. M. Stool antigen test for the diagnosis of Helicobacter pylori infection: A systematic review. Helicobacter. 9 (4), 347-368 (2004).
- Benigno, T. G. D. S., et al. pylori primary strains and virulence genotypes in the Northeastern region of Brazil. Rev Inst Med Trop Sao Paulo. 64, e47 (2022).
- Savoldi, A., Carrara, E., Graham, D. Y., Conti, M., Tacconelli, E. Prevalence of antibiotic resistance in Helicobacter pylori: A systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 155 (5), 1372-1382.e17 (2018).
- Kalach, N., et al. Usefulness of gastric biopsy-based real-time polymerase chain reaction for the diagnosis of Helicobacter pylori infection in children. J Pediatr Gastroenterol Nutr. 61 (3), 307-312 (2015).
- Han, X., et al. Quantitative PCR of string-test collected gastric material: A feasible approach to detect Helicobacter pylori and its resistance against clarithromycin and levofloxacin for susceptibility-guided therapy. Helicobacter. 28 (4), e12985 (2023).
- Zhong, Z., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am J Cancer Res. 11 (10), 5027-5037 (2021).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PloS One. 16 (8), e0256393 (2021).
- Bang, C. S., Lee, J. J., Baik, G. H. Artificial intelligence for the prediction of Helicobacter pylori infection in endoscopic images: Systematic review and meta-analysis of diagnostic test accuracy. J Med Internet Res. 22 (9), e21983 (2020).
- Liu, J., et al. Rapid and multi-target genotyping of Helicobacter pylori with digital microfluidics. Biosens Bioelectron. 256, 116282 (2024).
- Liu, H., Wang, J., Hu, X., Tang, X., Zhang, C. A rapid and high-throughput Helicobacter pylori RPA-CRISPR/Cas12a-based nucleic acid detection system. Clin Chim Acta. 540, 117201 (2023).
- Fan, C. J., et al. Diagnostic accuracy of a real-time PCR assay for detection of Helicobacter pylori and resistance to clarithromycin and levofloxacin directly from stool. Eur Rev Med Pharmacol Sci. 28 (12), 3836-3840 (2024).
- Patel, S. K., Pratap, C. B., Jain, A. K., Gulati, A. K., Nath, G. Diagnosis of Helicobacter pylori: what should be the gold standard. World J Gastroenterol. 20 (36), 12847-12859 (2014).
- Celiberto, F., et al. The state of the art of molecular fecal investigations for Helicobacter pylori (H. pylori) antibiotic resistances. Int J Mol Sci. 24 (5), 4361 (2023).
- Yang, H., Hu, B. Diagnosis of Helicobacter pylori infection and recent advances. Diagnostics. 11 (8), 1305 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon