Method Article
DNA Perdeleri Homolog Rekombinasyon Sırasında Karmaşık Moleküler Sistemlere Işık Tutuyor
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
DNA perdeleri, bir mikroakışkan numune odasının yüzeyinde hizalanmış DNA molekülleri ile etkileşime girerken yüzlerce hatta binlerce DNA bağlayıcı proteini gerçek zamanlı olarak görselleştirmek için yeni bir yöntem sunar.
Özet
Homolog rekombinasyon (HR), tüm organizmalarda çift sarmallı DNA kırılmalarının (DSB'ler) ve durmuş replikasyon çatallarının onarımı için önemlidir. HR'deki kusurlar, insan hücrelerinde genom bütünlüğü kaybı ve onkojenik transformasyon ile yakından ilişkilidir. HR, birçoğu tam olarak anlaşılmamış olan karmaşık bir protein kümesinin koordineli eylemlerini içerir. Burada açıklanan araştırmanın kilit yönü, hizalanmış DNA moleküllerinin bir mikroakışkan numune odasının yüzeyine monte edilmesine izin veren bir teknik olan "DNA perdeleri" adı verilen bir teknolojidir. Daha sonra toplam iç yansıma floresan mikroskobu (TIRFM) ile görselleştirilebilirler. DNA perdelerinin öncülüğünü laboratuvarımız yapmıştır ve diğer metodolojilerle kolayca ortaya çıkarılamayan milisaniye zaman ölçeklerinde ve nanometre ölçeğinde çözünürlükte uzay-zamansal bilgilere doğrudan erişim sağlar. DNA perdelerinin önemli bir avantajı, tek molekül deneylerinden istatistiksel olarak ilgili verilerin toplanmasını basitleştirmesidir. Bu araştırma, hücrelerin genom bütünlüğünü nasıl düzenlediğine ve koruduğuna dair yeni bilgiler vermeye devam ediyor.
Giriş
Genom bütünlüğünün korunması, tüm canlı hücrelerin düzgün çalışması için çok önemlidir1. Genom bütünlüğündeki kusurlar, çeşitli kanser türleri ve yaşa bağlı dejeneratif hastalıklar dahil olmak üzere ciddi sağlık koşullarına yol açabilir2. Homolog rekombinasyon (HR), DNA çift sarmallı kırılmaları (DSB), tek sarmallı DNA (ssDNA) boşluklarını ve zincirler arası DNA çapraz bağlarını onarmak için şablona bağlı DNA sentezini kullanır3. HR, durdurulan ve daraltılan çoğaltma çatallarının kurtarılması için de gereklidir 3,4. Ayrıca, HR, mayoz bölünme sırasında doğru kromozom ayrışması için gereklidir 5,6.
HR, birçoğu tam olarak anlaşılamayan karmaşık bir protein kümesinin koordineli eylemlerini içerir1. Örnekler arasında replikasyon proteini A (RPA), Rad51 ve Rad54 bulunur7. Hem prokaryotik hem de ökaryotik hücrelerdeki HR reaksiyonları, ssDNA bağlayıcı proteinler (prokaryotlarda SSB ve ökaryotlarda RPA) tarafından hızla kaplanan bir ssDNA ara ürünü içerir8. Bu proteinler ssDNA'yı nükleazlardan korur, ikincil yapıyı ortadan kaldırır ve aşağı akış faktörlerinin işe alınmasını teşvik eder 8,9. Rad51, tüm canlı organizmalarda bulunan, yüksek oranda korunmuş ATP'ye bağımlı Rad51 / RecA DNA rekombinaz ailesinin bir üyesidir1. Rad51, homolog dsDNA donörünün DNA ipliği istilasını teşvik eder. Önemi göz önüne alındığında, Rad51 yüksek düzeyde düzenlenir ve bu düzenleyici süreçlerdeki kusurlar genellikle genom bütünlüğü kaybı ve onkojenik dönüşüm7 ile ilişkilidir. Rad54, Swi2/Snf2 dsDNA translokazları ve kromatin yeniden modelleyicileri10,11 ailesinin bir üyesidir. Bu proteinler, temel Rad51 düzenleyici faktörler olarak işlev görür. Daha da önemlisi, Rad54, Rad51'i iplik istilasının dsDNA ürününden çıkarır ve ayrıca Rad51'in kromatin11 üzerinde yanlış birikmesini önlemek için gereklidir. Hem maya hem de bakteri hücrelerinde HR'de yer alan proteinler için moleküler aktiviteler, HR'deki işlevlerine ışık tutmuştur, ancak aktivitelerinin HR'ye tam olarak nasıl katkıda bulunduğu tam olarak anlaşılamamıştır 12.
DNA perdeleri, aksi takdirde erişilemeyecek olan moleküler mekanizmalara ve makromoleküler dinamiklere doğrudan erişim sağlayan benzersiz bir platform olarak ortaya çıkmıştır13,14. DNA perdelerini hazırlamak için, bir mikroakışkan odanın yüzeyi bir lipid çift tabakası ile kaplanır ve DNA molekülleri, bir biyotin-streptavidin bağlantısı yoluyla çift tabakaya bağlanır. Çift katman, doğal hücre zarlarını taklit ederek yüzeyi etkisiz hale getirir. Hidrodinamik kuvvet, DNA'yı nanofabrikasyon bariyerler boyunca hizalayarak, toplam iç yansıma floresan mikroskobu (TIRFM) ile yüzlerce molekülün tek bir görüş alanında görselleştirilmesine olanak tanır. Bariyerler elektron ışını litografisi ile yapılır ve bariyer tasarımındaki varyasyonlar, DNA'nın dağılımı ve bağlama geometrisi üzerinde hassas kontrol sağlar. Bu yaklaşımlar ssDNA veya dsDNA 13,14,15,16,17,18 ile kolayca uygulanabilir. Kaideler ayrıca, DNA'nın her iki ucunun da numune odası yüzeyine bağlanmasına izin vermek için nanofabrikasyon (bariyerlerle birlikte) olabilir, böylece tampon akışının yokluğunda kararlı durum deneyleri gerçekleştirilebilir.
Bireysel protein-nükleik asit komplekslerindeki zamana bağlı değişiklikler, gerçek zamanlı videoların incelenmesiyle ortaya çıkar ve proteinlerin zaman içinde DNA üzerindeki değişen konumunu sunan kimograflar kullanılarak basılı olarak temsil edilir. DNA perdeleri yaklaşımının önemli bir yönü, moleküler mekanizmalar hakkında a priori modeller veya varsayımlar gerektirmemesidir, çünkü bireysel reaksiyon bileşenlerinin davranışları gerçek zamanlı olarak gözlemlenebilir. Bu, moleküler davranışların doğrudan gözlemlenmesine izin verir. Burada, bu protokol, dsDNA substratları ile DNA perdelerinin nasıl hazırlanacağını ve homolog rekombinasyonda ara ürünleri incelemek için uygulanmasını açıklar.
Protokol
1. Lipid stoğunun hazırlanması
- 1 g 18: 1 (Δ9-Cis) PC, 100 mg 18: 1 PEG2000 PE ve 5 mg 18: 1 biyotinil başlık PE'yi 10 mL kloroformda çözün.
NOT: Hem DOPC hem de DPPE iyi karakterize edilmiştir ve (PEG modifikasyonu ile birlikte) akış hücresinin yüzeyine spesifik olmayan adsorpsiyonu en aza indirme yetenekleri nedeniyle seçilmiştir. Çözünmüş lipid ana karışımı alınabilir ve -20 ° C'de 12 aya kadar saklanabilir. - Organik çözücü bir cam şırıngayı temizlemek için kloroform kullanın, ardından lipit ana karışımının 200 μL'sini (istenen nihai hacmin% 10'u) cam şişeye aktarın.
- Çok yumuşak bir sıkıştırılmış nitrojen akışı kullanarak kloroformu cam şişeden buharlaştırın. Tüm kloroform izlerinin giderildiğinden emin olmak için buharlaşmanın sonuna doğru basıncı hafifçe artırın.
NOT: Buharlaştırma işlemi sırasında şişeyi eğin ve döndürün, böylece lipitler duvarda düzgün bir beyaz film tabakası olarak kurur. - Şişeyi gece boyunca vakum altında kapaksız olarak yerleştirin.
- Kurutulmuş lipid filme 2 mL lipid tamponu (10 mM Tris-HCl [pH 8.0], 100 mM NaCl) ekleyin ve şişeyi kapatın. Oda sıcaklığında (RT) en az 1 saat inkübe edin ve çözünene kadar girdap yapın.
- Çözeltiyi 5 mL polistiren yuvarlak tabanlı bir tüpe aktarın ve aşağıdaki parametrelere sahip bir mikro uç kullanarak buz üzerinde sonikat: genlik = 50, işlem süresi = 1.5 dakika, nabız = 15 s, zaman = 2 dakika. Sonikasyondan sonra çözüm berrak olacaktır (toplam enerji = 1.500-2.000 J).
- Çözeltiyi 0,22 μm PVDF şırınga filtresinden süzün ve lipid stoğunu 1 aya kadar 4 °C'de saklayın.
2. dsDNA substratının hazırlanması
- λ-DNA stoğunu (500 μg/mL) 65 °C'ye ısıtın.
- Aşağıdakileri içeren tavlama/ligasyon karışımı hazırlayın: 1.6 pM λ-DNA, her oligonükleotidin 100x molar fazlalığı (160 pM) (BioL: 5'Phos-AGG TCG CCG CCC-3Bio ve DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) ve 60 μL 10x T4 ligaz reaksiyon tamponu (toplam 600 μL hacimde).
NOT: BioL ve DigR oligonükleotidleri sırasıyla biyotinile edilir (Bio) ve digoksijenenin (Dig) ile modifiye edilir. Biyotinilasyon, DNA moleküllerinin streptavidin bağları yoluyla biyotinile lipidlere bağlanmasına izin verir. Dig modifikasyonu, kaideler üzerine kaplanmış anti-Dig antikorlarının Dig parçalarına bağlanması yoluyla çift bağlamayı mümkün kılar. - Reaksiyonu 65 ° C'de 10 dakika inkübe ederek oligonükleotidleri λ-DNA cos-ends'e tavlayın, ardından RT'ye yavaşça soğumaya bırakın.
- 5 μL T4 DNA ligaz ekleyin ve gece boyunca RT'de inkübe edin.
NOT: Tavlama ve ligasyon adımları, bir termal döngüleyicide de gerçekleştirilebilir. - 300 μL PEG-8000 çözeltisi (%30 w/v PEG-8000 ve 10 mM MgCl2) ekleyerek λ-DNA'yı çökeltin. 4 °C'de en az 1 saat inkübe edin.
- RT'de 5 dakika boyunca 14.000 x g'da santrifüjleyin. Peleti rahatsız etmeden süpernatanı dikkatlice çıkarın. İsteğe bağlı olarak, peleti 1x 500 μL soğutulmuş %70 etanol ile yıkayın.
- Peleti 40 μL TE tamponunda (10 mM Tris-HCl, 1 mM EDTA [pH 8.0]) yeniden süspanse edin.
3. Krom kalıplarının nanofabrikasyonu
- Masa üstü matkap presinde elmas kaplı 1 mm matkap ucu kullanarak 3" x 3" kuvars mikroskop slaytının ortasına (~3 cm aralıklı) iki delik açın (delme konumunun geometrisi için Şekil 1.4'e bakın).
- Delinmiş slaytları 200 mL Piranha çözeltisine (% 75 sülfürik asit [% 97] ve% 25 hidrojen peroksit) batırarak temizleyin. 30 dakika inkübe edin.
DİKKAT: Açıkta kalan cilt ile her türlü temayı önlemek için çok dikkatli olunmalıdır. Soğuk hidrojen peroksiti sülfürik aside çok yavaş bir şekilde karıştırarak çözeltiyi hazırlayın. Piranha atıklarını belirlenmiş bir atık kabına uygun şekilde atın. - Slaytları 200 mL çift damıtılmış suya batırarak durulayın. Kalan Piranha'yı ortadan kaldırmak için 2x tekrarlayın. Slaytları hafif bir sıkıştırılmış nitrojen akışıyla kurulayın.
- Bir spin kaplayıcı (45 sn için 4.000 rpm, rampa hızı 300 rpm/s) kullanarak, temiz kızakları fotorezist ve iletken polimer ile aşağıdaki gibi kaplayın:
- Slaytları %3 (a/h) polimetilmetakrilat (PMMA; 24.3K MW; anisol içinde çözülmüş) tabakası ile kaplayın.
- Slaytları %1,5 (a/h) polimetilmetakrilat (PMMA; 495K MW; anisol içinde çözülmüş) tabakası ile kaplayın.
- Elektron ışını litografisi için slaytları bir antistatik ajan tabakası ile kaplayın.
- Nanodesen oluşturma sistemi yazılımı ile donatılmış bir taramalı elektron mikroskobu ile elektron ışını litografisi kullanarak kaplanmış slaytlar üzerine bariyerler ve kaideler için özel desenler yazın.
- Önce elektron ışını litografisi için antistatik maddeyi çift damıtılmış su ile durulayarak slaytları geliştirin. Daha sonra, bunları gelişmekte olan çözeltiyi (% 75 metil izobütil keton [MIBK] ve% 25 izopropanol; -20 ° C'de soğuk tutun) içeren 50 mL'lik tüplere yerleştirin ve 1 dakika boyunca düşük güçte bir buz banyosunda sonikat yapın.
- Gelişmekte olan çözeltiyi izopropanol ile durulayın ve hafif bir sıkıştırılmış nitrojen akışı ile kurulayın.
- Bir elektron ışını evaporatörü kullanarak, desenli yüzeye 0,5 ş/ s'de 250 şkalınlığında bir krom tabakası bırakın.
- Bir fışkırtma şişesinden aseton kullanarak kromu ve kalan PMMA'yı kaldırın, ardından slaytları 10 dakika boyunca bir aseton banyosunda sonikleştirin. Bir fışkırtma şişesinden aseton ile tekrar yıkayın.
- Kalan asetondan herhangi bir tortu birikmesini önlemek için, slaytları izopropil alkolle durulayın ve hafif bir sıkıştırılmış nitrojen akışıyla kurulayın.
4. Akış hücresinin montajı
- 35 mm x 5 mm'lik dikdörtgen bir kağıt şablonu (krom desenini ve önceden delinmiş giriş/çıkış deliklerini kaplayacak) desenli tarafı yukarı bakacak şekilde kuvars sürgünün üzerine yerleştirin ( Şekil 1A).
- Kağıt şablonunu kaplayacak şekilde kuvars slaytın üzerine bir parça çift taraflı bant (1 mm kalınlığında) uygulayın, ardından dikdörtgen kağıt şablonunu kesmek için bir tıraş bıçağı kullanın ( Şekil 1A).
- Çift taraflı bandın üzerine bir cam lamel yerleştirin ve lamel bandı banda yapıştırmak için hafif bir baskı uygulayın ( Şekil 1A). Lamel ve sürgü arasındaki boşluk, çift taraflı bantla kapatılmış, mikroakışkan odayı oluşturur.
NOT: Deney sırasında sızıntıyı önlemek için kapak kızağı ile sürgü arasındaki contanın yeterince güvenli olması çok önemlidir. - Lamel-kaydıraklı sandviçi cam kızakların arasına yerleştirin ve basıncı eşit olarak dağıtmak için dört tarafa da bağlayıcı klipsler takın. Çift taraflı bandı eritmek ve hazneyi kapatmak için düzeneği 135 °C'de 60 dakika vakumlu fırına koyun.
- Fırından çıkarın, ardından bağlayıcı klipsleri ve cam slaytları serbest bırakın.
- Yapıştırılmış düzeneği 220 dakika boyunca bir ısıtma bloğuna (1 °C) yerleştirerek, çıkararak ve mikroakışkan portları düzgün bir şekilde kapatmak için dikkatlice hafif basınç uygulayarak mikroakışkan portları sıcak tutkal tabancasıyla önceden delinmiş giriş/çıkış deliklerine takın. Devam etmeden önce tutkalın tamamen kürlenmesine izin verin.
NOT: Monte edilmiş akış hücreleri 50 mL'lik tüplere yerleştirilebilir ve vakum altında saklanabilir.
5. dsDNA perdesinin montajı
- Giriş/çıkış borusunu akış hücresi mikroakışkan portlarına takın ve hazneyi iki adet 10 mL şırınga kullanarak 20 mL çift damıtılmış su ile yıkayın.
- Akış hücresinin her iki tarafına bağlı çift damıtılmış su ile doldurulmuş 10 mL şırınga ile, suyu giriş ve çıkış şırıngaları arasında itmek/çekmek için kuvvet uygulayın. Bu adım, akış hücresindeki tüm hava kabarcıklarını giderir.
- Hazneyi 3 mL lipid tamponu (10 mM Tris-HCl [pH 8.0], 100 mM NaCl) ile yıkayın.
NOT: Akış hücresine hava kabarcıkları girmesini önlemek için akış hücrelerine yapılan tüm bağlantıların damla damla olduğundan emin olun. - 40 μL lipozom çözeltisini 1 mL lipid tamponu ile karıştırın. Akış hücresine 300 μL lipit çözeltisi enjekte edin ve 5 dakika inkübe edin. Enjeksiyonu ve inkübasyonu 2 kez tekrarlayın.
NOT: Akış hücresine her şırınga bağlandığında enjeksiyon için giriş/çıkış tarafını değiştirin. - Hazneyi 3 mL lipid tamponu ile yıkayın ve 15 dakika inkübe edin.
NOT: Bu kuluçka süresi, deneylerin programlanmasına bağlı olarak 5 dakika ile 2 saat arasında değişebilir ve son perde montajında gözle görülür farklılıklar olabilir. - 40 μL 1 mg / mL anti-digoksigenin stok çözeltisini 200 μL lipid tamponu ile karıştırın. 100 μL çözelti enjekte edin ve 10 dakika inkübe edin. 3 mL lipid tamponu ile yıkamadan önce enjeksiyonu ve inkübasyonu 1 kez tekrarlayın.
NOT: Bu adım yalnızca çift bağlı dsDNA perdeleri için gereklidir. Tek bağlı dsDNA perdeleri için bu adımı atlayın. - Hazneyi 3 mL BSA tamponu (40 mM Tris-HCl [pH 8.0], 2 mM MgCl2, 1 mM DTT ve 0.2 mg / mL BSA) ile yıkayın.
- 10 μL 1 mg / mL streptavidin stok çözeltisini 990 μL BSA tamponu ile karıştırın. Her enjeksiyondan sonra 10 dakikalık bir inkübasyon ile iki ayrı 400 μL enjeksiyon olarak enjekte edin.
- Fazla streptavidini çıkarmak için hazneyi 3 mL BSA tamponu ile yıkayın.
- 25 ng dsDNA substrat stoğunu 1 mL BSA tamponunda seyreltin. DNA çözeltisini bir seferde 200 μL yavaşça enjekte edin, ardından her enjeksiyondan sonra 5 dakikalık bir inkübasyon yapın. İhtiyaç duyulan DNA miktarı, belirli deney türleri için optimize edilebilir.
- Akış hücresini mikroskop tablasına monte edin ve odağı gerektiği gibi ayarlayın.
- Giriş ve çıkış portlarını, bir şırınga pompası ve yüksek basınç anahtarı valfinden oluşan bir numune enjeksiyon sistemine bağlayın.
- Odayı 5 dakika boyunca 0,8 mL / dk'da BSA tamponu ile sürekli olarak yıkayarak DNA'yı kaidelere (12 μm ip uzunluğu) çift bağlayın.
NOT: Farklı çift bağlama uzunluklarını optimize etmek için akış hızlarını ve sürelerini ayarlamak gerekebilir. Bu adım sadece çift bağlı DNA perdeleri için gereklidir. Tek bağlı dsDNA perdeleri için bu adımı atlayın. - Perdeler artık deneyler için hazır.
6. Akış hücrelerinin geri dönüşümü
- Mikroakışkan portları ve lamel dikkatlice çıkarmak için bir tıraş bıçağı kullanmadan önce slaytları en az 30 dakika etanole batırın. Lameli uygun şekilde atın ve yeniden kullanılabilmeleri için mikroakışkan bağlantı noktalarında kalan yapıştırıcıyı temizleyin. Sürgü üzerindeki tutkal kalıntılarını ovalayarak temizleyin.
NOT: Slaytın ortasındaki desenli alanı çizmemeye dikkat edin. - Slaytları çift damıtılmış suyla iyice durulayın ve gece boyunca karıştırarak %2 alkali sıvı kuvars temizleme konsantresine batırın.
- Slaytları çift damıtılmış suyla iyice durulayın ve% 2 alkali sıvı kuvars temizleme konsantresi çözeltisine 80 ° C'de karıştırarak 1 saat daldırın, ardından 45 ° C'de 15 dakika sonikasyon yapın.
- Slaytları çift damıtılmış suyla iyice durulayın ve karıştırarak 30-60 dakika boyunca 1 M NaOH'ye batırın.
- Slaytları çift damıtılmış suyla iyice durulayın, ardından izopropil alkole batırın ve 45 ° C'de 15 dakika boyunca sonikat yapın.
- Slaytları etanol ile iyice durulayın ve karıştırarak 1 saat etanole batırın.
- Slaytları 135 °C'de kuruması için 1 saat vakumlu fırına koyun. Slaytlar artık akış hücrelerine yeniden monte edilmeye hazırdır.
Sonuçlar
Yukarıda açıklananlar, DNA onarım ara ürünlerindeki protein-DNA etkileşimlerinin incelenmesi bağlamında tek ve çift bağlı dsDNA perdelerinin hazırlanması, birleştirilmesi ve görüntülenmesidir. Şekil 1A , bir akış hücresindeki tüm bileşenleri, monte edilme sırasına göre katmanlanmış olarak göstermektedir. Şekil 1B , tek veya çift bağlı bir DNA perdesinin şemasını göstermektedir. Akış hücresinin yüzeyini pasifleştirmek için bir lipit çift tabakası kullanılır. DNA perdesi, bir ucunda lipit'e bağlı ve krom bariyerinde hizalanmış, akış yönüne dik olarak yönlendirilmiş paralel bir DNA molekülleri dizisinden oluşur. Çift bağlı DNA perdeleri için, DNA'nın diğer ucu, Digoksijenenin ve anti-DigoksİGENİN etkileşimleri yoluyla kaideye bağlanır, öyle ki, DNA uzamış kalırken akış yokluğunda görüntüleme gerçekleştirilebilir. dsDNA, floresan boya YoYo1 ile boyanabilir ve TIRF mikroskobu ile görüntülenebilir. Şekil 2A , YoYo1 ile boyanmış çift bağlı DNA perdesinin temsili bir geniş alan görüntüsünü göstermektedir.
DNA perdesi deneylerinden toplanan hızlandırılmış görüntü serileri, tipik olarak, ImageJ'de ilgilenilen her DNA molekülü için DNA molekülü boyunca dikey eksendeki konumu ve yatay eksendeki zamanı çizen bir kimografi oluşturularak analiz edilir. Şekil 2B, tek sarmallı DNA bağlayıcı protein RPA-mCherry (macenta) ve ATP varlığında maya rezeksiyon makineleri GFP-Sgs1 (görünmez), Top3-Rmi1 ve Dna2 tarafından YoYo1 ile boyanmış tek bağlı λ-DNA'nın (yeşil) rezeksiyonunu gösteren temsili kimografilerdir. YoYo1 sinyali, dsDNA'nın Sgs1-Dna2 tarafından serbest uçtan rezeke edilmesiyle zamanla kaybolur. Eşzamanlı olarak, mCherry sinyali, rezeksiyonun bir sonucu olarak ssDNA'nın üretildiği DNA uçları ile kolokalize olur. Rezeksiyonun hız ve süreç gibi biyofiziksel olarak ilgili özellikleri, YoYo1 sinyal kaybı yörüngelerinin eğimlerinin ölçülmesiyle çıkarılabilir. Şekil 2C,D, Sgs1-Dna2 ile rezeksiyonun sırasıyla hızlarının ve süreçselliğinin dağılımını göstermektedir.

Şekil 1: Akış hücresi düzeneği ve DNA perdelerinin şemaları. (A) Akış hücresi düzeneği için kademeli çizim. Kuvars sürgünün üstüne çift taraflı bant yerleştirilir ve merkezden dikdörtgen bir kanal kesmek için bir kağıt şablon kullanılır, bu kanal bir oda oluşturmak için üstte bir kapak kayma ile kapatılır. (B) Tamamen monte edilmiş DNA perdelerinin şemaları. Orta panel, tek bağlı bir akış hücresi sunarken, alt panel çift bağlı bir akış hücresi sunar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
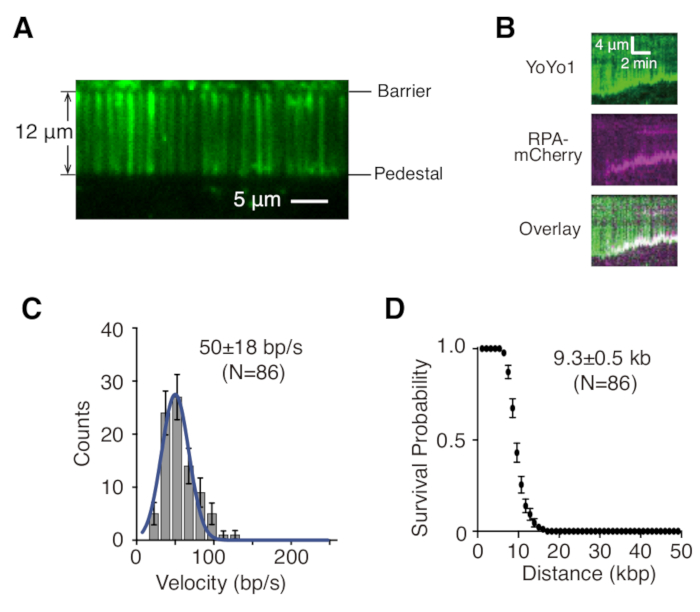
Şekil 2: Tek moleküllü DNA perdesi görselleştirme ve analizi. (A) YoYo1 (yeşil) ile boyanmış çift bağlı bir λ-DNA perdesinin temsili geniş alan görüntüsü. (B) Dna2 ile kovalandığında önceden bağlanmış GFP-Sgs1 (görünmez) varlığında, RPA-mCherry (macenta) ve 2 mM ATP varlığında YoYo1 ile boyanmış dsDNA'nın (yeşil) rezeksiyonunu gösteren temsili kimografiler. Sgs1-Dna2 ile dsDNA rezeksiyonunun hız dağılımı (C) ve süreçsellik grafiği (D), zaman içinde YoYo1 sinyal kaybının hızı ve derecesinin ölçülmesiyle elde edilir. (B), (C) ve (D) panelleri Xue ve ark.19'dan uyarlanmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
DNA perdelerinin, çeşitli DNA onarım süreçlerini incelemek için çok yönlü bir platform olduğu kanıtlanmıştır 13,14,15,16. Deney boyunca, sürekli tampon akışı veya kaideye çift bağlama yoluyla yüzeye yakın bir yerde uzatılmış kalan DNA molekülleri, tek molekül seviyesinde onarım ara ürünleri üzerindeki protein-DNA etkileşimlerinin TIRFM görüntülemesine izin verir. Yöntem, deneysel verim ve öngörülebilirlikte iyileşme sağlar, çünkü yüzlerce DNA molekülü, yüzeye spesifik olarak bağlanmamış olmak yerine nanofabrikasyon bariyerlerde düzenli bir şekilde hizalanır ve bu da büyük veri kümelerinin işlenmesine daha fazla fayda sağlar.
Elde edilen hızlandırılmış görüntü serilerinin dikkatli analizi, protein-DNA etkileşimlerinin kantitatif ölçümlerini sağlar (yani, bağlanma ömürleri, konumlar, stokiyometri, ayrışma kinetiği, translokasyon oranları, motor proteinlerin süreçleri ve proteinler ve/veya DNA arasındaki kolokalizasyonlar ve etkileşimler). DNA moleküllerini doğrudan yüzeye bağlayan veya konfokal görüntülemeyi ticari cihazlarda kuvvet manipülasyonu ile birleştiren diğer tek moleküllü görüntüleme teknikleriyle karşılaştırıldığında, daha yüksek verim için DNA perdelerinin en büyük ödünleşimi, ilk kurulum için gerekli tekniklerin oluşturulmasıdır 13,14,15,16. Bununla birlikte, bir kez kurulduktan sonra, tek bir akış hücresinde aynı anda yüzlerce molekül hakkında veri toplama yeteneği, veri toplama için harcanan zamanda önemli tasarruflara yol açar.
Diğer birçok tek moleküllü görüntüleme yönteminde olduğu gibi, temiz bir yüzey hazırlama yeteneği, kullanıcının DNA perdelerini güvenilir bir şekilde monte etme yeteneğini büyük ölçüde belirler. Bu durumda, temiz yüzeyler sadece temiz bir sürgü ile başlamayı değil, aynı zamanda akışkan bağlantılarından herhangi birini yaparken hazneye mikroskobik hava kabarcıkları sokmamaya dikkat etmeyi gerektirir. İlk olarak, slaytların temizliği ile ilgili olarak, yukarıda bölüm 6'da ayrıntıları verilen adımlar rutin bir temizleme işlemi olarak yeterli olacaktır. Filtrelenmiş çözeltilerin ve tamponların kullanılması da önerilir. İkinci olarak, akış hücresinin tampon enjeksiyon sistemine bağlanması sırasında damladan damlaya bağlantıların yapılması, kabarcıkların girmesini önlemek için çok önemlidir. Tampon yapmak için kullanılan çift damıtılmış suyun gazının alınması faydalı olabilir. Ayrıca, kabarcık olmadığından emin olmak için herhangi bir enjeksiyon işlemini dikkatlice gözlemlemeniz önerilir. Mikroskobik bir kabarcık, merkez desenli alana ulaşmadan önce sokulursa, kabarcığı geri itmek için karşı taraftaki mikroakışkan porttan tampon enjekte edilerek düzeltilebilir.
Herhangi bir floresan tabanlı deneyde göz önünde bulundurulması gereken bir diğer önemli nokta, sinyal-gürültüdür. Tek floresan proteinleri (yani GFP) gözlemlemenin bir anahtarı da temiz yüzeylerde bulunur. Uzun süreli kullanımdan sonra, protein agregaları tipik olarak krom bariyerlerde ve kaidelerde birikir. Bu nedenle, floresan proteinlerin eklenmesinden önce bariyerlerde güçlü bir floresan sinyali gözlenirse, slaytların Piranha çözeltisine 10 dakika daldırılarak tedavi edilmesi tavsiye edilir. Krom özelliklerinin aşınmasını önlemek için süre kısa tutulmuştur. Piranha yıkamasını düzenli temizlik prosedürü takip etmelidir.
Elektron ışını litografisi kullanılarak yapılan nanofabrikasyon, ekipman ve tesis gereksinimlerinin yanı sıra malzeme bilimindeki mevcut uzmanlığı nedeniyle zor olabilir. Alternatif olarak, DNA perdeleri için krom özelliklerinin üretilmesi için UV litografi tabanlı bir yöntem geliştirilmiştir20. Ayrıca bu protokolle birlikte, lambda DNA'sının 12 μm'de çift bağlanması için temel bir model de sağlanmıştır (ek bilgilere bakınız). Bu modelin tasarımının çeşitli yönleri, çeşitli deneysel ihtiyaçlara uyacak şekilde ayarlanabilir. Bunlar, bariyerler ve kaideler arasındaki mesafeyi, DNA yoğunluğunu kontrol etmek için bariyerdeki bitişik kuyucuklar arasındaki boşluğu ve çift bağlama verimliliğini optimize etmek ve çift bağlama uzunluklarındaki belirsizliği en aza indirmek için kaidelerin şeklini ve boyutunu içerir. Diğer pratik zorluklar arasında proteinler için floresan etiketleme stratejilerinin seçimi, manuel veri analizinde yanlılığın en aza indirilmesi ve daha akıcı veri işleme için komut dosyalarının geliştirilmesi yer alabilir.
Burada açıklanan protokol dsDNA substratlarını içerirken, ssDNA'ya dayalı DNA perdeleri de yaygın olarak kullanılmıştır 13,14,15,16. ssDNA substratlarının hazırlanması, M13 ssDNA plazmidinin biyotinile primerler13 ile yuvarlanan daire replikasyonunu kullanır. ssDNA perdesinin montajı ayrıca ikincil yapıyı ortadan kaldırmak ve ssDNA substratını uzatmak için üre ve ssDNA bağlayıcı protein RPA'nın kullanılmasını gerektirir13. RNA ile etkileşime giren proteinlerin çalışmaları için DNA perdelerini RNA veya DNA / RNA hibritlerini kullanacak şekilde genişletmek de mümkün olabilir. Şu anda olduğu gibi, DNA perdesi platformu, çoğu standart bir tampon setinin tekrarlayan enjeksiyonları olan bölüm 5'te ayrıntılı olarak açıklandığı gibi montaj sürecinde otomasyondan yararlanabilir. Bu, aynı anda birden fazla deneyin yürütülmesinde verimliliğin artmasına yol açabilir. Ek olarak, birleşik ve potansiyel olarak makine öğrenimi tabanlı bir analiz komut dosyaları paketi, minimum önyargı ile veri işlemeyi daha da hızlandıracaktır.
Açıklamalar
Yazarların ifşa edecek hiçbir şeyi yok.
Teşekkürler
Yazarlar, DNA perdeleri protokolünü geliştirdikleri, optimize ettikleri ve tartıştıkları ve bu el yazması hakkında yorum yaptıkları için Greene laboratuvarının geçmiş ve mevcut üyelerine teşekkür eder. Bu çalışma, Ulusal Bilim Vakfı (MCB1154511'den ECG'ye) ve Ulusal Sağlık Enstitüleri'nden (R01CA217973'den ECG'ye) alınan hibelerle desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Referanslar
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır