Method Article
Las cortinas de ADN arrojan luz sobre sistemas moleculares complejos durante la recombinación homóloga
* Estos autores han contribuido por igual
En este artículo
Resumen
Las cortinas de ADN presentan un método novedoso para visualizar cientos o incluso miles de proteínas de unión al ADN en tiempo real a medida que interactúan con las moléculas de ADN alineadas en la superficie de una cámara de muestras microfluídica.
Resumen
La recombinación homóloga (HR) es importante para la reparación de las roturas de ADN bicatenario (DSB) y las horquillas de replicación estancadas en todos los organismos. Los defectos en la HR están estrechamente asociados con la pérdida de la integridad del genoma y la transformación oncogénica en las células humanas. La HR implica acciones coordinadas de un conjunto complejo de proteínas, muchas de las cuales siguen siendo poco conocidas. El aspecto clave de la investigación aquí descrita es una tecnología llamada "cortinas de ADN", una técnica que permite el ensamblaje de moléculas de ADN alineadas en la superficie de una cámara de muestras microfluídica. A continuación, se pueden visualizar mediante microscopía de fluorescencia de reflexión interna total (TIRFM). Las cortinas de ADN fueron iniciadas por nuestro laboratorio y permiten el acceso directo a información espacio-temporal a escalas de tiempo de milisegundos y resolución a escala nanométrica, que no se puede revelar fácilmente a través de otras metodologías. Una de las principales ventajas de las cortinas de ADN es que simplifican la recopilación de datos estadísticamente relevantes de experimentos con moléculas individuales. Esta investigación continúa arrojando nuevos conocimientos sobre cómo las células regulan y preservan la integridad del genoma.
Introducción
El mantenimiento de la integridad del genoma es crucial para el correcto funcionamiento de todas las células vivas1. Los defectos en la integridad del genoma pueden provocar graves problemas de salud, incluidos varios tipos de cáncery enfermedades degenerativas relacionadas con la edad. La recombinación homóloga (HR) utiliza la síntesis de ADN dependiente de la plantilla para reparar las roturas de doble cadena (DSB) del ADN, las brechas de ADN monocatenario (ssDNA) y los enlaces cruzados de ADN entre hebras3. HR también es necesario para la recuperación de bifurcaciones de replicación estancadas y colapsadas 3,4. Además, la FC es esencial para la segregación cromosómica precisa durante la meiosis 5,6.
La HR implica las acciones coordinadas de un conjunto complejo de proteínas, muchas de las cuales siguen siendo poco conocidas1. Algunos ejemplos son la proteína de replicación A (RPA), Rad51 y Rad54, entre muchas otras7. Las reacciones HR tanto en células procariotas como eucariotas involucran un intermediario de ssDNA, que es rápidamente recubierto por proteínas de unión a ssDNA (SSB en procariotas y RPA en eucariotas)8. Estas proteínas protegen el ssDNA de las nucleasas, eliminan la estructura secundaria y promueven el reclutamiento de factores posteriores 8,9. Rad51 es un miembro de la familia de recombinasas de ADN Rad51/RecA dependientes de ATP altamente conservadas, que están presentes en todos los organismos vivos1. Rad51 promueve la invasión de la cadena de ADN del donante homólogo de dsDNA. Dada su importancia, Rad51 está altamente regulado, y los defectos en estos procesos regulatorios se asocian comúnmente con una pérdida de integridad del genoma y transformación oncogénica7. Rad54 es un miembro de la familia Swi2/Snf2 de translocasas de ADNd y remodeladores de cromatina10,11. Estas proteínas sirven como factores reguladores esenciales de Rad51. Es importante destacar que Rad54 elimina Rad51 del producto de dsDNA de la invasión de hebras y también es necesario para prevenir la acumulación incorrecta de Rad51 en la cromatina11. Las actividades moleculares de las proteínas implicadas en la HR, tanto en las células de levadura como en las bacterianas, han arrojado luz sobre su función en la HR, pero la forma exacta en que su actividad contribuye a la HR sigue siendo poco conocida.
Las cortinas de ADN se han convertido en una plataforma única que proporciona acceso directo a los mecanismos moleculares y a la dinámica macromolecular que, de otro modo, permanecerían inaccesibles13,14. Para preparar cortinas de ADN, la superficie de una cámara microfluídica se recubre con una bicapa lipídica y las moléculas de ADN se unen a la bicapa a través de un enlace biotina-estreptavidina. La bicapa hace que la superficie sea inerte imitando las membranas celulares naturales. La fuerza hidrodinámica alinea el ADN a lo largo de las barreras nanofabricadas, lo que permite la visualización de cientos de moléculas en un solo campo de visión mediante microscopía de fluorescencia de reflexión interna total (TIRFM). Las barreras se fabrican mediante litografía por haz de electrones, y las variaciones en el diseño de las barreras permiten un control preciso sobre la distribución y la geometría de anclaje del ADN. Estos enfoques son fácilmente aplicables con ssDNA o dsDNA 13,14,15,16,17,18. Los pedestales también pueden ser nanofabricados (junto con las barreras) para permitir que ambos extremos del ADN estén atados a la superficie de la cámara de muestras, de modo que se puedan llevar a cabo experimentos de estado estacionario en ausencia de flujo de tampón.
Los cambios dependientes del tiempo en los complejos individuales proteína-ácido nucleico se revelan mediante la inspección de videos en tiempo real y se representan en forma impresa mediante quimógrafos, que presentan la posición cambiante de las proteínas en el ADN a lo largo del tiempo. Un aspecto importante del enfoque de las cortinas de ADN es que no requiere necesariamente modelos a priori o suposiciones sobre los mecanismos moleculares, ya que el comportamiento de los componentes individuales de la reacción se puede observar en tiempo real. Esto permite la observación directa de los comportamientos moleculares. En este trabajo, se describe cómo preparar cortinas de ADN con sustratos de dsDNA, así como su aplicación para estudiar intermediarios en recombinación homóloga.
Protocolo
1. Preparación del caldo lipídico
- Disuelva 1 g de PC 18:1 (Δ9-Cis), 100 mg de PE PEG2000 18:1 y 5 mg de PE con tapón de biotinil 18:1 en 10 ml de cloroformo.
NOTA: Tanto el DOPC como el DPPE están bien caracterizados y (junto con la modificación del PEG) seleccionados por su capacidad para minimizar la adsorción no específica a la superficie de la celda de flujo. La mezcla maestra de lípidos disueltos puede alícuota y almacenarse a -20 °C hasta 12 meses. - Utilice cloroformo para limpiar una jeringa de vidrio con disolvente orgánico, luego transfiera 200 μL (10% del volumen final deseado) de la mezcla maestra de lípidos al vial de vidrio.
- Evapore el cloroformo del frasco de vidrio usando un chorro muy suave de nitrógeno comprimido. Aumente ligeramente la presión hacia el final de la evaporación para asegurarse de que se eliminen todos los rastros de cloroformo.
NOTA: Incline y gire el vial durante el proceso de evaporación para que los lípidos se sequen como una capa uniforme de película blanca en la pared. - Coloque el vial sin tapar al vacío durante la noche.
- Añada 2 mL de tampón lipídico (10 mM de Tris-HCl [pH 8,0], 100 mM de NaCl) a la película lipídica seca y tape el vial. Incubar a temperatura ambiente (RT) durante al menos 1 h y en vórtice hasta que se disuelva.
- Transfiera la solución a un tubo de poliestireno de fondo redondo de 5 mL y sonique sobre hielo con una micropunta con los siguientes parámetros: amplitud = 50, tiempo de proceso = 1,5 min, pulso = 15 s, tiempo de inactividad = 2 min. La solución será clara después de la sonicación (energía total = 1.500-2.000 J).
- Filtre la solución a través de un filtro de jeringa de PVDF de 0,22 μm y almacene el caldo de lípidos a 4 °C durante un máximo de 1 mes.
2. Preparación del sustrato de dsDNA
- Calentar el caldo de λ-ADN (500 μg/mL) a 65 °C.
- Prepare una mezcla de recocido/ligadura que contenga: 1,6 pM de λ-ADN, 100x exceso molar (160 pM) de cada oligonucleótido (BioL: 5'Phos-AGG TCG CCG CCC-3Bio y DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) y 60 μL de tampón de reacción de ligasa T4 10x (en un volumen total de 600 μL).
NOTA: Los oligonucleótidos BioL y DigR están biotinilados (Bio) y modificados con digoxigenina (Dig), respectivamente. La biotinilación permite la unión de moléculas de ADN a lípidos biotinilados a través de enlaces estreptavidina. La modificación Dig permite el doble anclaje a través de la unión de anticuerpos anti-Dig que están recubiertos en pedestales a las partes de Dig. - Recocer los oligonucleótidos a los extremos cósmicos del ADN λ incubando la reacción a 65 °C durante 10 min, luego dejar que se enfríe lentamente hasta RT.
- Añadir 5 μL de ADN ligasa T4 e incubar en RT durante la noche.
NOTA: Los pasos de recocido y ligadura también se pueden llevar a cabo en un termociclador. - Precipitar el λ-ADN añadiendo 300 μL de solución de PEG-8000 (30% p/v de PEG-8000 y 10 mM MgCl2). Incubar a 4 °C durante al menos 1 h.
- Centrifugar a 14.000 x g durante 5 min en RT. Retire con cuidado el sobrenadante sin alterar el pellet. Opcionalmente, lavar el pellet 1x con 500 μL de etanol refrigerado al 70%.
- Vuelva a suspender el pellet en 40 μL de tampón TE (10 mM de Tris-HCl, 1 mM de EDTA [pH 8,0]).
3. Nanofabricación de patrones de cromo
- Taladre dos orificios en el centro de un portaobjetos de microscopio de cuarzo de 1" x 3" (a ~3 cm de distancia) utilizando una broca de 1,4 mm recubierta de diamante en una taladradora de mesa (consulte la Figura 1 para ver la geometría de la posición de perforación).
- Limpie los portaobjetos perforados sumergiéndolos en 200 mL de solución de piraña (75% de ácido sulfúrico [97%] y 25% de peróxido de hidrógeno). Incubar durante 30 min.
PRECAUCIÓN: Se debe tener mucho cuidado para evitar todo contacto con la piel expuesta. Prepare la solución mezclando muy lentamente peróxido de hidrógeno frío en ácido sulfúrico. Deseche los desechos de pirañas correctamente en un contenedor de desechos designado. - Enjuague los portaobjetos sumergiéndolos en 200 ml de agua bidestilada. Repita 2 veces para eliminar cualquier piraña restante. Seque los portaobjetos con un suave chorro de nitrógeno comprimido.
- Utilizando un recubridor giratorio (4.000 rpm durante 45 s, velocidad de rampa de 300 rpm/s), recubra los portaobjetos limpios con fotorresistencia y polímero conductor de la siguiente manera:
- Recubra los portaobjetos con una capa de 3% (p/v) de polimetilmetacrilato (PMMA; 24.3K MW; disuelto en anisol).
- Recubrir los portaobjetos con una capa de polimetilmetacrilato (PMMA; 495K MW; disuelto en anisol) al 1,5% (p/v).
- Recubra los portaobjetos con una capa de agente antiestático para la litografía por haz de electrones.
- Escriba patrones personalizados para barreras y pedestales en los portaobjetos recubiertos utilizando litografía de haz de electrones con un microscopio electrónico de barrido equipado con software de sistema de generación de nanopatrones.
- Revele los portaobjetos enjuagando primero el agente antiestático para la litografía por haz de electrones con agua bidestilada. A continuación, colóquelos en tubos de 50 ml que contengan la solución en desarrollo (75% de metil isobutilcetona [MIBK] y 25% de isopropanol; manténgalos fríos a -20 °C) y sonicase en un baño de hielo a baja potencia durante 1 min.
- Enjuague la solución en desarrollo con isopropanol y seque con un chorro suave de nitrógeno comprimido.
- Utilizando un evaporador de haz de electrones, deposite una capa de cromo de 250 Å de espesor sobre la superficie modelada a 0,5 Å/s.
- Retire el cromo y el PMMA restante con acetona de una botella con atomizador, seguido de la sonicación de los portaobjetos en un baño de acetona durante 10 minutos. Lavar de nuevo con acetona de un frasco con atomizador.
- Para evitar que se depositen los restos de acetona, enjuague los portaobjetos con alcohol isopropílico y séquelos con un suave chorro de nitrógeno comprimido.
4. Montaje de la celda de flujo
- Coloque una plantilla de papel rectangular de 35 mm x 5 mm (cubriendo el patrón cromado y los orificios de entrada/salida preperforados) sobre el portaobjetos de cuarzo, con el lado estampado hacia arriba ( Figura 1A).
- Aplique un trozo de cinta adhesiva de doble cara (1 mm de grosor) sobre el portaobjetos de cuarzo, cubriendo la plantilla de papel, luego use una cuchilla de afeitar para cortar la plantilla de papel rectangular ( Figura 1A).
- Coloque un cubreobjetos de vidrio sobre la cinta adhesiva de doble cara y aplique una presión suave para sellar el cubreobjetos a la cinta ( Figura 1A). El espacio entre el cubreobjetos y el portaobjetos, sellado con cinta adhesiva de doble cara, forma la cámara microfluídica.
NOTA: Es crucial que el sello entre el cubreobjetos y la corredera esté lo suficientemente seguro para evitar fugas durante el experimento. - Coloque el sándwich de deslizamiento entre los portaobjetos de vidrio y coloque clips de carpeta en los cuatro lados para distribuir la presión de manera uniforme. Coloque el conjunto en un horno de vacío durante 60 minutos a 135 °C para derretir la cinta adhesiva de doble cara y sellar la cámara.
- Sácalo del horno y luego suelta los clips de carpeta y los portaobjetos de vidrio.
- Fije los puertos microfluídicos a los orificios de entrada/salida preperforados con una pistola de pegamento caliente colocando el conjunto pegado en un bloque calefactor (220 °C) durante 1 min, retirándolo y aplicando una presión suave con cuidado para sellar correctamente los puertos microfluídicos. Deje que el pegamento se cure por completo antes de continuar.
NOTA: Las celdas de flujo ensambladas pueden colocarse en tubos de 50 ml y almacenarse al vacío.
5. Montaje de la cortina dsDNA
- Conecte el tubo de entrada/salida a los puertos microfluídicos de la celda de flujo y lave la cámara con 20 ml de agua destilada doble con dos jeringas de 10 ml.
- Con una jeringa de 10 ml llena de agua destilada doble conectada a ambos lados de la celda de flujo, aplique fuerza para empujar/tirar del agua entre las jeringas de entrada y salida. Este paso elimina todas las burbujas de aire de la celda de flujo.
- Lave la cámara con 3 mL de tampón lipídico (10 mM de Tris-HCl [pH 8.0], 100 mM de NaCl).
NOTA: Asegúrese de que todas las conexiones a las celdas de flujo sean de gota a gota para evitar la introducción de burbujas de aire en la celda de flujo. - Mezclar 40 μL de solución de liposomas con 1 mL de tampón lipídico. Inyecte 300 μL de la solución lipídica en la celda de flujo e incube durante 5 min. Repita la inyección y la incubación 2 veces.
NOTA: Alterne el lado de entrada/salida para la inyección cada vez que se conecte una jeringa a la celda de flujo. - Lavar la cámara con 3 mL de tampón lipídico e incubar durante 15 min.
NOTA: Este período de incubación puede variar de 5 min a 2 h, dependiendo de la programación de los experimentos, sin diferencias notables en el montaje final de la cortina. - Mezclar 40 μL de 1 mg/mL de solución madre anti-digoxigenina con 200 μL de tampón lipídico. Inyectar 100 μL de la solución e incubar durante 10 min. Repita la inyección y la incubación 1 vez antes del lavado con 3 ml de tampón lipídico.
NOTA: Este paso solo es necesario para cortinas de dsDNA de doble atadura. Omita este paso para las cortinas de dsDNA de un solo cable. - Lave la cámara con 3 mL de tampón BSA (40 mM de Tris-HCl [pH 8.0], 2 mM de MgCl2, 1 mM de DTT y 0.2 mg/mL de BSA).
- Mezclar 10 μL de solución madre de estreptavidina de 1 mg/mL con 990 μL de tampón BSA. Inyecte como dos inyecciones separadas de 400 μL con una incubación de 10 minutos después de cada inyección.
- Lave la cámara con 3 mL de tampón BSA para eliminar el exceso de estreptavidina.
- Diluir 25 ng de sustrato de dsDNA en 1 mL de tampón BSA. Inyecte lentamente la solución de ADN 200 μL a la vez, seguido de una incubación de 5 minutos después de cada inyección. La cantidad de ADN necesaria puede optimizarse para tipos específicos de experimentos.
- Monte la celda de flujo en la platina del microscopio y ajuste el enfoque según sea necesario.
- Conecte los puertos de entrada y salida a un sistema de inyección de muestras compuesto por una bomba de jeringa y una válvula de interruptor de alta presión.
- Amarre dos veces el ADN a los pedestales (longitud de amarre de 12 μm) lavando continuamente la cámara con tampón BSA a 0,8 mL/min durante 5 min.
NOTA: Puede ser necesario ajustar los caudales y los tiempos para optimizar las diferentes longitudes de doble correa. Este paso solo es necesario para las cortinas de ADN de doble atadura. Omita este paso para las cortinas de dsDNA de un solo cable. - Las cortinas ya están listas para los experimentos.
6. Reciclaje de celdas de flujo
- Sumerja los portaobjetos en etanol durante al menos 30 minutos antes de usar una cuchilla de afeitar para quitar con cuidado los puertos microfluídicos y el cubreobjetos. Deseche correctamente el cubreobjetos y limpie cualquier resto de pegamento de los puertos microfluídicos para que puedan reutilizarse. Frote cualquier residuo de pegamento en el portaobjetos.
NOTA: Tenga cuidado de no rayar el área estampada en el centro de la diapositiva. - Enjuague bien los portaobjetos con agua bidestilada y sumérjalos en concentrado de limpieza de cuarzo líquido alcalino al 2% durante la noche, con agitación.
- Enjuague bien los portaobjetos con agua bidestilada y sumérjalos en una solución líquida concentrada de limpieza de cuarzo al 2% durante 1 h agitando a 80 °C, seguido de sonicación durante 15 min a 45 °C.
- Enjuague bien los portaobjetos con agua bidestilada y sumérjalos en NaOH 1 M durante 30-60 min, con agitación.
- Enjuague bien los portaobjetos con agua bidestilada, luego sumérjalos en alcohol isopropílico y soniquelos durante 15 minutos a 45 °C.
- Enjuague bien los portaobjetos con etanol y sumérjalos en etanol durante 1 h, agitando.
- Coloque los portaobjetos en un horno al vacío durante 1 h para que se sequen a 135 °C. Las guías ahora están listas para ser reensambladas en celdas de flujo.
Resultados
Lo anterior se describe como la preparación, el ensamblaje y la obtención de imágenes de cortinas de ADNd de anclaje simple y doble en el contexto del estudio de las interacciones proteína-ADN en los intermediarios de reparación del ADN. La Figura 1A muestra todos los componentes de una celda de flujo, en capas en el orden en que se ensamblan. La Figura 1B muestra el esquema de una cortina de ADN de una o dos ataduras. Se utiliza una bicapa lipídica para pasivar la superficie de la celda de flujo. La cortina de ADN consiste en una matriz paralela de moléculas de ADN atadas en un extremo al lípido y alineadas en la barrera de cromo, orientadas perpendicularmente a la dirección del flujo. En el caso de las cortinas de ADN de doble unión, el otro extremo del ADN está atado al pedestal a través de interacciones entre la digoxigenina y la anti-digoxigenina, de modo que las imágenes se pueden llevar a cabo en ausencia de flujo mientras el ADN permanece extendido. El dsDNA se puede teñir con colorante fluorescente YoYo1 y visualizar mediante microscopía TIRF. La Figura 2A muestra una imagen representativa de campo amplio de la cortina de ADN de doble anclaje teñida con YoYo1.
Las series de imágenes de lapso de tiempo recopiladas de los experimentos de cortina de ADN generalmente se analizan generando primero un quimógrafo, que traza la posición a lo largo de la molécula de ADN en el eje vertical frente al tiempo en el eje horizontal para cada molécula de ADN de interés en ImageJ. La Figura 2B son cimogramas representativos que muestran la resección del ADN λ (verde) teñido con YoYo1 por los mecanismos de resección de levaduras GFP-Sgs1 (no visible), Top3-Rmi1 y Dna2 en presencia de la proteína de unión al ADN monocatenario RPA-mCherry (magenta) y ATP. La señal YoYo1 se pierde con el tiempo a medida que el dsDNA es resecado desde el extremo libre por Sgs1-Dna2. Simultáneamente, la señal mCherry se colocaliza con los extremos del ADN en los que se genera el ssDNA como resultado de la resección. Las características biofísicamente relevantes de la resección, como la velocidad y la procesividad, se pueden extraer cuantificando las pendientes de las trayectorias de pérdida de señal de YoYo1. La Figura 2C,D muestra la distribución de velocidades y procesividad, respectivamente, de la resección por Sgs1-Dna2.

Figura 1: Esquemas del ensamblaje de la celda de flujo y las cortinas de ADN. (A) Ilustración escalonada para el ensamblaje de la celda de flujo. Se coloca cinta adhesiva de doble cara en la parte superior del portaobjetos de cuarzo, y se usa una plantilla de papel para cortar un canal rectangular desde el centro, que se sella con un cubreobjetos en la parte superior para formar una cámara. (B) Esquemas de cortinas de ADN completamente ensambladas. El panel central presenta una celda de flujo de un solo cableado, mientras que el panel inferior presenta una celda de flujo de doble conexión. Haga clic aquí para ver una versión más grande de esta figura.
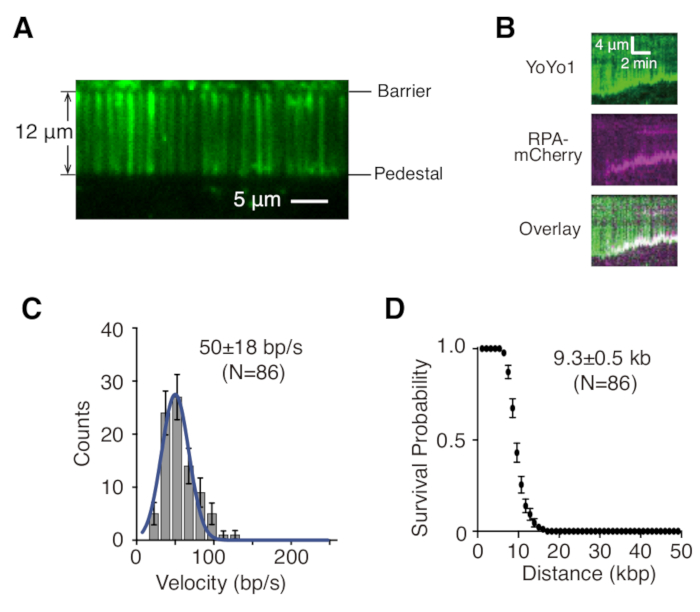
Figura 2: Visualización y análisis de una cortina de ADN de una sola molécula. (A) Imagen representativa de campo amplio de una cortina de λ-ADN de doble anclaje teñida con YoYo1 (verde). (B) Quimógrafos representativos que muestran la resección de dsDNA teñido con YoYo1 (verde) en presencia de GFP-Sgs1 preunido (no visible) cuando se persigue con Dna2, en presencia de RPA-mCherry (magenta) y 2 mM ATP. Distribución de velocidad (C) y gráfico de procesividad (D) de la resección de dsDNA por Sgs1-Dna2, obtenida cuantificando la tasa y extensión de la pérdida de señal de YoYo1 a lo largo del tiempo. Los paneles (B), (C) y (D) son una adaptación de Xue et al.19. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Las cortinas de ADN han demostrado ser una plataforma versátil para estudiar diversos procesos de reparación del ADN 13,14,15,16. Las moléculas de ADN que permanecen extendidas cerca de la superficie durante todo el experimento, ya sea a través de un flujo de tampón continuo o de doble anclaje al pedestal, permiten la obtención de imágenes TIRFM de las interacciones proteína-ADN en los intermediarios de reparación a nivel de una sola molécula. El método proporciona una mejora en el rendimiento experimental y la previsibilidad, ya que cientos de moléculas de ADN se alinean ordenadamente en barreras nanofabricadas en lugar de adherirse de forma no específica a la superficie, lo que beneficia aún más el procesamiento de grandes conjuntos de datos.
El análisis cuidadoso de la serie de imágenes de lapso de tiempo resultante proporciona mediciones cuantitativas de las interacciones proteína-ADN (es decir, tiempos de vida de unión, posiciones, estequiometría, cinética de disociación, tasas de translocación, procesividades de las proteínas motoras y colocalizaciones e interacciones entre proteínas y / o ADN). En comparación con otras técnicas de adquisición de imágenes de una sola molécula que atan las moléculas de ADN directamente a la superficie o combinan la obtención de imágenes confocales con la manipulación de la fuerza en instrumentos comerciales, la principal desventaja de las cortinas de ADN para un mayor rendimiento es establecer las técnicas necesarias para la configuración inicial 13,14,15,16. Sin embargo, una vez establecida, la capacidad de recopilar datos sobre cientos de moléculas simultáneamente en una celda de flujo conduce a un ahorro significativo en el tiempo dedicado a la recopilación de datos.
Al igual que con muchos otros métodos de adquisición de imágenes de una sola molécula, la capacidad de preparar una superficie limpia dicta en gran medida la capacidad del usuario para ensamblar de manera confiable cortinas de ADN. En este caso, las superficies limpias requieren no solo comenzar con un portaobjetos limpio, sino también asegurarse de no introducir burbujas de aire microscópicas en la cámara mientras se realiza ninguna de las conexiones fluídicas. En primer lugar, con respecto a la limpieza de los portaobjetos, los pasos detallados anteriormente en la sección 6 deberían ser suficientes como un proceso de limpieza de rutina. También se recomienda el uso de soluciones filtradas y tampones. En segundo lugar, es esencial realizar conexiones de gota a gota durante la conexión de la celda de flujo al sistema de inyección de tampón para evitar la introducción de burbujas. Puede ser útil desgasificar el agua bidestilada utilizada para hacer tampones. También se recomienda observar cuidadosamente cualquier proceso de inyección para asegurarse de que no haya burbujas. En caso de que se introduzca una burbuja microscópica antes de que alcance el área central del patrón, se puede remediar inyectando tampón desde el puerto microfluídico en el lado opuesto para empujar la burbuja hacia afuera.
Otro punto importante a tener en cuenta en cualquier experimento basado en fluorescencia es la relación señal-ruido. Una clave para observar proteínas fluorescentes individuales (es decir, GFP) también reside en superficies limpias. Después de un uso prolongado, los agregados de proteínas generalmente se acumulan en las barreras y pedestales de cromo. Por lo tanto, si se observa una fuerte señal de fluorescencia en las barreras antes de la adición de proteínas fluorescentes, se recomienda tratar los portaobjetos con una inmersión de 10 minutos en solución de piraña. El período de tiempo se mantiene corto para evitar erosionar las características de Chrome. El lavado de Piraña debe ir seguido del procedimiento de limpieza regular.
La nanofabricación mediante litografía por haz de electrones puede ser un reto, debido a los requisitos de equipo e instalaciones, así como a la experiencia existente en la ciencia de los materiales. Como alternativa, se ha desarrollado un método basado en litografía UV para la fabricación de características de cromo para cortinas de ADN20. También se proporciona con este protocolo un patrón básico para el ADN lambda de doble anclaje a 12 μm (ver información complementaria). Varios aspectos del diseño de este patrón se pueden ajustar para adaptarse a diversas necesidades experimentales. Estos incluyen la distancia entre barreras y pedestales, el espaciamiento entre pozos adyacentes en la barrera para controlar la densidad del ADN y la forma y el tamaño de los pedestales para optimizar la eficiencia de doble anclaje y minimizar la incertidumbre en las longitudes de doble anclaje. Otros desafíos prácticos pueden incluir la elección de estrategias de etiquetado fluorescente para proteínas, la minimización del sesgo en el análisis manual de datos y el desarrollo de scripts para un procesamiento de datos más optimizado.
Si bien el protocolo descrito aquí involucra sustratos de dsDNA, las cortinas de ADN basadas en ssDNA también se han utilizado ampliamente 13,14,15,16. La preparación de sustratos de ssDNA utiliza la replicación en círculo rodante del plásmido M13 ssDNA con cebadores biotinilados13. El ensamblaje de la cortina de ssDNA también requiere el uso de urea y la proteína de unión a ssDNA RPA para eliminar la estructura secundaria y extender el sustrato de ssDNA13. También puede ser posible ampliar las cortinas de ADN para utilizar ARN o híbridos de ADN/ARN para estudios de proteínas que interactúan con el ARN. En su estado actual, la plataforma de cortina DNA puede beneficiarse de la automatización en el proceso de montaje, como se detalla en la sección 5, la mayor parte del cual consiste en inyecciones repetitivas de un conjunto estándar de tampones. Esto puede conducir a una mayor eficiencia en la realización de múltiples experimentos simultáneamente. Además, un conjunto de scripts de análisis unificados y potencialmente basados en el aprendizaje automático aceleraría aún más el procesamiento de datos con un sesgo mínimo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen a los miembros pasados y actuales del laboratorio de Greene por desarrollar, optimizar y discutir el protocolo de cortinas de ADN, así como por comentar sobre este manuscrito. Este trabajo fue apoyado por subvenciones de la Fundación Nacional de Ciencias (MCB1154511 a E.C.G.) y los Institutos Nacionales de Salud (R01CA217973 a E.C.G.).
Materiales
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Referencias
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados