Method Article
Le tende di DNA fanno luce su sistemi molecolari complessi durante la ricombinazione omologa
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le tende di DNA rappresentano un nuovo metodo per visualizzare centinaia o addirittura migliaia di proteine che legano il DNA in tempo reale mentre interagiscono con le molecole di DNA allineate sulla superficie di una camera di campionamento microfluidica.
Abstract
La ricombinazione omologa (HR) è importante per la riparazione delle rotture del DNA a doppio filamento (DSB) e delle forcelle di replicazione bloccate in tutti gli organismi. I difetti dell'HR sono strettamente associati a una perdita di integrità del genoma e alla trasformazione oncogenica nelle cellule umane. La HR coinvolge azioni coordinate di un insieme complesso di proteine, molte delle quali rimangono poco comprese. L'aspetto chiave della ricerca qui descritta è una tecnologia chiamata "DNA curtains", una tecnica che consente l'assemblaggio di molecole di DNA allineate sulla superficie di una camera di campionamento microfluidica. Possono quindi essere visualizzati mediante microscopia a fluorescenza a riflessione interna totale (TIRFM). Le tende di DNA sono state sperimentate dal nostro laboratorio e consentono l'accesso diretto alle informazioni spazio-temporali su scale temporali di millisecondi e con risoluzione su scala nanometrica, che non possono essere facilmente rivelate attraverso altre metodologie. Uno dei principali vantaggi delle tende di DNA è che semplificano la raccolta di dati statisticamente rilevanti da esperimenti su singole molecole. Questa ricerca continua a produrre nuove intuizioni su come le cellule regolano e preservano l'integrità del genoma.
Introduzione
Il mantenimento dell'integrità del genoma è fondamentale per il corretto funzionamento di tutte le cellule viventi1. I difetti nell'integrità del genoma possono portare a gravi condizioni di salute, tra cui vari tipi di cancro e malattie degenerative legate all'età2. La ricombinazione omologa (HR) utilizza la sintesi del DNA dipendente dal modello per riparare le rotture del DNA a doppio filamento (DSB), le lacune del DNA a filamento singolo (ssDNA) e i legami incrociati del DNA tra filamenti3. L'HR è necessario anche per il recupero di fork di replicazione bloccate e collassate 3,4. Inoltre, l'HR è essenziale per l'accurata segregazione cromosomica durante la meiosi 5,6.
L'HR coinvolge le azioni coordinate di un complesso insieme di proteine, molte delle quali rimangono poco conosciute1. Gli esempi includono la proteina di replicazione A (RPA), Rad51 e Rad54, tra molte altre7. Le reazioni HR sia nelle cellule procariotiche che in quelle eucariotiche coinvolgono un intermedio di ssDNA, che viene rapidamente rivestito da proteine leganti l'ssDNA (SSB nei procarioti e RPA negli eucarioti)8. Queste proteine proteggono l'ssDNA dalle nucleasi, eliminano la struttura secondaria e promuovono il reclutamento dei fattori a valle 8,9. Rad51 è un membro della famiglia di ricombinasi del DNA Rad51/RecA, altamente conservata ATP-dipendente, che sono presenti in tutti gli organismi viventi1. Rad51 promuove l'invasione del filamento di DNA del donatore omologo di dsDNA. Data la sua importanza, Rad51 è altamente regolamentato e i difetti in questi processi regolatori sono comunemente associati a una perdita di integrità del genoma e alla trasformazione oncogenica7. Rad54 è un membro della famiglia Swi2/Snf2 di traslocasi dsDNA e rimodellatori di cromatina10,11. Queste proteine fungono da fattori regolatori essenziali di Rad51. È importante sottolineare che Rad54 rimuove Rad51 dal prodotto dsDNA dell'invasione dei filamenti ed è anche necessario per prevenire l'accumulo errato di Rad51 sulla cromatina11. Le attività molecolari delle proteine coinvolte nell'HR sia nel lievito che nelle cellule batteriche hanno fatto luce sulla loro funzione nell'HR, ma il modo esatto in cui la loro attività contribuisce all'HR rimane poco compreso 12.
Le tende di DNA sono emerse come una piattaforma unica che fornisce accesso diretto ai meccanismi molecolari e alle dinamiche macromolecolari che altrimenti rimarrebbero inaccessibili13,14. Per preparare le tende di DNA, la superficie di una camera microfluidica è rivestita con un doppio strato lipidico e le molecole di DNA sono legate al doppio strato attraverso un legame biotina-streptavidina. Il doppio strato rende inerte la superficie imitando le membrane cellulari naturali. La forza idrodinamica allinea il DNA lungo barriere nanofabbricate, consentendo la visualizzazione di centinaia di molecole in un unico campo visivo mediante microscopia a fluorescenza a riflessione interna totale (TIRFM). Le barriere sono realizzate mediante litografia a fascio di elettroni e le variazioni nel design delle barriere consentono un controllo preciso sulla distribuzione e sulla geometria di ancoraggio del DNA. Questi approcci sono facilmente applicabili sia con ssDNA che con dsDNA 13,14,15,16,17,18. I piedistalli possono anche essere nanofabbricati (insieme alle barriere) per consentire a entrambe le estremità del DNA di essere legate alla superficie della camera del campione in modo che gli esperimenti allo stato stazionario possano essere eseguiti in assenza di flusso tampone.
I cambiamenti dipendenti dal tempo nei singoli complessi proteina-acido nucleico sono rivelati dall'ispezione di video in tempo reale e sono rappresentati in stampa utilizzando chimografi, che presentano la posizione mutevole delle proteine sul DNA nel tempo. Un aspetto importante dell'approccio delle tende di DNA è che non richiede necessariamente modelli a priori o ipotesi sui meccanismi molecolari, perché il comportamento dei singoli componenti della reazione può essere osservato in tempo reale. Ciò consente l'osservazione diretta dei comportamenti molecolari. In questo documento, questo protocollo descrive come preparare tende di DNA con substrati di dsDNA e la sua applicazione per studiare gli intermedi nella ricombinazione omologa.
Protocollo
1. Preparazione dello stock lipidico
- Sciogliere 1 g di PC 18:1 (Δ9-Cis), 100 mg di EP 18:1 PEG2000 e 5 mg di PE con tappo biotinilico 18:1 in 10 mL di cloroformio.
NOTA: Sia DOPC che DPPE sono ben caratterizzati e (insieme alla modifica PEG) selezionati per la loro capacità di ridurre al minimo l'adsorbimento non specifico sulla superficie della cella a flusso. La miscela master lipidica disciolta può essere aliquotata e conservata a -20 °C fino a 12 mesi. - Utilizzare il cloroformio per pulire una siringa di vetro con solvente organico, quindi trasferire 200 μl (10% del volume finale desiderato) della miscela master lipidica nella fiala di vetro.
- Far evaporare il cloroformio dal flaconcino di vetro utilizzando un getto molto delicato di azoto compresso. Aumentare leggermente la pressione verso la fine dell'evaporazione per assicurarsi che tutte le tracce di cloroformio vengano rimosse.
NOTA: Inclinare e ruotare la fiala durante il processo di evaporazione in modo che i lipidi si asciughino come uno strato uniforme di pellicola bianca sulla parete. - Posizionare la fiala senza tappo sotto vuoto per una notte.
- Aggiungere 2 mL di tampone lipidico (10 mM Tris-HCl [pH 8,0], 100 mM NaCl) al film lipidico essiccato e tappare il flaconcino. Incubare a temperatura ambiente (RT) per almeno 1 ora e agitare fino alla dissoluzione.
- Trasferire la soluzione in una provetta di polistirene a fondo tondo da 5 mL e sonicare con ghiaccio utilizzando una micropunta con i seguenti parametri: ampiezza = 50, tempo di processo = 1,5 min, impulso = 15 s, tempo di spegnimento = 2 min. La soluzione sarà limpida dopo la sonicazione (energia totale = 1.500-2.000 J).
- Filtrare la soluzione attraverso un filtro per siringa in PVDF da 0,22 μm e conservare lo stock lipidico a 4 °C per un massimo di 1 mese.
2. Preparazione del substrato del dsDNA
- Riscaldare il materiale di produzione λ-DNA (500 μg/mL) a 65 °C.
- Preparare una miscela di ricottura/legatura contenente: 1,6 pM di λ-DNA, 100 volte l'eccesso molare (160 pM) di ciascun oligonucleotide (BioL: 5'Phos-AGG TCG CCG CCC-3Bio e DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) e 60 μL di tampone di reazione ligasi T4 10x (in un volume totale di 600 μL).
NOTA: Gli oligonucleotidi BioL e DigR sono biotinilati (Bio) e modificati con digossigenina (Dig), rispettivamente. La biotinilazione consente l'adesione delle molecole di DNA ai lipidi biotinilati tramite legami streptavidina. La modifica Dig consente il doppio legame attraverso il legame di anticorpi anti-Dig che sono rivestiti su piedistalli alle frazioni Dig. - Ricuocere gli oligonucleotidi alle estremità del λ-DNA incubando la reazione a 65 °C per 10 minuti, quindi lasciarla raffreddare lentamente a RT.
- Aggiungere 5 μl di DNA ligasi T4 e incubare a RT per una notte.
NOTA: Le fasi di ricottura e legatura possono essere eseguite anche in un termociclatore. - Precipitare il λ-DNA aggiungendo 300 μL di soluzione di PEG-8000 (30% p/v PEG-8000 e 10 mM di MgCl2). Incubare a 4 °C per almeno 1 ora.
- Centrifugare a 14.000 x g per 5 minuti a RT. Rimuovere con cautela il surnatante senza disturbare il pellet. Facoltativamente, lavare il pellet 1 volta con 500 μL di etanolo refrigerato al 70%.
- Risospendere il pellet in 40 μL di tampone TE (10 mM Tris-HCl, 1 mM EDTA [pH 8,0]).
3. Nanofabbricazione di modelli di cromo
- Praticare due fori al centro di un vetrino per microscopio al quarzo da 1" x 3" (~3 cm di distanza) utilizzando una punta da trapano diamantata da 1,4 mm su un trapano a colonna da tavolo (vedere la Figura 1 per la geometria della posizione di foratura).
- Pulire i vetrini perforati immergendoli in 200 mL di soluzione Piranha (75% di acido solforico [97%] e 25% di perossido di idrogeno). Incubare per 30 min.
ATTENZIONE: Prestare la massima attenzione per evitare qualsiasi contatto con la pelle esposta. Preparare la soluzione mescolando molto lentamente il perossido di idrogeno freddo con l'acido solforico. Smaltire correttamente i rifiuti Piranha in un contenitore per rifiuti designato. - Sciacquare i vetrini immergendoli in 200 ml di acqua bidistillata. Ripeti 2 volte per eliminare tutti i Piranha rimasti. Asciugare i vetrini con un getto delicato di azoto compresso.
- Utilizzando una centrifuga (4.000 giri/min per 45 s, velocità di rampa di 300 giri/min), rivestire le diapositive pulite con fotoresist e polimero conduttivo come segue:
- Rivestire i vetrini con uno strato di polimetilmetacrilato al 3% (p/v) (PMMA; 24,3K MW; disciolto in anisolo).
- Rivestire le guide con uno strato di polimetilmetacrilato all'1,5% (p/v) (PMMA; 495K MW; disciolto in anisolo).
- Rivestire i vetrini con uno strato di agente antistatico per litografia a fascio di elettroni.
- Scrivi modelli personalizzati per barriere e piedistalli sui vetrini rivestiti utilizzando la litografia a fascio di elettroni con un microscopio elettronico a scansione dotato di software per la generazione di nanopattern.
- Sviluppare i vetrini risciacquando prima l'agente antistatico per la litografia a fascio di elettroni con acqua a doppia distillazione. Quindi, metterli in provette da 50 mL contenenti la soluzione di sviluppo (75% di metilisobutilchetone [MIBK] e 25% di isopropanolo; conservare in frigorifero a -20 °C) e sonicare in un bagno di ghiaccio a bassa potenza per 1 minuto.
- Sciacquare la soluzione di sviluppo con isopropanolo e asciugare con un getto delicato di azoto compresso.
- Utilizzando un evaporatore a fascio di elettroni, depositare uno strato di cromo spesso 250 Å sulla superficie modellata a 0,5 Å/s.
- Sollevare il cromo e il PMMA rimanente usando l'acetone da un flacone a spruzzo, quindi sonicare i vetrini in un bagno di acetone per 10 minuti. Lavare nuovamente con acetone da un flacone a spruzzo.
- Per evitare depositi di acetone rimasto, sciacquare i vetrini con alcol isopropilico e asciugarli con un getto delicato di azoto compresso.
4. Assemblaggio della cella di flusso
- Posizionare una sagoma di carta rettangolare da 35 mm x 5 mm (che copra il motivo cromato e i fori di ingresso/uscita preforati) sopra il vetrino di quarzo, con il lato modellato rivolto verso l'alto (Figura 1A).
- Applicare un pezzo di nastro biadesivo (spessore 1 mm) sul vetrino di quarzo, coprendo la sagoma di carta, quindi utilizzare una lama di rasoio per ritagliare la sagoma di carta rettangolare (Figura 1A).
- Posizionare un vetrino coprioggetti di vetro sul nastro biadesivo e applicare una leggera pressione per sigillare il vetrino coprioggetti al nastro (Figura 1A). Lo spazio tra il vetrino coprioggetti e il vetrino, sigillato da nastro biadesivo, forma la camera microfluidica.
NOTA: È fondamentale che la guarnizione tra il vetrino coprioggetto e il vetrino sia sufficientemente sicura da evitare perdite durante l'esperimento. - Posizionare il sandwich vetrino coprioggetti tra i vetrini e fissare le clip del raccoglitore su tutti e quattro i lati per distribuire uniformemente la pressione. Mettere l'insieme in un forno sottovuoto per 60 minuti a 135 °C per sciogliere il nastro biadesivo e sigillare la camera.
- Rimuoverlo dal forno, quindi rilasciare le clip del raccoglitore e i vetrini.
- Fissare le porte microfluidiche ai fori di ingresso/uscita preforati con una pistola per colla a caldo posizionando il gruppo incollato su un blocco riscaldante (220 °C) per 1 minuto, rimuovendolo e applicando con cura una leggera pressione per sigillare correttamente le porte microfluidiche. Lasciare indurire completamente la colla prima di procedere.
NOTA: Le celle di flusso assemblate possono essere collocate in provette da 50 ml e conservate sotto vuoto.
5. Montaggio della tenda dsDNA
- Collegare il tubo di ingresso/uscita alle porte microfluidiche della cella di flusso e lavare la camera con 20 ml di acqua bidistillata utilizzando due siringhe da 10 ml.
- Con una siringa da 10 ml riempita con acqua bidistillata collegata a entrambi i lati della cella a flusso, applicare forza per spingere/tirare l'acqua tra le siringhe di ingresso e di uscita. Questo passaggio rimuove tutte le bolle d'aria dalla cella di flusso.
- Lavare la camera con 3 mL di tampone lipidico (10 mM Tris-HCl [pH 8,0], 100 mM NaCl).
NOTA: Assicurarsi che tutti i collegamenti alle celle di flusso siano goccia a goccia per evitare l'introduzione di bolle d'aria nella cella di flusso. - Miscelare 40 μL di soluzione liposomica con 1 mL di tampone lipidico. Iniettare 300 μL della soluzione lipidica nella cella a flusso e incubare per 5 minuti. Ripetere l'iniezione e l'incubazione 2 volte.
NOTA: Alternare il lato di ingresso/uscita per l'iniezione ogni volta che una siringa viene collegata alla cella a flusso. - Lavare la camera con 3 mL di tampone lipidico e incubare per 15 minuti.
NOTA: Questo periodo di incubazione può variare da 5 minuti a 2 ore, a seconda della programmazione degli esperimenti, senza differenze evidenti nell'assemblaggio finale della tenda. - Miscelare 40 μL di soluzione madre di anti-digossigenina da 1 mg/mL con 200 μL di tampone lipidico. Iniettare 100 μl della soluzione e incubare per 10 minuti. Ripetere l'iniezione e l'incubazione 1 volta prima del lavaggio con 3 ml di tampone lipidico.
NOTA: Questo passaggio è necessario solo per le tende dsDNA a doppio legame. Salta questo passaggio per le tende dsDNA single-tethered. - Lavare la camera con 3 mL di tampone BSA (40 mM Tris-HCl [pH 8,0], 2 mM MgCl2, 1 mM DTT e 0,2 mg/mL BSA).
- Miscelare 10 μL di soluzione madre di streptavidina da 1 mg/mL con 990 μL di tampone BSA. Iniettare in due iniezioni separate da 400 μL con un'incubazione di 10 minuti dopo ogni iniezione.
- Lavare la camera con 3 ml di tampone BSA per rimuovere l'eccesso di streptavidina.
- Diluire 25 ng di substrato di dsDNA in 1 mL di tampone BSA. Iniettare lentamente la soluzione di DNA 200 μl alla volta, seguita da un'incubazione di 5 minuti dopo ogni iniezione. La quantità di DNA necessaria può essere ottimizzata per tipi specifici di esperimenti.
- Montare la cella di flusso sul tavolino del microscopio e regolare la messa a fuoco secondo necessità.
- Collegare le porte di ingresso e di uscita a un sistema di iniezione del campione composto da una pompa a siringa e da una valvola di pressostato di alta pressione.
- Fissare il DNA ai piedistalli (lunghezza del cavo di 12 μm) lavando continuamente la camera con tampone BSA a 0,8 mL/min per 5 minuti.
NOTA: Potrebbe essere necessario regolare le portate e i tempi per ottimizzare le diverse lunghezze del doppio cavo. Questo passaggio è necessario solo per le tende di DNA a doppio legame. Salta questo passaggio per le tende dsDNA single-tethered. - Il sipario è ora pronto per gli esperimenti.
6. Riciclo delle celle a flusso
- Immergere i vetrini in etanolo per almeno 30 minuti prima di utilizzare una lama di rasoio per rimuovere con cura le porte microfluidiche e il vetrino coprioggetti. Smaltire correttamente il vetrino coprioggetti e pulire l'eventuale colla residua dalle porte microfluidiche in modo che possano essere riutilizzate. Strofinare per pulire eventuali residui di colla sul vetrino.
NOTA: Fare attenzione a non graffiare l'area modellata al centro della diapositiva. - Sciacquare accuratamente i vetrini con acqua bidistillata e immergerli in concentrato liquido alcalino al quarzo al 2% per una notte, agitando.
- Sciacquare accuratamente i vetrini con acqua bidistillata e immergerli in una soluzione concentrata di quarzo liquido alcalino al 2% per 1 ora agitando a 80 °C, quindi sonicando per 15 minuti a 45 °C.
- Sciacquare accuratamente i vetrini con acqua bidistillata e immergerli in 1 M di NaOH per 30-60 minuti, mescolando.
- Sciacquare accuratamente i vetrini con acqua bidistillata, quindi immergerli in alcol isopropilico e sonicare per 15 minuti a 45 °C.
- Sciacquare accuratamente i vetrini con etanolo e immergerli in etanolo per 1 ora, mescolando.
- Mettere i vetrini in forno sottovuoto per 1 ora ad asciugare a 135 °C. I vetrini sono ora pronti per essere riassemblati in celle di flusso.
Risultati
Sopra descritti sono la preparazione, l'assemblaggio e l'imaging di tende di dsDNA a singolo e doppio legame nel contesto dello studio delle interazioni proteina-DNA negli intermedi di riparazione del DNA. La Figura 1A mostra tutti i componenti di una cella di flusso, stratificati nell'ordine in cui sono assemblati. La Figura 1B illustra lo schema di una cortina di DNA a uno o due legami. Un doppio strato lipidico viene utilizzato per passivare la superficie della cella di flusso. La cortina di DNA è costituita da una serie parallela di molecole di DNA legate a un'estremità al lipide e allineate alla barriera del cromo, orientate perpendicolarmente alla direzione del flusso. Per le tende di DNA a doppio legame, l'altra estremità del DNA è legata al piedistallo attraverso le interazioni tra digossigenina e anti-digossigenina, in modo tale che l'imaging possa essere effettuato in assenza di flusso mentre il DNA rimane esteso. Il dsDNA può essere colorato con colorante fluorescente YoYo1 e visualizzato mediante microscopia TIRF. La Figura 2A mostra un'immagine rappresentativa a campo largo della tenda di DNA a doppio legame colorata con YoYo1.
Le serie di immagini time-lapse raccolte dagli esperimenti di DNA curtain vengono tipicamente analizzate generando prima un chimografo, che traccia la posizione lungo la molecola di DNA sull'asse verticale rispetto al tempo sull'asse orizzontale per ogni molecola di DNA di interesse in ImageJ. La Figura 2B sono chimografi rappresentativi che mostrano la resezione del λ-DNA single-tethered colorato con YoYo1 (verde) da parte dei macchinari di resezione del lievito GFP-Sgs1 (non visibile), Top3-Rmi1 e Dna2 in presenza della proteina legante il DNA a singolo filamento RPA-mCherry (magenta) e ATP. Il segnale YoYo1 viene perso nel tempo quando il dsDNA viene resecato dall'estremità libera da Sgs1-Dna2. Contemporaneamente, il segnale di mCherry colocalizza con le estremità del DNA in cui viene generato l'ssDNA come risultato della resezione. Le caratteristiche biofisicamente rilevanti della resezione, come la velocità e la processività, possono essere estratte quantificando le pendenze delle traiettorie di perdita del segnale YoYo1. La Figura 2C,D mostra la distribuzione delle velocità e la processività, rispettivamente, della resezione da parte di Sgs1-Dna2.

Figura 1: Schemi dell'assemblaggio delle celle a flusso e delle tende di DNA. (A) Illustrazione graduale per l'assemblaggio delle celle a flusso. Il nastro biadesivo viene posizionato sopra il vetrino di quarzo e un modello di carta viene utilizzato per ritagliare un canale rettangolare dal centro, che viene sigillato con un vetrino coprioggetto sulla parte superiore per formare una camera. (B) Schemi di tende di DNA completamente assemblate. Il pannello centrale presenta una cella di flusso a singolo lethered, mentre il pannello inferiore presenta una cella di flusso a doppio tethered. Clicca qui per visualizzare una versione più grande di questa figura.
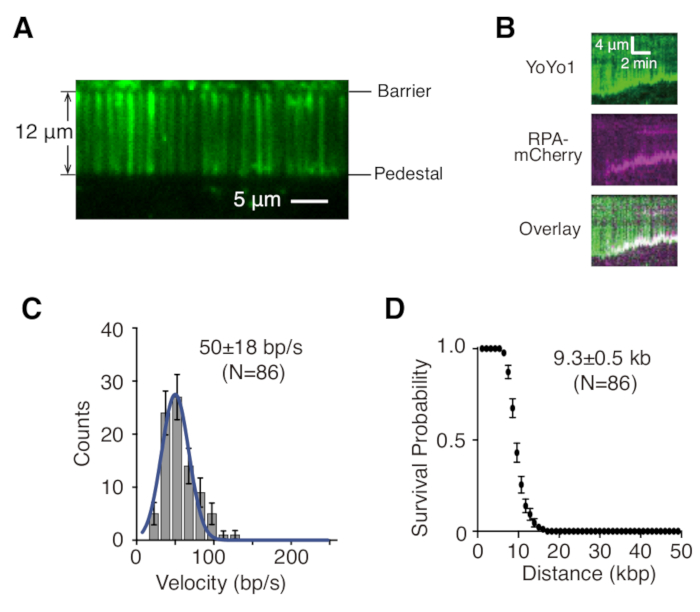
Figura 2: Visualizzazione e analisi della tenda di DNA a singola molecola. (A) Immagine rappresentativa a campo largo di una tenda di λ-DNA a doppio legame colorata con YoYo1 (verde). (B) Chimografi rappresentativi che mostrino la resezione del dsDNA colorato con YoYo1 (verde) in presenza di GFP-Sgs1 pre-legato (non visibile) quando inseguito con Dna2, in presenza di RPA-mCherry (magenta) e 2 mM ATP. Diagramma di distribuzione della velocità (C) e di processività (D) della resezione del dsDNA mediante Sgs1-Dna2, ottenuta quantificando la velocità e l'entità della perdita del segnale di YoYo1 nel tempo. I pannelli (B), (C) e (D) sono adattati da Xue et al.19. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Le tende di DNA hanno dimostrato di essere una piattaforma versatile per lo studio di vari processi di riparazione del DNA 13,14,15,16. Le molecole di DNA che rimangono estese vicino alla superficie durante l'esperimento, attraverso un flusso continuo di tampone o un doppio legame al piedistallo, consentono l'imaging TIRFM delle interazioni proteina-DNA su intermedi di riparazione a livello di singola molecola. Il metodo fornisce un miglioramento della produttività e della prevedibilità sperimentale, poiché centinaia di molecole di DNA sono allineate in modo ordinato su barriere nanofabbricate piuttosto che attaccate in modo non specifico alla superficie, il che avvantaggia ulteriormente l'elaborazione di grandi set di dati.
Un'attenta analisi della serie di immagini time-lapse risultante fornisce misurazioni quantitative delle interazioni proteina-DNA (ad esempio, tempi di vita dei legami, posizioni, stechiometria, cinetica di dissociazione, tassi di traslocazione, processività delle proteine motorie e colocalizzazioni e interazioni tra proteine e/o DNA). Rispetto ad altre tecniche di imaging a singola molecola che legano le molecole di DNA direttamente alla superficie o combinano l'imaging confocale con la manipolazione della forza in strumenti commerciali, il principale compromesso tra le tende di DNA per una maggiore produttività è stabilire le tecniche necessarie per la configurazione iniziale 13,14,15,16. Una volta stabilita, tuttavia, la capacità di raccogliere dati su centinaia di molecole contemporaneamente in una cella a flusso porta a un notevole risparmio di tempo dedicato alla raccolta dei dati.
Come con molti altri metodi di imaging a singola molecola, la capacità di preparare una superficie pulita determina in gran parte la capacità dell'utente di assemblare in modo affidabile le tende di DNA. In questo caso, le superfici pulite richiedono non solo di iniziare con un vetrino pulito, ma anche di assicurarsi di non introdurre microscopiche bolle d'aria nella camera durante la realizzazione di uno qualsiasi dei collegamenti fluidici. Innanzitutto, per quanto riguarda la pulizia dei vetrini, i passaggi descritti sopra nella sezione 6 dovrebbero essere sufficienti come processo di pulizia ordinaria. Si consiglia inoltre l'uso di soluzioni filtrate e tamponi. In secondo luogo, è essenziale effettuare collegamenti goccia a goccia durante il collegamento della cella di flusso al sistema di iniezione del tampone per evitare l'introduzione di bolle. Può essere utile degassare l'acqua a doppia distillazione utilizzata per la produzione di tamponi. Si raccomanda inoltre di osservare attentamente qualsiasi processo di iniezione per assicurarsi che non siano presenti bolle. Se una bolla microscopica viene introdotta prima che raggiunga l'area centrale modellata, si può rimediare iniettando un tampone dalla porta microfluidica sul lato opposto per spingere la bolla verso l'esterno.
Un altro importante punto da considerare in qualsiasi esperimento basato sulla fluorescenza è il rapporto segnale-rumore. Una chiave per osservare singole proteine fluorescenti (cioè GFP) risiede anche nelle superfici pulite. Dopo un uso prolungato, gli aggregati proteici si accumulano tipicamente alle barriere e ai piedistalli cromati. Pertanto, se si osserva un forte segnale di fluorescenza alle barriere prima dell'aggiunta di proteine fluorescenti, si consiglia di trattare i vetrini con un'immersione di 10 minuti in soluzione Piranha. Il periodo di tempo è breve per evitare di erodere le caratteristiche cromate. Il lavaggio Piranha deve essere seguito dalla regolare procedura di pulizia.
La nanofabbricazione mediante litografia a fascio di elettroni può essere impegnativa, a causa dei requisiti di attrezzature e strutture, nonché delle competenze esistenti nella scienza dei materiali. In alternativa, è stato sviluppato un metodo basato sulla litografia UV per la fabbricazione di caratteristiche di cromo per tende di DNA20. Questo protocollo include anche un modello di base per il doppio tethering del DNA lambda a 12 μm (vedi informazioni supplementari). Diversi aspetti della progettazione di questo modello possono essere adattati per soddisfare varie esigenze sperimentali. Questi includono la distanza tra barriere e piedistalli, la spaziatura tra i pozzetti adiacenti nella barriera per controllare la densità del DNA e la forma e le dimensioni dei piedistalli per ottimizzare l'efficienza del doppio tethering e ridurre al minimo l'incertezza nelle lunghezze del doppio tethering. Altre sfide pratiche possono includere la scelta di strategie di marcatura fluorescente per le proteine, la riduzione al minimo delle distorsioni nell'analisi manuale dei dati e lo sviluppo di script per un'elaborazione dei dati più snella.
Mentre il protocollo qui descritto coinvolge substrati di dsDNA, anche le tende di DNA basate su ssDNA sono state ampiamente utilizzate 13,14,15,16. La preparazione dei substrati di ssDNA utilizza la replicazione a cerchio rotante del plasmide ssDNA M13 con primer biotinilati13. L'assemblaggio della cortina di ssDNA richiede anche l'uso di urea e proteina legante l'ssDNA RPA per eliminare la struttura secondaria ed estendere il substrato di ssDNA13. Potrebbe anche essere possibile estendere le tende del DNA all'uso di RNA o ibridi DNA/RNA per studi di proteine che interagiscono con l'RNA. Allo stato attuale, la piattaforma DNA Curtain può beneficiare dell'automazione nel processo di assemblaggio, come descritto nella sezione 5, la maggior parte della quale è costituita da iniezioni ripetitive di un set standard di tamponi. Ciò può portare a una maggiore efficienza nell'esecuzione di più esperimenti contemporaneamente. Inoltre, una suite di script di analisi unificata e potenzialmente basata sull'apprendimento automatico accelererebbe ulteriormente l'elaborazione dei dati con una distorsione minima.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano i membri passati e attuali del laboratorio Greene per aver sviluppato, ottimizzato e discusso il protocollo delle tende del DNA, oltre a commentare questo manoscritto. Questo lavoro è stato sostenuto da sovvenzioni della National Science Foundation (MCB1154511 a E.C.G.) e del National Institutes of Health (R01CA217973 a E.C.G.).
Materiali
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Riferimenti
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon