Method Article
Cortinas de DNA lançam luz sobre sistemas moleculares complexos durante a recombinação homóloga
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
As cortinas de DNA apresentam um novo método para visualizar centenas ou mesmo milhares de proteínas de ligação ao DNA em tempo real, à medida que interagem com moléculas de DNA alinhadas na superfície de uma câmara de amostra microfluídica.
Resumo
A recombinação homóloga (HR) é importante para o reparo de quebras de DNA de fita dupla (DSBs) e garfos de replicação paralisados em todos os organismos. Defeitos na HR estão intimamente associados à perda da integridade do genoma e à transformação oncogênica em células humanas. A FC envolve ações coordenadas de um conjunto complexo de proteínas, muitas das quais permanecem pouco compreendidas. O aspecto chave da pesquisa descrita aqui é uma tecnologia chamada "cortinas de DNA", uma técnica que permite a montagem de moléculas de DNA alinhadas na superfície de uma câmara de amostra microfluídica. Eles podem então ser visualizados por microscopia de fluorescência de reflexão interna total (TIRFM). As cortinas de DNA foram pioneiras em nosso laboratório e permitem o acesso direto a informações espaço-temporais em escalas de tempo de milissegundos e resolução em escala nanométrica, que não podem ser facilmente reveladas por meio de outras metodologias. Uma grande vantagem das cortinas de DNA é que elas simplificam a coleta de dados estatisticamente relevantes de experimentos de molécula única. Esta pesquisa continua a produzir novos insights sobre como as células regulam e preservam a integridade do genoma.
Introdução
A manutenção da integridade do genoma é crucial para o bom funcionamento de todas as células vivas1. Defeitos na integridade do genoma podem levar a condições graves de saúde, incluindo vários tipos de câncer e doenças degenerativas relacionadas à idade2. A recombinação homóloga (HR) usa a síntese de DNA dependente do molde para reparar quebras de fita dupla (DSB) de DNA, lacunas de DNA de fita simples (ssDNA) e ligações cruzadas de DNA entre fitas3. A HR também é necessária para a recuperação de garfos de replicação paralisados e colapsados 3,4. Além disso, a FC é essencial para a segregação cromossômica precisa durante a meiose 5,6.
A FC envolve as ações coordenadas de um conjunto complexo de proteínas, muitas das quais permanecem pouco compreendidas1. Exemplos incluem proteína de replicação A (RPA), Rad51 e Rad54, entre muitos outros7. As reações HR em células procarióticas e eucarióticas envolvem um intermediário de ssDNA, que é rapidamente revestido por proteínas de ligação ao ssDNA (SSB em procariontes e RPA em eucariotos) 8. Essas proteínas protegem o ssDNA das nucleases, eliminam a estrutura secundária e promovem o recrutamento de fatores a jusante 8,9. Rad51 é um membro da família Rad51 / RecA altamente conservada e dependente de ATP de DNA recombinases, que estão presentes em todos os organismos vivos1. Rad51 promove a invasão da fita de DNA do doador homólogo de dsDNA. Dada a sua importância, o Rad51 é altamente regulado, e defeitos nesses processos regulatórios são comumente associados a uma perda de integridade do genoma e transformação oncogênica7. Rad54 é um membro da família Swi2 / Snf2 de translocases de dsDNA e remodeladores de cromatina10,11. Essas proteínas servem como fatores reguladores essenciais do Rad51. É importante ressaltar que o Rad54 remove o Rad51 do produto dsDNA da invasão da fita e também é necessário para evitar o acúmulo incorreto de Rad51 na cromatina11. As atividades moleculares das proteínas envolvidas na HR, tanto em células de levedura quanto em células bacterianas, lançaram luz sobre sua função na HR, mas exatamente como sua atividade contribui para a HR permanece pouco compreendida 12.
As cortinas de DNA surgiram como uma plataforma única que fornece acesso direto a mecanismos moleculares e dinâmicas macromoleculares que, de outra forma, permaneceriam inacessíveis13,14. Para preparar cortinas de DNA, a superfície de uma câmara microfluídica é revestida com uma bicamada lipídica e as moléculas de DNA são amarradas à bicamada por meio de uma ligação biotina-estreptavidina. A bicamada torna a superfície inerte imitando as membranas celulares naturais. A força hidrodinâmica alinha o DNA ao longo de barreiras nanofabricadas, permitindo a visualização de centenas de moléculas em um único campo de visão por microscopia de fluorescência de reflexão interna total (TIRFM). As barreiras são feitas por litografia por feixe de elétrons, e variações no design da barreira permitem um controle preciso sobre a distribuição e a geometria de amarração do DNA. Essas abordagens são prontamente aplicáveis com ssDNA ou dsDNA 13,14,15,16,17,18. Os pedestais também podem ser nanofabricados (junto com as barreiras) para permitir que ambas as extremidades do DNA sejam amarradas à superfície da câmara de amostra, de modo que experimentos em estado estacionário possam ser realizados na ausência de fluxo tampão.
Mudanças dependentes do tempo em complexos proteína-ácido nucléico individuais são reveladas pela inspeção de vídeos em tempo real e são representadas impressas usando quimógrafos, que apresentam a mudança de posição das proteínas no DNA ao longo do tempo. Um aspecto importante da abordagem das cortinas de DNA é que ela não requer necessariamente modelos a priori ou suposições sobre mecanismos moleculares, porque o comportamento de componentes de reação individuais pode ser observado em tempo real. Isso permite a observação direta de comportamentos moleculares. Aqui, este protocolo descreve como preparar cortinas de DNA com substratos de dsDNA, bem como sua aplicação para estudar intermediários em recombinação homóloga.
Protocolo
1. Preparação do estoque de lipídios
- Dissolva 1 g de 18: 1 (Δ9-Cis) PC, 100 mg de 18: 1 PEG2000 PE e 5 mg de 18: 1 biotinyl cap PE em 10 mL de clorofórmio.
NOTA: Tanto o DOPC quanto o DPPE são bem caracterizados e (juntamente com a modificação do PEG) selecionados por sua capacidade de minimizar a adsorção não específica na superfície da célula de fluxo. A mistura principal de lipídios dissolvidos pode ser aliquotada e armazenada a -20 °C por até 12 meses. - Use clorofórmio para limpar uma seringa de vidro com solvente orgânico e, em seguida, transfira 200 μL (10% do volume final desejado) da mistura principal de lipídios para o frasco de vidro.
- Evaporar o clorofórmio do frasco de vidro utilizando um jato muito suave de azoto comprimido. Aumente ligeiramente a pressão no final da evaporação para garantir que todos os vestígios de clorofórmio sejam removidos.
NOTA: Incline e gire o frasco durante o processo de evaporação para que os lipídios sequem como uma camada uniforme de filme branco na parede. - Coloque o frasco para injetáveis sem tampa sob vácuo durante a noite.
- Adicione 2 mL de tampão lipídico (10 mM Tris-HCl [pH 8,0], 100 mM NaCl) ao filme lipídico seco e tampe o frasco. Incubar à temperatura ambiente (RT) durante pelo menos 1 h e vórtice até dissolver.
- Transferir a solução para um tubo de fundo redondo de poliestireno de 5 ml e sonicar sobre gelo utilizando uma microponta com os seguintes parâmetros: amplitude = 50, tempo de processo = 1,5 min, pulso = 15 s, tempo de desligamento = 2 min. A solução ficará límpida após a sonicação (energia total = 1.500-2.000 J).
- Filtrar a solução através de um filtro de seringa de PVDF de 0,22 μm e conservar o depósito de lípidos a 4 °C durante um período máximo de 1 mês.
2. Preparação do substrato dsDNA
- Aquecer o stock de λ-ADN (500 μg/ml) a 65 °C.
- Prepare a mistura de recozimento / ligação contendo: 1,6 pM de λ-DNA, 100x excesso molar (160 pM) de cada oligonucleotídeo (BioL: 5'Phos-AGG TCG CCG CCC-3Bio e DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) e 60 μL de tampão de reação 10x T4 ligase (em um volume total de 600 μL).
NOTA: Os oligonucleotídeos BioL e DigR são biotinilados (Bio) e modificados com digoxigenina (Dig), respectivamente. A biotinilação permite a ligação de moléculas de DNA a lipídios biotinilados por meio de ligações estreptavidina. A modificação Dig permite amarração dupla por meio da ligação de anticorpos anti-Dig que são revestidos em pedestais para porções Dig. - Recozer os oligonucleótidos até às extremidades do λ-ADN, incubando a reacção a 65 °C durante 10 minutos e, em seguida, deixar arrefecer lentamente até à RT.
- Adicione 5 μL de T4 DNA ligase e incube em RT durante a noite.
NOTA: As etapas de recozimento e ligadura também podem ser realizadas em um termociclador. - Precipite o λ-DNA adicionando 300 μL de solução de PEG-8000 (30% p / v PEG-8000 e 10 mM MgCl2). Incubar a 4 °C durante pelo menos 1 h.
- Centrifugue a 14.000 x g por 5 min em RT. Remova cuidadosamente o sobrenadante sem perturbar o pellet. Opcionalmente, lave o pellet 1x com 500 μL de etanol 70% resfriado.
- Ressuspenda o pellet em 40 μL de tampão TE (10 mM Tris-HCl, 1 mM EDTA [pH 8,0]).
3. Nanofabricação de padrões de cromo
- Faça dois furos no centro de uma lâmina de microscópio de quartzo de 1" x 3" (~3 cm de distância) usando uma broca revestida de diamante de 1.4 mm em uma furadeira de mesa (consulte a Figura 1 para a geometria da posição de perfuração).
- Limpe as lâminas perfuradas submergindo-as em 200 mL de solução de Piranha (75% de ácido sulfúrico [97%] e 25% de peróxido de hidrogênio). Incubar por 30 min.
CUIDADO: Deve-se tomar extremo cuidado para evitar todo contato com a pele exposta. Prepare a solução misturando muito lentamente peróxido de hidrogênio frio em ácido sulfúrico. Descarte os resíduos de Piranha adequadamente em um recipiente de lixo designado. - Enxágue as lâminas submergindo em 200 mL de água bidestilada Repita 2x para eliminar qualquer Piranha restante. Seque as lâminas com um jato suave de nitrogênio comprimido.
- Usando um revestidor giratório (4.000 rpm por 45 s, taxa de rampa de 300 rpm/s), cubra as lâminas limpas com fotorresistente e polímero condutor da seguinte forma:
- Revestir lâminas com uma camada de polimetilmetacrilato a 3% (p/v) (PMMA; 24,3K MW; dissolvido em anisol).
- Revestir lâminas com uma camada de polimetilmetacrilato a 1,5% (p/v) (PMMA; 495K MW; dissolvido em anisol).
- Cubra as lâminas com uma camada de agente antiestático para litografia por feixe de elétrons.
- Escreva padrões personalizados para barreiras e pedestais nas lâminas revestidas usando litografia por feixe de elétrons com um microscópio eletrônico de varredura equipado com software de sistema de geração de nanopadrões.
- Desenvolva as lâminas enxaguando primeiro o agente antiestático para a litografia por feixe de elétrons com água bidestilada Em seguida, coloque-os em tubos de 50 mL contendo a solução reveladora (75% de metil isobutilcetona [MIBK] e 25% de isopropanol; mantenha resfriado a -20 ° C) e sonice em banho de gelo em baixa potência por 1 min.
- Enxágue a solução de revelação com isopropanol e seque com um jato suave de nitrogênio comprimido.
- Usando um evaporador de feixe de elétrons, deposite uma camada de cromo de 250 Å de espessura na superfície padronizada a 0.5 Å / s.
- Retire o cromo e o PMMA restante usando acetona de um frasco de esguicho, seguido de sonicação das lâminas em um banho de acetona por 10 min. Lave novamente com acetona de um frasco de esguicho.
- Para evitar depósitos da acetona restante, enxágue as lâminas com álcool isopropílico e seque com um jato suave de nitrogênio comprimido.
4. Montagem da célula de fluxo
- Coloque um modelo de papel retangular de 35 mm x 5 mm (cobrindo o padrão cromado, bem como os orifícios de entrada/saída pré-perfurados) sobre a lâmina de quartzo, com o lado estampado voltado para cima ( Figura 1A).
- Aplique um pedaço de fita dupla-face (1 mm de espessura) sobre a lâmina de quartzo, cobrindo o modelo de papel e, em seguida, use uma lâmina de barbear para cortar o modelo de papel retangular ( Figura 1A).
- Coloque uma lamínula de vidro sobre a fita dupla-face e aplique uma leve pressão para selar a lamínula na fita ( Figura 1A). O espaço entre a lamínula e a lâmina, selado por fita dupla-face, forma a câmara microfluídica.
NOTA: É crucial que a vedação entre a lamínula e a corrediça seja suficientemente segura para evitar vazamentos durante o experimento. - Coloque o sanduíche de lamínula entre as lâminas de vidro e prenda os clipes de fichário nos quatro lados para distribuir a pressão uniformemente. Coloque o conjunto em um forno a vácuo por 60 min a 135 °C para derreter a fita dupla face e selar a câmara.
- Retire-o do forno e solte os clipes de fichário e as lâminas de vidro.
- Conecte as portas microfluídicas aos orifícios de entrada/saída pré-perfurados com uma pistola de cola quente, colocando o conjunto colado em um bloco de aquecimento (220 °C) por 1 min, retirando-o e aplicando cuidadosamente uma leve pressão para vedar adequadamente as portas microfluídicas. Deixe a cola curar completamente antes de prosseguir.
NOTA: As células de fluxo montadas podem ser colocadas em tubos de 50 mL e armazenadas sob vácuo.
5. Montagem da cortina dsDNA
- Conecte a tubulação de entrada/saída às portas microfluídicas da célula de fluxo e lave a câmara com 20 mL de água bidestilada usando duas seringas de 10 mL.
- Com uma seringa de 10 mL cheia de água bidestilada conectada a ambos os lados da célula de fluxo, aplique força para empurrar/puxar a água entre as seringas de entrada e saída. Esta etapa remove todas as bolhas de ar da célula de fluxo.
- Lave a câmara com 3 mL de tampão lipídico (10 mM Tris-HCl [pH 8,0], 100 mM NaCl).
NOTA: Certifique-se de que todas as conexões com as células de fluxo sejam gota a gota para evitar a introdução de bolhas de ar na célula de fluxo. - Misture 40 μL de solução de lipossomas com 1 mL de tampão lipídico. Injete 300 μL da solução lipídica na célula de fluxo e incube por 5 min. Repita a injeção e incubação 2x.
NOTA: Alterne o lado de entrada/saída para injeção sempre que uma seringa for conectada à célula de fluxo. - Lave a câmara com 3 mL de tampão lipídico e incube por 15 min.
NOTA: Este período de incubação pode variar de 5 min a 2 h, dependendo do agendamento dos experimentos, sem diferenças perceptíveis na montagem final da cortina. - Misture 40 μL de solução estoque anti-digoxigenina 1 mg / mL com 200 μL de tampão lipídico. Injetar 100 μL da solução e incubar durante 10 min. Repita a injeção e a incubação 1x antes de lavar com 3 mL de tampão lipídico.
NOTA: Esta etapa é necessária apenas para cortinas dsDNA com amarração dupla. Pule esta etapa para cortinas dsDNA com amarração única. - Lave a câmara com 3 mL de tampão BSA (40 mM Tris-HCl [pH 8.0], 2 mM MgCl2, 1 mM DTT e 0.2 mg/mL BSA).
- Misture 10 μL de solução estoque de estreptavidina 1 mg / mL com 990 μL de tampão BSA. Injete em duas injeções separadas de 400 μL com uma incubação de 10 minutos após cada injeção.
- Lave a câmara com 3 mL de tampão BSA para remover qualquer excesso de estreptavidina.
- Diluir 25 ng de substrato dsDNA em 1 mL de tampão BSA. Injete lentamente a solução de DNA 200 μL de cada vez, seguida de uma incubação de 5 minutos após cada injeção. A quantidade de DNA necessária pode ser otimizada para tipos específicos de experimentos.
- Monte a célula de fluxo na platina do microscópio e ajuste o foco conforme necessário.
- Conecte as portas de entrada e saída a um sample sistema de injeção composto por uma bomba de seringa e válvula de interruptor de alta pressão.
- Amarre duas vezes o DNA aos pedestais (comprimento de amarração de 12 μm) lavando continuamente a câmara com tampão BSA a 0,8 mL / min por 5 min.
NOTA: Pode ser necessário ajustar as taxas e os tempos de fluxo para otimizar diferentes comprimentos de cabo duplo. Esta etapa é necessária apenas para cortinas de DNA com amarração dupla. Pule esta etapa para cortinas dsDNA com amarração única. - As cortinas estão agora prontas para experimentos.
6. Reciclagem de células de fluxo
- Mergulhe as lâminas em etanol por pelo menos 30 minutos antes de usar uma lâmina de barbear para remover cuidadosamente as portas microfluídicas e a lamínula. Descarte adequadamente a lamínula e limpe qualquer cola restante das portas microfluídicas para que possam ser reutilizadas. Esfregue qualquer resíduo de cola na lâmina.
NOTA: Tome cuidado para não arranhar a área padronizada no centro do slide. - Enxágue bem as lâminas com água bidestilada e mergulhe em concentrado de limpeza de quartzo líquido alcalino a 2% durante a noite, com agitação.
- Enxágue bem as lâminas com água bidestilada e mergulhe em solução concentrada de limpeza de quartzo líquido alcalino a 2% por 1 h com agitação a 80 °C, seguida de sonicação por 15 min a 45 °C.
- Enxágue bem as lâminas com água bidestilada e mergulhe em NaOH 1 M por 30-60 min, com agitação.
- Enxágue bem as lâminas com água bidestilada e, em seguida, mergulhe em álcool isopropílico e sonice por 15 min a 45 °C.
- Enxaguar bem as lâminas com etanol e mergulhar em etanol durante 1 h, agitando.
- Coloque as lâminas em um forno a vácuo por 1 h para secar a 135 °C. As lâminas agora estão prontas para serem remontadas em células de fluxo.
Resultados
Descritos acima estão a preparação, montagem e imagem de cortinas de dsDNA com amarração simples e dupla no contexto do estudo das interações proteína-DNA em intermediários de reparo de DNA. A Figura 1A mostra todos os componentes em uma célula de fluxo, em camadas na ordem em que são montados. A Figura 1B mostra o esquema de uma cortina de DNA com amarração simples ou dupla. Uma bicamada lipídica é usada para passivar a superfície da célula de fluxo. A cortina de DNA consiste em uma matriz paralela de moléculas de DNA amarradas em uma extremidade ao lipídio e alinhadas na barreira de cromo, orientadas perpendicularmente à direção do fluxo. Para cortinas de DNA duplamente amarradas, a outra extremidade do DNA é amarrada ao pedestal por meio de interações de digoxigenina e anti-digoxigenina, de modo que a imagem possa ser realizada na ausência de fluxo enquanto o DNA permanece estendido. O dsDNA pode ser corado com corante fluorescente YoYo1 e visualizado por microscopia TIRF. A Figura 2A mostra uma imagem representativa de campo amplo da cortina de DNA com corda dupla corada YoYo1.
As séries de imagens de lapso de tempo coletadas de experimentos de cortina de DNA são normalmente analisadas pela geração de um quimógrafo, que traça a posição ao longo da molécula de DNA no eixo vertical versus o tempo no eixo horizontal para cada molécula de DNA de interesse no ImageJ. A Figura 2B são quimógrafos representativos que mostram a ressecção de λ-DNA de corado único corado com YoYo1 (verde) pelas máquinas de ressecção de levedura GFP-Sgs1 (não visível), Top3-Rmi1 e Dna2 na presença de proteína de ligação ao DNA de fita simples RPA-mCherry (magenta) e ATP. O sinal YoYo1 é perdido ao longo do tempo à medida que o dsDNA é ressecado da extremidade livre por Sgs1-Dna2. Simultaneamente, o sinal mCherry colocaliza com as extremidades do DNA nas quais o ssDNA está sendo gerado como resultado da ressecção. Características biofisicamente relevantes da ressecção, como velocidade e processividade, podem ser extraídas quantificando as inclinações das trajetórias de perda de sinal YoYo1. A Figura 2C,D mostra a distribuição das velocidades e processividade, respectivamente, da ressecção por Sgs1-Dna2.

Figura 1: Esquemas de montagem de células de fluxo e cortinas de DNA. (A) Ilustração passo a passo para montagem de células de fluxo. A fita dupla-face é colocada em cima da lâmina de quartzo e um modelo de papel é usado para cortar um canal retangular do centro, que é selado com uma lamínula na parte superior para formar uma câmara. (B) Esquemas de cortinas de DNA totalmente montadas. O painel do meio apresenta uma célula de fluxo de amarração única, enquanto o painel inferior apresenta uma célula de fluxo de amarração dupla. Clique aqui para ver uma versão maior desta figura.
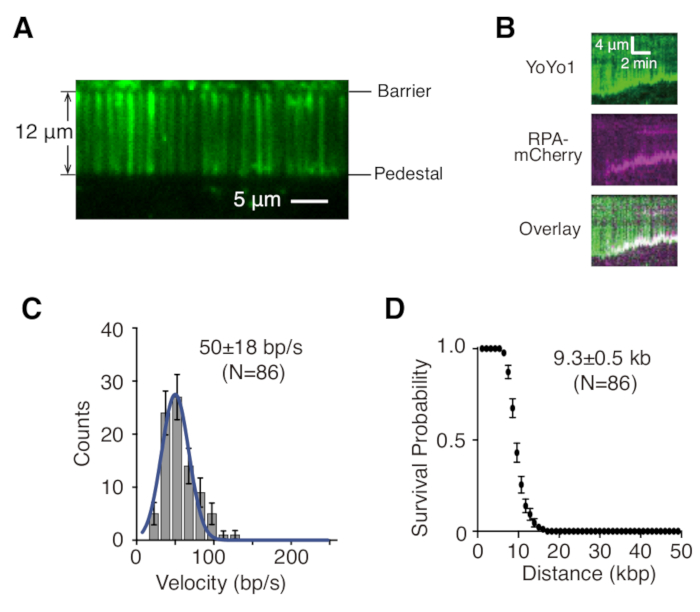
Figura 2: Visualização e análise de cortina de DNA de molécula única. (A) Imagem representativa de campo amplo de uma cortina de λ-DNA com fio duplo corado com YoYo1 (verde). (B) Quimógrafos representativos mostrando a ressecção de dsDNA corado com YoYo1 (verde) na presença de GFP-Sgs1 pré-ligado (não visível) quando perseguido com Dna2, na presença de RPA-mCherry (magenta) e 2 mM ATP. Distribuição de velocidade (C) e gráfico de processividade (D) da ressecção do dsDNA por Sgs1-Dna2, obtidos pela quantificação da taxa e extensão da perda de sinal YoYo1 ao longo do tempo. Os painéis (B), (C) e (D) são adaptados de Xue et al.19. Clique aqui para ver uma versão maior desta figura.
Discussão
As cortinas de DNA provaram ser uma plataforma versátil para estudar vários processos de reparo de DNA 13,14,15,16. As moléculas de DNA que permanecem estendidas perto da superfície durante todo o experimento, por meio de fluxo tampão contínuo ou amarração dupla ao pedestal, permitem imagens TIRFM de interações proteína-DNA em intermediários de reparo no nível de molécula única. O método proporciona melhoria no rendimento experimental e na previsibilidade, uma vez que centenas de moléculas de DNA são alinhadas ordenadamente em barreiras nanofabricadas, em vez de não especificamente ligadas à superfície, o que beneficia ainda mais o processamento de grandes conjuntos de dados.
A análise cuidadosa da série de imagens de lapso de tempo resultante fornece medições quantitativas das interações proteína-DNA (ou seja, tempos de vida de ligação, posições, estequiometria, cinética de dissociação, taxas de translocação, processividades de proteínas motoras e colocalizações e interações entre proteínas e / ou DNA). Em comparação com outras técnicas de imagem de molécula única que ligam moléculas de DNA diretamente à superfície ou combinam imagens confocais com manipulação de força em instrumentos comerciais, a principal compensação das cortinas de DNA por maior rendimento é estabelecer as técnicas necessárias para a configuração inicial 13,14,15,16. Uma vez estabelecida, no entanto, a capacidade de coletar dados sobre centenas de moléculas simultaneamente em uma célula de fluxo leva a uma economia significativa no tempo gasto na coleta de dados.
Tal como acontece com muitos outros métodos de imagem de molécula única, a capacidade de preparar uma superfície limpa determina em grande parte a capacidade do usuário de montar cortinas de DNA de forma confiável. Nesse caso, superfícies limpas exigem não apenas começar com uma lâmina limpa, mas também certificar-se de não introduzir bolhas de ar microscópicas na câmara ao fazer qualquer uma das conexões fluídicas. Em primeiro lugar, no que diz respeito à limpeza das lâminas, as etapas detalhadas acima na seção 6 devem ser suficientes como um processo de limpeza de rotina. O uso de soluções filtradas e tampões também é recomendado. Em segundo lugar, fazer conexões gota a gota durante a fixação da célula de fluxo ao sistema de injeção de tampão é essencial para evitar a introdução de bolhas. Pode ser útil desgaseificar a água bidestilada usada para fazer tampões. Também é recomendável observar cuidadosamente qualquer processo de injeção para garantir que não haja bolhas. Se uma bolha microscópica for introduzida antes de atingir a área padronizada central, ela pode ser remediada injetando tampão da porta microfluídica no lado oposto para empurrar a bolha de volta para fora.
Outro ponto importante a ser considerado em qualquer experimento baseado em fluorescência é a relação sinal-ruído. Uma chave para observar proteínas fluorescentes únicas (ou seja, GFP) também reside em superfícies limpas. Após o uso prolongado, os agregados de proteínas normalmente se acumulam nas barreiras e pedestais de cromo. Portanto, se um forte sinal de fluorescência for observado nas barreiras antes da adição de proteínas fluorescentes, é aconselhável tratar as lâminas com uma submersão de 10 min em solução de Piranha. O período de tempo é mantido curto para evitar a erosão dos recursos cromados. A lavagem da Piranha deve ser seguida pelo procedimento de limpeza regular.
A nanofabricação usando litografia por feixe de elétrons pode ser desafiadora, devido aos seus requisitos de equipamentos e instalações, bem como à experiência existente em ciência de materiais. Como alternativa, foi desenvolvido um método baseado em litografia UV para fabricação de características de cromo para cortinas de DNA20. Também é fornecido com este protocolo um padrão básico para DNA lambda de amarração dupla a 12 μm (consulte informações suplementares). Vários aspectos do design deste padrão podem ser ajustados para atender a várias necessidades experimentais. Isso inclui a distância entre barreiras e pedestais, espaçamento entre poços adjacentes na barreira para controlar a densidade do DNA e a forma e o tamanho dos pedestais para otimizar a eficiência da amarração dupla e minimizar a incerteza nos comprimentos de amarração dupla. Outros desafios práticos podem incluir a escolha de estratégias de marcação fluorescente para proteínas, minimizando o viés na análise manual de dados e o desenvolvimento de scripts para um processamento de dados mais simplificado.
Embora o protocolo descrito aqui envolva substratos de dsDNA, cortinas de DNA baseadas em ssDNA também têm sido amplamente utilizadas 13,14,15,16. A preparação de substratos de ssDNA utiliza replicação de círculo rolante do plasmídeo M13 ssDNA com primers biotinilados13. A montagem da cortina de ssDNA também requer o uso de ureia e proteína de ligação ao ssDNA RPA para eliminar a estrutura secundária e estender o substrato do ssDNA13. Também pode ser possível estender as cortinas de DNA para o uso de RNA ou híbridos de DNA / RNA para estudos de proteínas que interagem com RNA. Atualmente, a plataforma de cortina DNA pode se beneficiar da automação no processo de montagem, conforme detalhado na seção 5, a maioria das quais são injeções repetitivas de um conjunto padrão de buffers. Isso pode levar a uma maior eficiência na realização de vários experimentos simultaneamente. Além disso, um conjunto unificado e potencialmente baseado em aprendizado de máquina de
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Os autores agradecem aos membros anteriores e atuais do laboratório Greene por desenvolver, otimizar e discutir o protocolo de cortinas de DNA, bem como comentar este manuscrito. Este trabalho foi apoiado por doações da National Science Foundation (MCB1154511 para ECG) e dos Institutos Nacionais de Saúde (R01CA217973 para ECG).
Materiais
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Referências
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados