Method Article
Les rideaux d’ADN mettent en lumière des systèmes moléculaires complexes lors de la recombinaison homologue
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les rideaux d’ADN offrent une nouvelle méthode pour visualiser des centaines, voire des milliers de protéines de liaison à l’ADN en temps réel lorsqu’elles interagissent avec des molécules d’ADN alignées à la surface d’une chambre d’échantillonnage microfluidique.
Résumé
La recombinaison homologue (HR) est importante pour la réparation des cassures d’ADN double brin (CDB) et des fourches de réplication bloquées dans tous les organismes. Les défauts de HR sont étroitement associés à une perte d’intégrité du génome et à une transformation oncogénique dans les cellules humaines. La HR implique des actions coordonnées d’un ensemble complexe de protéines, dont beaucoup restent mal comprises. L’aspect clé de la recherche décrite ici est une technologie appelée « rideaux d’ADN », une technique qui permet d’assembler des molécules d’ADN alignées à la surface d’une chambre d’échantillonnage microfluidique. Ils peuvent ensuite être visualisés par microscopie à fluorescence à réflexion interne totale (TIRFM). Les rideaux d’ADN ont été mis au point par notre laboratoire et permettent un accès direct à des informations spatio-temporelles à des échelles de temps de l’ordre de la milliseconde et à une résolution nanométrique, qui ne peuvent pas être facilement révélées par d’autres méthodologies. L’un des principaux avantages des rideaux d’ADN est qu’ils simplifient la collecte de données statistiquement pertinentes à partir d’expériences sur une seule molécule. Cette recherche continue de fournir de nouvelles connaissances sur la façon dont les cellules régulent et préservent l’intégrité du génome.
Introduction
Le maintien de l’intégrité du génome est crucial pour le bon fonctionnement de toutes les cellules vivantes1. Les défauts d’intégrité du génome peuvent entraîner de graves problèmes de santé, notamment divers types de cancer et des maladies dégénératives liées à l’âge2. La recombinaison homologue (HR) utilise la synthèse de l’ADN dépendante de la matrice pour réparer les cassures double brin de l’ADN (DSB), les lacunes de l’ADN simple brin (ssDNA) et les réticulations de l’ADN interbrins3. La FC est également nécessaire pour la récupération des fourches de réplication bloquées et repliées 3,4. De plus, HR est essentiel pour la ségrégation précise des chromosomes pendant la méiose 5,6.
La FC implique les actions coordonnées d’un ensemble complexe de protéines, dont beaucoup restent mal comprises1. Les exemples incluent la protéine de réplication A (RPA), Rad51 et Rad54, parmi beaucoup d’autres7. Les réactions HR dans les cellules procaryotes et eucaryotes impliquent un intermédiaire de l’ADNsb, qui est rapidement recouvert par des protéines de liaison à l’ADNsb (SSB chez les procaryotes et RPA chez les eucaryotes)8. Ces protéines protègent l’ADNsb des nucléases, éliminent la structure secondaire et favorisent le recrutement des facteurs en aval 8,9. Rad51 fait partie de la famille des recombinases d’ADN Rad51/RecA, hautement dépendante de l’ATP, qui est présente dans tous les organismes vivants1. Rad51 favorise l’invasion des brins d’ADN du donneur homologue d’ADNdb. Compte tenu de son importance, Rad51 est hautement régulé, et les défauts de ces processus de régulation sont souvent associés à une perte d’intégrité du génome et à une transformation oncogénique7. Rad54 est un membre de la famille Swi2/Snf2 de translocases d’ADNdb et de remodeleurs de chromatine10,11. Ces protéines servent de facteurs régulateurs essentiels de Rad51. Il est important de noter que Rad54 élimine Rad51 du produit de l’ADNdb de l’invasion des brins et est également nécessaire pour prévenir la méaccumulation de Rad51 sur la chromatine11. Les activités moléculaires des protéines impliquées dans la RH à la fois dans les cellules de levure et les cellules bactériennes ont mis en lumière leur fonction dans la HR, mais la façon exacte dont leur activité contribue à la HR reste mal comprise 12.
Les rideaux d’ADN sont apparus comme une plate-forme unique offrant un accès direct aux mécanismes moléculaires et à la dynamique macromoléculaire qui resteraient autrement inaccessibles13,14. Pour préparer des rideaux d’ADN, la surface d’une chambre microfluidique est recouverte d’une bicouche lipidique, et les molécules d’ADN sont attachées à la bicouche par une liaison biotine-streptavidine. La bicouche rend la surface inerte en imitant les membranes cellulaires naturelles. La force hydrodynamique aligne l’ADN le long de barrières nanofabriquées, permettant de visualiser des centaines de molécules dans un seul champ de vision par microscopie à fluorescence à réflexion interne totale (TIRFM). Les barrières sont fabriquées par lithographie par faisceau d’électrons, et les variations dans la conception des barrières permettent un contrôle précis de la distribution et de la géométrie d’attache de l’ADN. Ces approches sont facilement applicables avec l’ADNss ou l’ADNdb 13,14,15,16,17,18. Les socles peuvent également être nanofabriqués (avec les barrières) pour permettre aux deux extrémités de l’ADN d’être attachées à la surface de la chambre d’échantillon, de sorte que des expériences à l’état stationnaire puissent être réalisées en l’absence d’écoulement tampon.
Les changements dépendants du temps dans les complexes protéiques-acides nucléiques individuels sont révélés par l’inspection de vidéos en temps réel et sont représentés par écrit à l’aide de kymographes, qui présentent la position changeante des protéines sur l’ADN au fil du temps. Un aspect important de l’approche des rideaux d’ADN est qu’elle ne nécessite pas nécessairement de modèles a priori ou d’hypothèses sur les mécanismes moléculaires, car le comportement des composants individuels de la réaction peut être observé en temps réel. Cela permet l’observation directe des comportements moléculaires. Ce protocole décrit ici comment préparer des rideaux d’ADN avec des substrats d’ADNdb ainsi que son application à l’étude des intermédiaires dans la recombinaison homologue.
Protocole
1. Préparation du stock lipidique
- Dissoudre 1 g de PC 18:1 (Δ9-Cis), 100 mg de PE PEG2000 18:1 et 5 mg de capsule de biotinyle PE 18:1 dans 10 mL de chloroforme.
REMARQUE : Le DOPC et le DPPE sont tous deux bien caractérisés et (avec la modification du PEG) sélectionnés pour leur capacité à minimiser l’adsorption non spécifique à la surface de la cellule d’écoulement. Le mélange maître lipidique dissous peut être aliquote et stocké à -20 °C jusqu’à 12 mois. - Utilisez du chloroforme pour nettoyer une seringue en verre à solvant organique, puis transférez 200 μL (10 % du volume final souhaité) du mélange maître lipidique dans le flacon en verre.
- Évaporez le chloroforme du flacon en verre à l’aide d’un jet très doux d’azote comprimé. Augmentez légèrement la pression vers la fin de l’évaporation pour vous assurer que toutes les traces de chloroforme sont éliminées.
REMARQUE : Inclinez et tournez le flacon pendant le processus d’évaporation afin que les lipides sèchent sous la forme d’une couche uniforme de film blanc sur la paroi. - Placez le flacon débouché sous vide pendant la nuit.
- Ajouter 2 mL de tampon lipidique (10 mM de Tris-HCl [pH 8,0], 100 mM de NaCl) dans le film lipidique séché et boucher le flacon. Incuber à température ambiante (RT) pendant au moins 1 h et agiter jusqu’à dissolution du mouton.
- Transvaser la solution dans un tube de polystyrène à fond rond de 5 mL et sonicer sur de la glace à l’aide d’une micro-pointe avec les paramètres suivants : amplitude = 50, temps de traitement = 1,5 min, impulsion = 15 s, temps d’arrêt = 2 min. La solution sera claire après sonication (énergie totale = 1 500-2 000 J).
- Filtrez la solution à l’aide d’un filtre à seringue en PVDF de 0,22 μm et conservez le stock lipidique à 4 °C jusqu’à 1 mois maximum.
2. Préparation du substrat d’ADNdb
- Réchauffer le stock d’ADN-λ (500 μg/mL) à 65 °C.
- Préparez un mélange de recuit/ligature contenant : 1,6 pM d’ADN-λ, 100 fois plus d’excès molaire (160 pM) de chaque oligonucléotide (BioL : 5'Phos-AGG TCG CCG CCC-3Bio et DigR : 5'Phos-GGG CGG CGA CCT-3Dig_N) et 60 μL de tampon de réaction 10x T4 ligase (dans un volume total de 600 μL).
REMARQUE : Les oligonucléotides BioL et DigR sont biotinylés (Bio) et modifiés avec de la digoxigénine (Dig), respectivement. La biotinylation permet la fixation de molécules d’ADN à des lipides biotinylés via des liaisons streptavidines. La modification Dig permet une double attache par liaison d’anticorps anti-Dig qui sont recouverts sur des socles à des fractions Dig. - Recuit les oligonucléotides aux extrémités de l’ADN-λ en incubant la réaction à 65 °C pendant 10 min, puis laissez-la refroidir lentement jusqu’à RT.
- Ajouter 5 μL d’ADN ligase T4 et incuber à RT pendant la nuit.
REMARQUE : Les étapes de recuit et de ligature peuvent également être effectuées dans un thermocycleur. - Précipitez l’ADN-λ en ajoutant 300 μL de solution de PEG-8000 (30 % p/v de PEG-8000 et 10 mM de MgCl2). Incuber à 4 °C pendant au moins 1 h.
- Centrifuger à 14 000 x g pendant 5 min à RT. Retirer délicatement le surnageant sans déranger la pastille. En option, lavez la pastille 1x avec 500 μL d’éthanol réfrigéré à 70 %.
- Remettre la pastille en suspension dans 40 μL de tampon TE (10 mM de Tris-HCl, 1 mM d’EDTA [pH 8,0]).
3. Nanofabrication de motifs de chrome
- Percez deux trous au centre d’une lame de microscope à quartz de 1 po x 3 po (à ~3 cm de distance) à l’aide d’un foret de 1,4 mm revêtu de diamant sur une perceuse à colonne de table (voir la figure 1 pour la géométrie de la position de perçage).
- Nettoyez les lames percées en les immergeant dans 200 mL de solution de Piranha (75 % d’acide sulfurique [97 %] et 25 % de peroxyde d’hydrogène). Incuber pendant 30 min.
ATTENTION : Des précautions extrêmes doivent être prises pour éviter tout contact avec la peau exposée. Préparez la solution en mélangeant très lentement du peroxyde d’hydrogène froid dans de l’acide sulfurique. Éliminez correctement les déchets de piranha dans un conteneur à déchets désigné. - Rincez les lames en les immergeant dans 200 mL d’eau doublement distillée. Répétez l’opération 2 fois pour éliminer les piranhas restants. Séchez les lames avec un léger jet d’azote comprimé.
- À l’aide d’une machine à revêtir (4 000 tr/min pendant 45 s, vitesse de rampe de 300 tr/min/s), enduire les lames propres d’une résine photosensible et d’un polymère conducteur comme suit :
- Enduire les lames d’une couche de polyméthacrylate de méthyle à 3 % (p/v) (PMMA ; 24,3 K MW ; dissous dans l’anisole).
- Enduire les lames d’une couche de polyméthacrylate de méthyle à 1,5 % (p/v) (PMMA ; 495K MW ; dissous dans l’anisole).
- Enduire les lames d’une couche d’agent antistatique pour la lithographie par faisceau d’électrons.
- Écrivez des motifs personnalisés pour les barrières et les socles sur les lames revêtues à l’aide de la lithographie par faisceau d’électrons avec un microscope électronique à balayage équipé d’un logiciel de système de génération de nanomotifs.
- Développer les lames en rinçant d’abord l’agent antistatique pour la lithographie par faisceau d’électrons avec de l’eau bidistillée. Ensuite, placez-les dans des tubes de 50 mL contenant la solution de développement (75 % de méthylisobutylcétone [MIBK] et 25 % d’isopropanol ; à conserver au frais à -20 °C) et faites du sonicate dans un bain de glace à faible puissance pendant 1 min.
- Rincez la solution de développement avec de l’isopropanol et séchez-la avec un léger jet d’azote comprimé.
- À l’aide d’un évaporateur à faisceau d’électrons, déposez une couche de chrome de 250 Å d’épaisseur sur la surface à motifs à 0,5 Å/s.
- Soulevez le chrome et le PMMA restant à l’aide de l’acétone d’un flacon pulvérisateur, puis sioninez les lames dans un bain d’acétone pendant 10 min. Lavez à nouveau avec de l’acétone à l’aide d’une bouteille à jeter.
- Pour éviter tout dépôt d’acétone restante, rincez les lames avec de l’alcool isopropylique et séchez-les avec un léger filet d’azote comprimé.
4. Assemblage de la cellule d’écoulement
- Placez un gabarit en papier rectangulaire de 35 mm x 5 mm (couvrant le motif chromé ainsi que les trous d’entrée/sortie pré-percés) sur la glissière en quartz, avec le côté à motifs vers le haut ( Figure 1A).
- Appliquez un morceau de ruban adhésif double face (1 mm d’épaisseur) sur la lame de quartz, en recouvrant le modèle en papier, puis utilisez une lame de rasoir pour découper le modèle en papier rectangulaire ( Figure 1A).
- Placez une lamelle en verre sur le ruban adhésif double face et appliquez une légère pression pour sceller la lamelle sur le ruban ( Figure 1A). L’espace entre la lamelle et la glissière, scellé par du ruban adhésif double face, forme la chambre microfluidique.
REMARQUE : Il est crucial que l’étanchéité entre la lamelle et la glissière soit suffisamment sûre pour éviter les fuites pendant l’expérience. - Placez le sandwich de lamelle entre les lames de verre et fixez les pinces à reliure sur les quatre côtés pour répartir uniformément la pression. Placez l’ensemble dans un four sous vide pendant 60 min à 135 °C pour faire fondre le ruban adhésif double face et sceller la chambre.
- Retirez-le du four, puis relâchez les pinces à reliure et les lames de verre.
- Fixez les orifices microfluidiques aux trous d’entrée/sortie pré-percés à l’aide d’un pistolet à colle chaude en plaçant l’ensemble collé sur un bloc chauffant (220 °C) pendant 1 min, en le retirant et en appliquant soigneusement une légère pression pour bien sceller les orifices microfluidiques. Laissez la colle durcir complètement avant de continuer.
REMARQUE : Les cellules d’écoulement assemblées peuvent être placées dans des tubes de 50 ml et stockées sous vide.
5. Assemblage du rideau dsDNA
- Fixez le tube d’entrée/sortie aux orifices microfluidiques de la cellule d’écoulement et lavez la chambre avec 20 ml d’eau bidistillée à l’aide de deux seringues de 10 ml.
- Avec une seringue de 10 ml remplie d’eau bidistillée reliée des deux côtés de la cellule d’écoulement, appliquez une force pour pousser/tirer l’eau entre les seringues d’entrée et de sortie. Cette étape élimine toutes les bulles d’air de la cellule d’écoulement.
- Laver la chambre avec 3 mL de tampon lipidique (10 mM de Tris-HCl [pH 8,0], 100 mM de NaCl).
REMARQUE : Assurez-vous que toutes les connexions aux cellules d’écoulement sont goutte à goutte pour éviter d’introduire des bulles d’air dans la cellule d’écoulement. - Mélanger 40 μL de solution de liposomes avec 1 mL de tampon lipidique. Injecter 300 μL de la solution lipidique dans la cellule d’écoulement et incuber pendant 5 min. Répétez l’injection et l’incubation 2 fois.
REMARQUE : Alternez le côté entrée/sortie pour l’injection chaque fois qu’une seringue est connectée à la cellule d’écoulement. - Laver la chambre avec 3 mL de tampon lipidique et incuber pendant 15 min.
REMARQUE : Cette période d’incubation peut varier de 5 min à 2 h, selon le calendrier des expériences, sans différences notables dans l’assemblage final du rideau. - Mélanger 40 μL de solution mère d’anti-digoxigénine à 1 mg/mL avec 200 μL de tampon lipidique. Injecter 100 μL de la solution et incuber pendant 10 min. Répétez l’injection et l’incubation 1 fois avant de laver avec 3 mL de tampon lipidique.
REMARQUE : Cette étape n’est nécessaire que pour les rideaux dsDNA à double attache. Ignorez cette étape pour les rideaux dsDNA mono-attachés. - Lavez la chambre avec 3 mL de tampon BSA (40 mM de Tris-HCl [pH 8,0], 2 mM de MgCl2, 1 mM de DTT et 0,2 mg/mL de BSA).
- Mélanger 10 μL de solution mère de streptavidine à 1 mg/mL avec 990 μL de tampon BSA. Injecter en deux injections distinctes de 400 μL avec une incubation de 10 minutes après chaque injection.
- Lavez la chambre avec 3 ml de tampon BSA pour éliminer tout excès de streptavidine.
- Diluer 25 ng de substrat d’ADNdb dans 1 mL de tampon BSA. Injectez lentement la solution d’ADN 200 μL à la fois, suivie d’une incubation de 5 minutes après chaque injection. La quantité d’ADN nécessaire peut être optimisée pour des types spécifiques d’expériences.
- Montez la cellule d’écoulement sur la platine du microscope et ajustez la mise au point si nécessaire.
- Connectez les orifices d’entrée et de sortie à un système d’injection d’échantillon composé d’une pompe à seringue et d’une vanne de commutation haute pression.
- Attachez deux fois l’ADN aux socles (longueur d’attache de 12 μm) en lavant continuellement la chambre avec un tampon BSA à 0,8 mL/min pendant 5 min.
REMARQUE : Il peut être nécessaire d’ajuster les débits et les temps pour optimiser les différentes longueurs de double attache. Cette étape n’est nécessaire que pour les rideaux d’ADN à double attache. Ignorez cette étape pour les rideaux dsDNA mono-attachés. - Les rideaux sont maintenant prêts pour les expériences.
6. Recyclage des cellules d’écoulement
- Immergez les lames dans de l’éthanol pendant au moins 30 minutes avant d’utiliser une lame de rasoir pour retirer soigneusement les orifices microfluidiques et la lamelle. Éliminez correctement la lamelle et nettoyez toute colle restante des orifices microfluidiques afin qu’ils puissent être réutilisés. Nettoyez tout résidu de colle sur la lame.
REMARQUE : Veillez à ne pas rayer la zone à motifs au centre de la diapositive. - Rincez abondamment les lames avec de l’eau bi-distillée et immergez-les dans un concentré de nettoyage en quartz liquide alcalin à 2 % pendant la nuit, en remuant.
- Rincez abondamment les lames avec de l’eau doublement distillée et immergez-les dans une solution de nettoyage concentrée de quartz liquide alcalin à 2 % pendant 1 h avec agitation à 80 °C, suivie d’une sonification pendant 15 min à 45 °C.
- Rincez abondamment les lames avec de l’eau bi-distillée et immergez-les dans 1 M de NaOH pendant 30 à 60 min, en remuant.
- Rincez abondamment les lames avec de l’eau bi-distillée, puis immergez-les dans de l’alcool isopropylique et du sonicate pendant 15 min à 45 °C.
- Rincez abondamment les lames avec de l’éthanol et immergez-les dans de l’éthanol pendant 1 h, en remuant.
- Placez les lames dans un four sous vide pendant 1 h pour qu’elles sèchent à 135 °C. Les lames sont maintenant prêtes à être réassemblées en cellules d’écoulement.
Résultats
La préparation, l’assemblage et l’imagerie de rideaux d’ADNdb à simple et double attache sont décrits ci-dessus dans le contexte de l’étude des interactions protéine-ADN dans les intermédiaires de réparation de l’ADN. La figure 1A montre tous les composants d’une cellule d’écoulement, superposés dans l’ordre dans lequel ils sont assemblés. La figure 1B représente le schéma d’un rideau d’ADN à simple ou double attache. Une bicouche lipidique est utilisée pour passiver la surface de la cellule d’écoulement. Le rideau d’ADN se compose d’un réseau parallèle de molécules d’ADN attachées à une extrémité au lipide et alignées au niveau de la barrière de chrome, orientées perpendiculairement à la direction de l’écoulement. Pour les rideaux d’ADN à double attache, l’autre extrémité de l’ADN est attachée au piédestal par des interactions de digoxigénine et d’anti-digoxigénine, de sorte que l’imagerie peut être effectuée en l’absence de flux tandis que l’ADN reste étendu. L’ADNdb peut être coloré avec le colorant fluorescent YoYo1 et visualisé par microscopie TIRF. La figure 2A montre une image représentative à grand champ d’un rideau d’ADN double attaché coloré par YoYo1.
Les séries d’images en accéléré recueillies à partir d’expériences sur le rideau d’ADN sont généralement analysées en générant d’abord un kymographe, qui trace la position le long de la molécule d’ADN sur l’axe vertical en fonction du temps sur l’axe horizontal pour chaque molécule d’ADN d’intérêt dans ImageJ. La figure 2B est un kymographe représentatif montrant la résection de l’ADN-λ mono-attaché coloré par YoYo1 (vert) par les machines de résection de levure GFP-Sgs1 (non visible), Top3-Rmi1 et Dna2 en présence de la protéine de liaison à l’ADN simple brin RPA-mCherry (magenta) et de l’ATP. Le signal YoYo1 est perdu au fil du temps lorsque l’ADNdb est réséqué à partir de l’extrémité libre par Sgs1-Dna2. Simultanément, le signal mCherry colocalise avec les extrémités de l’ADN où l’ADNss est généré à la suite de la résection. Les caractéristiques biophysiques pertinentes de la résection, telles que la vitesse et la processivité, peuvent être extraites en quantifiant les pentes des trajectoires de perte de signal YoYo1. La figure 2C,D montre la distribution des vitesses et de la processivité, respectivement, de la résection par Sgs1-Dna2.

Figure 1 : Schémas de l’assemblage des cellules d’écoulement et des rideaux d’ADN. (A) Illustration par étapes de l’assemblage des cellules d’écoulement. Du ruban adhésif double face est placé sur le dessus de la lame de quartz, et un gabarit en papier est utilisé pour découper un canal rectangulaire à partir du centre, qui est scellé avec une lamelle de recouvrement sur le dessus pour former une chambre. (B) Schémas de rideaux d’ADN entièrement assemblés. Le panneau central présente une cellule d’écoulement à simple attache, tandis que le panneau inférieur présente une cellule d’écoulement à double attache. Veuillez cliquer ici pour voir une version agrandie de cette figure.
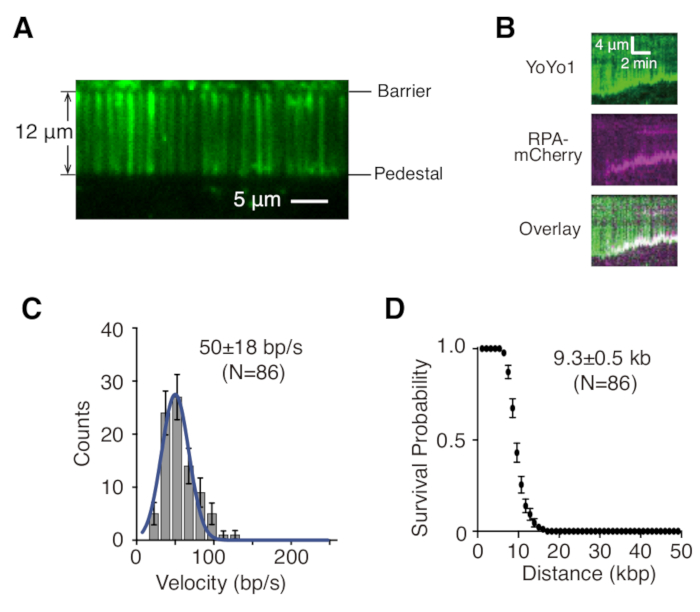
Figure 2 : Visualisation et analyse d’un rideau d’ADN d’une molécule unique. (A) Image représentative à grand champ d’un rideau d’ADN λ à double attache coloré avec YoYo1 (vert). (B) Kymographes représentatifs montrant la résection de l’ADNdb coloré à YoYo1 (vert) en présence de GFP-Sgs1 pré-lié (non visible) lorsqu’il est chassé avec de l’ADN2, en présence de RPA-mCherry (magenta) et de 2 mM ATP. Distribution de vitesse (C) et tracé de processivité (D) de la résection de l’ADNdb par Sgs1-Dna2, obtenus en quantifiant le taux et l’étendue de la perte de signal YoYo1 au cours du temps. Les panneaux (B), (C) et (D) sont adaptés de Xue et al.19. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Les rideaux d’ADN se sont avérés être une plate-forme polyvalente pour l’étude de divers processus de réparation de l’ADN 13,14,15,16. Les molécules d’ADN qui restent étendues près de la surface tout au long de l’expérience, soit par un flux tampon continu, soit par une double attache au piédestal, permettent l’imagerie TIRFM des interactions protéine-ADN sur les intermédiaires de réparation au niveau de la molécule unique. La méthode améliore le débit expérimental et la prévisibilité, car des centaines de molécules d’ADN sont alignées de manière ordonnée au niveau de barrières nanofabriquées plutôt que de ne pas être spécifiquement attachées à la surface, ce qui favorise le traitement de grands ensembles de données.
L’analyse minutieuse de la série d’images en accéléré qui en résulte fournit des mesures quantitatives des interactions protéine-ADN (c’est-à-dire les durées de vie de liaison, les positions, la stœchiométrie, la cinétique de dissociation, les taux de translocation, les processivités des protéines motrices et les colocalisations et interactions entre les protéines et/ou l’ADN). Par rapport à d’autres techniques d’imagerie à molécule unique qui attachent les molécules d’ADN directement à la surface ou combinent l’imagerie confocale avec la manipulation de force dans les instruments commerciaux, le principal compromis des rideaux d’ADN pour un débit plus élevé est l’établissement des techniques nécessaires à la configuration initiale 13,14,15,16. Cependant, une fois établie, la capacité de collecter des données sur des centaines de molécules simultanément dans une cellule d’écoulement permet de réaliser des économies significatives sur le temps consacré à la collecte de données.
Comme pour de nombreuses autres méthodes d’imagerie de molécules uniques, la capacité de préparer une surface propre dicte en grande partie la capacité de l’utilisateur à assembler de manière fiable des rideaux d’ADN. Dans ce cas, les surfaces propres nécessitent non seulement de commencer par une lame propre, mais aussi de s’assurer de ne pas introduire de bulles d’air microscopiques dans la chambre lors de l’une des connexions fluidiques. Tout d’abord, en ce qui concerne la propreté des diapositives, les étapes détaillées ci-dessus dans la section 6 devraient suffire comme processus de nettoyage de routine. L’utilisation de solutions filtrées et de tampons est également recommandée. Deuxièmement, il est essentiel d’effectuer des connexions goutte à goutte lors de la fixation de la cellule d’écoulement au système d’injection de tampon pour éviter l’introduction de bulles. Il peut être utile de dégazer l’eau bidistillée utilisée pour la fabrication de tampons. Il est également recommandé d’observer attentivement tout processus d’injection pour s’assurer qu’il n’y a pas de bulles. Si une bulle microscopique est introduite avant d’atteindre la zone centrale à motifs, il est possible d’y remédier en injectant un tampon à partir de l’orifice microfluidique du côté opposé pour repousser la bulle vers l’extérieur.
Un autre point important à prendre en compte dans toute expérience basée sur la fluorescence est le rapport signal/bruit. Une clé de l’observation des protéines fluorescentes uniques (c’est-à-dire la GFP) réside également dans les surfaces propres. Après une utilisation prolongée, les agrégats de protéines s’accumulent généralement au niveau des barrières de chrome et des piédestaux. Par conséquent, si un fort signal de fluorescence est observé aux barrières avant l’ajout de protéines fluorescentes, il est conseillé de traiter les lames avec une immersion de 10 minutes dans une solution de Piranha. La période est courte pour éviter d’éroder les fonctionnalités de chrome. Le lavage Piranha doit être suivi de la procédure de nettoyage régulière.
La nanofabrication à l’aide de la lithographie par faisceau d’électrons peut s’avérer difficile, en raison de ses exigences en matière d’équipement et d’installations, ainsi que de son expertise existante en science des matériaux. Comme alternative, une méthode basée sur la lithographie UV pour la fabrication de caractéristiques de chrome pour les rideaux d’ADN a été développée20. Ce protocole fournit également un modèle de base pour l’ADN lambda à double connexion à 12 μm (voir informations supplémentaires). Plusieurs aspects de la conception de ce modèle peuvent être ajustés pour répondre à divers besoins expérimentaux. Il s’agit notamment de la distance entre les barrières et les socles, de l’espacement entre les puits adjacents dans la barrière pour contrôler la densité de l’ADN, et de la forme et de la taille des socles pour optimiser l’efficacité de la double attache et minimiser l’incertitude des longueurs de double attache. D’autres défis pratiques peuvent inclure le choix de stratégies de marquage fluorescent pour les protéines, la minimisation des biais dans l’analyse manuelle des données et le développement de scripts pour un traitement plus rationalisé des données.
Bien que le protocole décrit ici implique des substrats d’ADNdb, les rideaux d’ADN basés sur l’ADNss ont également été largement utilisés 13,14,15,16. La préparation des substrats d’ADNss utilise la réplication en cercle roulant du plasmide d’ADNss M13 avec des amorces biotinylées13. L’assemblage du rideau d’ADNss nécessite également l’utilisation d’urée et de la protéine de liaison à l’ADNss RPA pour éliminer la structure secondaire et étendre le substrat de l’ADNsb13. Il pourrait également être possible d’étendre les rideaux d’ADN à l’utilisation d’ARN ou d’hybrides ADN/ARN pour l’étude des protéines interagissant avec l’ARN. Dans l’état actuel des choses, la plate-forme de rideau d’ADN peut bénéficier de l’automatisation du processus d’assemblage, comme détaillé dans la section 5, dont la plupart sont des injections répétitives d’un ensemble standard de tampons. Cela peut conduire à une amélioration de l’efficacité dans la réalisation de plusieurs expériences simultanément. De plus, une suite de scripts d’analyse unifiée et potentiellement basée sur l’apprentissage automatique accélérerait encore le traitement des données avec un minimum de biais.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient les membres passés et actuels du laboratoire Greene d’avoir développé, optimisé et discuté le protocole des rideaux d’ADN, ainsi que d’avoir commenté ce manuscrit. Ce travail a été soutenu par des subventions de la National Science Foundation (MCB1154511 à l’ECG) et des National Institutes of Health (R01CA217973 à l’ECG).
matériels
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Références
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon