Method Article
DNAカーテンは、相同組換え中の複雑な分子システムに光を当てます
要約
DNAカーテンは、マイクロ流体サンプルチャンバーの表面に整列したDNA分子と相互作用する数百または数千のDNA結合タンパク質をリアルタイムで視覚化する新しい方法を提供します。
要約
相同組換え(HR)は、すべての生物の二本鎖DNA切断(DSB)と失速した複製フォークの修復に重要です。HRの欠陥は、ヒト細胞におけるゲノムの完全性の喪失および発癌性形質転換と密接に関連しています。HRには、複雑なタンパク質セットの協調的な作用が含まれますが、その多くはまだ十分に理解されていません。ここで紹介する研究の鍵となるのは、「DNAカーテン」と呼ばれる技術で、これはマイクロ流体サンプルチャンバーの表面にDNA分子を整列させる技術です。その後、全反射蛍光顕微鏡(TIRFM)で可視化することができます。DNAカーテンは、私たちの研究室で開発されたもので、他の方法論では容易に明らかにできないミリ秒スケールとナノメートルスケールの解像度で時空間情報に直接アクセスすることができます。DNAカーテンの主な利点は、単一分子実験から統計的に関連性のあるデータの収集を簡素化することです。この研究は、細胞がゲノムの完全性をどのように調節し、維持するかについての新たな洞察をもたらし続けています。
概要
ゲノムの完全性の維持は、すべての生細胞が適切に機能するために重要です1。ゲノムの完全性の欠陥は、さまざまな種類のがんや加齢性変性疾患2など、深刻な健康状態につながる可能性があります。相同組換え(HR)は、テンプレート依存性DNA合成を使用して、DNA二本鎖切断(DSB)、一本鎖DNA(ssDNA)ギャップ、および鎖間DNA架橋を修復します3。HRは、停止および崩壊したレプリケーションフォークのリカバリにも必要です3,4。さらに、HRは減数分裂中の正確な染色体分離に不可欠です5,6。
HRには、複雑なタンパク質セットの協調的な作用が関与しており、その多くはまだ十分に理解されていません1。例としては、複製タンパク質A(RPA)、Rad51、Rad54など、多くのものがあります7。原核細胞と真核細胞の両方におけるHR反応には、ssDNA中間体が関与しており、これはssDNA結合タンパク質(原核生物ではSSB、真核生物ではRPA)によって急速にコーティングされています8。これらのタンパク質は、ヌクレアーゼからssDNAを保護し、二次構造を排除し、下流の因子の動員を促進します8,9。Rad51は、すべての生物に存在するDNAリコンビナーゼの高度に保存されたATP依存性Rad51/RecAファミリーのメンバーです1。Rad51は、相同dsDNAドナーのDNA鎖浸潤を促進します。その重要性を考えると、Rad51は高度に制御されており、これらの制御プロセスの欠陥は、一般的にゲノム完全性の喪失と発癌性形質転換と関連しています7。Rad54は、dsDNAトランスロカーゼおよびクロマチンリモデラー10,11のSwi2/Snf2ファミリーのメンバーである。これらのタンパク質は、必須のRad51調節因子として機能します。重要なことに、Rad54は鎖侵入のdsDNA産物からRad51を除去し、クロマチン11上のRad51の誤蓄積を防ぐためにも必要です。酵母細胞と細菌細胞の両方でHRに関与するタンパク質の分子活性は、HRにおけるそれらの機能に光を当てていますが、それらの活性がHRにどのように寄与するかは正確にはよくわかっていません12。
DNAカーテンは、他の方法ではアクセスできない分子メカニズムと高分子ダイナミクスへの直接アクセスを提供するユニークなプラットフォームとして登場しました13,14。DNAカーテンを調製するために、マイクロ流体チャンバーの表面を脂質二重膜でコーティングし、DNA分子をビオチン-ストレプトアビジン結合を介して二重層につなぎます。二重層は、天然の細胞膜を模倣することにより、表面を不活性にします。流体力学的力により、ナノ加工されたバリアに沿ってDNAが整列し、全反射蛍光顕微鏡(TIRFM)により、単一の視野で数百の分子を可視化することができます。バリアは電子ビームリソグラフィーによって作成され、バリア設計のバリエーションにより、DNAの分布とテザリング形状を正確に制御できます。これらのアプローチは、ssDNAまたはdsDNA 13,14,15,16,17,18のいずれかで容易に適用できます。また、DNAの両端をサンプルチャンバー表面につなぎ合わせることができるように、ペデスタルを(バリアとともに)ナノ加工して、バッファーフローがない状態で定常状態の実験を行うこともできます。
個々のタンパク質-核酸複合体の時間依存的な変化は、リアルタイムビデオの検査によって明らかになり、DNA上のタンパク質の位置が経時的に変化することを示すキモグラフを使用して印刷物で表されます。DNAカーテンアプローチの重要な側面は、個々の反応成分の挙動をリアルタイムで観察できるため、必ずしも 先験的な モデルや分子メカニズムの仮定を必要としないことです。これにより、分子の振る舞いを直接観察することができます。ここでは、このプロトコルは、dsDNA基質を有するDNAカーテンの調製方法、ならびに相同組換えにおける中間体を研究するためのその応用を説明する。
プロトコル
1. 脂質ストックの調製
- 18:1(Δ9-CIS)PC 1 g、18:1 PEG2000 PE 100 mg、18:1 ビオチニルキャップ PE 5 mg を 10 mL のクロロホルムに溶解します。
注:DOPCとDPPEはどちらも十分に特性評価されており、(PEG修飾とともに)フローセルの表面への非特異的吸着を最小限に抑える能力のために選択されています。溶解した脂質マスターミックスは、分注して-20°Cで最大12ヶ月間保存できます。 - クロロホルムを使用して有機溶媒ガラスシリンジを洗浄し、脂質マスターミックスの200μL(最終所望容量の10%)をガラスバイアルに移します。
- ガラスバイアルからクロロホルムを非常に穏やかな圧縮窒素の流れで蒸発させます。蒸発の終わりに向かって圧力をわずかに上げて、クロロホルムの痕跡がすべて除去されるようにします。
注:蒸発プロセス中にバイアルを傾けて回転させ、脂質が壁の白いフィルムの均一な層として乾燥するようにします。 - バイアルをキャップを外して真空下で一晩置きます。
- 乾燥した脂質フィルムに2 mLの脂質バッファー(10 mM Tris-HCl [pH 8.0]、100 mM NaCl)を加え、バイアルにキャップをします。室温(RT)で少なくとも1時間インキュベートし、溶解するまでボルテックスします。
- 溶液を5mLのポリスチレン丸底チューブに移し、次のパラメータでマイクロチップを使用して氷上で超音波処理します:振幅= 50、処理時間= 1.5分、パルス= 15秒、オフ時間= 2分。溶液は超音波処理後に透明になります(総エネルギー= 1,500〜2,000 J)。
- 0.22 μm PVDFシリンジフィルターで溶液をろ過し、脂質ストックを4°Cで最大1ヶ月間保存します。
2. dsDNA基質の調製
- λ-DNAストック(500 μg/mL)を65°Cに温めます。
- 1.6 pMのλ-DNA、各オリゴヌクレオチド(BioL:5'Phos-AGG TCG CCG CCC-3BioおよびDigR:5'Phos-GGG CGG CGA CCT-3Dig_N)の100倍モル過剰(160 pM)、および10x T4リガーゼ反応バッファー(総容量600 μL)60 μLを含むアニーリング/ライゲーションミックスを調製します。
注:BioLおよびDigRオリゴヌクレオチドは、それぞれビオチン化(Bio)およびジゴキシゲニン(Dig)で修飾されます。ビオチン化は、ストレプトアビジン結合を介してDNA分子をビオチン化脂質に結合させることを可能にします。Dig修飾は、台座にコーティングされた抗Dig抗体をDig部分に結合させることにより、ダブルテザリングを可能にします。 - 反応を65°Cで10分間インキュベートすることにより、オリゴヌクレオチドをλ-DNAcos末端にアニールし、その後、ゆっくりと室温まで冷却します。
- 5 μLのT4 DNAリガーゼを添加し、RTで一晩インキュベートします。
注:アニーリングおよびライゲーションステップは、サーマルサイクラーでも実行できます。 - 300 μL の PEG-8000 溶液 (30% w/v PEG-8000 および 10 mM MgCl2) を添加して、λ-DNA を沈殿させます。4°Cで少なくとも1時間インキュベートします。
- 14,000 x g でRTで5分間遠心分離し、ペレットを乱さずに上清を慎重に取り除きます。オプションで、ペレットを500μLの冷やした70%エタノールで1回洗浄します。
- ペレットを 40 μL の TE バッファー (10 mM Tris-HCl、1 mM EDTA [pH 8.0]) に再懸濁します。
3. クロムパターンの微細加工
- テーブルトップドリルプレスでダイヤモンドコーティングされた1.4mmドリルビットを使用して、1インチ x 3インチの石英顕微鏡スライド(~3cm間隔)の中央に2つの穴を開けます(穴あけ位置の形状については 図1 を参照)。
- ドリルで開けたスライドを200 mLのピラニア溶液(75%硫酸[97%]と25%過酸化水素)に浸して洗浄します。30分間インキュベートします。
注意: 露出した皮膚との接触を避けるために、細心の注意を払う必要があります。冷たい過酸化水素を硫酸に非常にゆっくりと混合して溶液を調製します。ピラニアの廃棄物は、指定された廃棄物容器に適切に廃棄してください。 - スライドを200mLの二重蒸留水に浸してすすぎます。2回繰り返して、残っているピラニアを排除します。圧縮された窒素の穏やかな流れでスライドを乾燥させます。
- スピンコーター(4,000 rpm、45 s、ランプレート300 rpm/s)を使用して、クリーンスライドをフォトレジストと導電性ポリマーで次のようにコーティングします。
- 3% (w/v) ポリメチルメタクリレート (PMMA; 24.3K MW; アニソールに溶解) の層でスライドをコーティングします。
- 1.5%(w / v)ポリメチルメタクリレート(PMMA;495K MW;アニソールに溶解)の層でスライドをコーティングします。
- スライドに帯電防止剤を塗布し、電子ビームリソグラフィーに使用します。
- コーティングされたスライドにバリアやペデスタルのカスタムパターンを、ナノパターン生成システムソフトウェアを搭載した走査型電子顕微鏡による電子ビームリソグラフィーで書き込みます。
- まず、電子線リソグラフィー用の帯電防止剤を二重蒸留水で洗い流してスライドを現像します。次に、現像液(75%メチルイソブチルケトン[MIBK]および25%イソプロパノール;-20°Cで冷やしておきます)を含む50 mLチューブに入れ、氷浴で1分間低温で超音波処理します。
- 現像液をイソプロパノールで洗い流し、圧縮窒素を穏やかに流して乾燥させます。
- 電子ビーム蒸着器を使用して、厚さ250 Åのクロム層をパターン化された表面に0.5 Å/sで堆積します。
- スクワートボトルからアセトンを使用してクロムと残りのPMMAを持ち上げ、続いてアセトン浴でスライドを10分間超音波処理します。噴出ボトルのアセトンで再度洗います。
- 残りのアセトンによる堆積物を防ぐために、スライドをイソプロピルアルコールですすぎ、圧縮窒素の穏やかな流れで乾燥させます。
4. フローセルの組立
- 35 mm x 5 mmの長方形の用紙テンプレート(クロームパターンと事前に開けられた入口/出口穴を覆う)を、パターン化された面を上に向けて、クォーツスライドの上に置きます( 図1A)。
- クォーツスライドに両面テープ(厚さ1 mm)を貼り付けて紙のテンプレートを覆い、かみそりの刃を使用して長方形の紙のテンプレートを切り取ります( 図1A)。
- 両面テープの上にガラスのカバースリップを置き、カバースリップをテープに密封するために穏やかな圧力を加えます( 図1A)。カバースリップとスライドの間のスペースは、両面テープでシールされ、マイクロ流体チャンバーを形成します。
注:実験中の漏れを防ぐために、カバースリップとスライドの間のシールが十分に固定されていることが重要です。 - カバースリップスライドサンドイッチをスライドガラスの間に置き、4つの側面すべてにバインダークリップを取り付けて、圧力を均等に分散します。アセンブリを真空オーブンに135°Cで60分間置き、両面テープを溶かしてチャンバーをシールします。
- オーブンから取り出し、バインダークリップとスライドガラスを外します。
- 接着されたアセンブリを加熱ブロック(220°C)に1分間置き、取り外し、慎重に穏やかな圧力を加えてマイクロ流体ポートを適切に密閉することにより、ホットグルーガンで事前に開けられた入口/出口穴にマイクロ流体ポートを取り付けます。続行する前に、接着剤が完全に硬化するのを待ちます。
注:組み立てたフローセルは、50 mLチューブに入れて真空下で保存できます。
5. dsDNAカーテンの組み立て
- インレット/アウトレットチューブをフローセルのマイクロ流体ポートに取り付け、2つの10 mLシリンジを使用して20 mLの二重蒸留水でチャンバーを洗浄します。
- フローセルの両側に接続された二重蒸留水で満たされた10 mLシリンジで、入口シリンジと出口シリンジの間に水を押したり引いたりする力を加えます。このステップでは、フローセルからすべての気泡が除去されます。
- チャンバーを3 mLの脂質バッファー(10 mM Tris-HCl [pH 8.0]、100 mM NaCl)で洗浄します。
注意: フローセルに気泡が入らないように、フローセルへのすべての接続がドロップツードロップであることを確認してください。 - 40 μLのリポソーム溶液と1 mLの脂質緩衝液を混合します。300 μLの脂質溶液をフローセルに注入し、5分間インキュベートします。注入とインキュベーションを2回繰り返します。
注:シリンジがフローセルに接続されるたびに、注入用の入口/出口側を交互にしてください。 - チャンバーを3 mLの脂質緩衝液で洗浄し、15分間インキュベートします。
注:このインキュベーション期間は、実験のスケジュールに応じて5分から2時間までさまざまですが、最終的なカーテンの組み立てに顕著な違いはありません。 - 40 μL の 1 mg/mL 抗ジゴキシゲニンストック溶液を 200 μL の脂質バッファーと混合します。溶液100μLを注入し、10分間インキュベートします。注入とインキュベーションを1回繰り返してから、3 mLの脂質バッファーで洗浄します。
注意: この手順は、ダブルテザーdsDNAカーテンの場合にのみ必要です。シングルテザーdsDNAカーテンの場合は、この手順をスキップします。 - チャンバーを3 mLのBSAバッファー(40 mM Tris-HCl [pH 8.0]、2 mM MgCl2、1 mM DTT、および0.2 mg/mL BSA)で洗浄します。
- 10 μL の 1 mg/mL ストレプトアビジン原液を 990 μL の BSA バッファーと混合します。400 μLを2回に分けて注入し、各注入後に10分間のインキュベーションを行います。
- チャンバーを3 mLのBSAバッファーで洗浄し、余分なストレプトアビジンを除去します。
- 25 ngのdsDNA基質ストックを1 mLのBSAバッファーで希釈します。DNA溶液を一度に200μLずつゆっくりと注入し、その後、各注入後に5分間インキュベーションします。必要なDNAの量は、特定のタイプの実験に最適化することができます。
- フローセルを顕微鏡ステージに取り付け、必要に応じてピントを調整します。
- 入力ポートと出力ポートを、シリンジポンプと高圧スイッチバルブで構成されるサンプル注入システムに接続します。
- DNAをペデスタル(テザー長12 μm)にダブルテザーし、チャンバーをBSAバッファーで0.8 mL/minで5分間連続的に洗浄します。
注:異なるダブルテザーの長さを最適化するために、流量と時間を調整する必要がある場合があります。このステップは、ダブルテザーDNAカーテンにのみ必要です。シングルテザーdsDNAカーテンの場合は、この手順をスキップします。 - これで、カーテンを実験する準備が整いました。
6. フローセルのリサイクル
- スライドをエタノールに少なくとも30分間浸してから、かみそりの刃を使用してマイクロ流体ポートとカバースリップを慎重に取り外します。カバースリップは適切に廃棄し、マイクロ流体ポートに残っている接着剤をすべて取り除いて、再利用できるようにします。スライド上の接着剤の残留物をこすり洗いします。
注意: スライドの中央にあるパターン化された領域を傷つけないように注意してください。 - スライドを二重蒸留水で十分にすすぎ、攪拌しながら2%アルカリ性液体石英洗浄濃縮物に一晩浸します。
- スライドを二重蒸留水で十分にすすぎ、80°Cで攪拌しながら2%アルカリ性液体石英洗浄濃縮液に1時間浸し、続いて45°Cで15分間超音波処理します。
- スライドを二重蒸留水で十分にすすぎ、攪拌しながら1 M NaOHに30〜60分間浸します。
- スライドを二重蒸留水で十分にすすぎ、次にイソプロピルアルコールに浸し、45°Cで15分間超音波処理します。
- スライドをエタノールで十分にすすぎ、攪拌しながらエタノールに1時間浸します。
- スライドを真空オーブンに1時間入れ、135°Cで乾燥させます。これで、スライドをフローセルに再組み立てする準備が整いました。
結果
上記で説明したのは、DNA修復中間体におけるタンパク質-DNA相互作用の研究という文脈で、シングルテザーおよびダブルテザーdsDNAカーテンの調製、組み立て、およびイメージングです。 図1A は、フローセル内のすべてのコンポーネントを、組み立てられた順序で層状に並べて示しています。 図1B は、シングルテザーまたはダブルテザーDNAカーテンの概略図を示しています。脂質二重層は、フローセルの表面を不動態化するために使用されます。DNAカーテンは、一端が脂質につながれ、クロム障壁で整列し、流れの方向に垂直に配向されたDNA分子の平行配列で構成されています。ダブルテザーDNAカーテンの場合、DNAのもう一方の端はジゴキシゲニンと抗ジゴキシゲニンの相互作用によって台座につながれているため、DNAが伸びたまま流れがない場合でもイメージングを行うことができます。dsDNAは、蛍光色素YoYo1で染色し、TIRF顕微鏡で可視化することができます。 図2A は、YoYo1染色されたダブルテザーDNAカーテンの代表的な広視野画像を示しています。
DNAカーテン実験から収集されたタイムラプス画像シリーズは、通常、ImageJで関心のある各DNA分子の縦軸上のDNA分子の位置と横軸上の時間に対して、最初にキモグラフを生成することによって分析されます。 図2B は、一本鎖DNA結合タンパク質RPA-mCherry(マゼンタ)およびATPの存在下で、酵母切除装置GFP-Sgs1(不明)、Top3-Rmi1、およびDna2によるYoYo1染色された単一連結λ-DNA(緑色)の切除を示す代表的なキモグラフである。YoYo1のシグナルは、dsDNAがSgs1-Dna2によって自由末端から切り取られるため、時間の経過とともに失われます。同時に、mCherryシグナルは、切除の結果としてssDNAが生成されているDNA末端と共局在します。速度や処理能力など、切除の生体物理学的に関連する特性は、YoYo1信号損失の軌跡の傾きを定量化することで抽出できます。 図2C、D は、Sgs1-Dna2による切除の速度と処理能力の分布をそれぞれ示しています。

図1:フローセルアセンブリとDNAカーテンの概略図(A)フローセルアセンブリの段階的な図。石英スライドの上に両面テープを貼り、中央から長方形の溝を紙のテンプレートで切り取り、上部にカバースリップでシールしてチャンバーを形成します。 (B)完全に組み立てられたDNAカーテンの概略図。中央のパネルはシングルテザーフローセルを示し、下部のパネルはダブルテザーフローセルを示しています。この図の拡大版を表示するには、ここをクリックしてください。
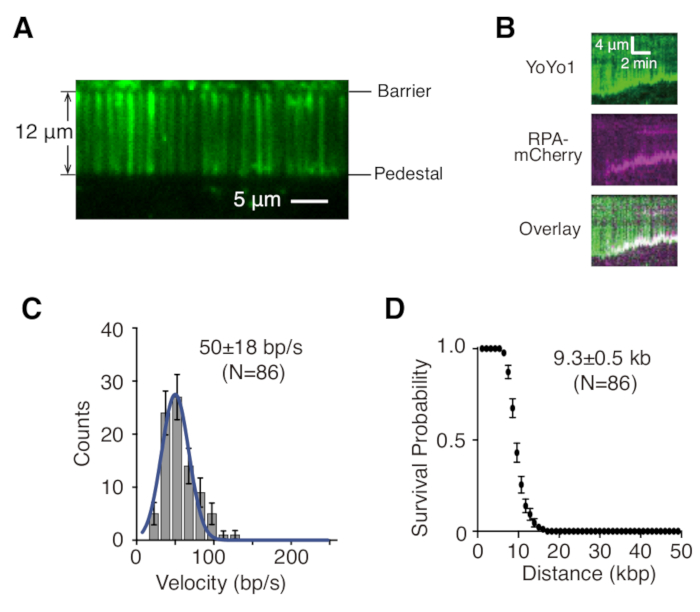
図2:1分子DNAカーテンの可視化と解析(A)YoYo1で染色した二重テザーλ-DNAカーテンの代表的な広視野画像(緑)。(B)RPA-mCherry(マゼンタ)および2 mM ATPの存在下で、Dna2で追跡したときに、事前に結合したGFP-Sgs1(見えない)の存在下でYoYo1で染色されたdsDNA(緑色)の切除を示す代表的なキモグラフ。経時的なYoYo1シグナル損失の速度と程度を定量化することによって得られた、Sgs1-Dna2によるdsDNA切除の速度分布(C)と処理能力プロット(D)。パネル(B)、(C)、および(D)は、Xue et al.19から採用されています。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
DNAカーテンは、さまざまなDNA修復プロセスを研究するための汎用性の高いプラットフォームであることが証明されています13,14,15,16。実験全体を通して表面近くに伸長したままのDNA分子は、連続的なバッファーフローまたはペデスタルへの二重テザリングのいずれかを通じて、修復中間体上のタンパク質-DNA相互作用の単一分子レベルでのTIRFMイメージングを可能にします。この方法では、数百のDNA分子が表面に非特異的に付着するのではなく、ナノファブリケーションされたバリアに整列しているため、実験のスループットと予測可能性が向上します。これにより、大規模なデータセットの処理がさらに向上します。
得られたタイムラプス画像シリーズを慎重に解析することで、タンパク質-DNA相互作用(結合寿命、位置、化学量論、解離速度、転座率、モータータンパク質の処理能力、タンパク質とDNA間の共局在と相互作用)を定量的に測定できます。DNA分子を直接表面につなぎ合わせるか、または市販の機器で共焦点イメージングと力操作を組み合わせる他の単一分子イメージング技術と比較して、より高いスループットのためのDNAカーテンの主なトレードオフは、初期設定13,14,15,16に必要な技術を確立することである.しかし、一度確立されると、1つのフローセルで数百の分子のデータを収集する能力が確立されると、データ収集に費やす時間が大幅に節約されます。
他の多くの単一分子イメージング法と同様に、クリーンな表面を調製する能力は、DNAカーテンを確実に組み立てるユーザーの能力を大きく左右します。この場合、表面をきれいにするには、きれいなスライドから始めるだけでなく、流体接続を行う際にチャンバーに微細な気泡を導入しないようにする必要があります。まず、スライドの清浄度に関しては、上記のセクション6で詳述した手順が定期的なクリーニングプロセスとして十分であるべきです。ろ過された溶液とバッファーの使用も推奨されます。次に、フローセルをバッファー注入システムに取り付けている間にドロップツードロップ接続を行うことは、気泡の発生を防ぐために不可欠です。バッファーの作成に使用される二重蒸留水を脱気すると役立つ場合があります。また、注入プロセスを注意深く観察して、気泡が存在しないことを確認することをお勧めします。微細な気泡が中央のパターン領域に到達する前に導入された場合は、反対側のマイクロ流体ポートからバッファーを注入して気泡を押し戻すことで改善できます。
蛍光ベースの実験で考慮すべきもう1つの重要なポイントは、S/N比です。単一の蛍光タンパク質(GFP)を観察するための鍵は、きれいな表面にも存在します。長期間使用すると、タンパク質凝集体は通常、クロムバリアとペデスタルに蓄積します。したがって、蛍光タンパク質を添加する前にバリアで強い蛍光シグナルが観察された場合は、スライドをピラニア溶液に10分間浸漬して処理することをお勧めします。クロームの特徴が損なわれないように、期間は短く保たれています。ピラニアウォッシュの後には、通常のクリーニング手順を実行する必要があります。
電子ビームリソグラフィーを用いたナノファブリケーションは、材料科学における既存の専門知識に加え、機器や設備を必要とするため、困難な場合があります。代替として、DNAカーテン用のクロム特徴を作製するためのUVリソグラフィーベースの方法が開発された20。このプロトコールには、12 μmでのラムダDNAのダブルテザリングの基本パターンも付属しています(補足情報を参照)。このパターンの設計のいくつかの側面は、さまざまな実験ニーズに合わせて調整できます。これには、バリアとペデスタルの間の距離、DNA密度を制御するためのバリア内の隣接するウェル間の間隔、ダブルテザリングの効率を最適化し、ダブルテザリングの長さの不確実性を最小限に抑えるためのペデスタルの形状とサイズが含まれます。その他の実用的な課題としては、タンパク質の蛍光標識戦略の選択、手動データ解析におけるバイアスの最小化、より効率的なデータ処理のためのスクリプトの開発などがあります。
ここで述べるプロトコルはdsDNA基質を含むが、ssDNAに基づくDNAカーテンもまた広く使用されてきた13,14,15,16。ssDNA基質の調製は、ビオチン化プライマー13によるM13 ssDNAプラスミドのローリングサークル複製を利用する。ssDNAカーテンの組み立てには、二次構造を排除し、ssDNA基質13を拡張するために、尿素およびssDNA結合タンパク質RPAの使用も必要である。また、RNA相互作用タンパク質の研究にDNAカーテンをRNAまたはDNA/RNAハイブリッドを使用するように拡張することも可能かもしれません。現状では、DNAカーテンプラットフォームは、セクション5で詳述されているように、アセンブリプロセスの自動化の恩恵を受けることができます。そのほとんどは、標準セットのバッファーの反復注入です。これにより、複数の実験を同時に行う際の効率が向上する可能性があります。さらに、機械学習ベースの可能性のある統合分析スクリプトスイートにより、バイアスを最小限に抑えながらデータ処理をさらに高速化できます。
開示事項
著者は何も開示していません。
謝辞
著者らは、DNAカーテンプロトコルの開発、最適化、議論、およびこの原稿へのコメントについて、Greeneラボの過去および現在のメンバーに感謝します。この研究は、全米科学財団(MCB1154511からE.C.G.へ)および国立衛生研究所(R01CA217973からE.C.G.へ)からの助成金によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
参考文献
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved