Method Article
DNA 窗帘揭示了同源重组过程中的复杂分子系统
摘要
DNA 幕提供了一种新方法,可以实时可视化数百甚至数千个 DNA 结合蛋白,因为它们与微流体样品室表面排列的 DNA 分子相互作用。
摘要
同源重组 (HR) 对于修复所有生物体中的双链 DNA 断裂 (DSB) 和停滞的复制叉都很重要。HR 缺陷与人类细胞中基因组完整性的丧失和致癌转化密切相关。HR 涉及一组复杂蛋白质的协调作用,其中许多蛋白质仍然知之甚少。这里描述的研究的关键方面是一种称为“DNA 幕”的技术,该技术允许在微流体样品室的表面上组装对齐的 DNA 分子。然后可以通过全内反射荧光显微镜 (TIRFM) 对它们进行可视化。DNA 幕布是由我们的实验室开创的,它允许在毫秒级时间和纳米级分辨率下直接访问时空信息,这是通过其他方法无法轻松揭示的。DNA 幕布的一个主要优点是它简化了从单分子实验中收集统计相关数据的过程。这项研究继续为细胞如何调节和保持基因组完整性提供新的见解。
引言
维持基因组完整性对于所有活细胞的正常运作至关重要1。基因组完整性缺陷会导致严重的健康状况,包括各种类型的癌症和与年龄相关的退行性疾病2。同源重组 (HR) 使用模板依赖性 DNA 合成来修复 DNA 双链断裂 (DSB)、单链 DNA (ssDNA) 间隙和链间 DNA 交联3。HR 对于恢复停滞和折叠的复制分叉 3,4 也是必要的。此外,HR 对于减数分裂期间的准确染色体分离至关重要 5,6。
HR 涉及一组复杂蛋白质的协调作用,其中许多蛋白质仍然知之甚少1。示例包括复制蛋白 A (RPA)、Rad51 和 Rad54 等7。原核细胞和真核细胞中的 HR 反应都涉及 ssDNA 中间体,该中间体被 ssDNA 结合蛋白(原核生物中的 SSB 和真核生物中的 RPA)快速包被8。这些蛋白保护 ssDNA 免受核酸酶的侵害,消除二级结构,并促进下游因子的募集 8,9。Rad51 是高度保守的 ATP 依赖性 DNA 重组酶 Rad51/RecA 家族的成员,存在于所有生物体中 1。Rad51 促进同源 dsDNA 供体的 DNA 链侵袭。鉴于其重要性,Rad51 受到高度调控,这些调控过程的缺陷通常与基因组完整性丧失和致癌转化有关7。Rad54 是 dsDNA 转位酶和染色质重塑基因 Swi2/Snf2 家族的成员10,11。这些蛋白质是 Rad51 必需的调节因子。重要的是,Rad54 从链侵袭的 dsDNA 产物中去除 Rad51,并且对于防止 Rad51 在染色质11 上错误积累也是必需的。酵母和细菌细胞中参与 HR 的蛋白质的分子活性揭示了它们在 HR 中的功能,但它们的活性如何对 HR 的贡献仍然知之甚少 12。
DNA 窗帘已成为一个独特的平台,可直接访问分子机制和大分子动力学,否则这些机制和大分子动力学将无法获得13,14。为了制备 DNA 幕,微流体室的表面涂有脂质双层,DNA 分子通过生物素-链霉亲和素键拴在双层上。双层通过模拟天然细胞膜使表面呈惰性。流体动力使 DNA 沿着纳米制造的屏障对齐,从而允许通过全内反射荧光显微镜 (TIRFM) 在单个视野中观察数百个分子。这些屏障是通过电子束光刻技术制成的,屏障设计的变化允许精确控制 DNA 的分布和栓系几何形状。这些方法很容易适用于 ssDNA 或 dsDNA 13,14,15,16,17,18。基座也可以进行纳米制造(连同屏障),以允许 DNA 的两端拴在样品室表面,以便可以在没有缓冲液流的情况下进行稳态实验。
通过实时视频的检查揭示单个蛋白质-核酸复合物的时间依赖性变化,并使用运动记录仪在打印中表示,运动记录仪显示蛋白质在 DNA 上的位置随时间的变化。DNA 窗帘方法的一个重要方面是它不一定需要关于分子机制 的先验 模型或假设,因为可以实时观察各个反应组分的行为。这允许直接观察分子行为。在此,该方案描述了如何用 dsDNA 底物制备 DNA 窗帘及其在研究同源重组中间体中的应用。
研究方案
1. 脂质原液的制备
- 将 1 g 18:1 (Δ9-Cis) PC、100 mg 18:1 PEG2000 PE 和 5 mg 18:1 生物素帽 PE 溶解在 10 mL 氯仿中。
注:DOPC 和 DPPE 均具有很好的特性,并且(连同 PEG 修饰)均因其能够最大限度地减少对流通池表面的非特异性吸附而被选中。溶解的脂质预混液可以分装并在 -20 °C 下储存长达 12 个月。 - 使用氯仿清洁有机溶剂玻璃注射器,然后将 200 μL(最终所需体积的 10%)脂质预混液转移到玻璃样品瓶中。
- 使用非常温和的压缩氮气流蒸发玻璃瓶中的氯仿。在蒸发结束时略微增加压力,以确保去除所有痕量的氯仿。
注:在蒸发过程中倾斜和旋转样品瓶,使脂质干燥,在壁上形成一层均匀的白色薄膜。 - 将未加盖的小瓶置于真空下过夜。
- 向干燥的脂质膜中加入 2 mL 脂质缓冲液(10 mM Tris-HCl [pH 8.0],100 mM NaCl)并盖上样品瓶。在室温 (RT) 下孵育至少 1 小时并涡旋直至溶解。
- 将溶液转移至 5 mL 聚苯乙烯圆底管中,并使用微尖在冰上超声处理,参数如下:振幅 = 50,处理时间 = 1.5 分钟,脉冲 = 15 秒,关闭时间 = 2 分钟。超声处理后溶液将变得透明(总能量 = 1,500-2,000 J)。
- 通过 0.22 μm PVDF 注射器过滤器过滤溶液,并将脂质原液在 4 °C 下储存长达 1 个月。
2. dsDNA 底物的制备
- 将 λ-DNA 原液 (500 μg/mL) 加热至 65 °C。
- 制备包含:1.6 pM λ-DNA、100x 摩尔过量 (160 pM) 的每种寡核苷酸(BioL:5'Phos-AGG TCG CCG CCC-3Bio 和 DigR:5'Phos-GGG CGG CGA CCT-3Dig_N)和 60 μL 10x T4 连接酶反应缓冲液(总体积为 600 μL)。
注:BioL 和 DigR 寡核苷酸分别被生物素化 (Bio) 和地高辛 (Dig) 修饰。生物素化允许 DNA 分子通过链霉亲和素键附着到生物素化脂质上。Dig 修饰通过将包被在基座上的抗 Dig 抗体与 Dig 基团结合来实现双重栓系。 - 通过在 65 °C 下孵育反应 10 分钟,将寡核苷酸退火到 λ-DNA cos 末端,然后使其缓慢冷却至 RT。
- 加入 5 μL T4 DNA 连接酶,并在 RT 下孵育过夜。
注意:退火和连接步骤也可以在热循环仪中进行。 - 通过添加 300 μL PEG-8000 溶液(30% w/v PEG-8000 和 10 mM MgCl2)沉淀 λ-DNA。在 4 °C 下孵育至少 1 小时。
- 在 RT 下以 14,000 x g 离心 5 分钟。小心去除上清液,不要干扰沉淀。或者,用 500 μL 冷冻的 70% 乙醇洗涤沉淀 1 次。
- 将沉淀重悬于 40 μL TE 缓冲液(10 mM Tris-HCl、1 mM EDTA [pH 8.0])中。
3. 铬模型的纳米加工
- 在台式钻床上使用金刚石涂层的 1.4 mm 钻头在 1“ x 3” 石英显微镜载玻片的中心(相距 ~3 cm)钻两个孔(钻孔位置的几何形状见 图 1 )。
- 将钻孔的载玻片浸入 200 mL 食人鱼溶液(75% 硫酸 [97%] 和 25% 过氧化氢)中,清洁它们。孵育 30 分钟。
注意: 必须格外小心,避免与裸露的皮肤接触。通过将冷的过氧化氢非常缓慢地混合到硫酸中来制备溶液。将食人鱼废物妥善丢弃在指定的废物容器中。 - 浸入 200 mL 双蒸水中冲洗玻片。重复 2 次以消除任何残留的食人鱼。用温和的压缩氮气流吹干载玻片。
- 使用旋涂机(4,000 rpm 持续 45 秒,斜坡速率为 300 rpm/s),用光刻胶和导电聚合物涂覆干净的载玻片,如下所示:
- 涂覆载玻片,涂有一层 3% (w/v) 聚甲基丙烯酸甲酯(PMMA;24.3K MW;溶于苯甲醚)。
- 用 1.5% (w/v) 聚甲基丙烯酸甲酯(PMMA;495K MW;溶于苯甲醚)的层包被载玻片。
- 用一层抗静电剂涂覆载玻片,用于电子束光刻。
- 使用电子束光刻技术和配备纳米图案生成系统软件的扫描电子显微镜为涂层载玻片上的屏障和基座编写自定义图案。
- 首先用双蒸水冲洗掉用于电子束光刻的抗静电剂,以显影载玻片。然后,将它们放入含有显影液(75% 甲基异丁基酮 [MIBK] 和 25% 异丙醇;在 -20 °C 下保持低温)的 50 mL 试管中,并在冰浴中以低功率超声处理 1 分钟。
- 用异丙醇冲洗掉显影液,然后用温和的压缩氮气流干燥。
- 使用电子束蒸发器,以 0.5 Å/s 的速度在图案表面上沉积一层 250 Å 厚的铬。
- 使用喷瓶中的丙酮去除铬和剩余的 PMMA,然后在丙酮浴中对载玻片进行超声处理 10 分钟。用喷瓶中的丙酮再次清洗。
- 为防止残留的丙酮沉积,请用异丙醇冲洗载玻片,然后用柔和的压缩氮气流干燥。
4. 流通池的组装
- 将 35 mm x 5 mm 矩形纸模板(覆盖镀铬图案以及预钻孔/出口孔)放在石英载玻片上,图案面朝上( 图 1A)。
- 在石英载玻片上贴上一条双面胶带(1 毫米厚),覆盖纸模板,然后用剃须刀片切出矩形纸模板( 图 1A)。
- 将玻璃盖玻片放在双面胶带上,轻轻按压以将盖玻片密封在胶带上( 图 1A)。盖玻片和载玻片之间的空间,用双面胶带密封,形成微流体室。
注意:盖玻片和载玻片之间的密封必须足够牢固,以防止实验过程中泄漏,这一点至关重要。 - 将盖玻片夹层放在载玻片之间,并在所有四个侧面连接粘合剂夹以均匀分配压力。将组件放入 135 °C 的真空烘箱中 60 分钟,以熔化双面胶带并密封腔室。
- 将其从烤箱中取出,然后松开活页夹和载玻片。
- 将胶合组件放在加热块 (220°C) 上 1 分钟,将其取下,然后小心地施加轻微的压力以正确密封微流体端口,从而用热胶枪将微流体端口连接到预先钻好的入口/出口孔。在继续之前,让胶水完全固化。
注:组装好的流通池可置于 50 mL 试管中并真空储存。
5. dsDNA 幕的组装
- 将入口/出口管连接到流通池微流体端口,并使用两个 10 mL 注射器用 20 mL 双蒸水清洗腔室。
- 将装满双蒸水的 10 mL 注射器连接到流通池的两侧,用力在入口和出口注射器之间推/拉水。此步骤可去除流通池中的所有气泡。
- 用 3 mL 脂质缓冲液(10 mM Tris-HCl [pH 8.0]、100 mM NaCl)清洗腔室。
注:确保与流通池的所有连接都是逐滴的,以避免将气泡引入流通池。 - 将 40 μL 脂质体溶液与 1 mL 脂质缓冲液混合。将 300 μL 脂质溶液注入流通池并孵育 5 分钟。重复注射和孵育 2 次。
注:每次将注射器连接到流通池时,请交替进样/出样侧进行进样。 - 用 3 mL 脂质缓冲液洗涤腔室并孵育 15 分钟。
注意:该孵育期可以从 5 分钟到 2 小时不等,具体取决于实验的安排,最终的帘幕组装没有明显差异。 - 将 40 μL 1 mg/mL 抗地高辛储备液与 200 μL 脂质缓冲液混合。注入 100 μL 溶液并孵育 10 分钟。重复注射和孵育 1 次,然后用 3 mL 脂质缓冲液洗涤。
注:此步骤仅适用于双栓系 dsDNA 帘。对于单栓系 dsDNA 窗帘,请跳过此步骤。 - 用 3 mL BSA 缓冲液(40 mM Tris-HCl [pH 8.0]、2 mM MgCl2、1 mM DTT 和 0.2 mg/mL BSA)清洗腔室。
- 将 10 μL 1 mg/mL 链霉亲和素储备液与 990 μL BSA 缓冲液混合。分两次单独进样 400 μL 进样,每次进样后孵育 10 分钟。
- 用 3 mL BSA 缓冲液清洗腔室,以去除任何多余的链霉亲和素。
- 在 1 mL BSA 缓冲液中稀释 25 ng dsDNA 底物储备液。缓慢注入 DNA 溶液,每次 200 μL,每次注射后孵育 5 分钟。所需的 DNA 量可以针对特定类型的实验进行优化。
- 将流通池安装到显微镜载物台上,并根据需要调整焦距。
- 将输入和输出端口连接到由注射泵和高压切换阀组成的样品进样系统。
- 用 BSA 缓冲液以 0.8 mL/min 的速度连续洗涤腔室 5 分钟,将 DNA 双系在基座上(系绳长度 12 μm)。
注意:可能需要调整流速和时间,以针对不同的双系绳长度进行优化。此步骤仅适用于双栓系 DNA 帘。对于单栓系 dsDNA 窗帘,请跳过此步骤。 - 窗帘现在已经准备好进行实验了。
6. 流通池的回收
- 将载玻片浸入乙醇中至少 30 分钟,然后使用剃须刀片小心地去除微流体端口和盖玻片。正确处理盖玻片并清除微流体端口上的任何残留胶水,以便它们可以重复使用。擦洗清洁玻片上的任何胶水残留物。
注意:注意不要刮伤幻灯片中心的图案区域。 - 用双蒸水彻底冲洗载玻片,并在搅拌下浸入 2% 碱性液体石英清洁浓缩液中过夜。
- 用双蒸水彻底冲洗载玻片,并在 80 °C 下搅拌浸入 2% 碱性液体石英清洁浓缩液中 1 小时,然后在 45 °C 下超声处理 15 分钟。
- 用双蒸水彻底冲洗载玻片,并在搅拌下浸入 1 M NaOH 中 30-60 分钟。
- 用双蒸水彻底冲洗载玻片,然后浸入异丙醇中并在 45 °C 下超声处理 15 分钟。
- 用乙醇彻底冲洗载玻片,并在搅拌下浸入乙醇中 1 小时。
- 将载玻片放入真空烘箱中 1 小时,以在 135 °C 下干燥。玻片现在可以重新组装到流通池中。
结果
上述是在研究 DNA 修复中间体中蛋白质-DNA 相互作用的背景下单栓系和双系 dsDNA 窗帘的制备、组装和成像。 图 1A 显示了流通池中的所有组件,按组装顺序分层。 图 1B 描述了单或双栓系 DNA 窗帘的示意图。脂质双层用于钝化流通池的表面。DNA 帘由一个平行的 DNA 分子阵列组成,这些分子的一端与脂质相连,并在铬屏障处对齐,垂直于流动方向。对于双栓系 DNA 幕,DNA 的另一端通过地高辛和抗地高辛相互作用拴在基座上,这样就可以在没有流动的情况下进行成像,同时 DNA 保持延伸。dsDNA 可以用荧光染料 YoYo1 染色,并通过 TIRF 显微镜观察。 图 2A 显示了 YoYo1 染色的双栓系 DNA 幕的代表性宽视野图像。
从 DNA 幕布实验中收集的延时图像序列通常通过首先生成运动图来分析,该运动图在 ImageJ 中绘制沿 DNA 分子在纵轴上的位置与横轴上的时间。图 2B 是代表性的运动图,显示了在单链 DNA 结合蛋白 RPA-mCherry(洋红色)和 ATP 存在下,酵母切除机制 GFP-Sgs1(不可见)、Top3-Rmi1 和 Dna2 切除 YoYo1 染色的单栓系 λ-DNA(绿色)。随着 dsDNA 被 Sgs1-DNA2 从自由端切除,YoYo1 信号会随着时间的推移而丢失。同时,mCherry 信号与 DNA 末端共定位,切除后在这些末端产生 ssDNA。通过量化 YoYo1 信号丢失轨迹的斜率,可以从切除的生物物理相关特征(例如速度和持续合成能力)中提取出来。图 2C,D 分别显示了 Sgs1-DNA 2 切除的速度和持续合成能力的分布。

图 1:流通池组装和 DNA 窗帘的示意图。 (A) 流通池组装的逐步示意图。在石英载玻片顶部放置双面胶带,用纸模板从中心切出一个矩形通道,顶部用盖玻片密封,形成腔室。 (B) 完全组装的 DNA 幕示意图。中间面板是一个单系系流动池,而底部面板是一个双系流式流通池。 请单击此处查看此图的较大版本。
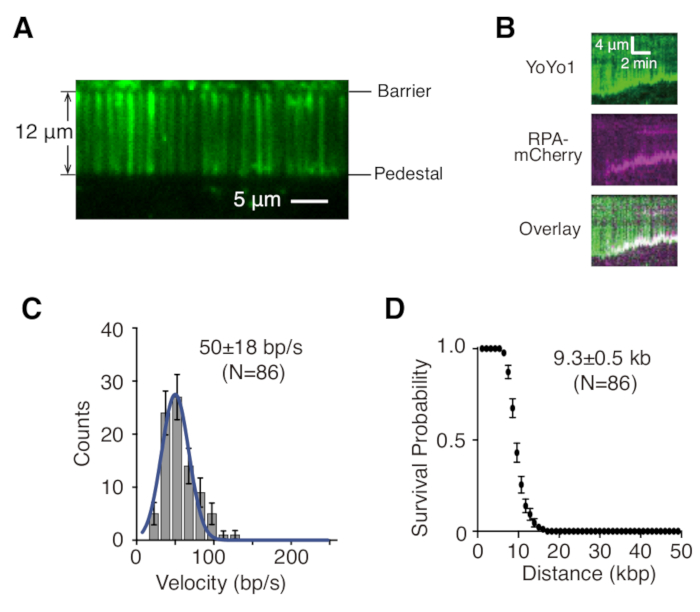
图 2:单分子 DNA 幕布可视化和分析。 (A) 用 YoYo1(绿色)染色的双栓系 λ-DNA 幕的代表性宽场图像。(B) 代表性运动图显示,在与 DNA 2 一起追踪时,在 RPA-mCherry(洋红色)和 2 mM ATP 存在下,在预结合的 GFP-Sgs1(不可见)存在下,YoYo1 染色的 dsDNA(绿色)被切除。通过 Sgs1-DNA 2 切除 dsDNA 的速度分布 (C) 和持续合成能力图 (D),通过量化 YoYo1 信号随时间丢失的速率和程度获得。面板 (B)、(C) 和 (D) 改编自 Xue 等人19。请单击此处查看此图的较大版本。
讨论
DNA 窗帘已被证明是研究各种 DNA 修复过程的多功能平台 13,14,15,16。在整个实验过程中,通过连续缓冲液流或双系系到基座,DNA 分子在表面附近延伸,允许在单分子水平上对蛋白质-DNA 相互作用进行 TIRFM 成像。该方法提高了实验通量和可预测性,因为数百个 DNA 分子在纳米制造的屏障处有序排列,而不是非特异性地附着在表面上,这进一步有利于大型数据集的处理。
对所得延时图像系列的仔细分析提供了蛋白质-DNA 相互作用的定量测量(即,结合寿命、位置、化学计量、解离动力学、易位速率、运动蛋白的持续合成能力以及蛋白质和/或 DNA 之间的共定位和相互作用)。与将 DNA 分子直接拴在表面或将共聚焦成像与商业仪器中的力作相结合的其他单分子成像技术相比,DNA 幕布为获得更高通量而的主要权衡是建立初始设置所需的技术 13,14,15,16.然而,一旦建立,在一个流动槽中同时收集数百个分子数据的能力可以显著节省数据收集时间。
与许多其他单分子成像方法一样,制备干净表面的能力在很大程度上决定了用户可靠地组装 DNA 帘的能力。在这种情况下,清洁的表面不仅需要从干净的载玻片开始,而且还需要在进行任何流体连接时不要将微小的气泡引入腔室。首先,关于载玻片的清洁度,上面第 6 节中详述的步骤应该足以作为常规清洁过程。还建议使用过滤后的溶液和缓冲液。其次,在将流通池连接到缓冲液进样系统期间进行即插式连接对于避免引入气泡至关重要。对用于制备缓冲液的双蒸水进行脱气可能会有所帮助。还建议仔细观察任何注射过程,以确保不存在气泡。如果在微小气泡到达中心图案区域之前被引入,可以通过从另一侧的微流体端口注入缓冲液将气泡推出来补救。
在任何基于荧光的实验中,另一个重要的考虑因素是信噪比。观察单个荧光蛋白(即 GFP)的关键也在于干净的表面。长时间使用后,蛋白质聚集体通常会积聚在铬屏障和基座上。因此,如果在添加荧光蛋白之前在屏障处观察到强烈的荧光信号,建议将载玻片浸没在 Piranha 溶液中 10 分钟。该时间段保持较短,以避免侵蚀 chrome 功能。食人鱼清洗后应进行定期清洁程序。
由于对设备和设施的要求以及材料科学方面的现有专业知识,使用电子束光刻技术进行纳米制造可能具有挑战性。作为替代方案,已经开发了基于 UV 光刻的方法来制造 DNA 幕的铬特征20。该方案还提供了用于 12 μm 双栓系 lambda DNA 的基本模式(参见补充信息)。这种模式设计的几个方面可以进行调整,以适应各种实验需求。这些因素包括屏障和基座之间的距离、屏障中相邻孔之间的间距以控制 DNA 密度,以及基座的形状和大小,以优化双栓系效率并最大限度地减少双栓系长度的不确定性。其他实际挑战可能包括蛋白质荧光标记策略的选择、最大限度地减少手动数据分析中的偏差以及开发脚本以简化数据处理。
虽然此处描述的方案涉及 dsDNA 底物,但基于 ssDNA 的 DNA 窗帘也已被广泛使用 13,14,15,16。ssDNA 底物的制备利用 M13 ssDNA 质粒和生物素化引物的滚动环复制13。ssDNA 幕的组装还需要使用尿素和 ssDNA 结合蛋白 RPA 来消除二级结构并延伸 ssDNA 底物13。也可以将 DNA 幕扩展到使用 RNA 或 DNA/RNA 杂交体来研究 RNA 相互作用的蛋白质。就目前而言,DNA 幕平台可以从组装过程的自动化中受益,如第 5 节所述,其中大部分是重复注射一组标准缓冲液。这可能会提高同时进行多个实验的效率。此外,一套统一的、可能基于机器学习的分析脚本将进一步加快数据处理速度,同时将偏差降至最低。
披露声明
作者没有什么可披露的。
致谢
作者感谢 Greene 实验室的前任和现任成员开发、优化和讨论 DNA 窗帘方案,并对本手稿发表评论。这项工作得到了美国国家科学基金会 (MCB1154511 给 ECG) 和美国国立卫生研究院 (R01CA217973 给 ECG 的资助)。
材料
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
参考文献
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。