Method Article
DNA-Vorhänge geben Aufschluss über komplexe molekulare Systeme während der homologen Rekombination
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
DNA-Vorhänge stellen eine neuartige Methode dar, um Hunderte oder sogar Tausende von DNA-bindenden Proteinen in Echtzeit zu visualisieren, während sie mit DNA-Molekülen interagieren, die auf der Oberfläche einer mikrofluidischen Probenkammer ausgerichtet sind.
Zusammenfassung
Die homologe Rekombination (HR) ist wichtig für die Reparatur von doppelsträngigen DNA-Brüchen (DSBs) und blockierten Replikationsgabeln in allen Organismen. Defekte in der HR sind eng mit einem Verlust der Genomintegrität und einer onkogenen Transformation in menschlichen Zellen verbunden. HR beinhaltet koordinierte Aktionen einer komplexen Reihe von Proteinen, von denen viele noch wenig verstanden sind. Der Schlüsselaspekt der hier beschriebenen Forschung ist eine Technologie namens "DNA-Vorhänge", eine Technik, die es ermöglicht, ausgerichtete DNA-Moleküle auf der Oberfläche einer mikrofluidischen Probenkammer zu assemblieren. Anschließend können sie mittels Totalreflexionsfluoreszenzmikroskopie (TIRFM) sichtbar gemacht werden. DNA-Vorhänge wurden von unserem Labor entwickelt und ermöglichen den direkten Zugang zu raumzeitlichen Informationen auf Millisekunden-Zeitskalen und mit einer Auflösung im Nanometerbereich, die mit anderen Methoden nicht einfach aufgedeckt werden können. Ein großer Vorteil von DNA-Vorhängen besteht darin, dass sie die Sammlung statistisch relevanter Daten aus Einzelmolekülexperimenten vereinfachen. Diese Forschung liefert weiterhin neue Erkenntnisse darüber, wie Zellen die Integrität des Genoms regulieren und erhalten.
Einleitung
Die Aufrechterhaltung der Integrität des Genoms ist entscheidend für das reibungslose Funktionieren aller lebenden Zellen1. Defekte in der Genomintegrität können zu schweren Gesundheitszuständen führen, einschließlich verschiedener Krebsarten und altersbedingter degenerativer Erkrankungen2. Die homologe Rekombination (HR) nutzt die Template-abhängige DNA-Synthese, um DNA-Doppelstrangbrüche (DSB), einzelsträngige DNA-Lücken (ssDNA) und DNA-Vernetzungen zwischen den Strängenzu reparieren 3. HR ist auch für die Wiederherstellung von blockierten und kollabierten Replikationsgabeln erforderlich 3,4. Darüber hinaus ist die HR essentiell für die genaue Chromosomensegregation während der Meiose 5,6.
HR beinhaltet die koordinierten Aktionen einer komplexen Reihe von Proteinen, von denen viele noch wenig verstanden sind1. Beispiele hierfür sind unter anderem das Replikationsprotein A (RPA), Rad51 und Rad547. HR-Reaktionen sowohl in prokaryotischen als auch in eukaryotischen Zellen beinhalten ein ssDNA-Zwischenprodukt, das schnell mit ssDNA-bindenden Proteinen (SSB in Prokaryoten und RPA in Eukaryoten) beschichtet wird8. Diese Proteine schützen die ssDNA vor Nukleasen, eliminieren die Sekundärstruktur und fördern die Rekrutierung nachgeschalteter Faktoren 8,9. Rad51 gehört zur hochkonservierten ATP-abhängigen Rad51/RecA-Familie von DNA-Rekombinasen, die in allen lebenden Organismen vorkommen1. Rad51 fördert die DNA-Stranginvasion des homologen dsDNA-Donors. Aufgrund seiner Bedeutung ist Rad51 stark reguliert, und Defekte in diesen regulatorischen Prozessen sind häufig mit einem Verlust der Genomintegrität und der onkogenen Transformation verbunden7. Rad54 ist ein Mitglied der Swi2/Snf2-Familie von dsDNA-Translokasen und Chromatin-Remodelern10,11. Diese Proteine dienen als essentielle Rad51-Regulationsfaktoren. Wichtig ist, dass Rad54 Rad51 aus dem dsDNA-Produkt der Stranginvasion entfernt und auch notwendig ist, um eine Fehlakkumulation von Rad51 auf Chromatin11 zu verhindern. Molekulare Aktivitäten von Proteinen, die an der HR beteiligt sind, sowohl in Hefe- als auch in Bakterienzellen, haben Aufschluss über ihre Funktion in der HR gegeben, aber wie genau ihre Aktivität zur HR beiträgt, ist noch wenig verstanden 12.
DNA-Vorhänge haben sich als einzigartige Plattform erwiesen, die einen direkten Zugang zu molekularen Mechanismen und makromolekularer Dynamik bietet, die sonst unzugänglich bleiben würden13,14. Zur Herstellung von DNA-Vorhängen wird die Oberfläche einer mikrofluidischen Kammer mit einer Lipiddoppelschicht beschichtet, und DNA-Moleküle werden durch eine Biotin-Streptavidin-Bindung an die Doppelschicht gebunden. Die Doppelschicht macht die Oberfläche inert, indem sie natürliche Zellmembranen nachahmt. Die hydrodynamische Kraft richtet die DNA entlang nanofabrizierter Barrieren aus und ermöglicht die Visualisierung von Hunderten von Molekülen in einem einzigen Sichtfeld durch Totalreflexions-Fluoreszenzmikroskopie (TIRFM). Die Barrieren werden durch Elektronenstrahllithographie hergestellt, und Variationen im Barrieredesign ermöglichen eine präzise Kontrolle über die Verteilung und Tethering-Geometrie der DNA. Diese Ansätze sind entweder mit ssDNA oder dsDNA 13,14,15,16,17,18 anwendbar. Sockel können auch nanofabriziert sein (zusammen mit den Barrieren), um zu ermöglichen, dass beide Enden der DNA an die Probenkammeroberfläche gebunden werden können, so dass stationäre Experimente ohne Pufferfluss durchgeführt werden können.
Zeitabhängige Veränderungen in einzelnen Protein-Nukleinsäure-Komplexen werden durch die Inspektion von Echtzeitvideos aufgedeckt und mit Hilfe von Kymographen, die die sich ändernde Position von Proteinen auf der DNA im Laufe der Zeit darstellen, im Druck dargestellt. Ein wichtiger Aspekt des DNA-Curtains-Ansatzes ist, dass er nicht unbedingt a priori Modelle oder Annahmen über molekulare Mechanismen erfordert, da das Verhalten einzelner Reaktionskomponenten in Echtzeit beobachtet werden kann. Dies ermöglicht die direkte Beobachtung molekularer Verhaltensweisen. Hierin beschreibt dieses Protokoll, wie DNA-Vorhänge mit dsDNA-Substraten hergestellt werden können, sowie deren Anwendung zur Untersuchung von Zwischenprodukten in homologer Rekombination.
Protokoll
1. Herstellung der Lipidbrühe
- 1 g 18:1 (Δ9-Cis) PC, 100 mg 18:1 PEG2000 PE und 5 mg 18:1 Biotinylkappe PE in 10 ml Chloroform lösen.
HINWEIS: Sowohl DOPC als auch DPPE sind gut charakterisiert und (zusammen mit der PEG-Modifikation) aufgrund ihrer Fähigkeit ausgewählt, die unspezifische Adsorption an der Oberfläche der Durchflusszelle zu minimieren. Der Mastermix für gelöste Lipide kann aliquotiert und bis zu 12 Monate bei -20 °C gelagert werden. - Verwenden Sie Chloroform, um eine Glasspritze mit organischen Lösungsmitteln zu reinigen, und übertragen Sie dann 200 μl (10 % des gewünschten Endvolumens) des Lipid-Mastermixes in das Glasfläschchen.
- Verdampfen Sie das Chloroform aus dem Glasfläschchen mit einem sehr sanften Strom komprimierten Stickstoffs. Erhöhen Sie den Druck gegen Ende der Verdampfung leicht, um sicherzustellen, dass alle Spuren von Chloroform entfernt werden.
HINWEIS: Kippen und drehen Sie das Fläschchen während des Verdampfungsvorgangs, so dass die Lipide als gleichmäßige Schicht aus weißem Film an der Wand trocknen. - Stellen Sie die Durchstechflasche unverschlossen über Nacht unter Vakuum.
- Geben Sie 2 ml Lipidpuffer (10 mM Tris-HCl [pH 8,0], 100 mM NaCl) in den getrockneten Lipidfilm und verschließen Sie das Fläschchen. Bei Raumtemperatur (RT) mindestens 1 h inkubieren und vortexen, bis es sich aufgelöst hat.
- Übertragen Sie die Lösung in ein 5-ml-Polystyrol-Rundbodenröhrchen und beschallen Sie sie auf Eis mit einem Mikrotip mit den folgenden Parametern: Amplitude = 50, Prozesszeit = 1,5 min, Impuls = 15 s, Auszeit = 2 min. Die Lösung wird nach der Beschallung klar sein (Gesamtenergie = 1.500-2.000 J).
- Die Lösung wird durch einen 0,22 μm PVDF-Spritzenvorsatzfilter filtriert und das Lipidmaterial bis zu 1 Monat bei 4 °C gelagert.
2. Vorbereitung des dsDNA-Substrats
- Erwärmen Sie den λ-DNA-Stamm (500 μg/mL) auf 65 °C.
- Bereiten Sie eine Annealing-/Ligationsmischung vor, die Folgendes enthält: 1,6 pM λ-DNA, 100x molaren Überschuss (160 pM) jedes Oligonukleotids (BioL: 5'Phos-AGG TCG CCG CCC-3Bio und DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) und 60 μL 10x T4-Ligase-Reaktionspuffer (bei einem Gesamtvolumen von 600 μL).
HINWEIS: BioL- und DigR-Oligonukleotide sind biotinyliert (Bio) bzw. mit Digoxigenin (Dig) modifiziert. Die Biotinylierung ermöglicht die Bindung von DNA-Molekülen an biotinylierte Lipide über Streptavidin-Bindungen. Die Dig-Modifikation ermöglicht ein doppeltes Tethering durch Bindung von Anti-Dig-Antikörpern, die auf Sockeln beschichtet sind, an Dig-Einheiten. - Glühen Sie die Oligonukleotide an die λ-DNA-cos-Enden, indem Sie die Reaktion 10 Minuten lang bei 65 °C inkubieren und dann langsam auf RT abkühlen lassen.
- Fügen Sie 5 μl T4-DNA-Ligase hinzu und inkubieren Sie über Nacht bei RT.
HINWEIS: Glüh- und Ligaturschritte können auch in einem Thermocycler durchgeführt werden. - Die λ-DNA wird durch Zugabe von 300 μl PEG-8000-Lösung (30 % w/v PEG-8000 und 10 mM MgCl2) gefällt. Bei 4 °C mindestens 1 h inkubieren.
- Zentrifugieren Sie bei 14.000 x g für 5 min bei RT. Entfernen Sie vorsichtig den Überstand, ohne das Pellet zu stören. Optional können Sie das Pellet 1x mit 500 μL gekühltem 70%igem Ethanol waschen.
- Resuspendieren Sie das Pellet in 40 μl TE-Puffer (10 mM Tris-HCl, 1 mM EDTA [pH 8,0]).
3. Nanofabrikation von Chrommustern
- Bohren Sie zwei Löcher in der Mitte eines 1" x 3" großen Quarz-Objektträgers (~3 cm Abstand) mit einem diamantbeschichteten 1,4-mm-Bohrer auf einer Tischbohrmaschine (siehe Abbildung 1 für die Geometrie der Bohrposition).
- Reinigen Sie die gebohrten Objektträger, indem Sie sie in 200 ml Piranha-Lösung (75 % Schwefelsäure [97 %] und 25 % Wasserstoffperoxid) tauchen. 30 min inkubieren.
ACHTUNG: Es ist äußerste Vorsicht geboten, um jeglichen Kontakt mit exponierter Haut zu vermeiden. Bereiten Sie die Lösung vor, indem Sie sehr langsam kaltes Wasserstoffperoxid in Schwefelsäure mischen. Entsorgen Sie Piranha-Abfälle ordnungsgemäß in einem dafür vorgesehenen Abfallbehälter. - Spülen Sie die Objektträger, indem Sie in 200 ml doppelt destilliertes Wasser eintauchen. Wiederholen Sie dies 2x, um alle verbleibenden Piranhas zu eliminieren. Trocknen Sie die Objektträger mit einem sanften Strahl komprimierten Stickstoffs.
- Mit einem Schleudercoater (4.000 U/min für 45 s, Rampenrate von 300 U/min/s) beschichten Sie die sauberen Objektträger wie folgt mit Fotolack und leitfähigem Polymer:
- Objektträger mit einer Schicht aus 3 % (w/v) Polymethylmethacrylat (PMMA; 24,3 K MW; in Anisol gelöst) beschichten.
- Die Objektträger werden mit einer Schicht aus 1,5 % (w/v) Polymethylmethacrylat (PMMA; 495 K MW; in Anisol gelöst) beschichtet.
- Beschichtung von Objektträgern mit einer Schicht aus Antistatikum für die Elektronenstrahllithographie.
- Schreiben Sie benutzerdefinierte Muster für Barrieren und Sockel auf die beschichteten Objektträger mithilfe der Elektronenstrahllithographie mit einem Rasterelektronenmikroskop, das mit einer Systemsoftware zur Generierung von Nanomustern ausgestattet ist.
- Entwickeln Sie die Objektträger, indem Sie zunächst das Antistatikum für die Elektronenstrahllithographie mit doppelt destilliertem Wasser abspülen. Geben Sie sie dann in 50-ml-Röhrchen mit der Entwicklungslösung (75 % Methylisobutylketon [MIBK] und 25 % Isopropanol; bei -20 °C kühl stellen) und beschallen Sie sie 1 Minute lang in einem Eisbad bei geringer Leistung.
- Die Entwicklungslösung mit Isopropanol abspülen und mit einem sanften Strom komprimierten Stickstoffs trocknen.
- Mit einem Elektronenstrahlverdampfer wird eine 250 Å dicke Chromschicht mit 0,5 Å/s auf die gemusterte Oberfläche aufgebracht.
- Heben Sie das Chrom und das restliche PMMA mit Aceton aus einer Spritzflasche ab und beschallen Sie die Objektträger anschließend 10 Minuten lang in einem Acetonbad. Nochmals mit Aceton aus einer Spritzflasche waschen.
- Um Ablagerungen durch das restliche Aceton zu vermeiden, spülen Sie die Objektträger mit Isopropylalkohol ab und trocknen Sie sie mit einem sanften Strahl komprimierten Stickstoffs.
4. Montage der Durchflusszelle
- Legen Sie eine rechteckige Papierschablone von 35 mm x 5 mm (die sowohl das Chrommuster als auch die vorgebohrten Einlass-/Auslasslöcher abdeckt) mit der gemusterten Seite nach oben über den Quarzschieber ( Abbildung 1A).
- Kleben Sie ein Stück doppelseitiges Klebeband (1 mm dick) über den Quarzobjektträger, decken Sie die Papierschablone ab und schneiden Sie dann mit einer Rasierklinge die rechteckige Papierschablone aus ( Abbildung 1A).
- Legen Sie ein Deckglas über das doppelseitige Klebeband und üben Sie leichten Druck aus, um das Deckglas mit dem Klebeband abzudichten ( Abbildung 1A). Der Raum zwischen Deckglas und Objektträger, der mit doppelseitigem Klebeband verschlossen ist, bildet die mikrofluidische Kammer.
HINWEIS: Es ist wichtig, dass die Dichtung zwischen Deckglas und Schieber ausreichend sicher ist, um ein Auslaufen während des Versuchs zu verhindern. - Legen Sie das Deckglas-Sandwich zwischen die Objektträger und befestigen Sie Bindeklammern an allen vier Seiten, um den Druck gleichmäßig zu verteilen. Stellen Sie die Baugruppe für 60 min bei 135 °C in einen Vakuumofen, um das doppelseitige Klebeband zu schmelzen und die Kammer abzudichten.
- Nehmen Sie es aus dem Ofen und lösen Sie dann die Bindeklammern und Glasobjektträger.
- Befestigen Sie mikrofluidische Anschlüsse mit einer Heißklebepistole an den vorgebohrten Einlass-/Auslasslöchern, indem Sie die geklebte Baugruppe 1 Minute lang auf einen Heizblock (220 °C) legen, ihn abnehmen und vorsichtig sanften Druck ausüben, um die mikrofluidischen Anschlüsse richtig abzudichten. Lassen Sie den Kleber vollständig aushärten, bevor Sie fortfahren.
HINWEIS: Zusammengebaute Durchflusszellen können in 50-ml-Röhrchen platziert und unter Vakuum gelagert werden.
5. Montage des dsDNA-Vorhangs
- Befestigen Sie den Einlass-/Auslassschlauch an den mikrofluidischen Anschlüssen der Durchflusszelle und waschen Sie die Kammer mit 20 ml doppelt destilliertem Wasser mit zwei 10-ml-Spritzen.
- Wenden Sie mit einer 10-ml-Spritze, die mit doppelt destilliertem Wasser gefüllt ist und mit beiden Seiten der Durchflusszelle verbunden ist, Kraft an, um das Wasser zwischen der Einlass- und der Auslassspritze zu schieben/ziehen. Dieser Schritt entfernt alle Luftblasen aus der Durchflusszelle.
- Waschen Sie die Kammer mit 3 mL Lipidpuffer (10 mM Tris-HCl [pH 8,0], 100 mM NaCl).
HINWEIS: Stellen Sie sicher, dass alle Verbindungen zu den Durchflusszellen von Tropfen zu Tropfen angeordnet sind, um das Einbringen von Luftblasen in die Durchflusszelle zu vermeiden. - Mischen Sie 40 μl Liposomenlösung mit 1 ml Lipidpuffer. Injizieren Sie 300 μl der Lipidlösung in die Durchflusszelle und inkubieren Sie sie 5 Minuten lang. Wiederholen Sie die Injektion und Inkubation 2x.
HINWEIS: Wechseln Sie die Einlass-/Auslassseite für die Injektion jedes Mal, wenn eine Spritze an die Durchflusszelle angeschlossen wird. - Waschen Sie die Kammer mit 3 ml Lipidpuffer und inkubieren Sie sie 15 Minuten lang.
HINWEIS: Diese Inkubationszeit kann je nach Zeitplan der Experimente zwischen 5 Minuten und 2 Stunden variieren, ohne dass es zu nennenswerten Unterschieden bei der endgültigen Montage des Vorhangs kommt. - Mischen Sie 40 μl 1 mg/ml Anti-Digoxigenin-Stammlösung mit 200 μl Lipidpuffer. Injizieren Sie 100 μl der Lösung und inkubieren Sie sie 10 Minuten lang. Wiederholen Sie die Injektion und Inkubation 1x, bevor Sie sie mit 3 ml Lipidpuffer waschen.
HINWEIS: Dieser Schritt ist nur für doppelt angebundene dsDNA-Vorhänge erforderlich. Überspringen Sie diesen Schritt für einfach angebundene dsDNA-Vorhänge. - Waschen Sie die Kammer mit 3 mL BSA-Puffer (40 mM Tris-HCl [pH 8,0], 2 mM MgCl2, 1 mM DTT und 0,2 mg/mL BSA).
- Mischen Sie 10 μl 1 mg/ml Streptavidin-Stammlösung mit 990 μl BSA-Puffer. Injizieren Sie in zwei separaten 400-μl-Injektionen mit einer 10-minütigen Inkubation nach jeder Injektion.
- Waschen Sie die Kammer mit 3 ml BSA-Puffer, um überschüssiges Streptavidin zu entfernen.
- Verdünnen Sie 25 ng dsDNA-Substrat in 1 ml BSA-Puffer. Injizieren Sie die DNA-Lösung langsam 200 μl auf einmal, gefolgt von einer 5-minütigen Inkubation nach jeder Injektion. Die benötigte Menge an DNA kann für bestimmte Arten von Experimenten optimiert werden.
- Montieren Sie die Durchflusszelle auf dem Mikroskoptisch und stellen Sie den Fokus nach Bedarf ein.
- Schließen Sie die Ein- und Ausgangsanschlüsse an ein Probeninjektionssystem an, das aus einer Spritzenpumpe und einem Hochdruck-Schaltventil besteht.
- Binden Sie die DNA doppelt an die Sockel (12 μm Haltegurtlänge), indem Sie die Kammer 5 Minuten lang kontinuierlich mit BSA-Puffer bei 0,8 mL/min waschen.
HINWEIS: Es kann erforderlich sein, die Durchflussraten und -zeiten anzupassen, um sie für unterschiedliche Doppelgurtlängen zu optimieren. Dieser Schritt ist nur für doppelt angebundene DNA-Vorhänge erforderlich. Überspringen Sie diesen Schritt für einfach angebundene dsDNA-Vorhänge. - Jetzt sind die Vorhänge bereit für Experimente.
6. Recycling von Durchflusszellen
- Tauchen Sie die Objektträger mindestens 30 Minuten lang in Ethanol, bevor Sie mit einer Rasierklinge vorsichtig die mikrofluidischen Öffnungen und das Deckglas entfernen. Entsorgen Sie das Deckglas ordnungsgemäß und entfernen Sie alle Klebstoffreste von den mikrofluidischen Anschlüssen, damit sie wiederverwendet werden können. Schrubben Sie alle Klebstoffreste auf der Rutsche.
HINWEIS: Achten Sie darauf, den gemusterten Bereich in der Mitte der Folie nicht zu zerkratzen. - Spülen Sie die Objektträger gründlich mit doppelt destilliertem Wasser ab und tauchen Sie sie über Nacht unter Rühren in 2% alkalisches flüssiges Quarzreinigungskonzentrat.
- Spülen Sie die Objektträger gründlich mit doppelt destilliertem Wasser ab und tauchen Sie sie 1 h lang in eine 2%ige alkalische flüssige Quarzreinigungskonzentratlösung unter Rühren bei 80 °C, gefolgt von einer Beschallung für 15 Minuten bei 45 °C.
- Spülen Sie die Objektträger gründlich mit doppelt destilliertem Wasser ab und tauchen Sie sie unter Rühren 30-60 min lang in 1 M NaOH.
- Spülen Sie die Objektträger gründlich mit doppelt destilliertem Wasser ab, tauchen Sie sie dann in Isopropylalkohol und beschallen Sie sie 15 Minuten lang bei 45 °C.
- Spülen Sie die Objektträger gründlich mit Ethanol ab und tauchen Sie sie 1 h lang unter Rühren in Ethanol.
- Legen Sie die Objektträger für 1 h in einen Vakuumofen, um bei 135 °C zu trocknen. Die Objektträger sind nun bereit, wieder zu Durchflusszellen zusammengebaut zu werden.
Ergebnisse
Oben beschrieben sind die Präparation, Assemblierung und Bildgebung von einfach- und doppelt angebundenen dsDNA-Vorhängen im Rahmen der Untersuchung von Protein-DNA-Wechselwirkungen in DNA-Reparatur-Zwischenprodukten. Abbildung 1A zeigt alle Komponenten in einer Durchflusszelle, die in der Reihenfolge geschichtet sind, in der sie zusammengebaut sind. Abbildung 1B zeigt das Schema eines einfach oder doppelt angebundenen DNA-Vorhangs. Eine Lipiddoppelschicht wird verwendet, um die Oberfläche der Durchflusszelle zu passivieren. Der DNA-Vorhang besteht aus einer parallelen Anordnung von DNA-Molekülen, die an einem Ende an das Lipid gebunden und an der Chrombarriere ausgerichtet sind, senkrecht zur Strömungsrichtung ausgerichtet. Bei doppelt angebundenen DNA-Vorhängen wird das andere Ende der DNA durch Digoxigenin- und Anti-Digoxigenin-Wechselwirkungen an den Sockel gebunden, so dass die Bildgebung ohne Fluss durchgeführt werden kann, während die DNA verlängert bleibt. dsDNA kann mit dem Fluoreszenzfarbstoff YoYo1 gefärbt und mittels TIRF-Mikroskopie sichtbar gemacht werden. Abbildung 2A zeigt ein repräsentatives Weitfeldbild eines YoYo1-gefärbten doppelt angebundenen DNA-Vorhangs.
Zeitraffer-Bildserien, die aus DNA-Vorhang-Experimenten gesammelt wurden, werden in der Regel analysiert, indem zunächst ein Kymograph erzeugt wird, der die Position entlang des DNA-Moleküls auf der vertikalen Achse im Vergleich zur Zeit auf der horizontalen Achse für jedes DNA-Molekül von Interesse in ImageJ darstellt. Abbildung 2B zeigt repräsentative Kymographien, die die Resektion von YoYo1-gefärbter einfach angebundener λ-DNA (grün) durch die Hefe-Resektionsmaschinen GFP-Sgs1 (nicht sichtbar), Top3-Rmi1 und Dna2 in Gegenwart des einzelsträngigen DNA-bindenden Proteins RPA-mCherry (magenta) und ATP zeigen. Das YoYo1-Signal geht mit der Zeit verloren, wenn dsDNA vom freien Ende durch Sgs1-DNA2 reseziert wird. Gleichzeitig kolokalisiert das mCherry-Signal mit den DNA-Enden, an denen die ssDNA als Ergebnis der Resektion erzeugt wird. Biophysikalisch relevante Eigenschaften der Resektion, wie Geschwindigkeit und Prozessivität, können durch Quantifizierung der Steigungen der YoYo1-Signalverlusttrajektorien extrahiert werden. Abbildung 2C,D zeigt die Verteilung der Geschwindigkeiten bzw. der Prozessivität der Resektion durch Sgs1-Dna2.

Abbildung 1: Schematische Darstellung des Zusammenbaus von Durchflusszellen und DNA-Vorhängen. (A) Schrittweise Illustration für den Aufbau von Durchflusszellen. Doppelseitiges Klebeband wird auf den Quarzobjektträger gelegt, und eine Papierschablone wird verwendet, um einen rechteckigen Kanal aus der Mitte auszuschneiden, der mit einem Deckglas oben verschlossen wird, um eine Kammer zu bilden. (B) Schematische Darstellungen von vollständig zusammengesetzten DNA-Vorhängen. Das mittlere Feld zeigt eine einfach angebundene Durchflusszelle, während das untere Feld eine doppelt angebundene Durchflusszelle darstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
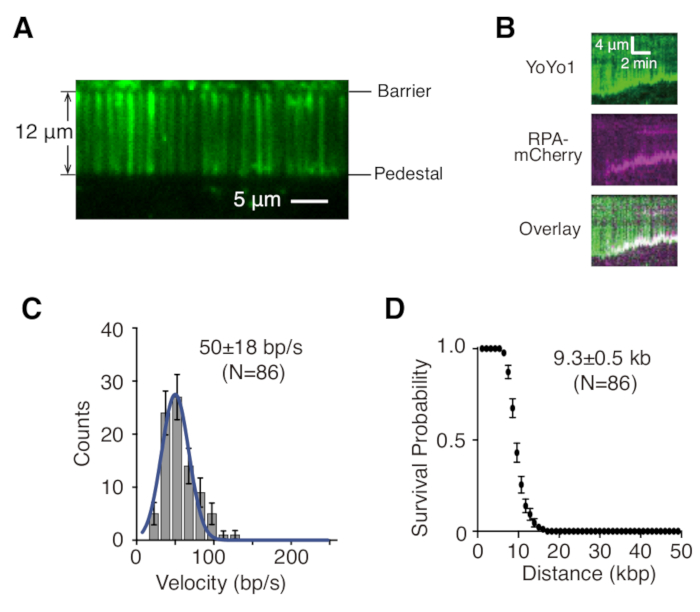
Abbildung 2: Visualisierung und Analyse eines Einzelmolekül-DNA-Vorhangs. (A) Repräsentatives Weitfeldbild eines doppelt angebundenen λ-DNA-Vorhangs, der mit YoYo1 gefärbt wurde (grün). (B) Repräsentative Kymographen, die die Resektion von YoYo1-gefärbter dsDNA (grün) in Gegenwart von vorgebundenem GFP-Sgs1 (nicht sichtbar) zeigen, wenn sie mit Dna2 gejagt wurde, in Gegenwart von RPA-mCherry (magenta) und 2 mM ATP. Geschwindigkeitsverteilung (C) und Prozessivitätsdiagramm (D) der dsDNA-Resektion durch Sgs1-Dna2, erhalten durch Quantifizierung der Rate und des Ausmaßes des YoYo1-Signalverlusts im Laufe der Zeit. Die Panels (B), (C) und (D) wurden von Xue et al.19 übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
DNA-Vorhänge haben sich als vielseitige Plattform für die Untersuchung verschiedener DNA-Reparaturprozesse erwiesen 13,14,15,16. DNA-Moleküle, die während des gesamten Experiments in der Nähe der Oberfläche ausgedehnt bleiben, entweder durch kontinuierlichen Pufferfluss oder durch doppelte Bindung an den Sockel, ermöglichen die TIRFM-Bildgebung von Protein-DNA-Wechselwirkungen auf Reparaturzwischenprodukten auf Einzelmolekülebene. Die Methode verbessert den experimentellen Durchsatz und die Vorhersagbarkeit, da Hunderte von DNA-Molekülen geordnet an nanofabrizierten Barrieren ausgerichtet sind und nicht unspezifisch an der Oberfläche befestigt sind, was der Verarbeitung großer Datensätze weiter zugute kommt.
Die sorgfältige Analyse der resultierenden Zeitraffer-Bildserien ermöglicht quantitative Messungen der Protein-DNA-Wechselwirkungen (d. h. Bindungslebensdauer, Positionen, Stöchiometrie, Dissoziationskinetik, Translokationsraten, Prozessivitäten von Motorproteinen sowie Kolokalisationen und Wechselwirkungen zwischen Proteinen und/oder DNA). Im Vergleich zu anderen Einzelmolekül-Bildgebungsverfahren, die DNA-Moleküle entweder direkt an die Oberfläche binden oder konfokale Bildgebung mit Kraftmanipulation in kommerziellen Instrumenten kombinieren, besteht der Hauptkompromiss von DNA-Vorhängen für einen höheren Durchsatz darin, die für die Ersteinrichtung erforderlichen Techniken zu etablieren 13,14,15,16. Einmal etabliert, führt die Möglichkeit, Daten von Hunderten von Molekülen gleichzeitig in einer Durchflusszelle zu sammeln, jedoch zu erheblichen Zeiteinsparungen bei der Datenerfassung.
Wie bei vielen anderen Einzelmolekül-Bildgebungsverfahren hängt die Fähigkeit, eine saubere Oberfläche zu präparieren, weitgehend über die Fähigkeit des Benutzers ab, DNA-Vorhänge zuverlässig zusammenzusetzen. In diesem Fall müssen saubere Oberflächen nicht nur mit einem sauberen Objektträger beginnen, sondern auch darauf achten, dass keine mikroskopisch kleinen Luftblasen in die Kammer gelangen, während eine der fluidischen Verbindungen hergestellt wird. Erstens, was die Sauberkeit der Rutschen betrifft, sollten die oben in Abschnitt 6 beschriebenen Schritte als routinemäßiger Reinigungsprozess ausreichen. Die Verwendung von filtrierten Lösungen und Puffern wird ebenfalls empfohlen. Zweitens ist es wichtig, während der Befestigung der Durchflusszelle an das Pufferinjektionssystem Drop-to-Drop-Verbindungen herzustellen, um das Einbringen von Blasen zu vermeiden. Es kann hilfreich sein, das doppeltdestillierte Wasser, das zur Herstellung von Puffern verwendet wird, zu entgasen. Es wird auch empfohlen, den Injektionsprozess sorgfältig zu beobachten, um sicherzustellen, dass keine Blasen vorhanden sind. Sollte eine mikroskopisch kleine Blase eingeführt werden, bevor sie den mittleren gemusterten Bereich erreicht, kann dies durch Injektion von Puffer aus dem mikrofluidischen Anschluss auf der gegenüberliegenden Seite behoben werden, um die Blase wieder herauszudrücken.
Ein weiterer wichtiger Punkt bei allen fluoreszenzbasierten Experimenten ist das Signal-Rausch-Verhältnis. Ein Schlüssel zur Beobachtung einzelner fluoreszierender Proteine (z. B. GFP) befindet sich ebenfalls in sauberen Oberflächen. Nach längerer Nutzung sammeln sich Proteinaggregate typischerweise an den Chrombarrieren und Sockeln an. Wenn daher vor der Zugabe von fluoreszierenden Proteinen ein starkes Fluoreszenzsignal an den Barrieren beobachtet wird, wird empfohlen, die Objektträger mit einem 10-minütigen Eintauchen in Piranha-Lösung zu behandeln. Der Zeitraum ist kurz gehalten, um eine Erosion der Chromfunktionen zu vermeiden. Nach der Piranha-Wäsche sollte die regelmäßige Reinigung durchgeführt werden.
Die Nanofabrikation mittels Elektronenstrahllithographie kann aufgrund der Anforderungen an die Ausrüstung und des vorhandenen Know-hows in den Materialwissenschaften eine Herausforderung darstellen. Als Alternative wurde ein auf UV-Lithographie basierendes Verfahren zur Herstellung von Chrommerkmalen für DNA-Vorhänge entwickelt20. Ebenfalls mit diesem Protokoll geliefert wird ein Grundmuster für das Double-Tethering von Lambda-DNA bei 12 μm (siehe ergänzende Informationen). Verschiedene Aspekte des Designs dieses Musters können angepasst werden, um verschiedenen experimentellen Anforderungen gerecht zu werden. Dazu gehören der Abstand zwischen Barrieren und Sockeln, der Abstand zwischen benachbarten Vertiefungen in der Barriere zur Kontrolle der DNA-Dichte sowie die Form und Größe der Sockel, um die Effizienz der doppelten Bindung zu optimieren und die Unsicherheit bei der doppelten Anbindungslänge zu minimieren. Zu den weiteren praktischen Herausforderungen gehören die Wahl von Fluoreszenzmarkierungsstrategien für Proteine, die Minimierung von Verzerrungen bei der manuellen Datenanalyse und die Entwicklung von Skripten für eine optimierte Datenverarbeitung.
Während das hier beschriebene Protokoll dsDNA-Substrate umfasst, wurden DNA-Vorhänge auf Basis von ssDNA auch ausgiebig verwendet 13,14,15,16. Bei der Herstellung von ssDNA-Substraten wird die Rollkreisreplikation des M13-ssDNA-Plasmids mit biotinylierten Primern13 verwendet. Der Assemblierung des ssDNA-Vorhangs erfordert auch die Verwendung von Harnstoff und ssDNA-bindendem Protein RPA, um die Sekundärstruktur zu eliminieren und das ssDNA-Substratzu verlängern 13. Es könnte auch möglich sein, DNA-Vorhänge zu erweitern, um RNA oder DNA/RNA-Hybride für die Untersuchung von RNA-interagierenden Proteinen zu verwenden. Nach derzeitigem Stand kann die DNA-Curtain-Plattform von der Automatisierung des Montageprozesses profitieren, wie in Abschnitt 5 beschrieben, wobei es sich bei den meisten um wiederholte Injektionen eines Standardsatzes von Puffern handelt. Dies kann zu einer verbesserten Effizienz bei der gleichzeitigen Durchführung mehrerer Experimente führen. Darüber hinaus würde eine einheitliche und potenziell auf maschinellem Lernen basierende Suite von Analyseskripten die Datenverarbeitung mit minimaler Verzerrung weiter beschleunigen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren danken den ehemaligen und aktuellen Mitgliedern des Greene-Labors für die Entwicklung, Optimierung und Diskussion des DNA-Vorhangprotokolls sowie für die Kommentierung dieses Manuskripts. Diese Arbeit wurde durch Zuschüsse der National Science Foundation (MCB1154511 to E.C.G.) und der National Institutes of Health (R01CA217973 to E.C.G.) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
Referenzen
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410 (2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten