Method Article
Medições de batimentos cardíacos sem anestesia em peixe-zebra em movimento livre
Neste Artigo
Resumo
O estudo visa desenvolver tecnologia para medições de batimentos cardíacos sem anestesia em peixes-zebra em movimento. Nossa abordagem combina imagens infravermelhas de ondas curtas e rastreamento do coração baseado em aprendizado de máquina. É uma técnica não invasiva, sem rótulos e fácil de usar que se adapta a uma ampla gama de estudos sobre o modelo de peixe-zebra.
Resumo
O peixe-zebra (Danio rerio) é um organismo modelo amplamente utilizado em pesquisas fisiológicas, farmacológicas e toxicológicas devido à sua semelhança genética com humanos e estágio embrionário transparente, o que facilita estudos cardiovasculares não invasivos. No entanto, os métodos atuais de avaliação da frequência cardíaca em peixe-zebra muitas vezes dependem da anestesia para imobilizar o sujeito, introduzindo alterações fisiológicas que comprometem a precisão e a reprodutibilidade dos dados. Este estudo apresenta uma nova técnica sem anestesia para medir os batimentos cardíacos em larvas de peixe-zebra em movimento livre, abordando uma limitação crítica na pesquisa cardiovascular. A abordagem proposta integra imagens infravermelhas de ondas curtas com rastreamento cardíaco baseado em aprendizado de máquina, permitindo o monitoramento preciso e contínuo da atividade cardíaca em espécimes não imobilizados. Uma rede neural convolucional foi treinada para detectar a região do coração e um sinal fotopletismográfico foi extraído das sequências de imagens para determinar a frequência cardíaca. A validação experimental demonstrou a confiabilidade e consistência do método em várias condições de teste. Um dos principais benefícios da metodologia é sua capacidade de preservar o estado fisiológico natural do peixe-zebra, minimizando os artefatos induzidos pelo estresse. Essa técnica não invasiva e sem rótulo oferece vantagens significativas para o estudo da fisiologia cardiovascular, cardiotoxicidade de drogas e toxicologia ambiental, expandindo as aplicações potenciais do peixe-zebra como modelo para pesquisa biomédica.
Introdução
O peixe-zebra (Danio rerio), um pequeno peixe ciprinídeo, tornou-se um organismo modelo essencial devido ao seu pequeno tamanho, alta taxa reprodutiva e facilidade de manipulação genética 1,2,3. A avaliação da freqüência cardíaca em embriões transparentes de peixe-zebra é cada vez mais utilizada em fisiologia, embriologia, toxicologia e outros campos 4,5,6,7,8. Por um lado, essa utilidade se deve ao fato de que o genoma do peixe-zebra inclui genes associados a doenças cardiovasculares humanas9, e o coração Danio rerio compartilha estruturas e vias de sinalização semelhantes com os humanos10,11. Isso torna o peixe-zebra um modelo inestimável para estudar o desenvolvimento e as doenças do coração 11,12,13. Por outro lado, a frequência cardíaca do peixe-zebra é sensível a influências externas, tornando-se um excelente modelo para estudos fisiológicos e toxicológicos, comparando a função cardíaca em peixes tratados e não tratados 7,8,14.
Progressos significativos foram feitos no desenvolvimento de métodos ópticos não invasivos para avaliar a frequência cardíaca em embriões de peixes transparentes15,16. Essas técnicas oferecem a vantagem de uma coleta rápida de dados de grandes tamanhos de amostra. Consequentemente, abordagens totalmente automatizadas para avaliação da frequência cardíaca em embriões de peixes foram desenvolvidas 4,5,6,17.
No entanto, certas limitações atualmente restringem o uso dessas técnicas ao período de 3-4 dpf. A primeira limitação é a perda de transparência devido à pigmentação do corpo do peixe. A segunda é o aumento da mobilidade dos embriões ao longo do tempo. Estender o período de desenvolvimento inicial do peixe-zebra durante o qual as abordagens ópticas podem ser usadas aumentaria sua utilidade, permitindo projetos experimentais de longo prazo para estudar cardiomiopatia, defeitos cardíacos congênitos e vários impactos no sistema cardiovascular, incluindo o rastreamento da dinâmica dos efeitos ao longo do tempo. Nosso grupo recentemente abordou a questão da perda de transparência empregando imagens na faixa de infravermelho de ondas curtas de 900-1700 nm18. Este artigo se concentra em abordar a questão da mobilidade embrionária.
Normalmente, anestésicos como o metanossulfonato de tricaína (MS-222) são usados para imobilizar embriões e larvas de peixes nadando livremente antes da imagem 14,19,20. No entanto, o MS-222 reduz significativamente a freqüência cardíaca21,22, assim como outros anestésicos23. Torna-se um desafio discernir se as alterações observadas na função cardíaca são devidas ao tratamento experimental, ao anestésico ou a uma interação entre os dois. Outra maneira de estender o período de baixa mobilidade do embrião é reduzindo a temperatura durante o desenvolvimento inicial8. No entanto, essa abordagem nem sempre é compatível com os objetivos da pesquisa e apenas estende minimamente o período de inscrição.
Neste estudo, apresentamos um novo método para abordar a mobilidade embrionária durante o registro da frequência cardíaca. Treinamos uma rede neural convolucional para identificar a região de interesse do coração em registros de embriões de peixe-zebra nadando livremente. A variação periódica na intensidade do pixel dentro desta área é utilizada para derivar o fotopletismograma (PPG), que é posteriormente usado para calcular a frequência cardíaca. O aplicativo de desktop desenvolvido, AutoHR, utilizou treinamento de rede neural e processamento de pilha de imagens, garantindo facilidade de uso e reprodutibilidade do protocolo.
Protocolo
Os peixes-zebra foram criados e criados de acordo com os protocolos ZFIN estabelecidos24. Todos os procedimentos foram aprovados pelo Comitê de Bioética do Centro Científico e Tecnológico de Instrumentação Única da Academia Russa de Ciências (STC UI RAS), protocolo #3/24, datado de 21/08/2024, e seguem as diretrizes de cuidados com o peixe-zebra do STC UI RAS. Manuais para versões individuais estão disponíveis mediante solicitação.
1. Preparação do equipamento para medição
- Seleção de equipamentos
- Escolha uma fonte de luz: Escolha uma fonte de luz LED que emita na faixa do infravermelho próximo, especificamente a partir de 800 nm, com uma potência mínima de 3 W. Este estudo utilizou LED com comprimento de onda central de 940 nm e largura de banda de 40 nm.
- Escolha uma câmera de vídeo: Escolha uma câmera equipada com um sensor sensível à faixa de comprimento de onda emitida pela fonte de luz e tenha uma taxa de quadros de pelo menos 60 fps. Consulte a folha de dados da câmera para determinar a distância entre pixels p, o tamanho do sensor ao longo de seu lado mais curto h e a taxa de quadros. Neste estudo, a câmera tem uma densidade de pixels de p = 5 μm e uma largura do sensor h = 5,12 mm.
- Escolha um tamanho de molde de agarose que seja pelo menos 1,5 vezes maior que o comprimento da larva B. Calcule as ampliações mínimas e máximas permitidas do sistema óptico usando as expressões fornecidas. Certifique-se de que Mmax ≤ Mmin. Se esta condição não for atendida, selecione uma câmera com um h maior e/ou um S menor e/ou menor.
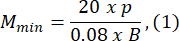
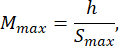
onde Smax é o comprimento do maior molde de agarose do conjunto de moldes. Um conjunto de moldes em vários tamanhos pode ser preparado. Este estudo utilizou S = 10 mm.
NOTA: As equações são derivadas com base na geometria do esquema óptico e nas proporções corporais do peixe-zebra. Explicações detalhadas são fornecidas na seção Discussão. - Escolha uma lente: Este estudo usou um sistema óptico com ampliações variáveis variando de MOSmin = 0,4x a MOSmax = 1,5x, uma distância focal máxima de fmax = 150 mm e uma distância de trabalho de mm.
NOTA: O símbolo x indica o fator de ampliação do sistema óptico.- Escolha um sistema do tipo microscópio de ampliação variável com uma faixa de ampliação (MOSmin - MOSmax) que permaneça dentro da faixa permitida (Mmin - Mmax). Consulte a folha de dados da lente para determinar a distância focal máxima fmax e a distância de trabalho correspondente L para MOSmax.
- Certifique-se de que a condição fmax ≤ Mmax • L seja satisfeita. Se esta condição não for atendida, selecione uma lente com um fmax e/ou L diferente. Certifique-se de que o tipo de montagem da câmera e da lente sejam compatíveis.
- Escolha um objeto de teste (uma régua ou uma grade) com um passo conhecido e um comprimento total de pelo menos . Escolha um objeto de teste na forma de uma régua ou grade com um passo conhecido e um tamanho total de pelo menos Smáx.
- Montagem de um suporte para aquisição de imagens (Figura 1).
- Monte a haste redonda em uma base estável. Monte o sistema de imagem conectando a lente à câmera. Monte o canal de imagem na haste com a janela de entrada voltada para cima.
- Monte o mecanismo de foco na haste. Fixe uma platina com uma superfície de trabalho de vidro com um diâmetro mínimo de 30 mm ao mecanismo de foco.
- Monte o iluminador: Coloque o LED em um substrato de metal para garantir uma dissipação de calor eficiente. Coloque uma lente colimadora plana convexa com um diâmetro de 23 mm e uma distância focal de 30 mm a uma distância de 30 mm do LED.
- Monte o iluminador no palco usando um suporte ajustável de três eixos, posicionando-o acima da superfície de trabalho com a lente voltada para baixo.
- Ajuste o iluminador e o sistema de imagem. Posicione o iluminador coaxialmente com o sistema de imagem. Ligue o iluminador e conecte a câmera ao computador.
- Inicie o aplicativo da câmera e ative o modo de visualização seguindo as instruções no manual da câmera. Coloque o objeto de teste no palco. Ajuste a distância de trabalho e a ampliação do microscópio para garantir que o tamanho se encaixe completamente no campo de visão.
NOTA: O tamanho deve caber inteiramente ao longo do lado curto da moldura para garantir a visualização completa do molde de agarose. - Ajuste a posição da platina em relação ao sistema de imagem para garantir que a imagem esteja focada e a posição do iluminador seja iluminada uniformemente.
- Preparação de molde de agarose
- Misture 20 mL de água destilada com 0,4 g de agarose de baixo ponto de fusão e aqueça a mistura até que a agarose se dissolva completamente. Por exemplo, em um microondas por 20 s a uma potência de 700 W.
- Molde o molde de agarose (Método 1): Despeje a solução aquecida na placa de Petri, garantindo uma altura de solução de 1,5-2 mm. Resfrie a placa de Petri até que a solução de agarose solidifique A uma temperatura de 17 °C, esse processo leva cerca de 5 min. Corte um quadrado com um comprimento lateral usando uma faca de papelaria e remova cuidadosamente a camada de agarose dentro da área delineada.
- Molde o molde de agarose (Método 2): Crie (S) × (S) × 10 mm, formas de plástico. Coloque a forma impressa com o lado quadrado em uma placa de Petri e encha-a com a solução aquecida, garantindo uma altura de solução de 1,5-2 mm. Resfrie a placa de Petri até que a solução de agarose solidifique e remova cuidadosamente a forma da solução solidificada.
NOTA: Os formulários podem ser facilmente impressos usando uma impressora 3D.
2. Aquisição de imagem
- Ajuste as configurações de captura de imagem.
- Conecte a câmera ao computador. Inicie o aplicativo da câmera e ative o modo de visualização. Ligue o iluminador.
NOTA: Se o tamanho do molde de agarose a ser usado for diferente do da etapa 1.3.3, repita a etapa 1.3.3 e remova o objeto de teste do estágio depois disso. - Coloque a larva do peixe-zebra em um molde de agarose por uma pipeta Pasteur. Coloque um molde de agarose no palco. Garanta a iluminação quase completa da cabeça da larva ajustando uma combinação de tempo de exposição e potência do iluminador (Figura 2A-C). O tempo de exposição não deve exceder 1,5 ms.
NOTA: Neste estudo, apresentamos dados para larvas de peixe-zebra do tipo selvagem com idade entre 12 e 21 dias após a fertilização (dpf).
- Conecte a câmera ao computador. Inicie o aplicativo da câmera e ative o modo de visualização. Ligue o iluminador.
- Realizar aquisição de imagem
- Defina a taxa de quadros para pelo menos 60 fps. Defina a profundidade de bits da imagem para 12 bits. Adquira imagens por pelo menos 10 seg. Defina o formato de nomenclatura do quadro como yyyy_mm_dd_hh_ss_mm_mss e o tipo de arquivo deve ser PNG ou TIFF, não BMP.
3. Treinando a rede neural para rotulagem
- Preparação de dados para rotulagem
NOTA: O processo de rotulagem no aplicativo Image Labeler, MATLAB, é descrito.- Reúna pelo menos 256 imagens adquiridas em um diretório separado. Execute o aplicativo. Importe as imagens navegando até Importar > do arquivo De e selecionando todas as imagens do diretório especificado.
- Crie classes para rotulagem de rede. Na guia Rótulos de ROI, clique em Rótulo, insira corpo no campo Nome do rótulo , selecione Rótulo de pixel no menu suspenso para o tipo de rótulo e clique em OK. Na guia Rótulos de ROI, clique em Rótulo, digite olho no campo Nome do rótulo , selecione Rótulo de pixel no menu suspenso para o tipo de rótulo e clique em OK. Na guia Rótulos de ROI, clique em Rótulo, insira peixe no campo Nome do rótulo , selecione Retângulo no menu suspenso para o tipo de rótulo e clique em OK.
- Etiquetagem dos quadros
- Na guia Rótulos de ROI, selecione a classe de anotação criada anteriormente, olho. Contorne um olho da amostra colocando pontos ao longo de seu perímetro usando o botão esquerdo do mouse. Para completar a anotação do olho, clique no ponto inicial do contorno (Figura 3B).
- Contorne outro olho da amostra da mesma maneira descrita na etapa 3.1.2.
- Na guia Rótulos de ROI, selecione a classe de anotação criada, body. Contorne o corpo do espécime, excluindo os olhos, colocando pontos ao longo de seu perímetro usando o botão esquerdo do mouse. Para completar a anotação do olho, clique no Ponto de Partida do Contorno.
- Na guia Rótulos de ROI, selecione a classe de anotação criada anteriormente, fish. Contorne todo o corpo da amostra desenhando uma caixa delimitadora retangular (Figura 4). Para fazer isso, clique em Um canto do retângulo pretendido com o botão esquerdo do mouse e arraste o cursor enquanto mantém o botão pressionado até que o retângulo envolva totalmente o peixe.
- Salve os contornos em um novo diretório chamado images clicando em Exportar > para arquivo na guia LABEL.
- Preparando conjuntos de dados para treinamento de rede neural
- Inicie o MATLAB. Abra o arquivo de script DatasetsPreparation.m (Arquivo Suplementar 1) selecionando Abrir > Abrir.
- Execute o script clicando em Executar. Na janela pop-up, selecione o diretório que contém as imagens delineadas, a pasta PixelLabelData e o arquivo gTruth.mat, que foi exportado na etapa 3.2.5.
NOTA: Após a conclusão da execução do script, duas pastas - RPN Training e CNN Training - serão geradas automaticamente. Eles são necessários para o treinamento subsequente da rede neural. Não modifique o conteúdo dessas pastas.
4. Treinando as redes neurais para detecção cardíaca
NOTA: Esta etapa é realizada uma vez para uma idade específica e condição de imagem. A GPU NVIDIA é fortemente recomendada para treinamento, pois acelera significativamente o processamento.
- Inicie o aplicativo AutoHR. Navegue até a guia Treinamento de Rede de Proposta de Região. Clique em Escolher diretório e selecione a pasta RPN Training criada na etapa 3.3.2. Clique em Iniciar treinamento.
NOTA: Após a conclusão da formação, será gerado um ficheiro de modelo denominado rpn_model.pt no mesmo diretório que o ficheiro AutoHR.exe. Este arquivo é necessário para processamento adicional da pilha de imagens. - Para treinar a Rede Neural Convolucional, navegue até a guia Treinamento de Rede Neural Convolucional. Clique em Escolher diretório e selecione a pasta CNN Training criada na etapa 3.3.2. Clique em Iniciar treinamento.
NOTA: Após a conclusão do treinamento, um arquivo de modelo chamado model.pt será gerado no mesmo diretório que o arquivo AutoHR.exe. Este arquivo é necessário para processamento adicional da pilha de imagens.
5. Quantificação da frequência cardíaca
- Grave uma sequência de imagens conforme descrito na etapa 2. Certifique-se de que as condições de imagem, como ampliação, tempo de exposição e iluminação, sejam idênticas às usadas para treinar a rede neural.
- Inicie o aplicativo AutoHR. Navegue até a guia Processamento. Selecione o diretório que contém modelos de rede neural treinados clicando em Escolher modelos. Clique em Escolher pasta para importar as imagens adquiridas para análise. O caminho de exportação será especificado automaticamente, mas você pode alterá-lo clicando em Escolher caminho de exportação. Clique em Processar para iniciar a análise.
NOTA: Após a conclusão, o valor da frequência cardíaca aparecerá no campo Frequência cardíaca na interface AutoHR. A pasta exportada incluirá os valores de frequência cardíaca (.xlsx), PPG (.xlsx), o primeiro quadro da sequência e as máscaras corporais e oculares do primeiro quadro (.png).
6. Verificação dos resultados do algoritmo
- Efectuar o ensaio com um peixe imóvel conforme descrito a seguir.
- Anestesiar as larvas com solução de MS-222 0,168 mg/L por 1 min. Adquira uma pilha de imagens de uma amostra anestesiada, conforme descrito na etapa 2. Realize uma contagem da frequência cardíaca por meio da observação visual.
- Utilize o software de RH conforme descrito na etapa 5. Repita as etapas para várias amostras. Verifique se os valores de frequência cardíaca obtidos são consistentes para todos os indivíduos dentro da precisão desejada.
- Realize o teste com um indivíduo móvel e teste de provocação, conforme descrito abaixo.
- Adquira uma pilha de imagens de uma amostra não anestesiada, conforme descrito na etapa 2. Realize um teste de provocação com o indivíduo para o qual a sequência foi gravada, usando um estímulo conhecido que afeta a frequência cardíaca. O estudo apresenta resultados para a salinização da água em concentrações de 5 mg/L.
- Adquira uma pilha de imagens da mesma amostra descrita na etapa 2 após o teste de provocação. Determine a frequência cardíaca em ambos os experimentos com o software de RH, conforme descrito na etapa 5. Verifique se as alterações observadas na frequência cardíaca estão alinhadas com os efeitos conhecidos do teste de provocação.
Resultados
A frequência cardíaca do peixe-zebra a 12 dpf foi determinada usando o protocolo descrito acima (Vídeo Suplementar 1). Os vídeos incluem uma sequência de imagens de larvas de peixe-zebra nadando livremente, um fotopletismograma derivado dessas sequências usando o protocolo proposto e a frequência cardíaca correspondente calculada a partir do fotopletismograma.
Os dados rotulados foram divididos aleatoriamente em conjuntos de treinamento, teste e validação em uma proporção de 3:2:1 durante o treinamento. A função de perda foi então implementada de acordo com a seguinte expressão:

onde x é logits de entrada, y é um alvo, tamanho do lote é o número de imagens processadas simultaneamente,

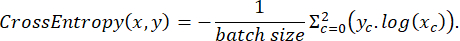
Para avaliar o desempenho do modelo durante o treinamento, foi empregada a função de precisão de predição, definida pela seguinte:


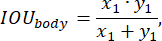
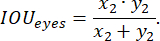
Aplicamos abordagens padrão para o desenvolvimento da função de perda, conforme detalhado, por exemplo, em25. A rede de atenção multiescala com arquitetura de modelo EfficientNet-B0 foi usada como codificador e camada de saída softmax. A rede neural foi treinada em uma amostra de teste por 50 épocas, utilizando uma taxa de aprendizado exponencialmente decrescente que começou em 0,02 e teve um fator de decaimento de 0,99.
A rede neural treinada foi usada para obter a máscara corporal e a máscara ocular (Figura 5С) do indivíduo em cada quadro de uma sequência arbitrária. O centro de massa da máscara ocular foi então calculado e a esqueletização foi aplicada à máscara corporal (Figura 5D). No cordão de peixe P resultante, foi identificado o ponto M, localizado mais próximo do centro de massa da máscara ocular (Figura 5F). A uma distância d do ponto M, igual à distância entre os olhos, o ponto N foi identificado ao longo da corda PPP em direção à cauda. Este ponto corresponde ao centro da área do coração. Essa área foi delimitada por um círculo com raio de r = d x 0,3, centralizado nesse ponto (Figura 5F).
O sinal PPG bruto foi calculado como o número médio de pixels dentro da área do coração em todos os quadros gravados (Figura 5G). Este sinal bruto foi então normalizado para seu valor médio. Posteriormente, o sinal foi filtrado usando um filtro Chebyshev Tipo II de segunda ordem com um design de4ª ordem, atenuação da banda de parada de 25 dB e uma frequência de borda da banda de parada de 12 Hz, consistente com os algoritmos de processamento padrão26,27 (Figura 6A). A frequência cardíaca foi determinada identificando-se a frequência dentro da faixa de 1-4 Hz que corresponde à potência de pico no módulo quadrado da transformada de Fourier do sinal filtrado (Figura 6A).
A equação (1) garante que a imagem do coração tenha aproximadamente 20 pixels x 20 pixels. Supõe-se que a dimensão longitudinal do coração seja responsável por aproximadamente 8% do comprimento total do corpo do peixe-zebra. Quando a imagem do coração é menor, o sinal resultante geralmente contém um componente de ruído significativo que supera o sinal útil, tornando-o inadequado para o cálculo da frequência cardíaca, conforme mostrado na Figura 6B.
Os resultados da aplicação do protocolo dependem significativamente da estabilidade da segmentação cardíaca para cada indivíduo. É crucial realizar experimentos de controle para demonstrar que as etapas 2 a 5 foram concluídas com sucesso e os dados obtidos refletem processos fisiológicos reais, em vez de resultados aleatórios. A validação do protocolo foi realizada seguindo o procedimento detalhado na etapa 6. No teste descrito na etapa 6.2, o sal foi adicionado à água a uma concentração de 5 mg/L como estímulo provocativo. Cinco larvas de peixe-zebra (12 pdf) foram incluídas em cada teste. A temperatura da água em um molde de agarose foi mantida em 23-25 °C. Os resultados indicam que a diferença entre a avaliação visual e o método proposto não ultrapassou 3% no primeiro teste. No segundo teste, a frequência cardíaca medida pelo método aumentou, como esperado (Figura 7).

Figura 1: Fluxo de trabalho esquemático. (A) Esquema e (B) aparência da configuração experimental Clique aqui para ver uma versão maior desta figura.

Figura 2: Imagens de larvas de peixe-zebra. Exemplos de (A) imagens de larvas de peixe-zebra de alta qualidade e (B-D) de baixa qualidade são (B) quadro subexposto, (С) quadro superexposto e (D) quadro desfocado. As imagens foram cortadas para destacar a larva. A barra de escala é de 1 mm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Imagens representativas. Exemplos de (A) imagens iniciais e (B) rotuladas. A barra de escala é de 1 mm. Clique aqui para ver uma versão maior desta figura.
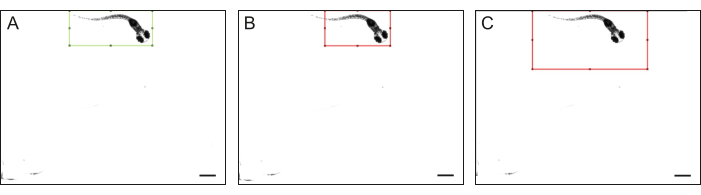
Figura 4: Contornos do corpo do peixe-zebra. Exemplos de (A) contornos de alta qualidade e (B-C) de baixa qualidade de todo o corpo do peixe-zebra: um peixe-zebra não se encaixa inteiramente no retângulo e o tamanho do retângulo é visivelmente maior que o do peixe-zebra. A barra de escala é de 1 mm. Clique aqui para ver uma versão maior desta figura.

Figura 5: Pipeline de processamento de imagem ilustrando os estágios de processamento primário e seus resultados intermediários correspondentes. Clique aqui para ver uma versão maior desta figura.

Figura 6: Quadros mostrando a frequência cardíaca larval. Exemplos de (A) quadros de alta qualidade e (B) de baixa qualidade com a área do coração larval com diâmetros de 20 pixels e 14 pixels, respectivamente, e espectros PPG e Fourier correspondentes de PPG. A barra de escala é de 1 mm. Abreviaturas: PPG = fotopletismografia. Clique aqui para ver uma versão maior desta figura.
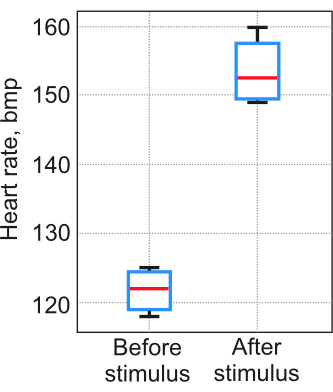
Figura 7: Medições da frequência cardíaca. As medidas foram obtidas pelo método proposto em um teste com estímulo provocador. Número da amostra. A caixa abrange os percentis 25e 75, a linha horizontal é a mediana, os bigodes pretos indicam os pontos de dados mais extremos não considerados discrepantes Clique aqui para ver uma versão maior desta figura.
Vídeo Suplementar 1: Ilustração do algoritmo de rede neural proposto para detectar e calcular a frequência cardíaca. O vídeo apresenta larvas de peixe-zebra em movimento livre, um fotopletismograma derivado pelo protocolo proposto e calculado a partir da frequência cardíaca das larvas do fotopletismograma Clique aqui para baixar este arquivo.
Arquivo Suplementar 1: Função MATLAB que executa o aumento da imagem. Clique aqui para baixar este arquivo.
Discussão
Neste estudo, apresentamos um protocolo experimental para medir os batimentos cardíacos de larvas de peixe-zebra nadando livremente. Avaliamos essa abordagem por meio de vários experimentos, demonstrando sua eficácia. Os principais componentes do método proposto incluem soluções de hardware e software. Primeiramente, utilizamos iluminação infravermelha para a imagem, que, como demonstrado anteriormente, evita problemas relacionados à pigmentação e aumenta a transparência do tecido, facilitando a determinação precisa dos batimentos cardíacos18. Em segundo lugar, propomos imagens dos peixes por baixo. Quando anestesiadas, as larvas do peixe-zebra geralmente ficam de lado devido à posição da bexiga natatória, resultando na diferença de orientação entre as configurações padrão e invertida do microscópio. No entanto, como o coração de um peixe nadador está sempre localizado ventralmente, o posicionamento do canal de imagem abaixo e do canal de iluminação acima aumenta a relação sinal-ruído. Finalmente, melhoramos significativamente o desempenho do processamento empregando uma rede neural para localizar o espécime dentro da imagem e identificar órgãos individuais. Usando conhecimento prévio e dados empíricos, desenvolvemos métodos para identificar o coração no corpo da larva do peixe-zebra, conforme detectado pela rede neural.
Outras vantagens da abordagem incluem o uso de uma fonte de luz LED, que, ao contrário das lâmpadas halógenas, não gera calor. A fixação do canal de iluminação no palco permite o alinhamento em relação ao tamanho do molde uma vez, eliminando a necessidade de ajustes de luz adicionais ao refocar. O algoritmo também pode ser aplicado para gerar grandes conjuntos de dados para o treinamento subsequente de uma rede neural para segmentação cardíaca. Ele acelera o processamento de dados e minimiza erros na determinação da posição do coração. Rotular diretamente o coração nas imagens apresenta um desafio significativo devido ao seu tamanho pequeno e às dificuldades em determinar com precisão seus limites e posição em um único quadro.
Vários fatores críticos devem ser abordados para alcançar os melhores resultados ao implementar este protocolo. Primeiro, o tempo de exposição não deve exceder 1,5 ms, pois tempos de exposição mais longos podem levar ao desfoque da imagem, principalmente durante movimentos rápidos de indivíduos. Esse desfoque dificultaria o cálculo preciso do fotopletismograma em pontos de tempo específicos (Figura 6B). Em segundo lugar, as forças de tensão superficial farão com que a superfície da água na cubeta se curve, levando à dispersão da luz e distorção nas imagens resultantes durante a captura de vídeo. Para mitigar esse efeito, uma camada fina e uniforme de água deve ser aplicada sobre a superfície da agarose para garantir um nível de água plano e consistente em toda a placa de Petri. Como a agarose absorve água gradualmente, recomendamos adicionar água aproximadamente a cada 10 minutos para manter um nível estável. Finalmente, a escolha da ampliação da lente deve ser adaptada ao estágio de desenvolvimento e aos requisitos específicos do experimento. Uma ampliação fixa dentro de uma faixa apropriada é suficiente para medições consistentes dentro da mesma faixa etária e sob condições uniformes. No entanto, uma lente de ampliação variável é mais adequada para estudos que envolvem diferentes estágios ou tarefas de desenvolvimento.
Se o teste revelar que o algoritmo falha, as etapas 2 e 3 devem ser revisitadas e reimplementadas. A etapa 3 geralmente apresenta desafios, principalmente durante a rotulagem de imagens. Garanta uma anotação cuidadosa dos olhos e do corpo, evitando qualquer sobreposição entre essas duas classes. Use imagens que capturem o indivíduo em várias posições, especialmente aquelas que exibem flexão corporal pronunciada, para aumentar a precisão da rotulagem. A etapa 2 deve ser repetida nos casos em que forem identificados problemas com intensidade de iluminação, tempo de exposição ou ampliação. Configurações incorretas podem reduzir a relação sinal-ruído, permitindo que o ruído sobrecarregue o sinal (Figura 6B). Além disso, pode surgir falha no algoritmo se os dados de treinamento e experimentais forem obtidos em diferentes configurações.
A configuração de hardware proposta utiliza uma câmera infravermelha com uma faixa de sensibilidade de 900-1700 nm. No entanto, as câmeras de alta resolução que operam nessa faixa espectral geralmente têm um custo proibitivo. Para resolver essa limitação, câmeras de vídeo industriais de espectro visível com sensibilidade do sensor que se estende até a faixa do infravermelho próximo podem ser empregadas após a remoção de seus filtros infravermelhos como uma alternativa econômica às câmeras infravermelhas. Além do uso de radiação infravermelha, os efeitos da pigmentação também podem ser minimizados com a aplicação de produtos químicos removedores de pigmento ou com o uso de peixes de linhagens genéticas específicas que carecem de pigmentação28,29. A versão atual do software foi projetada exclusivamente para medição da frequência cardíaca. No entanto, outros parâmetros de onda de pulso são igualmente essenciais para pesquisas abrangentes. Futuras atualizações de software se concentrarão na incorporação da análise da variabilidade da frequência cardíaca, um indicador-chave de várias doenças. Medições mais detalhadas, como o intervalo Q-T, são objeto de investigação adicional. Para desenvolver um modelo de rede neural universal capaz de operar com dados de várias idades e condições de aquisição de imagens, o conjunto de dados de treinamento deve incluir diversas amostras com pelo menos 128 imagens rotuladas de cada tipo.
Várias abordagens foram desenvolvidas anteriormente para detecção automatizada da área cardíaca e monitoramento da frequência cardíaca em embriões de peixe-zebra 4,6 e medaka 6,17. A marcação fluorescente do coração em peixe-zebra foi proposta para determinação da área cardíaca30. No entanto, todos os métodos publicados anteriormente limitam-se a trabalhar com embriões imóveis e transparentes durante o breve período pós-fertilização antes do início da motilidade embrionária. Esta é uma limitação significativa que reduz a aplicabilidade dessas técnicas. Outra questão descrita na literatura e em materiais complementares envolve o movimento brusco dos embriões durante o registro da frequência cardíaca 6,17. Esse movimento pode deslocar a área cardíaca alvo do software para o cálculo da frequência cardíaca. A abordagem descrita neste estudo aborda essas deficiências, permitindo o monitoramento de peixes-zebra móveis de até 30 dpf.
As vantagens dessa abordagem expandem significativamente as possibilidades de sua aplicação potencial. Nos últimos anos, o Danio rerio tornou-se um modelo amplamente difundido para o estudo de patologias cardiovasculares e cardiotoxicidade 11,12,31. A capacidade deste método de registrar de forma não invasiva a frequência cardíaca em diferentes estágios iniciais de desenvolvimento sem anestésicos oferece novas oportunidades para estudar a dinâmica das malformações cardíacas induzidas e os efeitos terapêuticos. Atualmente, o monitoramento da frequência cardíaca em embriões de peixe-zebra é usado para triagem de drogas em estudos pré-clínicos 32. As vantagens descritas do método proposto fornecem ferramentas adicionais para avaliar os efeitos retardados e a farmacodinâmica dos medicamentos. Finalmente, a utilização da freqüência cardíaca do peixe-zebra aumenta cada vez mais no monitoramento ecotoxicológico33,34. Neste campo, a abordagem proposta permite a avaliação dos efeitos tóxicos crônicos de poluentes em baixas concentrações durante períodos de exposição prolongados e os efeitos associados à bioacumulação.
Divulgações
Todos os autores revelaram quaisquer conflitos de interesse.
Agradecimentos
Este estudo foi apoiado pelo Programa de Tarefas do Estado Federal do STC UI RAS (FFNS-2025-0008). Este trabalho foi realizado utilizando o equipamento do Centro de Uso Coletivo do STC UI RAS [http:// https://ckp.ntcup.ru/en/].
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
Referências
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados