Method Article
Misurazioni del battito cardiaco senza anestesia in zebrafish in movimento libero
In questo articolo
Riepilogo
Lo studio mira a sviluppare una tecnologia per la misurazione del battito cardiaco senza anestesia nel pesce zebra in movimento. Il nostro approccio combina l'imaging a infrarossi a onde corte e il monitoraggio del cuore basato sull'apprendimento automatico. Si tratta di una tecnica non invasiva, priva di etichette e di facile utilizzo che si adatta a un'ampia gamma di studi sul modello di zebrafish.
Abstract
Il pesce zebra (Danio rerio) è un organismo modello ampiamente utilizzato nella ricerca fisiologica, farmacologica e tossicologica grazie alla sua somiglianza genetica con l'uomo e allo stadio embrionale trasparente, che facilita gli studi cardiovascolari non invasivi. Tuttavia, gli attuali metodi per la valutazione della frequenza cardiaca nel pesce zebra spesso si basano sull'anestesia per immobilizzare il soggetto, introducendo alterazioni fisiologiche che compromettono l'accuratezza e la riproducibilità dei dati. Questo studio presenta una nuova tecnica senza anestesia per misurare il battito cardiaco nelle larve di zebrafish che si muovono liberamente, affrontando una limitazione critica nella ricerca cardiovascolare. L'approccio proposto integra l'imaging a infrarossi a onde corte con il tracciamento cardiaco basato sull'apprendimento automatico, consentendo un monitoraggio preciso e continuo dell'attività cardiaca in campioni non immobilizzati. Una rete neurale convoluzionale è stata addestrata per rilevare la regione cardiaca e un segnale fotopletismografico è stato estratto dalle sequenze di immagini per determinare la frequenza cardiaca. La convalida sperimentale ha dimostrato l'affidabilità e la coerenza del metodo in più condizioni di test. Un vantaggio chiave della metodologia è la sua capacità di preservare lo stato fisiologico naturale del pesce zebra, riducendo al minimo gli artefatti indotti dallo stress. Questa tecnica non invasiva e priva di marcatura offre vantaggi significativi per lo studio della fisiologia cardiovascolare, della cardiotossicità dei farmaci e della tossicologia ambientale, ampliando le potenziali applicazioni del pesce zebra come modello per la ricerca biomedica.
Introduzione
Il pesce zebra (Danio rerio), un piccolo pesce ciprinide, è diventato un organismo modello essenziale grazie alle sue piccole dimensioni, all'alto tasso di riproduzione e alla facilità di manipolazione genetica 1,2,3. La valutazione della frequenza cardiaca negli embrioni trasparenti di zebrafish è sempre più utilizzata in fisiologia, embriologia, tossicologia e altri campi 4,5,6,7,8. Da un lato, questa utilità è dovuta al fatto che il genoma del pesce zebra include geni associati a malattie cardiovascolari umane9 e il cuore di Danio rerio condivide strutture e vie di segnalazione simili con gli esseri umani10,11. Rende il pesce zebra un modello inestimabile per lo studio dello sviluppo e delle malattie cardiache 11,12,13. D'altra parte, la frequenza cardiaca del pesce zebra è sensibile alle influenze esterne, il che lo rende un modello eccellente per studi fisiologici e tossicologici confrontando la funzione cardiaca nei pesci trattati e non trattati 7,8,14.
Sono stati compiuti progressi significativi nello sviluppo di metodi ottici non invasivi per la valutazione della frequenza cardiaca in embrioni di pesce trasparenti15,16. Queste tecniche offrono il vantaggio di una rapida raccolta di dati da campioni di grandi dimensioni. Di conseguenza, sono stati sviluppati approcci completamente automatizzati per la valutazione della frequenza cardiaca negli embrioni di pesce 4,5,6,17.
Tuttavia, alcune limitazioni attualmente limitano l'uso di queste tecniche al periodo di 3-4 dpf. La prima limitazione è una perdita di trasparenza dovuta alla pigmentazione del corpo del pesce. Il secondo è l'aumento della mobilità degli embrioni nel tempo. Estendere il periodo di sviluppo precoce del pesce zebra durante il quale gli approcci ottici possono essere utilizzati migliorerebbe la loro utilità, consentendo disegni sperimentali a lungo termine per studiare la cardiomiopatia, i difetti cardiaci congeniti e vari impatti sul sistema cardiovascolare, compreso il monitoraggio della dinamica degli effetti nel tempo. Il nostro gruppo ha recentemente affrontato il problema della perdita di trasparenza impiegando l'imaging nella gamma dell'infrarosso a onde corte di 900-1700 nm18. Questo articolo si concentra sull'affrontare la questione della mobilità degli embrioni.
Tipicamente, anestetici come la tricaina metanassonato (MS-222) vengono utilizzati per immobilizzare embrioni e larve di pesci che nuotano liberamente prima dell'imaging 14,19,20. Tuttavia, MS-222 riduce significativamente la frequenza cardiaca21,22, così come altri anestetici23. Diventa difficile discernere se i cambiamenti osservati nella funzione cardiaca sono dovuti a un trattamento sperimentale, all'anestetico o a un'interazione tra i due. Un altro modo per prolungare il periodo di bassa mobilità dell'embrione è ridurre la temperatura durante lo sviluppo precoce8. Tuttavia, questo approccio non è sempre compatibile con gli obiettivi della ricerca e prolunga solo in minima parte il periodo di registrazione.
In questo studio, introduciamo un nuovo metodo per affrontare la mobilità dell'embrione durante la registrazione della frequenza cardiaca. Abbiamo addestrato una rete neurale convoluzionale per identificare la regione di interesse del cuore nelle registrazioni di embrioni di pesce zebra che nuotano liberamente. La variazione periodica dell'intensità dei pixel all'interno di quest'area viene utilizzata per derivare il fotopletismogramma (PPG), che viene successivamente utilizzato per calcolare la frequenza cardiaca. L'applicazione desktop sviluppata, AutoHR, ha utilizzato sia l'addestramento della rete neurale che l'elaborazione dello stack di immagini, garantendo facilità d'uso e riproducibilità del protocollo.
Protocollo
I pesci zebra sono stati allevati e allevati secondo i protocolli ZFIN24 stabiliti. Tutte le procedure sono state approvate dal Comitato di Bioetica del Centro Scientifico e Tecnologico di Strumentazione Unica dell'Accademia Russa delle Scienze (STC UI RAS), protocollo #3/24, datato 08/21/2024, e seguono le linee guida per la cura del pesce zebra di STC UI RAS. Su richiesta sono disponibili manuali per le singole versioni.
1. Preparazione dell'attrezzatura per la misurazione
- Selezione dell'attrezzatura
- Scegli una sorgente luminosa: scegli una sorgente luminosa a LED che emetta nella gamma del vicino infrarosso, in particolare da 800 nm, con una potenza minima di uscita di 3 W. Questo studio ha utilizzato LED con una lunghezza d'onda centrale di 940 nm e una larghezza di banda di 40 nm.
- Scegli una videocamera: scegli una fotocamera dotata di un sensore sensibile alla gamma di lunghezze d'onda emesse dalla sorgente luminosa e con un frame rate di almeno 60 fps. Fare riferimento alla scheda tecnica della fotocamera per determinare il pixel pitch p, la dimensione del sensore lungo il lato più corto h e la frequenza dei fotogrammi. In questo studio, la fotocamera ha un passo dei pixel di p = 5 μm e una larghezza del sensore h = 5,12 mm.
- Scegli una dimensione dello stampo di agarosio che sia almeno 1,5 volte maggiore della lunghezza della larva B. Calcolare gli ingrandimenti minimi e massimi consentiti del sistema ottico utilizzando le espressioni fornite. Assicurarsi che Mmax ≤ Mmin. Se questa condizione non è soddisfatta, selezionare una telecamera con una h più grande e/o una S più piccola e/o più piccola.
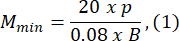
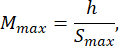
dove Smax è la lunghezza dello stampo di agarosio più grande del set di stampi. È possibile preparare una serie di stampi di varie dimensioni. Questo studio ha utilizzato S = 10 mm.
NOTA: Le equazioni sono derivate in base alla geometria dello schema ottico e alle proporzioni del corpo del pesce zebra. Spiegazioni dettagliate sono fornite nella sezione Discussione. - Scegli un obiettivo: questo studio ha utilizzato un sistema ottico con ingrandimenti variabili che vanno da MOSmin = 0,4x a MOSmax = 1,5x, una lunghezza focale massima di fmax = 150 mm e una distanza di lavoro di mm.
NOTA: Il simbolo x indica il fattore di ingrandimento del sistema ottico.- Scegliere un sistema di microscopio a ingrandimento variabile con un intervallo di ingrandimento (MOSmin - MOSmax) che rimane all'interno dell'intervallo consentito (Mmin - Mmax). Fare riferimento alla scheda tecnica dell'obiettivo per determinare la lunghezza focale massima fmax e la corrispondente distanza di lavoro L per MOSmax.
- Assicurarsi che la condizione fmax ≤ Mmax • L sia soddisfatta. Se questa condizione non viene soddisfatta, selezionare un obiettivo con un fmax e/o L diverso. Assicurarsi che il tipo di innesto della fotocamera e dell'obiettivo siano compatibili.
- Scegli un oggetto di prova (un righello o una griglia) con un passo noto e una lunghezza totale di almeno . Scegli un oggetto di prova sotto forma di righello o griglia con un passo noto e una dimensione totale di almeno Smax.
- Montaggio di un supporto per l'acquisizione delle immagini (Figura 1).
- Montare l'asta tonda su una base stabile. Assemblare il sistema di imaging collegando l'obiettivo alla fotocamera. Montare il canale di imaging sull'asta con la finestra di ingresso rivolta verso l'alto.
- Montare il meccanismo di messa a fuoco sull'asta. Fissare un tavolino con una superficie di lavoro in vetro con un diametro minimo di 30 mm al meccanismo di messa a fuoco.
- Assemblare l'illuminatore: posizionare il LED su un substrato metallico per garantire un'efficiente dissipazione del calore. Posizionare una lente di collimatore piatta convessa con un diametro di 23 mm e una lunghezza focale di 30 mm a una distanza di 30 mm dal LED.
- Montare l'illuminatore sul tavolino utilizzando una staffa regolabile su tre assi, posizionandolo sopra il piano di lavoro con la lente rivolta verso il basso.
- Regolare l'illuminatore e il sistema di imaging. Posizionare l'illuminatore coassialmente con il sistema di imaging. Accendere l'illuminatore e collegare la fotocamera al computer.
- Avvia l'applicazione della fotocamera e attiva la modalità di anteprima seguendo le istruzioni nel manuale della fotocamera. Posiziona l'oggetto di test sullo stage. Regolare la distanza di lavoro e l'ingrandimento del microscopio per assicurarsi che le dimensioni si adattino completamente al campo visivo.
NOTA: La dimensione deve adattarsi interamente al lato corto del telaio per garantire una visualizzazione completa dello stampo di agarosio. - Regolare la posizione del tavolino rispetto al sistema di imaging per garantire che l'immagine sia a fuoco e che la posizione dell'illuminatore sia illuminata in modo uniforme.
- Preparazione dello stampo di agarosio
- Mescolare 20 ml di acqua distillata con 0,4 g di agarosio a basso punto di fusione e scaldare la miscela fino a quando l'agarosio non si scioglie completamente. Ad esempio, in un forno a microonde per 20 s a una potenza di 700 W.
- Modellare lo stampo di agarosio (Metodo 1): Versare la soluzione riscaldata nella capsula di Petri, assicurandosi di un'altezza della soluzione di 1,5-2 mm. Raffreddare la capsula di Petri fino a quando la soluzione di agarosio non si sarà solidificata. A una temperatura di 17 °C, questo processo dura circa 5 minuti. Taglia un quadrato con una lunghezza laterale usando un coltello da cancelleria e rimuovi con cura lo strato di agarosio all'interno dell'area delineata.
- Modellare lo stampo di agarosio (Metodo 2): creare (S) × (S) × 10 mm, forme da plastica. Posizionare il modulo stampato con il lato quadrato in una capsula di Petri e riempirlo con la soluzione riscaldata, garantendo un'altezza della soluzione di 1,5-2 mm. Raffreddare la capsula di Petri fino a quando la soluzione di agarosio non si è solidificata e rimuovere con cura la forma dalla soluzione solidificata.
NOTA: I moduli possono essere facilmente stampati utilizzando una stampante 3D.
2. Acquisizione delle immagini
- Regolare le impostazioni di acquisizione delle immagini.
- Collegare la fotocamera al computer. Avvia l'applicazione della fotocamera e abilita la modalità di anteprima. Accendi l'illuminatore.
NOTA: Se la dimensione dello stampo di agarosio da utilizzare differisce da quella del passaggio 1.3.3, ripetere il passaggio 1.3.3 e rimuovere l'oggetto di prova dal tavolino successivo. - Mettere la larva di pesce zebra in uno stampo di agarosio con una pipetta Pasteur. Posizionare uno stampo di agarosio sul palco. Garantire un'illuminazione quasi completa della testa della larva regolando una combinazione di tempo di esposizione e potenza dell'illuminatore (Figura 2A-C). Il tempo di esposizione non deve superare 1,5 ms.
NOTA: In questo studio, presentiamo i dati per le larve di pesce zebra wild-type di età compresa tra 12 e 21 giorni dopo la fecondazione (dpf).
- Collegare la fotocamera al computer. Avvia l'applicazione della fotocamera e abilita la modalità di anteprima. Accendi l'illuminatore.
- Eseguire l'acquisizione delle immagini
- Imposta la frequenza dei fotogrammi su almeno 60 fps. Impostare la profondità di bit dell'immagine su 12 bit. Acquisire immagini per almeno 10 s. Impostate il formato di denominazione dei fotogrammi su yyyy_mm_dd_hh_ss_mm_mss e il tipo di file deve essere PNG o TIFF, non BMP.
3. Addestramento della rete neurale per l'etichettatura
- Preparazione dei dati per l'etichettatura
NOTA: Viene descritto il processo di etichettatura nell'app Image Labeler, MATLAB.- Raccogli almeno 256 immagini acquisite in una directory separata. Esegui l'app. Importa le immagini accedendo a Importa > da file e selezionando tutte le immagini dalla directory specificata.
- Creare classi per l'etichettatura di rete. Nella scheda Etichette ROI, fai clic su Etichetta, inserisci il corpo nel campo Nome etichetta , seleziona Etichetta pixel dal menu a discesa per il tipo di etichetta e fai clic su OK. Nella scheda Etichette ROI, fai clic su Etichetta, inserisci l'occhio nel campo Nome etichetta , seleziona Etichetta pixel dal menu a discesa per il tipo di etichetta e fai clic su OK. Nella scheda Etichette ROI, fai clic su Etichetta, inserisci fish nel campo Nome etichetta , seleziona Rettangolo dal menu a discesa per il tipo di etichetta e fai clic su OK.
- Etichettatura dei telai
- Nella scheda Etichette ROI, selezionare la classe di annotazione creata in precedenza, eye. Delinea un occhio del campione posizionando i punti lungo il suo perimetro usando il tasto sinistro del mouse. Per completare l'annotazione dell'occhio, fare clic sul punto iniziale del contorno (Figura 3B).
- Delineare un altro occhio del campione nello stesso modo descritto al punto 3.1.2.
- Nella scheda Etichette ROI, selezionare la classe di annotazione creata, il corpo. Delineare il corpo del campione, esclusi gli occhi, posizionando dei punti lungo il suo perimetro utilizzando il tasto sinistro del mouse. Per completare l'annotazione dell'occhio, fare clic sul punto iniziale del contorno.
- Nella scheda Etichette ROI, selezionare la classe di annotazione creata in precedenza, fish. Delineare l'intero corpo del campione disegnando un riquadro di delimitazione rettangolare (Figura 4). Per fare ciò, fare clic su un angolo del rettangolo desiderato con il tasto sinistro del mouse, quindi trascinare il cursore tenendo premuto il pulsante fino a quando il rettangolo non racchiude completamente il pesce.
- Salvare i contorni in una nuova directory denominata images facendo clic su Esporta > su file nella scheda ETICHETTA.
- Preparazione dei set di dati per l'addestramento delle reti neurali
- Avviare MATLAB. Aprire il file di script DatasetsPreparation.m (File supplementare 1) selezionando Apri > Apri.
- Eseguire lo script facendo clic su Esegui. Nella finestra pop-up, selezionare la directory contenente le immagini delineate, la cartella PixelLabelData e il file gTruth.mat, esportato nel passaggio 3.2.5.
NOTA: Al termine dell'esecuzione dello script, verranno generate automaticamente due cartelle: RPN Training e CNN Training. Sono necessari per il successivo addestramento della rete neurale. Non modificare il contenuto di queste cartelle.
4. Addestrare le reti neurali per il rilevamento del cuore
NOTA: Questo passaggio viene eseguito una volta per un'età e una condizione di imaging specifiche. La GPU NVIDIA è fortemente consigliata per l'addestramento in quanto accelera significativamente l'elaborazione.
- Avvia l'applicazione AutoHR. Vai alla scheda Formazione della rete di proposte regionali. Fare clic su Scegli directory e selezionare la cartella di formazione RPN creata nel passaggio 3.3.2. Fare clic su Inizia formazione.
NOTA: Al termine del training, verrà generato un file di modello denominato rpn_model.pt nella stessa directory del file AutoHR.exe. Questo file è necessario per l'ulteriore elaborazione dello stack di immagini. - Per addestrare la rete neurale convoluzionale, accedere alla scheda Addestramento rete neurale convoluzionale. Fare clic su Scegli directory e selezionare la cartella CNN Training creata nel passaggio 3.3.2. Fare clic su Inizia formazione.
NOTA: Al termine del training, verrà generato un file di modello denominato model.pt nella stessa directory del file AutoHR.exe. Questo file è necessario per l'ulteriore elaborazione dello stack di immagini.
5. Quantificazione della frequenza cardiaca
- Registrare una sequenza di immagini come descritto al punto 2. Assicurarsi che le condizioni di imaging, come l'ingrandimento, il tempo di esposizione e l'illuminazione, siano identiche a quelle utilizzate per l'addestramento della rete neurale.
- Avvia l'applicazione AutoHR. Passare alla scheda Elaborazione. Selezionare la directory contenente i modelli di rete neurale addestrati facendo clic su Scegli modelli. Fare clic su Scegli cartella per importare le immagini acquisite per l'analisi. Il percorso di esportazione verrà specificato automaticamente, ma è possibile modificarlo facendo clic su Scegli percorso di esportazione. Fare clic su Elabora per avviare l'analisi.
NOTA: Al termine, il valore della frequenza cardiaca apparirà nel campo Frequenza cardiaca all'interno dell'interfaccia AutoHR. La cartella esportata includerà i valori della frequenza cardiaca (.xlsx), PPG (.xlsx), il primo fotogramma della sequenza e le maschere del corpo e degli occhi per il primo fotogramma (.png).
6. Verifica dei risultati dell'algoritmo
- Eseguire il test con un pesce immobile come descritto di seguito.
- Anestetizzare le larve con una soluzione di MS-222 0,168 mg/L per 1 minuto. Acquisire una pila di immagini di un campione anestetizzato come descritto nel passaggio 2. Eseguire un conteggio della frequenza cardiaca attraverso l'osservazione visiva.
- Utilizza il software HR come descritto nel passaggio 5. Ripetere i passaggi per diversi campioni. Verificare se i valori della frequenza cardiaca ottenuti sono coerenti per tutti gli individui entro la precisione desiderata.
- Eseguire il test con un individuo mobile e il test di provocazione come descritto di seguito.
- Acquisire una pila di immagini di un campione non anestetizzato come descritto nel passaggio 2. Condurre un test di provocazione con l'individuo per il quale è stata registrata la sequenza, utilizzando uno stimolo noto che influisce sulla frequenza cardiaca. Lo studio presenta i risultati della salinizzazione dell'acqua a concentrazioni di 5 mg/L.
- Acquisire una pila di immagini dello stesso campione come descritto nel passaggio 2 dopo il test di provocazione. Determinare la frequenza cardiaca in entrambi gli esperimenti con il software HR come descritto al punto 5. Verificare se le variazioni osservate nella frequenza cardiaca sono in linea con gli effetti noti del test di provocazione.
Risultati
La frequenza cardiaca del pesce zebra a 12 dpf è stata determinata utilizzando il protocollo sopra descritto (Video supplementare 1). I video includono una sequenza di immagini di larve di pesce zebra che nuotano liberamente, un fotopletismogramma derivato da queste sequenze utilizzando il protocollo proposto e la frequenza cardiaca corrispondente calcolata dal fotopletismogramma.
I dati etichettati sono stati suddivisi in modo casuale in set di addestramento, test e convalida in un rapporto 3:2:1 durante l'addestramento. La funzione di perdita è stata quindi implementata secondo la seguente espressione:

dove x è la logica di input, y è un target, la dimensione del batch è il numero di immagini elaborate contemporaneamente,

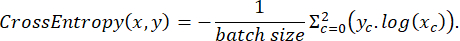
Per valutare le prestazioni del modello durante l'addestramento, è stata impiegata la funzione di accuratezza della previsione, definita da quanto segue:


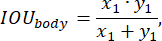
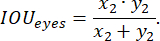
Abbiamo applicato approcci standard allo sviluppo della funzione di perdita, come dettagliato, ad esempio, in25. La rete di attenzione multiscala con architettura del modello EfficientNet-B0 è stata utilizzata come encoder e livello di output softmax. La rete neurale è stata addestrata su un campione di prova per 50 epoche, utilizzando un tasso di apprendimento esponenzialmente decrescente che iniziava a 0,02 e aveva un fattore di decadimento di 0,99.
La rete neurale addestrata è stata utilizzata per ottenere la maschera corporea e la maschera oculare (Figura 5С) dell'individuo in ogni fotogramma di una sequenza arbitraria. È stato quindi calcolato il centro di massa della maschera per gli occhi e la scheletratura è stata applicata alla maschera per il corpo (Figura 5D). Sulla corda di pesce risultante P, è stato identificato il punto M, situato più vicino al centro di massa della maschera oculare (Figura 5F). Ad una distanza d dal punto M, uguale alla distanza tra gli occhi, il punto N è stato identificato lungo la corda PPP verso la coda. Questo punto corrisponde al centro dell'area del cuore. Quest'area era delineata da un cerchio con un raggio di r = d x 0,3, centrato in questo punto (Figura 5F).
Il segnale PPG grezzo è stato calcolato come il numero medio di pixel all'interno dell'area del cuore in tutti i fotogrammi registrati (Figura 5G). Questo segnale grezzo è stato quindi normalizzato al suo valore medio. Successivamente, il segnale è stato filtrato utilizzando un filtro Chebyshev di tipo II di secondo ordine con un design del 4° ordine, un'attenuazione della banda di arresto di 25 dB e una frequenza del bordo della banda di arresto di 12 Hz, coerente con gli algoritmi di elaborazione standard26,27 (Figura 6A). La frequenza cardiaca è stata determinata identificando la frequenza all'interno dell'intervallo 1-4 Hz che corrisponde alla potenza di picco nel modulo quadrato della trasformata di Fourier del segnale filtrato (Figura 6A).
L'equazione (1) assicura che l'immagine del cuore sia di circa 20 pixel x 20 pixel. Si presume che la dimensione longitudinale del cuore rappresenti circa l'8% della lunghezza totale del corpo del pesce zebra. Quando l'immagine del cuore è più piccola, il segnale risultante contiene spesso una componente di rumore significativa che supera il segnale utile, rendendolo inadatto per il calcolo della frequenza cardiaca, come mostrato nella Figura 6B.
I risultati dell'applicazione del protocollo dipendono in modo significativo dalla stabilità della segmentazione cardiaca per ciascun individuo. È fondamentale condurre esperimenti di controllo per dimostrare che le fasi 2-5 sono state completate con successo e che i dati ottenuti riflettono processi fisiologici reali piuttosto che risultati casuali. La convalida del protocollo è stata eseguita seguendo la procedura descritta nel passaggio 6. Nel test descritto al punto 6.2, il sale è stato aggiunto all'acqua a una concentrazione di 5 mg/L come stimolo provocatorio. In ogni test sono state incluse cinque larve di pesce zebra (12 pdf). La temperatura dell'acqua in una forma di agarosio è stata mantenuta a 23-25 °C. I risultati indicano che la differenza tra la valutazione visiva e il metodo proposto non ha superato il 3% nel primo test. Nel secondo test, la frequenza cardiaca misurata con il metodo è aumentata, come previsto (Figura 7).

Figura 1: Flusso di lavoro schematico. (A) Schema e (B) aspetto della configurazione sperimentale Fare clic qui per visualizzare una versione più ampia di questa figura.

Figura 2: Immagini di larve di pesce zebra. Esempi di (A) immagini di larve di pesce zebra di alta qualità e (B-D) di bassa qualità sono (B) fotogramma sottoesposto, (С) fotogramma sovraesposto e (D) fotogramma sfocato. Le immagini sono state ritagliate per evidenziare la larva. La barra della scala è di 1 mm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Immagini rappresentative. Esempi di (A) immagini iniziali e (B) con etichetta. La barra della scala è di 1 mm. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 4: Contorni del corpo del pesce zebra. Esempi di (A) contorni di alta qualità e (B-C) di bassa qualità dell'intero corpo del pesce zebra: un pesce zebra non si adatta interamente all'interno del rettangolo e la dimensione del rettangolo è notevolmente più grande del pesce zebra. La barra della scala è di 1 mm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Pipeline di elaborazione delle immagini che illustra le fasi di elaborazione primarie e i risultati intermedi corrispondenti. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Fotogrammi che mostrano la frequenza cardiaca larvale. Esempi di (A) fotogrammi di alta qualità e (B) di bassa qualità con l'area del cuore larvale con diametri di 20 pixel e 14 pixel, rispettivamente, e corrispondenti spettri PPG e Fourier di PPG. La barra della scala è di 1 mm. Abbreviazioni: PPG = fotopletismografia. Clicca qui per visualizzare una versione più grande di questa figura.
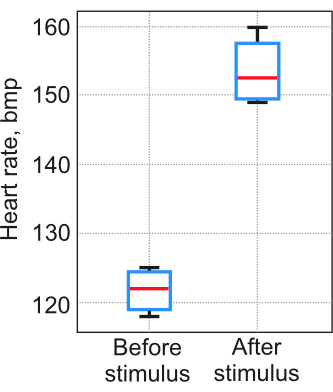
Figura 7: Misurazioni della frequenza cardiaca. Le misurazioni sono state ottenute utilizzando il metodo proposto in un test con uno stimolo provocatorio. Numero di campione . La casella si estende tra il 25° e il 75° percentile, la linea orizzontale è la mediana, i baffi neri indicano i punti dati più estremi non considerati valori anomali Fare clic qui per visualizzare una versione più grande di questa figura.
Video supplementare 1: Illustrazione dell'algoritmo di rete neurale proposto per il rilevamento e il calcolo della frequenza cardiaca. Il video presenta larve di pesce zebra in movimento libero, un fotopletismogramma derivato dal protocollo proposto e calcolato dalla frequenza cardiaca delle larve di fotopletismogramma Clicca qui per scaricare questo file.
File supplementare 1: funzione MATLAB che esegue l'aumento dell'immagine. Clicca qui per scaricare questo file.
Discussione
In questo studio, presentiamo un protocollo sperimentale per misurare il battito cardiaco delle larve di pesce zebra che nuotano liberamente. Abbiamo valutato questo approccio attraverso diversi esperimenti, dimostrandone l'efficacia. I componenti chiave del metodo proposto includono sia soluzioni hardware che software. In primo luogo, abbiamo utilizzato l'illuminazione a infrarossi per l'imaging che, come dimostrato in precedenza, evita problemi legati alla pigmentazione e migliora la trasparenza dei tessuti, facilitando la determinazione accurata del battito cardiaco18. In secondo luogo, proponiamo di fotografare il pesce dal basso. Quando sono anestetizzate, le larve di zebrafish giacciono tipicamente su un fianco a causa della posizione della vescica natatoria, con conseguente differenza di orientamento tra le configurazioni standard e del microscopio invertito trascurabile. Tuttavia, poiché il cuore di un pesce che nuota si trova sempre ventralmente, il posizionamento del canale di imaging al di sotto e del canale di illuminazione al di sopra migliora il rapporto segnale/rumore. Infine, abbiamo migliorato significativamente le prestazioni di elaborazione utilizzando una rete neurale per localizzare il campione all'interno dell'immagine e identificare i singoli organi. Utilizzando sia le conoscenze pregresse che i dati empirici, abbiamo sviluppato metodi per individuare il cuore nel corpo della larva di pesce zebra come rilevato dalla rete neurale.
Altri vantaggi dell'approccio includono l'uso di una sorgente luminosa a LED, che, a differenza delle lampade alogene, non genera calore. Il fissaggio del canale di illuminazione sul tavolino consente l'allineamento relativo alle dimensioni dello stampo una volta, eliminando la necessità di ulteriori regolazioni della luce durante la rimessa a fuoco. L'algoritmo può anche essere applicato per generare grandi set di dati per il successivo addestramento di una rete neurale per la segmentazione del cuore. Accelera l'elaborazione dei dati e riduce al minimo gli errori nella determinazione della posizione del cuore. Etichettare direttamente il cuore sulle immagini rappresenta una sfida significativa a causa delle sue dimensioni ridotte e delle difficoltà nel determinare con precisione i suoi confini e la sua posizione all'interno di un singolo fotogramma.
Diversi fattori critici devono essere affrontati per ottenere risultati ottimali nell'attuazione di questo protocollo. Innanzitutto, il tempo di esposizione non deve superare 1,5 ms, poiché tempi di esposizione più lunghi possono portare alla sfocatura dell'immagine, in particolare durante i movimenti rapidi degli individui. Tale sfocatura ostacolerebbe un calcolo accurato del fotopletismogramma in punti temporali specifici (Figura 6B). In secondo luogo, le forze di tensione superficiale causeranno la curvatura della superficie dell'acqua nella cuvetta, portando alla dispersione della luce e alla distorsione nelle immagini risultanti durante l'acquisizione video. Per mitigare questo effetto, è necessario applicare uno strato d'acqua sottile e uniforme sulla superficie dell'agarosio per garantire un livello d'acqua piatto e costante in tutta la capsula di Petri. Poiché l'agarosio assorbe gradualmente l'acqua, si consiglia di aggiungere acqua ogni 10 minuti circa per mantenere un livello stabile. Infine, la scelta dell'ingrandimento della lente dovrebbe essere adattata allo stadio di sviluppo e ai requisiti specifici dell'esperimento. Un ingrandimento fisso all'interno di un intervallo appropriato è sufficiente per misurazioni coerenti all'interno della stessa fascia di età e in condizioni uniformi. Tuttavia, una lente a ingrandimento variabile è più adatta per studi che coinvolgono diverse fasi o attività di sviluppo.
Se il test rivela che l'algoritmo fallisce, i passaggi 2 e 3 devono essere rivisti e reimplementati. Il passaggio 3 presenta spesso delle sfide, in particolare durante l'etichettatura delle immagini. Assicurati di annotare attentamente gli occhi e il corpo, evitando qualsiasi sovrapposizione tra queste due classi. Utilizza immagini che catturino l'individuo in varie posizioni, in particolare quelle che mostrano una pronunciata flessione del corpo, per migliorare la precisione dell'etichettatura. Il passaggio 2 deve essere ripetuto nei casi in cui vengono identificati problemi con l'intensità dell'illuminazione, il tempo di esposizione o l'ingrandimento. Impostazioni errate possono ridurre il rapporto segnale/rumore, consentendo al rumore di sopraffare il segnale (Figura 6B). Inoltre, può verificarsi un errore dell'algoritmo se i dati di addestramento e sperimentali vengono ottenuti con impostazioni diverse.
La configurazione hardware proposta utilizza una telecamera a infrarossi con una gamma di sensibilità di 900-1700 nm. Tuttavia, le telecamere ad alta risoluzione che operano in questa gamma spettrale sono spesso proibitive dal punto di vista dei costi. Per ovviare a questa limitazione, le videocamere industriali a spettro visibile con sensibilità del sensore che si estende nella gamma del vicino infrarosso possono essere impiegate dopo aver rimosso i filtri a infrarossi come alternativa economica alle telecamere a infrarossi. Oltre a utilizzare la radiazione infrarossa, gli effetti della pigmentazione possono anche essere minimizzati applicando sostanze chimiche che rimuovono il pigmento o utilizzando pesci provenienti da specifiche linee genetiche prive di pigmentazione28,29. L'attuale versione del software è progettata esclusivamente per la misurazione della frequenza cardiaca. Tuttavia, altri parametri dell'onda del polso sono altrettanto essenziali per una ricerca completa. I futuri aggiornamenti del software si concentreranno sull'integrazione dell'analisi della variabilità della frequenza cardiaca, un indicatore chiave di varie malattie. Misurazioni più dettagliate, come l'intervallo Q-T, sono oggetto di ulteriori indagini. Per sviluppare un modello di rete neurale universale in grado di funzionare con dati di varie età e condizioni di acquisizione delle immagini, il set di dati di addestramento dovrebbe includere campioni diversi con almeno 128 immagini etichettate di ciascun tipo.
In precedenza sono stati sviluppati diversi approcci per il rilevamento automatico dell'area cardiaca e il monitoraggio della frequenza cardiaca negli embrioni di zebrafish 4,6 e medaka 6,17. La marcatura fluorescente del cuore nel pesce zebra è stata proposta per la determinazione dell'area cardiaca30. Tuttavia, tutti i metodi precedentemente pubblicati si limitano a lavorare con embrioni immobili e trasparenti durante il breve periodo post-fecondazione prima dell'inizio della motilità embrionale. Si tratta di una limitazione significativa che riduce l'applicabilità di queste tecniche. Un altro problema descritto nella letteratura e nei materiali supplementari riguarda il movimento improvviso degli embrioni durante la registrazione della frequenza cardiaca 6,17. Tale movimento può spostare l'area cardiaca presa di mira dal software per il calcolo della frequenza cardiaca. L'approccio descritto in questo studio affronta queste carenze, consentendo il monitoraggio di pesci zebra mobili fino a 30 dpf.
I vantaggi di questo approccio ampliano notevolmente le possibilità della sua potenziale applicazione. Negli ultimi anni, Danio rerio è diventato un modello diffuso per lo studio delle patologie cardiovascolari e della cardiotossicità 11,12,31. La capacità di questo metodo di registrare in modo non invasivo la frequenza cardiaca in diverse fasi iniziali dello sviluppo senza anestesia offre nuove opportunità per studiare le dinamiche delle malformazioni cardiache indotte e gli effetti terapeutici. Al giorno d'oggi, il monitoraggio della frequenza cardiaca negli embrioni di zebrafish viene utilizzato per lo screening farmacologico negli studi preclinici 32. I vantaggi descritti del metodo proposto forniscono ulteriori strumenti per valutare gli effetti ritardati e la farmacodinamica dei farmaci. Infine, l'utilizzo della frequenza cardiaca del pesce zebra aumenta costantemente nel monitoraggio ecotossicologico 33,34. In questo campo, l'approccio proposto consente di valutare gli effetti tossici cronici degli inquinanti a basse concentrazioni per periodi di esposizione prolungati e gli effetti associati al bioaccumulo.
Divulgazioni
Tutti gli autori hanno segnalato eventuali conflitti di interesse.
Riconoscimenti
Questo studio è stato supportato dal Federal State Task Program di STC UI RAS (FFNS-2025-0008). Questo lavoro è stato eseguito utilizzando l'attrezzatura del Centro per l'uso collettivo di STC UI RAS [http:// https://ckp.ntcup.ru/en/].
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
Riferimenti
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551(2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813(2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046(2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703(2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083(2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13(2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552(2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268(2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329(2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045(2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664(2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884(2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111(2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061(2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65(2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , 4th ed, Univ. of Oregon Press. Eugene. (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. 2024 26th Int Conf Digital Signal Proc Appl DSPA 2024, , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532(2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865(2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852(2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8(2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695(2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075(2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703(2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon