Method Article
自由に動くゼブラフィッシュにおける麻酔不要の心拍測定
要約
この研究は、動くゼブラフィッシュの麻酔なしで心拍を測定する技術を開発することを目的としています。私たちのアプローチは、短波赤外線イメージングと機械学習ベースの心臓追跡を組み合わせたものです。これは、非侵襲的でラベルフリーでユーザーフレンドリーな手法であり、ゼブラフィッシュモデルに関する幅広い研究に適しています。
要約
ゼブラフィッシュ(Danio rerio)は、ヒトとの遺伝的類似性と透明な胚期により、非侵襲的な心血管研究を容易にするため、生理学的、薬理学的、毒物学的研究において広く使用されているモデル生物です。しかし、現在のゼブラフィッシュの心拍数評価方法は、被験者を固定するために麻酔に頼ることが多く、データの精度と再現性を損なう生理学的変化をもたらします。この研究は、自由に動くゼブラフィッシュの幼生の心拍を測定するための新しい麻酔不要の技術を提示し、心血管研究における重大な制限に対処します。提案されたアプローチは、短波赤外線イメージングと機械学習ベースの心臓追跡を統合し、固定されていない標本の心臓活動を正確かつ継続的に監視することを可能にします。畳み込みニューラルネットワークを訓練して心臓領域を検出し、画像配列から光電式容積脈波信号を抽出して心拍数を決定しました。実験的検証により、この分析法の信頼性と一貫性が複数の試験条件で実証されました。この方法論の主な利点は、ゼブラフィッシュの自然な生理学的状態を維持し、ストレスによるアーティファクトを最小限に抑える能力です。この非侵襲的でラベルフリーの技術は、心血管生理学、薬物心毒性、および環境毒性学の研究に大きな利点をもたらし、生物医学研究のモデルとしてのゼブラフィッシュの潜在的な応用を拡大します。
概要
ゼブラフィッシュ(Danio rerio)は、小型のコイ科魚類で、その小型で繁殖率が高く、遺伝子操作が容易であることから、必須のモデル生物となっています1,2,3。透明なゼブラフィッシュの胚における心拍数の評価は、生理学、発生学、毒物学、およびその他の分野でますます利用されています4,5,6,7,8。一方では、この有用性は、ゼブラフィッシュのゲノムがヒトの心血管疾患に関連する遺伝子を含んでおり9、ダニオ・レリオ心臓がヒトと同様の構造とシグナル伝達経路を共有しているという事実によるものである10,11。これにより、ゼブラフィッシュは心臓の発達と病気を研究するための非常に貴重なモデルになります11,12,13。一方、ゼブラフィッシュの心拍数は外部からの影響に敏感であり、処理された魚と未処理の魚の心機能を比較することにより、生理学的および毒物学的研究の優れたモデルとなっています7,8,14。
透明な魚の胚15,16の心拍数を評価するための非侵襲的な光学的方法の開発において、大きな進歩が見られました。これらの手法には、大きなサンプルサイズから迅速にデータを収集できるという利点があります。その結果、魚の胚における心拍数評価のための完全に自動化されたアプローチが開発されました4,5,6,17。
ただし、現在、特定の制限により、これらの手法の使用は 3 〜 4 dpf の期間に制限されています。最初の制限は、魚の体の色素沈着による透明性の喪失です。2つ目は、時間の経過とともに胚の可動性が増加することです。ゼブラフィッシュの初期発生期間を光学的アプローチが利用できる期間を延長することで、その有用性が高まり、心筋症、先天性心疾患、および心血管系へのさまざまな影響を研究するための長期的な実験デザインが可能になります。私たちのグループは最近、900-1700 nm18の短波赤外領域でのイメージングを採用することにより、透明性損失の問題に対処しました。この論文は、胚の移動性の問題に対処することに焦点を当てています。
典型的には、トリカインメタンスルホン酸(MS-222)のような麻酔薬は、イメージング14,19,20の前に、自由に泳ぐ魚の胚や幼生を固定するために使用される。しかし、MS-222は、他の麻酔薬23と同様に、心拍数21,22を有意に減少させる。観察された心機能の変化が実験的治療によるものなのか、麻酔薬によるものなのか、それとも両者の相互作用によるものなのかを見極めるのは難しくなります。胚の低可動性期間を延長する別の方法は、発生初期に温度を下げることである8。ただし、このアプローチは必ずしも研究目標と互換性があるわけではなく、登録期間の延長は最小限に抑えられます。
本研究では、心拍登録時の胚の可動性に対処するための新しい方法を紹介します。私たちは、自由に泳ぐゼブラフィッシュの胚の記録における心臓の関心領域を特定するために、畳み込みニューラルネットワークを訓練しました。この領域内のピクセル強度の周期的な変動は、光電式容積脈波(PPG)を導き出すために利用され、その後、心拍数を計算するために使用されます。開発したデスクトップアプリケーションであるAutoHRは、ニューラルネットワークの学習と画像スタック処理の両方を活用し、使いやすさとプロトコルの再現性を確保しました。
プロトコル
ゼブラフィッシュは、確立されたZFINプロトコル24に従って飼育および飼育されました。すべての手順は、ロシア科学アカデミーの科学技術センター・オブ・ユニーク・インスツルメンテーション(STC UI RAS)の生命倫理委員会によって承認され、2024年8月21日付けのプロトコル#3/24、STC UI RASのゼブラフィッシュケアガイドラインに従っています。個々のバージョンのマニュアルは、ご要望に応じてご利用いただけます。
1. 測定機器の準備
- 機器の選択
- 光源を選択する: 近赤外線範囲、特に 800 nm から放射され、最小出力が 3 W の LED 光源を選択します。この研究では、中心波長940nm、帯域幅40nmのLEDを使用しました。
- ビデオカメラを選択する:光源から放射される波長範囲に感度があり、フレームレートが60fps以上のセンサーを搭載したカメラを選択します。カメラのデータシートを参照して、ピクセルピッチ p、短辺 hに沿ったセンサーサイズ、およびフレームレートを決定します。この研究では、カメラのピクセルピッチは p = 5 μm、センサー幅は h = 5.12 mmです。
- 幼虫Bの長さの少なくとも1.5倍大きいアガロースの型のサイズを選択してください。指定された式を使用して、光学系の最小許容倍率と最大許容倍率を計算します。M最大 ≤M最小を確認します。この条件を満たさない場合は、hが大きいカメラ、Sが小さいカメラ、または小さいSカメラを選択してください。
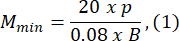
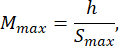
ここで、 Smax は、モールドセットの最大のアガロースモールドの長さです。様々なサイズの型をセットで用意できます。この研究では 、S = 10 mmを使用しました。
注:方程式は、光学スキームの形状とゼブラフィッシュの体の割合に基づいて導き出されます。詳細な説明は、ディスカッションセクションに記載されています。 - レンズの選択:この研究では、 MOSmin = 0.4x から MOSmax = 1.5x までの可変倍率、 fmax = 150 mmの最大焦点距離、および作動距離 mm の可変倍率の光学系を使用しました。
注意: 記号xは、光学系の倍率を示します。- 倍率可変顕微鏡タイプのシステムは、倍率範囲(MOSmin - MOSmax)が許容範囲(Mmin - Mmax)に収まるようにしてください。レンズのデータシートを参照して、M OSmaxの最大焦点距離f maxと対応する作動距離Lを決定します。
- 条件fmax≤Mmax •Lが満たされていることを確認します。この条件を満たさない場合は、FmaxやLが異なるレンズを選んでください。カメラとレンズのマウントタイプに互換性があることを確認してください。
- 既知のピッチと少なくともの全長を持つテストオブジェクト(定規またはグリッド)を選択します。定規またはグリッドの形式で、ピッチが既知で、合計サイズが Smax 以上のテストオブジェクトを選択します。
- 画像取得用のスタンドの組み立て(図1)。
- 丸棒を安定したベースに取り付けます。レンズをカメラに取り付けて、イメージングシステムを組み立てます。インプットウィンドウを上に向けて、イメージングチャンネルをロッドに取り付けます。
- フォーカシング機構をロッドに取り付けます。集光機構に最小直径30mmのガラス製作業面を持つステージを取り付けます。
- イルミネーターの組み立て:LEDを金属基板に配置して、効率的な熱放散を確保します。直径23mm、焦点距離30mmの凸型フラットコリメータレンズをLEDから30mmの距離に置きます。
- イルミネーターは、3軸調整可能なブラケットを使用してステージに取り付け、レンズを下に向けて作業面の上に配置します。
- イルミネーターとイメージングシステムを調整します。イルミネーターをイメージングシステムと同軸に配置します。イルミネーターの電源を入れ、カメラをコンピューターに接続します。
- カメラアプリケーションを起動し、プレビューモードをアクティブにしますview カメラのマニュアルの指示に従って。テストオブジェクトをステージ上に置きます。顕微鏡の作動距離と倍率を調整して、サイズが視野内に完全に収まるようにします。
注:サイズは、アガロース型が完全に視覚化されるように、フレームの短辺に完全に収まる必要があります。 - イメージングシステムに対するステージの位置を調整して、画像にピントが合い、イルミネーターの位置が均一に照らされるようにします。
- アガロース型の準備
- 20 mLの蒸留水と0.4 gの低融点アガロースを混合し、アガロースが完全に溶解するまで混合物を加熱します。たとえば、700 Wの電力で20秒間の電子レンジで。
- アガロース型の形を整える(方法1):加熱した溶液をペトリ皿に注ぎ、溶液の高さを1.5〜2mmにします。アガロース溶液が固まるまでペトリ皿を冷やします。17°Cの温度では、このプロセスには約5分かかります。文房具ナイフを使用して一辺の長さの正方形を切り取り、輪郭を描かれた領域内のアガロース層を慎重に取り除きます。
- アガロース型を成形する(方法2):プラスチックから10mm×(S)×(S)を作成します。印刷されたフォームを正方形の面をシャーレに入れ、加熱した溶液で満たし、溶液の高さを1.5〜2mmにします。アガロース溶液が固まるまでシャーレを冷却し、固化した溶液からフォームを慎重に取り除きます。
注:フォームは3Dプリンターを使用して簡単に印刷できます。
2. 画像取得
- 画像キャプチャ設定を調整します。
- カメラをコンピューターに接続します。カメラアプリケーションを起動し、プレビューモードを有効にします。イルミネーターの電源を入れます。
注:使用するアガロース型のサイズが手順1.3.3のサイズと異なる場合は、手順1.3.3を繰り返して、その後のステージからテスト対象物を取り外してください。 - ゼブラフィッシュの幼生をパスツールピペットでアガロースの型に入れます。アガロースの型をステージに置きます。露光時間とイルミネーターの電力の組み合わせを調整することにより、幼虫の頭部のほぼ完全な照明を確保します(図2A-C)。露光時間は1.5ミリ秒を超えてはなりません。
注:この研究では、受精後12〜21日齢の野生型ゼブラフィッシュの幼生(dpf)のデータを提示します。
- カメラをコンピューターに接続します。カメラアプリケーションを起動し、プレビューモードを有効にします。イルミネーターの電源を入れます。
- 画像取得の実行
- フレームレートを 60 fps 以上に設定します。画像のビット深度を 12 ビットに設定します。少なくとも 10 秒間の画像を取得します。フレームの命名形式を yyyy_mm_dd_hh_ss_mm_mss に設定し、ファイルの種類は BMP ではなく PNG または TIFF にする必要があります。
3. ラベリングのためのニューラルネットワークのトレーニング
- ラベリング用データの準備
注: イメージ ラベラー アプリ MATLAB でのラベル付けのプロセスについて説明します。- 取得した画像を少なくとも 256 個別のディレクトリに集めます。アプリを実行します。[ Import of File ] に移動し、指定したディレクトリからすべての画像を選択して、画像をインポートし>。
- ネットワーク ラベリングのクラスを作成します。ROI Labelsタブで Labelをクリックし、 Label Name フィールドにbodyと入力し、ラベルタイプのドロップダウンメニューから Pixel Label を選択して、 OKをクリックします。「ROI ラベル」タブで、「 ラベル」をクリックし、「 ラベル名 」フィールドに「eye」と入力し、ラベル・タイプのドロップダウン・メニューから 「ピクセル・ラベル 」を選択して 、「OK」をクリックします。「ROI ラベル」タブで、「 ラベル」をクリックし、「 ラベル名 」フィールドに「魚」と入力し、ラベル・タイプのドロップダウン・メニューから 「長方形 」を選択して、「OK」をクリックします。
- フレームのラベリング
- [ROI ラベル] タブで、以前に作成した注釈クラス eye を選択します。マウスの左ボタンを使用して、標本の周囲に沿って点を配置し、試験片の片方の目の輪郭を描きます。目の注釈を完了するには、アウトラインの始点をクリックします(図3B)。
- ステップ3.1.2で説明したのと同じ方法で、標本の別の目の輪郭を描きます。
- [ROI ラベル] タブで、作成した注釈クラス body を選択します。目を除く標本の体の輪郭を描くには、マウスの左ボタンを使用して標本の周囲に点を配置します。目の注釈を完成させるには、 アウトラインの始点をクリックします。
- [ROI ラベル] タブで、以前に作成した注釈クラス fish を選択します。長方形のバウンディングボックスを描画して、試験片のボディ全体の輪郭を描きます(図4)。これを行うには、マウスの左ボタンで目的の長方形の 1つのコーナー をクリックし、長方形が魚を完全に囲むまでボタンを押しながらカーソルをドラッグします。
- アウトラインを images という名前の新しいディレクトリに保存するには、「ラベル」タブの 「> をファイルにエクスポート 」をクリックします。
- ニューラルネットワーク学習用のデータセットの準備
- MATLAB を起動します。[開く] > [開く] を選択して、スクリプト ファイル DatasetsPreparation.m (補足ファイル 1) を開きます。
- [ 実行] をクリックしてスクリプトを実行します。ポップアップウィンドウで、アウトライン化された画像を含むディレクトリ、フォルダPixelLabelData、および手順3.2.5でエクスポートしたファイルgTruth.matを選択します。
注: スクリプトの実行が完了すると、RPN Training と CNN Training の 2 つのフォルダが自動的に生成されます。これらは、その後のニューラルネットワークのトレーニングに必要です。これらのフォルダの内容は変更しないでください。
4. 心臓検出のためのニューラルネットワークのトレーニング
注:この手順は、特定の年齢と画像条件に対して1回実行されます。NVIDIA GPUは、処理を大幅に高速化するため、トレーニングに強くお勧めします。
- AutoHRアプリケーションを起動します。「地域提案ネットワーク研修」タブに移動します。 「ディレクトリの選択 」をクリックし、手順 3.3.2 で作成した RPN Training フォルダを選択します。 「トレーニングを開始」をクリックします。
注: トレーニングが完了すると、rpn_model.pt という名前のモデル ファイルが AutoHR.exe ファイルと同じディレクトリに生成されます。このファイルは、さらに画像スタックを処理するために必要です。 - 畳み込みニューラル ネットワークに学習させるには、畳み込みニューラル ネットワークの学習タブに移動します。 「ディレクトリの選択 」をクリックし、手順 3.3.2 で作成した CNN Training フォルダーを選択します。 「トレーニングを開始」をクリックします。
注: トレーニングが完了すると、model.pt という名前のモデル ファイルが AutoHR.exe ファイルと同じディレクトリに生成されます。このファイルは、さらに画像スタックを処理するために必要です。
5. 心拍数の定量化
- ステップ 2 で説明したように、一連の画像を記録します。倍率、露光時間、照明などのイメージング条件が、ニューラルネットワークの学習に使用した条件と同じであることを確認してください。
- AutoHRアプリケーションを起動します。[処理] タブに移動します。学習済みのニューラル ネットワーク モデルを含むディレクトリを選択する には、[モデルの選択] をクリックします。[ フォルダの選択 ]をクリックして、取得した画像を分析用にインポートします。エクスポートパスは自動的に指定されますが、[ エクスポートパスの選択]をクリックして変更できます。 「プロセス 」をクリックして、分析を開始します。
注意: 完了すると、心拍数の値がAutoHRインターフェース内の心拍数フィールドに表示されます。エクスポートされたフォルダには、心拍数の値(.xlsx)、PPG(.xlsx)、シーケンスの最初のフレーム、および最初のフレーム(.png)のボディマスクとアイマスクが含まれます。
6. アルゴリズムの結果の検証
- 以下に説明するように、動かない魚でテストを実行します。
- 0.168 mg/L MS-222溶液で幼虫に1分間麻酔をかけます。ステップ2で説明したように、麻酔をかけた標本の画像スタックを取得します。目視観察による心拍数のカウントを行います。
- ステップ5で説明したように、HRソフトウェアを利用します。いくつかの試験片に対して手順を繰り返します。得られた心拍数の値が、目的の精度内ですべての個人で一貫しているかどうかを確認します。
- 以下に説明するように、モバイル個人および挑発テストを使用してテストを実行します。
- ステップ2で説明したように、麻酔されていない標本の画像スタックを取得します。シーケンスが記録された個人に対して、心拍数に影響を与える既知の刺激を使用して挑発テストを実施します。この研究では、5 mg / Lの濃度への水の塩類化の結果が示されています。
- 挑発試験後のステップ2で説明したのと同じ標本の画像スタックを取得します。ステップ5で説明したように、HRソフトウェアを使用した両方の実験で心拍数を測定します。観察された心拍数の変化が、挑発テストの既知の効果と一致しているかどうかを確認します。
結果
12 dpfでのゼブラフィッシュの心拍数は、上記のプロトコルを使用して決定しました(補足ビデオ1)。ビデオには、自由に泳ぐゼブラフィッシュの幼生の一連の画像、提案されたプロトコルを使用してこれらの配列から得られたフォトプレチスモグラム、およびフォトプレチスモグラムから計算された対応する心拍数が含まれています。
ラベル付けされたデータは、トレーニング中に 3:2:1 の比率でトレーニング、テスト、および検証セットにランダムに分割されました。次に、loss 関数は次の式に従って実装されました。

ここで、 x は入力ロジット、 y はターゲット、 バッチサイズは 同時に処理される画像の数、

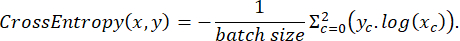
トレーニング中のモデルのパフォーマンスを評価するために、次のように定義される予測精度関数が使用されました。


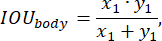
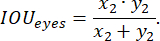
損失関数の開発には、たとえば25で詳述されているように、標準的なアプローチを適用しました。EfficientNet-B0モデルアーキテクチャを使用したマルチスケールアテンションネットワークは、エンコーダーとソフトマックス出力レイヤーとして使用されました。ニューラルネットワークは、0.02から始まり、0.99の減衰係数を持つ指数関数的に減衰する学習率を利用して、50エポックのテストサンプルでトレーニングされました。
訓練されたニューラルネットワークを使用して、任意のシーケンスの各フレームにおける個人のボディマスクとアイマスク(図5C)を取得しました。次に、アイマスクの重心を計算し、ボディマスクにスケルトン化を適用しました(図5D)。得られた魚の弦P上で、アイマスクの重心に最も近い位置にある点Mが同定されました(図5F)。点Mからの距離dで、目の間の距離に等しい点Nは、尾に向かって弦PPPに沿って識別されました。この点は心臓領域の中心に対応します。この領域は、この点を中心とする半径r = d x 0.3の円で描かれました(図5F)。
生のPPG信号は、記録されたすべてのフレームの心臓領域内の平均ピクセル数として計算されました(図5G)。次に、この生の信号をその平均値に正規化しました。続いて、信号は、4次設計、25dBの阻止帯域減衰、および12Hzの阻止帯域エッジ周波数を備えた2次チェビシェフタイプIIフィルタを使用してフィルタリングされました。これは、標準的な処理アルゴリズム26,27と一致しています(図6A)。心拍数は、フィルタリングされた信号のフーリエ変換の二乗係数のピークパワーに対応する1〜4 Hzの範囲内の周波数を特定することによって決定されました(図6A)。
式 (1) は、ハートの画像が約 20 ピクセル x 20 ピクセルであることを保証します。心臓の縦方向の寸法は、ゼブラフィッシュの全身の長さの約8%を占めると想定されています。心臓の画像が小さいと、結果として得られる信号には、有用な信号を上回る大きなノイズ成分が含まれていることが多く、 図6Bに示すように、心拍数の計算には適していません。
プロトコール適用の結果は、各個人の心臓セグメンテーションの安定性に大きく依存します。ステップ2〜5が成功裏に完了し、得られたデータがランダムな結果ではなく実際の生理学的プロセスを反映していることを示すために、対照実験を実施することが重要です。プロトコルの検証は、ステップ6で詳述した手順に従って実施しました。ステップ6.2で説明した試験では、挑発的な刺激として5 mg / Lの濃度で塩を水に加えました。各試験には、5匹のゼブラフィッシュの幼生(12 pdf)が含まれていました。アガロース型内の水温は23〜25°Cに維持されました。 その結果、最初のテストでは、視覚的評価と提案手法との差が3%を超えなかったことが示されています。2回目のテストでは、この方法で測定された心拍数が予想通り増加しました(図7)。

図1:概略図のワークフロー (A)スキームと(B)実験セットアップの外観 この図の拡大版を見るには、ここをクリックしてください。

図2:ゼブラフィッシュの幼生の画像。 (A)高品質および(B-D)低品質のゼブラフィッシュ幼生画像の例としては、(B)露出不足のフレーム、(C)露出オーバーのフレーム、(D)ぼやけたフレームがあります。画像は幼虫を強調するためにトリミングされています。スケールバーは1mmです。 この図の拡大版を見るには、ここをクリックしてください。

図3:代表的な画像。 (A)初期画像と(B)ラベル付き画像の例。スケールバーは1mmです。 この図の拡大版を見るには、ここをクリックしてください。
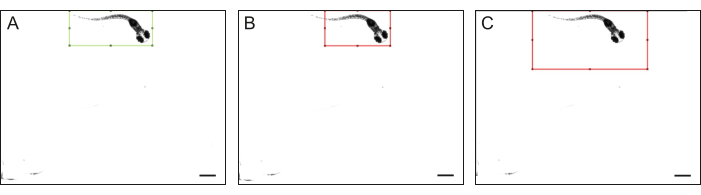
図4:ゼブラフィッシュの体外見。 ゼブラフィッシュの体全体の(A)高品質および(B-C)低品質のアウトラインの例:ゼブラフィッシュは長方形に完全に収まらず、長方形のサイズはゼブラフィッシュよりも著しく大きい。スケールバーは1mmです。 この図の拡大版を見るには、ここをクリックしてください。

図5:主要な処理ステージとそれに対応する中間結果を示す画像処理パイプライン。 この図の拡大版を表示するには、ここをクリックしてください。

図6:幼虫の心拍数を示すフレーム。 (A)高品質フレームと(B)直径がそれぞれ20ピクセルと14ピクセルの幼虫の心臓領域、および対応するPPGとPPGのフーリエスペクトルを持つフレームの例。スケールバーは1mmです。略語:PPG =光電式容積脈波記録法。 この図の拡大版を表示するには、ここをクリックしてください。
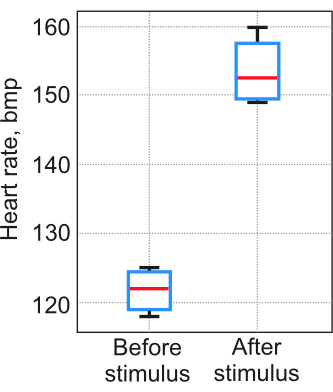
図7:心拍数の測定値。 測定値は、挑発刺激を用いた試験で提案された方法を用いて得られた。サンプル番号。ボックスは25パー センタイルと75パー センタイルにまたがり、水平線は中央値、黒いひげは外れ値とは見なされない最も極端なデータポイントを示していますこの 図の拡大版を表示するには、ここをクリックしてください。
補足ビデオ1: 心拍数の検出と計算のために提案されたニューラルネットワークアルゴリズムの図。ビデオは自由に動くゼブラフィッシュの幼生、提案されたプロトコルによって導き出され、光電式容積脈波の幼虫の心拍数から計算された光電式容積脈波を示しています このファイルをダウンロードするにはここをクリックしてください。
補足ファイル1:画像拡張を行うMATLAB関数。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
本研究では、自由に泳ぐゼブラフィッシュの幼生の心拍を測定するための実験プロトコルを提示する。このアプローチをいくつかの実験を通じて評価し、その有効性を実証しました。提案された方法の主要なコンポーネントには、ハードウェアとソフトウェアの両方のソリューションが含まれます。まず、イメージングに赤外線照明を使用したが、これは、以前に示したように、色素沈着に関連する問題を回避し、組織の透明性を高め、正確な心拍測定を容易にする18。次に、魚を下からイメージングすることを提案します。麻酔をかけると、ゼブラフィッシュの幼生は通常、遊泳用膀胱の位置により横向きになり、標準的な顕微鏡構成と倒立顕微鏡構成の向きの違いはごくわずかです。しかし、泳ぐ魚の心臓部は常に腹側に位置しているため、イメージングチャンネルを下に、照明チャンネルを上に配置することで、S/N比が向上します。最後に、ニューラルネットワークを使用して画像内の標本を特定し、個々の臓器を識別することにより、処理パフォーマンスが大幅に向上しました。私たちは、事前知識と経験的データの両方を用いて、ニューラルネットワークによって検出されたゼブラフィッシュの幼生の体内の心臓をピンポイントで特定する方法を開発しました。
このアプローチのその他の利点には、ハロゲンランプとは異なり、熱を発生しないLED光源の使用が含まれます。照明チャンネルをステージに固定すると、金型サイズに対する位置合わせが一度可能になり、ピントを合わせ直す際に追加の照明調整が不要になります。このアルゴリズムは、心臓セグメンテーションのためのニューラルネットワークのその後のトレーニングのための大規模なデータセットを生成するためにも適用できます。データ処理を高速化し、心臓の位置決定のエラーを最小限に抑えます。画像上で心臓に直接ラベルを付けることは、サイズが小さく、1つのフレーム内でその境界と位置を正確に決定するのが難しいため、大きな課題を提示します。
このプロトコルを実装する際に最適な結果を得るには、いくつかの重要な要素に対処する必要があります。まず、露光時間が長いと、特に個人の急速な動きの間に画像がぼやける可能性があるため、露光時間は1.5ミリ秒を超えないようにする必要があります。このようなぼやけは、特定の時点での正確な光電式容積脈波計算を妨げます(図6B)。次に、表面張力によりキュベット内の水面が湾曲し、ビデオキャプチャ中に結果として得られる画像に光散乱と歪みが生じます。この影響を軽減するには、薄く均一な水の層をアガロース表面に適用して、ペトリ皿全体の水位を平らで一定に保つ必要があります。アガロースは徐々に水分を吸収するため、安定して水位を保つために、約10分おきに水を加えることをおすすめします。最後に、レンズ倍率の選択は、開発段階と実験の特定の要件に合わせて調整する必要があります。適切な範囲内の固定倍率は、同じ年齢層内で均一な条件下で一貫した測定を行うのに十分です。ただし、可変倍率レンズは、さまざまな発達段階やタスクを含む研究に適しています。
テストでアルゴリズムが失敗することが判明した場合は、ステップ 2 と 3 を再検討して再実装する必要があります。ステップ3は、特に画像のラベリング中に、しばしば課題を提示します。目と体には注意深く注釈を付け、これら 2 つのクラスが重ならないようにしてください。さまざまな位置にいる個人、特に顕著な体の曲げを示す個人を捉える画像を使用して、ラベリングの精度を高めます。ステップ2は、照明の強度、露光時間、または倍率の問題が特定された場合に繰り返す必要があります。設定が正しくないと、信号対雑音比が低下し、ノイズが信号を圧倒する可能性があります(図6B)。さらに、トレーニングデータと実験データが異なる設定で取得された場合、アルゴリズムの失敗が発生する可能性があります。
提案されたハードウェアセットアップは、900〜1700nmの感度範囲の赤外線カメラを利用しています。ただし、このスペクトル範囲で動作する高解像度カメラは、多くの場合、法外なコストがかかります。この制限に対処するために、赤外線フィルターを取り外した後、センサー感度が近赤外線範囲にまで及ぶ産業用可視スペクトルビデオカメラを、赤外線カメラの費用対効果の高い代替品として採用できます。赤外線を使用することに加えて、色素沈着の影響は、色素除去化学物質を適用するか、色素沈着を欠く特定の遺伝系統の魚を使用することによっても最小限に抑えることができる28,29。現在のソフトウェアバージョンは、心拍数測定専用に設計されています。しかし、他の脈波パラメータは、包括的な研究にも同様に不可欠です。将来のソフトウェアアップグレードでは、さまざまな疾患の重要な指標である心拍変動分析の組み込みに重点が置かれるようになります。Q-T間隔などのより詳細な測定は、さらなる調査の対象です。さまざまな年齢や画像取得条件のデータで動作できるユニバーサルニューラルネットワークモデルを開発するには、トレーニングデータセットに、各タイプのラベル付き画像が少なくとも128個ある多様なサンプルを含める必要があります。
ゼブラフィッシュ4,6およびメダカ胚6,17の心臓領域の自動検出および心拍数モニタリングのために、これまでにいくつかのアプローチが開発されてきました。ゼブラフィッシュにおける心臓の蛍光標識は、心臓面積決定のために提案されている30。しかし、これまでに発表されたすべての方法は、胚の運動性が始まる前の短い受精後の期間に、動かない透明な胚を扱うことに限定されています。これは、これらの手法の適用性を低下させる重大な制限です。文献や補足資料に記載されている別の問題は、心拍数記録中の胚の突然の動きに関するものである6,17。このような動きは、心拍数計算のためにソフトウェアが目標とする心臓領域を変位させることができます。この研究で説明したアプローチは、これらの欠点に対処し、最大30 dpfの移動性ゼブラフィッシュのモニタリングを可能にします。
このアプローチの利点は、その潜在的なアプリケーションの可能性を大幅に拡大します。近年、Danio rerioは、心血管病理学および心毒性11,12,31を研究するための広範なモデルとなっている。この方法の能力は、麻酔薬を使用せずにさまざまな初期発達段階にわたって心拍数を非侵襲的に記録する能力であり、誘発された心臓奇形のダイナミクスと治療効果を研究するための新たな機会を提供します。今日では、ゼブラフィッシュ胚の心拍数モニタリングが前臨床試験の薬物スクリーニングに利用されている32。提案された方法の記載された利点は、薬物の遅延効果および薬力学を評価するための追加のツールを提供する。最後に、ゼブラフィッシュの心拍数の利用は、生態毒性学的モニタリング33,34でますます増加しています。この分野では、提案されたアプローチにより、低濃度の汚染物質の長期暴露期間にわたる慢性毒性の影響と、生物蓄積に関連する影響の評価が可能になります。
開示事項
すべての著者は、利益相反を開示しています。
謝辞
この研究は、STC UI RAS (FFNS-2025-0008) の連邦国家タスク プログラムの支援を受けました。この作業は、STC UI RAS [http:// https://ckp.ntcup.ru/en/] の共同利用センターの機器を使用して行われました。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
参考文献
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved