Method Article
자유롭게 움직이는 제브라피시의 마취 없는 심장 박동 측정
요약
이 연구는 움직이는 제브라피시에서 마취 없는 심장 박동 측정을 위한 기술을 개발하는 것을 목표로 합니다. 우리의 접근 방식은 단파 적외선 이미징과 기계 학습 기반 심장 추적을 결합합니다. 이는 비침습적이고 라벨이 없으며 사용자 친화적인 기술로, 제브라피시 모델에 대한 광범위한 연구에 적합합니다.
초록
제브라피시(Danio rerio)는 인간과 유전적 유사성과 투명한 배아 단계로 인해 생리학, 약리학 및 독성학 연구에서 널리 사용되는 모델 유기체로 비침습적 심혈관 연구를 용이하게 합니다. 그러나 현재 제브라피시의 심박수 평가 방법은 피험자를 움직이지 못하게 하기 위해 마취에 의존하는 경우가 많아 데이터 정확성과 재현성을 손상시키는 생리학적 변화가 발생합니다. 이 연구는 자유롭게 움직이는 제브라피시 유충의 심장 박동을 측정하기 위한 새로운 무마취 기술을 제시하여 심혈관 연구의 중요한 한계를 해결합니다. 제안된 접근 방식은 단파 적외선 이미징과 머신 러닝 기반 심장 추적을 통합하여 고정되지 않은 표본에서 정확하고 지속적인 심장 활동 모니터링을 가능하게 합니다. 심장 영역을 감지하기 위해 컨볼루션 신경망을 훈련시켰고, 심박수를 결정하기 위해 이미지 시퀀스에서 광혈류 신호를 추출했습니다. 실험적 검증은 여러 테스트 조건에서 방법의 신뢰성과 일관성을 입증했습니다. 이 방법론의 주요 이점은 제브라피시의 자연스러운 생리적 상태를 보존하여 스트레스로 인한 인공물을 최소화할 수 있다는 것입니다. 이 비침습적이고 라벨이 없는 기술은 심혈관 생리학, 약물 심장 독성 및 환경 독성학 연구에 상당한 이점을 제공하여 생물 의학 연구의 모델로서 제브라피시의 잠재적 응용 분야를 확장합니다.
서문
작은 사이프리니드 물고기인 제브라피시(Danio rerio)는 작은 크기, 높은 번식률, 유전자 조작의 용이성으로 인해 필수 모델 유기체가 되었습니다 1,2,3. 투명 제브라피시 배아의 심박수 평가는 생리학, 발생학, 독성학 및 기타 분야에서 점점 더 많이 활용되고 있습니다 4,5,6,7,8. 한편으로는, 제브라피시 게놈(genome)이 인간의 심혈관 질환과 관련된 유전자를 포함하고있으며9, 다니오 레리오 심장(Danio rerio heart)이 인간과 유사한 구조와 신호 경로를 공유한다는 사실에 기인합니다10,11. 이는 제브라피시를 심장 발달 및 질병 연구를 위한 귀중한 모델로 만듭니다 11,12,13. 반면에, 제브라피시 심박수는 외부 영향에 민감하기 때문에 처리된 물고기와 치료되지 않은 물고기의 심장 기능을 비교함으로써 생리학 및 독성학 연구를 위한 훌륭한 모델입니다 7,8,14.
투명한 어류 배아에서 심박수를 평가하기 위한 비침습적 광학 방법을 개발하는 데 상당한 진전이 이루어졌습니다15,16. 이러한 기법은 큰 표본 크기에서 데이터를 빠르게 수집할 수 있는 이점을 제공합니다. 그 결과, 어류 배아의 심박수 평가를 위한 완전 자동화된 접근법이 개발되었습니다 4,5,6,17.
그러나 현재 이러한 기술의 사용을 3-4 dpf 기간으로 제한하는 특정 제한 사항이 있습니다. 첫 번째 한계는 물고기 몸체의 색소 침착으로 인한 투명도 손실입니다. 두 번째는 시간이 지남에 따라 배아의 이동성이 증가한다는 것입니다. 광학 접근 방식을 사용할 수 있는 제브라피쉬의 초기 개발 기간을 연장하면 유용성이 향상되어 장기 실험 설계에서 심근병증, 선천성 심장 결함 및 시간 경과에 따른 영향의 역학 추적을 포함하여 심혈관계에 대한 다양한 영향을 연구할 수 있습니다. 우리 그룹은 최근 900-1700 nm의 단파 적외선 범위에서 이미징을 사용하여 투명도 손실 문제를 해결했습니다18. 이 논문은 배아 이동성 문제를 해결하는 데 중점을 둡니다.
일반적으로 트리카인 메탄설포네이트(MS-222)와 같은 마취제는 이미징 전에 자유롭게 수영하는 물고기 배아와 유충을 고정시키는 데 사용됩니다 14,19,20. 그러나 MS-222는 다른 마취제와 마찬가지로 심박수를 현저히 감소시킵니다21,22 . 관찰된 심장 기능의 변화가 실험적 치료에 의한 것인지, 마취제 때문인지, 아니면 둘 사이의 상호 작용에 의한 것인지 구분하기가 어려워집니다. 배아의 낮은 이동성 기간을 연장하는 또 다른 방법은 초기 발달 기간 동안 온도를 낮추는 것입니다8. 그러나 이 접근 방식은 연구 목표와 항상 양립할 수 있는 것은 아니며 등록 기간을 최소한으로 연장할 뿐입니다.
이 연구에서는 심박수 등록 중 배아 이동성을 해결하는 새로운 방법을 소개합니다. 우리는 자유롭게 헤엄치는 제브라피시 배아의 기록에서 심장의 관심 영역을 식별하기 위해 컨볼루션 신경망을 훈련시켰습니다. 이 영역 내에서 픽셀 강도의 주기적 변화는 광용적맥파(PPG)를 도출하는 데 사용되며, 이는 이후에 심박수를 계산하는 데 사용됩니다. 개발된 데스크톱 애플리케이션인 AutoHR은 신경망 교육과 이미지 스택 처리를 모두 활용하여 사용 편의성과 프로토콜 재현성을 보장했습니다.
프로토콜
제브라피쉬는 확립된 ZFIN 프로토콜24에 따라 사육되고 길러졌습니다. 모든 절차는 2024년 8월 21일자 러시아 과학 아카데미(STC UI RAS)의 고유 계측 과학 기술 센터(STC UI RAS)의 생명윤리 위원회(Bioethics Committee)의 승인을 받았으며 STC UI RAS의 제브라피시 관리 지침을 따릅니다. 개별 버전에 대한 설명서는 요청 시 제공됩니다.
1. 측정을 위한 장비 준비
- 장비 선택
- 광원 선택: 근적외선 범위, 특히 800nm에서 방출되는 LED 광원을 선택하고 최소 전력 출력은 3W입니다. 이 연구에서는 중심 파장이 940nm이고 대역폭이 40nm인 LED를 사용했습니다.
- 비디오 카메라 선택: 광원에서 방출되는 파장 범위에 민감한 센서가 장착되어 있고 프레임 속도가 60fps 이상인 카메라를 선택하십시오. 카메라의 데이터시트를 참조하여 픽셀 피치 p, 짧은 면 h를 따른 센서 크기 및 프레임 속도를 확인하십시오. 이 연구에서 카메라의 픽셀 피치는 p = 5μm이고 센서 너비는 h = 5.12mm입니다.
- 유충 B의 길이보다 최소 1.5배 큰 아가로스 곰팡이 크기를 선택하십시오. 제공된 표현식을 사용하여 광학 시스템의 최소 및 최대 허용 배율을 계산합니다. M최대 ≤ M 최소인지 확인하십시오. 이 조건이 충족되지 않으면 h가 더 크거나 S가 더 작거나 작은 카메라를 선택하십시오.
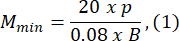
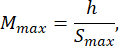
여기서 Smax 는 금형 세트의 가장 큰 아가로스 금형의 길이입니다. 다양한 크기의 금형 세트를 준비 할 수 있습니다. 이 연구에서는 S = 10mm를 사용했습니다.
참고: 방정식은 광학 구성표 형상과 제브라피쉬의 신체 비율을 기반으로 파생됩니다. 자세한 설명은 토론 섹션에서 확인할 수 있습니다. - 렌즈 선택: 이 연구는 MOSmin = 0.4x에서 MOSmax = 1.5x 범위의 다양한 배율, fmax = 150mm의 최대 초점 거리 및 mm 작동 거리를 가진 광학 시스템을 사용했습니다.
참고: 기호 x는 광학 시스템의 배율을 나타냅니다.- 허용 범위(Mmin - M max) 내에서 유지되는 배율 범위(MOSmin - MOSmax)를 가진 가변 배율 현미경 유형 시스템을 선택하십시오. 렌즈 데이터시트를 참조하여 MOSmax에 대한 최대 초점 거리 fmax와 해당 working distance L을 확인하십시오.
- 조건 fmax ≤ Mmax • L이 충족되는지 확인합니다. 이 조건이 충족되지 않으면 fmax 및/또는 L이 다른 렌즈를 선택하십시오. 카메라와 렌즈의 마운트 유형이 호환되는지 확인하십시오.
- 알려진 피치와 총 길이가 최소 인 테스트 개체(눈금자 또는 그리드)를 선택합니다. 알려진 피치와 총 크기가 최대 S 이상인 눈금자 또는 그리드 형태의 테스트 개체를 선택합니다.
- 이미지 획득을 위한 스탠드 조립(그림 1).
- 안정된 바닥에 둥근 막대를 장착합니다. 렌즈를 카메라에 부착하여 이미징 시스템을 조립합니다. 입력 창이 위쪽을 향하도록 하여 로드에 이미징 채널을 장착합니다.
- 포커싱 메커니즘을 로드에 장착합니다. 최소 직경 30mm의 유리 작업 표면이 있는 스테이지를 포커싱 메커니즘에 부착합니다.
- 조명기 조립: 효율적인 방열을 보장하기 위해 금속 기판에 LED를 놓습니다. 직경이 23mm이고 초점 거리가 30mm인 볼록 평면 콜리메이터 렌즈를 LED에서 30mm 거리에 배치합니다.
- 3축 조정 가능한 브래킷을 사용하여 조명기를 s에 장착하고 렌즈가 아래를 향하도록 하여 작업 표면 위에 배치합니다.
- 조명기와 이미징 시스템을 조정합니다. 조명기를 이미징 시스템과 동축으로 배치합니다. 조명을 켜고 카메라를 컴퓨터에 연결합니다.
- 카메라 응용 프로그램을 실행하고 사전을 활성화합니다.view 카메라 설명서의 지침에 따라 모드를 사용합니다. 테스트 개체를 스테이지에 놓습니다. 현미경의 작동 거리와 배율을 조정하여 크기가 시야에 완전히 맞도록 합니다.
참고: 크기는 아가로스 몰드의 완전한 시각화를 보장하기 위해 프레임의 짧은 면을 따라 완전히 맞아야 합니다. - 이미징 시스템을 기준으로 스테이지의 위치를 조정하여 이미지의 초점이 맞춰지고 조명기의 위치가 균일하게 조명되도록 합니다.
- 아가로스 곰팡이의 준비
- 증류수 20mL와 저융점 아가로스 0.4g을 섞어 아가로스가 완전히 녹을 때까지 혼합물을 가열합니다. 예를 들어, 전자 레인지에서 20W의 전력으로 700 초 동안.
- 아가로스 몰드 모양(방법 1): 가열된 용액을 페트리 접시에 붓고 용액 높이가 1.5-2mm가 되도록 합니다. 아가로스 용액이 굳을 때까지 페트리 접시를 식힙니다. 17°C의 온도에서 이 과정은 약 5분이 소요됩니다. 문구용 칼을 사용하여 한 변의 길이로 사각형을 자르고 윤곽이 그려진 영역 내의 아가로스 층을 조심스럽게 제거합니다.
- 아가로스 몰드 형성(방법 2): 플라스틱으로 (S) × (S) × 10mm 형태를 만듭니다. 정사각형 면이 있는 인쇄된 양식을 페트리 접시에 놓고 가열된 용액으로 채워 용액 높이가 1.5-2mm가 되도록 합니다. 아가로스 용액이 응고될 때까지 페트리 접시를 식히고 응고된 용액에서 형태를 조심스럽게 제거합니다.
참고: 양식은 3D 프린터를 사용하여 쉽게 인쇄할 수 있습니다.
2. 이미지 획득
- 이미지 캡처 설정을 조정합니다.
- 카메라를 컴퓨터에 연결합니다. 카메라 응용 프로그램을 실행하고 사전을 활성화합니다.view 모드. 조명기를 켭니다.
참고: 사용할 아가로스 몰드의 크기가 1.3.3 단계와 다른 경우 1.3.3 단계를 반복하고 그 후 단계에서 테스트 개체를 제거합니다. - 제브라 피쉬 유충을 파스퇴르 피펫으로 아가로스 틀에 넣습니다. 무대에 아가로스 몰드를 놓습니다. 노출 시간과 조명기 전력의 조합을 조정하여 유충 머리의 거의 완전한 조명을 보장합니다(그림 2A-C). 노출 시간은 1.5ms를 초과해서는 안 됩니다.
참고: 이 연구에서는 수정 후 12일에서 21일(dpf)까지 생후 12일에서 21일 사이의 야생형 제브라피시 유충에 대한 데이터를 제시합니다.
- 카메라를 컴퓨터에 연결합니다. 카메라 응용 프로그램을 실행하고 사전을 활성화합니다.view 모드. 조명기를 켭니다.
- 이미지 획득 수행
- 프레임 속도를 60fps 이상으로 설정합니다. 이미지 비트 심도를 12비트로 설정합니다. 최소 10초 동안 이미지를 획득합니다. 프레임 명명 형식을 yyyy_mm_dd_hh_ss_mm_mss로 설정하고 파일 형식은 BMP가 아닌 PNG 또는 TIFF여야 합니다.
3. 라벨링을 위한 신경망 훈련
- 라벨링을 위한 데이터 준비
참고: Image Labeler 앱 MATLAB에서 라벨링하는 과정에 대해 설명합니다.- 별도의 디렉터리에 256개 이상의 획득한 이미지를 수집합니다. 앱을 실행합니다. Import > From file(파일에서 가져오기 )로 이동하고 지정된 디렉터리에서 모든 이미지를 선택하여 이미지를 가져옵니다.
- 네트워크 레이블 지정을 위한 클래스를 만듭니다. ROI Labels 탭에서 Label을 클릭하고, Label Name 필드에 body를 입력하고, 라벨 유형에 대한 드롭다운 메뉴에서 Pixel Label 을 선택하고, OK를 클릭합니다. ROI Labels 탭에서 Label을 클릭하고, Label Name 필드에 eye를 입력하고, 레이블 유형에 대한 드롭다운 메뉴에서 Pixel Label 을 선택하고, OK를 클릭합니다. ROI Labels 탭에서 Label을 클릭하고, Label Name 필드에 fish를 입력하고, 레이블 유형에 대한 드롭다운 메뉴에서 Rectangle 을 선택하고, OK를 클릭합니다.
- 프레임의 라벨링
- ROI Labels 탭에서 이전에 생성한 주석 클래스 eye를 선택합니다. 마우스 왼쪽 버튼을 사용하여 표본의 둘레를 따라 점을 배치하여 표본의 한쪽 눈의 윤곽을 그립니다. 눈의 주석을 완성하려면 윤곽선의 시작점을 클릭하십시오(그림 3B).
- 3.1.2단계에서 설명한 것과 같은 방식으로 표본의 다른 눈의 윤곽을 그립니다.
- ROI Labels 탭에서 생성된 주석 클래스 body를 선택합니다. 눈을 제외한 표본의 몸체의 윤곽을 잡고, 마우스 왼쪽 버튼을 사용하여 표본의 둘레를 따라 점을 배치합니다. 눈의 주석을 완성하려면 윤곽선의 시작점을 클릭하십시오.
- ROI 레이블 탭에서 이전에 생성된 주석 클래스 fish를 선택합니다. 직사각형 경계 상자를 그려 표본의 전체 몸체를 윤곽을 그립니다(그림 4). 이렇게 하려면 마우스 왼쪽 버튼으로 원하는 사각형의 One Corner 를 클릭한 다음 사각형이 물고기를 완전히 둘러쌀 때까지 버튼을 누른 상태에서 커서를 드래그합니다.
- LABEL 탭에서 Export > To File 을 클릭하여 images라는 새 디렉토리에 개요를 저장합니다.
- 신경망 훈련을 위한 데이터셋 준비하기
- MATLAB을 실행합니다. 열기(Open) > 열기(Open)를 선택하여 스크립트 파일 DatasetsPreparation.m(Supplementary File 1)을 엽니다.
- 실행을 클릭하여 스크립트를 실행합니다. 팝업 창에서 윤곽선이 설정된 이미지가 포함된 디렉터리, 폴더 PixelLabelData, 3.2.5단계에서 내보낸 gTruth.mat 파일을 선택합니다.
참고: 스크립트 실행이 완료되면 RPN Training과 CNN Training이라는 두 개의 폴더가 자동으로 생성됩니다. 후속 신경망 학습에 필요합니다. 이러한 폴더의 내용을 수정하지 마십시오.
4. 심장 감지를 위한 신경망 훈련
참고: 이 단계는 특정 연령 및 이미징 상태에 대해 한 번 수행됩니다. NVIDIA GPU는 처리 속도를 크게 높이므로 교육에 강력히 권장됩니다.
- AutoHR 애플리케이션을 실행합니다. Region Proposal Network Training(지역 제안 네트워크 교육) 탭으로 이동합니다. 디렉토리 선택을 클릭하고 3.3.2단계에서 생성한 RPN 교육 폴더를 선택합니다. Start Training(교육 시작)을 클릭합니다.
참고: 학습이 완료되면 AutoHR.exe 파일과 동일한 디렉터리에 rpn_model.pt라는 모델 파일이 생성됩니다. 이 파일은 추가 이미지 스택 처리에 필요합니다. - Convolutional Neural Network를 훈련시키려면 Convolutional Neural Network Training 탭으로 이동합니다. 디렉터리 선택을 클릭하고 3.3.2단계에서 만든 CNN 교육 폴더를 선택합니다. Start Training(교육 시작)을 클릭합니다.
참고: 학습이 완료되면 AutoHR.exe 파일과 동일한 디렉터리에 model.pt 라는 모델 파일이 생성됩니다. 이 파일은 추가 이미지 스택 처리에 필요합니다.
5. 심박수 정량화
- 2단계에서 설명한 대로 일련의 이미지를 기록합니다. 배율, 노출 시간, 조명과 같은 이미징 조건이 신경망 훈련에 사용되는 것과 동일한지 확인합니다.
- AutoHR 애플리케이션을 실행합니다. 처리 탭으로 이동합니다. Choose Models(모델 선택)를 클릭하여 학습된 신경망 모델이 포함된 디렉터리를 선택합니다. Choose Folder 를 클릭하여 분석을 위해 획득한 이미지를 가져옵니다. 내보내기 경로는 자동으로 지정되지만 내보내기 경로 선택을 클릭하여 변경할 수 있습니다. 프로세스를 클릭하여 해석을 시작합니다.
참고: 완료되면 심박수 값이 AutoHR 인터페이스의 심박수 필드에 나타납니다. 내보낸 폴더에는 심박수 값(.xlsx), PPG(.xlsx), 시퀀스의 첫 번째 프레임, 첫 번째 프레임(.png)의 바디 및 아이 마스크가 포함됩니다.
6. 알고리즘 결과 검증
- 아래 설명된 대로 움직이지 않는 물고기로 테스트를 수행합니다.
- 0.168 mg/L MS-222 용액으로 유충을 1분 동안 마취합니다. 2단계에서 설명한 대로 마취된 표본의 이미지 스택을 획득합니다. 육안 관찰을 통해 심박수 수를 계산합니다.
- 5단계에서 설명한 대로 HR 소프트웨어를 활용합니다. 여러 시편에 대해 단계를 반복합니다. 얻은 심박수 값이 원하는 정확도 내에서 모든 개인에 대해 일관되는지 확인합니다.
- 아래 설명된 대로 모바일 개인 및 도발 테스트로 테스트를 수행합니다.
- 2단계에서 설명한 대로 마취되지 않은 표본의 이미지 스택을 획득합니다. 심박수에 영향을 미치는 알려진 자극을 사용하여 시퀀스가 기록된 개인과 함께 도발 테스트를 수행합니다. 이 연구는 5mg/L의 농도로 물 염분화에 대한 결과를 제시합니다.
- 도발 테스트 후 2단계에서 설명한 것과 동일한 표본의 이미지 스택을 획득합니다. 5단계에 설명된 대로 HR 소프트웨어를 사용한 두 실험 모두에서 심박수를 확인합니다. 관찰된 심박수 변화가 유발 테스트의 알려진 효과와 일치하는지 확인합니다.
결과
12 dpf에서 제브라피쉬의 심박수는 위에서 설명한 프로토콜을 사용하여 결정되었습니다(보충 비디오 1). 비디오에는 자유롭게 헤엄치는 제브라피시 유충의 이미지 시퀀스, 제안된 프로토콜을 사용하여 이러한 시퀀스에서 파생된 광용류측정, 광용적맥파에서 계산된 해당 심박수가 포함되어 있습니다.
레이블이 지정된 데이터는 훈련 중에 3:2:1 비율로 훈련, 테스트 및 검증 세트로 무작위로 분할되었습니다. 그런 다음 손실 함수는 다음 표현식에 따라 구현되었습니다.

여기서 x 는 입력 로짓, y 는 목표값, 배치 크기는 동시에 처리되는 이미지의 수입니다.

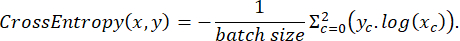
훈련 중 모델의 성능을 평가하기 위해 다음과 같이 정의된 예측 정확도 함수가 사용되었습니다.


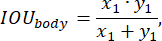
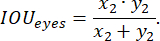
우리는 손실 함수의 개발에 표준 접근 방식을 적용했으며, 예를 들어25에 자세히 설명되어 있습니다. EfficientNet-B0 모델 아키텍처를 사용하는 Multi-scale Attention Network가 인코더 및 소프트맥스 출력 계층으로 사용되었습니다. 신경망은 0.02에서 시작하여 0.99의 감쇠 계수를 가진 기하급수적으로 감소하는 학습률을 활용하여 50개의 epoch 동안 테스트 샘플에 대해 훈련되었습니다.
훈련된 신경망을 사용하여 임의의 시퀀스의 각 프레임에서 개인의 바디 마스크와 아이 마스크(그림 5С)를 얻었습니다. 그런 다음 아이 마스크의 질량 중심을 계산하고 바디 마스크에 스켈레톤화를 적용했습니다(그림 5D). 그 결과 생성된 물고기 현 P에서 아이 마스크의 질량 중심에 가장 가까운 점 M이 식별되었습니다(그림 5F). 눈 사이의 거리와 동일한 점 M으로부터의 거리 d에서, 점 N은 꼬리를 향한 현 PPP를 따라 식별되었습니다. 이 지점은 심장 영역의 중심에 해당합니다. 이 영역은 이 지점을 중심으로 반지름이 r = d x 0.3인 원으로 표시되었습니다(그림 5F).
원시 PPG 신호는 기록된 모든 프레임에서 심장 영역 내의 평균 픽셀 수로 계산되었습니다(그림 5G). 그런 다음 이 원시 신호를 평균값으로 정규화했습니다. 그 후, 신호는 표준 처리 알고리즘26,27과 일치하는4차 설계, 25dB 저지대역 감쇠 및 12Hz의 저지대역 에지 주파수를 갖는 2차 체비쇼프 유형 II 필터를 사용하여 필터링되었습니다. 심박수는 필터링된 신호의 푸리에 변환의 제곱 계수의 피크 전력에 해당하는 1-4Hz 범위 내의 주파수를 식별하여 결정되었습니다(그림 6A).
방정식 (1)은 심장 이미지가 약 20픽셀 x 20픽셀임을 보장합니다. 심장의 세로 치수는 제브라피쉬의 전체 몸 길이의 약 8%를 차지하는 것으로 가정합니다. 심장 영상이 더 작을 경우, 결과 신호에는 유용한 신호를 능가하는 상당한 잡음 성분이 포함되는 경우가 많아 그림 6B와 같이 심박수 계산에 적합하지 않습니다.
프로토콜 적용의 결과는 각 개인의 심장 세분화 안정성에 따라 크게 달라집니다. 2-5단계가 성공적으로 완료되었으며 얻은 데이터는 무작위 결과가 아닌 실제 생리학적 과정을 반영한다는 것을 입증하기 위해 대조 실험을 수행하는 것이 중요합니다. 프로토콜 검증은 6단계에서 자세히 설명된 절차에 따라 수행되었습니다. 6.2단계에서 설명한 테스트에서 도발적인 자극으로 5mg/L의 농도로 물에 소금을 첨가했습니다. 각 테스트에는 5마리의 제브라피시 유충(12pdf)이 포함되었습니다. 아가로스 주형의 수온은 23-25°C로 유지되었습니다. 결과는 첫 번째 테스트에서 시각적 평가와 제안된 방법의 차이가 3%를 초과하지 않았음을 나타냅니다. 두 번째 테스트에서는 이 방법으로 측정한 심박수가 예상대로 증가했습니다(그림 7).

그림 1: 회로도 워크플로우. (A) 체계 및 (B) 실험 설정의 모양 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 제브라피시 유충 이미지. (A) 고품질 및 (B-D) 저품질 제브라피시 유충 이미지의 예로는 (B) 노출 부족 프레임, (С) 노출 과다 프레임, (D) 흐릿한 프레임이 있습니다. 유충을 강조하기 위해 이미지가 잘렸습니다. 눈금 막대는 1mm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 대표 이미지. (A) 초기 이미지와 (B) 레이블이 지정된 이미지의 예. 눈금 막대는 1mm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
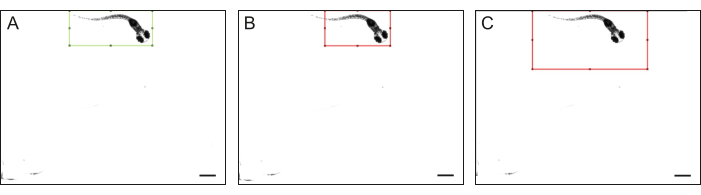
그림 4: 제브라피시 몸체 윤곽. 전체 제브라피시 몸체의 (A) 고품질 및 (B-C) 저품질 윤곽선의 예: 제브라피쉬는 사각형 안에 완전히 맞지 않으며 사각형의 크기가 제브라피쉬보다 눈에 띄게 큽니다. 눈금 막대는 1mm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: 기본 처리 단계와 해당 중간 결과를 보여주는 이미지 처리 파이프라인. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: 유충의 심박수를 보여주는 프레임. (A) 직경이 각각 20픽셀과 14픽셀인 유충 심장 영역과 PPG의 해당 PPG 및 푸리에 스펙트럼을 가진 고품질 및 (B) 저품질 프레임의 예. 눈금 막대는 1mm입니다. 약어: PPG = 광용적맥파. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
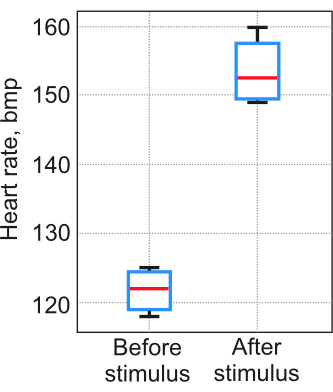
그림 7: 심박수 측정. 측정값은 도발 자극이 있는 테스트에서 제안된 방법을 사용하여 얻어졌습니다. 표본 번호 . 상자는 25번째 와 75번째 백분위수에 걸쳐 있고, 수평선은 중앙값이며, 검은색 수염은 이상값으로 간주되지 않는 가장 극단적인 데이터 포인트를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 비디오 1: 심박수를 감지하고 계산하기 위해 제안된 신경망 알고리즘의 그림. 이 비디오는 자유롭게 움직이는 제브라피시 유충을 보여주며, 제안된 프로토콜에 의해 파생되고 광용류매각 유충의 심박수에서 계산된 광용류측정법입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: 이미지 증강을 수행하는 MATLAB 함수. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 연구에서는 자유롭게 수영하는 제브라피시 유충의 심장 박동을 측정하기 위한 실험 프로토콜을 제시합니다. 우리는 여러 가지 실험을 통해 이 접근 방식을 평가하여 그 효과를 입증했습니다. 제안된 방법의 핵심 구성 요소에는 하드웨어 및 소프트웨어 솔루션이 모두 포함됩니다. 첫째, 이미징을 위해 적외선 조명을 사용했는데, 이는 앞서 설명한 바와 같이 색소 침착과 관련된 문제를 피하고 조직 투명도를 향상시켜 정확한 심장 박동 결정을 용이하게 합니다18. 둘째, 아래에서 물고기를 이미징하는 것을 제안합니다. 마취 시 제브라피시 유충은 일반적으로 부레의 위치로 인해 옆으로 누워 있기 때문에 표준 현미경과 도립 현미경 구성 간의 방향 차이는 무시할 수 있습니다. 그러나 헤엄치는 물고기의 심장은 항상 복부에 위치하기 때문에 이미징 채널을 아래에 배치하고 조명 채널을 위에 배치하면 신호 대 잡음비가 향상됩니다. 마지막으로, 신경망을 사용하여 이미지 내에서 표본을 찾고 개별 장기를 식별함으로써 처리 성능을 크게 개선했습니다. 사전 지식과 경험적 데이터를 모두 사용하여 신경망에서 감지된 제브라피시 유충 몸체의 심장을 정확히 찾아내는 방법을 개발했습니다.
이 접근 방식의 다른 장점으로는 할로겐 램프와 달리 열을 발생시키지 않는 LED 광원을 사용한다는 것입니다. 무대에 조명 채널을 고정하면 금형 크기를 기준으로 한 번 정렬할 수 있으므로 다시 초점을 맞출 때 추가 조명 조정이 필요하지 않습니다. 이 알고리즘은 또한 심장 분할을 위한 신경망의 후속 훈련을 위한 대규모 데이터 세트를 생성하는 데 적용할 수 있습니다. 데이터 처리를 가속화하고 심장 위치 결정의 오류를 최소화합니다. 이미지에 하트를 직접 라벨링하는 것은 크기가 작고 단일 프레임 내에서 경계와 위치를 정확하게 결정하기 어렵기 때문에 상당한 도전이 됩니다.
이 프로토콜을 구현할 때 최적의 결과를 얻기 위해 몇 가지 중요한 요소를 해결해야 합니다. 첫째, 노출 시간이 길면 특히 개인의 빠른 움직임 중에 이미지가 흐려질 수 있으므로 노출 시간이 1.5ms를 초과해서는 안 됩니다. 이러한 흐릿함은 특정 시점에서 정확한 광용류량 계산을 방해합니다(그림 6B). 둘째, 표면 장력으로 인해 큐벳의 물 표면이 구부러져 비디오 캡처 중에 결과 이미지에서 광 산란 및 왜곡이 발생합니다. 이 효과를 완화하려면 아가로스 표면 위에 얇고 균일한 물층을 적용하여 페트리 접시 전체에 걸쳐 평평하고 일관된 수위를 보장해야 합니다. 아가로스는 서서히 물을 흡수하기 때문에 안정적인 수준을 유지하기 위해 약 10분마다 물을 추가하는 것이 좋습니다. 마지막으로, 렌즈 배율의 선택은 개발 단계와 실험의 특정 요구 사항에 맞게 조정되어야 합니다. 적절한 범위 내의 고정 배율은 동일한 연령 그룹 내에서 균일한 조건에서 일관된 측정을 위해 충분합니다. 그러나 가변 배율 렌즈는 다양한 발달 단계 또는 작업과 관련된 연구에 더 적합합니다.
테스트 결과 알고리즘이 실패한 것으로 확인되면 2단계와 3단계를 다시 검토하고 다시 구현해야 합니다. 3단계는 특히 이미지 라벨링 중에 문제가 발생하는 경우가 많습니다. 눈과 몸에 대한 주의 깊은 주석을 확인하여 이 두 클래스 간의 겹침을 피하십시오. 다양한 위치에 있는 개인, 특히 뚜렷한 신체 구부러짐을 보이는 이미지를 캡처하는 이미지를 사용하여 라벨링 정확도를 높입니다. 조명 강도, 노출 시간 또는 배율 문제가 식별된 경우 2단계를 반복해야 합니다. 잘못된 설정은 신호 대 잡음비를 줄여 잡음이 신호를 압도할 수 있습니다(그림 6B). 또한 서로 다른 설정에서 학습 및 실험 데이터를 얻는 경우 알고리즘 오류가 발생할 수 있습니다.
제안된 하드웨어 설정은 감도 범위가 900-1700nm인 적외선 카메라를 사용합니다. 그러나 이 스펙트럼 범위에서 작동하는 고해상도 카메라는 종종 비용이 많이 듭니다. 이러한 한계를 극복하기 위해, 적외선 필터를 제거한 후 근적외선 범위로 센서 감도가 확장되는 산업용 가시 스펙트럼 비디오 카메라를 적외선 카메라의 비용 효율적인 대안으로 사용할 수 있습니다. 적외선을 사용하는 것 외에도 색소 제거 화학 물질을 적용하거나 색소 침착이 없는 특정 유전 계통의 물고기를 사용하여 색소 침착의 영향을 최소화할 수 있습니다28,29. 현재 소프트웨어 버전은 심박수 측정 전용으로 설계되었습니다. 그러나 다른 맥파 매개변수도 포괄적인 연구에 똑같이 필수적입니다. 향후 소프트웨어 업그레이드는 다양한 질병의 핵심 지표인 심박 변이도 분석을 통합하는 데 중점을 둘 것입니다. Q-T 간격과 같은 보다 자세한 측정은 추가 조사 대상입니다. 다양한 연령 및 이미지 획득 조건의 데이터로 작동할 수 있는 범용 신경망 모델을 개발하려면 훈련 데이터셋에 각 유형의 레이블이 지정된 이미지가 128개 이상 있는 다양한 샘플이 포함되어야 합니다.
제브라피시4,6 및 메다카 배 아 6,17에서 심장 부위의 자동 감지 및 심박수 모니터링을 위한 여러 접근 방식이 이전에 개발되었습니다. 제브라피시의 심장에 대한 형광 표지는 심장 면적 측정을 위해 제안되었습니다30. 그러나 이전에 발표된 모든 방법은 배아 운동성이 시작되기 전의 짧은 수정 후 기간 동안 움직이지 않고 투명한 배아를 대상으로 하는 것으로 제한됩니다. 이는 이러한 기술의 적용 가능성을 줄이는 중요한 제한 사항입니다. 문헌 및 보충 자료에 설명된 또 다른 문제는 심박수 기록 중 배아의 갑작스러운 움직임에 관한것입니다 6,17. 이러한 움직임은 심박수 계산을 위해 소프트웨어가 목표로 하는 심장 영역을 대체할 수 있습니다. 이 연구에서 설명한 접근 방식은 이러한 단점을 해결하여 최대 30dpf의 이동식 제브라피시를 모니터링할 수 있습니다.
이 접근 방식의 장점은 잠재적인 응용 가능성의 가능성을 크게 확장합니다. 최근 몇 년 동안 다니오 레리오는 심혈관 병리학 및 심장독성을 연구하기 위한 광범위한 모델이 되었다 11,12,31. 마취제 없이 다양한 초기 발달 단계에 걸쳐 심박수를 비침습적으로 기록하는 이 방법의 능력은 유도된 심장 기형 및 치료 효과의 역학을 연구할 수 있는 새로운 기회를 제공합니다. 요즘에는 제브라피시 배아의 심박수 모니터링이 전임상 연구에서 약물 스크리닝에 사용됩니다 32. 제안된 방법의 설명된 이점은 약물의 지연 효과 및 약력학을 평가하기 위한 추가 도구를 제공합니다. 마지막으로, 생태독성학적 모니터링에서 제브라피시 심박수의 활용은 계속 증가하고 있습니다33,34. 이 분야에서 제안된 접근 방식은 장기간의 노출 기간 동안 저농도 오염 물질의 만성 독성 영향 및 생물 축적과 관련된 영향을 평가할 수 있습니다.
공개
모든 저자는 이해 상충을 공개했습니다.
감사의 말
이 연구는 STC UI RAS(FFNS-2025-0008)의 연방 국가 과제 프로그램의 지원을 받았습니다. 이 작업은 STC UI RAS [http:// https://ckp.ntcup.ru/en/ 의 집합적 사용 센터(Center for Collective Use of STC UI RAS)의 장비를 사용하여 수행되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
참고문헌
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유