Method Article
생체 내 부분적으로 구속된 유충 제브라피시에서 감각 자극에 의해 유도된 신경 활동의 공초점 형광 이미징
요약
여기에서는 컨포칼 현미경을 사용하여 GCaMP 칼슘 지표를 발현하는 형질전환 제브라피쉬의 뇌 영역에서 신경 활동을 조사하기 위한 자세한 프로토콜을 제시합니다.
초록
제브라피시 유충은 행동의 신경 메커니즘을 연구하기 위한 유망한 척추동물 모델 시스템입니다. 그들의 반투명함과 상대적으로 단순한 신경 회로는 행동의 세포 분석에서 광유전학 기술의 사용을 용이하게 합니다. GCaMP6와 같은 생체 내 신경 활동의 형광 지표는 유충 제브라피시의 간단한 행동과 관련된 신경 활동을 연구하는 데 널리 사용되었습니다. 여기에서는 형질전환 계통 Tg(elav3:GCaMP6s)를 사용하여 반억제된 제브라피시 유충에서 감각 유도 활동을 감지하기 위한 프로토콜을 제시합니다. 특히, 우리는 화학 작용제인 알릴 이소티오시아네이트를 사용하여 후뇌와 척수 경계에 있는 뇌 영역에서 강력하고 재현 가능한 형광 반응을 유도합니다. 우리는 다양한 행동 패러다임에서 신경 활동의 광학 모니터링을 위한 GCaMP6의 잠재적인 사용과 이 기술의 한계에 대해 논의합니다. 우리의 프로토콜은 유충 제브라피시 뇌에서 역동적이고 행동과 관련된 생체 내 신경 활동을 모니터링하기 위한 접근 가능한 접근 방식을 간략하게 설명합니다.
서문
제브라피쉬는 상세한 세포-분자 신경생물학적 연구를 위한 다루기 쉬운 척추동물 모델을 나타냅니다. 유충 제브라피쉬는 수정 후 5일(dpf)에 ~100,000개의 뉴런을 가지고 있으며, 이는 포유류의 뇌보다 현저히 적습니다. 또한, 제브라피쉬는 상대적으로 반투명하여 신경 구조 및 기능에 대한 광학 연구를 용이하게 하는 특성입니다 1,2,3,4,5. 제브라피쉬에 사용하기 위해 고충실도 칼슘 지표6, 전압 센서 7,8, 신경 활동의 활동 의존적 마커 9,10,11,12,13 등 여러 광유전학 도구가 개발되었습니다. 이러한 도구는 유전자 변형에 대한 편의성 14,15,16,17 및 제브라피시 유충이 목욕 용액에 존재하는 화학 물질을 흡수할 준비성 18,19,20,21과 같은 이 모델이 가지고 있는 다른 이점을 보완합니다.
제브라피시 광학 생리학에는 다양한 방법, 특히 이광자, 광시트 및 컨포칼 현미경 검사에 유용합니다. 이러한 각 기술은 해상도의 두 가지 관련 문제, 즉 주변 조직에 의한 광 산란을 포함한 광학 액세스와 특히 밀리초 미만의 규모에서 활동 전위 역학을 캡처하기 위한 샘플링 속도의 균형을 맞춰야 합니다22. 이광자 현미경을 사용하여 생체 내 칼슘 이미징이 극적으로 개선되었지만, 이 방법은 종종 <1mm2의 시야로 제한되며 일반적으로 단일 깊이 평면만 획득할 수 있으므로 신경 회로22의 넓은 영역에 걸친 활동 캡처가 제한됩니다. 광시트 현미경의 경우 뇌의 거의 모든 뉴런의 활동을 기록할 수 있는 잠재력은 이광자 현미경 검사의 시야 제한을 해결하지만, 현재 카메라 속도는 유충 제브라피시 1,23의 뇌 부피당 40평면에서 초당 약 3개의 뇌 부피로 캡처를 물리적으로 제한합니다. 컨포칼 현미경 검사는 이광자 및 광시트 현미경 검사보다 깊이 해상도와 캡처 속도 모두에서 열등합니다. 컨포칼 현미경 검사는 전 세계 실험실에 대한 광범위한 접근성과 cFos 및 p-ERK9와 같은 신경 활동 리포터를 사용하여 신경 활동의 전체 뇌 재구성을 달성할 수 있는 장점이 있습니다. 또한 작은 뇌 영역이 표적이 되는 경우 컨포칼 현미경은 신경 활동의 적절한 시간 해상도를 제공할 수 있습니다.
본 논문은 컨포칼 현미경을 사용하여 GCaMP6를 범신경으로 발현하는 형질전환 제브라피시의 신경 활동을 기록하는 방법을 설명합니다. 신경 경로 24,25,26,27,28,29의 기능을 이해하기 위해 제브라피시 유충을 사용하는 몇 가지 유사한 프로토콜이 개발되었습니다. 타임 랩스 이미징, 칼슘 역학의 형광 지표 및 라이브 이미징과 같은 이러한 프로토콜 중 일부의 주요 기능은 혐오성 화학 자극 물질인 AITC(Allyl Isothiocyanate)에 반응하여 제브라피시 중추 신경계의 소규모 뉴런 집단에서 신경 활동을 측정하기 위해 결합되었습니다 11,26,27,29,30,31. AITC는 후뇌 영역에 집중된 뇌 전반의 반응을 이끌어낸다11. 뒷뇌의 꼬리 바로 옆에 있는 뉴런의 한 무리는 운동과 AITC에 대한 장기적인 반응에 중요한 역할을 합니다. 이 반응은 혐오적 자극이 제거된 것보다 더 오래 지속된다30. 시야를 제한함으로써 우리는 GCaMP6를 발현하는 뉴런의 형광 변화에 의해 반영된 이 신경 클러스터의 신경 활동을 감지하는 데 성공했습니다. 우리는 컨포칼 현미경을 사용하여 충분한 시공간 해상도를 달성하기 위한 기술, 지침 및 모범 사례를 제공합니다. 또한 광학 기록 방법의 한계에 대해 논의합니다. 이러한 한계에도 불구하고 이 방법은 기억 및 감각 운동 처리를 포함한 다양한 신경생물학적 현상을 조사할 수 있어야 합니다.
프로토콜
동물을 사용하는 모든 절차는 풀러턴에 있는 캘리포니아 주립대학교의 기관 동물 관리 사용 위원회(프로토콜 # 2023-1310)의 승인을 받았습니다.
1. 저융점 아가로스에서 유충 제브라피시 스테이징
- 기관 동물 관리 지침에 따라 2명의 수컷에서 2명의 암컷으로 범신경으로 발현하는 Tg(elav3:GCaMP6s)6의 성체 형질전환 동물을 사육합니다. 성체는 특수 사육실에 배치되어 방출된 알은 성인이 접근할 수 없는 가짜 바닥이 있는 아래쪽 방에서 포획됩니다. 처음 밝을 때나 분리 칸막이가 제거되면 암컷은 알을 낳고 수컷은 가짜 바닥에 붙잡힌 알을 수정시킵니다.
- 성체를 제거하고 난자 여과기를 사용하여 수정란을 채취하고 배아(E3) 배지(5mM NaCl, 0.33mM MgSO4, 0.33mM CaCl2, 0.17mM KCl, 1mM HEPES, 0.00001% 메틸렌 블루, pH 7.0)30 , 인큐베이터 내부 28.5°C에서 14시간 조명/10시간 어둡게 놓습니다.
참고: 사용된 E3 레시피는 형광에 영향을 미치거나 중요한 자동 형광 신호를 유발하지 않는 메틸렌 블루의 농도가 낮습니다. GCaMP6s 형질전환 라인이 제공하는 자연스러운 반투명으로 인해 프로토콜에 명시된 발달 연령에서 1-페닐 2-티오우레아(PTU)의 사용을 요구하지 않습니다. - E3 배지에 용해된 3% 저융점 아가로스를 사용하여 수정 후 2-7일(dpf)에 동물을 장착합니다. 열은 제브라피쉬가 유기체에 대한 손상 없이 조작될 수 있는 온도로 아가올라갑니다. 낮은 융점 아가로스가 냉각되어 응고되기 전에(<1분) 그림 1과 같이 검사할 뇌 영역이 대물렌즈에 최대한 가깝도록 바닥이 유리인 페트리 접시에 유충의 등쪽이 위로 향하게 놓습니다. 실험에 따라 다르지만 일반적으로 실험 그룹당 8-10마리의 제브라피시로 충분합니다.
참고: 이 프로토콜에서 동물은 마취되지 않았습니다. - 아가로스가 유충과 함께 굳은 후(약 2-3분) E3 용액 5mL를 추가합니다. 그런 다음 실험 프로토콜에서 요구하는 대로 메스로 아가로스를 자릅니다.
참고: 제브라피쉬의 어떤 부분이 구속에서 벗어나거나 다른 유형의 자극을 받아야 하는지에 따라 한천의 다른 부분을 잘라내야 합니다. - 제브라피쉬가 들어 있는 페트리 접시를 컨포칼 현미경 스테이지로 옮기고 물고기가 20분 동안 적응하도록 합니다.
2. 자극 적용을 사용한 컨포칼 현미경 검사에서 설정 및 이미징
- 적응 기간이 끝나면 광원(~475nm)이 있는 명시야를 사용하여 실온에서 40x(NA 1.0) 침수 대물렌즈 아래에 물고기를 중앙에 놓습니다.
- 컨포칼 현미경 획득 설정은 형광 분자의 품질 및 발현 수준, 조직의 깊이 및 광학적 특성, 시야의 크기, 기록 속도에 따라 달라집니다. 실험적 요구 사항에 따라 방출된 빛을 최적으로 포착할 수 있도록 범위 표시기를 사용하여 적절한 설정을 결정합니다.
- 레이저 출력과 마스터 게인을 적절하게 조정하여 최적의 범위에서 형광등을 캡처함으로써 GCaMP6s 의존 형광 신호의 불필요한 손실을 방지합니다. 아래에 제공된 주요 공초점 설정 및 요인 목록을 사용하여 적절한 설정을 결정하십시오.
- 핀홀 크기: 사용된 대물렌즈에 따라 이를 결정하고 다른 초점면을 제한하면서 단일 초점면을 수용하도록 설정합니다. 이 프로토콜에는 32μm의 핀홀이 사용됩니다.
- 레이저 출력 및 파장: 형광 분자의 발현 수준과 조직의 깊이 및 광학 특성에 따라 레이저 출력을 조정합니다. 범위 표시기를 사용하여 레이저가 광검출기의 범위를 초과하지 않는지 확인하십시오. excitation 파장을 형광 분자를 최적으로 여기하는 빛의 파장으로 설정합니다. 이 실험에서는 물고기가 GCaMP6s 전이유전자에 대해 이형접합적이었고 발현 수준이 낮았기 때문에 488nm 파장에서 5% 레이저 출력, 650 마스터 게인을 사용했습니다. 강력한 전이유전자 발현으로 레이저 출력 범위는 1.5%에서 3%이며 이는 일반적으로 충분합니다.
참고: 레이저 출력을 너무 낮게 설정하면 정보(광전자 증배관에서 감지된 빛)가 손실됩니다. 레이저 출력을 너무 높게 설정하면 방출되는 빛이 감지기의 범위를 초과하거나 샘플(광표백제)을 손상시킬 수 있습니다. - 마스터 게인: 마스터 게인은 광 검출기의 감도를 결정합니다. 범위 표시기를 사용하여 마스터 게인이 너무 높게 설정되어 광검출기의 범위를 초과하지 않는지 확인하십시오. 이 실험에서는 마스터 게인을 650으로 설정합니다.
- 분야의 view: 관심 있는 조직을 캡처할 수 있을 만큼 시야가 충분히 큰지 확인합니다. 이는 일반적으로 컨포칼 현미경의 주요 제한 사항인데, 그림 2에서 볼 수 있듯이 시야의 크기가 캡처 속도를 제한하기 때문입니다. AITC와 관련된 실험의 경우 시야를 79.86μm2로 설정합니다.
- 캡처 속도: 캡처 속도가 빨라지면 광검출기가 포착하는 빛의 양이 줄어듭니다. 캡처되는 빛이 적을수록 이미지 품질이 저하됩니다. 캡처 속도가 이미지 품질과 관심 있는 생리학적 이벤트를 캡처하는 데 필요한 속도의 균형을 유지하는지 확인합니다. 여기서 캡처 속도는 1.20 f/s로 설정되어 있습니다.
- 시야를 척수의 상부 부분에 있는 commissura infima Halleri의 꼬리 신경 클러스터30의 중앙에 놓습니다. 이 클러스터는 ~2.5 dpf로 매우 작지만 나중에 개발(7 dpf)에서 훨씬 더 큽니다.
참고: 1단계에서 아가로스 장착이 최적화되지 않은 경우, 즉 동물이 아가로스에 비스듬히 위치하거나 너무 깊숙이 위치하면 이미지 품질이 저하됩니다. 그림 3은 잘 배치된 동물의 제브라피시 척수 이미지와 최적이 아닌 위치에 있는 제브라피쉬의 척수 대조 이미지를 보여줍니다. - 픽셀당 0.119μm2의 공간 해상도로 1.20fps(초당 프레임)에서 79.86μm2의 시야를 사용하여 시계열 스캔을 수행합니다. 원하는 응용 프로그램에 따라 시야의 크기와 이미지 획득 속도를 조정합니다.
- 이미징 시작 후 2분에 AITC(10μM 최종 농도)의 원액 또는 E3 용액 중 41.67μL를 피펫으로 접시에 추가합니다. 타임랩스 이미징으로 관심 영역에서 GCaMP6 활동의 변화를 관찰하기 위해 ~30초 동안 계속 기록합니다. 녹음이 완료된 후 아가로스에서 동물을 풀어주고 기관 동물 관리 지침에 따라 안락사시킵니다.
참고: GCaMP6s 활동을 기록하는 동안 컨포칼 광 자체가 자극이 될 수 있습니다. 이는 이미징 결과 분석에서 고려되어야 합니다. 기록 시간은 프레임 수와 각 프레임의 캡처 속도의 함수이며, 원하는 시간에 따라 조정할 수 있습니다.
3. FIJI를 이용한 GCaMP 신호 분석
- 기록 후 출력이 다운스트림 FIJI 분석을 위한 .tif 파일 형식이 아닌 경우 현미경 소프트웨어 제품군에서 이미지 파일을 .tif 파일로 내보냅니다.
- 신경 추적 분석을 위해 FIJI를 다운로드하십시오. FIJI를 열고 .czi 파일을 프로그램으로 가져온 다음 메시지가 표시되면 BioFormats 플러그인의 기본 설정을 사용합니다.
- 관심 있는 영역/뉴런을 Circle 또는 Freehand 도구를 사용하여 각각의 아이콘을 클릭하여 강조 표시합니다. (아이콘 위로 마우스를 가져 가면 프로그램에서 도구 이름이 깜박입니다.)
참고: 관심 영역의 크기가 다양하기 때문에 고정된 영역/원은 사용할 수 없습니다. Thunder package32와 같은 복셀로 시간 경과에 따른 형광 변화를 등록할 수 있는 소프트웨어는 개별 뉴런을 감지할 수 있습니다. 그러나 현재 실험에서 ~20개의 뉴런의 경우 시각 장애인 관찰자에 의한 막 경계 감지가 자동 감지 시스템보다 우수할 수 있습니다. - ROI를 선택한 후 FIJI 도구 모음에서 > 분석 도구 > ROI 관리자를 클릭합니다. 그런 다음 [t] 추가를 클릭하면 선택한 ROI가 ROI Manager 창에 추가됩니다.
- ROI Manager(ROI 관리자)에서 More > Multi Measure(다중 측정)를 클릭합니다. 설정을 기본값으로 두고 확인을 클릭합니다. FIJI는 Python을 사용하여 CSV 파일로 조작하거나 스프레드시트의 원시 데이터로 조작할 수 있는 원시 데이터를 생성합니다.
- 원시 데이터를 정규화하고 지정된 관심 영역에 대한 신경 추적으로 나타내려면 자극 전 2분 창의 마지막 ~30초 값을 평균하고 모든 자극 후 값을 해당 평균 기준 값으로 정규화합니다.
정규화된 형광 강도 = (자극 후 주어진 관심 영역의 순간 형광)/(자극 전 2분 간격의 ~30초의 평균 형광) - 그림 4와 같이 데이터를 신경 추적으로 플로팅합니다.
참고: 뇌의 부피가 기록되는 광시트 현미경과 같은 일부 유형의 현미경에서는 운동 아티팩트가 참조 뇌 부피 1,11에 대한 후처리 공간 등록을 통해 처리됩니다. 그러나 단일 평면에서만 기록하기 때문에 이동 아티팩트를 조정할 수 없습니다. 따라서 여기에 표시된 결과의 경우 프레임이 제거되지 않았습니다. 그러나 움직임 아티팩트가 결과를 크게 변경하는 경우 자동화된 시스템이 움직임 아티팩트1, 11이 있는 프레임을 식별하고 수정하거나 눈가린 관찰자가 이러한 프레임을 제거할 수 있습니다.
결과
알릴 이소티오시아네이트의 투여는 유충 제브라피시에서 칼슘 관련 신경 신호를 유발합니다.
AITC의 투여(2.6단계)는 유충 제브라피시11,30의 뇌 전반에 걸쳐 GCaMP6s 관련 신경 활동의 광범위한 증가를 유발합니다. 그림 4와 같이 AITC를 적용한 후 뇌의 작은 영역에서 형광 신호가 증가하는 것을 관찰했습니다.
칼슘 관련 신경 신호의 해상도, 포획 속도 및 포획 영역의 균형
캡처 속도를 높이면 해상도가 낮아지며, 이는 실험 설계에서 고려해야 합니다. 그림 5에서는 ~2.5 dpf에서 제브라피시 배아를 사용하여 다양한 속도와 해당 해상도를 제시합니다. 신경 활동을 기록할 때 캡처 속도는 활동을 기록할 수 있을 만큼 충분히 빨라야 합니다. 활동전위로 인한 전압 변화는 상대적으로 빠르지만(1-2ms), GCaMP와의 칼슘 상호 작용은 활동전위 신호의 지속 시간을 연장합니다. 여기에 사용된 분자인 GCaMP6s는 특히 느린 동역학을 가지고 있습니다. GCaMP 분자를 사용하는 단일 활동 전위는 세포 배양6에서 검출되고 상관될 수 있지만, 이러한 신호 검출의 정밀도가 유충 제브라피시와 함께 생체 내에서 발생할 수 있는지는 불분명합니다. 그러나 캡처 속도가 높을수록 신호 감지가 더 높은 캡처 속도로 인해 크게 감소하지 않는다고 가정할 때 신경 활동 관련 시간 정보가 캡처될 가능성이 높아집니다. 또한 관찰 영역의 크기는 표준 컨포칼 현미경을 사용한 캡처 속도에도 영향을 미칩니다. 시야를 줄임으로써 캡처 속도는 증가하지만 다른 뇌 영역의 신경 활동에 대한 정보를 잃게 됩니다. GCaMP6 신호를 캡처하려면 컨포칼 현미경을 사용하는 응용 분야는 작은 뇌 영역(50-150μm2)으로 제한되어야 합니다. 앨리어싱을 줄이기 위해 캡처 속도가 관찰되는 신호보다 빨라야 합니다. 그러나 컨포칼 현미경 검사의 캡처 속도는 필연적으로 방출된 빛의 형태로 일부 정보를 손실하는 결과를 초래할 것입니다. 이러한 상대적 비용과 편익은 원하는 실험 질문을 효과적으로 해결하기 위해 항상 고려되어야 합니다.

그림 1: 이미징을 위해 부분적으로 구속된 유충 제브라피쉬의 위치 묘사. 유충은 낮은 융점 아가로스에 박혀 있고 등쪽이 위로 향하게 위치합니다. 제브라피시 피부가 AITC에 노출될 수 있도록 아가로스의 배부 및 꼬리 부분을 제거했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
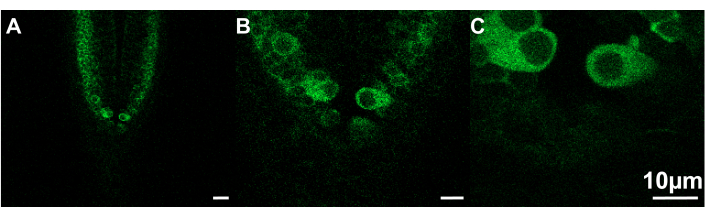
그림 2: 다양한 캡처 속도에서 볼 수 있는 다양한 크기의 이미지 예. (A) 프레임 크기 = 319.45 x 319.45μm; 캡처 속도 = .30fps. (B) 프레임 크기 = 106.48 x 106.48 μm; 캡처 속도 = 7.67fps. (C) 프레임 크기 = 53.24 x 53.24 μm; 캡처 속도 = 9.80fps. 제브라피쉬, ~2.5dpf; 눈금 막대 10 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
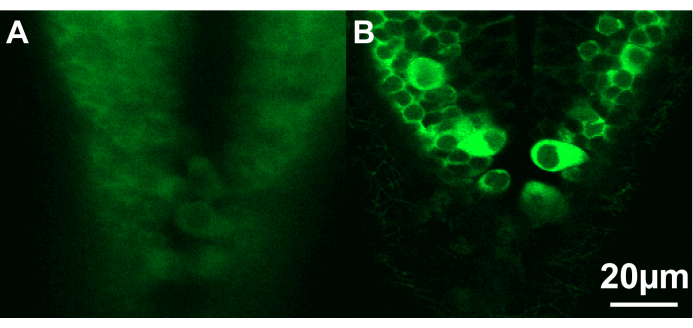
그림 3: 두 제브라피시 유충의 뒷뇌의 꼬리 부분의 컨포칼 이미지. (A) 유충(~2.5 dpf)이 이상적이지 않은 각도로 아가로스에 너무 깊숙이 위치하여 뉴런의 선명한 이미징을 방해합니다. (B) 여기서 유충(~2.5dpf)은 아가로스 표면 근처에 적절하게 위치합니다. 눈금 막대 20 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
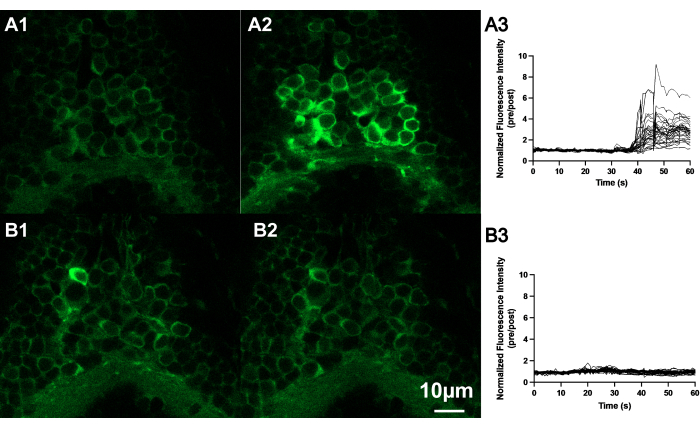
그림 4: AITC는 제브라피시 뇌의 후뇌와 척수 경계에 위치한 뉴런의 형광 강도를 증가시킵니다. (A) AITC 또는 E3에 노출되었을 때 제브라피시(7dpf) 뉴런의 대표적인 컨포칼 이미지. AITC를 적용하기 전(A1)과 후(A2)의 뉴런 이미지(A2)를 대조 용액(E3)의 (B1) 적용 전(B1) 및 (B2) 적용 후의 뉴런 이미지와 비교했습니다. 스케일 바 10μM. AITC(A3) 또는 E3(B3)에 노출된 배아 제브라피쉬의 뒷뇌에 위치한 여러 뉴런의 정규화된 형광 상태의 흔적. AITC를 수조에 적용한 후 뉴런은 형광 증가를 보인 반면, E3를 적용한 후 뉴런에서는 형광 증가가 관찰되지 않았습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: GCaMP6의 캡처 속도에 따라 캡처된 빛의 양과 이미지의 최종 해상도가 결정됩니다. (A) 10.13초(0.10fps)마다 캡처된 뉴런 이미지는 후뇌와 척수에 있는 뉴런의 상대적으로 높은 해상도를 제공합니다. 캡처 속도가 증가하면 시간 정보는 얻을 수 있지만 공간 해상도는 손실됩니다: (B) 캡처 속도 = 0.79fps, (C) 캡처 속도 = 3.16fps, (D) 캡처 속도 = 7.67fps. 제브라피쉬, 2 dpf; 눈금 막대 20 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
우리는 GCaMP6s와 컨포칼 현미경을 함께 사용하여 제브라피시 유충의 뇌에서 신경 활동을 기록할 수 있음을 보여주었습니다. GCaMP의 느린 동역학으로 인해 필요한 낮은 캡처 속도는 관찰된 뇌 영역을 줄임으로써 보상할 수 있습니다6. 더 빠른 시간 역학(즉, GCaMP6f)을 가진 기자를 사용할 수 있지만, 우수한 시간 해상도는 일반적으로 형광 신호가 감소하는 대가로 발생합니다6. 컨포칼 현미경은 상대적으로 느린 기록 속도(22)로 제한되므로 컨포칼 현미경 검사의 시간적 한계를 고려하여 리포터 분자를 선택해야 합니다.
이 프로토콜은 두 가지 주요 특징, 즉 1) 기록 중 동물을 아가로스에 넣어야 한다는 요구 사항과 2) 컨포칼 현미경 검사의 상대적으로 느린 포획 속도에 의해 제한됩니다. 시야각과 캡처 속도의 균형을 맞추는 것은 주어진 응용 분야에 대해 충분한 해상도로 신경 활동을 캡처하는 데 중요합니다. AITC로 인한 활동이 중단되거나 감소하면 유충 제브라피쉬가 자극을 감지하지 못하거나 AITC가 충분히 강력하지 않을 수 있음을 나타낼 수 있습니다. 후자의 경우 스톡을 새로 고치고 -20 °C에서 냉동 유지하면 효능이 확장됩니다. 물고기가 자극을 감지할 수 없다면, 그 동물이 한천에 너무 깊이 이식된 것일 수 있습니다. 중요한 단계는 프로토콜의 1단계에서 물고기의 위치를 파악하는 것인데, 이는 약물 적용 중 최적의 광학 접근을 위해 중요하기 때문입니다.
컨포칼 현미경의 스캔 속도가 상대적으로 느리기 때문에 전뇌 기록은 기술적으로 불가능합니다. 의미심장하게도, 여기에 설명된 프로토콜은 보완 기술로 식별된 후 작은 뇌 영역을 측정하는 데 사용할 수 있습니다. 특히, 활동 의존적 리포터 9,11로 신경 활동을 표시하는 분자를 활용하는 기술을 사용함으로써, 실험자는 이 기사에 설명된 방법의 적용을 위해 주요 뇌 관심 영역을 표적으로 삼을 수 있습니다. 그런 다음 관심 있는 뇌 영역을 주석이 달린 제브라피시 뇌 지도, z Brain Atlas11로 참조할 수 있습니다.
약간의 조정으로 여기에서 논의된 기술과 지침을 사용하여 감각 운동 처리, 학습 및 기억, 감각 및 지각을 포함한 다양한 뇌 기능을 조사할 수 있습니다.
공개
저자는 이 연구가 잠재적인 이해 상충으로 해석될 수 있는 상업적 또는 재정적 관계가 없는 상태에서 수행되었다고 선언합니다.
감사의 말
이 작업은 미국 국립보건원(National Institutes of Health, SC2GM1304854)의 ACR 보조금과 미국 국립과학재단(National Science Foundation, 2050850)의 DLG 보조금으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Low Melting Point Agarose | Invitrogen | 16520-100 | Diluted to 3% |
| Allyl Isothiocyanate (AITC) | Sigma Aldrich | 377430 | Chemical stimulant |
| E3 | N/A | N/A | Water-medium for zebrafish larvae |
| Glass Bottom Dishes | Thermo Fisher Scientific | 12-567-400 | Used to hold zebrafish during imaging experiments |
| Micropipette (10-100 uL) | Cole-Parmer | 21600-14 | Apparatus used for creating AITC dilutions |
| Microscope Slides | Fisherbrand | 12-550-A3 | Used to screen for phenotype |
| Mirror Finish Forceps | DUMONT | 11251-23 | Used to orient zebrafish in agarose |
| myTEMP Mini Digital Incubators | Benchmark | H2200-HC | Holding area for zebrafish; set to 28.5°C |
| Nitrile Gloves | MedPRIDE | MPR-50504 | Basic PPE |
| Petri Dishes | VWR | 89107-632 | Container for zebrafish |
| Posi-Click Tubes | DENVILLE | C-2171 | Used for AITC dilution |
| Samco Polyurethane Transfer Pipettes | Thermo Fisher Scientific | 225 | Apparatus used to select animal/administer diluted bolus of AITC |
| Stemi SV11 Apo Microscope | Zeiss | 1.25496E+11 | Used to stage zebrafish |
| Transgenic Larval Zebrafish (2 to 7 DPF) | N/A | N/A | Animal test subjects; Tg(elav3:GCaMP6s) strain |
| Zeiss Confocal Microscope (Model LSM9) | Zeiss | 3523004097 | Imaging of fish |
참고문헌
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nat Method. 10 (5), 413-420 (2013).
- Meyer, M. P., Smith, S. J. Evidence from in vivo imaging that synaptogenesis guides the growth and branching of axonal arbors by two distinct mechanisms. J Neurosci. 26 (13), 3604-3614 (2006).
- Sagasti, A., Guido, M. R., Raible, D. W., Schier, A. F. Repulsive interactions shape the morphologies and functional arrangement of zebrafish peripheral sensory arbors. Curr Biol. 15 (9), 804-814 (2005).
- Son, J. H., et al. Transgenic FingRs for live mapping of synaptic dynamics in genetically-defined neurons. Sci Rep. 6, 18734 (2016).
- Zada, D., Tovin, A., Lerer-Goldshtein, T., Vatine, G. D., Appelbaum, L. Altered behavioral performance and live imaging of circuit-specific neural deficiencies in a zebrafish model for psychomotor retardation. PLoS Genet. 10 (9), e1004615 (2014).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Abdelfattah, A. S., et al. and photostable chemigenetic indicators for extended in vivo voltage imaging. Science. 365 (6454), 699-704 (2019).
- Abdelfattah, A. S., et al. Sensitivity optimization of a rhodopsin-based fluorescent voltage indicator. Neuron. 111 (10), 1547-1563.e9 (2023).
- Gao, Y. J., Ji, R. R. c-Fos and pERK, which is a better marker for neuronal activation and central sensitization after noxious stimulation and tissue injury. Open Pain J. 2, 11-17 (2009).
- Kawashima, T., Okuno, H., Bito, H. A new era for functional labeling of neurons: activity-dependent promoters have come of age. Front Neural Circuits. 8, 37 (2014).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nat Methods. 12, 1039-1046 (2015).
- Reijmers, L. G., Perkins, B. L., Matsuo, N., Mayford, M. Localization of a stable neural correlate of associative memory. Science. 317 (5842), 1230-1233 (2007).
- Mohr, M. A., Argast, P., Pantazis, P. Labeling cellular structures in vivo using confined primed conversion of photoconvertible fluorescent proteins. Nature Protoc. 11 (12), 2419-2431 (2016).
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc Natl Acad Sci U S A. 106 (42), 17968-17973 (2009).
- Wyart, C., Del Bene, F. Let there be light: zebrafish neurobiology and the optogenetic revolution. Rev Neurosci. 22 (1), 121-130 (2011).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr Biol. 18 (15), 1133-1137 (2008).
- Portugues, R., Severi, K. E., Wyart, C., Ahrens, M. B. Optogenetics in a transparent animal: circuit function in the larval zebrafish. Curr Opinion Neurobiol. 23 (1), 119-126 (2013).
- Best, J. D., Alderton, W. K. Zebrafish: An in vivo. model for the study of neurological diseases. Neuropsychiat Dis Treatment. 4 (3), 567-576 (2008).
- Goldsmith, P. Zebrafish as a pharmacological tool: the how, why and when. Curr Opinion Pharmacol. 4 (5), 504-512 (2004).
- Roberts, A. C., et al. Habituation of the C-start response in larval zebrafish exhibits several distinct phases and sensitivity to NMDA receptor blockade. PloS One. 6 (12), e29132 (2011).
- Wolman, M. A., Jain, R. A., Liss, L., Granato, M. Chemical modulation of memory formation in larval zebrafish. Proc Natl Acad Sci U S A. 108 (37), 15468-15473 (2011).
- Ji, N., Freeman, J., Smith, S. L. Technologies for imaging neural activity in large volumes. Nat Neurosci. 19 (9), 1154-1164 (2016).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Front Neural Circuits. 7, 65 (2013).
- Distel, M., Köster, R. W. In vivo time-lapse imaging of zebrafish embryonic development. CSH Protoc. 2007, (2007).
- Liu, T. T., Hou, H., Du, J. L. A protocol for simultaneous Ca2+ and morphology imaging of brain endothelial tip cells in larval zebrafish. STAR Protoc. 2 (1), 100388 (2021).
- Wong, H. C., Drerup, C. M. Using fluorescent indicators for in vivo quantification of spontaneous or evoked motor neuron presynaptic activity in transgenic zebrafish. STAR Protoc. 3 (4), 101766 (2022).
- Graeden, E., Sive, H. Live imaging of the zebrafish embryonic brain by confocal microscopy. J Vis Exp. (26), e1217 (2009).
- Hirsinger, E., Steventon, B. A versatile mounting method for long term imaging of zebrafish development. J Vis Exp. (119), e55210 (2017).
- Renaud, O., Herbomel, P., Kissa, K. Studying cell behavior in whole zebrafish embryos by confocal live imaging: application to hematopoietic stem cells. Nat Protoc. 6 (12), 1897-1904 (2011).
- Roberts, A. C., et al. Induction of short-term sensitization by an aversive chemical stimulus in zebrafish larvae. eNeuro. 7 (6), (2020).
- Prober, D. A., et al. Zebrafish TRPA1 channels are required for chemosensation but not for thermosensation or mechanosensory hair cell function. J Neurosci. 28 (40), 10102-10110 (2008).
- Freeman, J., et al. Mapping brain activity at scale with cluster computing. Nat Methods. 11, 941-950 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유