Method Article
In vivo Imagerie confocale par fluorescence de l’activité neuronale induite par la stimulation sensorielle chez des larves de poisson-zèbre partiellement restreintes
Dans cet article
Résumé
Ici, nous présentons un protocole détaillé pour examiner l’activité neuronale dans les régions cérébrales du poisson-zèbre transgénique qui expriment des indicateurs calciques GCaMP à l’aide de la microscopie confocale.
Résumé
Les larves de poisson-zèbre sont un système modèle de vertébré prometteur pour l’étude des mécanismes neuronaux du comportement. Leur translucidité et leurs circuits neuronaux relativement simples facilitent l’utilisation de techniques optogénétiques dans l’analyse cellulaire du comportement. Les indicateurs fluorescents de l’activité neuronale in vivo , tels que les GCaMP6, ont été largement utilisés pour étudier l’activité neuronale associée à des comportements simples chez les larves de poisson-zèbre. Ici, nous présentons un protocole de détection de l’activité induite par des capteurs chez des larves de poisson-zèbre semi-restreintes en utilisant la lignée transgénique Tg(elav3 :GCaMP6s). En particulier, nous utilisons l’agent chimique allyl isothiocyanate pour induire une réponse fluorescente robuste et reproductible dans une région du cerveau à la frontière du cerveau postérieur et de la moelle épinière. Nous discutons des utilisations potentielles des GCaMP6 pour la surveillance optique de l’activité neuronale au cours d’une gamme de paradigmes comportementaux et des limites de cette technique. Notre protocole décrit une approche accessible pour surveiller l’activité neuronale in vivo dynamique et liée au comportement dans le cerveau des larves de poisson-zèbre.
Introduction
Le poisson-zèbre représente un modèle animal vertébré avec une traçabilité pour des études neurobiologiques cellulaires-moléculaires détaillées. Les larves de poisson-zèbre possèdent ~100 000 neurones à 5 jours après la fécondation (dpf), nettement moins que les cerveaux de mammifères. De plus, les poissons-zèbres sont relativement translucides, une propriété qui facilite les études optiques de la structure et de la fonction neuronales 1,2,3,4,5. Plusieurs outils optogénétiques ont été développés pour être utilisés chez le poisson zèbre, notamment des indicateurs de calcium haute fidélité6, des capteurs de tension 7,8 et des marqueurs dépendants de l’activité neuronale 9,10,11,12,13. Ces outils sont complémentaires à d’autres avantages possédés par ce modèle, tels que la facilité d’adaptation aux modifications génétiques 14,15,16,17 et la facilité avec laquelle les larves de poisson-zèbre absorbent les produits chimiques présents dans les solutions de bain 18,19,20,21.
Diverses méthodes sont utiles pour la physiologie optique du poisson-zèbre, en particulier la microscopie à deux photons, la microscopie à feuillet de lumière et la microscopie confocale. Chacune de ces technologies doit équilibrer deux problèmes de résolution connexes : l’accès optique, y compris la diffusion de la lumière par les tissus environnants, et la vitesse d’échantillonnage, en particulier pour capturer la cinétique du potentiel d’action à l’échelle inférieure à la milliseconde22. Des améliorations spectaculaires ont été observées dans l’imagerie calcique in vivo à l’aide de la microscopie à deux photons, mais cette méthode est souvent limitée à un champ de vision de <1mm2, et généralement, un seul plan de profondeur peut être acquis, limitant ainsi la capture de l’activité dans de grandes régions de circuits neuronaux22. Pour la microscopie à feuillet de lumière, la possibilité d’enregistrer l’activité de presque tous les neurones du cerveau résout la limitation du champ de vision de la microscopie à deux photons, mais les vitesses actuelles des caméras limitent physiquement la capture à environ trois volumes cérébraux par seconde à 40 plans par volume cérébral chez le poisson-zèbre larvaire 1,23. La microscopie confocale est inférieure en termes de résolution en profondeur et de vitesse de capture à la microscopie à deux photons et à feuillet de lumière. La microscopie confocale présente l’avantage d’être largement accessible aux laboratoires du monde entier et de pouvoir réaliser des reconstructions de l’activité neuronale sur l’ensemble du cerveau à l’aide de rapporteurs de l’activité neuronale, tels que cFos et p-ERK9. De plus, si de petites régions cérébrales sont ciblées, le microscope confocal peut fournir une résolution temporelle adéquate de l’activité neuronale.
Le présent article décrit une méthode qui utilise la microscopie confocale pour enregistrer l’activité neuronale chez le poisson-zèbre transgénique exprimant les GCaMP6 de manière panneuronale. Plusieurs protocoles similaires utilisant des larves de poisson-zèbre ont été développés pour comprendre la fonction des voies neuronales 24,25,26,27,28,29. Les principales caractéristiques de plusieurs de ces protocoles, telles que l’imagerie en accéléré, les indicateurs fluorescents de la dynamique du calcium et l’imagerie en direct, ont été combinées pour mesurer l’activité neuronale dans une petite population de neurones du système nerveux central du poisson-zèbre en réponse à l’isothiocyanate d’allyle (AITC), un irritant chimique aversif 11,26,27,29,30,31. L’AITC suscite une réponse à l’échelle du cerveau concentrée dans la zone du cerveau postérieur11. Un groupe de neurones juste caudaux au cerveau postérieur joue un rôle dans la locomotion et une réponse prolongée à l’AITC. Cette réponse dure plus longtemps que la suppression du stimulus aversif30. En restreignant le champ de vision, nous avons réussi à détecter une activité neuronale dans ce groupe neuronal comme en témoigne le changement de fluorescence dans les neurones exprimant GCaMP6s. Nous fournissons des techniques, des lignes directrices et les meilleures pratiques pour obtenir une résolution spatio-temporelle suffisante à l’aide de la microscopie confocale. De plus, nous discutons des limites de notre méthode d’enregistrement optique. Malgré ces limites, la méthode devrait permettre d’étudier une variété de phénomènes neurobiologiques, y compris la mémoire et le traitement sensorimoteur.
Protocole
Toutes les procédures utilisant des animaux ont été approuvées par l’Institutional Animal Care Use Committee de l’Université d’État de Californie, Fullerton (Protocole # 2023-1310).
1. Mise en scène des larves de poisson-zèbre dans l’agarose à bas point de fusion
- Élever des animaux transgéniques adultes exprimant panneuronalement Tg(elav3 :GCaMP6s)6 avec deux mâles à deux femelles conformément aux directives institutionnelles en matière de soins aux animaux. Les adultes sont placés dans des chambres de reproduction spéciales afin que les œufs libérés soient capturés dans une chambre inférieure avec un faux fond qui n’est pas accessible aux adultes. Aux premières lueurs du jour ou lorsqu’un séparateur est retiré, les femelles libèrent des œufs et les mâles fécondent les œufs capturés dans le faux fond.
- Prélever les adultes et recueillir les œufs fécondés à l’aide d’une passoire à œufs et les placer dans un milieu embryonnaire (E3) (5 mM de NaCl, 0,33 mM de MgSO4, 0,33 mM de CaCl2, 0,17 mM de KCl, 1 mM d’HEPES, 0,00001 % de bleu de méthylène, pH de 7,0)30 avec 14 h de lumière et 10 h d’obscurité à 28,5 °C à l’intérieur d’un incubateur
REMARQUE : La recette E3 utilisée a une faible concentration de bleu de méthylène qui n’a pas d’impact sur la fluorescence et n’entraîne aucun signal d’auto-fluorescence significatif. Nous n’exigeons pas l’utilisation de la 1-phényl-2-thiourée (PTU) aux âges de développement spécifiés dans le protocole en raison de la translucidité naturelle offerte par la lignée transgénique GCaMP6s. - Montez les animaux 2 à 7 jours après la fécondation (dpf) en utilisant 3 % d’agarose à bas point de fusion dissoute dans un milieu E3. La chaleur atteint une température à laquelle le poisson-zèbre peut être manipulé sans endommager l’organisme. Avant que l’agarose à bas point de fusion ne refroidisse et ne se solidifie (<1 min), placez les larves côté dorsal vers le haut dans une boîte de Pétri à fond en verre de manière à ce que la région du cerveau à inspecter soit aussi proche que possible de l’objectif, comme le montre la figure 1. Selon l’expérience, 8 à 10 poissons-zèbres par groupe expérimental sont généralement suffisants.
REMARQUE : Les animaux n’ont pas été anesthésiés dans ce protocole. - Une fois que l’agarose a pris et que la larve est en place (environ 2 à 3 min), ajoutez 5 mL de solution E3. Ensuite, coupez l’agarose à l’aide d’un scalpel comme l’exige le protocole expérimental.
REMARQUE : Différentes parties de la gélose doivent être coupées, en fonction des parties du poisson-zèbre qui doivent être libérées de la contention et/ou recevoir un type de stimulus différent. - Déplacez la boîte de Pétri contenant le poisson-zèbre vers la platine du microscope confocal et laissez le poisson s’acclimater pendant 20 min.
2. Mise en place et imagerie en microscopie confocale avec application de stimulus
- Après la période d’acclimatation, centrez le poisson sous l’objectif d’immersion dans l’eau 40x (NA 1.0) à température ambiante à l’aide d’un fond clair avec une source lumineuse (~475 nm).
- Les paramètres d’acquisition du microscope confocal dépendent des qualités et du niveau d’expression de la molécule fluorescente, de la profondeur et des propriétés optiques du tissu, de la taille du champ de vision et de la vitesse d’enregistrement. Déterminez les paramètres appropriés à l’aide de l’indicateur de portée pour assurer une capture optimale de la lumière émise en fonction des besoins expérimentaux.
- Assurez-vous d’un réglage correct de la puissance laser et du gain principal pour capturer la lumière fluorescente dans la plage optimale, évitant ainsi la perte inutile du signal de fluorescence dépendant du GCaMP6s. Utilisez la liste des principaux paramètres et facteurs confocaux ci-dessous pour déterminer les paramètres appropriés.
- Taille du sténopé : Déterminez cela en fonction de l’objectif utilisé et réglé pour accepter un seul plan focal tout en limitant les autres plans focaux. Pour ce protocole, un sténopé de 32 μm est utilisé.
- Puissance et longueur d’onde du laser : ajustez la puissance du laser en fonction du niveau d’expression de la molécule fluorescente et de la profondeur et des propriétés optiques du tissu. Utilisez l’indicateur de portée pour vous assurer que le laser ne dépasse pas la portée du photodétecteur. Réglez la longueur d’onde d’excitation sur la longueur d’onde de la lumière qui excite de manière optimale la molécule fluorescente. Pour cette expérience, parce que les poissons étaient hétérozygotes pour le transgène GCaMP6s et que les niveaux d’expression étaient faibles, utilisez une puissance laser de 5 % à une longueur d’onde de 488 nm, un gain de maître de 650. Avec une forte expression transgénique, la puissance laser varie de 1,5 % à 3 %, ce qui est généralement suffisant.
REMARQUE : Si la puissance du laser est réglée trop faible, des informations (lumière détectée par le tube photomultiplicateur) seront perdues. Si la puissance laser est trop élevée, la lumière émise peut dépasser la portée du détecteur ou endommager l’échantillon (photoblanchiment). - Gain principal : Le gain principal détermine la sensibilité du photodétecteur. Utilisez l’indicateur de portée pour vous assurer que le gain principal n’est pas réglé trop haut, dépassant ainsi la portée du photodétecteur. Pour cette expérience, le gain principal est réglé sur 650.
- Champ de vision : Assurez-vous que le champ de vision est suffisamment grand pour capturer le tissu d’intérêt. Il s’agit d’une limitation majeure pour les microscopes confocaux en général, car la taille du champ de vision limite la vitesse de capture, comme le montre la figure 2. Pour les expériences impliquant l’AITC, régler le champ de vision à 79,86 μm2.
- Vitesse de capture : à mesure que la vitesse de capture augmente, la quantité de lumière capturée par le photodétecteur diminue. Comme moins de lumière est capturée, la qualité de l’image sera dégradée. Assurez-vous que la vitesse de capture équilibre la qualité de l’image et la vitesse requise pour capturer l’événement physiologique d’intérêt. Ici, la vitesse de capture est réglée sur 1,20 f/s.
- Centrer le champ de vision sur un cluster neuronal caudal à la commissura infima Halleri dans la partie rostrale de la moelle épinière30. Ce groupe est assez petit à ~2,5 dpf mais est beaucoup plus grand plus tard dans le développement (7 dpf).
REMARQUE : Si le montage de l’agarose à l’étape 1 n’est pas optimal, c’est-à-dire que l’animal est positionné à un angle ou trop profondément dans l’agarose, la qualité de l’image sera affectée. La figure 3 montre une image de la moelle épinière du poisson-zèbre chez un animal bien placé et une image contrastée de la moelle épinière d’un poisson zèbre mal placé. - Effectuez un balayage de série chronologique à l’aide d’un champ de vision de 79,86 μm2 à 1,20 images par s (fps) avec une résolution spatiale de 0,119 μm2 par pixel. Ajustez la taille du champ de vision et la vitesse d’acquisition de l’image en fonction de l’application souhaitée.
- 2 minutes après le début de l’imagerie, ajouter 41,67 μL d’une solution mère d’AITC (concentration finale de 10 μM) ou d’une solution E3 dans la boîte à l’aide d’une pipette. Poursuivez l’enregistrement pendant ~30 s pour observer tout changement dans l’activité des GCaMP6 dans la région d’intérêt avec l’imagerie en accéléré. Une fois l’enregistrement terminé, libérez l’animal de l’agarose et euthanasiez-le conformément aux directives de l’établissement en matière de soins aux animaux.
REMARQUE : Lors de l’enregistrement de l’activité du GCaMP6, la lumière confocale elle-même peut être un stimulus. Cela doit être pris en compte dans l’analyse des résultats d’imagerie. La durée d’enregistrement est fonction du nombre d’images et de la vitesse de capture de chaque image, qui peut être ajustée si nécessaire pour le temps souhaité.
3. Analyse du signal GCaMP à l’aide de FIJI
- Après l’enregistrement, si la sortie n’est pas dans le format de fichier .tif pour l’analyse FIJI en aval, exportez les fichiers d’image sous forme de fichiers .tif de la suite logicielle de microscope.
- Téléchargez FIJI pour l’analyse des traces neuronales. Ouvrez FIJI, importez les fichiers .czi dans le programme et, lorsque vous y êtes invité, utilisez les paramètres par défaut du plugin BioFormats.
- Mettez en surbrillance une zone/un neurone d’intérêt à l’aide des outils Cercle ou À main levée en cliquant sur leurs icônes respectives. (En survolant les icônes, le nom de l’outil dans le programme clignotera.)
REMARQUE : Étant donné que la zone d’intérêt varie en taille, les zones/cercles fixes ne sont pas possibles. Les logiciels qui permettent d’enregistrer les changements fluorescents au fil du temps sous forme de voxels, tels que le package Thunder32, pourraient permettre la détection de neurones individuels ; Cependant, pour les ~20 neurones de l’expérience actuelle, la détection des bords de la membrane par un observateur aveugle serait probablement supérieure à un système de détection automatisé. - Une fois qu’un retour sur investissement a été sélectionné, dans la barre d’outils FIJI, cliquez sur Analyser les outils > > Gestionnaire de retour sur investissement. Après cela, cliquez sur Ajouter [t] et le retour sur investissement sélectionné sera ajouté à la fenêtre du gestionnaire de retour sur investissement.
- Dans le Gestionnaire de retour sur investissement, cliquez sur Plus > Multi Mesure. Laissez les paramètres par défaut et cliquez sur OK. FIJI produira des données brutes qui pourront être manipulées sous forme de fichier CSV à l’aide de Python ou de données brutes dans une feuille de calcul.
- Pour normaliser les données brutes et les représenter sous forme de trace neuronale pour une région d’intérêt donnée, faites la moyenne des valeurs des dernières ~30 secondes de la fenêtre de 2 minutes avant le stimulus, et normalisez toutes les valeurs post-stimulus à cette valeur de référence moyenne :
intensité de fluorescence normalisée = (fluorescence instantanée d’une région d’intérêt donnée après le stimulus)/(fluorescence moyenne de ~30 s d’un intervalle de 2 minutes avant le stimulus) - Tracez les données sous forme de traces neuronales, comme illustré à la figure 4.
REMARQUE : Dans certains types de microscopies, telles que la microscopie à feuillet de lumière, où un volume du cerveau est enregistré, les artefacts de mouvement sont adressés par post-traitement de l’enregistrement spatial à un volume cérébral de référence 1,11. Cependant, comme nous n’enregistrons qu’à partir d’un seul plan, aucun ajustement ne peut être effectué pour les artefacts de mouvement. Par conséquent, pour les résultats présentés ici, aucune image n’a été supprimée. Cependant, si les artefacts de mouvement modifiaient considérablement les résultats, les systèmes automatisés pourraient identifier et corriger les images avec les artefactsde mouvement 1, 11, ou un observateur aveuglé pourrait supprimer ces images.
Résultats
L’administration d’isothiocyanate d’allyle provoque un signal neuronal associé au calcium chez les larves de poisson-zèbre
L’administration d’AITC (étape 2.6) provoque une augmentation généralisée de l’activité neuronale associée aux GCaMP6s dans le cerveau de la larve de poisson-zèbre11,30. Nous avons observé une augmentation du signal fluorescent dans une petite région du cerveau après l’application de l’AITC, comme le montre la figure 4.
Équilibrage de la résolution des signaux neuronaux associés au calcium, de la vitesse de capture et de la zone de capture
L’augmentation de la vitesse de capture réduit la résolution, qui doit être prise en compte dans la conception expérimentale. Nous présentons une variété de vitesses et leurs résolutions correspondantes en utilisant des embryons de poisson-zèbre à ~2,5 dpf dans la figure 5. Lors de l’enregistrement de l’activité neuronale, la vitesse de capture doit être suffisamment rapide pour enregistrer l’activité. Bien que les changements de tension dus à un potentiel d’action soient relativement rapides (1-2 ms), les interactions du calcium avec GCaMP prolongeront la durée du signal de potentiel d’action. GCaMP6s, la molécule utilisée ici, a une cinétique particulièrement lente. Des potentiels d’action unique utilisant des molécules de GCaMP peuvent être détectés et corrélés dans la culture cellulaire6, mais il n’est pas clair si cette précision de détection du signal pourrait se produire in vivo avec des larves de poisson-zèbre. Cependant, plus la vitesse de capture est élevée, plus il est probable que des informations temporelles liées à l’activité neuronale soient capturées, en supposant que la détection du signal n’est pas significativement réduite par les vitesses de capture plus élevées. De plus, la taille du champ d’observation influencera également les vitesses de capture avec un microscope confocal standard. En réduisant le champ de vision, on augmente la vitesse de capture mais on perd des informations sur l’activité neuronale dans d’autres zones du cerveau. Pour capturer les signaux GCaMP6, les applications utilisant la microscopie confocale doivent être limitées à une petite zone cérébrale (50-150 μm2). La vitesse de capture doit être plus rapide que le signal observé pour réduire le repliement. Cependant, les vitesses de capture de la microscopie confocale entraîneront inévitablement la perte de certaines informations sous forme de lumière émise. Ces coûts et avantages relatifs doivent toujours être pris en compte pour répondre efficacement à la question expérimentale souhaitée.

Figure 1 : Représentation de la position d’une larve de poisson-zèbre partiellement restreinte pour l’imagerie. La larve est nourrie dans une agarose à bas point de fusion et positionnée côté dorsal vers le haut. Les parties rostrale et caudale de l’agarose ont été retirées pour permettre l’exposition de la peau du poisson-zèbre à l’AITC. Veuillez cliquer ici pour voir une version agrandie de cette figure.
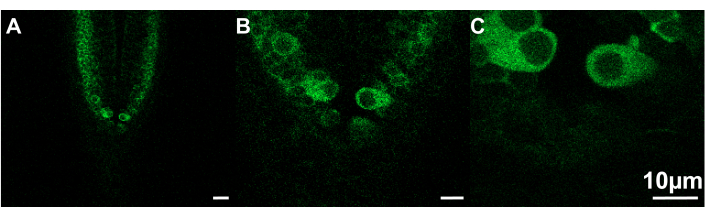
Figure 2 : Exemples d’images de différentes tailles qui peuvent être visualisées à différentes vitesses de capture. (A) Taille de l’image = 319,45 x 319,45 μm ; Vitesse de capture = 0,30 ips. (B) Taille du cadre = 106,48 x 106,48 μm ; Vitesse de capture = 7,67 ips. (C) Taille du cadre = 53,24 x 53,24 μm ; Vitesse de capture = 9,80 ips. Poisson-zèbre, ~2,5 dpf ; barre d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
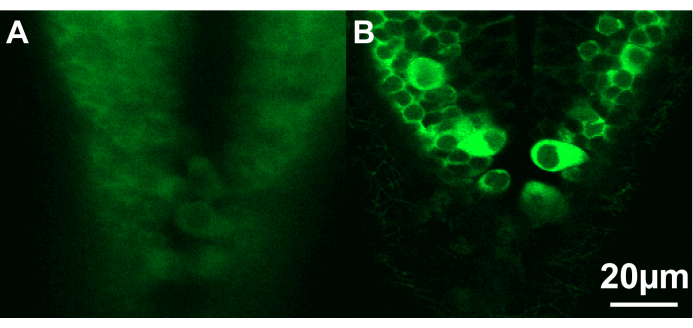
Figure 3 : Images confocales de la partie caudale du cerveau postérieur de deux larves de poisson-zèbre. (A) La larve (~2,5 dpf) est positionnée à un angle non idéal et trop profondément dans l’agarose, empêchant une imagerie nette des neurones. (B) Ici, la larve (~2,5 dpf) est correctement positionnée près de la surface de l’agarose. Barre d’échelle 20 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
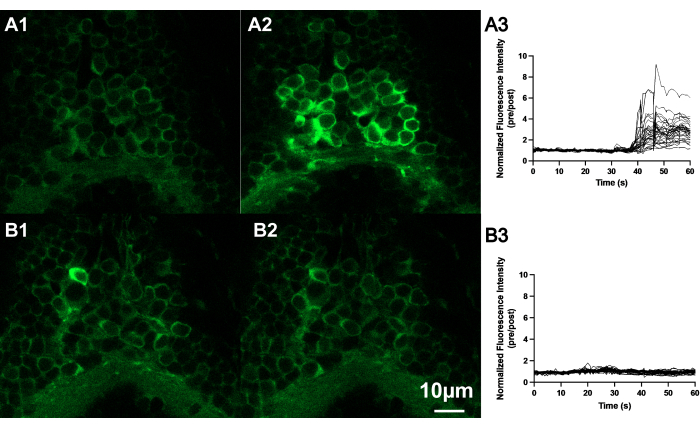
Figure 4 : L’AITC augmente l’intensité de fluorescence des neurones situés à la limite du cerveau du poisson zèbre et de la moelle épinière du cerveau du poisson-zèbre. (A) Images confocales représentatives des neurones du poisson-zèbre (7 dpf) lorsqu’ils sont exposés à l’AITC ou à l’E3. Images de neurones avant (A1) et après (A2) l’application de l’AITC comparées aux images (B) des neurones avant (B1) et après (B2) l’application de la solution de contrôle (E3). Barre d’échelle 10 μM. Traces de l’état fluorescent normalisé de plusieurs neurones situés dans le cerveau postérieur de poissons-zèbres embryonnaires exposés à l’AITC (A3) ou E3 (B3). Notez que les neurones ont montré une fluorescence accrue après l’application d’AITC dans le bain, alors qu’une augmentation de la fluorescence n’a pas été observée dans les neurones après l’application de E3. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : La vitesse de capture des GCaMP6 détermine la quantité de lumière capturée et la résolution résultante de l’image. (A) Les images des neurones capturées toutes les 10,13 s (0,10 fps) donnent une résolution relativement élevée des neurones du cerveau postérieur et de la moelle épinière. Avec une augmentation de la vitesse de capture, des informations temporelles sont obtenues, mais la résolution spatiale est perdue : (B) vitesse de capture = 0,79 ips, (C) vitesse de capture = 3,16 ips et (D) vitesse de capture = 7,67 ips. Poisson zèbre, 2 dpf ; barre d’échelle 20 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Discussion
Nous avons montré que l’activité neuronale peut être enregistrée dans le cerveau des larves de poisson-zèbre à l’aide de GCaMP6 associés à la microscopie confocale ; les vitesses de capture plus faibles requises en raison de la cinétique plus lente des GCaMP peuvent être compensées par la réduction de la surface cérébrale observée6. Des rapporteurs avec une dynamique temporelle plus rapide (c’est-à-dire GCaMP6f) sont disponibles, mais la résolution temporelle supérieure se fait généralement au prix d’un signal de fluorescence réduit6. Le microscope confocal est limité à des vitesses d’enregistrement relativement plus lentes22, de sorte que les molécules rapporteures doivent être sélectionnées en tenant compte des limites temporelles de la microscopie confocale.
Le protocole est limité par deux caractéristiques clés : 1) la nécessité de placer l’animal dans de l’agarose pendant l’enregistrement et 2) la vitesse de capture relativement lente de la microscopie confocale. L’équilibre entre le champ de vision et les vitesses de capture est essentiel pour capturer l’activité neuronale avec une résolution suffisante pour une application donnée. Si l’activité induite par l’AITC est tronquée ou diminuée, cela pourrait indiquer que les larves de poisson-zèbre ne sont peut-être pas en mesure de détecter le stimulus ou que l’AITC n’est peut-être pas suffisamment puissante. Dans ce dernier cas, rafraîchir le bouillon et le maintenir congelé à -20 °C prolongera son efficacité. Si le poisson ne peut pas détecter le stimulus, il se peut que l’animal ait été implanté trop profondément dans la gélose. Une étape critique est le positionnement d’un poisson au cours de l’étape 1 du protocole, car cela est crucial pour un accès optique optimal lors de l’application du médicament.
En raison des vitesses de balayage relativement lentes de la microscopie confocale, les enregistrements du cerveau entier ne sont pas techniquement possibles. De manière significative, le protocole décrit ici peut être utilisé pour mesurer de petites zones cérébrales après identification avec des techniques complémentaires. En particulier, en utilisant des techniques qui exploitent des molécules qui marquent l’activité neuronale avec des rapporteurs dépendants de l’activité 9,11, l’expérimentateur peut cibler des zones cérébrales clés d’intérêt pour l’application des méthodes décrites dans cet article. Les zones cérébrales d’intérêt peuvent ensuite être référencées à l’atlas cérébral annoté du poisson-zèbre, z brain atlas11.
Avec des ajustements mineurs, les techniques et les directives discutées ici peuvent être utilisées pour étudier une gamme de fonctions cérébrales, y compris le traitement sensorimoteur, l’apprentissage et la mémoire, ainsi que la sensation et la perception.
Déclarations de divulgation
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Ce travail a été soutenu par une subvention à l’ACR des National Institutes of Health (SC2GM1304854) et une subvention à la DLG de la National Science Foundation (2050850).
matériels
| Name | Company | Catalog Number | Comments |
| Low Melting Point Agarose | Invitrogen | 16520-100 | Diluted to 3% |
| Allyl Isothiocyanate (AITC) | Sigma Aldrich | 377430 | Chemical stimulant |
| E3 | N/A | N/A | Water-medium for zebrafish larvae |
| Glass Bottom Dishes | Thermo Fisher Scientific | 12-567-400 | Used to hold zebrafish during imaging experiments |
| Micropipette (10-100 uL) | Cole-Parmer | 21600-14 | Apparatus used for creating AITC dilutions |
| Microscope Slides | Fisherbrand | 12-550-A3 | Used to screen for phenotype |
| Mirror Finish Forceps | DUMONT | 11251-23 | Used to orient zebrafish in agarose |
| myTEMP Mini Digital Incubators | Benchmark | H2200-HC | Holding area for zebrafish; set to 28.5°C |
| Nitrile Gloves | MedPRIDE | MPR-50504 | Basic PPE |
| Petri Dishes | VWR | 89107-632 | Container for zebrafish |
| Posi-Click Tubes | DENVILLE | C-2171 | Used for AITC dilution |
| Samco Polyurethane Transfer Pipettes | Thermo Fisher Scientific | 225 | Apparatus used to select animal/administer diluted bolus of AITC |
| Stemi SV11 Apo Microscope | Zeiss | 1.25496E+11 | Used to stage zebrafish |
| Transgenic Larval Zebrafish (2 to 7 DPF) | N/A | N/A | Animal test subjects; Tg(elav3:GCaMP6s) strain |
| Zeiss Confocal Microscope (Model LSM9) | Zeiss | 3523004097 | Imaging of fish |
Références
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nat Method. 10 (5), 413-420 (2013).
- Meyer, M. P., Smith, S. J. Evidence from in vivo imaging that synaptogenesis guides the growth and branching of axonal arbors by two distinct mechanisms. J Neurosci. 26 (13), 3604-3614 (2006).
- Sagasti, A., Guido, M. R., Raible, D. W., Schier, A. F. Repulsive interactions shape the morphologies and functional arrangement of zebrafish peripheral sensory arbors. Curr Biol. 15 (9), 804-814 (2005).
- Son, J. H., et al. Transgenic FingRs for live mapping of synaptic dynamics in genetically-defined neurons. Sci Rep. 6, 18734(2016).
- Zada, D., Tovin, A., Lerer-Goldshtein, T., Vatine, G. D., Appelbaum, L. Altered behavioral performance and live imaging of circuit-specific neural deficiencies in a zebrafish model for psychomotor retardation. PLoS Genet. 10 (9), e1004615(2014).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Abdelfattah, A. S., et al. and photostable chemigenetic indicators for extended in vivo voltage imaging. Science. 365 (6454), 699-704 (2019).
- Abdelfattah, A. S., et al. Sensitivity optimization of a rhodopsin-based fluorescent voltage indicator. Neuron. 111 (10), 1547-1563.e9 (2023).
- Gao, Y. J., Ji, R. R. c-Fos and pERK, which is a better marker for neuronal activation and central sensitization after noxious stimulation and tissue injury. Open Pain J. 2, 11-17 (2009).
- Kawashima, T., Okuno, H., Bito, H. A new era for functional labeling of neurons: activity-dependent promoters have come of age. Front Neural Circuits. 8, 37(2014).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nat Methods. 12, 1039-1046 (2015).
- Reijmers, L. G., Perkins, B. L., Matsuo, N., Mayford, M. Localization of a stable neural correlate of associative memory. Science. 317 (5842), 1230-1233 (2007).
- Mohr, M. A., Argast, P., Pantazis, P. Labeling cellular structures in vivo using confined primed conversion of photoconvertible fluorescent proteins. Nature Protoc. 11 (12), 2419-2431 (2016).
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc Natl Acad Sci U S A. 106 (42), 17968-17973 (2009).
- Wyart, C., Del Bene, F. Let there be light: zebrafish neurobiology and the optogenetic revolution. Rev Neurosci. 22 (1), 121-130 (2011).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr Biol. 18 (15), 1133-1137 (2008).
- Portugues, R., Severi, K. E., Wyart, C., Ahrens, M. B. Optogenetics in a transparent animal: circuit function in the larval zebrafish. Curr Opinion Neurobiol. 23 (1), 119-126 (2013).
- Best, J. D., Alderton, W. K. Zebrafish: An in vivo. model for the study of neurological diseases. Neuropsychiat Dis Treatment. 4 (3), 567-576 (2008).
- Goldsmith, P. Zebrafish as a pharmacological tool: the how, why and when. Curr Opinion Pharmacol. 4 (5), 504-512 (2004).
- Roberts, A. C., et al. Habituation of the C-start response in larval zebrafish exhibits several distinct phases and sensitivity to NMDA receptor blockade. PloS One. 6 (12), e29132(2011).
- Wolman, M. A., Jain, R. A., Liss, L., Granato, M. Chemical modulation of memory formation in larval zebrafish. Proc Natl Acad Sci U S A. 108 (37), 15468-15473 (2011).
- Ji, N., Freeman, J., Smith, S. L. Technologies for imaging neural activity in large volumes. Nat Neurosci. 19 (9), 1154-1164 (2016).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Front Neural Circuits. 7, 65(2013).
- Distel, M., Köster, R. W. In vivo time-lapse imaging of zebrafish embryonic development. CSH Protoc. 2007, (2007).
- Liu, T. T., Hou, H., Du, J. L. A protocol for simultaneous Ca2+ and morphology imaging of brain endothelial tip cells in larval zebrafish. STAR Protoc. 2 (1), 100388(2021).
- Wong, H. C., Drerup, C. M. Using fluorescent indicators for in vivo quantification of spontaneous or evoked motor neuron presynaptic activity in transgenic zebrafish. STAR Protoc. 3 (4), 101766(2022).
- Graeden, E., Sive, H. Live imaging of the zebrafish embryonic brain by confocal microscopy. J Vis Exp. (26), e1217(2009).
- Hirsinger, E., Steventon, B. A versatile mounting method for long term imaging of zebrafish development. J Vis Exp. (119), e55210(2017).
- Renaud, O., Herbomel, P., Kissa, K. Studying cell behavior in whole zebrafish embryos by confocal live imaging: application to hematopoietic stem cells. Nat Protoc. 6 (12), 1897-1904 (2011).
- Roberts, A. C., et al. Induction of short-term sensitization by an aversive chemical stimulus in zebrafish larvae. eNeuro. 7 (6), (2020).
- Prober, D. A., et al. Zebrafish TRPA1 channels are required for chemosensation but not for thermosensation or mechanosensory hair cell function. J Neurosci. 28 (40), 10102-10110 (2008).
- Freeman, J., et al. Mapping brain activity at scale with cluster computing. Nat Methods. 11, 941-950 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon