Method Article
Im lebenden Organismus Konfokale Fluoreszenzbildgebung der durch sensorische Stimulation induzierten neuronalen Aktivität bei teilweise gefesselten Zebrafischlarven
In diesem Artikel
Zusammenfassung
Hier stellen wir ein detailliertes Protokoll vor, um die neuronale Aktivität in Gehirnregionen transgener Zebrafische, die GCaMP-Kalziumindikatoren exprimieren, mittels konfokaler Mikroskopie zu untersuchen.
Zusammenfassung
Zebrafischlarven sind ein vielversprechendes Modellsystem für Wirbeltiere, um die neuronalen Mechanismen des Verhaltens zu untersuchen. Ihre Transluzenz und relativ einfachen neuronalen Schaltkreise erleichtern den Einsatz optogenetischer Techniken bei zellulären Verhaltensanalysen. Fluoreszierende Indikatoren für die neuronale Aktivität in vivo , wie z. B. GCaMP6s, wurden häufig verwendet, um die neuronale Aktivität zu untersuchen, die mit einfachen Verhaltensweisen bei Zebrafischlarven verbunden ist. In dieser Arbeit stellen wir ein Protokoll zum Nachweis sensorisch induzierter Aktivität in halb zurückgehaltenen Zebrafischlarven unter Verwendung der transgenen Linie Tg(elav3:GCaMP6s) vor. Insbesondere verwenden wir den chemischen Wirkstoff Allylisothiocyanat, um eine robuste, reproduzierbare Fluoreszenzreaktion in einer Gehirnregion an der Grenze zwischen Hinterhirn und Rückenmark zu induzieren. Wir diskutieren die potenziellen Anwendungen von GCaMP6s für die optische Überwachung der neuronalen Aktivität während einer Reihe von Verhaltensparadigmen und die Grenzen dieser Technik. Unser Protokoll skizziert einen zugänglichen Ansatz zur Überwachung dynamischer, verhaltensbezogener in vivo neuronaler Aktivität im Gehirn von Zebrafischlarven.
Einleitung
Der Zebrafisch stellt ein Wirbeltiermodell dar, das sich für detaillierte zellulär-molekulare neurobiologische Untersuchungen steuern lässt. Zebrafischlarven besitzen 5 Tage nach der Befruchtung (dpf) ~100.000 Neuronen, deutlich weniger als Säugetiergehirne. Darüber hinaus sind Zebrafische relativ durchscheinend, eine Eigenschaft, die optische Untersuchungen der neuronalen Struktur und Funktion erleichtert 1,2,3,4,5. Mehrere optogenetische Werkzeuge wurden für den Einsatz im Zebrafisch entwickelt, darunter High-Fidelity-Kalziumindikatoren6, Spannungssensoren 7,8 und aktivitätsabhängige Marker der neuronalen Aktivität 9,10,11,12,13. Diese Werkzeuge ergänzen andere Vorteile dieses Modells, wie z. B. die Zugänglichkeit für genetische Veränderungen 14,15,16,17 und die Bereitschaft, mit der Zebrafischlarven in Badelösungen enthaltene Chemikalien aufnehmen 18,19,20,21.
Für die optische Physiologie des Zebrafisches sind eine Vielzahl von Methoden nützlich, insbesondere die Zwei-Photonen-, Lichtblatt- und konfokale Mikroskopie. Jede dieser Technologien muss zwei miteinander verbundene Auflösungsprobleme in Einklang bringen: den optischen Zugang, einschließlich der Lichtstreuung durch umgebendes Gewebe, und die Probenahmegeschwindigkeit, insbesondere für die Erfassung der Kinetik des Aktionspotenzials im Sub-Millisekunden-Bereich22. Es gab dramatische Verbesserungen bei der In-vivo-Kalziumbildgebung mit Hilfe der Zwei-Photonen-Mikroskopie, aber diese Methode ist oft auf ein Sichtfeld von <1 mm2 beschränkt, und typischerweise kann nur eine einzige Tiefenebene erfasst werden, wodurch die Erfassung der Aktivität in großen Regionen neuronaler Schaltkreise eingeschränktwird 22. Für die Lichtblattmikroskopie löst das Potenzial, die Aktivität fast aller Neuronen im Gehirn aufzuzeichnen, die Einschränkung des Sichtfelds der Zwei-Photonen-Mikroskopie, aber die derzeitigen Kamerageschwindigkeiten begrenzen die Erfassung physikalisch auf etwa drei Gehirnvolumina pro Sekunde bei 40 Ebenen pro Hirnvolumen bei der Zebrafischlarve 1,23. Die konfokale Mikroskopie ist der Zwei-Photonen- und Lichtblattmikroskopie sowohl in der Tiefenauflösung als auch in der Einfanggeschwindigkeit unterlegen. Die konfokale Mikroskopie hat die Vorteile eines breiten Zugangs zu Laboratorien weltweit und der Möglichkeit, Rekonstruktionen der neuronalen Aktivität im gesamten Gehirn mit Hilfe von Reportern der neuronalen Aktivität wie cFos und p-ERK9 zu erzielen. Darüber hinaus kann das konfokale Mikroskop, wenn kleine Hirnregionen angesprochen werden, eine angemessene zeitliche Auflösung der neuronalen Aktivität liefern.
Die vorliegende Arbeit beschreibt eine Methode, die konfokale Mikroskopie verwendet, um die neuronale Aktivität in transgenen Zebrafischen aufzuzeichnen, die GCaMP6s panneuronal exprimieren. Mehrere ähnliche Protokolle mit Zebrafischlarven wurden entwickelt, um die Funktion der Nervenbahnen 24,25,26,27,28,29 zu verstehen. Schlüsselmerkmale mehrerer dieser Protokolle, wie z. B. Zeitraffer-Bildgebung, fluoreszierende Indikatoren für die Kalziumdynamik und Live-Bildgebung, wurden kombiniert, um die neuronale Aktivität in einer kleinen Population von Neuronen im zentralen Nervensystem des Zebrafisches als Reaktion auf Allylisothiocyanat (AITC), einen aversiven chemischen Reizstoff, zu messen 11,26,27,29,30,31. AITC löst eine hirnweite Reaktion aus, die sich auf den Hinterhirnbereichkonzentriert 11. Eine Gruppe von Neuronen nur kaudal zum Hinterhirn spielt eine Rolle bei der Fortbewegung und einer verlängerten Reaktion auf AITC. Diese Reaktion überdauert die Entfernung des aversiven Reizes30. Durch die Einschränkung des Sichtfeldes ist es uns gelungen, die neuronale Aktivität in diesem neuronalen Cluster zu detektieren, die sich in der Fluoreszenzänderung von Neuronen widerspiegelt, die GCaMP6s exprimieren. Wir stellen Techniken, Richtlinien und Best Practices zur Verfügung, um mit Hilfe der konfokalen Mikroskopie eine ausreichende raumzeitliche Auflösung zu erreichen. Darüber hinaus gehen wir auf die Grenzen unserer optischen Aufzeichnungsmethode ein. Trotz dieser Einschränkungen soll die Methode die Untersuchung einer Vielzahl von neurobiologischen Phänomenen ermöglichen, einschließlich des Gedächtnisses und der sensomotorischen Verarbeitung.
Protokoll
Alle Verfahren mit Tieren wurden vom Institutional Animal Care Use Committee an der California State University, Fullerton, genehmigt (Protokoll # 2023-1310).
1. Inszenierung von Zebrafischlarven in Agarose mit niedrigem Schmelzpunkt
- Zucht adulter transgener Tiere mit panneuronal exprimierendem Tg(elav3:GCaMP6s)6 mit zwei Männchen auf zwei Weibchen gemäß den Richtlinien der institutionellen Tierpflege. Die erwachsenen Tiere werden in spezielle Brutkammern gebracht, so dass die freigesetzten Eier in einer unteren Kammer mit doppeltem Boden aufgefangen werden, die für erwachsene Tiere nicht zugänglich ist. Beim ersten Licht oder wenn ein Trennteiler entfernt wird, geben die Weibchen Eier frei, und die Männchen befruchten die Eier, die sie im doppelten Boden gefangen haben.
- Entnehmen Sie adulte Eizellen und entnehmen Sie befruchtete Eizellen mit einem Eisieb und legen Sie sie in das Medium des Embryos (E3) (5 mM NaCl, 0,33 mM MgSO4, 0,33 mM CaCl2, 0,17 mM KCl, 1 mM HEPES, 0,00001 % Methylenblau, pH 7,0)30 mit 14 h hell/10 h dunkel bei 28,5 °C in einem Inkubator
HINWEIS: Das verwendete E3-Rezept enthält eine niedrige Konzentration an Methylenblau, das die Fluoreszenz nicht beeinflusst oder zu einem signifikanten Autofluoreszenzsignal führt. Aufgrund der natürlichen Transluzenz, die die transgene Linie GCaMP6s bietet, ist die Verwendung von 1-Phenyl-2-Thioharnstoff (PTU) in den im Protokoll angegebenen Entwicklungsaltern nicht erforderlich. - Tiere 2-7 Tage nach der Befruchtung (dpf) mit 3 % Agarose bei niedrigem Schmelzpunkt, gelöst in E3-Medium, einbetten. Die Hitze agarose auf eine Temperatur, bei der der Zebrafisch manipuliert werden kann, ohne den Organismus zu schädigen. Bevor die Agarose bei niedrigem Schmelzpunkt abkühlt und erstarrt (<1 min), positionieren Sie die Larven mit der Rückenseite nach oben in einer Petrischale mit Glasboden, so dass die zu untersuchende Hirnregion so nah wie möglich am Objektiv ist, wie in Abbildung 1 gezeigt. Je nach Versuch sind in der Regel 8 bis 10 Zebrafische pro Versuchsgruppe ausreichend.
HINWEIS: Die Tiere wurden in diesem Protokoll nicht betäubt. - Nachdem die Agarose mit der Larve an Ort und Stelle ist (ca. 2 bis 3 Minuten), fügen Sie 5 ml E3-Lösung hinzu. Schneiden Sie dann die Agarose mit einem Skalpell ein, wie es das Versuchsprotokoll vorschreibt.
HINWEIS: Verschiedene Teile des Agars sollten weggeschnitten werden, je nachdem, welche Teile des Zebrafisches von der Fessel befreit werden müssen und/oder eine andere Art von Reiz erhalten müssen. - Stellen Sie die Petrischale mit dem Zebrafisch auf den konfokalen Mikroskoptisch und lassen Sie den Fisch 20 Minuten lang akklimatisieren.
2. Aufbau und Bildgebung unter konfokaler Mikroskopie mit Stimulusapplikation
- Nach der Eingewöhnungsphase zentrieren Sie den Fisch unter dem 40x (NA 1.0) Wasserimmersionsobjektiv bei Raumtemperatur unter Verwendung eines Hellfeldes mit einer Lichtquelle (~475 nm).
- Die Einstellungen für die Aufnahme des konfokalen Mikroskops hängen von den Qualitäten und dem Expressionsniveau des fluoreszierenden Moleküls, der Tiefe und den optischen Eigenschaften des Gewebes, der Größe des Sichtfelds und der Aufnahmegeschwindigkeit ab. Bestimmen Sie mit der Reichweitenanzeige die geeigneten Einstellungen, um eine optimale Erfassung des emittierten Lichts entsprechend den experimentellen Anforderungen zu gewährleisten.
- Stellen Sie sicher, dass die Laserleistung und die Master-Verstärkung richtig eingestellt werden, um Fluoreszenzlicht im optimalen Bereich einzufangen und so unnötige Verluste des GCaMP6s-abhängigen Fluoreszenzsignals zu vermeiden. Verwenden Sie die unten aufgeführte Liste der wichtigsten konfokalen Einstellungen und Faktoren, um die geeigneten Einstellungen zu bestimmen.
- Lochblendengröße: Bestimmen Sie diese durch das verwendete Objektiv und stellen Sie es so ein, dass es eine einzelne Fokusebene akzeptiert, während andere Fokusebenen eingeschränkt werden. Für dieses Protokoll wird eine Lochblende von 32 μm verwendet.
- Laserleistung und Wellenlänge: Passen Sie die Laserleistung basierend auf dem Expressionsniveau des fluoreszierenden Moleküls sowie der Tiefe und den optischen Eigenschaften des Gewebes an. Verwenden Sie die Entfernungsanzeige, um sicherzustellen, dass der Laser die Reichweite des Fotodetektors nicht überschreitet. Stellen Sie die Anregungswellenlänge auf die Wellenlänge des Lichts ein, das das fluoreszierende Molekül optimal anregt. Da die Fische für das GCaMP6s-Transgen heterozygot waren und die Expressionsniveaus niedrig waren, verwenden Sie für dieses Experiment eine Laserleistung von 5 % bei einer Wellenlänge von 488 nm und einer Master-Verstärkung von 650. Bei starker Transgenexpression liegt die Laserleistung zwischen 1,5 % und 3 %, was in der Regel ausreichend ist.
HINWEIS: Wenn die Laserleistung zu niedrig eingestellt ist, gehen Informationen (von der Photomultiplier-Röhre erkanntes Licht) verloren. Wenn die Laserleistung zu hoch eingestellt ist, kann das emittierte Licht die Reichweite des Detektors überschreiten oder die Probe beschädigen (Photobleiche). - Master-Gain: Der Master-Gain bestimmt die Empfindlichkeit des Fotodetektors. Verwenden Sie die Bereichsanzeige, um sicherzustellen, dass die Master-Verstärkung nicht zu hoch eingestellt ist und dadurch die Reichweite des Fotodetektors überschritten wird. Für dieses Experiment wird die Master-Verstärkung auf 650 eingestellt.
- Sichtfeld: Stellen Sie sicher, dass das Sichtfeld groß genug ist, um das interessierende Gewebe zu erfassen. Dies ist im Allgemeinen eine große Einschränkung für konfokale Mikroskope, da die Größe des Sichtfelds die Aufnahmegeschwindigkeit einschränkt, wie in Abbildung 2 gezeigt. Für die Experimente mit AITC stellen Sie das Sichtfeld auf 79,86 μm2 ein.
- Erfassungsgeschwindigkeit: Wenn die Erfassungsgeschwindigkeit erhöht wird, wird die vom Fotodetektor erfasste Lichtmenge reduziert. Wenn weniger Licht eingefangen wird, verschlechtert sich die Bildqualität. Stellen Sie sicher, dass die Aufnahmegeschwindigkeit die Qualität des Bildes und die Geschwindigkeit, die für die Erfassung des interessierenden physiologischen Ereignisses erforderlich ist, in Einklang bringt. Hier ist die Aufnahmegeschwindigkeit auf 1,20 f/s eingestellt.
- Zentrieren Sie das Gesichtsfeld auf einem neuronalen Cluster kaudal zur Commissura infima Halleri im rostralen Teil des Rückenmarks30. Dieser Cluster ist mit ~2,5 dpf recht klein, wird aber später in der Entwicklung viel größer (7 dpf).
HINWEIS: Wenn die Agarose-Montage in Schritt 1 suboptimal ist, d. h. wenn das Tier schräg oder zu tief in der Agarose positioniert ist, wird die Bildqualität beeinträchtigt. In Abbildung 3 sind ein Bild des Zebrafisch-Rückenmarks bei einem gut platzierten Tier und ein kontrastierendes Bild des Rückenmarks bei einem suboptimal platzierten Zebrafisch dargestellt. - Führen Sie einen Zeitreihenscan mit einem Sichtfeld von 79,86 μm2 bei 1,20 Bildern pro s (fps) und einer räumlichen Auflösung von 0,119 μm2 pro Pixel durch. Passen Sie die Größe des Sichtfelds und die Geschwindigkeit der Bildaufnahme je nach gewünschter Anwendung an.
- 2 Minuten nach Beginn der Bildgebung geben Sie mit einer Pipette 41,67 μl einer Stammlösung von AITC (10 μM Endkonzentration) oder E3-Lösung in die Schale. Setzen Sie die Aufzeichnung für ~30 s fort, um Änderungen in der GCaMP6s-Aktivität im interessierenden Bereich mit Zeitrafferaufnahmen zu beobachten. Nachdem die Aufzeichnung abgeschlossen ist, befreien Sie das Tier von der Agarose und euthanasieren Sie es gemäß den Richtlinien der institutionellen Tierpflege.
HINWEIS: Bei der Aufzeichnung der GCaMP6-Aktivität kann das konfokale Licht selbst ein Stimulus sein. Dies muss bei der Analyse der bildgebenden Ergebnisse berücksichtigt werden. Die Aufnahmezeit ist eine Funktion der Anzahl der Bilder und der Aufnahmerate jedes Bildes, die nach Bedarf für die gewünschte Zeit angepasst werden kann.
3. Analyse des GCaMP-Signals mit FIJI
- Wenn die Ausgabe nach der Aufzeichnung nicht im .tif Dateiformat für die nachgelagerte FIJI-Analyse vorliegt, exportieren Sie die Bilddateien als .tif Dateien aus der Mikroskop-Software-Suite.
- Laden Sie FIJI für die Analyse von neuronalen Spuren herunter. Öffnen Sie FIJI, importieren Sie die .czi-Dateien in das Programm und verwenden Sie bei Aufforderung die Standardeinstellungen im BioFormats-Plugin.
- Markieren Sie einen interessanten Bereich/ein Neuron mit den Kreis- oder Freihandwerkzeugen, indem Sie auf die entsprechenden Symbole klicken. (Wenn Sie mit der Maus über die Symbole fahren, blinkt der Name des Werkzeugs im Programm auf.)
HINWEIS: Da der Interessenbereich unterschiedlich groß ist, sind feste Bereiche/Kreise nicht möglich. Software, die die Registrierung von Fluoreszenzänderungen über die Zeit als Voxel ermöglicht, wie das Thunder-Paket32, könnte die Detektion einzelner Neuronen ermöglichen; Für die ~20 Neuronen im aktuellen Experiment wäre die Detektion der Membrangrenzen durch einen blinden Beobachter jedoch wahrscheinlich einem automatisierten Detektionssystem überlegen. - Nachdem Sie einen ROI ausgewählt haben, klicken Sie in der Symbolleiste "FIJI " auf "Analyze > Tools > ROI Manager". Klicken Sie danach auf Hinzufügen [t], und der ausgewählte ROI wird dem ROI-Manager-Fenster hinzugefügt.
- Klicken Sie im ROI-Manager auf Mehr > Multi-Measure. Behalten Sie die Standardeinstellungen bei, und klicken Sie auf OK. Fidschi wird Rohdaten produzieren, die als CSV-Datei mit Python oder als Rohdaten in einer Tabellenkalkulation bearbeitet werden können.
- Um die Rohdaten zu normalisieren und als neuronale Spur für einen bestimmten interessierenden Bereich darzustellen, mitteln Sie die letzten ~30 s-Werte des 2-Minuten-Fensters vor dem Stimulus und normalisieren Sie alle Post-Stimulus-Werte auf diesen durchschnittlichen Ausgangswert:
Normalisierte Fluoreszenzintensität = (momentane Fluoreszenz einer gegebenen Region von Interesse nach dem Stimulus)/(durchschnittliche Fluoreszenz von ~30 s eines 2-minütigen Intervalls vor dem Stimulus) - Stellen Sie die Daten als neuronale Kurven dar, wie in Abbildung 4 dargestellt.
HINWEIS: Bei einigen Arten von Mikroskopie, wie z. B. der Lichtblattmikroskopie, bei denen ein Volumen des Gehirns aufgezeichnet wird, werden Bewegungsartefakte durch Nachbearbeitung der räumlichen Registrierung auf ein Referenzvolumendes Gehirns 1,11 adressiert. Da wir jedoch nur von einer einzigen Ebene aus aufnehmen, können keine Anpassungen für Bewegungsartefakte vorgenommen werden. Daher wurden für die hier gezeigten Ergebnisse keine Frames entfernt. Sollten jedoch Bewegungsartefakte die Ergebnisse signifikant verändern, könnten automatisierte Systeme Frames mit den Bewegungsartefakten1, 11 identifizieren und korrigieren oder ein verblindeter Beobachter könnte solche Frames entfernen.
Ergebnisse
Die Verabreichung von Allylisothiocyanat verursacht ein Kalzium-assoziiertes neuronales Signal in Zebrafischlarven
Die Verabreichung von AITC (Schritt 2.6) führt zu einem weit verbreiteten Anstieg der GCaMP6s-assoziierten neuronalen Aktivität im Gehirn der Zebrafischlarven11,30. Wir beobachteten ein erhöhtes Fluoreszenzsignal in einer kleinen Region des Gehirns nach der Anwendung von AITC, wie in Abbildung 4 gezeigt.
Ausgleich der Auflösung von Kalzium-assoziierten neuronalen Signalen, der Erfassungsgeschwindigkeit und des Erfassungsbereichs
Durch die Erhöhung der Aufnahmegeschwindigkeit verringert sich die Auflösung, was bei der Versuchsplanung berücksichtigt werden muss. In Abbildung 5 zeigen wir eine Vielzahl von Geschwindigkeiten und die entsprechenden Auflösungen anhand von Zebrafischembryonen bei ~2,5 dph. Bei der Aufzeichnung neuronaler Aktivität muss die Erfassungsgeschwindigkeit ausreichend hoch sein, um die Aktivität aufzuzeichnen. Obwohl die Spannungsänderungen aufgrund eines Aktionspotentials relativ schnell sind (1-2 ms), verlängern Kalziumwechselwirkungen mit GCaMP die Dauer des Aktionspotentialsignals. GCaMP6s, das hier verwendete Molekül, weist eine besonders langsame Kinetik auf. Einzelaktionspotentiale mit GCaMP-Molekülen können in Zellkultur nachgewiesen und korreliert werden6, aber es ist unklar, ob diese Präzision der Signaldetektion in vivo bei Zebrafischlarven erfolgen kann. Je höher jedoch die Erfassungsgeschwindigkeit ist, desto wahrscheinlicher ist es, dass neuronale aktivitätsbezogene zeitliche Informationen erfasst werden, vorausgesetzt, die Signalerkennung wird durch die höheren Erfassungsgeschwindigkeiten nicht signifikant reduziert. Darüber hinaus beeinflusst die Größe des Beobachtungsfeldes auch die Aufnahmegeschwindigkeiten mit einem konfokalen Standardmikroskop. Durch die Verkleinerung des Sichtfeldes erhöht man die Erfassungsgeschwindigkeit, verliert aber Informationen über die neuronale Aktivität in anderen Gehirnbereichen. Um GCaMP6-Signale zu erfassen, müssen Anwendungen mit konfokaler Mikroskopie auf ein kleines Hirnareal (50-150 μm2) beschränkt werden. Die Erfassungsgeschwindigkeit sollte schneller sein als das beobachtete Signal, um Aliasing zu reduzieren. Die Einfanggeschwindigkeiten der konfokalen Mikroskopie führen jedoch unweigerlich zum Verlust einiger Informationen in Form von emittiertem Licht. Diese relativen Kosten und Vorteile müssen immer berücksichtigt werden, um die gewünschte experimentelle Fragestellung effektiv zu beantworten.

Abbildung 1: Darstellung der Position einer Zebrafischlarve, die für die Bildgebung teilweise eingeschränkt ist. Die Larve wird in eine Agarose mit niedrigem Schmelzpunkt eingebettet und mit der Rückenseite nach oben positioniert. Rostrale und kaudale Anteile der Agarose wurden entfernt, damit die Haut des Zebrafisches AITC ausgesetzt werden konnte. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
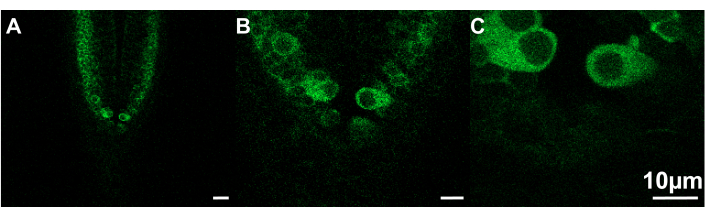
Abbildung 2: Beispiele für Bilder unterschiedlicher Größe, die mit unterschiedlichen Aufnahmegeschwindigkeiten angezeigt werden können. (A) Bildgröße = 319,45 x 319,45 μm; Aufnahmegeschwindigkeit = .30 fps. (B) Rahmengröße = 106,48 x 106,48 μm; Aufnahmegeschwindigkeit = 7,67 fps. (C) Rahmengröße = 53,24 x 53,24 μm; Aufnahmegeschwindigkeit = 9,80 fps. Zebrafisch, ~2,5 dpf; Maßstabsleiste 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
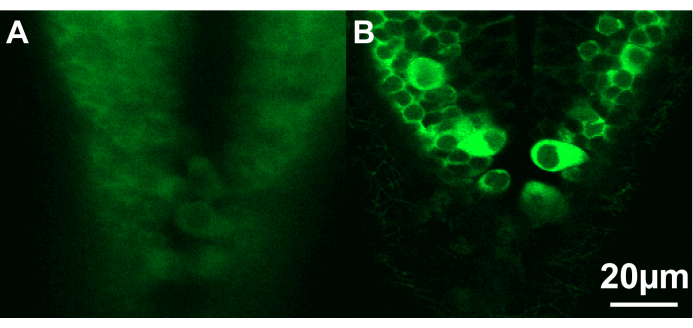
Abbildung 3: Konfokale Bilder des kaudalen Teils des Hinterhirns von zwei Zebrafischlarven. (A) Die Larve (~2,5 dpf) ist in einem unidealen Winkel und zu tief in der Agarose positioniert, was eine scharfe Abbildung der Neuronen verhindert. (B) Hier ist die Larve (~2,5 dpf) richtig in der Nähe der Oberfläche der Agarose positioniert. Maßstabsleiste 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
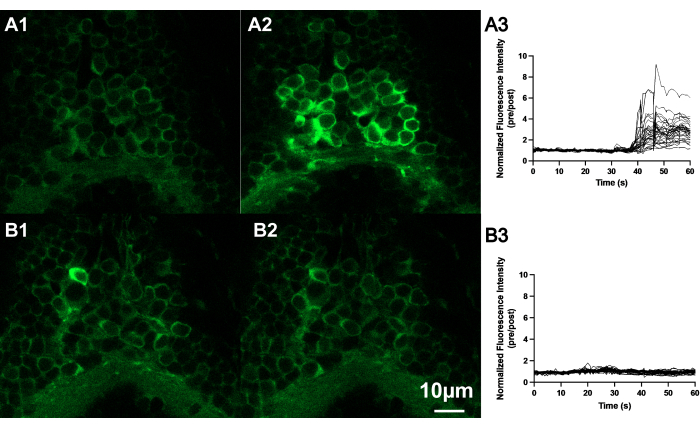
Abbildung 4: AITC erhöht die Fluoreszenzintensität von Neuronen, die sich an der Grenze zwischen Hinterhirn und Rückenmark des Zebrafischgehirns befinden. (A) Repräsentative konfokale Bilder von Zebrafisch-Neuronen (7 dpf) bei Exposition gegenüber AITC oder E3. Bilder von Neuronen vor (A1) und nach (A2) Applikation von AITC im Vergleich zu (B) Bildern von Neuronen vor (B1) und nach (B2) Applikation der Kontrolllösung (E3). Maßstabsbalken 10 μM. Spuren des normalisierten Fluoreszenzzustands mehrerer Neuronen im Hinterhirn von embryonalen Zebrafischen, die AITC (A3) oder E3 (B3) ausgesetzt waren. Es ist zu beachten, dass die Neuronen nach dem Auftragen von AITC auf das Bad eine erhöhte Fluoreszenz aufwiesen, während bei den Neuronen nach der Anwendung von E3 keine erhöhte Fluoreszenz beobachtet wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Die Aufnahmegeschwindigkeit von GCaMP6s bestimmt die Menge des eingefangenen Lichts und die resultierende Auflösung des Bildes. (A) Bilder von Neuronen, die alle 10,13 s (0,10 fps) aufgenommen werden, ergeben eine relativ hohe Auflösung von Neuronen im Hinterhirn und Rückenmark. Mit zunehmender Aufnahmegeschwindigkeit werden zeitliche Informationen gewonnen, aber die räumliche Auflösung geht verloren: (B) Aufnahmegeschwindigkeit = 0,79 fps, (C) Aufnahmegeschwindigkeit = 3,16 fps und (D) Aufnahmegeschwindigkeit = 7,67 fps. Zebrafisch, 2 dpf; Maßstabsleiste 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Wir haben gezeigt, dass die neuronale Aktivität im Gehirn von Zebrafischlarven mit Hilfe von GCaMP6s zusammen mit konfokaler Mikroskopie aufgezeichnet werden kann; Die geringeren Aufnahmegeschwindigkeiten, die aufgrund der langsameren Kinetik von GCaMPs erforderlich sind, können durch eine Reduzierung des beobachteten Gehirnbereichs kompensiert werden6. Reporter mit schnellerer zeitlicher Dynamik (z. B. GCaMP6f) sind verfügbar, aber die überlegene zeitliche Auflösung geht in der Regel auf Kosten eines reduzierten Fluoreszenzsignals6. Das konfokale Mikroskop ist auf relativ langsamere Aufnahmegeschwindigkeitenbeschränkt 22, daher sollten Reportermoleküle unter Berücksichtigung der zeitlichen Grenzen der konfokalen Mikroskopie ausgewählt werden.
Das Protokoll ist durch zwei Hauptmerkmale eingeschränkt: 1) die Anforderung, dass das Tier während der Aufzeichnung in Agarose gelegt werden muss, und 2) die relativ langsame Aufnahmegeschwindigkeit der konfokalen Mikroskopie. Das Gleichgewicht zwischen Sichtfeld und Erfassungsgeschwindigkeit ist der Schlüssel zur Erfassung neuronaler Aktivität mit ausreichender Auflösung für eine bestimmte Anwendung. Wenn die AITC-induzierte Aktivität verkürzt oder vermindert ist, könnte dies darauf hindeuten, dass die Zebrafischlarve den Reiz nicht erkennen kann oder dass der AITC nicht ausreichend wirksam ist. Im letzteren Fall verlängert das Auffrischen der Brühe und das Einfrieren bei -20 °C die Wirksamkeit. Wenn der Fisch den Reiz nicht erkennen kann, wurde das Tier möglicherweise zu tief in den Agar implantiert. Ein kritischer Schritt ist die Positionierung eines Fisches während Schritt 1 des Protokolls, da dies für einen optimalen optischen Zugang während der Anwendung des Medikaments entscheidend ist.
Aufgrund der relativ langsamen Scangeschwindigkeiten der konfokalen Mikroskopie sind Ganzhirnaufnahmen technisch nicht möglich. Bezeichnenderweise kann das hier skizzierte Protokoll verwendet werden, um kleine Hirnareale nach der Identifizierung mit komplementären Techniken zu messen. Insbesondere durch die Verwendung von Techniken, die Moleküle nutzen, die die neuronale Aktivität mit aktivitätsabhängigen Reportern markieren 9,11, kann der Experimentator Schlüsselbereiche des Gehirns ansprechen, die für die Anwendung der in diesem Artikel beschriebenen Methoden von Interesse sind. Die interessierenden Hirnareale können dann auf den annotierten Zebrafisch-Gehirnatlas, z Hirnatlas11, bezogen werden.
Mit geringfügigen Anpassungen können die hier besprochenen Techniken und Richtlinien eingesetzt werden, um eine Reihe von Gehirnfunktionen zu untersuchen, einschließlich sensomotorischer Verarbeitung, Lernen und Gedächtnis sowie Empfindung und Wahrnehmung.
Offenlegungen
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Diese Arbeit wurde durch einen Zuschuss der National Institutes of Health (SC2GM1304854) an die ACR und einen Zuschuss der National Science Foundation (2050850) an die DLG unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Low Melting Point Agarose | Invitrogen | 16520-100 | Diluted to 3% |
| Allyl Isothiocyanate (AITC) | Sigma Aldrich | 377430 | Chemical stimulant |
| E3 | N/A | N/A | Water-medium for zebrafish larvae |
| Glass Bottom Dishes | Thermo Fisher Scientific | 12-567-400 | Used to hold zebrafish during imaging experiments |
| Micropipette (10-100 uL) | Cole-Parmer | 21600-14 | Apparatus used for creating AITC dilutions |
| Microscope Slides | Fisherbrand | 12-550-A3 | Used to screen for phenotype |
| Mirror Finish Forceps | DUMONT | 11251-23 | Used to orient zebrafish in agarose |
| myTEMP Mini Digital Incubators | Benchmark | H2200-HC | Holding area for zebrafish; set to 28.5°C |
| Nitrile Gloves | MedPRIDE | MPR-50504 | Basic PPE |
| Petri Dishes | VWR | 89107-632 | Container for zebrafish |
| Posi-Click Tubes | DENVILLE | C-2171 | Used for AITC dilution |
| Samco Polyurethane Transfer Pipettes | Thermo Fisher Scientific | 225 | Apparatus used to select animal/administer diluted bolus of AITC |
| Stemi SV11 Apo Microscope | Zeiss | 1.25496E+11 | Used to stage zebrafish |
| Transgenic Larval Zebrafish (2 to 7 DPF) | N/A | N/A | Animal test subjects; Tg(elav3:GCaMP6s) strain |
| Zeiss Confocal Microscope (Model LSM9) | Zeiss | 3523004097 | Imaging of fish |
Referenzen
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nat Method. 10 (5), 413-420 (2013).
- Meyer, M. P., Smith, S. J. Evidence from in vivo imaging that synaptogenesis guides the growth and branching of axonal arbors by two distinct mechanisms. J Neurosci. 26 (13), 3604-3614 (2006).
- Sagasti, A., Guido, M. R., Raible, D. W., Schier, A. F. Repulsive interactions shape the morphologies and functional arrangement of zebrafish peripheral sensory arbors. Curr Biol. 15 (9), 804-814 (2005).
- Son, J. H., et al. Transgenic FingRs for live mapping of synaptic dynamics in genetically-defined neurons. Sci Rep. 6, 18734 (2016).
- Zada, D., Tovin, A., Lerer-Goldshtein, T., Vatine, G. D., Appelbaum, L. Altered behavioral performance and live imaging of circuit-specific neural deficiencies in a zebrafish model for psychomotor retardation. PLoS Genet. 10 (9), e1004615 (2014).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Abdelfattah, A. S., et al. and photostable chemigenetic indicators for extended in vivo voltage imaging. Science. 365 (6454), 699-704 (2019).
- Abdelfattah, A. S., et al. Sensitivity optimization of a rhodopsin-based fluorescent voltage indicator. Neuron. 111 (10), 1547-1563.e9 (2023).
- Gao, Y. J., Ji, R. R. c-Fos and pERK, which is a better marker for neuronal activation and central sensitization after noxious stimulation and tissue injury. Open Pain J. 2, 11-17 (2009).
- Kawashima, T., Okuno, H., Bito, H. A new era for functional labeling of neurons: activity-dependent promoters have come of age. Front Neural Circuits. 8, 37 (2014).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nat Methods. 12, 1039-1046 (2015).
- Reijmers, L. G., Perkins, B. L., Matsuo, N., Mayford, M. Localization of a stable neural correlate of associative memory. Science. 317 (5842), 1230-1233 (2007).
- Mohr, M. A., Argast, P., Pantazis, P. Labeling cellular structures in vivo using confined primed conversion of photoconvertible fluorescent proteins. Nature Protoc. 11 (12), 2419-2431 (2016).
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc Natl Acad Sci U S A. 106 (42), 17968-17973 (2009).
- Wyart, C., Del Bene, F. Let there be light: zebrafish neurobiology and the optogenetic revolution. Rev Neurosci. 22 (1), 121-130 (2011).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr Biol. 18 (15), 1133-1137 (2008).
- Portugues, R., Severi, K. E., Wyart, C., Ahrens, M. B. Optogenetics in a transparent animal: circuit function in the larval zebrafish. Curr Opinion Neurobiol. 23 (1), 119-126 (2013).
- Best, J. D., Alderton, W. K. Zebrafish: An in vivo. model for the study of neurological diseases. Neuropsychiat Dis Treatment. 4 (3), 567-576 (2008).
- Goldsmith, P. Zebrafish as a pharmacological tool: the how, why and when. Curr Opinion Pharmacol. 4 (5), 504-512 (2004).
- Roberts, A. C., et al. Habituation of the C-start response in larval zebrafish exhibits several distinct phases and sensitivity to NMDA receptor blockade. PloS One. 6 (12), e29132 (2011).
- Wolman, M. A., Jain, R. A., Liss, L., Granato, M. Chemical modulation of memory formation in larval zebrafish. Proc Natl Acad Sci U S A. 108 (37), 15468-15473 (2011).
- Ji, N., Freeman, J., Smith, S. L. Technologies for imaging neural activity in large volumes. Nat Neurosci. 19 (9), 1154-1164 (2016).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Front Neural Circuits. 7, 65 (2013).
- Distel, M., Köster, R. W. In vivo time-lapse imaging of zebrafish embryonic development. CSH Protoc. 2007, (2007).
- Liu, T. T., Hou, H., Du, J. L. A protocol for simultaneous Ca2+ and morphology imaging of brain endothelial tip cells in larval zebrafish. STAR Protoc. 2 (1), 100388 (2021).
- Wong, H. C., Drerup, C. M. Using fluorescent indicators for in vivo quantification of spontaneous or evoked motor neuron presynaptic activity in transgenic zebrafish. STAR Protoc. 3 (4), 101766 (2022).
- Graeden, E., Sive, H. Live imaging of the zebrafish embryonic brain by confocal microscopy. J Vis Exp. (26), e1217 (2009).
- Hirsinger, E., Steventon, B. A versatile mounting method for long term imaging of zebrafish development. J Vis Exp. (119), e55210 (2017).
- Renaud, O., Herbomel, P., Kissa, K. Studying cell behavior in whole zebrafish embryos by confocal live imaging: application to hematopoietic stem cells. Nat Protoc. 6 (12), 1897-1904 (2011).
- Roberts, A. C., et al. Induction of short-term sensitization by an aversive chemical stimulus in zebrafish larvae. eNeuro. 7 (6), (2020).
- Prober, D. A., et al. Zebrafish TRPA1 channels are required for chemosensation but not for thermosensation or mechanosensory hair cell function. J Neurosci. 28 (40), 10102-10110 (2008).
- Freeman, J., et al. Mapping brain activity at scale with cluster computing. Nat Methods. 11, 941-950 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten