Method Article
In vivo Imágenes de fluorescencia confocal de la actividad neuronal inducida por estimulación sensorial en larvas de pez cebra parcialmente restringidas
En este artículo
Resumen
Aquí, presentamos un protocolo detallado para examinar la actividad neuronal en regiones cerebrales de pez cebra transgénico que expresan indicadores de calcio GCaMP utilizando microscopía confocal.
Resumen
Las larvas de pez cebra son un prometedor sistema modelo de vertebrados para estudiar los mecanismos neuronales del comportamiento. Su translucidez y circuitos neuronales relativamente simples facilitan el uso de técnicas optogenéticas en los análisis celulares del comportamiento. Los indicadores fluorescentes de la actividad neuronal in vivo , como los GCaMP6, se han utilizado ampliamente para estudiar la actividad neuronal asociada con comportamientos simples en las larvas de pez cebra. Aquí, presentamos un protocolo para detectar la actividad inducida por la sensorialidad en larvas de pez cebra semi-restringidas utilizando la línea transgénica Tg(elav3:GCaMP6s). En particular, utilizamos el agente químico isotiocianato de alilo para inducir una respuesta fluorescente robusta y reproducible en una región del cerebro en el borde del cerebro posterior y la médula espinal. Discutimos los usos potenciales de GCaMP6s para el monitoreo óptico de la actividad neuronal durante una variedad de paradigmas de comportamiento y las limitaciones de esta técnica. Nuestro protocolo describe un enfoque accesible para monitorear la actividad neuronal in vivo dinámica y relacionada con el comportamiento en el cerebro de las larvas de pez cebra.
Introducción
El pez cebra representa un modelo animal de vertebrado con capacidad de tratamiento para investigaciones neurobiológicas celulares y moleculares detalladas. Las larvas de pez cebra poseen ~ 100,000 neuronas a los 5 días después de la fertilización (dpf), significativamente menos que los cerebros de los mamíferos. Además, los peces cebra son relativamente translúcidos, una propiedad que facilita los estudios ópticos de la estructura y función neuronal 1,2,3,4,5. Se han desarrollado varias herramientas optogenéticas para su uso en el pez cebra, incluidos los indicadores de calciode alta fidelidad 6, los sensores de voltaje 7,8 y los marcadores dependientes de la actividad neuronal 9,10,11,12,13. Estas herramientas son complementarias a otras ventajas que posee este modelo, como la susceptibilidad a las modificaciones genéticas 14,15,16,17 y la prontitud con la que las larvas de pez cebra absorben los químicos presentes en las soluciones de baño 18,19,20,21.
Una variedad de métodos son útiles para la fisiología óptica del pez cebra, particularmente la microscopía de dos fotones, de lámina de luz y confocal. Cada una de estas tecnologías debe equilibrar dos problemas relacionados de resolución: el acceso óptico, incluida la dispersión de la luz por el tejido circundante, y la velocidad de muestreo, especialmente para capturar la cinética del potencial de acción en una escala inferior al milisegundo22. Ha habido mejoras dramáticas en la obtención de imágenes de calcio in vivo utilizando microscopía de dos fotones, pero este método a menudo está restringido a un campo de visión de <1mm2 y, por lo general, solo se puede adquirir un solo plano de profundidad, lo que limita la captura de actividad a través de grandes regiones de circuitos neuronales22. Para la microscopía de lámina de luz, el potencial de registrar la actividad de casi todas las neuronas en el cerebro resuelve la limitación del campo de visión de la microscopía de dos fotones, pero las velocidades actuales de las cámaras limitan físicamente la captura a aproximadamente tres volúmenes cerebrales por segundo a 40 planos por volumen cerebral en las larvas de pez cebra 1,23. La microscopía confocal es inferior tanto en profundidad como en resolución y velocidad de captura a la microscopía de dos fotones y lámina de luz. La microscopía confocal tiene las ventajas de una amplia accesibilidad a los laboratorios de todo el mundo y la capacidad de lograr reconstrucciones de la actividad neuronal de todo el cerebro utilizando indicadores de la actividad neuronal, como cFos y p-ERK9. Además, si se dirigen a pequeñas regiones cerebrales, el microscopio confocal puede proporcionar una resolución temporal adecuada de la actividad neuronal.
El presente artículo describe un método que utiliza la microscopía confocal para registrar la actividad neuronal en peces cebra transgénicos que expresan GCaMP6s panneuronalmente. Se han desarrollado varios protocolos similares con larvas de pez cebra para comprender la función de las vías neuronales 24,25,26,27,28,29. Las características clave de varios de estos protocolos, como las imágenes de lapso de tiempo, los indicadores fluorescentes de la dinámica del calcio y las imágenes en vivo, se han combinado para medir la actividad neuronal en una pequeña población de neuronas en el sistema nervioso central del pez cebra en respuesta al isotiocianato de alilo (AITC), un irritante químico aversivo 11,26,27,29,30,31 . AITC provoca una respuesta de todo el cerebro centrada en el área posterior del cerebro11. Un grupo de neuronas justo caudal al cerebro posterior tiene un papel en la locomoción y una respuesta prolongada a la AITC. Esta respuesta dura más que la eliminación del estímulo aversivo30. Al restringir el campo de visión, hemos logrado detectar la actividad neuronal en este grupo neuronal, reflejada por el cambio de fluorescencia en las neuronas que expresan GCaMP6s. Proporcionamos técnicas, pautas y mejores prácticas para lograr una resolución espacio-temporal suficiente utilizando microscopía confocal. Además, discutimos las limitaciones de nuestro método de grabación óptica. A pesar de estas limitaciones, el método debería permitir la investigación de una variedad de fenómenos neurobiológicos, incluyendo la memoria y el procesamiento sensoriomotor.
Protocolo
Todos los procedimientos con animales fueron aprobados por el Comité Institucional de Uso de Cuidado de Animales de la Universidad Estatal de California, Fullerton (Protocolo # 2023-1310).
1. Estadificación de larvas de pez cebra en agarosa de bajo punto de fusión
- Cría animales transgénicos adultos de Tg(elav3:GCaMP6s)6 panneuronalmente con dos machos y dos hembras de acuerdo con las pautas institucionales de cuidado animal. Los adultos se colocan en cámaras especiales de cría para que los huevos liberados se capturen en una cámara inferior con un falso fondo que no es accesible para los adultos. Con la primera luz del día o cuando se retira un divisor de separación, las hembras liberan huevos y los machos fertilizan los huevos capturados en el falso fondo.
- Retire los adultos y recoja los óvulos fertilizados con un colador de huevos y colóquelos en el medio embrionario (E3) (5 mM de NaCl, 0,33 mM de MgSO4, 0,33 mM de CaCl2, 0,17 mM de KCl, 1 mM de HEPES, 0,00001% de azul de metileno, pH 7,0)30 con 14 h de luz/10 h de oscuridad a 28,5 °C dentro de una incubadora
NOTA: La receta E3 utilizada tiene una baja concentración de azul de metileno que no afecta la fluorescencia ni conduce a ninguna señal de autofluorescencia significativa. No requerimos el uso de 1-fenil-2-tiourea (PTU) en las edades de desarrollo especificadas en el protocolo debido a la translucidez natural que proporciona la línea transgénica GCaMP6s. - Monte los animales 2-7 días después de la fertilización (dpf) utilizando un 3% de agarosa de bajo punto de fusión disuelta en medio E3. Calentar la agarosa a una temperatura a la que el pez cebra pueda ser manipulado sin dañar el organismo. Antes de que la agarosa del punto de fusión bajo se enfríe y solidifique (<1 min), coloque las larvas con el lado dorsal hacia arriba en una placa de Petri con fondo de vidrio para que la región del cerebro a inspeccionar esté lo más cerca posible del objetivo, como se muestra en la Figura 1. Dependiendo del experimento, por lo general, de 8 a 10 peces cebra por grupo experimental es suficiente.
NOTA: Los animales no fueron anestesiados en este protocolo. - Después de que la agarosa se haya cuajado con la larva en su lugar (aproximadamente 2 a 3 minutos), agregue 5 ml de solución E3. A continuación, corte la agarosa con un bisturí como exige el protocolo experimental.
NOTA: Se deben cortar diferentes partes del agar, dependiendo de qué partes del pez cebra necesiten liberarse de la restricción y / o recibir un tipo diferente de estímulo. - Mueva la placa de Petri que contiene el pez cebra a la etapa de microscopio confocal y deje que el pez se aclimate durante 20 minutos.
2. Configuración e imagen bajo microscopía confocal con aplicación de estímulos
- Después del período de aclimatación, centre a los peces bajo el objetivo de inmersión en agua de 40x (NA 1.0) a temperatura ambiente utilizando un campo claro con una fuente de luz (~475 nm).
- Los ajustes de adquisición del microscopio confocal dependerán de las cualidades y el nivel de expresión de la molécula fluorescente, la profundidad y las propiedades ópticas del tejido, el tamaño del campo de visión y la velocidad de registro. Determine los ajustes apropiados utilizando el indicador de rango para garantizar una captura óptima de la luz emitida de acuerdo con las necesidades experimentales.
- Garantice el ajuste adecuado de la potencia del láser y la ganancia maestra para capturar la luz fluorescente en el rango óptimo, evitando así la pérdida innecesaria de la señal de fluorescencia dependiente de GCaMP6s. Utilice la lista de configuraciones confocales clave y factores que se detallan a continuación para determinar la configuración adecuada.
- Tamaño del agujero de alfiler: Determine esto por el objetivo utilizado y ajústelo para que acepte un solo plano focal mientras se restringen otros planos focales. Para este protocolo, se utiliza un orificio de 32 μm.
- Potencia del láser y longitud de onda: Ajuste la potencia del láser en función del nivel de expresión de la molécula fluorescente y la profundidad y las propiedades ópticas del tejido. Utilice el indicador de rango para asegurarse de que el láser no exceda el alcance del fotodetector. Ajuste la longitud de onda de excitación a la longitud de onda de la luz que excita de manera óptima la molécula fluorescente. Para este experimento, debido a que los peces eran heterocigotos para el transgén GCaMP6s y los niveles de expresión eran bajos, se utilizó una potencia láser del 5% a una longitud de onda de 488 nm, una ganancia maestra de 650. Con una fuerte expresión de transgenes, la potencia del láser oscila entre el 1,5% y el 3%, lo que suele ser suficiente.
NOTA: Si la potencia del láser se establece demasiado baja, se perderá la información (luz detectada por el tubo fotomultiplicador). Si la potencia del láser se establece demasiado alta, la luz emitida podría exceder el alcance del detector o dañar la muestra (fotoblanqueo). - Ganancia maestra: La ganancia maestra determina la sensibilidad del fotodetector. Utilice el indicador de rango para asegurarse de que la ganancia maestra no se establezca demasiado alta, excediendo así el alcance del fotodetector. Para este experimento, la ganancia maestra se establece en 650.
- Campo de visión: asegúrese de que el campo de visión sea lo suficientemente grande como para capturar el tejido de interés. Esta es una limitación importante para los microscopios confocales en general porque el tamaño del campo de visión limitará la velocidad de captura, como se muestra en la Figura 2. Para los experimentos con AITC, establezca el campo de visión en 79,86 μm2.
- Velocidad de captura: A medida que se aumenta la velocidad de captura, se reducirá la cantidad de luz capturada por el fotodetector. A medida que se capture menos luz, la calidad de la imagen se degradará. Asegúrese de que la velocidad de captura equilibre la calidad de la imagen y la velocidad necesaria para capturar el evento fisiológico de interés. Aquí, la velocidad de captura se establece en 1,20 f/s.
- Centre el campo de visión en un grupo neuronal caudal a la comisura infima Halleri en la porción rostral de la médula espinal30. Este cúmulo es bastante pequeño a ~2,5 dpf, pero es mucho más grande más adelante en el desarrollo (7 dpf).
NOTA: Si el montaje con agarosa en el paso 1 no es óptimo, es decir, el animal está colocado en ángulo o demasiado profundamente en la agarosa, la calidad de la imagen se verá afectada negativamente. En la Figura 3 se muestra una imagen de la médula espinal del pez cebra en un animal bien ubicado y una imagen contrastante de la médula espinal en un pez cebra colocado de manera subóptima. - Realice un escaneo de series temporales con un campo de visión de 79,86 μm2 a 1,20 fotogramas por s (fps) con una resolución espacial de 0,119 μm2 por píxel. Ajuste el tamaño del campo de visión y la velocidad de adquisición de imágenes de acuerdo con la aplicación deseada.
- A los 2 minutos después del inicio de la imagen, agregue 41,67 μL de una solución madre de AITC (concentración final de 10 μM) o E3 a la placa con una pipeta. Continúe grabando durante ~30 s para observar cualquier cambio en la actividad de GCaMP6s en la región de interés con imágenes de lapso de tiempo. Una vez completada la grabación, libere al animal de la agarosa y eutanasiarlo según las pautas institucionales de cuidado animal.
NOTA: Al registrar la actividad de GCaMP6s, la luz confocal en sí misma puede ser un estímulo. Esto debe tenerse en cuenta en el análisis de los resultados de las imágenes. El tiempo de grabación es una función del número de fotogramas y la velocidad de captura de cada fotograma, que se puede ajustar según sea necesario para el tiempo deseado.
3. Análisis de la señal GCaMP usando FIJI
- Después de la grabación, si la salida no está en el formato de archivo .tif para el análisis FIJI posterior, exporte los archivos de imagen como archivos .tif desde el paquete de software de microscopio.
- Descargue FIJI para el análisis de trazas neuronales. Abra FIJI, importe los archivos .czi en el programa y, cuando se le solicite, use la configuración predeterminada en el complemento BioFormats.
- Resalte un área/neurona de interés utilizando las herramientas Círculo o Mano alzada haciendo clic en sus respectivos iconos. (Al pasar el cursor sobre los iconos, parpadeará el nombre de la herramienta en el programa).
NOTA: Debido a que el área de interés varía en tamaño, no son posibles las áreas fijas/círculos. El software que permite el registro de cambios fluorescentes a lo largo del tiempo como vóxeles, como el paquete Thunder32, podría permitir la detección de neuronas individuales; Sin embargo, para las ~20 neuronas en el experimento actual, la detección de los bordes de la membrana por parte de un observador ciego probablemente sería superior a un sistema de detección automatizado. - Una vez seleccionado un ROI, en la barra de herramientas FIJI, haga clic en Analizar herramientas > > Administrador de ROI. Después de eso, haga clic en Agregar [t] y el ROI seleccionado se agregará a la ventana del Administrador de ROI.
- En el Administrador de ROI, haga clic en Más > Multi Measure. Deje la configuración predeterminada y haga clic en Aceptar. FIJI producirá datos sin procesar que se pueden manipular como un archivo CSV usando Python o como datos sin procesar en una hoja de cálculo.
- Para normalizar los datos sin procesar y representarlos como un rastro neuronal para una región de interés determinada, promedie los últimos ~30 s de los últimos ~30 s de la ventana de 2 minutos antes del estímulo y normalice todos los valores posteriores al estímulo a ese valor de referencia promedio:
Intensidad de fluorescencia normalizada = (fluorescencia instantánea de una región de interés dada después del estímulo)/(fluorescencia promedio de ~30 s de un intervalo de 2 minutos antes del estímulo) - Represente los datos como trazas neuronales, como se muestra en la figura 4.
NOTA: En algunos tipos de microscopía, como la microscopía de lámina de luz, en la que se registra un volumen del cerebro, los artefactos de movimiento se abordan mediante el registro espacial de posprocesamiento a un volumen cerebral de referencia 1,11. Sin embargo, debido a que solo estamos grabando desde un solo plano, no se pueden realizar ajustes para los artefactos de movimiento. Por lo tanto, para los resultados que se muestran aquí, no se eliminó ningún fotograma. Sin embargo, si los artefactos de movimiento alteran significativamente los resultados, los sistemas automatizados podrían identificar y corregir los fotogramas con los artefactos de movimiento1, 11, o un observador ciego podría eliminar dichos fotogramas.
Resultados
La administración de isotiocianato de alilo provoca una señal neuronal asociada al calcio en las larvas de pez cebra
La administración de AITC (paso 2.6) provoca un aumento generalizado de la actividad neuronal asociada a GCaMP6s en todo el cerebro de las larvas de pez cebra11,30. Observamos un aumento de la señal fluorescente en una pequeña región del cerebro después de aplicar AITC, como se muestra en la Figura 4.
Equilibrar la resolución de las señales neuronales asociadas al calcio, la velocidad de captura y el área de captura
El aumento de la velocidad de captura reduce la resolución, lo que debe tenerse en cuenta en el diseño experimental. Presentamos una variedad de velocidades y sus correspondientes resoluciones utilizando embriones de pez cebra a ~2.5 dpf en la Figura 5. Al registrar la actividad neuronal, la velocidad de captura debe ser lo suficientemente rápida como para registrar la actividad. Aunque los cambios de voltaje debidos a un potencial de acción son relativamente rápidos (1-2 ms), las interacciones del calcio con GCaMP prolongarán la duración de la señal de potencial de acción. GCaMP6s, la molécula utilizada aquí, tiene una cinética particularmente lenta. Los potenciales de acción individuales utilizando moléculas GCaMP pueden detectarse y correlacionarse en el cultivo celular6, pero no está claro si esta precisión de la detección de señales podría ocurrir in vivo con larvas de pez cebra. Sin embargo, cuanto mayor sea la velocidad de captura, más probable es que se capture información temporal relacionada con la actividad neuronal, suponiendo que la detección de señales no se reduzca significativamente con las velocidades de captura más altas. Además, el tamaño del campo de observación también influirá en las velocidades de captura con un microscopio confocal estándar. Al reducir el campo de visión, se aumenta la velocidad de captura, pero se pierde información sobre la actividad neuronal en otras áreas del cerebro. Para capturar señales GCaMP6, las aplicaciones que utilizan microscopía confocal deben limitarse a un área cerebral pequeña (50-150 μm2). La velocidad de captura debe ser más rápida que la señal que se observa para reducir el solapamiento. Sin embargo, las velocidades de captura de la microscopía confocal resultarán inevitablemente en la pérdida de cierta información en forma de luz emitida. Estos costos y beneficios relativos siempre deben ser considerados para abordar la pregunta experimental deseada de manera efectiva.

Figura 1: Representación de la posición de una larva de pez cebra parcialmente restringida para la toma de imágenes. La larva está incrustada en un agarosa de bajo punto de fusión y colocada con el lado dorsal hacia arriba. Las porciones rostral y caudal de la agarosa se eliminaron para permitir que la piel del pez cebra quedara expuesta al AITC. Haga clic aquí para ver una versión más grande de esta figura.
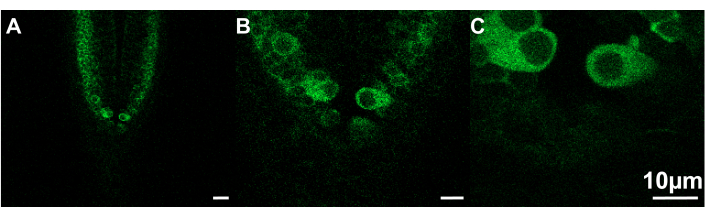
Figura 2: Ejemplos de imágenes de diferentes tamaños que se pueden ver a diferentes velocidades de captura. (A) Tamaño del fotograma = 319,45 x 319,45 μm; Velocidad de captura = .30 fps. (B) Tamaño del marco = 106,48 x 106,48 μm; Velocidad de captura = 7,67 fps. (C) Tamaño del marco = 53,24 x 53,24 μm; Velocidad de captura = 9,80 fps. Pez cebra, ~2.5 dpf; barra de escala de 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
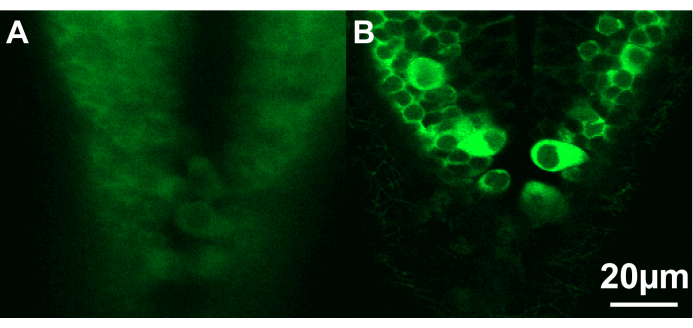
Figura 3: Imágenes confocales de la porción caudal del cerebro posterior de dos larvas de pez cebra. (A) La larva (~2,5 dpf) está colocada en un ángulo no ideal y demasiado profundamente en la agarosa, lo que impide obtener imágenes nítidas de las neuronas. (B) Aquí, la larva (~2.5 dpf) está correctamente colocada cerca de la superficie de la agarosa. Barra de escala de 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
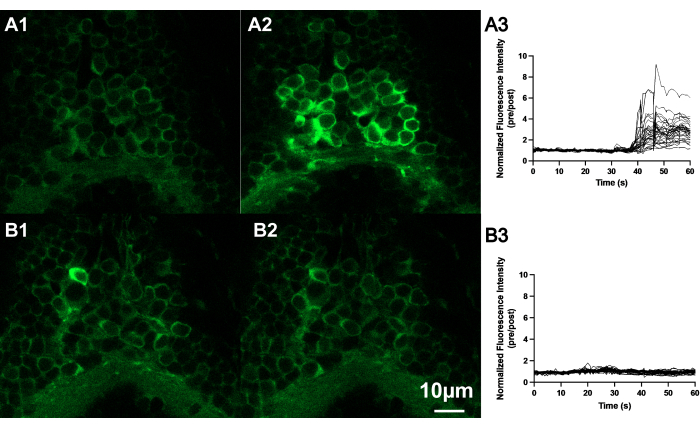
Figura 4: AITC aumenta la intensidad de fluorescencia de las neuronas ubicadas en el borde del cerebro posterior y la médula espinal del cerebro del pez cebra. (A) Imágenes confocales representativas de neuronas de pez cebra (7 dpf) cuando se exponen a AITC o E3. Imágenes de neuronas antes (A1) y después (A2) de la aplicación de AITC comparadas con (B) imágenes de neuronas antes (B1) y después (B2) de la aplicación de la solución de control (E3). Barra de escala 10 μM. Trazas del estado fluorescente normalizado de varias neuronas localizadas en el cerebro posterior de pez cebra embrionario expuesto a AITC (A3) o E3 (B3). Nótese que las neuronas mostraron un aumento de la fluorescencia después de aplicar AITC al baño, mientras que el aumento de la fluorescencia no se observó en las neuronas después de aplicar E3. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: La velocidad de captura de GCaMP6s determina la cantidad de luz capturada y la resolución resultante de la imagen. (A) Las imágenes de neuronas capturadas cada 10,13 s (0,10 fps) dan una resolución relativamente alta de las neuronas en el cerebro posterior y la médula espinal. Con un aumento en la velocidad de captura, se obtiene información temporal, pero se pierde la resolución espacial: (B) velocidad de captura = 0,79 fps, (C) velocidad de captura = 3,16 fps y (D) velocidad de captura = 7,67 fps. Pez cebra, 2 dpf; barra de escala de 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Hemos demostrado que la actividad neuronal se puede registrar en el cerebro de larvas de pez cebra utilizando GCaMP6s junto con microscopía confocal; las velocidades de captura más bajas requeridas debido a la cinética más lenta de los GCaMP pueden compensarse reduciendo el área cerebral observada6. Existen reporteros con una dinámica temporal más rápida (es decir, GCaMP6f), pero la resolución temporal superior generalmente se produce a costa de una señal de fluorescencia reducida6. El microscopio confocal está limitado a velocidades de registro relativamente más lentas22, por lo que las moléculas indicadoras deben seleccionarse teniendo en cuenta las limitaciones temporales de la microscopía confocal.
El protocolo está limitado por dos características clave: 1) el requisito de que el animal se coloque en agarosa durante el registro y 2) la velocidad de captura relativamente lenta de la microscopía confocal. Equilibrar el campo de visión y las velocidades de captura es clave para capturar la actividad neuronal con suficiente resolución para una aplicación determinada. Si la actividad inducida por el AITC se trunca o disminuye, esto podría indicar que las larvas de pez cebra pueden no ser capaces de detectar el estímulo o que el AITC puede ser insuficientemente potente. En este último caso, refrescar el caldo y mantenerlo congelado a -20 °C prolongará su eficacia. Si el pez no puede detectar el estímulo, es posible que el animal haya sido implantado demasiado profundamente en el agar. Un paso crítico es el posicionamiento de un pez durante el Paso 1 del protocolo, ya que esto es crucial para un acceso óptico óptimo durante la aplicación del medicamento.
Debido a las velocidades de exploración relativamente lentas de la microscopía confocal, las grabaciones de todo el cerebro no son técnicamente posibles. Significativamente, el protocolo descrito aquí se puede utilizar para medir pequeñas áreas del cerebro después de la identificación con técnicas complementarias. En particular, mediante el uso de técnicas que aprovechan las moléculas que marcan la actividad neuronal con reporteros dependientes de la actividad 9,11, el experimentador puede apuntar a áreas clave del cerebro de interés para la aplicación de los métodos descritos en este artículo. Las áreas de interés del cerebro se pueden referenciar al atlas del cerebro del pez cebra anotado, z brain atlas11.
Con pequeños ajustes, las técnicas y pautas discutidas aquí se pueden emplear para investigar una variedad de funciones cerebrales, incluido el procesamiento sensoriomotor, el aprendizaje y la memoria, y la sensación y la percepción.
Divulgaciones
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Agradecimientos
Este trabajo fue apoyado por una subvención a ACR de los Institutos Nacionales de Salud (SC2GM1304854) y una subvención a DLG de la Fundación Nacional de Ciencias (2050850).
Materiales
| Name | Company | Catalog Number | Comments |
| Low Melting Point Agarose | Invitrogen | 16520-100 | Diluted to 3% |
| Allyl Isothiocyanate (AITC) | Sigma Aldrich | 377430 | Chemical stimulant |
| E3 | N/A | N/A | Water-medium for zebrafish larvae |
| Glass Bottom Dishes | Thermo Fisher Scientific | 12-567-400 | Used to hold zebrafish during imaging experiments |
| Micropipette (10-100 uL) | Cole-Parmer | 21600-14 | Apparatus used for creating AITC dilutions |
| Microscope Slides | Fisherbrand | 12-550-A3 | Used to screen for phenotype |
| Mirror Finish Forceps | DUMONT | 11251-23 | Used to orient zebrafish in agarose |
| myTEMP Mini Digital Incubators | Benchmark | H2200-HC | Holding area for zebrafish; set to 28.5°C |
| Nitrile Gloves | MedPRIDE | MPR-50504 | Basic PPE |
| Petri Dishes | VWR | 89107-632 | Container for zebrafish |
| Posi-Click Tubes | DENVILLE | C-2171 | Used for AITC dilution |
| Samco Polyurethane Transfer Pipettes | Thermo Fisher Scientific | 225 | Apparatus used to select animal/administer diluted bolus of AITC |
| Stemi SV11 Apo Microscope | Zeiss | 1.25496E+11 | Used to stage zebrafish |
| Transgenic Larval Zebrafish (2 to 7 DPF) | N/A | N/A | Animal test subjects; Tg(elav3:GCaMP6s) strain |
| Zeiss Confocal Microscope (Model LSM9) | Zeiss | 3523004097 | Imaging of fish |
Referencias
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nat Method. 10 (5), 413-420 (2013).
- Meyer, M. P., Smith, S. J. Evidence from in vivo imaging that synaptogenesis guides the growth and branching of axonal arbors by two distinct mechanisms. J Neurosci. 26 (13), 3604-3614 (2006).
- Sagasti, A., Guido, M. R., Raible, D. W., Schier, A. F. Repulsive interactions shape the morphologies and functional arrangement of zebrafish peripheral sensory arbors. Curr Biol. 15 (9), 804-814 (2005).
- Son, J. H., et al. Transgenic FingRs for live mapping of synaptic dynamics in genetically-defined neurons. Sci Rep. 6, 18734(2016).
- Zada, D., Tovin, A., Lerer-Goldshtein, T., Vatine, G. D., Appelbaum, L. Altered behavioral performance and live imaging of circuit-specific neural deficiencies in a zebrafish model for psychomotor retardation. PLoS Genet. 10 (9), e1004615(2014).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Abdelfattah, A. S., et al. and photostable chemigenetic indicators for extended in vivo voltage imaging. Science. 365 (6454), 699-704 (2019).
- Abdelfattah, A. S., et al. Sensitivity optimization of a rhodopsin-based fluorescent voltage indicator. Neuron. 111 (10), 1547-1563.e9 (2023).
- Gao, Y. J., Ji, R. R. c-Fos and pERK, which is a better marker for neuronal activation and central sensitization after noxious stimulation and tissue injury. Open Pain J. 2, 11-17 (2009).
- Kawashima, T., Okuno, H., Bito, H. A new era for functional labeling of neurons: activity-dependent promoters have come of age. Front Neural Circuits. 8, 37(2014).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nat Methods. 12, 1039-1046 (2015).
- Reijmers, L. G., Perkins, B. L., Matsuo, N., Mayford, M. Localization of a stable neural correlate of associative memory. Science. 317 (5842), 1230-1233 (2007).
- Mohr, M. A., Argast, P., Pantazis, P. Labeling cellular structures in vivo using confined primed conversion of photoconvertible fluorescent proteins. Nature Protoc. 11 (12), 2419-2431 (2016).
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc Natl Acad Sci U S A. 106 (42), 17968-17973 (2009).
- Wyart, C., Del Bene, F. Let there be light: zebrafish neurobiology and the optogenetic revolution. Rev Neurosci. 22 (1), 121-130 (2011).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr Biol. 18 (15), 1133-1137 (2008).
- Portugues, R., Severi, K. E., Wyart, C., Ahrens, M. B. Optogenetics in a transparent animal: circuit function in the larval zebrafish. Curr Opinion Neurobiol. 23 (1), 119-126 (2013).
- Best, J. D., Alderton, W. K. Zebrafish: An in vivo. model for the study of neurological diseases. Neuropsychiat Dis Treatment. 4 (3), 567-576 (2008).
- Goldsmith, P. Zebrafish as a pharmacological tool: the how, why and when. Curr Opinion Pharmacol. 4 (5), 504-512 (2004).
- Roberts, A. C., et al. Habituation of the C-start response in larval zebrafish exhibits several distinct phases and sensitivity to NMDA receptor blockade. PloS One. 6 (12), e29132(2011).
- Wolman, M. A., Jain, R. A., Liss, L., Granato, M. Chemical modulation of memory formation in larval zebrafish. Proc Natl Acad Sci U S A. 108 (37), 15468-15473 (2011).
- Ji, N., Freeman, J., Smith, S. L. Technologies for imaging neural activity in large volumes. Nat Neurosci. 19 (9), 1154-1164 (2016).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Front Neural Circuits. 7, 65(2013).
- Distel, M., Köster, R. W. In vivo time-lapse imaging of zebrafish embryonic development. CSH Protoc. 2007, (2007).
- Liu, T. T., Hou, H., Du, J. L. A protocol for simultaneous Ca2+ and morphology imaging of brain endothelial tip cells in larval zebrafish. STAR Protoc. 2 (1), 100388(2021).
- Wong, H. C., Drerup, C. M. Using fluorescent indicators for in vivo quantification of spontaneous or evoked motor neuron presynaptic activity in transgenic zebrafish. STAR Protoc. 3 (4), 101766(2022).
- Graeden, E., Sive, H. Live imaging of the zebrafish embryonic brain by confocal microscopy. J Vis Exp. (26), e1217(2009).
- Hirsinger, E., Steventon, B. A versatile mounting method for long term imaging of zebrafish development. J Vis Exp. (119), e55210(2017).
- Renaud, O., Herbomel, P., Kissa, K. Studying cell behavior in whole zebrafish embryos by confocal live imaging: application to hematopoietic stem cells. Nat Protoc. 6 (12), 1897-1904 (2011).
- Roberts, A. C., et al. Induction of short-term sensitization by an aversive chemical stimulus in zebrafish larvae. eNeuro. 7 (6), (2020).
- Prober, D. A., et al. Zebrafish TRPA1 channels are required for chemosensation but not for thermosensation or mechanosensory hair cell function. J Neurosci. 28 (40), 10102-10110 (2008).
- Freeman, J., et al. Mapping brain activity at scale with cluster computing. Nat Methods. 11, 941-950 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados