Method Article
Dosaggio del numero più probabile modificato per quantificare la salmonella nei prodotti a base di pollo crudo e pronto da cuocere
In questo articolo
Riepilogo
Quantificare con precisione la Salmonella nel pollame a bassi livelli è una sfida industriale e normativa attuale. Questo protocollo descrive un test MPN che consente la quantificazione della Salmonella nei prodotti avicoli crudi e pronti per la cottura. Questo metodo è veloce, sensibile e si allinea con le linee guida FSIS, migliorando la sicurezza alimentare e sostenendo gli sforzi per la salute pubblica.
Abstract
La salmonella è una delle principali cause di malattie di origine alimentare negli Stati Uniti, in particolare nei prodotti a base di pollame. I metodi tradizionali per rilevare la Salmonella si concentrano sulla prevalenza piuttosto che sulla quantificazione, il che limita la loro utilità nella valutazione dei livelli di contaminazione e dei rischi. Questo studio introduce un nuovo test del numero più probabile (MPN) progettato per quantificare la Salmonella nei prodotti avicoli pronti da cuocere, come il cordon bleu di pollo. Il metodo prevede il lavaggio del campione di pollame, la concentrazione del risciacquo attraverso la centrifugazione e la diluizione in serie in un blocco di 48 pozzetti. Il test MPN è integrato con il metodo di amplificazione isotermica mediata da loop (LAMP) per fornire una quantificazione sensibile, accurata e rapida della contaminazione da Salmonella entro lo stesso periodo di tempo dei protocolli FSIS (Food Safety and Inspection Service) esistenti. I risultati mostrano una forte correlazione lineare tra le misurazioni MPN-LAMP e i livelli teorici di inoculazione (R² = 0,933). Tuttavia, la variabilità a concentrazioni più basse evidenzia le sfide per rilevare con precisione la Salmonella a questi livelli, con il limite inferiore di rilevabilità pratico stimato a circa 300 CFU/g. I potenziali perfezionamenti per migliorare l'applicabilità del protocollo includono l'aumento della quantità campionata per migliorare ulteriormente il limite di rilevamento, l'ottimizzazione delle formulazioni dei terreni di arricchimento e l'espansione del rilevamento molecolare per mirare a più sierotipi di Salmonella . Nel complesso, questo studio rappresenta uno strumento pratico per l'industria alimentare, che consente una quantificazione affidabile della contaminazione da Salmonella nei prodotti avicoli, contribuendo a migliorare la sicurezza alimentare e la salute pubblica.
Introduzione
Come principale causa di malattie di origine alimentare, ospedalizzazione e morte negli Stati Uniti, la Salmonella ha un impatto significativo sulla salute pubblica e sull'economia. L'onere economico stimato dell'agente patogeno nel solo 2013 è stato di 3,67 miliardi didollari1. Sebbene le recenti iniziative normative mirino a ridurre la salmonellosi del 25 % entro il 20302 , rimangono evidenti le lacune nelle attuali strategie di rilevamento e mitigazione, in particolare per quanto riguarda l'allineamento della sorveglianza degli impianti di lavorazione con i risultati in materia di salute pubblica3 .
I prodotti avicoli surgelati pronti per la cottura, che sono stati implicati in molteplici focolai di Salmonella , sono una preoccupazione significativa per la salute pubblica. In risposta, il Food Safety and Inspection Service (FSIS) ha classificato la Salmonella come adulterante in questi prodotti. Attualmente, la FSIS Microbiology Laboratory Guidebook (MLG) 4.15 si concentra esclusivamente sulla determinazione della prevalenza di Salmonella nei prodotti avicoli4. In base a questa linea guida, i campioni raccolti vengono arricchiti per 18-24 ore e quindi sottoposti a screening utilizzando il sistema di rilevamento molecolare (MDS), che identifica la presenza o l'assenza di Salmonella ma non fornisce informazioni sul livello di contaminazione. Sebbene questo approccio sia prezioso per rilevare la presenza di agenti patogeni, non riesce a fornire informazioni quantitative che potrebbero aiutare le aziende di trasformazione alimentare a valutare i rischi di contaminazione in modo più accurato e ad adottare azioni correttive mirate.
In questo studio, abbiamo sviluppato un metodo per aumentare il rilevamento dalla prevalenza alla quantificazione dei patogeni microbici. È stato progettato per una perfetta integrazione nei processi esistenti per rilevare la Salmonella nei prodotti avicoli con un'interruzione minima degli attuali protocolli FSIS. Invece di arricchire semplicemente il campione di massa, il metodo inizia lavando i prodotti avicoli utilizzando terreni coerenti con gli attuali metodi FSIS. Il risciacquo viene quindi distribuito nella prima colonna di un blocco di pozzi profondo 48 pezzi. Le diluizioni seriali vengono eseguite sulle restanti cinque colonne e il blocco viene incubato per 18-24 ore, allineandosi con il protocollo MLG 4.15. Dopo l'incubazione, i pozzetti vengono testati per la presenza di Salmonella e i risultati vengono utilizzati per calcolare il numero più probabile (MPN)5,6. Questo approccio consente di quantificare la contaminazione nello stesso lasso di tempo dell'attuale processo FSIS, rendendolo un'opzione pratica sia per l'industria che per l'uso normativo. La Figura 1 illustra un diagramma a blocchi che riassume il test MPN modificato. La figura include le fotografie scattate in fasi specifiche, il blocco di 48 pozzetti utilizzato per la diluizione e la crescita delle repliche e le tre tecniche utilizzate come parametri di riferimento per valutare il numero più probabile di Salmonella presente nel pollo macinato. Nella prima fase di questo studio, abbiamo utilizzato pollo macinato irradiato per ridurre al minimo l'impatto della microflora di fondo e l'incertezza delle misurazioni relative all'inoculo verificato prima di applicare il protocollo a campioni di pollo non irradiati.
Protocollo
NOTA: Tutto il lavoro associato a questo protocollo deve essere condotto all'interno di un laboratorio di biosicurezza di livello 2 (BSL-2). Se del caso, questo protocollo deve essere condotto all'interno di una cabina di sicurezza biologica (BSC) per mantenere le condizioni asettiche e ridurre al minimo il rischio di contaminazione del campione o l'esposizione dell'operatore a patogeni microbici. Quando si trasferiscono i campioni al di fuori del BSC, utilizzare contenitori sigillati per mantenere l'integrità del campione ed evitare fuoriuscite in caso di cadute accidentali. Preferibilmente, i componenti monouso dovrebbero essere utilizzati durante tutta la procedura per mitigare la possibilità di contaminazione incrociata. Nei casi in cui i prodotti monouso non siano fattibili, assicurarsi che tutte le attrezzature e i materiali siano sterili prima dell'uso. Una corretta gestione dei rifiuti è fondamentale; Tutti i componenti usa e getta usati devono essere smaltiti come rifiuti a rischio biologico. Autoclavare i materiali riutilizzabili prima del riutilizzo per garantire una corretta sterilizzazione e contenimento dei materiali potenzialmente pericolosi. L'osservanza di queste precauzioni non solo salvaguarda l'integrità del campione, ma riduce anche al minimo il rischio di esposizione dell'operatore a patogeni microbici.
1. Preparazione dei campioni di carne
- Acquisizione e lavorazione di campioni di carne
- Carne fresca
- Acquista il pollo macinato dal reparto carne fresca dei rivenditori locali. Trasferire tutti i campioni in un deposito a 4 °C ed elaborarli entro 24 ore dal ricevimento. Dividere asetticamente la carne in campioni da 25 g.
- Sigillare sottovuoto e irradiare il campione. Qui, il Texas A&M AgriLife National Center for Electron Beam Research ha irradiato la carne sottoposta a una dose di ~25 kGy.
NOTA: Sebbene l'irradiazione sia stata utilizzata come misura di controllo in questo studio per garantire l'eliminazione della microflora di fondo, non è un prerequisito per il protocollo nelle applicazioni pratiche, come dimostrato dai prodotti pronti da cuocere non irradiati utilizzati nella sezione successiva. In contesti di campo, metodi alternativi come i terreni selettivi o la specificità della diagnostica molecolare possono affrontare la potenziale interferenza di microrganismi non bersaglio.
- Prodotti a base di pollo pronti da cuocere
- Acquista prodotti a base di pollo pronti da cucinare dal reparto surgelati dei rivenditori locali. Dividere asetticamente in campioni da 25 g.
- Raccogli i campioni dal centro dei singoli pezzi per assicurarti che tutti gli ingredienti (ad esempio, panatura e formaggio) siano inclusi.
- Carne fresca
- Preparazione dei terreni
- Preparare l'acqua peptonica tamponata (BPW) sciogliendo 25 g di polvere di BPW in 1 L di nanopuro H2O.
- Preparare le piastre per infusione cerebrale e cardiaca (BHI). Per fare ciò, sciogliere 37 g di polvere di BHI in 1 L di nanopure H2O e aggiungere 15 g di agar nella soluzione di BHI. Sterilizzare tutti i terreni in autoclave a 121 °C per 15 minuti. Versare da 20 a 25 mL di terreno in piastre di Petri con coperchio trasparente (100 mm x 15 mm).
NOTA: È meglio versare le piastre all'interno di una cabina di sicurezza biologica per mantenere le condizioni asettiche.
2. Coltura cellulare
- Preparare la coltura iniziale strisciando il sierotipo di Salmonella enterica Typhimurium ATCC 14028 su una piastra di agar BHI e incubare a 37 °C per una notte.
- Preparare le colture durante la notte inoculando 25 ml di brodo BHI con una colonia di Salmonella appena cresciuta. Colture a crescita aerobica durante la notte a 37 °C con agitazione a 100 giri/min.
3. Inoculazione di campioni di pollame
- Diluizione e placcatura della coltura
- Preparare una serie di diluizioni di 10 volte della coltura notturna in BPW per ottenere concentrazioni finali di circa 1 x 108 a 1 x 101 CFU/mL. Si supponga che la concentrazione della coltura notturna di Salmonella sia di 1 x 109 CFU/mL.
- Trasferire 0,5 mL della coltura notturna in 4,5 mL di BPW, mescolare, quindi trasferire 0,5 mL della diluizione in 4,5 mL di BPW per ogni diluizione aggiuntiva.
- Distribuire 10 μL della diluizione 1 x 103 CFU/mL su una piastra di agar BHI in triplicato per l'enumerazione delle cellule per calcolare la concentrazione della coltura durante la notte.
- Inoculazione di campioni di carne
- Trasferire asetticamente 25 g di pollo macinato irradiato in una sacca sterile per lo stomaco in duplicato. La borsa misura 7,5 x 12 pollici ed è etichettata come 1,63 L. Il sacchetto contiene una partizione filtrante con un diametro del foro di 330 μm e ce ne sono 285 per cm quadrato.
- Inoculare ogni campione con 1 mL della diluizione della concentrazione target. Ad esempio, aggiungere 1 mL di coltura dalla diluizione di 1 x 10³ CFU/mL per ottenere un livello di contaminazione di circa 1.000 cellule/25 g di pollo. Distribuire delicatamente l'inoculo liquido sulla superficie dei campioni di pollo utilizzando un divaricatore di cellule sterili e lasciarlo riposare per 1 ora a 4 °C.
- Preparare i campioni di controllo negativi aggiungendo 1 mL di BPW sterile.
4. Elaborazione del campione
- Aggiungere 225 mL di BPW a ciascun campione. Il rapporto tra volume e media è stato selezionato per allinearsi a FSIS MLG 4.154.
- Omogeneizzare i campioni utilizzando lo Stomacher7 a velocità normale e una durata di 120 s.
- Centrifugazione e risospensione
- Rimuovere con cautela il liquido dal lato filtrato della sacca utilizzando una pipetta da 50 ml. Dividere il liquido in due flaconi da centrifuga sterili.
- Bilanciare le bottiglie con BPW sterile per garantire pesi uguali. Centrifugare i campioni a 10.000 x g per 10 minuti. Scartare il surnatante. Risospendere il pellet cellulare in 3 mL di brodo BPW con una spatola sterile.
- Aggiungere altri 27 ml di brodo BPW e mescolare accuratamente mescolando con una spatola. Combinare il contenuto di entrambi i flaconi da centrifuga in un flacone per ogni campione.
5. Configurazione del blocco MPN
NOTA: La Tabella 1 illustra uno schema delle diluizioni in un blocco di 48 pozzetti.
- Aggiungere 3 mL del campione risospeso a ciascun pozzetto nella colonna 1 del blocco da 48 pozzetti (8 repliche).
- Preparare una serie di diluizioni di 10 volte sulle colonne 1-6 all'interno del blocco utilizzando una pipetta a otto canali.
- Aggiungere 0,3 mL di campione in 2,7 mL di pipetta BPW per miscelare. Ripetere per ogni diluizione. Incubare i blocchi per una notte (~18 h) a 37 °C con agitazione a ~100 giri/min.
6. Impiattamento ed enumerazione
- Enumerazione della piastra di caduta modificata
- Piastra 7 μl di campione coltivato durante la notte di ciascuna diluizione in una griglia 4 x 6 su una piastra di agar utilizzando una pipetta multicanale (Figura 2). L'utilizzo di una griglia 4 x 6 su due piastre ospita meglio 8 campioni, rispetto alla tipica griglia di goccioline 6 x 68.
- Lasciare asciugare le piastre all'aria per 10 minuti prima dell'incubazione. Incubare le piastre di agar per una notte (~18-24 h) a 37 °C. Dopo l'incubazione, contare il numero di colonie su ogni piastra.
7. Rilevamento qPCR di Salmonella
- Estrazione del DNA utilizzando un kit commerciale
- Mescolate le colture nel blocco da 48 pozzetti pipettando più volte su e giù. Pipettare 200 μl di ciascuna coltura in una piastra PCR a 96 pozzetti.
- Sigillare e centrifugare la piastra a 6.600 x g per 10 min. Rimuovere il surnatante e aggiungere 20 μl del reagente del kit al pellet.
- Risospendere il pellet pipettandolo su e giù. Sigillare e riscaldare la piastra a 99 °C per 10 minuti, quindi raffreddarla a 20 °C.
- Centrifugare nuovamente a 6.600 x g per 10 min. Utilizzare 2 μl di surnatante per l'analisi qPCR.
- Impostazione della piastra
- Preparare la miscela di reazione qPCR secondo il protocollo9 stabilito come segue: 10 μl di 2x Master Mix; 0,4 μL di ciascun primer e sonda (soluzione di lavoro da 10 μM): invA in avanti: 5'-GTTGAGGATGTTATTCGCAAAGG-3', invA in senso inverso: 5'-GGAGGCTTCCGGGTCAAG-3', sonda invA: 5'-CCGTCAGACCTCTGGCAGTACCTTCCTC-3' marcata con il colorante fluoreni Cal Fluor Orange 560; 0,2 μl di modello di controllo dell'amplificazione interna (IAC) (6 x 104 copie/μl)9; 0,4 μL di ciascun primer e sonda IAC (10 μM): IAC in avanti: 5'-GGCGCGCCTAACACATCT-3', IAC in retromarcia: 5'-TGGAAGCAATGCCAAATGTGTA-3', sonda IAC: 5'-TTACAACGGGAGAAGACAATGCCACCA-3' marcata con colorante TAFRA. Regolare il volume con ddH2O a 20 μL totali.
- Eseguire la PCR in tempo reale con le seguenti condizioni cicliche9: 95 °C per 10 minuti (denaturazione iniziale del DNA e attivazione della polimerasi hot-start), 40 cicli di 95 °C per 15 s e 60 °C per 1 minuto, utilizzare le impostazioni Ct predefinite per esportare i risultati per l'analisi.
8. Rilevamento mediante saggio 3M MDS
- Seguire il protocollo del kit per il test di rilevamento molecolare Salmonella . Mescolate le colture nel blocco da 48 pozzetti pipettando più volte su e giù. Pipettare 20 μl di ciascun campione nella provetta di lisi fornita in kit.
- Riscaldare i campioni a 100 °C per 15 minuti. La soluzione passerà dal rosa al giallo. Incubare i campioni per 10 minuti a temperatura ambiente. La soluzione cambierà da gialla a rosa.
- Trasferire 20 μl di lisato in una provetta reattiva e caricare le provette reattive nel supporto.
- Aggiungi il supporto allo strumento MDS e configura il software per comunicare le informazioni sul kit e sul campione. Lo strumento richiede che ogni pozzetto sia etichettato con il numero di lotto per il saggio e un nome del campione. Eseguire il software MDS ed esportare il report.
9. Analisi dei dati
- Classificazione dei risultati positivi e negativi.
- Per la placcatura a goccia 4 x 6, valutare come positivi i punti sulle piastre di agar con almeno 1 colonia e i punti sulle piastre di agar senza crescita come negativi.
- Per la qPCR, valutare i pozzetti che hanno un Ct inferiore o uguale a 30 come positivi e i pozzetti che hanno un Ct maggiore di 30 come negativi.
- Per MDS, utilizzare i risultati del sistema MDS, riportati come positivi o negativi.
- Calcolo MPN
- Analizza positivi e negativi annotati utilizzando il metodo della risoluzione di massima probabilità semplice (SMPR) descritto in precedenza6 o calcolatori MPN verificati alternativi. 10
Risultati
Carne irradiata
Nell'analisi di regressione, una pendenza di 1 indica che per ogni aumento unitario della variabile indipendente (asse x), la variabile dipendente (asse y) aumenta esattamente di 1 unità. Ciò suggerisce una relazione proporzionale tra le due variabili, il che significa che la variazione della variabile dipendente rispecchia la variazione della variabile indipendente. Un'intercetta di 0 significa che quando la variabile indipendente è 0, anche la variabile dipendente è 0. Ciò suggerisce che non esiste un offset fisso o una distorsione nella relazione tra le due variabili. Insieme, una pendenza di 1 e un'intercetta di 0 suggeriscono un accordo tra le variabili11,12. Questo scenario ideale non indica alcun errore sistematico o distorsione e sarebbe il risultato atteso in un sistema ben calibrato in cui le misurazioni si allineano perfettamente con le previsioni.
La Figura 3A presenta il saggio MPN-LAMP (asse y) tracciato rispetto all'inoculo teorico verificato dall'analisi della piastra di diffusione discussa nella nota al punto 3.1.1. (asse x). L'equazione della retta di regressione, y = -72,36 + 0,161x, con un valore R² di 0,978, indica una forte correlazione lineare tra i risultati di MPN-LAMP e i livelli batterici inoculati. L'elevato valore di R² suggerisce che il test MPN-LAMP è altamente affidabile nella previsione quantitativa della contaminazione batterica. Tuttavia, la pendenza di 0,161 si discosta significativamente dalla pendenza ideale di 1, indicando che il metodo MPN-LAMP sottostima l'inoculo, prevedendo solo il 16,1% della concentrazione batterica nell'inoculo teorico. Inoltre, l'intercetta negativa di -72,36 suggerisce una distorsione negativa a concentrazioni più basse, che si discosta ulteriormente dall'ideale.
La Figura 3B illustra il metodo MPN-qPCR (asse y) tracciato rispetto all'inoculo teorico (asse x). La linea di regressione, y = -46,84 + 0,0395x, con un valore R² di 0,978, dimostra una forte relazione lineare tra il test MPN-qPCR e l'inoculo teorico. Sebbene l'alto R² indichi una robusta correlazione, la pendenza di 0,0395 è ancora più piccola di quella dei risultati MPN-LAMP, suggerendo un maggior grado di sottostima della contaminazione batterica da MPN-qPCR. L'intercetta negativa di -46,84 indica ulteriormente una potenziale distorsione al ribasso a concentrazioni più basse.
La Figura 3C presenta i risultati della placcatura MPN (asse y) tracciati rispetto all'inoculo teorico (asse x), con un'equazione di regressione di y = -330,4 + 0,3138x e un valore R² di 0,944. Sebbene questo valore di R² indichi una forte relazione lineare, è leggermente inferiore a quelli dei risultati MPN-LAMP e MPN-qPCR, suggerendo una maggiore variabilità nella quantificazione delle colonie. La pendenza di 0,3138, sebbene superiore alle pendenze osservate negli altri metodi, indica ancora una sottostima della concentrazione batterica rispetto all'inoculo teorico. Inoltre, l'intercetta negativa di -330,4 indica una notevole distorsione, soprattutto a concentrazioni più basse, che può indicare potenziali imprecisioni nelle tecniche di placcatura o nella manipolazione dei campioni. Questa leggera riduzione di R² e la variabilità osservata potrebbero indicare che il processo di placcatura tradizionale è più variabile rispetto ai metodi molecolari.
Metodi di confronto
Tutti e tre i metodi (MPN-LAMP, MPN-plating e MPN-qPCR) hanno mostrato una tendenza a sottostimare i livelli batterici rispetto all'inoculazione teorica. Questa sottostima può essere attribuita a problemi di recupero cellulare, in cui la Salmonella potrebbe non staccarsi completamente dalla superficie del pollo o dal particolato durante la lavorazione, riducendo il suo trasferimento nel risciacquo o nel test. Poiché la Salmonella è stata inoculata direttamente sul pollo, una parte delle cellule batteriche può legarsi fortemente alla superficie, limitando il recupero nella fase di risciacquo. Inoltre, alcune celle possono rimanere attaccate al particolato nel pellet dopo la centrifugazione, riducendo ulteriormente il recupero. Approcci come trattamenti enzimatici, processi meccanici o protocolli di stomaco ottimizzati potrebbero migliorare il distacco batterico. Queste strategie sono discusse ulteriormente nella sezione di discussione, sottolineando il loro potenziale per migliorare i tassi di recupero
La Figura 4A presenta il recupero sull'asse y rispetto a tre metodi MPN sull'asse x. La percentuale di recupero, calcolata come rapporto tra il valore MPN e il livello teorico di inoculazione, ha prodotto i seguenti risultati: il metodo MPN-LAMP ha avuto un recupero medio del 15,19% con una deviazione standard del 10,04%; il metodo MPN-plating ha avuto un recupero medio del 13,13% con una deviazione standard dell'11,45%; e il metodo MPN-qPCR ha avuto un recupero medio del 6,67% con una deviazione standard del 3,19%. Un test Tukey-Kramer HSD non ha identificato una differenza statisticamente significativa nel recupero tra i metodi (P > 0,1969), suggerendo che tutti e tre i metodi hanno funzionato in modo simile in termini di recupero durante la lavorazione del pollo inoculato con Salmonella.
Nonostante il trattamento identico tra i metodi per separare la Salmonella dalla matrice di pollo, sono state osservate variazioni nel rilevamento tra MPN-LAMP, MPN-qPCR e MPN-plating. Ciò suggerisce che fattori che vanno oltre la separazione fisica dei batteri dal campione possono influire sul rilevamento. La presenza di inibitori nella matrice del campione potrebbe interferire con i metodi molecolari - qPCR e LAMP - influenzandone la sensibilità e le prestazioni complessive. Inoltre, la vitalità batterica potrebbe aver influito sulla capacità della Salmonella di crescere su piastre di agar solide (MPN plating) rispetto al brodo liquido (qPCR, LAMP), portando a variazioni nel recupero tra i metodi.
Per esplorare ulteriormente la sensibilità dei metodi, abbiamo prima esaminato il numero di risposte positive a ciascuna diluizione per MPN-LAMP, MPN-qPCR e MPN-plating in sette studi indipendenti (Tabella 2). A diluizioni più basse (1 x 10-5 e 1 x 10-4), MPN-LAMP ha dimostrato un numero maggiore di risposte positive rispetto sia alla MPN-plating che alla MPN-qPCR, suggerendo che il metodo LAMP è più sensibile a basse concentrazioni di Salmonella. Quando i campioni erano più concentrati (diluizioni inferiori a 1 x 10-3), tutti e tre i metodi hanno mostrato un aumento delle rilevazioni positive, con una convergenza che si è verificata intorno alla diluizione 1 x 10-2 , dove tutti i metodi hanno rilevato un numero simile di campioni positivi.
La Figura 4B presenta la percentuale di misurazioni positive a ciascuna diluizione tra i tre metodi, fornendo un confronto diretto della loro sensibilità nel rilevare la Salmonella. La percentuale di risposte positive offre una visualizzazione più chiara delle prestazioni del metodo in tutta la serie di diluizioni e le barre di errore che rappresentano ± intervalli di confidenza di Clopper-Pearson (CI) del 95% forniscono informazioni sulla variabilità a ciascun livello. A diluizioni più basse, gli intervalli di confidenza più ampi indicano una maggiore variabilità nelle prestazioni di rilevamento di ciascun metodo. Coerentemente con il numero grezzo di rilevamenti positivi, MPN-LAMP rileva una percentuale più elevata di campioni positivi alle concentrazioni più diluite (1 x 10-5), mentre MPN-qPCR e MPN-plating rilevano meno positivi. All'aumentare della concentrazione batterica, i metodi convergono, con tutti e tre gli approcci che mostrano un rilevamento vicino al 100% a concentrazioni più elevate (ad esempio, 1 x 10-2 e oltre). Il restringimento degli intervalli di confidenza a queste concentrazioni più elevate indica una maggiore coerenza tra tutti i metodi, sottolineando le loro prestazioni comparabili quando i livelli batterici sono più alti.
I risultati della Figura 3 suggeriscono che la costante sottostima (quantitativamente) di tutti e tre i metodi potrebbe essere guidata dalla separazione incompleta di Salmonella dalla matrice del campione. Tuttavia, gli inibitori molecolari o i fattori di vitalità batterica potrebbero guidare la variazione osservata nel rilevamento a diverse diluizioni (qualitativamente) e attraverso diversi metodi (qualitativamente e quantitativamente). Gli intervalli di confidenza più stretti a concentrazioni più elevate nella Figura 4B indicano una minore variabilità e prestazioni qualitative più coerenti tra i metodi, mentre gli intervalli più ampi a concentrazioni più basse indicano una maggiore incertezza nella rilevazione qualitativa.
Rilevamento nel pollo pronto da cuocere
Abbiamo scelto il metodo LAMP per il rilevamento della Salmonella nel pollo pronto da cuocere in quanto mostrava un numero maggiore di campioni positivi (14,3%) rispetto ad altri metodi testati alla più bassa diluizione di Salmonella . Alla diluizione più bassa di Salmonella (1 x 10-5), nessuno dei campioni è stato rilevato come positivo con il metodo qPCR interno. D'altra parte, alcuni positivi sono stati rilevati dalla placcatura (7,1%, la metà del numero rilevato da LAMP). Tuttavia, i risultati di questo metodo basato sulla coltura possono essere influenzati quando si passa a prodotti più complessi come il pollo pronto da cuocere, dove la presenza di microflora di fondo potrebbe distorcere i risultati perché i campioni non vengono irradiati. Per questi motivi, abbiamo deciso di studiare il metodo MPN-LAMP per la quantificazione della Salmonella da pollo pronto da cuocere.
La Figura 5 illustra i risultati del test MPN-LAMP utilizzato per quantificare la Salmonella in campioni di cordon bleu di pollo inoculati, congelati e pronti per la cottura. Il livello teorico di inoculazione (CFU/g) è stato tracciato sull'asse x, mentre le corrispondenti misure MPN-LAMP sono state tracciate sull'asse y. L'analisi di regressione ha prodotto l'equazione y = -15,04 + 0,3869x, con un coefficiente di determinazione (R²) di 0,933, indicando una forte correlazione lineare tra l'inoculo teorico e i risultati MPN-LAMP. Questo alto valore di R² suggerisce che il metodo MPN-LAMP quantifica in modo affidabile la Salmonella in questi campioni, sebbene la pendenza di 0,3869 indichi che il metodo sottostima il livello effettivo di inoculo. L'area ombreggiata intorno alla linea di regressione rappresenta l'intervallo di confidenza del 95%.
Per illustrare meglio le prestazioni di MPN-LAMP a livelli di inoculazione più bassi, il grafico nel riquadro nella Figura 5 ingrandisce i punti dati inferiori a 300 CFU/g. Sulla base dell'equazione di regressione, il limite inferiore di rilevabilità può essere stimato a circa 40 CFU/g (38,9 CFU/g). Tuttavia, l'intervallo di confidenza del 95% suggerisce che il limite inferiore pratico di rilevamento è più vicino a 325 CFU/g, poiché qualsiasi valore MPN-LAMP negativo dovrebbe essere interpretato come zero. La linea rossa nel riquadro rappresenta lo zero e, sebbene nessun punto dati sia sceso al di sotto di questa linea, la possibilità che i valori scendano sotto lo zero, in particolare a livelli di inoculo più bassi, introduce la possibilità di risultati falsi negativi. Questa tendenza evidenzia le difficoltà di quantificare con precisione la Salmonella a basse concentrazioni e sarebbero necessari ulteriori dati per stabilire definitivamente il limite inferiore di rilevazione.

Figura 1: Diagramma a blocchi del test MPN modificato per il rilevamento della Salmonella. Una panoramica schematica del test del numero più probabile (MPN) modificato. Delinea le fasi chiave del processo, a partire dall'inoculazione di campioni di pollo macinato irradiati, seguita dall'omogeneizzazione del campione, dalla centrifugazione, dalla distribuzione delle aliquote e dall'incubazione in condizioni specificate. La figura include le fotografie del blocco di 48 pozzetti utilizzato per la diluizione e la crescita replicata, insieme a tre tecniche utilizzate per produrre risultati positivi e negativi per il calcolo della Simple Maximum Probability Resolution (SMPR). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Schema del saggio su piastra a goccia. La configurazione del saggio su piastra a goccia per una diluizione specifica (ad es. non diluita, 10-1, 10-2, ecc.) dal blocco a 48 pozzetti. Ogni campione viene placcato in un formato a griglia 4 x 6 su una piastra di agar utilizzando una pipetta multicanale. Il layout è stato modificato dalla tradizionale griglia 6 x 6 a due griglie 4 x 6 per garantire che potessero essere ospitate sei repliche da otto diluizioni. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Confronto tra inoculo e metodi MPN. (A) Un grafico della concentrazione di MPN-LAMP (asse y) rispetto all'inoculo teorico verificato mediante placcatura (asse x). Una retta di regressione approssimata (y = -72,36 + 0,161x) con un coefficiente di determinazione (R² = 0,978) dimostra una relazione lineare forte. (B) Un grafico della concentrazione di MPN-qPCR (asse y) rispetto all'inoculo teorico (asse x), con una linea di regressione approssimata (y = -46,84 + 0,0395x) e R² = 0,978, che mostra una correlazione altrettanto forte. (C) Un grafico della concentrazione di MPN-plating (asse y) rispetto all'inoculo teorico (asse x), con una linea di regressione (y = -330,4 + 0,3138x) e R² = 0,944, che indica una correlazione forte ma leggermente inferiore rispetto a LAMP e qPCR. L'equazione di regressione e il coefficiente di determinazione per ogni figura sono presentati come un inserto nell'angolo in alto a sinistra dei grafici. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Confronto tra i metodi MPN. (A) La figura presenta la percentuale di recupero, calcolata come rapporto tra il valore MPN e il livello teorico di inoculazione per i metodi MPN-LAMP, MPN-plating e MPN-qPCR. Il metodo MPN-LAMP ha avuto un recupero medio del 15,19% (±10,04%), il metodo MPN-plating ha avuto un recupero medio del 13,13% (± 11,45%) e il metodo MPN-qPCR ha avuto un recupero medio del 6,67% (± 3,19%). I box plot evidenziano la distribuzione dei dati di recupero, con i valori anomali indicati. Le barre di errore rappresentano l'intervallo di dati entro 1,5 volte l'intervallo interquartile (IQR). (B) Percentuale di rilevamenti positivi di Salmonella a ciascuna diluizione in tre metodi: MPN-LAMP (blu), MPN-plating (rosso) e MPN-qPCR (verde). L'asse x rappresenta i livelli di diluizione (da 10-6 a 10) e l'asse y mostra la percentuale di misurazioni positive. Le barre di errore indicano gli intervalli di confidenza di Clopper-Pearson del 95% ±, che riflettono la variabilità nel rilevamento per ciascun metodo. MPN-LAMP mostra percentuali di rilevamenti più elevate rispetto ad altri metodi a diluizioni inferiori, mentre tutti i metodi convergono vicino al 100% di rilevamenti a concentrazioni più elevate. Clicca qui per visualizzare una versione più grande di questa figura.
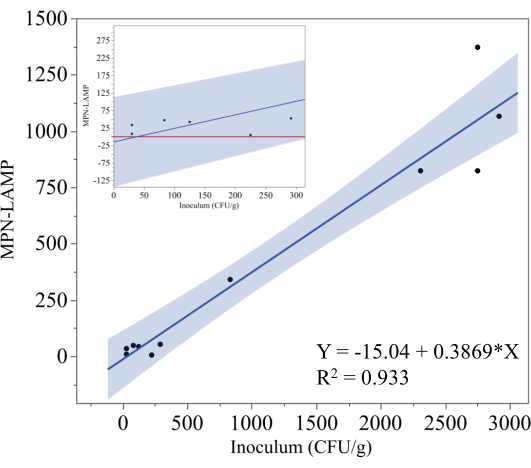
Figura 5: Metodo MPN per pollo pronto da cuocere. Analisi di regressione dei risultati di MPN-LAMP rispetto al livello teorico di inoculazione di Salmonella in campioni di cordon bleu di pollo congelati e pronti per la cottura. La linea di regressione è rappresentata da y = -15,04 + 0,3869x con un R² di 0,933, che indica una forte relazione lineare. L'area ombreggiata rappresenta l'intervallo di confidenza del 95%. L'inserto si concentra sulle misurazioni MPN-LAMP a livelli di inoculazione più bassi. Clicca qui per visualizzare una versione più grande di questa figura.
| 1 | 2 | 3 | 4 | 5 | 6 | |
| Un | n=1 | n=1 | n=1 | n=1 | n=1 | n=1 |
| B | 2 | 2 | 2 | 2 | 2 | 2 |
| C | 3 | 3 | 3 | 3 | 3 | 3 |
| D | 4 | 4 | 4 | 4 | 4 | 4 |
| E | 5 | 5 | 5 | 5 | 5 | 5 |
| F | 6 | 6 | 6 | 6 | 6 | 6 |
| G | 7 | 7 | 7 | 7 | 7 | 7 |
| H | 8 | 8 | 8 | 8 | 8 | 8 |
| 100 | 10-1 | 10-2 | 10-3 | 10-4 | 10-5 |
Tabella 1: Schema delle repliche e delle diluizioni in un blocco a 48 pozzetti. Questa figura illustra la disposizione delle repliche e delle diluizioni seriali di 10 volte all'interno del blocco a 48 pozzetti. Ogni colonna rappresenta un fattore di diluizione specifico, che va da 100 a 10-5 con repliche tecniche per ogni diluizione distribuite sulle otto righe.
| Diluizione | ||||||
| 10-5 | 10-4 | 10-3 | 10-2 | 10-1 | 100 | |
| LAMPADA MPN | 8 | 16 | 31 | 3 | 56 | 56 |
| MPN-placcatura | 4 | 11 | 32 | 55 | 56 | 56 |
| MPN-qPCR | 0 | 7 | 26 | 55 | 56 | 56 |
Tabella 2: Numero di eventi positivi. La tabella mostra il numero di reazioni positive (oltre 56 repliche totali). I risultati sono organizzati in una tabella in cui le colonne rappresentano la diluizione e le righe sono rappresentative del metodo.
Discussione
Significato del protocollo
La salmonella rimane una delle principali preoccupazioni per la sicurezza alimentare, in particolare per i prodotti avicoli, che sono spesso implicati in focolai di malattie di origine alimentare13,14. Essendo una delle principali cause di malattie batteriche di origine alimentare negli Stati Uniti, metodi affidabili per rilevare la Salmonella nei prodotti avicoli freschi e pronti da cuocere sono fondamentali per garantire la sicurezza alimentare15. La capacità di quantificare la Salmonella a bassi livelli è fondamentale per identificare la contaminazione che altrimenti potrebbe non essere rilevata. La sensibilità al metodo è particolarmente importante per la conformità normativa e la salute pubblica, poiché anche basse dosi di Salmonella possono provocare malattie, soprattutto nelle popolazioni vulnerabili.
Il test MPN-LAMP utilizza il sistema MDS, attualmente utilizzato da FSIS in MLG 4.15, per determinare la prevalenza della contaminazione4. Il test qui presentato fornisce un'estensione dell'attuale metodo FSIS per quantificare la Salmonella vitale, anche a bassi livelli, fornendo dati più precisi sul livello di contaminazione. I risultati di questo studio hanno dimostrato una forte correlazione tra il metodo MPN-LAMP e i valori teorici di inoculazione, con un R² di 0,933 (Figura 5). Ciò suggerisce che il metodo è affidabile per quantificare la Salmonella negli alimenti, anche se una certa sottostima si è verificata a livelli di inoculazione più bassi.
Passaggi critici
Il protocollo per la quantificazione della Salmonella richiede precisione nella preparazione del campione, nell'inoculazione e nell'analisi molecolare. Un'adeguata tecnica asettica, diluizioni accurate e aderenza alle condizioni di incubazione sono fondamentali per un rilevamento e un conteggio affidabili della Salmonella. Garantire condizioni sterili durante il trasferimento di campioni e terreni di coltura è essenziale per prevenire la contaminazione.
Altrettanto importante è l'omogeneizzazione dei campioni durante lo stomaching, seguita da un'attenta risospensione del pellet dopo la centrifugazione per separare i batteri dalla matrice alimentare. Una corretta manipolazione durante queste fasi garantisce che le cellule vitali vengano recuperate per ulteriori analisi. Un pipettaggio accurato durante il processo di diluizione seriale per la configurazione MPN, insieme a condizioni di incubazione costanti, è fondamentale per ottenere risultati riproducibili.
Infine, i metodi di rilevamento molecolare, come la qPCR, richiedono precisione e riproducibilità nell'estrazione del DNA e nel ciclo della qPCR per evitare falsi negativi. Sebbene l'estrazione del DNA sia intrinsecamente variabile, i reagenti e le procedure standardizzate garantiscono la ripetibilità riducendo al minimo l'interferenza dell'inibitore e migliorando il recupero del DNA. Questi passaggi sono fondamentali per confermare la presenza di Salmonella e per individuare specifici marcatori genetici che possono aiutare a superare i limiti dei metodi tradizionali di placcatura e arricchimento.
Potenziali insidie e limitazioni
Vitalità batterica
Una delle sfide principali dell'implementazione di un test basato sulla crescita come la placcatura MPN è la vitalità batterica, e questo è particolarmente rilevante per campioni di pollo complessi pronti da cuocere. I prodotti a base di pollo congelati, impanati e ripieni, come il cordon bleu di pollo, sono particolarmente soggetti a fluttuazioni di temperatura e periodi di conservazione prolungati, entrambi fattori che possono stressare le cellule di Salmonella e rendere l'agente patogeno più difficile da rilevare nei saggi basati sulla crescita. Inoltre, la presenza di ingredienti diversi dal pollo (ad esempio, formaggio, panatura, prosciutto, spezie, burro) introduce ulteriore complessità nella matrice alimentare e può inibire la crescita di Salmonella durante le fasi di arricchimento, complicando una quantificazione accurata. Inoltre, la Salmonella deve competere con i microbi di fondo nei metodi basati sulla coltura16, il che è particolarmente rilevante per i campioni pronti da cuocere con più ingredienti che aggiungono diversità al microbiota rispetto al pollo crudo. Infine, la manipolazione e la lavorazione di fattori di stress, come gli antimicrobici, possono influenzare il recupero della Salmonella 17.
La vitalità batterica potrebbe aver influito sulle prestazioni dei metodi testati in questo studio. Tutti i metodi hanno sottostimato le concentrazioni di Salmonella a bassi livelli e hanno avuto una maggiore variabilità di rilevamento a concentrazioni più basse di patogeni (Figura 4A, B). Tuttavia, il metodo MPN-LAMP su base molecolare ha superato la MPN-plating e la MPN-qPCR nella rilevazione qualitativa a livelli di inoculazione inferiori. I risultati sottolineano la necessità di raffinazione a basse concentrazioni di agenti patogeni.
Eterogeneità del campione
Un'altra importante limitazione riguarda l'eterogeneità dei campioni alimentari, che può introdurre variabilità nei risultati del rilevamento della Salmonella . Il protocollo è stato progettato per disperdere 30 mL di campione omogeneizzato, consentendo la distribuzione di 8 repliche come aliquote da 3 mL (24 mL in totale, 6 mL in eccesso) in tutto il blocco. Sebbene questo approccio conservativo (che si traduce in un campione aggiuntivo) tenga conto delle potenziali perdite, introduce il sottocampionamento poiché l'intero volume non viene testato.
L'eterogeneità del campione alimentare può influire sull'accuratezza della misurazione durante il trasferimento dei campioni dalla miscela ridispersa nel blocco e l'esecuzione delle diluizioni18,19. Secondo la teoria del campionamento (TOS), gli errori di campionamento fondamentali possono essere ridotti aumentando la massa del campione o riducendo l'eterogeneità composizionale attraverso la riduzione delle dimensioni delle particelle20. Tuttavia, mentre alcuni studi indicano che la rottura della matrice mediante stomaco non influisce in modo significativo sul recupero batterico dalla contaminazione superficiale, è stato dimostrato che la riduzione della dimensione delle particelle della matrice alimentare riduce il recupero batterico in alcuni casi 21,22,23. In questo studio, la dimensione delle particelle è stata ridotta mediante stomaco prima dell'arricchimento; Sebbene ciò possa ridurre l'errore di campionamento riducendo l'eterogeneità composizionale nei campioni, lo stomaco e la centrifugazione possono influire sul recupero batterico.
Infine, la composizione e la morfologia eterogenee dell'alimento, nonché il fatto che la contaminazione sia interna o superficiale, possono portare a variabilità nel recupero dei patogeni. Lavori precedenti18,22 hanno dimostrato che un forte legame batterico alle superfici dei tessuti può ridurre i tassi di recupero. Studi in cui enzimi come la tripsina, la collagenasi e l'endopeptidasi sono stati utilizzati per rilasciare batteri dalle matrici di carne e pollame24,25 e dalla pectinasi e dalla cellulasi alle verdure a foglia verde19 dimostrano il potenziale per migliorare il recupero della Salmonella dalle matrici alimentari.
Suggerimenti per la risoluzione dei problemi
Per evitare problemi, assicurarsi che tutti i terreni e i reagenti siano preparati al momento e sterili. I materiali scaduti o sterilizzati in modo improprio possono portare alla contaminazione o influenzare la crescita della Salmonella. Se si osserva una contaminazione, verificare che la tecnica asettica sia stata seguita in modo coerente durante tutto il protocollo, in particolare durante l'inoculazione, la diluizione, l'aliquotazione e la placcatura del campione.
Quando la conta delle colonie è inferiore al previsto, verificare l'accuratezza delle diluizioni seriali e delle concentrazioni di inoculo può aiutare a identificare gli errori. Le condizioni di incubazione, tra cui temperatura, tempo e agitazione, devono essere attentamente monitorate per supportare una crescita ottimale della Salmonella . In caso di crescita bassa o assente, uno stomaco inadeguato o la fuoriuscita dalle sacche potrebbero contribuire a una distribuzione non uniforme dei batteri.
Se la rilevazione molecolare produce risultati incoerenti, il controllo delle procedure di estrazione del DNA e la verifica della qualità dei primer e delle sonde sono passaggi fondamentali. Inoltre, la variabilità tra le matrici alimentari può presentare diversi livelli di inibizione, che possono essere monitorati utilizzando il controllo interno dell'amplificazione (IAC). La corretta conservazione e manipolazione dei reagenti di estrazione è fondamentale per prevenire falsi negativi dovuti a degradazione o contaminazione. Possono anche essere giustificati metodi di estrazione del DNA più efficienti.
L'incorporazione di trattamenti come il sistema GentleMACS26, che impiega azioni meccaniche, termiche ed enzimatiche, può ottimizzare ulteriormente il recupero dei batteri dal cibo. Questo sistema è in grado di ridurre l'eterogeneità della matrice alimentare, facilitando il trasferimento di campioni uniformi nel blocco e l'esecuzione di accurate diluizioni seriali.
Potenziale di perfezionamento ed espansione
Ci sono diverse opportunità per perfezionare ed espandere questo protocollo. L'esplorazione di formulazioni alternative di terreni di arricchimento potrebbe migliorare il recupero di cellule di Salmonella stressate o subletali o danneggiate in modo subletale, in particolare da matrici alimentari complesse come il cordon bleu di pollo. Sebbene l'agar selettivo sia comunemente usato per colpire la Salmonella, le cellule danneggiate o stressate a causa delle condizioni di lavorazione e conservazione possono portare a falsi negativi su tali terreni. Per superare questa sfida, l'ottimizzazione dell'equilibrio tra pressione selettiva e recupero microbico potrebbe migliorare il rilevamento di sierotipi di Salmonella che sono meno competitivi in condizioni standard di arricchimento. Questo perfezionamento potrebbe migliorare la sensibilità complessiva dei saggi, in particolare nei prodotti alimentari difficili in cui lo stress microbico è comune. Accorciare i tempi di incubazione potrebbe accelerare il tempo per ottenere i risultati, anche se ciò potrebbe ridurre la sensibilità e limitare il rilevamento. Il lavoro futuro dovrebbe esplorare i progressi nei terreni di arricchimento per supportare la rapida replicazione batterica, garantendo una quantificazione accurata e soddisfacendo al contempo i requisiti delle parti interessate.
Inoltre, lo studio di diverse temperature e durate di incubazione per il test MPN potrebbe ottimizzare il recupero della Salmonella da vari prodotti avicoli. Ciò potrebbe espandere l'applicabilità del protocollo ad altri agenti patogeni di origine alimentare che richiedono condizioni di crescita distinte, ampliando così la sua utilità nei test di sicurezza alimentare. L'automazione potrebbe migliorare la scalabilità di questo protocollo integrando manipolatori robotici di liquidi per le fasi di aliquotazione e diluizione. Le centrifughe a flusso continuo o i sistemi di centrifughe ad alta capacità possono anche risolvere i limiti dell'elaborazione in batch, consentendo applicazioni ad alta produttività in ambienti industriali.
L'espansione dell'aspetto del rilevamento molecolare per includere la PCR multiplex potrebbe consentire il rilevamento simultaneo di più agenti patogeni, migliorando la produttività negli ambienti di sorveglianza e test. Inoltre, l'integrazione del sequenziamento di nuova generazione (NGS) potrebbe fornire preziose informazioni sul sierotipo e sulla virulenza direttamente dalle colture di arricchimento di MPN, facilitando le indagini epidemiologiche e gli sforzi di tracciamento delle fonti.
Infine, il limite di quantificazione/rilevazione può essere ampliato aumentando la quantità di campioni utilizzati nell'MPN. Ad esempio, se il peso del campione di carne è stato aumentato da 25 g a 325 g, il limite di rilevabilità potrebbe essere estrapolato a 4 CFU/g utilizzando l'equazione di regressione lineare. Tuttavia, assumendo lo stesso intervallo di confidenza relativa al 95%, il limite inferiore pratico di quantificazione è di >30 CFU/g.
Conclusione
Lo sviluppo del test MPN-LAMP in questo studio offre uno strumento prezioso e pratico per quantificare la contaminazione da Salmonella nei prodotti avicoli. Con una forte correlazione con i livelli teorici di inoculazione (R² = 0,933), il metodo rileva in modo affidabile la Salmonella a bassi livelli, il che è fondamentale per la sicurezza alimentare e la conformità normativa. Questa capacità è particolarmente importante per rilevare contaminazioni che altrimenti potrebbero passare inosservate, consentendo ai produttori di affrontare in modo proattivo i potenziali rischi. Il test MPN-LAMP fornisce un'alternativa quantitativa ai metodi tradizionali basati sulla prevalenza, offrendo un livello di contaminazione da Salmonella in una varietà di prodotti avicoli, compresi i prodotti pronti da cuocere più complessi.
Uno dei punti di forza di questo metodo risiede nella sua adattabilità a diverse matrici alimentari, in particolare a prodotti surgelati e trasformati come il cordon bleu di pollo, dove i metodi tradizionali potrebbero avere difficoltà a causa della presenza di ingredienti aggiuntivi come panatura e formaggio. L'integrazione di tecniche molecolari, come LAMP e qPCR, con il test MPN garantisce un approccio completo al rilevamento, superando le sfide poste dai microbi di fondo e dalle condizioni di elaborazione. Questi metodi molecolari migliorano la sensibilità del protocollo, rendendolo uno strumento versatile nei test di sicurezza alimentare di routine e nella sorveglianza della contaminazione, con il potenziale di ridurre il rischio di focolai correlati alla Salmonella.
Sebbene non esista un singolo metodo di rilevamento privo di limitazioni, il test MPN-LAMP dimostra prestazioni costanti in una gamma di livelli di inoculo e matrici alimentari. La variabilità a livelli di inoculazione più bassi può essere affrontata attraverso futuri perfezionamenti, come l'ottimizzazione della preparazione dei campioni e delle formulazioni dei terreni per massimizzare il recupero, ma ciò non toglie nulla alla robustezza complessiva del protocollo. Fornendo un rilevamento accurato delle cellule di Salmonella , anche in condizioni difficili, il metodo MPN-LAMP fornisce all'industria alimentare uno strumento inestimabile per la salvaguardia della salute pubblica.
Guardando al futuro, c'è spazio per ulteriori miglioramenti, come l'espansione del test per includere più sierotipi di Salmonella o l'incorporazione del sequenziamento di nuova generazione per una maggiore caratterizzazione dei patogeni. Ciononostante, questo protocollo rappresenta già un progresso significativo nel rilevamento e nella quantificazione della Salmonella , fornendo un metodo pratico, efficiente e affidabile che soddisfa le crescenti esigenze dei test di sicurezza alimentare.
Divulgazioni
Tutti gli autori dichiarano che non vi è alcun conflitto di interessi.
Riconoscimenti
Questa ricerca è stata supportata dal Dipartimento dell'Agricoltura degli Stati Uniti, Servizio di Ricerca Agricola (USDA-ARS), Programma Nazionale 108, Corrente Sistema Informativo di Ricerca numeri 8072-42000-093-000-D e 8072-42000-094-000-D. La menzione di nomi commerciali o prodotti commerciali in questo articolo ha il solo scopo di fornire informazioni specifiche e non implica una raccomandazione o un'approvazione da parte del Dipartimento dell'Agricoltura degli Stati Uniti. L'USDA è un fornitore e datore di lavoro per le pari opportunità.
Materiali
| Name | Company | Catalog Number | Comments |
| 48 deep well block 4.6ml | Fisher Scientific International, Inc | NC1964628 | |
| Agar - Solidifying Agent (Difco) | Becton, Dickinson and Company (BD) | 281230 | |
| Analytical Balance | Mettler Toledo | JL602-G/L | Equipment |
| Analytical Balance | Mettler Toledo | AB54-S | Equipment |
| Autoclave - Amsco Lab250, Laboratory Steam Sterilizer | Steris plc | LV-250 | Equipment |
| Biological Safety Cabinet, Type A2, Purifier Logic+ | Labconco Corporation | 302411101 | Equipment |
| Brain Heart Infusion (BHI) Broth | Becton, Dickinson and Company (BD) | 237500 | |
| Buffered Peptone Water | Bio-Rad Laboratories Inc. | 3564684 | |
| Cell Spreader - L-shaped | VWR | 76208-438 | |
| Centrifuge Microcentrifuge 5424 | Eppendorf | 5424 | Equipment |
| Centrifuge, Avanti J-25 | Beckman Coulter, Inc. | Equipment | |
| DNA Extraction - PreMan Ultra Sample Preparation Reagent | Thermo Fisher Scientific Inc. | 4318930 | |
| Ground Chicken | Local retailers | ||
| IAC forward primer: 5'-GGCGCGCCTAACACATCT-3' | Integrated DNA Technologies | ||
| IAC probe: 5'-TTACAACGGGAGAAGACAATGC CACCA-3' labeled with 5' TAMRA/3' BHQ-2 | Biosearch Technologies | ||
| IAC reverse primer: 5'-TGGAAGCAATGCCAAATGTGTA-3' | Integrated DNA Technologies | ||
| Incubator - Inova 4230 incubator shaker | New Brunswick Scientific | 4230 | Equipment |
| Inoculating Loop - Combi Loop 10µL and 1µL | Fisher Scientific International, Inc | 22-363-602 | |
| invA forward primer: 5'-GTTGAGGATGTTATTCGCAAAG G-3' | Integrated DNA Technologies | ||
| invA probe: 5'-CCGTCAGACCTCTGGCAGTAC CTTCCTC-3' labeled with 5' Cal Fluor Orange 560/3' BHQ-1 | Biosearch Technologies | ||
| invA reverse primer: 5'-GGAGGCTTCCGGGTCAAG-3' | Integrated DNA Technologies | ||
| Irradiation Treatment | Texas A&M Agrilife Research National Center for Electron Beam Research | Service | |
| Luria Bertani (LB) Broth | Becton, Dickinson and Company (BD) | 244620 | |
| Manual pipette Pipet-Lite LTS Pipette L-1000XLS+ | Mettler Toledo | 17014382 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-100XLS+ | Mettler Toledo | 17014384 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-10XLS+ | Mettler Toledo | 17014388 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-200XLS+ | Mettler Toledo | 17014391 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-20XLS+ | Mettler Toledo | 17014392 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-200XLS+ | Mettler Toledo | 17013805 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-20XLS+ | Mettler Toledo | 17013803 | Equipment |
| Media Storage Bottle -PYREX 1L Square Glass Bottle, with GL45 Screw Cap | Corning Inc. | 1396-1L | Equipment |
| Media Storage Bottle -PYREX 2L Round Wide Mouth Bottle, with GLS80 Screw Cap | Corning Inc. | 1397-2L | Equipment |
| Microtiter plate, 96 well plate, flat bottom, polystyrene, 0.34cm2, sterile, 108/cs | MilliporeSigma | Z707902 | |
| Mixer - Vortex Genie 2 | Scientific Industries Inc. | SI-0236 | Equipment |
| Molecular Detection Assay 2-Salmonella kit | Neogen | MDA2SAL96 | |
| Molecular Detection Instrument | Neogen | MDS100 | Equipment |
| Motorized pipette controller, PIPETBOY2 | INTEGRA Biosciences Corp. | 155019 | Equipment |
| PCR Mastermix 2× TaqMan Gene Expression | Thermo Fisher Scientific Inc. | 4369542 | |
| Petri Dish Rotator - bioWORLD Inoculation Turntable | Fisher Scientific International, Inc | 3489E20 | Equipment |
| Petri Dishes with Clear Lid (100 mm x 15mm) | Fisher Scientific International, Inc | FB0875713 | |
| Pipette Tips GP LTS 1000µL S 768A/8 | Mettler Toledo | 30389273 | |
| Pipette Tips GP LTS 20µL 960A/10 | Mettler Toledo | 30389270 | |
| Pipette Tips GP LTS 200µL F 960A/10 | Mettler Toledo | 30389276 | |
| Ready to cook chicken products | Local retailers | ||
| Reagent Reservoir, 25 mL sterile reservoir used with multichannel pipettors | Thermo Fisher Scientific Inc. | 8093-11 | |
| Realtime PCR - 7500 Real-Time PCR system | (Applied Biosystems, Foster City, CA) | 2750036476 | Equipment |
| Serological Pipettes, Nunc Serological Pipettes (10 mL) | Thermo Fisher Scientific Inc. | 170356N | |
| Serological Pipettes, Nunc Serological Pipettes (2 mL) | Thermo Fisher Scientific Inc. | 170372N | |
| Serological Pipettes, Nunc Serological Pipettes (25 mL) | Thermo Fisher Scientific Inc. | 170357N | |
| Serological Pipettes, Nunc Serological Pipettes (50 mL) | Thermo Fisher Scientific Inc. | 170376N | |
| Spreader - Fisherbrand L-Shaped Cell Spreaders | Fisher Scientific International, Inc | 14-665-230 | |
| Stomacher bag, Nasco Whirl-Pak Write-On Homogenizer Blender Filter Bags | Thermo Fisher Scientific Inc. | 01-812 | |
| Stomacher 80 Biomaster Lab Blender | Seward | 30010019 | Equipment |
| Thermocycler (GeneAmp PCR system 9700) | Applied Biosystems | 535028293 | Equipment |
| Water Filtration - Elga Veolia Purelab Flex | Elga LabWater | PF2XXXXM1-US | Equipment |
| Whirlpak bags 1.63L | VWR | 11216-777 |
Riferimenti
- Batz, M., Hoffmann, S., Morris, J. G. Disease-outcome trees, eq-5d scores, and estimated annual losses of quality-adjusted life years (qalys) for 14 foodborne pathogens in the united states. Foodborne Pathogens and Disease. 11 (5), 395-402 (2014).
- . The Grand Challenge: Salmonella Available from: https://tellus.ars.usda.gov/stories/articles/the-grand-challenge-salmonella (2024)
- National Advisory Committee on Microbiological Criteria in Foods (NACMCF). Response to questions posed by the food safety and inspection service: Enhancing Salmonella control in poultry products. J Food Prot. 82 (4), 645-668 (2019).
- Food Safety and Inspection Service. . 4.15 Isolation and identification of Salmonella from meat, poultry, pasteurized egg, siluriformes (Fish) products and carcass and environmental sponges. , (2024).
- Irwin, P., Reed, S., Brewster, J., Nguyen, L., He, Y. P. Non-stochastic sampling error in quantal analyses for campylobacter species on poultry products. Analytical and Bioanalytical Chemistry. 405 (7), 2353-2369 (2013).
- Irwin, P., Tu, S., Damert, W., Phillips, J. A modified gauss-newton algorithm and ninety-six well micro-technique for calculating mpn using excel spreadsheets. Journal of Rapid Methods & Automation in Microbiology. 8 (3), 171-191 (2000).
- Ravishankar, S., Ahmed, E. Y., Carlstrom, C. Food microbiology: A laboratory manual. Food Microbiology. 21, 489 (2004).
- Chen, C. Y., Nace, G. W., Irwin, P. L. A 6 x 6 drop plate method for simultaneous colony counting and mpn enumeration of campylobacter jejuni, listeria monocytogenes, and escherichia coli. J Microbiol Methods. 55 (2), 475-479 (2003).
- Suo, B., He, Y., Tu, S. I., Shi, X. A multiplex real-time polymerase chain reaction for simultaneous detection of salmonella spp., escherichia coli o157, and listeria monocytogenes in meat products. Foodborne Pathogens and Disease. 7 (6), 619-628 (2010).
- Jarvis, B., Wilrich, C., Wilrich, P. T. Reconsideration of the derivation of most probable numbers, their standard deviations, confidence bounds and rarity values. J Appl Microbiol. 109 (5), 1660-1667 (2010).
- Stevens, R., Poppe, K. Validation of clinical prediction models: What does the "calibration slope" really measure. Journal of Clinical Epidemiology. 118, (2019).
- Miller, M. E., Hui, S. L., Tierney, W. M. Validation techniques for logistic regression models. Statistics in Medicine. 10 (8), 1213-1226 (1991).
- Galán-Relaño, &. #. 1. 9. 3. ;., et al. Salmonella and salmonellosis: An update on public health implications and control strategies. Animals. 13 (23), 3666 (2023).
- Gorski, L., et al. Growth assessment of salmonella enterica multi-serovar populations in poultry rinsates with commonly used enrichment and plating media. Food Microbiology. 119, 104431 (2024).
- Schmidt, J. W., et al. Evaluation of methods for identifying poultry wing rinses with salmonella concentrations greater than or equal to 10 cfu/ml. J Food Prot. 87 (11), 100362 (2024).
- Gorski, A., Liang, L. S. Effect of enrichment medium on real-time detection of salmonella enterica from lettuce and tomato enrichment cultures. Journal of Food Protection. 73 (6), 1047-1056 (2010).
- Guillén, S., Nadal, L., Álvarez, I., Mañas, P., Cebrián, G. Impact of the resistance responses to stress conditions encountered in food and food processing environments on the virulence and growth fitness of non-typhoidal salmonellae. Foods. 10 (3), 617 (2021).
- Rohde, A., Hammerl, J. A., Appel, B., Dieckmann, R., Al Dahouk, S. Sampling and homogenization strategies significantly influence the detection of foodborne pathogens in meat. BioMed Research International. 2015, (2015).
- Wang, D., Wang, Z., He, F., Kinchla, A. J., Nugen, S. R. Enzymatic digestion for improved bacteria separation from leafy green vegetables. Journal of Food Protection. 79 (8), 1378-1386 (2016).
- Pitard, F. F. . Theory of sampling and sampling practice. , (2019).
- Sharpe, A. . in Detecting pathogens in food. , 52-68 (2003).
- Hannah, J., et al. Effect of stomaching on numbers of bacteria recovered from chicken skin. Poultry Science. 90 (2), 491-493 (2011).
- Mcmeekin, T., Thomas, C. Retention of bacteria on chicken skin after immersion in bacterial suspensions. Journal of Applied Bacteriology. 45 (3), 383-387 (1978).
- Rodrigues-Szulc, U., Ventoura, G., Mackey, B., Payne, M. Rapid physicochemical detachment, separation and concentration of bacteria from beef surfaces. Journal of Applied Bacteriology. 80 (6), 673-681 (1996).
- Vibbert, H. B., et al. Accelerating sample preparation through enzyme-assisted microfiltration of salmonella in chicken extract. Biotechnol Prog. 31 (6), 1551-1562 (2015).
- Armstrong, C. M., et al. Use of a commercial tissue dissociation system to detect salmonella-contaminated poultry products. Analytical and Bioanalytical Chemistry. 416 (3), 621-626 (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon