Method Article
Modifizierter Most Probable Number Assay zur Quantifizierung von Salmonellen in rohen und küchenfertigen Hühnerprodukten
In diesem Artikel
Zusammenfassung
Die genaue Quantifizierung von Salmonellen bei Geflügel in geringen Mengen ist eine aktuelle industrielle und regulatorische Herausforderung. Dieses Protokoll beschreibt einen MPN-Assay, der die Quantifizierung von Salmonellen in rohen und küchenfertigen Geflügelprodukten ermöglicht. Diese Methode ist schnell, empfindlich und entspricht den FSIS-Richtlinien, verbessert die Lebensmittelsicherheit und unterstützt die Bemühungen um die öffentliche Gesundheit.
Zusammenfassung
Salmonellen sind eine der Hauptursachen für lebensmittelbedingte Krankheiten in den Vereinigten Staaten, insbesondere bei Geflügelprodukten. Herkömmliche Methoden zum Nachweis von Salmonellen konzentrieren sich eher auf die Prävalenz als auf die Quantifizierung, was ihren Nutzen bei der Bewertung von Kontaminationsgraden und -risiken einschränkt. In dieser Studie wird ein neuartiger MPN-Assay (Most Likely Number) vorgestellt, der zur Quantifizierung von Salmonellen in küchenfertigen Geflügelprodukten wie z. B. Chicken Cordon Bleu entwickelt wurde. Bei der Methode wird die Geflügelprobe gewaschen, die Spülung durch Zentrifugation konzentriert und in einem 48-Well-Block seriell verdünnt. Der MPN-Assay ist in die LAMP-Methode (Loop-Mediated Isothermal Amplification) integriert, um eine empfindliche, genaue und schnelle Quantifizierung der Salmonellenkontamination innerhalb des gleichen Zeitrahmens wie bestehende FSIS-Protokolle (Food Safety and Inspection Service) zu ermöglichen. Die Ergebnisse zeigen eine starke lineare Korrelation zwischen den MPN-LAMP-Messungen und den theoretischen Impfwerten (R² = 0,933). Die Variabilität bei niedrigeren Konzentrationen verdeutlicht jedoch die Herausforderung beim genauen Nachweis von Salmonellen in diesen Konzentrationen, wobei die praktische untere Nachweisgrenze auf etwa 300 KBE/g geschätzt wird. Zu den möglichen Verfeinerungen zur Verbesserung der Anwendbarkeit des Protokolls gehören die Erhöhung der Probenmenge, um die Nachweisgrenze weiter zu verbessern, die Optimierung der Formulierungen von Anreicherungsmedien und die Ausweitung des molekularen Nachweises auf mehrere Salmonella-Serovare . Insgesamt stellt diese Studie ein praktisches Instrument für die Lebensmittelindustrie dar, das eine zuverlässige Quantifizierung der Salmonellenkontamination in Geflügelprodukten ermöglicht und so zu einer verbesserten Lebensmittelsicherheit und öffentlichen Gesundheit beiträgt.
Einleitung
Als eine der Hauptursachen für lebensmittelbedingte Krankheiten, Krankenhausaufenthalte und Todesfälle in den USA haben Salmonellen erhebliche Auswirkungen auf die öffentliche Gesundheit und die Wirtschaft. Die geschätzte wirtschaftliche Belastung des Erregers belief sich allein im Jahr 2013 auf 3,67 Milliarden US-Dollar1. Obwohl die jüngsten Regulierungsinitiativen darauf abzielen, die Salmonellose bis 2030 um 25 % zu reduzieren2, sind nach wie vor Lücken in den derzeitigen Erkennungs- und Eindämmungsstrategien offensichtlich, insbesondere bei der Abstimmung der Überwachung von Verarbeitungsbetrieben auf die Ergebnisse der öffentlichen Gesundheit3 .
Gefrorene küchenfertige Geflügelprodukte, die mit mehreren Salmonellenausbrüchen in Verbindung gebracht wurden, stellen ein erhebliches Problem für die öffentliche Gesundheit dar. Als Reaktion darauf stufte der Food Safety and Inspection Service (FSIS) Salmonellen als Verfälschungsmittel in diesen Produkten ein. Derzeit konzentriert sich das FSIS Microbiology Laboratory Guidebook (MLG) 4.15 ausschließlich auf die Bestimmung der Prävalenz von Salmonellen in Geflügelprodukten4. Gemäß dieser Richtlinie werden die gesammelten Proben 18-24 Stunden lang angereichert und dann mit dem Molecular Detection System (MDS) gescreent, das das Vorhandensein oder Fehlen von Salmonellen identifiziert, aber keinen Einblick in den Grad der Kontamination bietet. Dieser Ansatz ist zwar wertvoll für den Nachweis von Krankheitserregern, liefert aber keine quantitativen Informationen, die Lebensmittelverarbeitern helfen könnten, Kontaminationsrisiken genauer einzuschätzen und gezielte Korrekturmaßnahmen zu ergreifen.
In dieser Studie haben wir eine Methode entwickelt, um den Nachweis von mikrobiellen Krankheitserregern von der Prävalenz bis zur Quantifizierung zu erweitern. Es wurde für die nahtlose Integration in bestehende Prozesse zum Nachweis von Salmonellen in Geflügelprodukten mit minimaler Unterbrechung der aktuellen FSIS-Protokolle entwickelt. Anstatt einfach nur die Massenprobe anzureichern, beginnt die Methode mit dem Waschen der Geflügelprodukte mit Medien, die den aktuellen FSIS-Methoden entsprechen. Die Spülung wird dann in die erste Säule eines 48 tiefe Brunnenblocks verteilt. Serielle Verdünnungen werden über die verbleibenden fünf Säulen durchgeführt, und der Block wird 18-24 Stunden lang inkubiert, in Übereinstimmung mit dem MLG 4.15-Protokoll. Nach der Inkubation werden die Vertiefungen auf Salmonellen untersucht, und die Ergebnisse werden zur Berechnung der wahrscheinlichsten Zahl (MPN)5,6 verwendet. Dieser Ansatz ermöglicht die Quantifizierung der Kontamination innerhalb des gleichen Zeitrahmens wie das derzeitige FSIS-Verfahren, was ihn zu einer praktischen Option sowohl für die Industrie als auch für den behördlichen Einsatz macht. Abbildung 1 zeigt ein Blockdiagramm, das den modifizierten MPN-Assay zusammenfasst. Die Abbildung umfasst Fotos, die in bestimmten Schritten aufgenommen wurden, den 48-Well-Block, der für die Verdünnung und das Wachstum von Replikaten verwendet wird, und die drei Techniken, die als Benchmarks zur Bewertung der wahrscheinlichsten Anzahl von Salmonellen in Hackfleisch verwendet werden. In der ersten Phase dieser Studie verwendeten wir bestrahltes Hackfleisch, um die Auswirkungen der Hintergrundmikroflora und die Unsicherheit der Messungen in Bezug auf das verifizierte Inokulum zu minimieren, bevor wir das Protokoll auf nicht bestrahlte Hühnerproben anwendeten.
Protokoll
HINWEIS: Alle Arbeiten im Zusammenhang mit diesem Protokoll sollten in einem Labor der Biosicherheitsstufe 2 (BSL-2) durchgeführt werden. Gegebenenfalls sollte dieses Protokoll in einer biologischen Sicherheitswerkbank (BSC) durchgeführt werden, um aseptische Bedingungen aufrechtzuerhalten und das Risiko einer Probenkontamination oder einer Exposition des Bedieners gegenüber mikrobiellen Krankheitserregern zu minimieren. Wenn Sie Proben außerhalb des BSC transportieren, verwenden Sie versiegelte Behälter, um die Integrität der Proben zu wahren und ein Verschütten im Falle eines versehentlichen Herunterfallens zu verhindern. Vorzugsweise sollten während des gesamten Verfahrens Einwegkomponenten verwendet werden, um die Möglichkeit einer Kreuzkontamination zu verringern. In Fällen, in denen Einwegartikel nicht möglich sind, stellen Sie sicher, dass alle Geräte und Materialien vor der Verwendung steril sind. Eine ordnungsgemäße Abfallbewirtschaftung ist von entscheidender Bedeutung. Alle verwendeten Einwegkomponenten sollten als biologisch gefährlicher Abfall entsorgt werden. Autoklavieren Sie wiederverwendbare Materialien vor der Wiederverwendung, um eine ordnungsgemäße Sterilisation und Eindämmung potenziell gefährlicher Materialien zu gewährleisten. Die Einhaltung dieser Vorsichtsmaßnahmen schützt nicht nur die Integrität der Probe, sondern minimiert auch das Risiko einer Exposition des Bedieners gegenüber mikrobiellen Krankheitserregern.
1. Vorbereitung der Fleischproben
- Gewinnung und Verarbeitung von Fleischproben
- Frischfleisch
- Erwerben Sie Hackfleisch in der Frischfleischabteilung des örtlichen Einzelhandels. Alle Proben werden bei 4 °C gelagert und innerhalb von 24 Stunden nach Erhalt verarbeitet. Fleisch aseptisch in 25 g Proben aufteilen.
- Vakuumieren Sie die Probe und bestrahlen Sie sie. Hier bestrahlte das Texas A&M AgriLife National Center for Electron Beam Research Fleisch einer Dosis von ~25 kGy.
ANMERKUNG: Obwohl die Bestrahlung in dieser Studie als Kontrollmaßnahme verwendet wurde, um die Beseitigung der Hintergrundmikroflora zu gewährleisten, ist sie in der Praxis keine Voraussetzung für das Protokoll, wie die im folgenden Abschnitt verwendeten nicht bestrahlten küchenfertigen Produkte zeigen. Im Feld können alternative Methoden wie selektive Medien oder Spezifität der molekularen Diagnostik potenzielle Interferenzen durch Nichtziel-Mikroorganismen berücksichtigen.
- Kochfertige Hähnchenprodukte
- Erwerben Sie küchenfertige Hähnchenprodukte in der Tiefkühlabteilung des örtlichen Einzelhandels. Aseptisch in 25 g Proben aufteilen.
- Entnehmen Sie Proben aus der Mitte der einzelnen Stücke, um sicherzustellen, dass alle Zutaten (z. B. Panade und Käse) enthalten sind.
- Frischfleisch
- Vorbereitung der Medien
- Bereiten Sie gepuffertes Peptonwasser (BPW) vor, indem Sie 25 g BPW-Pulver in 1 L nanoreinem H2O auflösen.
- Bereiten Sie Platten für die Gehirn-Herz-Infusion (BHI) vor. Dazu lösen Sie 37 g BHI-Pulver in 1 L nanoreinem H2O auf und geben Sie 15 g Agar in BHI-Lösung. Sterilisieren Sie alle Medien durch Autoklavieren bei 121 °C für 15 Minuten. Gießen Sie 20 bis 25 ml Medium in Petrischalen mit durchsichtigem Deckel (100 mm x 15 mm).
HINWEIS: Es ist am besten, die Platten in eine biologische Sicherheitswerkbank zu gießen, um aseptische Bedingungen aufrechtzuerhalten.
2. Zellkultur
- Die erste Kultur wird vorbereitet, indem Salmonella enterica serovar Typhimurium ATCC 14028 auf eine BHI-Agarplatte gestreift und über Nacht bei 37 °C inkubiert wird.
- Bereiten Sie Übernachtkulturen vor, indem Sie 25 ml BHI-Brühe mit einer Kolonie frisch gezüchteter Salmonellen beimpfen. Züchten Sie die Kulturen aerob über Nacht bei 37 °C und schütteln Sie sie bei 100 U/min.
3. Inokulation von Geflügelproben
- Kulturverdünnung und -plattierung
- Bereiten Sie eine Reihe von 10-fachen Verdünnungen der Übernachtkultur in BPW vor, um Endkonzentrationen von ca. 1 x 108 bis 1 x 101 KBE/ml zu erreichen. Es wird angenommen, dass die Konzentration der Salmonellen-Übernachtkultur 1 x 109 KBE/ml beträgt.
- 0,5 ml der Übernachtkultur in 4,5 ml BPW überführen, mischen und dann 0,5 ml der Verdünnung in 4,5 ml BPW für jede weitere Verdünnung übertragen.
- Verteilen Sie 10 μl der 1 x 103 KBE/ml-Verdünnung auf einer BHI-Agarplatte in dreifacher Ausfertigung für die Zellzählung, um die Kulturkonzentration über Nacht zu berechnen.
- Inokulation von Fleischproben
- 25 g bestrahltes Hähnchenhackfleisch in zweifacher Ausfertigung aseptisch in einen sterilen Magenbeutel umfüllen. Die Tasche ist 7,5 x 12 Zoll groß und mit 1,63 l gekennzeichnet. Der Beutel enthält eine Filtertrennwand mit einem Lochdurchmesser von 330 μm, davon sind es 285 pro Quadratzentimeter.
- Beimpfen Sie jede Probe mit 1 mL der Verdünnung der Zielkonzentration. Fügen Sie beispielsweise 1 ml Kultur aus der Verdünnung von 1 x 10 μm KBE/ml hinzu, um einen Kontaminationsgrad von ca. 1.000 Zellen/25 g Huhn zu erreichen. Verteilen Sie das flüssige Inokulum vorsichtig mit einem sterilen Zellverteiler auf der Oberfläche der Hühnerproben und lassen Sie es 1 h bei 4 °C stehen.
- Bereiten Sie negative Kontrollproben vor, indem Sie 1 ml steriles BPW hinzufügen.
4. Probenverarbeitung
- Geben Sie 225 mL BPW zu jeder Probe. Das Verhältnis von Volumen zu Medium wurde so gewählt, dass es sich an FSIS MLG 4.154 orientiert.
- Homogenisieren Sie die Proben mit dem Stomacher7 bei normaler Geschwindigkeit und einer Dauer von 120 s.
- Zentrifugation und Resuspension
- Entfernen Sie die Flüssigkeit vorsichtig mit einer 50-ml-Pipette von der gefilterten Seite des Beutels. Teilen Sie die Flüssigkeit in zwei sterile Zentrifugenflaschen auf.
- Gleichen Sie die Flaschen mit sterilem BPW aus, um ein gleichmäßiges Gewicht zu gewährleisten. Zentrifugieren Sie die Proben bei 10.000 x g für 10 min. Entsorgen Sie den Überstand. Das Zellpellet in 3 mL BPW-Bouillon mit einem sterilen Spatel resuspendieren.
- Fügen Sie weitere 27 ml BPW-Brühe hinzu und mischen Sie sie gründlich unter Rühren mit einem Spatel. Kombinieren Sie den Inhalt beider Zentrifugenflaschen in einer Flasche für jede Probe.
5. Einrichtung des MPN-Blocks
HINWEIS: Tabelle 1 zeigt ein Schema der Verdünnungen in einem 48-Well-Block.
- Geben Sie 3 ml der resuspendierten Probe in jede Vertiefung in Spalte 1 des 48-Well-Blocks (8 Replikate).
- Bereiten Sie mit einer Achtkanalpipette eine Reihe von 10-fachen Verdünnungen über die Säulen 1 bis 6 innerhalb des Blocks vor.
- 0,3 mL der Probe in 2,7 mL BPW Pipette geben, um zu mischen. Wiederholen Sie den Vorgang für jede Verdünnung. Inkubieren Sie die Blöcke über Nacht (~18 h) bei 37 °C und schütteln Sie sie bei ~100 U/min.
6. Plattieren und Aufzählen
- Geänderte Aufzählung von Fallplatten
- Platte 7 μl einer über Nacht gewachsenen Probe jeder Verdünnung in einem 4 x 6-Gitter auf einer Agarplatte mit einer Mehrkanalpipette (Abbildung 2). Die Verwendung eines 4 x 6-Gitters auf zwei Platten bietet Platz für 8 Proben im Gegensatz zum typischen 6 x 6-Gitter von Tröpfchen8.
- Lassen Sie die Platten vor der Inkubation 10 Minuten an der Luft trocknen. Inkubieren Sie die Agarplatten über Nacht (~18-24 h) bei 37 °C. Zählen Sie nach der Inkubation die Anzahl der Kolonien auf jeder Platte.
7. qPCR-Nachweis von Salmonellen
- DNA-Extraktion mit einem kommerziellen Kit
- Mischen Sie die Kulturen im 48-Well-Block, indem Sie mehrmals auf und ab pipettieren. Pipettieren Sie 200 μl jeder Kultur in eine 96-Well-PCR-Platte.
- Verschließen Sie die Platte und zentrifugieren Sie sie anschließend bei 6.600 x g für 10 min. Entfernen Sie den Überstand und fügen Sie dem Pellet 20 μl des Kit-Reagenzes hinzu.
- Resuspendieren Sie das Pellet, indem Sie auf und ab pipettieren. Die Platte verschließen und 10 Minuten lang bei 99 °C erhitzen, anschließend auf 20 °C abkühlen lassen.
- Erneut bei 6.600 x g für 10 min zentrifugieren. Verwenden Sie 2 μl des Überstands für die qPCR-Analyse.
- Platten-Setup
- Bereiten Sie das qPCR-Reaktionsgemisch gemäß dem festgelegten Protokoll9 wie folgt vor: 10 μl 2x Master Mix; 0,4 μl von jedem Primer und jeder Sonde (10 μM Arbeitslösung): invA vorne: 5'-GTTGAGGATGTTATTCGCAAAGG-3', invA rückwärts: 5'-GGAGGCTTCCGGGTCAAG-3', invA-Sonde: 5'-CCGTCAGACCTCTGGCAGTACCTTCCTC-3', markiert mit dem Cal Fluor Orange 560 Fluorenfarbstoff; 0,2 μl Vorlage für die interne Amplifikationskontrolle (IAC) (6 x 104 Kopien/μl)9; 0,4 μl jedes IAC-Primers und jeder Sonde (10 μM): IAC vorne: 5'-GGCGCGCCTAACACATCT-3', IAC rückwärts: 5'-TGGAAGCAATGCCAAATGTGTA-3', IAC-Sonde: 5'-TTACAACGGGAGAAGACAATGCCACCA-3', markiert mit TAMRA-Farbstoff. Stellen Sie das Volumen mit ddH2O bis insgesamt 20 μl ein.
- Führen Sie die Echtzeit-PCR unter den folgenden Zyklusbedingungendurch 9: 95 °C für 10 min (initiale Denaturierung der DNA und Aktivierung der Heißstart-Polymerase), 40 Zyklen von 95 °C für 15 s und 60 °C für 1 min, verwenden Sie die Standard-Ct-Einstellungen, um die Ergebnisse für die Analyse zu exportieren.
8. Nachweis mit dem 3M MDS-Assay
- Befolgen Sie das Protokoll des Salmonella-Kits für den molekularen Nachweisassay. Mischen Sie die Kulturen im 48-Well-Block, indem Sie mehrmals auf und ab pipettieren. Pipettieren Sie 20 μl jeder Probe in das im Kit enthaltene Analyseröhrchen.
- Die Proben 15 min lang auf 100 °C erhitzen. Die Lösung färbt sich von rosa zu gelb. Inkubieren Sie die Proben 10 Minuten lang bei Raumtemperatur. Die Lösung wechselt von gelb zu rosa.
- Übertragen Sie 20 μl Lysat in ein Reagenzröhrchen und laden Sie die Reagenzröhrchen in den Halter.
- Fügen Sie den Halter zum MDS-Gerät hinzu und konfigurieren Sie die Software so, dass die Informationen über das Kit und die Probe übermittelt werden. Das Gerät erfordert, dass jede Vertiefung mit der Chargennummer für den Assay und einem Probennamen beschriftet ist. Führen Sie die MDS-Software aus, und exportieren Sie den Bericht.
9. Datenanalyse
- Klassifizierung von positiven und negativen Ergebnissen.
- Bei einer 4 x 6-Tropfen-Plattierung sind Flecken auf Agarplatten mit mindestens 1 Kolonie als positiv und Flecken auf Agarplatten ohne Wachstum als negativ zu bewerten.
- Bewerten Sie für die qPCR Vertiefungen mit einem Ct von weniger als oder gleich 30 als positiv und Vertiefungen mit einem Ct von mehr als 30 als negativ.
- Verwenden Sie für MDS die Ergebnisse aus dem MDS-System, die als positiv oder negativ gemeldet wurden.
- MPN-Berechnung
- Analysieren Sie kommentierte positive und negative Werte mit derzuvor beschriebenen einfachen SMPR-Methode (Maximum Probability Resolution) oder alternativen verifizierten MPN-Rechnern. 10
Ergebnisse
Bestrahltes Fleisch
In der Regressionsanalyse bedeutet eine Steigung von 1, dass für jede Einheitenzunahme der unabhängigen Variablen (x-Achse) die abhängige Variable (y-Achse) um genau 1 Einheit zunimmt. Dies deutet auf eine proportionale Beziehung zwischen den beiden Variablen hin, was bedeutet, dass die Änderung der abhängigen Variablen die Änderung der unabhängigen Variablen widerspiegelt. Ein Knotenpunkt von 0 bedeutet, dass, wenn die unabhängige Variable 0 ist, die abhängige Variable ebenfalls 0 ist. Dies deutet darauf hin, dass es keinen festen Versatz oder Bias in der Beziehung zwischen den beiden Variablen gibt. Zusammen deuten eine Steigung von 1 und ein Schnittpunkt von 0 auf eine Übereinstimmung zwischen den Variablen11,12 hin. Dieses ideale Szenario deutet auf keinen systematischen Fehler oder Verzerrungen hin und wäre das erwartete Ergebnis in einem gut kalibrierten System, bei dem die Messungen perfekt mit den Vorhersagen übereinstimmen.
Abbildung 3A zeigt den MPN-LAMP-Assay (y-Achse), aufgetragen gegen das theoretische Inokulum, das durch die in der Anmerkung in Schritt 3.1.1 beschriebene Spreizplattenanalyse verifiziert wurde. (x-Achse). Die Regressionsliniengleichung y = -72,36 + 0,161x mit einem R²-Wert von 0,978 weist auf eine starke lineare Korrelation zwischen den MPN-LAMP-Ergebnissen und den inokulierten Bakterienkonzentrationen hin. Der hohe R²-Wert deutet darauf hin, dass der MPN-LAMP-Assay bei der quantitativen Vorhersage einer bakteriellen Kontamination sehr zuverlässig ist. Die Steigung von 0,161 weicht jedoch deutlich von der idealen Steigung von 1 ab, was darauf hindeutet, dass die MPN-LAMP-Methode das Inokulum unterschätzt und nur 16,1 % der Bakterienkonzentration im theoretischen Inokulum vorhersagt. Darüber hinaus deutet der negative Abschnitt von -72,36 auf eine negative Verzerrung bei niedrigeren Konzentrationen hin, die weiter vom Ideal abweicht.
Abbildung 3B zeigt die MPN-qPCR-Methode (y-Achse), aufgetragen gegen das theoretische Inokulum (x-Achse). Die Regressionslinie y = -46,84 + 0,0395x mit einem R²-Wert von 0,978 zeigt eine starke lineare Beziehung zwischen dem MPN-qPCR-Assay und dem theoretischen Inokulum. Obwohl der hohe R² auf eine robuste Korrelation hinweist, ist die Steigung von 0,0395 noch kleiner als die der MPN-LAMP-Ergebnisse, was auf eine stärkere Unterschätzung der bakteriellen Kontamination durch MPN-qPCR hindeutet. Der negative Abschnitt von -46,84 deutet weiterhin auf eine mögliche Abwärtstendenz bei niedrigeren Konzentrationen hin.
Abbildung 3C zeigt die MPN-Plattierungsergebnisse (y-Achse) aufgetragen gegen das theoretische Inokulum (x-Achse), mit einer Regressionsgleichung von y = -330,4 + 0,3138x und einem R²-Wert von 0,944. Dieser R²-Wert deutet zwar auf eine starke lineare Beziehung hin, ist aber etwas niedriger als die der MPN-LAMP- und MPN-qPCR-Ergebnisse, was auf eine größere Variabilität bei der Quantifizierung der Kolonie hindeutet. Die Steigung von 0,3138 ist zwar höher als die in den anderen Methoden beobachteten Steigungen, deutet aber immer noch auf eine Unterschätzung der Bakterienkonzentration im Vergleich zum theoretischen Inokulum hin. Darüber hinaus deutet der negative Abschnitt von -330,4 auf eine beträchtliche Verzerrung hin, insbesondere bei niedrigeren Konzentrationen, was auf mögliche Ungenauigkeiten bei den Beschichtungstechniken oder der Probenhandhabung hinweisen könnte. Diese leichte Verringerung von R² und die beobachtete Variabilität könnten darauf hindeuten, dass der traditionelle Beschichtungsprozess variabler ist als die molekularen Methoden.
Vergleichsmethoden
Alle drei Methoden (MPN-LAMP, MPN-Plating und MPN-qPCR) zeigten eine Tendenz, die Bakterienkonzentration im Vergleich zur theoretischen Inokulation zu unterschätzen. Diese Unterschätzung kann auf Probleme bei der Zellrückgewinnung zurückgeführt werden, bei denen sich Salmonellen während der Verarbeitung möglicherweise nicht vollständig von der Hühneroberfläche oder den Partikeln lösen, wodurch der Transfer in die Spülung oder den Assay verringert wird. Da die Salmonellen direkt auf das Huhn geimpft wurden, kann ein Teil der Bakterienzellen stark an die Oberfläche binden, was die Erholung im Spülschritt einschränkt. Darüber hinaus können einige Zellen nach der Zentrifugation an Partikeln im Pellet haften, was die Rückgewinnung weiter verringert. Ansätze wie enzymatische Behandlungen, mechanische Verarbeitung oder optimierte Magenprotokolle könnten die bakterielle Ablösung verbessern. Diese Strategien werden im Diskussionsteil näher erläutert, wobei ihr Potenzial zur Verbesserung der Rückgewinnungsraten hervorgehoben wird
Abbildung 4A zeigt die Wiederherstellung auf der y-Achse im Vergleich zu drei MPN-Methoden auf der x-Achse. Die prozentuale Wiederfindung, berechnet als Verhältnis des MPN-Wertes zum theoretischen Impfniveau, ergab folgende Ergebnisse: Die MPN-LAMP-Methode hatte eine mittlere Wiederfindung von 15,19 % mit einer Standardabweichung von 10,04 %; die MPN-Plattierungsmethode hatte eine mittlere Wiederfindung von 13,13 % mit einer Standardabweichung von 11,45 %; und die MPN-qPCR-Methode hatte eine mittlere Wiederfindung von 6,67 % mit einer Standardabweichung von 3,19 %. Ein Tukey-Kramer-HSD-Test ergab keinen statistisch signifikanten Unterschied in der Wiederfindung zwischen den Methoden (P > 0,1969), was darauf hindeutet, dass alle drei Methoden bei der Verarbeitung von Salmonellen-geimpftem Hühnchen in Bezug auf die Wiederfindung ähnlich abschnitten.
Trotz identischer Behandlung bei allen Methoden zur Trennung von Salmonellen aus der Hühnermatrix wurden Unterschiede beim Nachweis zwischen MPN-LAMP, MPN-qPCR und MPN-Plattierung beobachtet. Dies deutet darauf hin, dass Faktoren, die über die physikalische Trennung von Bakterien aus der Probe hinausgehen, die Detektion beeinflussen können. Das Vorhandensein von Inhibitoren in der Probenmatrix könnte die molekularen Methoden qPCR und LAMP beeinträchtigen und deren Empfindlichkeit und Gesamtleistung beeinträchtigen. Darüber hinaus könnte die bakterielle Lebensfähigkeit die Fähigkeit von Salmonellen beeinflusst haben, auf festen Agarplatten (MPN-Beschichtung) im Vergleich zu flüssiger Brühe (qPCR, LAMP) zu wachsen, was zu einer unterschiedlichen Wiederfindung zwischen den Methoden führte.
Um die Sensitivität der Methoden weiter zu untersuchen, untersuchten wir zunächst die Anzahl der positiven Reaktionen bei jeder Verdünnung für MPN-LAMP, MPN-qPCR und MPN-Plattierung in sieben unabhängigen Studien (Tabelle 2). Bei niedrigeren Verdünnungen (1 x 10-5 und 1 x 10-4) zeigte MPN-LAMP eine höhere Anzahl positiver Reaktionen im Vergleich zu MPN-Plattierung und MPN-qPCR, was darauf hindeutet, dass die LAMP-Methode empfindlicher auf niedrige Konzentrationen von Salmonellen reagiert. Bei stärkerer Konzentration der Proben (Verdünnungen unter 1 x 10-3) zeigten alle drei Methoden eine Zunahme positiver Nachweise, wobei die Konvergenz um die 1 x 10-2-Verdünnung herum erfolgte, wobei alle Methoden eine ähnliche Anzahl positiver Proben detektierten.
Abbildung 4B zeigt den Prozentsatz positiver Messungen bei jeder Verdünnung für die drei Methoden und bietet einen direkten Vergleich ihrer Empfindlichkeit beim Nachweis von Salmonellen. Der Prozentsatz der positiven Reaktionen bietet eine klarere Visualisierung der Methodenleistung über die Verdünnungsreihe hinweg, und Fehlerbalken, die ± Clopper-Pearson-Konfidenzintervalle (CI) von 95 % darstellen, geben Einblick in die Variabilität auf jeder Ebene. Bei niedrigeren Verdünnungen weisen die breiteren Konfidenzintervalle auf eine größere Variabilität in der Nachweisleistung der einzelnen Methoden hin. In Übereinstimmung mit der reinen Anzahl positiver Nachweise erkennt MPN-LAMP einen höheren Prozentsatz positiver Proben bei den am stärksten verdünnten Konzentrationen (1 x 10-5), während MPN-qPCR und MPN-Plating weniger positive Nachweise aufweisen. Mit zunehmender Bakterienkonzentration konvergieren die Methoden, wobei alle drei Ansätze bei höheren Konzentrationen (z. B. 1 x 10-2 und höher) einen Nachweis von nahezu 100 % zeigen. Die Verengung der Konfidenzintervalle bei diesen höheren Konzentrationen deutet auf eine größere Konsistenz aller Methoden hin, was ihre vergleichbare Leistung bei höheren Bakteriengehalten unterstreicht.
Die Ergebnisse in Abbildung 3 deuten darauf hin, dass die konsistente Unterprognose (quantitativ) aller drei Methoden auf eine unvollständige Trennung der Salmonellen von der Probenmatrix zurückzuführen sein könnte. Molekulare Inhibitoren oder bakterielle Viabilitätsfaktoren könnten jedoch die beobachtete Variation des Nachweises bei verschiedenen Verdünnungen (qualitativ) und zwischen verschiedenen Methoden (qualitativ und quantitativ) antreiben. Die engeren Konfidenzintervalle bei höheren Konzentrationen in Abbildung 4B deuten auf eine geringere Variabilität und eine konsistentere qualitative Leistung zwischen den Methoden hin, während die breiteren Intervalle bei niedrigeren Konzentrationen auf eine erhöhte Unsicherheit bei der qualitativen Detektion hinweisen.
Detektion in küchenfertigem Hähnchen
Wir wählten die LAMP-Methode zum Nachweis von Salmonellen in küchenfertigem Hähnchen, da sie eine größere Anzahl positiver Proben (14,3%) aufwies als andere Methoden, die mit der geringsten Salmonellenverdünnung getestet wurden. Bei der niedrigsten Salmonellenverdünnung (1 x 10-5) wurde keine der Proben mit der hauseigenen qPCR-Methode als positiv nachgewiesen. Auf der anderen Seite wurden einige positive Aspekte durch Plattierung nachgewiesen (7,1 %, die Hälfte der von LAMP nachgewiesenen Anzahl). Die Ergebnisse dieser kulturbasierten Methode können jedoch beeinträchtigt werden, wenn auf komplexere Produkte wie küchenfertiges Hähnchen umgestellt wird, bei denen das Vorhandensein von Hintergrundmikroflora die Ergebnisse verzerren könnte, da die Proben nicht bestrahlt werden. Aus diesen Gründen haben wir uns entschlossen, die MPN-LAMP-Methode zur Quantifizierung von Salmonellen aus küchenfertigem Hähnchen zu untersuchen.
Abbildung 5 zeigt die Ergebnisse des MPN-LAMP-Assays, der zur Quantifizierung von Salmonellen in inokulierten, gefrorenen, küchenfertigen Hühnchen-Cordon-Bleu-Proben verwendet wurde. Das theoretische Niveau der Inokulation (KBE/g) wurde auf der x-Achse aufgetragen, während die entsprechenden MPN-LAMP-Messungen auf der y-Achse aufgetragen wurden. Die Regressionsanalyse ergab die Gleichung y = -15,04 + 0,3869x mit einem Bestimmungskoeffizienten (R²) von 0,933, was auf eine starke lineare Korrelation zwischen dem theoretischen Inokulum und den MPN-LAMP-Ergebnissen hinweist. Dieser hohe R²-Wert deutet darauf hin, dass die MPN-LAMP-Methode die Salmonellen in diesen Proben zuverlässig quantifiziert, obwohl die Steigung von 0,3869 darauf hindeutet, dass die Methode den tatsächlichen Inokulumspiegel unterschätzt. Der schattierte Bereich um die Regressionslinie stellt das Konfidenzintervall von 95 % dar.
Um die Leistung von MPN-LAMP bei niedrigeren Impfwerten besser zu veranschaulichen, zoomt das Nebendiagramm in Abbildung 5 auf Datenpunkte unter 300 KBE/g. Basierend auf der Regressionsgleichung kann die untere Nachweisgrenze auf etwa 40 KBE/g (38,9 KBE/g) geschätzt werden. Das Konfidenzintervall von 95 % deutet jedoch darauf hin, dass die praktische untere Nachweisgrenze eher bei 325 KBE/g liegt, da negative MPN-LAMP-Werte als Null interpretiert werden sollten. Die rote Linie im Einschub stellt Null dar, und obwohl keine Datenpunkte unter diese Linie fielen, besteht die Möglichkeit, dass die Werte unter Null fallen, insbesondere bei niedrigeren Inokulumwerten, die Möglichkeit falsch-negativer Ergebnisse. Dieser Trend verdeutlicht die Schwierigkeiten bei der genauen Quantifizierung von Salmonellen bei niedrigen Konzentrationen, und es wären zusätzliche Daten erforderlich, um die untere Nachweisgrenze endgültig festzulegen.

Abbildung 1: Blockdiagramm des modifizierten MPN-Assays für den Salmonellennachweis .Ein schematischer Überblick über den modifizierten MPN-Assay (Most Probable Number). Es werden die wichtigsten Schritte des Prozesses beschrieben, beginnend mit der Inokulation von bestrahlten Hühnerhackfleischproben, gefolgt von der Probenhomogenisierung, Zentrifugation, aliquoten Verteilung und Inkubation unter bestimmten Bedingungen. Die Abbildung enthält Fotos des 48-Well-Blocks, der für die Verdünnung und das Replikationswachstum verwendet wird, sowie drei Techniken, die verwendet werden, um positive und negative Ergebnisse für die Berechnung der Simple Maximum Probability Resolution (SMPR) zu erzielen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Schematische Darstellung des Drop-Plate-Assays. Der Drop-Plate-Assay-Aufbau für eine bestimmte Verdünnung (z. B. unverdünnt, 10-1, 10-2 usw.) aus dem 48-Well-Block. Jede Probe wird in einem 4 x 6-Rasterformat mit einer Mehrkanalpipette auf einer Agarplatte plattiert. Das Layout wurde vom traditionellen 6 x 6-Raster auf zwei 4 x 6-Raster umgestellt, um sicherzustellen, dass sechs Replikate aus acht Verdünnungen untergebracht werden können. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Vergleich von Inoculum mit MPN-Methoden. (A) Ein Diagramm der MPN-LAMP-Konzentration (y-Achse) gegen das theoretische Inokulum, verifiziert durch Plattierung (x-Achse). Eine angepasste Regressionslinie (y = -72,36 + 0,161x) mit einem Bestimmtheitskoeffizienten (R² = 0,978) zeigt eine starke lineare Beziehung. (B) Ein Diagramm der MPN-qPCR-Konzentration (y-Achse) gegen das theoretische Inokulum (x-Achse), mit einer angepassten Regressionslinie (y = -46,84 + 0,0395x) und R² = 0,978, die eine ähnlich starke Korrelation zeigt. (C) Ein Diagramm der MPN-Plattierungskonzentration (y-Achse) gegen das theoretische Inokulum (x-Achse), mit einer Regressionslinie (y = -330,4 + 0,3138x) und R² = 0,944, was auf eine starke, aber etwas geringere Korrelation als bei LAMP und qPCR hinweist. Die Regressionsgleichung und der Bestimmtheitskoeffizient für jede Zahl werden als Einlage in der oberen linken Ecke der Diagramme dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Vergleich der MPN-Methoden. (A) Die Abbildung zeigt die prozentuale Wiederfindung, berechnet als Verhältnis des MPN-Werts zum theoretischen Impfniveau für MPN-LAMP-, MPN-Plating- und MPN-qPCR-Methoden. Die MPN-LAMP-Methode hatte eine mittlere Wiederfindung von 15,19 % (±10,04 %), die MPN-Plattierungsmethode eine mittlere Wiederfindung von 13,13 % (± 11,45 %) und die MPN-qPCR-Methode eine mittlere Wiederfindung von 6,67 % (± 3,19 %). Die Boxplots verdeutlichen die Verteilung der Wiederherstellungsdaten, wobei Ausreißer angezeigt sind. Fehlerbalken stellen den Datenbereich innerhalb des 1,5-fachen des Interquartilsabstands (IQR) dar. (B) Prozentsatz positiver Salmonellennachweise bei jeder Verdünnung bei drei Methoden: MPN-LAMP (blau), MPN-Beschichtung (rot) und MPN-qPCR (grün). Die x-Achse stellt die Verdünnungsstufen (10-6 bis 10) dar, und die y-Achse zeigt den Prozentsatz der positiven Messungen. Fehlerbalken zeigen die ± Clopper-Pearson-Konfidenzintervalle von 95 % an, die die Variabilität der Erkennung für jede Methode widerspiegeln. MPN-LAMP zeigt bei niedrigeren Verdünnungen einen höheren Prozentsatz an Detektionen als andere Methoden, während alle Methoden bei höheren Konzentrationen nahe 100 % Detektion konvergieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
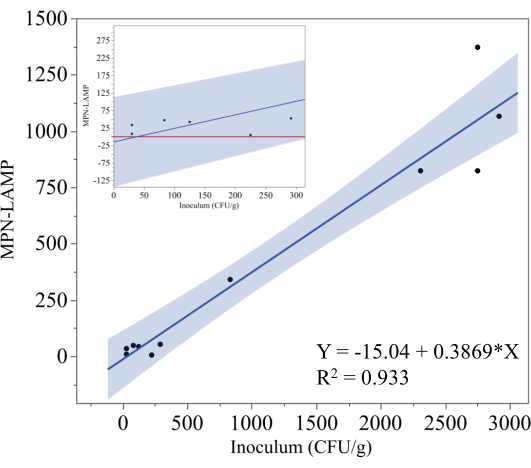
Abbildung 5: MPN-Methode für küchenfertiges Hähnchen. Regressionsanalyse der MPN-LAMP-Ergebnisse gegen die theoretische Inokulationsrate von Salmonellen in gefrorenen, küchenfertigen Hühnchen-Cordon-Bleu-Proben. Die Regressionslinie wird durch y = -15,04 + 0,3869x mit einem R² von 0,933 dargestellt, was auf eine starke lineare Beziehung hinweist. Der schattierte Bereich stellt das Konfidenzintervall von 95 % dar. Der Einschub konzentriert sich auf die MPN-LAMP-Messungen bei niedrigeren Impfstufen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| 1 | 2 | 3 | 4 | 5 | 6 | |
| Ein | n=1 | n=1 | n=1 | n=1 | n=1 | n=1 |
| B | 2 | 2 | 2 | 2 | 2 | 2 |
| C | 3 | 3 | 3 | 3 | 3 | 3 |
| D | 4 | 4 | 4 | 4 | 4 | 4 |
| E | 5 | 5 | 5 | 5 | 5 | 5 |
| F | 6 | 6 | 6 | 6 | 6 | 6 |
| G | 7 | 7 | 7 | 7 | 7 | 7 |
| H | 8 | 8 | 8 | 8 | 8 | 8 |
| 100 | 10-1 | 10-2 | 10-3 kg | 10-4 kg | 10-5 kg |
Tabelle 1: Schematische Darstellung von Replikaten und Verdünnungen in einem 48-Well-Block. Diese Abbildung veranschaulicht die Anordnung von Replikaten und seriellen 10-fachen Verdünnungen innerhalb des 48-Well-Blocks. Jede Spalte stellt einen spezifischen Verdünnungsfaktor dar, der von 100 bis 10-5 reicht, wobei technische Replikate für jede Verdünnung auf die acht Zeilen verteilt sind.
| Verdünnung | ||||||
| 10-5 kg | 10-4 kg | 10-3 kg | 10-2 | 10-1 | 100 | |
| MPN-LAMPE | 8 | 16 | 31 | 3 | 56 | 56 |
| MPN-Beschichtung | 4 | 11 | 32 | 55 | 56 | 56 |
| MPN-qPCR | 0 | 7 | 26 | 55 | 56 | 56 |
Tabelle 2: Anzahl der positiven Ereignisse. In der Tabelle wird die Anzahl der positiven Reaktionen (insgesamt mehr als 56 Wiederholungen) angezeigt. Die Ergebnisse werden in einer Tabelle angeordnet, in der die Spalten die Verdünnung darstellen und die Zeilen für die Methode repräsentativ sind.
Diskussion
Bedeutung des Protokolls
Salmonellen sind nach wie vor ein großes Problem für die Lebensmittelsicherheit, insbesondere bei Geflügelerzeugnissen, die häufig mit lebensmittelbedingten Krankheitsausbrüchen in Verbindung gebracht werden13,14. Als eine der Hauptursachen für bakterielle lebensmittelbedingte Krankheiten in den Vereinigten Staaten sind zuverlässige Methoden zum Nachweis von Salmonellen sowohl in frischen als auch in küchenfertigen Geflügelprodukten von entscheidender Bedeutung für die Gewährleistung der Lebensmittelsicherheit15. Die Fähigkeit, Salmonellen in geringen Mengen zu quantifizieren, ist entscheidend für die Identifizierung von Kontaminationen, die sonst unentdeckt bleiben könnten. Die Empfindlichkeit der Methode ist besonders wichtig für die Einhaltung gesetzlicher Vorschriften und die öffentliche Gesundheit, da selbst niedrige Dosen von Salmonellen zu Krankheiten führen können, insbesondere in gefährdeten Bevölkerungsgruppen.
Der MPN-LAMP-Assay verwendet das MDS-System, das derzeit von FSIS in MLG 4.15 verwendet wird, um die Prävalenz von Kontaminationenzu bestimmen 4. Der hier vorgestellte Assay stellt eine Erweiterung der aktuellen FSIS-Methode dar, um lebensfähige Salmonellen auch in niedrigen Konzentrationen zu quantifizieren und genauere Daten über den Grad der Kontamination zu liefern. Die Ergebnisse dieser Studie zeigten eine starke Korrelation zwischen der MPN-LAMP-Methode und den theoretischen Impfwerten mit einem R² von 0,933 (Abbildung 5). Dies deutet darauf hin, dass die Methode für die Quantifizierung von Salmonellen in Lebensmitteln zuverlässig ist, obwohl bei niedrigeren Impfmengen eine gewisse Unterschätzung auftrat.
Kritische Schritte
Das Protokoll für die Quantifizierung von Salmonellen erfordert Präzision bei der Probenvorbereitung, Inokulation und molekularen Analyse. Die richtige aseptische Technik, genaue Verdünnungen und die Einhaltung der Inkubationsbedingungen sind entscheidend für den zuverlässigen Nachweis und die Zählung von Salmonellen. Die Gewährleistung steriler Bedingungen während des Transfers von Geflügelproben und -medien ist unerlässlich, um eine Kontamination zu vermeiden.
Ebenso wichtig ist die Homogenisierung der Proben während des Magens, gefolgt von einer sorgfältigen Resuspension des Pellets nach der Zentrifugation, um die Bakterien von der Nahrungsmatrix zu trennen. Die richtige Handhabung während dieser Schritte stellt sicher, dass lebensfähige Zellen für die weitere Analyse zurückgewonnen werden. Präzises Pipettieren während des seriellen Verdünnungsprozesses für die MPN-Einrichtung in Verbindung mit konsistenten Inkubationsbedingungen ist entscheidend für das Erzielen reproduzierbarer Ergebnisse.
Schließlich erfordern molekulare Nachweismethoden wie die qPCR Präzision und Reproduzierbarkeit bei der DNA-Extraktion und dem qPCR-Zyklus, um falsch negative Ergebnisse zu vermeiden. Obwohl die DNA-Extraktion von Natur aus variabel ist, gewährleisten standardisierte Reagenzien und Verfahren die Wiederholbarkeit, indem sie die Interferenz von Inhibitoren minimieren und die DNA-Rückgewinnung verbessern. Diese Schritte sind von grundlegender Bedeutung, um das Vorhandensein von Salmonellen zu bestätigen und spezifische genetische Marker zu identifizieren, die dazu beitragen können, Einschränkungen bei herkömmlichen Plattier- und Anreicherungsmethoden zu überwinden.
Mögliche Fallstricke und Einschränkungen
Bakterielle Lebensfähigkeit
Eine der größten Herausforderungen bei der Implementierung eines wachstumsbasierten Assays wie der MPN-Plattierung ist die Lebensfähigkeit der Bakterien, und dies ist besonders relevant für komplexe küchenfertige Hühnerproben. Gefrorene, panierte und gefüllte Hühnerprodukte, wie z. B. Chicken Cordon bleu, sind besonders anfällig für Temperaturschwankungen und längere Lagerzeiten, die beide Salmonellenzellen belasten und den Erreger in wachstumsbasierten Assays schwerer nachweisbar machen können. Darüber hinaus führt das Vorhandensein von Nicht-Hühnerzutaten (z. B. Käse, Panade, Schinken, Gewürze, Butter) zu einer weiteren Komplexität in der Lebensmittelmatrix und kann das Wachstum von Salmonellen während der Anreicherungsphasen hemmen, was eine genaue Quantifizierung erschwert. Darüber hinaus müssen Salmonellen in kulturbasierten Methoden16 mit Hintergrundmikroben konkurrieren, was besonders für küchenfertige Proben mit mehreren Zutaten relevant ist, was die Mikrobiota im Vergleich zu rohem Huhn vielfältiger macht. Schließlich kann der Umgang mit und die Verarbeitung von Stressfaktoren, wie z. B. antimikrobiellen Mitteln, die Erholung von Salmonellen beeinflussen17.
Die bakterielle Lebensfähigkeit könnte sich auf die Leistung der in dieser Studie getesteten Methoden ausgewirkt haben. Alle Methoden unterschätzten die Salmonellenkonzentrationen bei niedrigen Konzentrationen und wiesen eine höhere Nachweisvariabilität bei niedrigeren Erregerkonzentrationen auf (Abbildung 4A,B). Die molekularbasierte MPN-LAMP-Methode übertraf jedoch die MPN-Beschichtung und die MPN-qPCR beim qualitativen Nachweis bei niedrigeren Inokulationsniveaus. Die Ergebnisse unterstreichen die Notwendigkeit einer Verfeinerung bei niedrigen Erregerkonzentrationen.
Heterogenität der Stichprobe
Eine weitere wichtige Einschränkung betrifft die Heterogenität der Lebensmittelproben, die zu einer Variabilität der Salmonellennachweisergebnisse führen kann. Das Protokoll wurde entwickelt, um 30 mL homogenisierter Probe zu dispergieren, was die Verteilung von 8 Replikaten als 3 mL Aliquots (24 mL insgesamt, 6 mL Überschuss) über den Block ermöglicht. Dieser konservative Ansatz (der zu einer zusätzlichen Stichprobe führt) berücksichtigt zwar potenzielle Verluste, führt jedoch zu einer Unterabtastung, da nicht das gesamte Volumen getestet wird.
Die Heterogenität der Lebensmittelprobe kann die Messgenauigkeit beim Transfer von Proben aus dem redispergierten Gemisch in den Block und bei der Durchführung von Verdünnungen beeinträchtigen18,19. Gemäß der Theory of Sampling (TOS) können grundlegende Probenahmefehler reduziert werden, indem die Probenmasse erhöht oder die Heterogenität der Zusammensetzung durch Partikelgrößenreduzierungreduziert wird 20. Während einige Studien jedoch darauf hindeuten, dass der Abbau der Matrix durch Magen die bakterielle Erholung von Oberflächenkontaminationen nicht signifikant beeinflusst, hat sich gezeigt, dass die Verringerung der Partikelgröße der Lebensmittelmatrix in einigen Fällen die bakterielle Erholung verringert 21,22,23. In dieser Studie wurde die Partikelgröße durch Magenbildung vor der Anreicherung reduziert; Während dies den Probenahmefehler verringern kann, indem die Heterogenität der Zusammensetzung der Proben verringert wird, können Magenbehandlung und Zentrifugation die bakterielle Erholung beeinträchtigen.
Schließlich können die heterogene Zusammensetzung und Morphologie des Lebensmittels sowie die Frage, ob die Kontamination intern oder auf der Oberfläche erfolgt, zu einer Variabilität bei der Wiederfindung von Krankheitserregern führen. Frühere Arbeiten18,22 haben gezeigt, dass eine starke bakterielle Bindung an Gewebeoberflächen die Wiederfindungsraten verringern kann. Studien, in denen Enzyme wie Trypsin, Kollagenase und Endopeptidase verwendet wurden, um Bakterien aus Fleisch- und Geflügelmatrizen24,25 und Pektinase und Cellulase aus Blattgemüse19 freizusetzen, zeigen das Potenzial, die Salmonellenrückgewinnung aus Lebensmittelmatrizen zu verbessern.
Vorschläge zur Problembehandlung
Um Probleme zu vermeiden, stellen Sie sicher, dass alle Medien und Reagenzien frisch vorbereitet und steril sind. Abgelaufene oder unsachgemäß sterilisierte Materialien können zu einer Kontamination führen oder das Wachstum von Salmonellen beeinträchtigen. Wenn eine Kontamination festgestellt wird, stellen Sie sicher, dass die aseptische Technik während des gesamten Protokolls konsequent befolgt wurde, insbesondere während der Inokulation, Verdünnung, Aliquotierung und Plattierung der Probe.
Wenn die Koloniezahlen niedriger als erwartet sind, kann die Überprüfung der Genauigkeit von seriellen Verdünnungen und Inokulumkonzentrationen helfen, Fehler zu identifizieren. Die Inkubationsbedingungen, einschließlich Temperatur, Zeit und Bewegung, müssen sorgfältig überwacht werden, um ein optimales Salmonellenwachstum zu unterstützen. In Fällen von geringem oder keinem Wachstum kann eine unzureichende Magenbildung oder ein Auslaufen aus den Beuteln zu einer ungleichmäßigen Verteilung der Bakterien beitragen.
Wenn der molekulare Nachweis zu widersprüchlichen Ergebnissen führt, sind die Überprüfung der DNA-Extraktionsverfahren und die Überprüfung der Qualität von Primern und Sonden wichtige Schritte. Außerdem kann die Variabilität zwischen den Lebensmittelmatrizen ein unterschiedliches Maß an Hemmung aufweisen, das mit Hilfe der internen Amplifikationskontrolle (IAC) überwacht werden kann. Die richtige Lagerung und Handhabung von Extraktionsreagenzien ist entscheidend, um falsch negative Ergebnisse aufgrund von Degradation oder Kontamination zu vermeiden. Effizientere DNA-Extraktionsmethoden können ebenfalls gerechtfertigt sein.
Durch die Einbeziehung von Behandlungen wie dem GentleMACS-System26, das mechanische, thermische und enzymatische Maßnahmen einsetzt, kann die Bakterienrückgewinnung aus Lebensmitteln weiter optimiert werden. Dieses System kann die Heterogenität der Lebensmittelmatrix reduzieren, was es einfacher macht, gleichmäßige Proben in den Block zu überführen und genaue serielle Verdünnungen durchzuführen.
Mögliche Verfeinerung und Erweiterung
Es gibt mehrere Möglichkeiten, dieses Protokoll zu verfeinern und zu erweitern. Die Erforschung alternativer Anreicherungsmedienformulierungen könnte die Gewinnung von gestressten oder subletal geschädigten Salmonellenzellen verbessern, insbesondere aus komplexen Lebensmittelmatrizen wie Chicken Cordon bleu. Während selektiver Agar häufig verwendet wird, um Salmonellen zu bekämpfen, können Zellen, die aufgrund von Verarbeitungs- und Lagerbedingungen verletzt oder gestresst sind, zu falsch negativen Ergebnissen auf solchen Medien führen. Um diese Herausforderung zu meistern, könnte die Optimierung des Gleichgewichts zwischen Selektionsdruck und mikrobieller Rückgewinnung die Detektion von Salmonella-Serovaren verbessern, die unter Standardanreicherungsbedingungen weniger wettbewerbsfähig sind. Diese Verfeinerung könnte die allgemeine Sensitivität des Assays verbessern, insbesondere bei anspruchsvollen Lebensmitteln, bei denen mikrobieller Stress häufig vorkommt. Eine Verkürzung der Inkubationszeiten könnte die Zeit bis zu den Ergebnissen verkürzen, obwohl dies die Empfindlichkeit verringern und die Detektion einschränken kann. Zukünftige Arbeiten sollten Fortschritte bei Anreicherungsmedien untersuchen, um eine schnelle bakterielle Replikation zu unterstützen und eine genaue Quantifizierung zu gewährleisten und gleichzeitig die Anforderungen der Interessengruppen zu erfüllen.
Darüber hinaus könnte die Untersuchung unterschiedlicher Inkubationstemperaturen und -dauern für den MPN-Assay die Salmonellengewinnung aus verschiedenen Geflügelprodukten optimieren. Dies könnte die Anwendbarkeit des Protokolls auf andere lebensmittelbedingte Krankheitserreger erweitern, die unterschiedliche Wachstumsbedingungen erfordern, und damit seinen Nutzen bei Lebensmittelsicherheitstests erweitern. Die Automatisierung könnte die Skalierbarkeit dieses Protokolls durch die Integration von robotergestützten Liquid Handlern für Aliquotierungs- und Verdünnungsschritte verbessern. Zentrifugen mit kontinuierlichem Durchfluss oder Zentrifugensysteme mit hoher Kapazität können ebenfalls Einschränkungen bei der Chargenverarbeitung beheben und so Anwendungen mit hohem Durchsatz in industriellen Umgebungen ermöglichen.
Die Erweiterung des molekularen Nachweisaspekts um die Multiplex-PCR könnte den gleichzeitigen Nachweis mehrerer Krankheitserreger ermöglichen und den Durchsatz in Überwachungs- und Testumgebungen verbessern. Darüber hinaus könnte die Einbeziehung von Next-Generation-Sequencing (NGS) wertvolle Serotyp- und Virulenzinformationen direkt aus MPN-Anreicherungskulturen liefern, was epidemiologische Untersuchungen und die Verfolgung der Quelle erleichtert.
Schließlich kann die Quantifizierungs-/Nachweisgrenze erweitert werden, indem die Anzahl der im MPN verwendeten Proben erhöht wird. Wenn beispielsweise das Gewicht der Fleischprobe von 25 g auf 325 g erhöht wird, kann die Nachweisgrenze mit Hilfe der linearen Regressionsgleichung auf 4 KBE/g extrapoliert werden. Unter der Annahme des gleichen relativen Konfidenzintervalls von 95 % liegt die praktische untere Bestimmungsgrenze jedoch bei >30 KBE/g.
Schlussfolgerung
Die Entwicklung des MPN-LAMP-Assays in dieser Studie bietet ein wertvolles und praktisches Werkzeug zur Quantifizierung der Salmonellenkontamination in Geflügelprodukten. Mit einer starken Korrelation zu den theoretischen Impfmengen (R² = 0,933) weist die Methode Salmonellen zuverlässig in niedrigen Konzentrationen nach, was für die Lebensmittelsicherheit und die Einhaltung gesetzlicher Vorschriften von entscheidender Bedeutung ist. Diese Fähigkeit ist besonders wichtig bei der Erkennung von Kontaminationen, die sonst unbemerkt bleiben könnten, und ermöglicht es den Produzenten, potenzielle Risiken proaktiv anzugehen. Der MPN-LAMP-Assay bietet eine quantitative Alternative zu herkömmlichen prävalenzbasierten Methoden, da er ein gewisses Maß an Salmonellenkontamination in einer Vielzahl von Geflügelprodukten bietet, einschließlich komplexerer küchenfertiger Artikel.
Eine der wichtigsten Stärken dieser Methode liegt in ihrer Anpassungsfähigkeit an verschiedene Lebensmittelmatrizen, insbesondere an gefrorene und verarbeitete Produkte wie Hähnchen-Cordon Bleu, bei denen traditionelle Methoden aufgrund des Vorhandenseins zusätzlicher Zutaten wie Panade und Käse Schwierigkeiten haben könnten. Die Integration molekularer Techniken wie LAMP und qPCR in den MPN-Assay gewährleistet einen umfassenden Nachweisansatz und überwindet die Herausforderungen, die durch Hintergrundmikroben und Verarbeitungsbedingungen entstehen. Diese molekularen Methoden erhöhen die Empfindlichkeit des Protokolls und machen es zu einem vielseitigen Instrument für routinemäßige Lebensmittelsicherheitstests und die Überwachung von Kontaminationen, mit dem Potenzial, das Risiko von Salmonellen-bedingten Ausbrüchen zu verringern.
Obwohl keine einzelne Nachweismethode ohne Einschränkungen ist, zeigt der MPN-LAMP-Assay eine konsistente Leistung über eine Reihe von Inokulumkonzentrationen und Lebensmittelmatrizen. Die Variabilität bei niedrigeren Impfmengen kann durch zukünftige Verfeinerungen behoben werden, wie z. B. die Optimierung der Probenvorbereitung und der Medienformulierungen zur Maximierung der Wiederfindung, aber dies beeinträchtigt nicht die allgemeine Robustheit des Protokolls. Durch den präzisen Nachweis von Salmonellenzellen , auch unter schwierigen Bedingungen, gibt die MPN-LAMP-Methode der Lebensmittelindustrie ein unschätzbares Werkzeug zum Schutz der öffentlichen Gesundheit an die Hand.
Mit Blick auf die Zukunft gibt es Raum für weitere Verbesserungen, wie z. B. die Erweiterung des Assays um mehr Salmonella-Serovare oder die Einbeziehung von Next-Generation-Sequencing für eine bessere Charakterisierung der Krankheitserreger. Nichtsdestotrotz stellt dieses Protokoll bereits einen bedeutenden Fortschritt beim Nachweis und der Quantifizierung von Salmonellen dar und bietet eine praktische, effiziente und zuverlässige Methode, die den wachsenden Anforderungen an die Lebensmittelsicherheitsprüfung gerecht wird.
Offenlegungen
Alle Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Forschung wurde unterstützt durch das U.S. Department of Agriculture, Agricultural Research Service (USDA-ARS), National Program 108, Current Research Information System Nummern 8072-42000-093-000-D und 8072-42000-094-000-D. Die Erwähnung von Handelsnamen oder Handelsprodukten in diesem Artikel dient ausschließlich dem Zweck, spezifische Informationen bereitzustellen, und impliziert keine Empfehlung oder Billigung durch das US-Landwirtschaftsministerium. USDA ist ein Anbieter und Arbeitgeber für Chancengleichheit.
Materialien
| Name | Company | Catalog Number | Comments |
| 48 deep well block 4.6ml | Fisher Scientific International, Inc | NC1964628 | |
| Agar - Solidifying Agent (Difco) | Becton, Dickinson and Company (BD) | 281230 | |
| Analytical Balance | Mettler Toledo | JL602-G/L | Equipment |
| Analytical Balance | Mettler Toledo | AB54-S | Equipment |
| Autoclave - Amsco Lab250, Laboratory Steam Sterilizer | Steris plc | LV-250 | Equipment |
| Biological Safety Cabinet, Type A2, Purifier Logic+ | Labconco Corporation | 302411101 | Equipment |
| Brain Heart Infusion (BHI) Broth | Becton, Dickinson and Company (BD) | 237500 | |
| Buffered Peptone Water | Bio-Rad Laboratories Inc. | 3564684 | |
| Cell Spreader - L-shaped | VWR | 76208-438 | |
| Centrifuge Microcentrifuge 5424 | Eppendorf | 5424 | Equipment |
| Centrifuge, Avanti J-25 | Beckman Coulter, Inc. | Equipment | |
| DNA Extraction - PreMan Ultra Sample Preparation Reagent | Thermo Fisher Scientific Inc. | 4318930 | |
| Ground Chicken | Local retailers | ||
| IAC forward primer: 5'-GGCGCGCCTAACACATCT-3' | Integrated DNA Technologies | ||
| IAC probe: 5'-TTACAACGGGAGAAGACAATGC CACCA-3' labeled with 5' TAMRA/3' BHQ-2 | Biosearch Technologies | ||
| IAC reverse primer: 5'-TGGAAGCAATGCCAAATGTGTA-3' | Integrated DNA Technologies | ||
| Incubator - Inova 4230 incubator shaker | New Brunswick Scientific | 4230 | Equipment |
| Inoculating Loop - Combi Loop 10µL and 1µL | Fisher Scientific International, Inc | 22-363-602 | |
| invA forward primer: 5'-GTTGAGGATGTTATTCGCAAAG G-3' | Integrated DNA Technologies | ||
| invA probe: 5'-CCGTCAGACCTCTGGCAGTAC CTTCCTC-3' labeled with 5' Cal Fluor Orange 560/3' BHQ-1 | Biosearch Technologies | ||
| invA reverse primer: 5'-GGAGGCTTCCGGGTCAAG-3' | Integrated DNA Technologies | ||
| Irradiation Treatment | Texas A&M Agrilife Research National Center for Electron Beam Research | Service | |
| Luria Bertani (LB) Broth | Becton, Dickinson and Company (BD) | 244620 | |
| Manual pipette Pipet-Lite LTS Pipette L-1000XLS+ | Mettler Toledo | 17014382 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-100XLS+ | Mettler Toledo | 17014384 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-10XLS+ | Mettler Toledo | 17014388 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-200XLS+ | Mettler Toledo | 17014391 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-20XLS+ | Mettler Toledo | 17014392 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-200XLS+ | Mettler Toledo | 17013805 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-20XLS+ | Mettler Toledo | 17013803 | Equipment |
| Media Storage Bottle -PYREX 1L Square Glass Bottle, with GL45 Screw Cap | Corning Inc. | 1396-1L | Equipment |
| Media Storage Bottle -PYREX 2L Round Wide Mouth Bottle, with GLS80 Screw Cap | Corning Inc. | 1397-2L | Equipment |
| Microtiter plate, 96 well plate, flat bottom, polystyrene, 0.34cm2, sterile, 108/cs | MilliporeSigma | Z707902 | |
| Mixer - Vortex Genie 2 | Scientific Industries Inc. | SI-0236 | Equipment |
| Molecular Detection Assay 2-Salmonella kit | Neogen | MDA2SAL96 | |
| Molecular Detection Instrument | Neogen | MDS100 | Equipment |
| Motorized pipette controller, PIPETBOY2 | INTEGRA Biosciences Corp. | 155019 | Equipment |
| PCR Mastermix 2× TaqMan Gene Expression | Thermo Fisher Scientific Inc. | 4369542 | |
| Petri Dish Rotator - bioWORLD Inoculation Turntable | Fisher Scientific International, Inc | 3489E20 | Equipment |
| Petri Dishes with Clear Lid (100 mm x 15mm) | Fisher Scientific International, Inc | FB0875713 | |
| Pipette Tips GP LTS 1000µL S 768A/8 | Mettler Toledo | 30389273 | |
| Pipette Tips GP LTS 20µL 960A/10 | Mettler Toledo | 30389270 | |
| Pipette Tips GP LTS 200µL F 960A/10 | Mettler Toledo | 30389276 | |
| Ready to cook chicken products | Local retailers | ||
| Reagent Reservoir, 25 mL sterile reservoir used with multichannel pipettors | Thermo Fisher Scientific Inc. | 8093-11 | |
| Realtime PCR - 7500 Real-Time PCR system | (Applied Biosystems, Foster City, CA) | 2750036476 | Equipment |
| Serological Pipettes, Nunc Serological Pipettes (10 mL) | Thermo Fisher Scientific Inc. | 170356N | |
| Serological Pipettes, Nunc Serological Pipettes (2 mL) | Thermo Fisher Scientific Inc. | 170372N | |
| Serological Pipettes, Nunc Serological Pipettes (25 mL) | Thermo Fisher Scientific Inc. | 170357N | |
| Serological Pipettes, Nunc Serological Pipettes (50 mL) | Thermo Fisher Scientific Inc. | 170376N | |
| Spreader - Fisherbrand L-Shaped Cell Spreaders | Fisher Scientific International, Inc | 14-665-230 | |
| Stomacher bag, Nasco Whirl-Pak Write-On Homogenizer Blender Filter Bags | Thermo Fisher Scientific Inc. | 01-812 | |
| Stomacher 80 Biomaster Lab Blender | Seward | 30010019 | Equipment |
| Thermocycler (GeneAmp PCR system 9700) | Applied Biosystems | 535028293 | Equipment |
| Water Filtration - Elga Veolia Purelab Flex | Elga LabWater | PF2XXXXM1-US | Equipment |
| Whirlpak bags 1.63L | VWR | 11216-777 |
Referenzen
- Batz, M., Hoffmann, S., Morris, J. G. Disease-outcome trees, eq-5d scores, and estimated annual losses of quality-adjusted life years (qalys) for 14 foodborne pathogens in the united states. Foodborne Pathogens and Disease. 11 (5), 395-402 (2014).
- . The Grand Challenge: Salmonella Available from: https://tellus.ars.usda.gov/stories/articles/the-grand-challenge-salmonella (2024)
- National Advisory Committee on Microbiological Criteria in Foods (NACMCF). Response to questions posed by the food safety and inspection service: Enhancing Salmonella control in poultry products. J Food Prot. 82 (4), 645-668 (2019).
- Food Safety and Inspection Service. . 4.15 Isolation and identification of Salmonella from meat, poultry, pasteurized egg, siluriformes (Fish) products and carcass and environmental sponges. , (2024).
- Irwin, P., Reed, S., Brewster, J., Nguyen, L., He, Y. P. Non-stochastic sampling error in quantal analyses for campylobacter species on poultry products. Analytical and Bioanalytical Chemistry. 405 (7), 2353-2369 (2013).
- Irwin, P., Tu, S., Damert, W., Phillips, J. A modified gauss-newton algorithm and ninety-six well micro-technique for calculating mpn using excel spreadsheets. Journal of Rapid Methods & Automation in Microbiology. 8 (3), 171-191 (2000).
- Ravishankar, S., Ahmed, E. Y., Carlstrom, C. Food microbiology: A laboratory manual. Food Microbiology. 21, 489 (2004).
- Chen, C. Y., Nace, G. W., Irwin, P. L. A 6 x 6 drop plate method for simultaneous colony counting and mpn enumeration of campylobacter jejuni, listeria monocytogenes, and escherichia coli. J Microbiol Methods. 55 (2), 475-479 (2003).
- Suo, B., He, Y., Tu, S. I., Shi, X. A multiplex real-time polymerase chain reaction for simultaneous detection of salmonella spp., escherichia coli o157, and listeria monocytogenes in meat products. Foodborne Pathogens and Disease. 7 (6), 619-628 (2010).
- Jarvis, B., Wilrich, C., Wilrich, P. T. Reconsideration of the derivation of most probable numbers, their standard deviations, confidence bounds and rarity values. J Appl Microbiol. 109 (5), 1660-1667 (2010).
- Stevens, R., Poppe, K. Validation of clinical prediction models: What does the "calibration slope" really measure. Journal of Clinical Epidemiology. 118, (2019).
- Miller, M. E., Hui, S. L., Tierney, W. M. Validation techniques for logistic regression models. Statistics in Medicine. 10 (8), 1213-1226 (1991).
- Galán-Relaño, &. #. 1. 9. 3. ;., et al. Salmonella and salmonellosis: An update on public health implications and control strategies. Animals. 13 (23), 3666 (2023).
- Gorski, L., et al. Growth assessment of salmonella enterica multi-serovar populations in poultry rinsates with commonly used enrichment and plating media. Food Microbiology. 119, 104431 (2024).
- Schmidt, J. W., et al. Evaluation of methods for identifying poultry wing rinses with salmonella concentrations greater than or equal to 10 cfu/ml. J Food Prot. 87 (11), 100362 (2024).
- Gorski, A., Liang, L. S. Effect of enrichment medium on real-time detection of salmonella enterica from lettuce and tomato enrichment cultures. Journal of Food Protection. 73 (6), 1047-1056 (2010).
- Guillén, S., Nadal, L., Álvarez, I., Mañas, P., Cebrián, G. Impact of the resistance responses to stress conditions encountered in food and food processing environments on the virulence and growth fitness of non-typhoidal salmonellae. Foods. 10 (3), 617 (2021).
- Rohde, A., Hammerl, J. A., Appel, B., Dieckmann, R., Al Dahouk, S. Sampling and homogenization strategies significantly influence the detection of foodborne pathogens in meat. BioMed Research International. 2015, (2015).
- Wang, D., Wang, Z., He, F., Kinchla, A. J., Nugen, S. R. Enzymatic digestion for improved bacteria separation from leafy green vegetables. Journal of Food Protection. 79 (8), 1378-1386 (2016).
- Pitard, F. F. . Theory of sampling and sampling practice. , (2019).
- Sharpe, A. . in Detecting pathogens in food. , 52-68 (2003).
- Hannah, J., et al. Effect of stomaching on numbers of bacteria recovered from chicken skin. Poultry Science. 90 (2), 491-493 (2011).
- Mcmeekin, T., Thomas, C. Retention of bacteria on chicken skin after immersion in bacterial suspensions. Journal of Applied Bacteriology. 45 (3), 383-387 (1978).
- Rodrigues-Szulc, U., Ventoura, G., Mackey, B., Payne, M. Rapid physicochemical detachment, separation and concentration of bacteria from beef surfaces. Journal of Applied Bacteriology. 80 (6), 673-681 (1996).
- Vibbert, H. B., et al. Accelerating sample preparation through enzyme-assisted microfiltration of salmonella in chicken extract. Biotechnol Prog. 31 (6), 1551-1562 (2015).
- Armstrong, C. M., et al. Use of a commercial tissue dissociation system to detect salmonella-contaminated poultry products. Analytical and Bioanalytical Chemistry. 416 (3), 621-626 (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten