Method Article
Dosage modifié du nombre le plus probable pour quantifier la présence de Salmonella dans les produits de poulet crus et prêts à cuire
Dans cet article
Résumé
Quantifier avec précision les salmonelles présentes à de faibles niveaux chez les volailles est un défi industriel et réglementaire actuel. Ce protocole décrit un dosage de la NPP qui permet de quantifier la présence de Salmonella dans les produits de volaille crus et prêts à cuire. Cette méthode est rapide, sensible et s’aligne sur les directives du FSIS, améliorant la sécurité alimentaire et soutenant les efforts de santé publique.
Résumé
La salmonelle est l’une des principales causes de maladies d’origine alimentaire aux États-Unis, en particulier dans les produits à base de volaille. Les méthodes traditionnelles de détection de Salmonella se concentrent sur la prévalence plutôt que sur la quantification, ce qui limite leur utilité dans l’évaluation des niveaux de contamination et des risques. Cette étude présente un nouveau test du nombre le plus probable (NPP) conçu pour quantifier Salmonella dans les produits de volaille prêts à cuire, tels que le cordon bleu de poulet. La méthode consiste à laver l’échantillon de volaille, à concentrer le rinçage par centrifugation et à le diluer en série dans un bloc de 48 puits. Le test NPP est intégré à la méthode d’amplification isotherme médiée en boucle (LAMP) afin de fournir une quantification sensible, précise et rapide de la contamination par Salmonella dans le même délai que les protocoles existants du Food Safety and Inspection Service (FSIS). Les résultats montrent une forte corrélation linéaire entre les mesures MPN-LAMP et les niveaux théoriques d’inoculation (R² = 0,933). Cependant, la variabilité à des concentrations plus faibles met en évidence les défis liés à la détection précise de Salmonella à ces niveaux, la limite inférieure pratique de détection étant estimée à environ 300 UFC/g. Parmi les améliorations qui pourraient être apportées pour améliorer l’applicabilité du protocole, mentionnons l’augmentation de la quantité échantillonnée afin d’améliorer davantage la limite de détection, l’optimisation des formulations de milieux d’enrichissement et l’élargissement de la détection moléculaire pour cibler plusieurs sérovars de Salmonella . Dans l’ensemble, cette étude présente un outil pratique pour l’industrie alimentaire, permettant une quantification fiable de la contamination par Salmonella dans les produits de volaille, contribuant ainsi à l’amélioration de la sécurité alimentaire et de la santé publique.
Introduction
En tant que principale cause de maladies d’origine alimentaire, d’hospitalisations et de décès aux États-Unis, Salmonella a un impact significatif sur la santé publique et l’économie. Le fardeau économique estimé de l’agent pathogène en 2013 seulement était de 3,67 milliards de dollars1. Bien que les récentes initiatives réglementaires visent à réduire la salmonellose de 25 % d’ici 20302, les stratégies actuelles de détection et d’atténuation présentent des lacunes évidentes, notamment en ce qui concerne l’harmonisation de la surveillance des usines de transformation avec les résultats en matière de santé publique3 .
Les produits de volaille congelés prêts à cuire, qui ont été impliqués dans plusieurs éclosions de Salmonella , sont une préoccupation importante pour la santé publique. En réponse, le Food Safety and Inspection Service (FSIS) a classé Salmonella comme adultérant dans ces produits. À l’heure actuelle, le FSIS Microbiology Laboratory Guidebook (MLG) 4.15 se concentre uniquement sur la détermination de la prévalence de Salmonella dans les produits de volaille4. En vertu de cette ligne directrice, les échantillons prélevés sont enrichis pendant 18 à 24 heures, puis analysés à l’aide du système de détection moléculaire (SMD), qui identifie la présence ou l’absence de Salmonella , mais ne donne pas d’aperçu du niveau de contamination. Bien que cette approche soit utile pour détecter la présence d’agents pathogènes, elle ne fournit pas d’informations quantitatives qui pourraient aider les transformateurs d’aliments à évaluer les risques de contamination avec plus de précision et à prendre des mesures correctives ciblées.
Dans cette étude, nous avons développé une méthode pour augmenter la détection de la prévalence à la quantification des agents pathogènes microbiens. Il a été conçu pour s’intégrer sans heurts dans les processus existants de détection de Salmonella dans les produits de volaille avec une perturbation minimale des protocoles actuels du FSIS. Au lieu de simplement enrichir l’échantillon en vrac, la méthode commence par laver les produits de volaille à l’aide de milieux conformes aux méthodes actuelles du FSIS. Le rinçage est ensuite distribué dans la première colonne d’un bloc de puits de 48 profondeurs. Des dilutions en série sont effectuées sur les cinq colonnes restantes, et le bloc est incubé pendant 18 à 24 h, conformément au protocole MLG 4.15. Après l’incubation, les puits sont analysés pour détecter la présence de Salmonella, et les résultats sont utilisés pour calculer le nombre le plus probable (NPP)5,6. Cette approche permet de quantifier la contamination dans le même laps de temps que le processus actuel du FSIS, ce qui en fait une option pratique pour l’industrie et la réglementation. La figure 1 représente un schéma fonctionnel résumant le dosage du NPP modifié. La figure comprend des photographies prises à des étapes précises, le bloc de 48 puits utilisé pour la dilution et la croissance des répétitions, et les trois techniques utilisées comme points de référence pour évaluer le nombre le plus probable de Salmonella présent dans le poulet haché. Dans la première phase de cette étude, nous avons utilisé du poulet haché irradié pour minimiser l’impact de la microflore de fond et l’incertitude des mesures par rapport à l’inoculum vérifié avant d’appliquer le protocole à des échantillons de poulet non irradiés.
Protocole
REMARQUE : Tous les travaux associés à ce protocole doivent être effectués dans un laboratoire de niveau de biosécurité 2 (BSL-2). S’il y a lieu, ce protocole doit être mis en œuvre au sein d’une enceinte de sécurité biologique (ESB) afin de maintenir des conditions aseptiques et de réduire au minimum le risque de contamination de l’échantillon ou d’exposition de l’opérateur à des agents pathogènes microbiens. Lorsque vous transférez des échantillons à l’extérieur de l’ESB, utilisez des contenants scellés pour maintenir l’intégrité de l’échantillon et prévenir les déversements en cas de chute accidentelle. De préférence, des composants jetables doivent être utilisés tout au long de la procédure pour atténuer le risque de contamination croisée. Dans les cas où les produits jetables ne sont pas réalisables, assurez-vous que tout l’équipement et le matériel sont stériles avant de l’utiliser. Une bonne gestion des déchets est cruciale ; Tous les composants jetables usagés doivent être jetés comme déchets biologiques. Autoclavez les matériaux réutilisables avant de les réutiliser pour assurer une stérilisation et un confinement appropriés des matériaux potentiellement dangereux. Le respect de ces précautions permet non seulement de protéger l’intégrité de l’échantillon, mais aussi de minimiser le risque d’exposition de l’opérateur à des agents pathogènes microbiens.
1. Préparation des échantillons de viande
- Acquisition et transformation d’échantillons de viande
- Viande fraîche
- Achetez du poulet haché au rayon des viandes fraîches des détaillants locaux. Transférez tous les échantillons dans un stockage à 4 °C et traitez-les dans les 24 heures suivant leur réception. Divisez aseptiquement la viande en échantillons de 25 g.
- Scellez sous vide et irradiez l’échantillon. Ici, le Texas A&M AgriLife National Center for Electron Beam Research a irradié de la viande soumise à une dose de ~25 kGy.
REMARQUE : Bien que l’irradiation ait été utilisée comme mesure de contrôle dans cette étude pour assurer l’élimination de la microflore de fond, elle n’est pas une condition préalable au protocole dans des applications pratiques, comme le montrent les produits prêts à cuire non irradiés utilisés dans la section suivante. Sur le terrain, d’autres méthodes telles que les milieux sélectifs ou la spécificité des diagnostics moléculaires peuvent traiter les interférences potentielles provenant de micro-organismes non cibles.
- Produits de poulet prêts à cuisiner
- Acquérez des produits de poulet prêts à cuisiner dans la section des aliments surgelés des détaillants locaux. Diviser aseptiquement en échantillons de 25 g.
- Prélevez des échantillons au centre de chaque morceau pour vous assurer que tous les ingrédients (p. ex., la panure et le fromage) sont inclus.
- Viande fraîche
- Préparation des milieux
- Préparez de l’eau peptonée tamponnée (EPB) en dissolvant 25 g de poudre d’EPP dans 1 L de nanopure H2O.
- Préparez des plaques de perfusion cerveau-cœur (BHI). Pour ce faire, dissolvez 37 g de poudre de BHI dans 1 L deH2O nanopure et ajoutez 15 g de gélose dans la solution de BHI. Stériliser tous les milieux en autoclave à 121 °C pendant 15 min. Verser 20 à 25 ml de média dans des boîtes de Pétri avec couvercle transparent (100 mm x 15 mm).
REMARQUE : Il est préférable de verser les plaques dans une enceinte de sécurité biologique pour maintenir des conditions aseptiques.
2. Culture cellulaire
- Préparez la culture initiale en striant le sérovar Typhimurium ATCC 14028 de Salmonella enterica sur une plaque de gélose BHI et incubez à 37 °C pendant la nuit.
- Préparez des cultures de nuit en inoculant 25 ml de bouillon BHI avec une colonie de Salmonella fraîchement cultivée. Cultivez des cultures en aérobie pendant la nuit à 37 °C en secouant à 100 tr/min.
3. Inoculation d’échantillons de volaille
- Dilution de la culture et placage
- Préparer une série de dilutions de 10 fois de la culture de nuit dans l’EPA pour obtenir des concentrations finales d’environ 1 x 108 à 1 x 101 UFC/mL. Supposons que la concentration de la culture de nuit de Salmonella est de 1 x 109 UFC/mL.
- Transvaser 0,5 mL de la culture de nuit dans 4,5 mL d’EPT, mélanger, puis transférer 0,5 mL de dilution dans 4,5 mL d’EPT pour chaque dilution supplémentaire.
- Étaler 10 μL de la dilution de 1 x 103 UFC/mL sur une plaque de gélose BHI en trois exemplaires pour le dénombrement cellulaire afin de calculer la concentration de culture pendant la nuit.
- Inoculation d’échantillons de viande
- Transférez aseptiquement 25 g de poulet haché irradié dans un sac stomacher stérile en deux exemplaires. Le sac mesure 7,5 x 12 pouces et est étiqueté comme 1,63 L. Le sac contient une cloison filtrante d’un diamètre de trou de 330 μm, et il y en a 285 par cm carré.
- inoculer chaque échantillon avec 1 mL de la dilution de concentration cible. Par exemple, ajoutez 1 mL de culture à partir de la dilution de 1 x 10³ UFC/mL pour obtenir un niveau de contamination d’environ 1 000 cellules/25 g de poulet. Répartissez délicatement l’inoculum liquide sur la surface des échantillons de poulet à l’aide d’un épandeur de cellules stériles et laissez-le reposer pendant 1 h à 4 °C.
- Préparez des échantillons témoins négatifs en ajoutant 1 mL de DMP stérile.
4. Traitement des échantillons
- Ajouter 225 mL d’EPT à chaque échantillon. Le rapport entre le volume et le support a été choisi pour s’aligner sur la MLG 4.154 du FSIS.
- Homogénéiser les échantillons à l’aide du Stomacher7 à vitesse normale et d’une durée de 120 s.
- Centrifugation et remise en suspension
- Retirer délicatement le liquide du côté filtré du sac à l’aide d’une pipette de 50 ml. Divisez le liquide en deux flacons de centrifugation stériles.
- Équilibrez les bouteilles avec du BPW stérile pour assurer des poids égaux. Centrifuger les échantillons à 10 000 x g pendant 10 min. Jetez le surnageant. Remettre la pastille en suspension dans 3 mL de bouillon d’EPT à l’aide d’une spatule stérile.
- Ajouter 27 ml de bouillon BPW et bien mélanger en remuant à l’aide d’une spatule. Combinez le contenu des deux flacons de centrifugation en un seul flacon pour chaque échantillon.
5. Configuration du bloc MPN
REMARQUE : Le tableau 1 présente un schéma des dilutions dans un bloc de 48 puits.
- Ajouter 3 mL de l’échantillon remis en suspension dans chaque puits de la colonne 1 du bloc de 48 puits (8 répétitions).
- Préparez une série de dilutions 10 fois sur les colonnes 1 à 6 à l’intérieur du bloc à l’aide d’une pipette à huit canaux.
- Ajouter 0,3 mL d’échantillon dans 2,7 mL de pipette BPW pour mélanger. Répétez l’opération pour chaque dilution. Incuber les blocs pendant la nuit (~18 h) à 37 °C avec agitation à ~100 tr/min.
6. Placage et dénombrement
- Énumération modifiée des plaques de chute
- Plaque de 7 μL d’échantillon cultivé pendant la nuit de chaque dilution dans une grille de 4 x 6 sur une plaque de gélose à l’aide d’une pipette multicanaux (figure 2). L’utilisation d’une grille 4 x 6 sur deux plaques permet de mieux traiter 8 échantillons, par opposition à la grille typique de 6 x 6 gouttelettes8.
- Laissez les plaques sécher à l’air libre pendant 10 minutes avant l’incubation. Incuber les plaques de gélose pendant une nuit (~18-24 h) à 37 °C. Après l’incubation, comptez le nombre de colonies sur chaque plaque.
7. Détection de Salmonella par qPCR
- Extraction d’ADN à l’aide d’un kit commercial
- Mélangez les cultures dans le bloc de 48 puits en pipetant plusieurs fois de haut en bas. Pipeter 200 μL de chaque culture dans une plaque de PCR à 96 puits.
- Scellez puis centrifugez la plaque à 6 600 x g pendant 10 min. Retirer le surnageant et ajouter 20 μL du réactif du kit à la pastille.
- Remettez le granulé en suspension en le pipetant de haut en bas. Fermez et chauffez la plaque à 99 °C pendant 10 min, puis refroidissez à 20 °C.
- Centrifuger à nouveau à 6 600 x g pendant 10 min. Utilisez 2 μL de surnageant pour l’analyse qPCR.
- Configuration de la plaque
- Préparez le mélange réactionnel qPCR conformément au protocole9 établi comme suit : 10 μL de 2x Master Mix ; 0,4 μL de chaque amorce et sonde (solution de travail de 10 μM) : invA avant : 5'-GTTGAGGATGTTATTCGCAAAGG-3', invA arrière : 5'-GGAGGCTTCCGGGTCAAG-3', sonde invA : 5'-CCGTCAGACCTCTGGCAGTACCTTCCTC-3' marquée avec le colorant fluorènes Cal Fluor Orange 560 ; 0,2 μL de modèle de contrôle d’amplification interne (IAC) (6 x 104 copies/μL)9 ; 0,4 μL de chaque amorce et sonde IAC (10 μM) : IAC avant : 5'-GGCGCGCCTAACACATCT-3', IAC arrière : 5'-TGGAAGCAATGCCAAATGTGTA-3', Sonde IAC : 5'-TTACAACGGGAGAAGACAATGCCACCA-3' marquée avec le colorant TAMA. Ajustez le volume avec jdH2O à 20 μL au total.
- Effectuez une PCR en temps réel avec les conditions de cycle suivantes9 : 95 °C pendant 10 min (dénaturation initiale de l’ADN et activation de la polymérase à démarrage à chaud), 40 cycles de 95 °C pendant 15 s et 60 °C pendant 1 min, utilisez les paramètres Ct par défaut pour exporter les résultats à des fins d’analyse.
8. Détection à l’aide du test 3M MDS
- Suivez le protocole de la trousse de détection moléculaire de Salmonella . Mélangez les cultures dans le bloc de 48 puits en pipetant plusieurs fois de haut en bas. Pipeter 20 μL de chaque échantillon dans le tube de lyse fourni par le kit.
- Chauffer les échantillons à 100 °C pendant 15 min. La solution passera du rose au jaune. Incuber les échantillons pendant 10 min à température ambiante. La solution passera du jaune au rose.
- Transférez 20 μL de lysat dans un tube de réactif et chargez les tubes de réactif dans le support.
- Ajoutez le support à l’instrument MDS et configurez le logiciel pour communiquer les informations sur le kit et l’échantillon. L’instrument exige que chaque puits soit étiqueté avec le numéro de lot de l’essai et le nom de l’échantillon. Exécutez le logiciel MDS et exportez le rapport.
9. Analyse des données
- Classification des résultats positifs et négatifs.
- Pour un placage en goutte 4 x 6, évaluer les taches sur les plaques de gélose avec au moins 1 colonie comme positives et les taches sur les plaques de gélose sans croissance comme négatives.
- Pour la qPCR, évaluez les puits dont l’indice Ct est inférieur ou égal à 30 comme positifs et les puits dont l’indice Ct est supérieur à 30 comme négatifs.
- Pour les DMS, utilisez les résultats du système MDS, déclarés positifs ou négatifs.
- Calcul du MPN
- Analysez les points positifs et négatifs annotés à l’aide de la méthode SMPR (Simple Maximum Probability Resolution) décrite précédemment6 ou d’autres calculatrices MPN vérifiées. 10
Résultats
Viande irradiée
Dans l’analyse de régression, une pente de 1 indique que pour chaque unité d’augmentation de la variable indépendante (axe des x), la variable dépendante (axe des y) augmente d’exactement 1 unité. Cela suggère une relation proportionnelle entre les deux variables, ce qui signifie que la variation de la variable dépendante reflète la variation de la variable indépendante. Une ordonnée à l’origine de 0 signifie que lorsque la variable indépendante est 0, la variable dépendante est également 0. Cela suggère qu’il n’y a pas de décalage ou de biais fixe dans la relation entre les deux variables. Ensemble, une pente de 1 et une ordonnée à l’origine de 0 suggèrent une concordance entre les variables11,12. Ce scénario idéal n’indique aucune erreur ou biais systématique et serait le résultat attendu dans un système bien calibré où les mesures s’alignent parfaitement avec les prédictions.
La figure 3A présente le dosage MPN-LAMP (axe y) tracé par rapport à l’inoculum théorique vérifié par l’analyse sur plaque étalée dont il est question dans la note à l’étape 3.1.1. (axe des x). L’équation de la droite de régression, y = -72,36 + 0,161x, avec une valeur R² de 0,978, indique une forte corrélation linéaire entre les résultats MPN-LAMP et les niveaux de bactéries inoculées. La valeur élevée de R² suggère que le test MPN-LAMP est très fiable dans la prédiction quantitative de la contamination bactérienne. Cependant, la pente de 0,161 s’écarte considérablement de la pente idéale de 1, ce qui indique que la méthode MPN-LAMP sous-estime l’inoculum, ne prédisant que 16,1 % de la concentration bactérienne dans l’inoculum théorique. De plus, l’ordonnée à l’origine négative de -72,36 suggère un biais négatif à des concentrations plus faibles, s’écartant davantage de l’idéal.
La figure 3B illustre la méthode MPN-qPCR (axe des y) tracée par rapport à l’inoculum théorique (axe des x). La droite de régression, y = -46,84 + 0,0395x, avec une valeur R² de 0,978, démontre une forte relation linéaire entre le test MPN-qPCR et l’inoculum théorique. Bien que le R² élevé indique une corrélation robuste, la pente de 0,0395 est encore plus petite que celle des résultats MPN-LAMP, suggérant un plus grand degré de sous-estimation de la contamination bactérienne par MPN-qPCR. L’ordonnée à l’origine négative de -46,84 indique en outre un biais baissier potentiel à des concentrations plus faibles.
La figure 3C présente les résultats de placage MPN (axe y) tracés par rapport à l’inoculum théorique (axe x), avec une équation de régression de y = -330,4 + 0,3138x et une valeur R² de 0,944. Bien que cette valeur R² indique une forte relation linéaire, elle est légèrement inférieure à celles des résultats MPN-LAMP et MPN-qPCR, ce qui suggère une plus grande variabilité dans la quantification des colonies. La pente de 0,3138, bien que supérieure aux pentes observées dans les autres méthodes, indique tout de même une sous-estimation de la concentration bactérienne par rapport à l’inoculum théorique. De plus, l’intersection négative de -330,4 indique un biais considérable, en particulier à des concentrations plus faibles, ce qui peut indiquer des inexactitudes potentielles dans les techniques de placage ou la manipulation des échantillons. Cette légère réduction de R² et la variabilité observée pourraient indiquer que le procédé de placage traditionnel est plus variable que les méthodes moléculaires.
Méthodes de comparaison
Les trois méthodes (MPN-LAMP, MPN-plating et MPN-qPCR) ont montré une tendance à sous-estimer les niveaux bactériens par rapport à l’inoculation théorique. Cette sous-estimation peut être attribuée à des problèmes de récupération cellulaire, où Salmonella peut ne pas se détacher complètement de la surface du poulet ou des matières particulaires pendant le traitement, réduisant ainsi son transfert dans le rinçage ou le dosage. Comme la bactérie Salmonella a été inoculée directement sur le poulet, une partie des cellules bactériennes peut se lier fortement à la surface, ce qui limite la récupération lors de l’étape de rinçage. De plus, certaines cellules peuvent rester attachées aux particules dans le granulé après la centrifugation, ce qui réduit encore la récupération. Des approches telles que les traitements enzymatiques, le traitement mécanique ou les protocoles d’estomac optimisés pourraient améliorer le détachement bactérien. Ces stratégies sont abordées plus en détail dans la section de discussion, en mettant l’accent sur leur potentiel d’amélioration des taux de récupération
La figure 4A présente la récupération sur l’axe des y par rapport à trois méthodes MPN sur l’axe des x. Le pourcentage de récupération, calculé comme le rapport entre la valeur MPN et le niveau d’inoculation théorique, a donné les résultats suivants : la méthode MPN-LAMP avait une récupération moyenne de 15,19 % avec un écart-type de 10,04 % ; la méthode de placage MPN avait une récupération moyenne de 13,13 % avec un écart-type de 11,45 % ; et la méthode MPN-qPCR a eu une récupération moyenne de 6,67 % avec un écart-type de 3,19 %. Un test HSD de Tukey-Kramer n’a pas identifié de différence statistiquement significative dans la récupération entre les méthodes (P > 0,1969), ce qui suggère que les trois méthodes ont donné des résultats similaires en termes de récupération lors de la transformation du poulet inoculé à Salmonella.
Malgré un traitement identique entre les méthodes de séparation de Salmonella de la matrice de poulet, des variations dans la détection entre le MPN-LAMP, MPN-qPCR et le MPN-platage ont été observées. Cela suggère que des facteurs autres que la séparation physique des bactéries de l’échantillon peuvent avoir une incidence sur la détection. La présence d’inhibiteurs dans la matrice de l’échantillon pourrait interférer avec les méthodes moléculaires - qPCR et LAMP - affectant leur sensibilité et leurs performances globales. De plus, la viabilité bactérienne peut avoir eu une incidence sur la capacité de Salmonella à se développer sur des plaques de gélose solide (placage NPP) par rapport à un bouillon liquide (qPCR, LAMP), ce qui a entraîné une variation de la récupération entre les méthodes.
Pour explorer davantage la sensibilité des méthodes, nous avons d’abord examiné le nombre de réponses positives à chaque dilution pour la MPN-LAMP, MPN-qPCR et le MPN-plating dans sept essais indépendants (Tableau 2). À des dilutions plus faibles (1 x 10-5 et 1 x 10-4), le MPN-LAMP a montré un nombre plus élevé de réponses positives par rapport au placage MPN et au MPN-qPCR, ce qui suggère que la méthode LAMP est plus sensible aux faibles concentrations de Salmonella. Lorsque les échantillons étaient plus concentrés (dilutions inférieures à 1 x 10-3), les trois méthodes ont montré une augmentation des détections positives, avec une convergence se produisant autour de la dilution 1 x 10-2 , où toutes les méthodes ont détecté un nombre similaire d’échantillons positifs.
La figure 4B présente le pourcentage de mesures positives à chaque dilution selon les trois méthodes, ce qui permet une comparaison directe de leur sensibilité dans la détection de Salmonella. Le pourcentage de réponses positives offre une visualisation plus claire des performances de la méthode dans la série de dilution, et les barres d’erreur représentant ± intervalles de confiance (IC) de Clopper-Pearson à 95 % donnent un aperçu de la variabilité à chaque niveau. À des dilutions plus faibles, les intervalles de confiance plus larges indiquent une plus grande variabilité dans les performances de détection de chaque méthode. Conformément au nombre brut de détections positives, le MPN-LAMP détecte un pourcentage plus élevé d’échantillons positifs aux concentrations les plus diluées (1 x 10-5), tandis que le MPN-qPCR et le MPN-plating détectent moins de positifs. À mesure que la concentration bactérienne augmente, les méthodes convergent, les trois approches montrant une détection proche de 100 % à des concentrations plus élevées (p. ex., 1 x 10-2 et plus). Le rétrécissement des intervalles de confiance à ces concentrations plus élevées indique une plus grande cohérence entre toutes les méthodes, mettant l’accent sur leur performance comparable lorsque les niveaux bactériens sont plus élevés.
Les résultats de la figure 3 suggèrent que la sous-estimation constante (quantitative) des trois méthodes pourrait être due à une séparation incomplète de Salmonella de la matrice de l’échantillon. Cependant, des inhibiteurs moléculaires ou des facteurs de viabilité bactérienne pourraient être à l’origine de la variation observée dans la détection à différentes dilutions (qualitativement) et entre différentes méthodes (qualitativement et quantitativement). Les intervalles de confiance plus serrés à des concentrations plus élevées dans la figure 4B indiquent une moindre variabilité et un rendement qualitatif plus cohérent entre les méthodes, tandis que les intervalles plus larges à des concentrations plus faibles indiquent une incertitude accrue dans la détection qualitative.
Détection dans le poulet prêt à cuire
Nous avons choisi la méthode LAMP pour la détection de Salmonella dans le poulet prêt à cuire, car elle a révélé un plus grand nombre d’échantillons positifs (14,3 %) que les autres méthodes testées à la plus faible dilution de Salmonella . À la dilution la plus faible de Salmonella (1 x 10-5), aucun des échantillons n’a été détecté positif par la méthode interne de qPCR. En revanche, certains positifs ont été détectés par placage (7,1 %, la moitié du nombre détecté par LAMP). Cependant, les résultats de cette méthode basée sur la culture peuvent être affectés lors du passage à des produits plus complexes tels que le poulet prêt à cuire, où la présence d’une microflore de fond pourrait fausser les résultats parce que les échantillons ne sont pas irradiés. Pour ces raisons, nous avons décidé d’étudier la méthode MPN-LAMP pour la quantification de Salmonella à partir de poulet prêt à cuire.
La figure 5 illustre les résultats de l’essai NPP-LAMP utilisé pour quantifier Salmonella dans des échantillons de poulet cordon bleu inoculé, congelé et prêt à cuire. Le niveau théorique d’inoculation (UFC/g) a été tracé sur l’axe des abscisses, tandis que les mesures MPN-LAMP correspondantes ont été tracées sur l’axe des ordonnées. L’analyse de régression a donné l’équation y = -15,04 + 0,3869x, avec un coefficient de détermination (R²) de 0,933, indiquant une forte corrélation linéaire entre l’inoculum théorique et les résultats MPN-LAMP. Cette valeur élevée de R² suggère que la méthode MPN-LAMP quantifie de manière fiable les Salmonella dans ces échantillons, bien que la pente de 0,3869 indique que la méthode sous-estime le niveau réel d’inoculum. La zone ombrée autour de la droite de régression représente l’intervalle de confiance à 95 %.
Pour mieux illustrer la performance du NPP-LAMP à des niveaux d’inoculation plus faibles, le graphique en médaillon de la figure 5 zoome sur les points de données inférieurs à 300 UFC/g. D’après l’équation de régression, la limite inférieure de détection peut être estimée à environ 40 UFC/g (38,9 UFC/g). Cependant, l’intervalle de confiance à 95 % suggère que la limite inférieure pratique de détection est plus proche de 325 UFC/g, car toute valeur MPN-LAMP négative doit être interprétée comme nulle. La ligne rouge dans l’encart représente zéro, et bien qu’aucun point de données ne soit inférieur à cette ligne, la possibilité que les valeurs tombent en dessous de zéro, en particulier à des niveaux d’inoculum inférieurs, introduit la possibilité de résultats faussement négatifs. Cette tendance met en évidence les difficultés qu’il est difficile de quantifier avec précision les faibles concentrations de Salmonella, et des données supplémentaires seraient nécessaires pour établir définitivement la limite inférieure de détection.

Figure 1 : Schéma fonctionnel du dosage MPN modifié pour la détection de Salmonella. Un aperçu schématique du dosage du nombre le plus probable (NPP) modifié. Il décrit les étapes clés du processus, en commençant par l’inoculation d’échantillons de poulet haché irradié, suivie de l’homogénéisation des échantillons, de la centrifugation, de la distribution d’aliquotes et de l’incubation dans des conditions spécifiées. La figure comprend des photographies du bloc de 48 puits utilisé pour la dilution et la croissance répétée, ainsi que trois techniques utilisées pour produire des résultats positifs et négatifs pour le calcul de la résolution SMPR (Simple Maximum Probability Resolution). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Schéma de l’essai sur plaque de chute. La configuration du dosage sur plaque de chute pour une dilution spécifique (par exemple, non diluée, 10-1, 10-2, etc.) à partir du bloc de 48 puits. Chaque échantillon est plaqué dans un format de grille 4 x 6 sur une plaque de gélose à l’aide d’une pipette multicanaux. La disposition a été modifiée, passant de la grille traditionnelle de 6 x 6 à deux grilles de 4 x 6 pour s’assurer que six répliques de huit dilutions pouvaient être prises en charge. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Comparaison de l’Inoculum avec les méthodes NPP. (A) Un graphique de la concentration MPN-LAMP (axe y) par rapport à l’inoculum théorique vérifié par placage (axe x). Une droite de régression ajustée (y = -72,36 + 0,161x) avec un coefficient de détermination (R² = 0,978) démontre une forte relation linéaire. (B) Un graphique de la concentration MPN-qPCR (axe y) par rapport à l’inoculum théorique (axe x), avec une droite de régression ajustée (y = -46,84 + 0,0395x) et R² = 0,978, montrant une corrélation tout aussi forte. (C) Un graphique de la concentration de placage MPN (axe y) par rapport à l’inoculum théorique (axe x), avec une droite de régression (y = -330,4 + 0,3138x) et R² = 0,944, indiquant une corrélation forte mais légèrement inférieure à celle de LAMP et qPCR. L’équation de régression et le coefficient de détermination de chaque figure sont présentés sous forme d’encart dans le coin supérieur gauche des graphiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Comparaison des méthodes MPN. (A) La figure présente le pourcentage de récupération, calculé comme le rapport entre la valeur du NMP et le niveau d’inoculation théorique pour les méthodes MPN-LAMP, MPN-placage et MPN-qPCR. La méthode MPN-LAMP a eu une récupération moyenne de 15,19 % (±10,04 %), la méthode MPN-plating a eu une récupération moyenne de 13,13 % (± 11,45 %) et la méthode MPN-qPCR a eu une récupération moyenne de 6,67 % (± 3,19 %). Les boîtes à moustaches mettent en évidence la distribution des données de récupération, avec les valeurs aberrantes indiquées. Les barres d’erreur représentent la plage de données comprise dans 1,5 fois l’intervalle interquartile (IQR). (B) Pourcentage de détections positives de Salmonella à chaque dilution selon trois méthodes : NPP-LAMP (bleu), placage NPP (rouge) et NPP-qPCR (vert). L’axe des x représente les niveaux de dilution (10-6 à 10) et l’axe des y montre le pourcentage de mesures positives. Les barres d’erreur indiquent les intervalles de confiance ± à 95 % de Clopper-Pearson, qui reflètent la variabilité de la détection pour chaque méthode. MPN-LAMP montre des pourcentages de détections plus élevés que les autres méthodes à des dilutions plus faibles, tandis que toutes les méthodes convergent vers une détection proche de 100 % à des concentrations plus élevées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
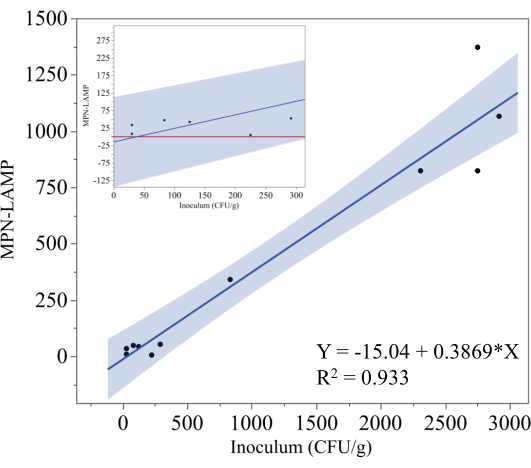
Figure 5 : Méthode NPP pour le poulet prêt à cuire. L’analyse de régression des résultats MPN-LAMP par rapport au niveau théorique d’inoculation de Salmonella dans des échantillons de poulet cordon bleu congelés et prêts à cuire. La droite de régression est représentée par y = -15,04 + 0,3869x avec un R² de 0,933, indiquant une forte relation linéaire. La région ombrée représente l’intervalle de confiance à 95 %. L’encadré se concentre sur les mesures MPN-LAMP à des niveaux d’inoculation inférieurs. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| 1 | 2 | 3 | 4 | 5 | 6 | |
| Un | n = 1 | n = 1 | n = 1 | n = 1 | n = 1 | n = 1 |
| B | 2 | 2 | 2 | 2 | 2 | 2 |
| C | 3 | 3 | 3 | 3 | 3 | 3 |
| D | 4 | 4 | 4 | 4 | 4 | 4 |
| E | 5 | 5 | 5 | 5 | 5 | 5 |
| F | 6 | 6 | 6 | 6 | 6 | 6 |
| G | 7 | 7 | 7 | 7 | 7 | 7 |
| H | 8 | 8 | 8 | 8 | 8 | 8 |
| 100 | 10-1 | 10-2 | 10-3 | 10-4 | 10-5 |
Tableau 1 : Schéma des répétitions et des dilutions dans un bloc de 48 puits. Cette figure illustre la disposition des répétitions et des dilutions en série de 10 fois dans le bloc de 48 puits. Chaque colonne représente un facteur de dilution spécifique, allant de 100 à 10-5 , les répétitions techniques pour chaque dilution étant réparties sur les huit rangées.
| Dilution | ||||||
| 10-5 | 10-4 | 10-3 | 10-2 | 10-1 | 100 | |
| LAMPE MPN | 8 | 16 | 31 | 3 | 56 | 56 |
| Placage MPN | 4 | 11 | 32 | 55 | 56 | 56 |
| MPN-qPCR | 0 | 7 | 26 | 55 | 56 | 56 |
Tableau 2 : Nombre d’événements positifs. Le tableau affiche le nombre de réactions positives (sur un total de 56 répétitions). Les résultats sont disposés dans un tableau où les colonnes représentent la dilution et les rangées sont représentatives de la méthode.
Discussion
Importance du protocole
La salmonelle demeure une préoccupation majeure en matière de salubrité des aliments, en particulier dans le secteur des produits de volaille, qui sont souvent impliqués dans des éclosions de maladies d’origine alimentaire13,14. En tant que principale cause de maladies bactériennes d’origine alimentaire aux États-Unis, des méthodes fiables de détection de Salmonella dans les produits de volaille frais et prêts à cuire sont essentielles pour garantir la sécurité alimentaire15. La capacité de quantifier Salmonella à de faibles niveaux est essentielle pour identifier la contamination qui pourrait autrement passer inaperçue. La sensibilité de la méthode est particulièrement importante pour la conformité réglementaire et la santé publique, car même de faibles doses de Salmonella peuvent entraîner des maladies, en particulier chez les populations vulnérables.
Le test MPN-LAMP utilise le système MDS, qui est actuellement utilisé par le FSIS dans le MLG 4.15, pour déterminer la prévalence de la contamination4. Le test présenté ici fournit une extension de la méthode actuelle du FSIS pour quantifier les Salmonella viables, même à de faibles niveaux, fournissant des données plus précises sur le niveau de contamination. Les résultats de cette étude ont mis en évidence une forte corrélation entre la méthode MPN-LAMP et les valeurs théoriques d’inoculation, avec un R² de 0,933 (Figure 5). Cela suggère que la méthode est fiable pour quantifier Salmonella dans les aliments, bien qu’une certaine sous-estimation se soit produite à des niveaux d’inoculation inférieurs.
Étapes critiques
Le protocole de quantification de Salmonella nécessite une précision dans la préparation des échantillons, l’inoculation et l’analyse moléculaire. Une technique d’asepsie appropriée, des dilutions précises et le respect des conditions d’incubation sont essentiels pour une détection et un dénombrement fiables de Salmonella. Il est essentiel de garantir des conditions stériles lors du transfert des échantillons et des milieux de volaille pour prévenir la contamination.
Tout aussi importante est l’homogénéisation des échantillons pendant l’estomac, suivie d’une remise en suspension soigneuse des granulés après la centrifugation pour séparer les bactéries de la matrice alimentaire. Une manipulation appropriée au cours de ces étapes permet de s’assurer que les cellules viables sont récupérées pour une analyse plus approfondie. Un pipetage précis pendant le processus de dilution en série pour la configuration MPN, associé à des conditions d’incubation constantes, est crucial pour obtenir des résultats reproductibles.
Enfin, les méthodes de détection moléculaire, telles que la qPCR, nécessitent précision et reproductibilité dans l’extraction de l’ADN et le cycle de la qPCR pour éviter les faux négatifs. Bien que l’extraction de l’ADN soit intrinsèquement variable, les réactifs et les procédures standardisés assurent la répétabilité en minimisant l’interférence des inhibiteurs et en améliorant la récupération de l’ADN. Ces étapes sont fondamentales pour confirmer la présence de Salmonella et pour cibler des marqueurs génétiques spécifiques qui peuvent aider à surmonter les limites des méthodes traditionnelles de placage et d’enrichissement.
Pièges et limites potentiels
Viabilité bactérienne
L’un des principaux défis de la mise en œuvre d’un test basé sur la croissance tel que le placage MPN est la viabilité bactérienne, ce qui est particulièrement pertinent pour les échantillons complexes de poulet prêts à cuire. Les produits de poulet congelés, panés et farcis, tels que le poulet cordon bleu, sont particulièrement sujets aux fluctuations de température et aux périodes de stockage prolongées, qui peuvent tous deux stresser les cellules de Salmonella et rendre l’agent pathogène plus difficile à détecter dans les tests basés sur la croissance. De plus, la présence d’ingrédients autres que ceux du poulet (p. ex. fromage, panure, jambon, épices, beurre) introduit une complexité supplémentaire dans la matrice alimentaire et peut inhiber la croissance de Salmonella pendant les phases d’enrichissement, ce qui complique la quantification précise. De plus, Salmonella doit rivaliser avec les microbes de fond dans les méthodes basées sur la culture16, ce qui est particulièrement pertinent pour les échantillons prêts à cuisiner avec plusieurs ingrédients qui ajoutent de la diversité au microbiote par rapport au poulet cru. Enfin, la manipulation et le traitement des facteurs de stress, tels que les antimicrobiens, peuvent influencer la récupération de Salmonella 17.
La viabilité bactérienne peut avoir eu une incidence sur la performance des méthodes testées dans cette étude. Toutes les méthodes sous-estimaient les concentrations de Salmonella à de faibles concentrations et présentaient une plus grande variabilité de détection à des concentrations d’agents pathogènes plus faibles (figures 4A et B). Cependant, la méthode MPN-LAMP moléculaire a surpassé le placage MPN et le MPN-qPCR en termes de détection qualitative à des niveaux d’inoculation inférieurs. Les résultats soulignent la nécessité d’affiner à de faibles concentrations d’agents pathogènes.
Hétérogénéité de l’échantillon
Une autre limitation importante concerne l’hétérogénéité des échantillons d’aliments, qui peut introduire une variabilité dans les résultats de détection de Salmonella . Le protocole a été conçu pour disperser 30 mL d’échantillon homogénéisé, ce qui permet la distribution de 8 répétitions sous forme d’aliquotes de 3 mL (24 mL au total, 6 mL en excès) dans le bloc. Bien que cette approche conservatrice (qui permet d’obtenir un échantillon supplémentaire) tienne compte des pertes potentielles, elle introduit un sous-échantillonnage car le volume entier n’est pas testé.
L’hétérogénéité de l’échantillon alimentaire peut affecter la précision de la mesure lors du transfert d’échantillons du mélange redispersé dans le bloc et de la réalisation de dilutions18,19. Selon la théorie de l’échantillonnage (TOS), les erreurs d’échantillonnage fondamentales peuvent être réduites en augmentant la masse de l’échantillon ou en réduisant l’hétérogénéité de la composition grâce à la réduction de la taille des particules20. Cependant, alors que certaines études indiquent que la décomposition de la matrice par l’estomac n’a pas d’impact significatif sur la récupération bactérienne à partir d’une contamination de surface, il a été démontré que la réduction de la taille des particules de la matrice alimentaire diminue la récupération bactérienne dans certains cas 21,22,23. Dans cette étude, la taille des particules a été réduite par l’estomac avant l’enrichissement ; Bien que cela puisse réduire les erreurs d’échantillonnage en réduisant l’hétérogénéité de la composition des échantillons, la diluation et la centrifugation peuvent avoir un impact sur la récupération bactérienne.
Enfin, la composition et la morphologie hétérogènes des aliments, ainsi que le fait que la contamination soit interne ou en surface, peuvent entraîner une variabilité de la récupération des agents pathogènes. Des travaux antérieurs18,22 ont démontré qu’une forte liaison bactérienne aux surfaces des tissus peut réduire les taux de récupération. Des études où des enzymes telles que la trypsine, la collagénase et l’endopeptidase ont été utilisées pour libérer des bactéries à partir de matrices de viande et de volaille24,25 et de pectinase et cellulase dans les légumes-feuilles19 démontrent le potentiel d’amélioration de la récupération de Salmonella à partir des matrices alimentaires.
Suggestions de dépannage
Pour éviter tout problème, assurez-vous que tous les milieux et réactifs sont fraîchement préparés et stériles. Les matériaux périmés ou mal stérilisés peuvent entraîner une contamination ou affecter la croissance de Salmonella. Si une contamination est observée, vérifier que la technique aseptique a été suivie de façon uniforme tout au long du protocole, en particulier lors de l’inoculation, de la dilution, de l’aliquotage et du placage de l’échantillon.
Lorsque le nombre de colonies est inférieur aux prévisions, la vérification de l’exactitude des dilutions en série et des concentrations d’inoculum peut aider à identifier les erreurs. Les conditions d’incubation, y compris la température, le temps et l’agitation, doivent être soigneusement surveillées pour favoriser une croissance optimale de Salmonella . En cas de croissance faible ou nulle, un estomac inadéquat ou des fuites des poches peuvent contribuer à une distribution inégale de la bactérie.
Si la détection moléculaire produit des résultats incohérents, la vérification des procédures d’extraction de l’ADN et la vérification de la qualité des amorces et des sondes sont des étapes clés. De plus, la variabilité entre les matrices alimentaires peut présenter différents niveaux d’inhibition, qui peuvent être surveillés à l’aide du contrôle d’amplification interne (IAC). Le stockage et la manipulation appropriés des réactifs d’extraction sont essentiels pour éviter les faux négatifs dus à la dégradation ou à la contamination. Des méthodes d’extraction d’ADN plus efficaces peuvent également être justifiées.
L’incorporation de traitements tels que le système GentleMACS26, qui utilise des actions mécaniques, thermiques et enzymatiques, peut optimiser davantage la récupération des bactéries dans les aliments. Ce système peut réduire l’hétérogénéité de la matrice alimentaire, ce qui facilite le transfert d’échantillons uniformes dans le bloc et la réalisation de dilutions en série précises.
Potentiel de raffinement et d’expansion
Il existe plusieurs possibilités d’affiner et d’étendre ce protocole. L’exploration de formulations alternatives de milieux d’enrichissement pourrait améliorer la récupération des cellules de Salmonella stressées ou sublétales, en particulier à partir de matrices alimentaires complexes telles que le cordon bleu de poulet. Bien que la gélose sélective soit couramment utilisée pour cibler Salmonella, les cellules blessées ou stressées en raison des conditions de traitement et de stockage peuvent entraîner des faux négatifs sur ces milieux. Pour surmonter ce défi, l’optimisation de l’équilibre entre la pression sélective et la récupération microbienne pourrait améliorer la détection des sérovars de Salmonella qui sont moins compétitifs dans des conditions d’enrichissement standard. Ce raffinement pourrait améliorer la sensibilité globale des essais, en particulier dans les produits alimentaires difficiles où le stress microbien est courant. Le raccourcissement des temps d’incubation pourrait accélérer le délai d’obtention des résultats, bien que cela puisse réduire la sensibilité et limiter la détection. Les travaux futurs devraient explorer les progrès réalisés dans les milieux d’enrichissement pour soutenir la réplication bactérienne rapide, assurant une quantification précise tout en répondant aux exigences des parties prenantes.
De plus, l’étude de différentes températures et durées d’incubation pour le test MPN pourrait optimiser la récupération de Salmonella à partir de divers produits de volaille. Cela pourrait étendre l’applicabilité du protocole à d’autres agents pathogènes d’origine alimentaire qui nécessitent des conditions de croissance distinctes, élargissant ainsi son utilité dans les tests de sécurité alimentaire. L’automatisation pourrait améliorer l’évolutivité de ce protocole en intégrant des manipulateurs de liquides robotisés pour les étapes d’aliquotage et de dilution. Les centrifugeuses à flux continu ou les systèmes de centrifugation à grande capacité peuvent également résoudre les limitations du traitement par lots, permettant des applications à haut débit dans les environnements industriels.
L’élargissement de l’aspect de la détection moléculaire pour inclure la PCR multiplex pourrait permettre la détection simultanée de plusieurs agents pathogènes, améliorant ainsi le débit dans les environnements de surveillance et d’essai. De plus, l’intégration du séquençage de nouvelle génération (NGS) pourrait fournir des informations précieuses sur le sérotype et la virulence directement à partir de cultures d’enrichissement du NMP, facilitant ainsi les enquêtes épidémiologiques et les efforts de suivi à la source.
Enfin, la limite de quantification/détection peut être repoussée en augmentant la quantité d’échantillons utilisés dans le NPP. Par exemple, si le poids de l’échantillon de viande était augmenté de 25 g à 325 g, la limite de détection pourrait être extrapolée à 4 UFC/g à l’aide de l’équation de régression linéaire. Cependant, en supposant le même intervalle de confiance relatif à 95 %, la limite inférieure pratique de quantification est de >30 UFC/g.
Conclusion
La mise au point du test MPN-LAMP dans cette étude offre un outil précieux et pratique pour quantifier la contamination par Salmonella dans les produits de volaille. Avec une forte corrélation avec les niveaux d’inoculation théoriques (R² = 0,933), la méthode détecte de manière fiable Salmonella à de faibles niveaux, ce qui est essentiel pour la sécurité alimentaire et la conformité réglementaire. Cette capacité est particulièrement importante pour détecter une contamination qui pourrait autrement passer inaperçue, ce qui permet aux producteurs de gérer de manière proactive les risques potentiels. Le test MPN-LAMP offre une alternative quantitative aux méthodes traditionnelles basées sur la prévalence, offrant un niveau de contamination à Salmonella dans une variété de produits de volaille, y compris des articles prêts à cuire plus complexes.
L’un des principaux points forts de cette méthode réside dans son adaptabilité à différentes matrices alimentaires, en particulier aux produits surgelés et transformés comme le poulet cordon bleu, où les méthodes traditionnelles peuvent avoir du mal en raison de la présence d’ingrédients supplémentaires tels que la panure et le fromage. L’intégration de techniques moléculaires, telles que la LAMP et la qPCR, avec le test MPN garantit une approche globale de la détection, surmontant les défis posés par les microbes de fond et les conditions de traitement. Ces méthodes moléculaires améliorent la sensibilité du protocole, ce qui en fait un outil polyvalent pour les tests de routine de sécurité alimentaire et la surveillance de la contamination, avec le potentiel de réduire le risque d’éclosions liées à Salmonella.
Bien qu’aucune méthode de détection ne soit exempte de limites, le test MPN-LAMP démontre des performances constantes sur une gamme de niveaux d’inoculum et de matrices alimentaires. La variabilité à des niveaux d’inoculation plus faibles peut être abordée par des améliorations futures, telles que l’optimisation de la préparation des échantillons et des formulations des milieux pour maximiser la récupération, mais cela n’enlève rien à la robustesse globale du protocole. En permettant une détection précise des cellules de Salmonella , même dans des conditions difficiles, la méthode MPN-LAMP dote l’industrie alimentaire d’un outil inestimable pour la protection de la santé publique.
À l’avenir, il y a place à d’autres améliorations, comme l’élargissement de l’essai pour inclure davantage de sérovars de Salmonella ou l’intégration du séquençage de nouvelle génération pour une meilleure caractérisation des agents pathogènes. Néanmoins, ce protocole représente déjà une avancée significative dans la détection et la quantification de Salmonella , fournissant une méthode pratique, efficace et fiable qui répond aux exigences croissantes des tests de sécurité alimentaire.
Déclarations de divulgation
Tous les auteurs déclarent qu’il n’y a pas de conflit d’intérêts.
Remerciements
Cette recherche a été financée par le département de l’Agriculture des États-Unis, Agricultural Research Service (USDA-ARS), National Program 108, Current Research Information System numéros 8072-42000-093-000-D et 8072-42000-094-000-D. La mention de noms commerciaux ou de produits commerciaux dans cet article est uniquement dans le but de fournir des informations spécifiques et n’implique pas une recommandation ou une approbation par le département de l’Agriculture des États-Unis. L’USDA est un fournisseur et un employeur qui souscrit au principe de l’égalité des chances.
matériels
| Name | Company | Catalog Number | Comments |
| 48 deep well block 4.6ml | Fisher Scientific International, Inc | NC1964628 | |
| Agar - Solidifying Agent (Difco) | Becton, Dickinson and Company (BD) | 281230 | |
| Analytical Balance | Mettler Toledo | JL602-G/L | Equipment |
| Analytical Balance | Mettler Toledo | AB54-S | Equipment |
| Autoclave - Amsco Lab250, Laboratory Steam Sterilizer | Steris plc | LV-250 | Equipment |
| Biological Safety Cabinet, Type A2, Purifier Logic+ | Labconco Corporation | 302411101 | Equipment |
| Brain Heart Infusion (BHI) Broth | Becton, Dickinson and Company (BD) | 237500 | |
| Buffered Peptone Water | Bio-Rad Laboratories Inc. | 3564684 | |
| Cell Spreader - L-shaped | VWR | 76208-438 | |
| Centrifuge Microcentrifuge 5424 | Eppendorf | 5424 | Equipment |
| Centrifuge, Avanti J-25 | Beckman Coulter, Inc. | Equipment | |
| DNA Extraction - PreMan Ultra Sample Preparation Reagent | Thermo Fisher Scientific Inc. | 4318930 | |
| Ground Chicken | Local retailers | ||
| IAC forward primer: 5'-GGCGCGCCTAACACATCT-3' | Integrated DNA Technologies | ||
| IAC probe: 5'-TTACAACGGGAGAAGACAATGC CACCA-3' labeled with 5' TAMRA/3' BHQ-2 | Biosearch Technologies | ||
| IAC reverse primer: 5'-TGGAAGCAATGCCAAATGTGTA-3' | Integrated DNA Technologies | ||
| Incubator - Inova 4230 incubator shaker | New Brunswick Scientific | 4230 | Equipment |
| Inoculating Loop - Combi Loop 10µL and 1µL | Fisher Scientific International, Inc | 22-363-602 | |
| invA forward primer: 5'-GTTGAGGATGTTATTCGCAAAG G-3' | Integrated DNA Technologies | ||
| invA probe: 5'-CCGTCAGACCTCTGGCAGTAC CTTCCTC-3' labeled with 5' Cal Fluor Orange 560/3' BHQ-1 | Biosearch Technologies | ||
| invA reverse primer: 5'-GGAGGCTTCCGGGTCAAG-3' | Integrated DNA Technologies | ||
| Irradiation Treatment | Texas A&M Agrilife Research National Center for Electron Beam Research | Service | |
| Luria Bertani (LB) Broth | Becton, Dickinson and Company (BD) | 244620 | |
| Manual pipette Pipet-Lite LTS Pipette L-1000XLS+ | Mettler Toledo | 17014382 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-100XLS+ | Mettler Toledo | 17014384 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-10XLS+ | Mettler Toledo | 17014388 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-200XLS+ | Mettler Toledo | 17014391 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-20XLS+ | Mettler Toledo | 17014392 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-200XLS+ | Mettler Toledo | 17013805 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-20XLS+ | Mettler Toledo | 17013803 | Equipment |
| Media Storage Bottle -PYREX 1L Square Glass Bottle, with GL45 Screw Cap | Corning Inc. | 1396-1L | Equipment |
| Media Storage Bottle -PYREX 2L Round Wide Mouth Bottle, with GLS80 Screw Cap | Corning Inc. | 1397-2L | Equipment |
| Microtiter plate, 96 well plate, flat bottom, polystyrene, 0.34cm2, sterile, 108/cs | MilliporeSigma | Z707902 | |
| Mixer - Vortex Genie 2 | Scientific Industries Inc. | SI-0236 | Equipment |
| Molecular Detection Assay 2-Salmonella kit | Neogen | MDA2SAL96 | |
| Molecular Detection Instrument | Neogen | MDS100 | Equipment |
| Motorized pipette controller, PIPETBOY2 | INTEGRA Biosciences Corp. | 155019 | Equipment |
| PCR Mastermix 2× TaqMan Gene Expression | Thermo Fisher Scientific Inc. | 4369542 | |
| Petri Dish Rotator - bioWORLD Inoculation Turntable | Fisher Scientific International, Inc | 3489E20 | Equipment |
| Petri Dishes with Clear Lid (100 mm x 15mm) | Fisher Scientific International, Inc | FB0875713 | |
| Pipette Tips GP LTS 1000µL S 768A/8 | Mettler Toledo | 30389273 | |
| Pipette Tips GP LTS 20µL 960A/10 | Mettler Toledo | 30389270 | |
| Pipette Tips GP LTS 200µL F 960A/10 | Mettler Toledo | 30389276 | |
| Ready to cook chicken products | Local retailers | ||
| Reagent Reservoir, 25 mL sterile reservoir used with multichannel pipettors | Thermo Fisher Scientific Inc. | 8093-11 | |
| Realtime PCR - 7500 Real-Time PCR system | (Applied Biosystems, Foster City, CA) | 2750036476 | Equipment |
| Serological Pipettes, Nunc Serological Pipettes (10 mL) | Thermo Fisher Scientific Inc. | 170356N | |
| Serological Pipettes, Nunc Serological Pipettes (2 mL) | Thermo Fisher Scientific Inc. | 170372N | |
| Serological Pipettes, Nunc Serological Pipettes (25 mL) | Thermo Fisher Scientific Inc. | 170357N | |
| Serological Pipettes, Nunc Serological Pipettes (50 mL) | Thermo Fisher Scientific Inc. | 170376N | |
| Spreader - Fisherbrand L-Shaped Cell Spreaders | Fisher Scientific International, Inc | 14-665-230 | |
| Stomacher bag, Nasco Whirl-Pak Write-On Homogenizer Blender Filter Bags | Thermo Fisher Scientific Inc. | 01-812 | |
| Stomacher 80 Biomaster Lab Blender | Seward | 30010019 | Equipment |
| Thermocycler (GeneAmp PCR system 9700) | Applied Biosystems | 535028293 | Equipment |
| Water Filtration - Elga Veolia Purelab Flex | Elga LabWater | PF2XXXXM1-US | Equipment |
| Whirlpak bags 1.63L | VWR | 11216-777 |
Références
- Batz, M., Hoffmann, S., Morris, J. G. Disease-outcome trees, eq-5d scores, and estimated annual losses of quality-adjusted life years (qalys) for 14 foodborne pathogens in the united states. Foodborne Pathogens and Disease. 11 (5), 395-402 (2014).
- . The Grand Challenge: Salmonella Available from: https://tellus.ars.usda.gov/stories/articles/the-grand-challenge-salmonella (2024)
- National Advisory Committee on Microbiological Criteria in Foods (NACMCF). Response to questions posed by the food safety and inspection service: Enhancing Salmonella control in poultry products. J Food Prot. 82 (4), 645-668 (2019).
- Food Safety and Inspection Service. . 4.15 Isolation and identification of Salmonella from meat, poultry, pasteurized egg, siluriformes (Fish) products and carcass and environmental sponges. , (2024).
- Irwin, P., Reed, S., Brewster, J., Nguyen, L., He, Y. P. Non-stochastic sampling error in quantal analyses for campylobacter species on poultry products. Analytical and Bioanalytical Chemistry. 405 (7), 2353-2369 (2013).
- Irwin, P., Tu, S., Damert, W., Phillips, J. A modified gauss-newton algorithm and ninety-six well micro-technique for calculating mpn using excel spreadsheets. Journal of Rapid Methods & Automation in Microbiology. 8 (3), 171-191 (2000).
- Ravishankar, S., Ahmed, E. Y., Carlstrom, C. Food microbiology: A laboratory manual. Food Microbiology. 21, 489 (2004).
- Chen, C. Y., Nace, G. W., Irwin, P. L. A 6 x 6 drop plate method for simultaneous colony counting and mpn enumeration of campylobacter jejuni, listeria monocytogenes, and escherichia coli. J Microbiol Methods. 55 (2), 475-479 (2003).
- Suo, B., He, Y., Tu, S. I., Shi, X. A multiplex real-time polymerase chain reaction for simultaneous detection of salmonella spp., escherichia coli o157, and listeria monocytogenes in meat products. Foodborne Pathogens and Disease. 7 (6), 619-628 (2010).
- Jarvis, B., Wilrich, C., Wilrich, P. T. Reconsideration of the derivation of most probable numbers, their standard deviations, confidence bounds and rarity values. J Appl Microbiol. 109 (5), 1660-1667 (2010).
- Stevens, R., Poppe, K. Validation of clinical prediction models: What does the "calibration slope" really measure. Journal of Clinical Epidemiology. 118, (2019).
- Miller, M. E., Hui, S. L., Tierney, W. M. Validation techniques for logistic regression models. Statistics in Medicine. 10 (8), 1213-1226 (1991).
- Galán-Relaño, &. #. 1. 9. 3. ;., et al. Salmonella and salmonellosis: An update on public health implications and control strategies. Animals. 13 (23), 3666 (2023).
- Gorski, L., et al. Growth assessment of salmonella enterica multi-serovar populations in poultry rinsates with commonly used enrichment and plating media. Food Microbiology. 119, 104431 (2024).
- Schmidt, J. W., et al. Evaluation of methods for identifying poultry wing rinses with salmonella concentrations greater than or equal to 10 cfu/ml. J Food Prot. 87 (11), 100362 (2024).
- Gorski, A., Liang, L. S. Effect of enrichment medium on real-time detection of salmonella enterica from lettuce and tomato enrichment cultures. Journal of Food Protection. 73 (6), 1047-1056 (2010).
- Guillén, S., Nadal, L., Álvarez, I., Mañas, P., Cebrián, G. Impact of the resistance responses to stress conditions encountered in food and food processing environments on the virulence and growth fitness of non-typhoidal salmonellae. Foods. 10 (3), 617 (2021).
- Rohde, A., Hammerl, J. A., Appel, B., Dieckmann, R., Al Dahouk, S. Sampling and homogenization strategies significantly influence the detection of foodborne pathogens in meat. BioMed Research International. 2015, (2015).
- Wang, D., Wang, Z., He, F., Kinchla, A. J., Nugen, S. R. Enzymatic digestion for improved bacteria separation from leafy green vegetables. Journal of Food Protection. 79 (8), 1378-1386 (2016).
- Pitard, F. F. . Theory of sampling and sampling practice. , (2019).
- Sharpe, A. . in Detecting pathogens in food. , 52-68 (2003).
- Hannah, J., et al. Effect of stomaching on numbers of bacteria recovered from chicken skin. Poultry Science. 90 (2), 491-493 (2011).
- Mcmeekin, T., Thomas, C. Retention of bacteria on chicken skin after immersion in bacterial suspensions. Journal of Applied Bacteriology. 45 (3), 383-387 (1978).
- Rodrigues-Szulc, U., Ventoura, G., Mackey, B., Payne, M. Rapid physicochemical detachment, separation and concentration of bacteria from beef surfaces. Journal of Applied Bacteriology. 80 (6), 673-681 (1996).
- Vibbert, H. B., et al. Accelerating sample preparation through enzyme-assisted microfiltration of salmonella in chicken extract. Biotechnol Prog. 31 (6), 1551-1562 (2015).
- Armstrong, C. M., et al. Use of a commercial tissue dissociation system to detect salmonella-contaminated poultry products. Analytical and Bioanalytical Chemistry. 416 (3), 621-626 (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon