Method Article
Ensayo modificado del número más probable para cuantificar la salmonela en productos de pollo crudos y listos para cocinar
En este artículo
Resumen
Cuantificar con precisión la Salmonella en las aves de corral a niveles bajos es un desafío industrial y regulatorio actual. Este protocolo describe un ensayo de NMP que permite la cuantificación de Salmonella en productos avícolas crudos y listos para cocinar. Este método es rápido, sensible y se alinea con las pautas del FSIS, mejorando la seguridad alimentaria y apoyando los esfuerzos de salud pública.
Resumen
La salmonela es una de las principales causas de enfermedades transmitidas por los alimentos en los Estados Unidos, particularmente en los productos avícolas. Los métodos tradicionales para detectar Salmonella se centran en la prevalencia más que en la cuantificación, lo que limita su utilidad para evaluar los niveles y riesgos de contaminación. Este estudio presenta un nuevo ensayo del número más probable (NMP) diseñado para cuantificar la Salmonella en productos avícolas listos para cocinar, como el pollo cordon bleu. El método consiste en lavar la muestra de aves de corral, concentrar el enjuague a través de la centrifugación y diluirlo en serie en un bloque de 48 pocillos. El ensayo MPN se integra con el método de amplificación isotérmica mediada por bucle (LAMP) para proporcionar una cuantificación sensible, precisa y rápida de la contaminación por Salmonella dentro del mismo período de tiempo que los protocolos existentes del Servicio de Inspección y Seguridad Alimentaria (FSIS). Los resultados muestran una fuerte correlación lineal entre las mediciones de MPN-LAMP y los niveles teóricos de inoculación (R² = 0,933). Sin embargo, la variabilidad a concentraciones más bajas pone de relieve los desafíos para detectar con precisión Salmonella a estos niveles, con el límite inferior práctico de detección estimado en aproximadamente 300 UFC/g. Los posibles refinamientos para mejorar la aplicabilidad del protocolo incluyen el aumento de la cantidad muestreada para mejorar aún más el límite de detección, la optimización de las formulaciones de los medios de enriquecimiento y la expansión de la detección molecular para dirigirse a múltiples serovares de Salmonella . En general, este estudio presenta una herramienta práctica para la industria alimentaria, que permite la cuantificación fiable de la contaminación por Salmonella en productos avícolas, contribuyendo a mejorar la seguridad alimentaria y la salud pública.
Introducción
Como una de las principales causas de enfermedades, hospitalizaciones y muertes transmitidas por los alimentos en los EE. UU., la salmonela tiene un impacto económico y de salud pública significativo. La carga económica estimada del patógeno solo en 2013 fue de 3.670millones de dólares. Aunque las recientes iniciativas regulatorias tienen como objetivo reducir la salmonelosis en un 25% para 20302, las brechas en las estrategias actuales de detección y mitigación siguen siendo evidentes, particularmente en la alineación de la vigilancia de las plantas de procesamiento con los resultados de salud pública3 .
Los productos avícolas congelados listos para cocinar, que se han visto implicados en múltiples brotes de Salmonella , son una preocupación importante para la salud pública. En respuesta, el Servicio de Inspección y Seguridad Alimentaria (FSIS, por sus siglas en inglés) clasificó a la Salmonella como adulterante en estos productos. Actualmente, la Guía de Laboratorio de Microbiología (MLG) 4.15 del FSIS se centra únicamente en determinar la prevalencia de Salmonella en productos avícolas4. Según esta directriz, las muestras recogidas se enriquecen durante 18-24 horas y luego se analizan utilizando el Sistema de Detección Molecular (MDS), que identifica la presencia o ausencia de Salmonella pero no ofrece información sobre el nivel de contaminación. Si bien este enfoque es valioso para detectar la presencia de patógenos, no proporciona información cuantitativa que podría ayudar a los procesadores de alimentos a evaluar los riesgos de contaminación con mayor precisión y tomar medidas correctivas específicas.
En este estudio, desarrollamos un método para aumentar la detección desde la prevalencia hasta la cuantificación de patógenos microbianos. Fue diseñado para una integración perfecta en los procesos existentes para detectar Salmonella en productos avícolas con una interrupción mínima de los protocolos actuales del FSIS. En lugar de simplemente enriquecer la muestra a granel, el método comienza lavando los productos avícolas utilizando medios consistentes con los métodos actuales del FSIS. Luego, el enjuague se distribuye en la primera columna de un bloque de pozos de 48 profundidades. Se realizan diluciones seriadas en las cinco columnas restantes, y el bloque se incuba durante 18-24 h, alineándose con el protocolo MLG 4.15. Después de la incubación, los pocillos se analizan para detectar Salmonella y los resultados se utilizan para calcular el número más probable (NMP)5,6. Este enfoque permite la cuantificación de la contaminación dentro del mismo marco de tiempo que el proceso FSIS actual, lo que lo convierte en una opción práctica tanto para la industria como para el uso regulatorio. La Figura 1 muestra un diagrama de bloques que resume el ensayo MPN modificado. La figura incluye fotografías tomadas en etapas específicas, el bloque de 48 pocillos utilizado para la dilución y el crecimiento de réplicas, y las tres técnicas utilizadas como puntos de referencia para evaluar el número más probable de Salmonella presente en el pollo molido. En la primera fase de este estudio, utilizamos pollo molido irradiado para minimizar el impacto de la microflora de fondo y la incertidumbre de las mediciones relativas al inóculo verificado antes de aplicar el protocolo a muestras de pollo no irradiadas.
Protocolo
NOTA: Todo el trabajo asociado con este protocolo debe realizarse dentro de un laboratorio de Bioseguridad Nivel 2 (BSL-2). Cuando sea apropiado, este protocolo debe llevarse a cabo dentro de una cabina de seguridad biológica (BSC) para mantener las condiciones asépticas y minimizar el riesgo de contaminación de la muestra o exposición del operador a patógenos microbianos. Al transferir muestras fuera del BSC, utilice recipientes sellados para mantener la integridad de la muestra y evitar derrames en caso de caídas accidentales. Preferiblemente, se deben usar componentes desechables durante todo el procedimiento para mitigar la posibilidad de contaminación cruzada. En los casos en que los desechables no sean factibles, asegúrese de que todos los equipos y materiales sean estériles antes de usarlos. La gestión adecuada de los residuos es crucial; Todos los componentes desechables usados deben desecharse como residuos de riesgo biológico. Materiales reutilizables en autoclave antes de su reutilización para garantizar una esterilización y contención adecuadas de materiales potencialmente peligrosos. El cumplimiento de estas precauciones no solo salvaguarda la integridad de la muestra, sino que también minimiza el riesgo de exposición del operador a patógenos microbianos.
1. Preparación de muestras de carne
- Adquisición y procesamiento de muestras de carne
- Carne fresca
- Compre pollo molido en el departamento de carne fresca de los minoristas locales. Transfiera todas las muestras al almacenamiento a 4 °C y procese dentro de las 24 horas posteriores a la recepción. Divida asépticamente la carne en muestras de 25 g.
- Sellar al vacío e irradiar la muestra. Aquí, el Centro Nacional de Investigación por Haz de Electrones de Texas A&M AgriLife irradió carne sometida a una dosis de ~25 kGy.
NOTA: Si bien la irradiación se utilizó como medida de control en este estudio para garantizar la eliminación de la microflora de fondo, no es un requisito previo para el protocolo en aplicaciones prácticas, como lo demuestran los productos listos para cocinar no irradiados utilizados en la sección siguiente. En entornos de campo, los métodos alternativos, como los medios selectivos o la especificidad del diagnóstico molecular, pueden abordar la posible interferencia de microorganismos no objetivo.
- Productos de pollo listos para cocinar
- Adquiera productos de pollo listos para cocinar en la sección de alimentos congelados de los minoristas locales. Dividir asépticamente en muestras de 25 g.
- Recoja muestras del centro de las piezas individuales para asegurarse de que todos los ingredientes (por ejemplo, empanizado y queso) estén incluidos.
- Carne fresca
- Preparación de los medios
- Prepare agua de peptona tamponada (BPW) disolviendo 25 g de polvo de BPW en 1 L de H2O nanopuro.
- Prepare placas de infusión cerebro-corazón (BHI). Para ello, disuelva 37 g de polvo de BHI en 1 L de nanopure H2O y añada 15 g de agar en la solución de BHI. Esterilice todos los medios esterilizándolos en autoclave a 121 °C durante 15 min. Vierta de 20 a 25 ml de medio en placas de Petri con tapa transparente (100 mm x 15 mm).
NOTA: Lo mejor es verter las placas dentro de una cabina de seguridad biológica para mantener las condiciones asépticas.
2. Cultivo celular
- Preparar el cultivo inicial rayando Salmonella enterica serovar Typhimurium ATCC 14028 en una placa de agar BHI e incubar a 37 °C durante la noche.
- Prepare cultivos durante la noche inoculando 25 mL de caldo BHI con una colonia de Salmonella recién cultivada. Cultivo aeróbico durante la noche a 37 °C con agitación a 100 rpm.
3. Inoculación de muestras de aves de corral
- Dilución y recubrimiento de cultivos
- Prepare una serie de diluciones 10 veces del cultivo nocturno en BPW para lograr concentraciones finales de aproximadamente 1 x 108 a 1 x 101 UFC /mL. Supongamos que la concentración del cultivo nocturno de Salmonella es de 1 x 109 UFC/mL.
- Transfiera 0,5 mL del cultivo nocturno a 4,5 mL de BPW, mezcle, luego transfiera 0,5 mL de la dilución a 4,5 mL de BPW por cada dilución adicional.
- Extienda 10 μL de la dilución de 1 x 103 UFC/mL en una placa de agar BHI por triplicado para la enumeración de células para calcular la concentración de cultivo durante la noche.
- Inoculación de muestras de carne
- Transfiera asépticamente 25 g de pollo molido irradiado a una bolsa estomacal estéril por duplicado. La bolsa mide 7,5 x 12 pulgadas y está etiquetada como 1,63 L. La bolsa contiene una partición de filtro con un diámetro de orificio de 330 μm, y hay 285 por cm cuadrado.
- Inocular cada muestra con 1 mL de la concentración objetivo de dilución. Por ejemplo, agregue 1 mL de cultivo de la dilución de 1 x 10³ UFC/mL para lograr un nivel de contaminación de aproximadamente 1,000 células/25 g de pollo. Distribuya suavemente el inóculo líquido sobre la superficie de las muestras de pollo utilizando un esparcidor de células estéril y déjelo reposar durante 1 h a 4 °C.
- Prepare muestras de control negativas agregando 1 mL de BPW estéril.
4. Procesamiento de muestras
- Agregue 225 mL de BPW a cada muestra. La relación entre el volumen y el medio se seleccionó para alinearse con FSIS MLG 4.154.
- Homogeneizar las muestras utilizando el Stomacher7 a velocidad normal y una duración de 120 s.
- Centrifugación y resuspensión
- Retire con cuidado el líquido del lado filtrado de la bolsa con una pipeta de 50 ml. Divida el líquido en dos botellas de centrífuga estériles.
- Equilibre los frascos con BPW estéril para garantizar pesos iguales. Centrifugar las muestras a 10.000 x g durante 10 min. Deseche el sobrenadante. Vuelva a suspender el pellet celular en 3 mL de caldo BPW con una espátula estéril.
- Agregue 27 ml adicionales de caldo BPW y mezcle bien revolviendo con una espátula. Combine el contenido de ambos frascos de centrífuga en un frasco para cada muestra.
5. Configuración de bloques MPN
NOTA: La Tabla 1 muestra un esquema de las diluciones en un bloque de 48 pocillos.
- Agregue 3 mL de la muestra resuspendida a cada pocillo en la columna 1 del bloque de 48 pocillos (8 repeticiones).
- Prepare una serie de diluciones de 10 veces en las columnas 1-6 dentro del bloque utilizando una pipeta de ocho canales.
- Añada 0,3 mL de muestra en 2,7 mL de pipeta BPW para mezclar. Repita para cada dilución. Incubar los bloques durante la noche (~18 h) a 37 °C con agitación a ~100 rpm.
6. Enchapado y enumeración
- Enumeración de placa de caída modificada
- Placa de 7 μL de muestra cultivada durante la noche de cada dilución en una rejilla de 4 x 6 en una placa de agar utilizando una pipeta multicanal (Figura 2). El uso de una cuadrícula de 4 x 6 en dos placas acomoda mejor 8 muestras, a diferencia de la típica cuadrícula de 6 x 6 de gotas8.
- Deje que las placas se sequen al aire durante 10 minutos antes de la incubación. Incubar las placas de agar durante la noche (~18-24 h) a 37 °C. Después de la incubación, cuente el número de colonias en cada plato.
7. Detección de Salmonella por qPCR
- Extracción de ADN con un kit comercial
- Mezcle los cultivos en el bloque de 48 pocillos pipeteando hacia arriba y hacia abajo varias veces. Pipetear 200 μL de cada cultivo en una placa de PCR de 96 pocillos.
- Selle y luego centrifugue la placa a 6.600 x g durante 10 min. Retire el sobrenadante y añada 20 μL del reactivo del kit al pellet.
- Vuelva a suspender el pellet pipeteando hacia arriba y hacia abajo. Selle y caliente la placa a 99 °C durante 10 minutos, seguido de un enfriamiento a 20 °C.
- Vuelva a centrifugar a 6.600 x g durante 10 min. Utilice 2 μL del sobrenadante para el análisis de qPCR.
- Configuración de la placa
- Prepare la mezcla de reacción de qPCR de acuerdo conel protocolo 9 establecido de la siguiente manera: 10 μL de 2x Master Mix; 0,4 μL de cada cebador y sonda (10 μM de solución de trabajo): invA adelante: 5'-GTTGAGGATGTTATTCGCAAAGG-3', invA reverso: 5'-GGAGGCTTCCGGGTCAAG-3', sonda invA: 5'-CCGTCAGACCTCTGGCAGTACCTCTC-3' etiquetados con el colorante de fluorenos Cal Fluor Orange 560; 0,2 μL de plantilla de control de amplificación interna (IAC) (6 x 104 copias/μL)9; 0,4 μL de cada cebador y sonda IAC (10 μM): IAC hacia adelante: 5'-GGCGCGCCTAACACATCT-3', IAC hacia atrás: 5'-TGGAAGCAATGCCAAATGTA-3', sonda IAC: 5'-TTACAACGGGAGAAGACAATGCCACCA-3' marcada con colorante TAMRA. Ajuste el volumen con ddH2O a 20 μL en total.
- Realice PCR en tiempo real con las siguientes condiciones de ciclo9: 95 °C durante 10 min (desnaturalización inicial del ADN y activación de la polimerasa de arranque en caliente), 40 ciclos de 95 °C durante 15 s y 60 °C durante 1 min, utilice la configuración predeterminada de Ct para exportar los resultados para el análisis.
8. Detección mediante el ensayo MDS de 3M
- Siga el protocolo del kit de ensayo de detección molecular de Salmonella . Mezcle los cultivos en el bloque de 48 pocillos pipeteando hacia arriba y hacia abajo varias veces. Pipetear 20 μL de cada muestra en el tubo de lisis suministrado por el kit.
- Calentar las muestras a 100 °C durante 15 min. La solución cambiará de rosa a amarillo. Incubar las muestras durante 10 min a temperatura ambiente. La solución cambiará de amarillo a rosa.
- Transfiera 20 μL de lisado a un tubo de reactivo y cargue los tubos de reactivo en el soporte.
- Agregue el soporte al instrumento MDS y configure el software para comunicar la información sobre el kit y la muestra. El instrumento requiere que cada pocillo esté etiquetado con el número de lote del ensayo y un nombre de muestra. Ejecute el software MDS y exporte el informe.
9. Análisis de datos
- Clasificación de resultados positivos y negativos.
- Para el recubrimiento de 4 x 6 gotas, evalúe las manchas en las placas de agar con al menos 1 colonia como positivas y las manchas en las placas de agar sin crecimiento como negativas.
- Para la qPCR, evalúe los pocillos que tienen un Ct menor o igual a 30 como positivos y los pocillos que tienen un Ct mayor que 30 como negativos.
- En el caso de MDS, utilice los resultados del sistema MDS, notificados como positivos o negativos.
- Cálculo de MPN
- Analice los positivos y negativos anotados utilizando el método de resolución de probabilidad máxima simple (SMPR) descrito anteriormente 6 o calculadoras MPN verificadas alternativas. 10
Resultados
Carne irradiada
En el análisis de regresión, una pendiente de 1 indica que por cada unidad de aumento en la variable independiente (eje x), la variable dependiente (eje y) aumenta exactamente 1 unidad. Esto sugiere una relación proporcional entre las dos variables, lo que significa que el cambio en la variable dependiente refleja el cambio en la variable independiente. Una intersección con el cliente 0 significa que cuando la variable independiente es 0, la variable dependiente también es 0. Esto sugiere que no hay un desplazamiento fijo o un sesgo en la relación entre las dos variables. Juntas, una pendiente de 1 y una intersección de 0 sugieren concordancia entre las variables11,12. Este escenario ideal no indica ningún error sistemático o sesgo y sería el resultado esperado en un sistema bien calibrado en el que las mediciones se alinean perfectamente con las predicciones.
La Figura 3A presenta el ensayo MPN-LAMP (eje Y) trazado contra el inóculo teórico verificado por el análisis de la placa extendida discutido en la nota en el paso 3.1.1. (eje x). La ecuación de la línea de regresión, y = -72,36 + 0,161x, con un valor de R² de 0,978, indica una fuerte correlación lineal entre los resultados de MPN-LAMP y los niveles de bacterias inoculadas. El alto valor de R² sugiere que el ensayo MPN-LAMP es altamente confiable en la predicción cuantitativa de la contaminación bacteriana. Sin embargo, la pendiente de 0,161 se desvía significativamente de la pendiente ideal de 1, lo que indica que el método MPN-LAMP subestima el inóculo, prediciendo solo el 16,1% de concentración bacteriana en el inóculo teórico. Además, la intersección negativa de -72.36 sugiere un sesgo negativo a concentraciones más bajas, desviándose aún más del ideal.
La Figura 3B ilustra el método MPN-qPCR (eje y) trazado contra el inóculo teórico (eje x). La línea de regresión, y = -46,84 + 0,0395x, con un valor de R² de 0,978, demuestra una fuerte relación lineal entre el ensayo MPN-qPCR y el inóculo teórico. Aunque el alto R² indica una correlación robusta, la pendiente de 0,0395 es incluso menor que la de los resultados de MPN-LAMP, lo que sugiere un mayor grado de subestimación en la contaminación bacteriana por MPN-qPCR. La intersección negativa de -46.84 indica además un posible sesgo a la baja a concentraciones más bajas.
En la Figura 3C se presentan los resultados de la siembra de NMP (eje y) trazados contra el inóculo teórico (eje x), con una ecuación de regresión de y = -330,4 + 0,3138x y un valor de R² de 0,944. Si bien este valor de R² indica una fuerte relación lineal, es ligeramente inferior a los de los resultados de MPN-LAMP y MPN-qPCR, lo que sugiere una mayor variabilidad en la cuantificación de colonias. La pendiente de 0,3138, aunque mayor que las pendientes observadas en los otros métodos, todavía indica una subestimación de la concentración bacteriana en relación con el inóculo teórico. Además, la intersección negativa de -330.4 apunta a un sesgo considerable, especialmente a concentraciones más bajas, lo que puede indicar posibles imprecisiones en las técnicas de siembra o en el manejo de muestras. Esta ligera reducción del R² y la variabilidad observada podrían indicar que el proceso de recubrimiento tradicional es más variable que los métodos moleculares.
Métodos de comparación
Los tres métodos (MPN-LAMP, MPN-plating y MPN-qPCR) mostraron una tendencia a subestimar los niveles bacterianos en relación con la inoculación teórica. Esta subestimación puede atribuirse a problemas de recuperación celular, en los que la Salmonella puede no desprenderse completamente de la superficie del pollo o de las partículas durante el procesamiento, lo que reduce su transferencia al enjuague o al ensayo. Dado que la Salmonella se inoculó directamente sobre el pollo, una porción de células bacterianas puede unirse fuertemente a la superficie, lo que limita la recuperación en el paso de enjuague. Además, algunas células pueden permanecer adheridas a las partículas en el gránulo después de la centrifugación, lo que reduce aún más la recuperación. Enfoques como los tratamientos enzimáticos, el procesamiento mecánico o los protocolos optimizados de estómago podrían mejorar el desprendimiento bacteriano. Estas estrategias se discuten más adelante en la sección de discusión, enfatizando su potencial para mejorar las tasas de recuperación
La Figura 4A presenta la recuperación en el eje Y frente a tres métodos MPN en el eje X. El porcentaje de recuperación, calculado como la relación entre el valor de MPN y el nivel teórico de inoculación, arrojó los siguientes resultados: el método MPN-LAMP tuvo una recuperación media de 15,19% con una desviación estándar de 10,04%; el método de recubrimiento MPN tuvo una recuperación media de 13,13% con una desviación estándar de 11,45%; y el método MPN-qPCR tuvo una recuperación media del 6,67% con una desviación estándar del 3,19%. Una prueba de HSD de Tukey-Kramer no identificó una diferencia estadísticamente significativa en la recuperación entre los métodos (P > 0.1969), lo que sugiere que los tres métodos tuvieron un desempeño similar en términos de recuperación cuando se procesó pollo inoculado con Salmonella.
A pesar del tratamiento idéntico en todos los métodos para separar Salmonella de la matriz de pollo, se observaron variaciones en la detección entre MPN-LAMP, MPN-qPCR y MPN-plateado. Esto sugiere que factores más allá de la separación física de las bacterias de la muestra pueden afectar la detección. La presencia de inhibidores en la matriz de la muestra podría interferir con los métodos moleculares (qPCR y LAMP), afectando su sensibilidad y rendimiento general. Además, la viabilidad bacteriana puede haber afectado la capacidad de Salmonella para crecer en placas de agar sólido (placas de MPN) en comparación con el caldo líquido (qPCR, LAMP), lo que lleva a variaciones en la recuperación entre los métodos.
Para explorar más a fondo la sensibilidad de los métodos, primero examinamos el número de respuestas positivas en cada dilución para MPN-LAMP, MPN-qPCR y MPN-plating en siete ensayos independientes (Tabla 2). A diluciones más bajas (1 x 10-5 y 1 x 10-4), MPN-LAMP demostró un mayor número de respuestas positivas en comparación con la placa MPN y la MPN-qPCR, lo que sugiere que el método LAMP es más sensible a bajas concentraciones de Salmonella. Cuando las muestras estaban más concentradas (diluciones menores que 1 x 10-3), los tres métodos mostraron un aumento en las detecciones positivas, con convergencia en torno a la dilución 1 x 10-2 , donde todos los métodos detectaron un número similar de muestras positivas.
La Figura 4B presenta el porcentaje de mediciones positivas en cada dilución en los tres métodos, proporcionando una comparación directa de su sensibilidad en la detección de Salmonella. El porcentaje de respuestas positivas ofrece una visualización más clara del rendimiento del método en toda la serie de dilución, y las barras de error que representan ± intervalos de confianza (IC) de Clopper-Pearson del 95% proporcionan información sobre la variabilidad en cada nivel. A diluciones más bajas, los intervalos de confianza más amplios indican una mayor variabilidad en el rendimiento de detección de cada método. De acuerdo con el número bruto de detecciones positivas, MPN-LAMP detecta un mayor porcentaje de muestras positivas en las concentraciones más diluidas (1 x 10-5), mientras que MPN-qPCR y MPN-plateing detectan menos positivos. A medida que aumenta la concentración bacteriana, los métodos convergen, y los tres enfoques muestran una detección cercana al 100% a concentraciones más altas (por ejemplo, 1 x 10-2 y más). El estrechamiento de los intervalos de confianza a estas concentraciones más altas indica una mayor consistencia en todos los métodos, enfatizando su rendimiento comparable cuando los niveles bacterianos son más altos.
Los hallazgos de la Figura 3 sugieren que la subpredicción constante (cuantitativamente) de los tres métodos podría deberse a la separación incompleta de Salmonella de la matriz de la muestra. Sin embargo, los inhibidores moleculares o los factores de viabilidad bacteriana podrían estar impulsando la variación observada en la detección a diferentes diluciones (cualitativamente) y a través de diferentes métodos (cualitativa y cuantitativamente). Los intervalos de confianza más ajustados a concentraciones más altas en la Figura 4B indican menos variabilidad y un rendimiento cualitativo más consistente entre los métodos, mientras que los intervalos más amplios a concentraciones más bajas apuntan a una mayor incertidumbre en la detección cualitativa.
Detección en pollo listo para cocinar
Seleccionamos el método LAMP para la detección de Salmonella en pollo listo para cocinar, ya que mostró un mayor número de muestras positivas (14,3%) que otros métodos probados con la dilución más baja de Salmonella . Con la dilución más baja de Salmonella (1 x 10-5), ninguna de las muestras se detectó como positiva mediante el método de qPCR interno. Por otro lado, se detectaron algunos positivos por enchapado (7,1%, la mitad de lo detectado por LAMP). Sin embargo, los resultados de este método basado en el cultivo pueden verse afectados cuando se pasa a productos más complejos, como el pollo listo para cocinar, donde la presencia de microflora de fondo podría sesgar los resultados porque las muestras no se irradian. Por estas razones, decidimos investigar el método MPN-LAMP para la cuantificación de Salmonella a partir de pollo listo para cocinar.
La Figura 5 muestra los resultados del ensayo MPN-LAMP utilizado para cuantificar Salmonella en muestras de cordon bleu de pollo inoculadas, congeladas y listas para cocinar. El nivel teórico de inoculación (UFC/g) se graficó en el eje x, mientras que las mediciones correspondientes de MPN-LAMP se trazaron en el eje y. El análisis de regresión arrojó la ecuación y = -15.04 + 0.3869x, con un coeficiente de determinación (R²) de 0.933, lo que indica una fuerte correlación lineal entre el inóculo teórico y los resultados de MPN-LAMP. Este alto valor de R² sugiere que el método MPN-LAMP cuantifica de forma fiable la Salmonella en estas muestras, aunque la pendiente de 0,3869 indica que el método subestima el nivel real de inóculo. El área sombreada alrededor de la línea de regresión representa el intervalo de confianza del 95%.
Para ilustrar mejor el rendimiento de MPN-LAMP a niveles de inoculación más bajos, el gráfico inserto de la Figura 5 se centra en los puntos de datos por debajo de 300 UFC/g. Sobre la base de la ecuación de regresión, el límite inferior de detección puede estimarse en aproximadamente 40 UFC/g (38,9 UFC/g). Sin embargo, el intervalo de confianza del 95% sugiere que el límite inferior práctico de detección está más cerca de 325 UFC/g, ya que cualquier valor negativo de MPN-LAMP debe interpretarse como cero. La línea roja en el recuadro representa cero, y aunque ningún punto de datos cayó por debajo de esta línea, la posibilidad de que los valores caigan por debajo de cero, particularmente en niveles de inóculo más bajos, introduce la posibilidad de resultados falsos negativos. Esta tendencia pone de manifiesto las dificultades de cuantificar con precisión la Salmonella a bajas concentraciones, y se necesitarían datos adicionales para establecer definitivamente el límite inferior de detección.

Figura 1: Diagrama de bloques del ensayo de NMP modificado para la detección de Salmonella. Una descripción esquemática del ensayo del número más probable (MPN) modificado. Describe los pasos clave del proceso, comenzando con la inoculación de muestras de pollo molido irradiadas, seguidas de la homogeneización de la muestra, la centrifugación, la distribución de alícuotas y la incubación en condiciones específicas. La figura incluye fotografías del bloque de 48 pozos utilizado para la dilución y el crecimiento replicado, junto con tres técnicas utilizadas para producir resultados positivos y negativos para el cálculo de la Resolución de Máxima Probabilidad Simple (SMPR). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Esquema del ensayo de la placa de caída. La configuración del ensayo de placa de caída para una dilución específica (por ejemplo, sin diluir, 10-1, 10-2, etc.) del bloque de 48 pocillos. Cada muestra se coloca en un formato de cuadrícula de 4 x 6 en una placa de agar utilizando una pipeta multicanal. El diseño se modificó de la cuadrícula tradicional de 6 x 6 a dos cuadrículas de 4 x 6 para garantizar que se pudieran acomodar seis réplicas de ocho diluciones. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Comparación del inóculo con los métodos MPN. (A) Un gráfico de la concentración de MPN-LAMP (eje y) frente al inóculo teórico verificado por siembra (eje x). Una línea de regresión ajustada (y = -72,36 + 0,161x) con un coeficiente de determinación (R² = 0,978) demuestra una fuerte relación lineal. (B) Un gráfico de la concentración de MPN-qPCR (eje y) contra el inóculo teórico (eje x), con una línea de regresión ajustada (y = -46,84 + 0,0395x) y R² = 0,978, que muestra una correlación igualmente fuerte. (C) Un gráfico de la concentración de MPN-plating (eje y) frente al inóculo teórico (eje x), con una línea de regresión (y = -330,4 + 0,3138x) y R² = 0,944, lo que indica una correlación fuerte pero ligeramente menor que LAMP y qPCR. La ecuación de regresión y el coeficiente de determinación para cada figura se presentan como un inserto en la esquina superior izquierda de los gráficos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Comparación de métodos MPN. (A) La figura presenta el porcentaje de recuperación, calculado como la relación entre el valor de MPN y el nivel teórico de inoculación para los métodos MPN-LAMP, MPN-plating y MPN-qPCR. El método MPN-LAMP tuvo una recuperación media del 15,19% (±10,04%), el método de siembra de MPN tuvo una recuperación media del 13,13% (± 11,45%) y el método MPN-qPCR tuvo una recuperación media del 6,67% (± 3,19%). Los diagramas de caja resaltan la distribución de los datos de recuperación, con los valores atípicos indicados. Las barras de error representan el rango de datos dentro de 1,5 veces el rango intercuartílico (IQR). (B) Porcentaje de detecciones positivas de Salmonella en cada dilución a través de tres métodos: MPN-LAMP (azul), MPN-plateado (rojo) y MPN-qPCR (verde). El eje x representa los niveles de dilución (10-6 a 10) y el eje y muestra el porcentaje de mediciones positivas. Las barras de error indican los intervalos de confianza de Clopper-Pearson ± 95%, que reflejan la variabilidad en la detección para cada método. MPN-LAMP muestra porcentajes más altos de detecciones que otros métodos a diluciones más bajas, mientras que todos los métodos convergen cerca del 100% de detección a concentraciones más altas. Haga clic aquí para ver una versión más grande de esta figura.
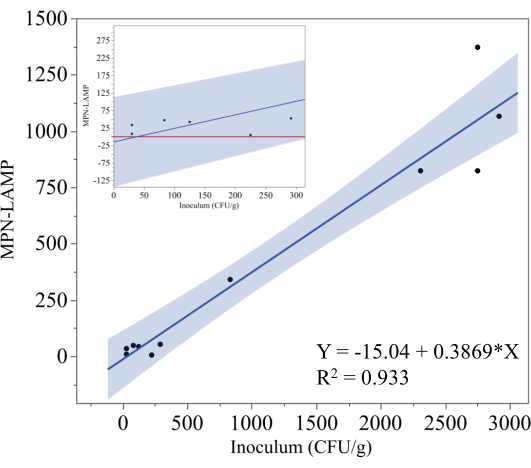
Figura 5: Método MPN para pollo listo para cocinar. Análisis de regresión de los resultados de MPN-LAMP frente al nivel teórico de inoculación de Salmonella en muestras de cordon bleu de pollo congeladas listas para cocinar. La línea de regresión está representada por y = -15,04 + 0,3869x con un R² de 0,933, lo que indica una fuerte relación lineal. La región sombreada representa el intervalo de confianza del 95%. El recuadro se centra en las mediciones de MPN-LAMP a niveles de inoculación más bajos. Haga clic aquí para ver una versión más grande de esta figura.
| 1 | 2 | 3 | 4 | 5 | 6 | |
| Un | n=1 | n=1 | n=1 | n=1 | n=1 | n=1 |
| B | 2 | 2 | 2 | 2 | 2 | 2 |
| C | 3 | 3 | 3 | 3 | 3 | 3 |
| D | 4 | 4 | 4 | 4 | 4 | 4 |
| E | 5 | 5 | 5 | 5 | 5 | 5 |
| F | 6 | 6 | 6 | 6 | 6 | 6 |
| G | 7 | 7 | 7 | 7 | 7 | 7 |
| H | 8 | 8 | 8 | 8 | 8 | 8 |
| 100 | 10-1 | 10-2 | 10-3 | 10-4 | 10-5 |
Tabla 1: Esquema de réplicas y diluciones en un bloque de 48 pocillos. Esta figura ilustra la disposición de las réplicas y las diluciones en serie de 10 veces dentro del bloque de 48 pocillos. Cada columna representa un factor de dilución específico, que va de 100 a 10-5 con réplicas técnicas para cada dilución distribuidas en las ocho filas.
| Dilución | ||||||
| 10-5 | 10-4 | 10-3 | 10-2 | 10-1 | 100 | |
| MPN-LÁMPARA | 8 | 16 | 31 | 3 | 56 | 56 |
| Recubrimiento MPN | 4 | 11 | 32 | 55 | 56 | 56 |
| MPN-qPCR | 0 | 7 | 26 | 55 | 56 | 56 |
Tabla 2: Número de eventos positivos. La tabla muestra el número de reacciones positivas (más de 56 réplicas en total). Los resultados se organizan en una tabla en la que las columnas representan la dilución y las filas son representativas del método.
Discusión
Importancia del protocolo
La salmonela sigue siendo una preocupación importante en la inocuidad de los alimentos, particularmente dentro de los productos avícolas, que a menudo están implicados en brotes de enfermedades transmitidas por los alimentos13,14. Como una de las principales causas de enfermedades bacterianas transmitidas por los alimentos en los Estados Unidos, los métodos confiables para detectar Salmonella en productos avícolas frescos y listos para cocinar son fundamentales para garantizar la inocuidad de los alimentos15. La capacidad de cuantificar la Salmonella a niveles bajos es vital para identificar la contaminación que, de otro modo, podría pasar desapercibida. La sensibilidad del método es particularmente importante para el cumplimiento regulatorio y la salud pública, ya que incluso dosis bajas de Salmonella pueden provocar enfermedades, especialmente en poblaciones vulnerables.
El ensayo MPN-LAMP utiliza el sistema MDS, que actualmente es utilizado por FSIS en MLG 4.15, para determinar la prevalencia de contaminación4. El ensayo presentado aquí proporciona una extensión del método actual del FSIS para cuantificar Salmonella viable, incluso a niveles bajos, proporcionando datos más precisos sobre el nivel de contaminación. Los resultados de este estudio demostraron una fuerte correlación entre el método MPN-LAMP y los valores teóricos de inoculación, con un R² de 0,933 (Figura 5). Esto sugiere que el método es confiable para cuantificar la Salmonella en los alimentos, aunque se produjo cierta subestimación a los niveles más bajos de inoculación.
Pasos críticos
El protocolo para la cuantificación de Salmonella requiere precisión en la preparación de la muestra, la inoculación y el análisis molecular. La técnica aséptica adecuada, las diluciones precisas y el cumplimiento de las condiciones de incubación son fundamentales para la detección y enumeración confiables de Salmonella. Garantizar condiciones estériles durante la transferencia de muestras y medios de aves de corral es esencial para evitar la contaminación.
Igualmente importante es la homogeneización de las muestras durante el estómago, seguida de una cuidadosa resuspensión del pellet después de la centrifugación para separar las bacterias de la matriz alimentaria. El manejo adecuado durante estos pasos garantiza que las células viables se recuperen para su posterior análisis. El pipeteo preciso durante el proceso de dilución en serie para la configuración de MPN, junto con condiciones de incubación consistentes, es crucial para obtener resultados reproducibles.
Por último, los métodos de detección molecular, como la qPCR, requieren precisión y reproducibilidad en la extracción de ADN y en el ciclo de la qPCR para evitar falsos negativos. Aunque la extracción de ADN es inherentemente variable, los reactivos y procedimientos estandarizados garantizan la repetibilidad al minimizar la interferencia de los inhibidores y mejorar la recuperación del ADN. Estos pasos son fundamentales para confirmar la presencia de Salmonella y para identificar marcadores genéticos específicos que pueden ayudar a superar las limitaciones de los métodos tradicionales de siembra y enriquecimiento.
Posibles escollos y limitaciones
Viabilidad bacteriana
Uno de los principales desafíos de la implementación de un ensayo basado en el crecimiento, como el recubrimiento de MPN, es la viabilidad bacteriana, y esto es especialmente relevante para muestras complejas de pollo listas para cocinar. Los productos de pollo congelados, empanizados y rellenos, como el pollo cordon bleu, son particularmente propensos a las fluctuaciones de temperatura y a los períodos de almacenamiento prolongados, los cuales pueden estresar las células de Salmonella y hacer que el patógeno sea más difícil de detectar en ensayos basados en el crecimiento. Además, la presencia de ingredientes distintos del pollo (por ejemplo, queso, empanizado, jamón, especias, mantequilla) introduce una mayor complejidad en la matriz alimentaria y puede inhibir el crecimiento de Salmonella durante las fases de enriquecimiento, lo que complica la cuantificación precisa. Además, la Salmonella debe competir contra los microbios de fondo en los métodos basados en el cultivo16, lo que es especialmente relevante para las muestras listas para cocinar con múltiples ingredientes, lo que añade diversidad a la microbiota en relación con el pollo crudo. Por último, los factores estresantes de manipulación y procesamiento, como los antimicrobianos, pueden influir en la recuperación de Salmonella 17.
La viabilidad bacteriana puede haber afectado el rendimiento de los métodos probados en este estudio. Todos los métodos subestimaron las concentraciones de Salmonella a niveles bajos y tuvieron una mayor variabilidad de detección a concentraciones más bajas de patógenos (Figura 4A, B). Sin embargo, el método MPN-LAMP de base molecular superó a la placa MPN y a la MPN-qPCR en la detección cualitativa a niveles de inoculación más bajos. Los resultados enfatizan la necesidad de refinamiento a bajas concentraciones de patógenos.
Heterogeneidad de la muestra
Otra limitación importante es la heterogeneidad de las muestras de alimentos, lo que puede introducir variabilidad en los resultados de la detección de Salmonella . El protocolo fue diseñado para dispersar 30 mL de muestra homogeneizada, permitiendo la distribución de 8 réplicas como alícuotas de 3 mL (24 mL en total, 6 mL en exceso) a través del bloque. Si bien este enfoque conservador (que da como resultado una muestra adicional) tiene en cuenta las pérdidas potenciales, introduce el submuestreo, ya que no se analiza todo el volumen.
La heterogeneidad de la muestra de alimento puede afectar la precisión de la medición cuando se transfieren muestras de la mezcla redispersada al bloque y se realizan diluciones18,19. De acuerdo con la Teoría del Muestreo (TOS), los errores fundamentales de muestreo pueden reducirse aumentando la masa de la muestra o reduciendo la heterogeneidad de la composición a través de la reducción del tamaño de partícula20. Sin embargo, mientras que algunos estudios indican que la descomposición de la matriz por estómago no impacta significativamente la recuperación bacteriana de la contaminación superficial, se ha demostrado que la reducción del tamaño de partícula de la matriz alimentaria disminuye la recuperación bacteriana en algunos casos 21,22,23. En este estudio, el tamaño de partícula se redujo mediante el estómago antes del enriquecimiento; Si bien esto puede reducir el error de muestreo al reducir la heterogeneidad de la composición en las muestras, el estómago y la centrifugación pueden afectar la recuperación bacteriana.
Por último, la composición heterogénea y la morfología del alimento, así como si la contaminación es interna o superficial, puede dar lugar a variabilidad en la recuperación de patógenos. Trabajos previos 18,22 han demostrado que una fuerte unión bacteriana a las superficies de los tejidos puede reducir las tasas de recuperación. Los estudios en los que se utilizaron enzimas como la tripsina, la colagenasa y la endopeptidasa para liberar bacterias de las matrices de carne y avesde corral 24,25 y pectinasa y celulasa a las verduras de hoja verde19 demuestran el potencial para mejorar la recuperación de Salmonella de las matrices alimentarias.
Sugerencias para la solución de problemas
Para evitar problemas, asegúrese de que todos los medios y reactivos estén recién preparados y estériles. Los materiales caducados o esterilizados incorrectamente pueden provocar contaminación o afectar el crecimiento de Salmonella. Si se observa contaminación, verifique que la técnica aséptica se haya seguido de manera consistente durante todo el protocolo, particularmente durante la inoculación de la muestra, la dilución, la alícuota y la siembra.
Cuando los recuentos de colonias son más bajos de lo esperado, la verificación de la precisión de las diluciones en serie y las concentraciones de inóculo puede ayudar a identificar errores. Las condiciones de incubación, incluida la temperatura, el tiempo y la agitación, deben controlarse cuidadosamente para apoyar el crecimiento óptimo de Salmonella . En casos de crecimiento bajo o nulo, el estómago inadecuado o las fugas de las bolsas podrían contribuir a una distribución desigual de las bacterias.
Si la detección molecular produce resultados inconsistentes, la verificación de los procedimientos de extracción de ADN y la verificación de la calidad de los cebadores y sondas son pasos clave. Además, la variabilidad entre matrices de alimentos puede presentar diferentes niveles de inhibición, que pueden ser monitoreados utilizando el control de amplificación interna (IAC). El almacenamiento y la manipulación adecuados de los reactivos de extracción son cruciales para evitar falsos negativos debidos a la degradación o la contaminación. También pueden justificarse métodos de extracción de ADN más eficientes.
La incorporación de tratamientos como el sistema GentleMACS26, que emplea acciones mecánicas, térmicas y enzimáticas, puede optimizar aún más la recuperación de bacterias de los alimentos. Este sistema puede reducir la heterogeneidad de la matriz alimentaria, lo que facilita la transferencia de muestras uniformes al bloque y la realización de diluciones seriadas precisas.
Potencial de refinamiento y expansión
Hay varias oportunidades para refinar y expandir este protocolo. La exploración de formulaciones alternativas de medios de enriquecimiento podría mejorar la recuperación de las células de Salmonella estresadas o lesionadas subletalmente, particularmente de matrices alimentarias complejas como el pollo cordon bleu. Si bien el agar selectivo se usa comúnmente para atacar la Salmonella, las células que están lesionadas o estresadas debido a las condiciones de procesamiento y almacenamiento pueden provocar falsos negativos en dichos medios. Para superar este desafío, la optimización del equilibrio entre la presión selectiva y la recuperación microbiana podría mejorar la detección de serovares de Salmonella que son menos competitivos en condiciones de enriquecimiento estándar. Este refinamiento podría mejorar la sensibilidad general del ensayo, particularmente en productos alimenticios desafiantes donde el estrés microbiano es común. Acortar los tiempos de incubación podría acelerar el tiempo de obtención de resultados, aunque esto puede reducir la sensibilidad y limitar la detección. El trabajo futuro debe explorar avances en los medios de enriquecimiento para respaldar la replicación bacteriana rápida, asegurando una cuantificación precisa y cumpliendo con los requisitos de las partes interesadas.
Además, la investigación de diferentes temperaturas y duraciones de incubación para el ensayo MPN podría optimizar la recuperación de Salmonella de varios productos avícolas. Esto podría ampliar la aplicabilidad del protocolo a otros patógenos transmitidos por los alimentos que requieren condiciones de crecimiento distintas, ampliando así su utilidad en las pruebas de inocuidad de los alimentos. La automatización podría mejorar la escalabilidad de este protocolo mediante la integración de manipuladores de líquidos robóticos para los pasos de alícuota y dilución. Las centrífugas de flujo continuo o los sistemas de centrífugas de alta capacidad también pueden abordar las limitaciones del procesamiento por lotes, lo que permite aplicaciones de alto rendimiento en entornos industriales.
La ampliación del aspecto de la detección molecular para incluir la PCR multiplex podría permitir la detección simultánea de múltiples patógenos, mejorando el rendimiento en los entornos de vigilancia y prueba. Además, la incorporación de la secuenciación de próxima generación (NGS) podría proporcionar información valiosa sobre el serotipo y la virulencia directamente de los cultivos de enriquecimiento de NMP, lo que facilitaría las investigaciones epidemiológicas y los esfuerzos de seguimiento de la fuente.
Por último, el límite de cuantificación/detección puede ampliarse aumentando la cantidad de muestras utilizadas en el NMP. Por ejemplo, si el peso de la muestra de carne se incrementó de 25 g a 325 g, entonces el límite de detección podría extrapolarse a 4 UFC/g utilizando la ecuación de regresión lineal. Sin embargo, suponiendo el mismo intervalo de confianza relativo del 95%, el límite inferior práctico de cuantificación es >30 UFC/g.
Conclusión
El desarrollo del ensayo MPN-LAMP en este estudio ofrece una herramienta valiosa y práctica para cuantificar la contaminación por Salmonella en productos avícolas. Con una fuerte correlación con los niveles teóricos de inoculación (R² = 0,933), el método detecta de forma fiable la Salmonella a niveles bajos, lo que es fundamental para la seguridad alimentaria y el cumplimiento de la normativa. Esta capacidad es especialmente importante para detectar contaminación que, de otro modo, podría pasar desapercibida, lo que permite a los productores abordar de forma proactiva los riesgos potenciales. El ensayo MPN-LAMP proporciona una alternativa cuantitativa a los métodos tradicionales basados en la prevalencia, ya que ofrece un nivel de contaminación por Salmonella en una variedad de productos avícolas, incluidos los artículos listos para cocinar más complejos.
Una de las fortalezas clave de este método radica en su adaptabilidad a diferentes matrices de alimentos, particularmente productos congelados y procesados como el pollo cordon bleu, donde los métodos tradicionales pueden tener dificultades debido a la presencia de ingredientes adicionales como el empanizado y el queso. La integración de técnicas moleculares, como LAMP y qPCR, con el ensayo MPN garantiza un enfoque integral de la detección, superando los desafíos planteados por los microbios de fondo y las condiciones de procesamiento. Estos métodos moleculares mejoran la sensibilidad del protocolo, convirtiéndolo en una herramienta versátil en las pruebas rutinarias de seguridad alimentaria y la vigilancia de la contaminación, con el potencial de reducir el riesgo de brotes relacionados con Salmonella.
Si bien ningún método de detección está exento de limitaciones, el ensayo MPN-LAMP demuestra un rendimiento constante en una variedad de niveles de inóculo y matrices de alimentos. La variabilidad a niveles de inoculación más bajos se puede abordar a través de refinamientos futuros, como la optimización de la preparación de muestras y las formulaciones de los medios para maximizar la recuperación, pero esto no resta valor a la solidez general del protocolo. Al proporcionar una detección precisa de células de Salmonella , incluso en condiciones difíciles, el método MPN-LAMP equipa a la industria alimentaria con una herramienta invaluable para salvaguardar la salud pública.
De cara al futuro, hay margen para seguir mejorando, como ampliar el ensayo para incluir más serovares de Salmonella o incorporar la secuenciación de próxima generación para una mayor caracterización de los patógenos. No obstante, este protocolo ya representa un avance significativo en la detección y cuantificación de Salmonella , proporcionando un método práctico, eficiente y confiable que satisface las crecientes demandas de las pruebas de seguridad alimentaria.
Divulgaciones
Todos los autores declaran que no existe conflicto de intereses.
Agradecimientos
Esta investigación fue apoyada por el Departamento de Agricultura de los Estados Unidos, Servicio de Investigación Agrícola (USDA-ARS), Programa Nacional 108, Número del Sistema de Información de Investigación Actual 8072-42000-093-000-D y 8072-42000-094-000-D. La mención de nombres comerciales o productos comerciales en este artículo es únicamente con el propósito de proporcionar información específica y no implica una recomendación o respaldo por parte del Departamento de Agricultura de los Estados Unidos. El USDA es un proveedor y empleador que ofrece igualdad de oportunidades.
Materiales
| Name | Company | Catalog Number | Comments |
| 48 deep well block 4.6ml | Fisher Scientific International, Inc | NC1964628 | |
| Agar - Solidifying Agent (Difco) | Becton, Dickinson and Company (BD) | 281230 | |
| Analytical Balance | Mettler Toledo | JL602-G/L | Equipment |
| Analytical Balance | Mettler Toledo | AB54-S | Equipment |
| Autoclave - Amsco Lab250, Laboratory Steam Sterilizer | Steris plc | LV-250 | Equipment |
| Biological Safety Cabinet, Type A2, Purifier Logic+ | Labconco Corporation | 302411101 | Equipment |
| Brain Heart Infusion (BHI) Broth | Becton, Dickinson and Company (BD) | 237500 | |
| Buffered Peptone Water | Bio-Rad Laboratories Inc. | 3564684 | |
| Cell Spreader - L-shaped | VWR | 76208-438 | |
| Centrifuge Microcentrifuge 5424 | Eppendorf | 5424 | Equipment |
| Centrifuge, Avanti J-25 | Beckman Coulter, Inc. | Equipment | |
| DNA Extraction - PreMan Ultra Sample Preparation Reagent | Thermo Fisher Scientific Inc. | 4318930 | |
| Ground Chicken | Local retailers | ||
| IAC forward primer: 5'-GGCGCGCCTAACACATCT-3' | Integrated DNA Technologies | ||
| IAC probe: 5'-TTACAACGGGAGAAGACAATGC CACCA-3' labeled with 5' TAMRA/3' BHQ-2 | Biosearch Technologies | ||
| IAC reverse primer: 5'-TGGAAGCAATGCCAAATGTGTA-3' | Integrated DNA Technologies | ||
| Incubator - Inova 4230 incubator shaker | New Brunswick Scientific | 4230 | Equipment |
| Inoculating Loop - Combi Loop 10µL and 1µL | Fisher Scientific International, Inc | 22-363-602 | |
| invA forward primer: 5'-GTTGAGGATGTTATTCGCAAAG G-3' | Integrated DNA Technologies | ||
| invA probe: 5'-CCGTCAGACCTCTGGCAGTAC CTTCCTC-3' labeled with 5' Cal Fluor Orange 560/3' BHQ-1 | Biosearch Technologies | ||
| invA reverse primer: 5'-GGAGGCTTCCGGGTCAAG-3' | Integrated DNA Technologies | ||
| Irradiation Treatment | Texas A&M Agrilife Research National Center for Electron Beam Research | Service | |
| Luria Bertani (LB) Broth | Becton, Dickinson and Company (BD) | 244620 | |
| Manual pipette Pipet-Lite LTS Pipette L-1000XLS+ | Mettler Toledo | 17014382 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-100XLS+ | Mettler Toledo | 17014384 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-10XLS+ | Mettler Toledo | 17014388 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-200XLS+ | Mettler Toledo | 17014391 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-20XLS+ | Mettler Toledo | 17014392 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-200XLS+ | Mettler Toledo | 17013805 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-20XLS+ | Mettler Toledo | 17013803 | Equipment |
| Media Storage Bottle -PYREX 1L Square Glass Bottle, with GL45 Screw Cap | Corning Inc. | 1396-1L | Equipment |
| Media Storage Bottle -PYREX 2L Round Wide Mouth Bottle, with GLS80 Screw Cap | Corning Inc. | 1397-2L | Equipment |
| Microtiter plate, 96 well plate, flat bottom, polystyrene, 0.34cm2, sterile, 108/cs | MilliporeSigma | Z707902 | |
| Mixer - Vortex Genie 2 | Scientific Industries Inc. | SI-0236 | Equipment |
| Molecular Detection Assay 2-Salmonella kit | Neogen | MDA2SAL96 | |
| Molecular Detection Instrument | Neogen | MDS100 | Equipment |
| Motorized pipette controller, PIPETBOY2 | INTEGRA Biosciences Corp. | 155019 | Equipment |
| PCR Mastermix 2× TaqMan Gene Expression | Thermo Fisher Scientific Inc. | 4369542 | |
| Petri Dish Rotator - bioWORLD Inoculation Turntable | Fisher Scientific International, Inc | 3489E20 | Equipment |
| Petri Dishes with Clear Lid (100 mm x 15mm) | Fisher Scientific International, Inc | FB0875713 | |
| Pipette Tips GP LTS 1000µL S 768A/8 | Mettler Toledo | 30389273 | |
| Pipette Tips GP LTS 20µL 960A/10 | Mettler Toledo | 30389270 | |
| Pipette Tips GP LTS 200µL F 960A/10 | Mettler Toledo | 30389276 | |
| Ready to cook chicken products | Local retailers | ||
| Reagent Reservoir, 25 mL sterile reservoir used with multichannel pipettors | Thermo Fisher Scientific Inc. | 8093-11 | |
| Realtime PCR - 7500 Real-Time PCR system | (Applied Biosystems, Foster City, CA) | 2750036476 | Equipment |
| Serological Pipettes, Nunc Serological Pipettes (10 mL) | Thermo Fisher Scientific Inc. | 170356N | |
| Serological Pipettes, Nunc Serological Pipettes (2 mL) | Thermo Fisher Scientific Inc. | 170372N | |
| Serological Pipettes, Nunc Serological Pipettes (25 mL) | Thermo Fisher Scientific Inc. | 170357N | |
| Serological Pipettes, Nunc Serological Pipettes (50 mL) | Thermo Fisher Scientific Inc. | 170376N | |
| Spreader - Fisherbrand L-Shaped Cell Spreaders | Fisher Scientific International, Inc | 14-665-230 | |
| Stomacher bag, Nasco Whirl-Pak Write-On Homogenizer Blender Filter Bags | Thermo Fisher Scientific Inc. | 01-812 | |
| Stomacher 80 Biomaster Lab Blender | Seward | 30010019 | Equipment |
| Thermocycler (GeneAmp PCR system 9700) | Applied Biosystems | 535028293 | Equipment |
| Water Filtration - Elga Veolia Purelab Flex | Elga LabWater | PF2XXXXM1-US | Equipment |
| Whirlpak bags 1.63L | VWR | 11216-777 |
Referencias
- Batz, M., Hoffmann, S., Morris, J. G. Disease-outcome trees, eq-5d scores, and estimated annual losses of quality-adjusted life years (qalys) for 14 foodborne pathogens in the united states. Foodborne Pathogens and Disease. 11 (5), 395-402 (2014).
- . The Grand Challenge: Salmonella Available from: https://tellus.ars.usda.gov/stories/articles/the-grand-challenge-salmonella (2024)
- National Advisory Committee on Microbiological Criteria in Foods (NACMCF). Response to questions posed by the food safety and inspection service: Enhancing Salmonella control in poultry products. J Food Prot. 82 (4), 645-668 (2019).
- Food Safety and Inspection Service. . 4.15 Isolation and identification of Salmonella from meat, poultry, pasteurized egg, siluriformes (Fish) products and carcass and environmental sponges. , (2024).
- Irwin, P., Reed, S., Brewster, J., Nguyen, L., He, Y. P. Non-stochastic sampling error in quantal analyses for campylobacter species on poultry products. Analytical and Bioanalytical Chemistry. 405 (7), 2353-2369 (2013).
- Irwin, P., Tu, S., Damert, W., Phillips, J. A modified gauss-newton algorithm and ninety-six well micro-technique for calculating mpn using excel spreadsheets. Journal of Rapid Methods & Automation in Microbiology. 8 (3), 171-191 (2000).
- Ravishankar, S., Ahmed, E. Y., Carlstrom, C. Food microbiology: A laboratory manual. Food Microbiology. 21, 489 (2004).
- Chen, C. Y., Nace, G. W., Irwin, P. L. A 6 x 6 drop plate method for simultaneous colony counting and mpn enumeration of campylobacter jejuni, listeria monocytogenes, and escherichia coli. J Microbiol Methods. 55 (2), 475-479 (2003).
- Suo, B., He, Y., Tu, S. I., Shi, X. A multiplex real-time polymerase chain reaction for simultaneous detection of salmonella spp., escherichia coli o157, and listeria monocytogenes in meat products. Foodborne Pathogens and Disease. 7 (6), 619-628 (2010).
- Jarvis, B., Wilrich, C., Wilrich, P. T. Reconsideration of the derivation of most probable numbers, their standard deviations, confidence bounds and rarity values. J Appl Microbiol. 109 (5), 1660-1667 (2010).
- Stevens, R., Poppe, K. Validation of clinical prediction models: What does the "calibration slope" really measure. Journal of Clinical Epidemiology. 118, (2019).
- Miller, M. E., Hui, S. L., Tierney, W. M. Validation techniques for logistic regression models. Statistics in Medicine. 10 (8), 1213-1226 (1991).
- Galán-Relaño, &. #. 1. 9. 3. ;., et al. Salmonella and salmonellosis: An update on public health implications and control strategies. Animals. 13 (23), 3666 (2023).
- Gorski, L., et al. Growth assessment of salmonella enterica multi-serovar populations in poultry rinsates with commonly used enrichment and plating media. Food Microbiology. 119, 104431 (2024).
- Schmidt, J. W., et al. Evaluation of methods for identifying poultry wing rinses with salmonella concentrations greater than or equal to 10 cfu/ml. J Food Prot. 87 (11), 100362 (2024).
- Gorski, A., Liang, L. S. Effect of enrichment medium on real-time detection of salmonella enterica from lettuce and tomato enrichment cultures. Journal of Food Protection. 73 (6), 1047-1056 (2010).
- Guillén, S., Nadal, L., Álvarez, I., Mañas, P., Cebrián, G. Impact of the resistance responses to stress conditions encountered in food and food processing environments on the virulence and growth fitness of non-typhoidal salmonellae. Foods. 10 (3), 617 (2021).
- Rohde, A., Hammerl, J. A., Appel, B., Dieckmann, R., Al Dahouk, S. Sampling and homogenization strategies significantly influence the detection of foodborne pathogens in meat. BioMed Research International. 2015, (2015).
- Wang, D., Wang, Z., He, F., Kinchla, A. J., Nugen, S. R. Enzymatic digestion for improved bacteria separation from leafy green vegetables. Journal of Food Protection. 79 (8), 1378-1386 (2016).
- Pitard, F. F. . Theory of sampling and sampling practice. , (2019).
- Sharpe, A. . in Detecting pathogens in food. , 52-68 (2003).
- Hannah, J., et al. Effect of stomaching on numbers of bacteria recovered from chicken skin. Poultry Science. 90 (2), 491-493 (2011).
- Mcmeekin, T., Thomas, C. Retention of bacteria on chicken skin after immersion in bacterial suspensions. Journal of Applied Bacteriology. 45 (3), 383-387 (1978).
- Rodrigues-Szulc, U., Ventoura, G., Mackey, B., Payne, M. Rapid physicochemical detachment, separation and concentration of bacteria from beef surfaces. Journal of Applied Bacteriology. 80 (6), 673-681 (1996).
- Vibbert, H. B., et al. Accelerating sample preparation through enzyme-assisted microfiltration of salmonella in chicken extract. Biotechnol Prog. 31 (6), 1551-1562 (2015).
- Armstrong, C. M., et al. Use of a commercial tissue dissociation system to detect salmonella-contaminated poultry products. Analytical and Bioanalytical Chemistry. 416 (3), 621-626 (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados