Method Article
Hipokampal Piramidal Nöronlardaki Rolleri İçin Pozisyona Özgü GABAA Reseptör Alt Birimi Yanlış Algılama Varyantlarının Tanımlanması ve Sınıflandırılması
Bu Makalede
Özet
Bu çalışma, DNA'dan protein fonksiyonuna ve nöral davranışa kadar uzanan çok ölçekli bir çerçeve sunmaktadır. GABAA reseptör alt biriminde öngörülen patojenik mutasyonları araştırmak için yeni bir yaklaşım sunar ve patojenik olarak tahmin edilen epileptojenik mutasyonların ve proksimal mutasyonların CA1 piramidal nöron modeli üzerinde benzer etkiler üretebileceğini varsaymaktadır.
Özet
Epilepsi ile ilişkili genlerde fonksiyonel olarak bilinmeyen varyantların etkilerini anlamak, hastalık patofizyolojisinin aydınlatılması ve kişiselleştirilmiş terapötiklerin geliştirilmesi için çok önemlidir. DNA dizisinden protein fonksiyonuna ve nöral davranışa kadar uzanan çok ölçekli bir çerçeveyle, GABAA reseptör alt birimindeki epileptojenik mutasyonların ve yakındaki tahmin edilen mutasyonların CA1 piramidal nöron modeli üzerinde benzer etkiler üretebileceğini varsayarak, patojenik mutasyonları tahmin etmek ve araştırmak için yeni bir yaklaşım tanımlıyoruz. Çalışma, tahmin edilen patojenik mutasyonlar ile proksimal epileptojenik mutasyonlar arasındaki karakteristik ilişkileri araştırarak, epileptojenik mutasyonların hipokampal piramidal nöron simülasyonları üzerindeki etkilerine dayalı olarak tahmin edilen mutasyonların etkilerini tahmin etmeyi amaçlamaktadır.
Metodoloji, GABAA reseptörü γ2 alt birim genetik verilerinin toplanmasıyla başlar, ardından özel bir komut dosyası kullanılarak R'de gerçekleştirilen veri temizleme ve biçimlendirme ile devam eder. Daha sonra, γ2 alt biriminin patojenik yanlış anlamlı varyantlarını tanımlamak ve önceliklendirmek için topluluk tahmincileri uygulanacaktır. Epileptojenik mutasyonlar tarafından paylaşılan alt birim yapısal alanlara spesifik bir patojenik varyantın (tahmin edilen) haritalanması, etkilerinin moleküler modellemesi ve evrimsel korumanın değerlendirilmesi ile birlikte gösterilecektir. Daha sonra, varyanta özgü meta-analiz ve parametre normalizasyonu gerçekleştirilecek, ardından tahmin edilen mutasyonlar ile proksimal epileptojenik mutasyonlar arasındaki anlamlı ilişkileri belirlemek için korelasyon analizi yapılacaktır. Python tabanlı bir nöral simülatör kullanılarak, vahşi tip ve epileptojenik mutantların etkisini yansıtan çok bölmeli iletkenlik tabanlı nöron modeli açıklanacaktır. Epileptojenik GABA tarafından üretilen nöral tepkilerin simülasyonuBir reseptör alt tipi, tahmin edilen patojenik varyantların nöral yanıt üzerindeki etkisinin kaba tahmini için dikkate alınacaktır. Bildiğimiz kadarıyla, bu, epilepsi araştırmaları için çok önemli olan GABAA reseptör varyantlarının nöronal davranış üzerindeki etkilerini tahmin etmek için çok ölçekli bir çerçeveyi araştıran ilk protokoldür. Bu protokol, epilepsi ile ilişkili GABAA reseptörlerinin potansiyel olarak patojenik varyantlarının neden olduğu hücresel fenotiplerin tahminlerini geliştirmek için bir temel görevi görebilir.
Giriş
Neredeyse tüm insan hastalıkları için, genetik çeşitlilik bireysel duyarlılıkta önemli bir rol oynar. Bu nedenle, dizi varyasyonlarının hastalık riski ile nasıl ilişkili olduğunu anlamak, hastalık gelişiminde yer alan temel süreçleri ortaya çıkarmak ve önleme ve tedavi için yeni yaklaşımları belirlemek için değerli bir yol sunar1. Bu aynı zamanda pediatrik birinci basamakta en yaygın kronik tıbbi durumlar arasında yer alan nörogelişimsel bozukluklar için de geçerlidir2. Otizm spektrum bozukluğu, zihinsel engellilik ve epilepsi gibi durumlar, genetik çeşitliliğin gelişim sırasında bireysel duyarlılığı nasıl önemli ölçüde etkilediğini göstermektedir3.
Gelişmekte olan beyin, uyarılma ve inhibisyon arasındaki kritik dengede genetik olarak programlanmış nörogelişimsel uyumsuzluk nedeniyle epileptik nöbetlere yetişkin beyninden daha duyarlıdır4. Yetişkin beynindeki birincil inhibitör nörotransmitter olan GABA (gama-aminobütirik asit), embriyonik ve erken doğum sonrası gelişim sırasında uyarıcı olduğundan, bu genç beyinlerde nöbetleri önlemek için gereken stabilite için uygun değildir. K-Cl ko-taşıyıcılarının5 yeterli ekspresyonunun olmamasından kaynaklanan bu geçici durum, işlevsiz GABAA reseptörlerinin varlığında nöbet aktivitesi riskinin artmasına katkıda bulunabilir. GABAA reseptörleri, Cl-ion 6'nın hücre içi konsantrasyonuna bağlı olarak GABA'nın uyarıcı ve inhibitör etkilerine aracılık eder. Bu nedenle, beyin olgunlaştıkça, GABAA reseptörü kodlayan genlerdeki ve diğer iyon kanallarındaki mutasyonlar, uyarılabilirliği bozar ve nöronal metabolizma, hücre sinyalizasyonu vesinaps oluşumu 7 ile ilgili genlerdeki mutasyonlar, çocukluk çağı epilepsisi8 gibi durumlara neden olabilir.
Klinik müdahaleler, nörogelişimsel bozuklukların tedavisinde hassasiyeti artırmak için genetik analizden giderek daha fazla yararlanmaktadır2. Pediatrik epilepside genetik testler, tedavi kararlarını yönlendirmede genetik varyantların önemini vurgulayan hassas tıp yaklaşımları9 için potansiyel hedefler sunmaktadır. Ek olarak, de novo mutasyonları olan epilepsi hastalarının ~%25'i, hassas tıp için potansiyel hedefleri belirleyen genetik tanılar alır ve bu da tedavi kararlarına rehberlik etmede genetik varyantların önemli değerinin altını çizer10. Bu, genetik keşifleri önemli ölçüde hızlandıran hedeflenen gen panelleri, tüm ekzom dizilimi ve tüm genom dizilimi gibi yeni nesil dizileme teknolojilerindeki gelişmelerle desteklenmiştir11. Bununla birlikte, artan sayıda yeni gen keşfi, sonuçlar, varyantın hastalık patogenezindeki moleküler rolüne ilişkin çelişkili kanıtları veya yetersiz bilgiyi yansıtan bir sınıflandırma olan bilinmeyen öneme sahip bir varyant (VUS) verdiğinde bir zorlukla birlikte gelir. VUS olarak sınıflandırılan varyantlar, Amerikan Tıbbi Genetik ve Genomik Koleji (ACMG) ve Moleküler Patoloji Derneği (AMP) tarafından önerilen beş katmanlı varyant sınıflandırma sistemi içinde bir kategoriye karşılık gelir12.
İşlevsel olarak bilinmeyen genetik varyantların zorluğunu ele almak, iki temel boyutta çaba gerektirir: klinik uygulama ve araştırma. Klinik olarak, VUS'u çevreleyen belirsizlik hasta yönetimini ve karar vermeyi zorlaştırabilir13. Bilimsel araştırma perspektifinden bakıldığında, önemi belirsiz artan sayıda varyant arasında patojenik varyantların tanımlanması ve bunların hastalık patofizyolojisi ve fenotipik etkilerindeki rollerinin belirlenmesi çok önemlidir1. İdeal bir senaryo, işlevsel olarak karakterize edilmemiş tüm varyantların moleküler, nöronal ve ağ düzeyindeki etkilerini doğru bir şekilde tahmin etmeyi ve böylece laboratuvar tabanlı araştırmalar için gereken kaynakları, zamanı ve çabayı en aza indirmeyi içerir. Bu hususlar, genetik epilepsilerin kesin teşhisini sağlamak, kişiselleştirilmiş tedaviyi desteklemek ve potansiyel farmakolojik hedeflerin keşfedilmesini kolaylaştırmak için genetik varyantların doğru bir şekilde sınıflandırılmasının önemini vurgulamaktadır. Mevcut öngörücü araçlar 14,15,16,17 nispeten doğrudur, ancak tipik olarak yalnızca ikili sınıflandırmalar (patojenik ve iyi huylu) sağlar ve moleküler patofizyoloji, fenotipik sonuçlar ve altta yatan mekanizmalar hakkında hastalığa özgü içgörülerden yoksundur. Seçilmiş GABAA reseptör alt birimi kodlayan genlerin bilinmeyen yanlış anlamlı varyantlarına odaklanan bu makale, moleküler, evrimsel ve yapısal yönler gibi varyantların bağlamsal faktörlerinin yanı sıra epilepsi ile ilişkili mutasyonların in vitro biyofiziksel verilerinden türetilen nöral patoloji simülasyonlarını dahil ederek araştırma rehberliğini geliştirmeyi amaçlayan bir çerçeve sunmaktadır. Metodolojimiz, epilepsi 18,19,20'nin patofizyolojisinde yer alan önemli bir alt birim olan GABA A reseptörünün γ2 alt biriminin bilinmeyen patojenik varyantlarının tanımlanmasını ele almaktadır. Bunu, bu tahmin edilen varyantların, yapısal ve elektrofizyolojik verilerle karakterize edilen epilepsi ile ilişkili mutasyonlarla pozisyona özgü eşleşmesinin araştırılması takip eder. Bu veriler daha sonra, hızlı sinaptik inhibisyondan sorumlu γ2, α1 ve β3 alt birimlerinden (γ2-GABAA reseptörleri) oluşan bir GABAA reseptör alt tipini eksprese eden bir hipokampal piramidal nöron modeli üzerindeki varyant etkisini tahmin etmek için kullanılır6. GABAA reseptörlerinin büyük bir alt birim havuzundan (α1-α6, β1-β3, γ1-γ3, δ, Ε, θ, π ve ρ1-ρ3) bir araya geldiğini ve alt birim bileşimine bağlı olarak, GABAA reseptörlerinin modülasyonları, biyofiziksel özellikleri ve ayrıca bölgesel, hücresel ve hücre altı ekspresyon modellerinde farklılık gösterdiğini ve belirli işlevlerle birleştiğini not etmek önemlidir 6,21,22,23, 24,25. Bu nedenle, bu çalışma sadece γ2-GABAA reseptörlerine veya γ2 içeren GABAA reseptörlerine odaklanmaktadır.
GABAA reseptör alt birimleri, karakteristik yapısal özelliklerden oluşur - uzun bir N-terminal hücre dışı alan (ECD), dört transmembran yayılma alanı (TM1 ila TM4), TM1 ve TM2'yi bağlayan bir hücre içi bağlayıcı, TM2 ve TM3'ü bağlayan bir hücre dışı bağlayıcı, TM3 ve TM4 arasında büyük bir hücre içi döngü (TM3-TM4 döngüsü) ve kısa bir hücre dışı C terminali 6,26, 27. GABAA reseptörünün, GABA bağlanmasının β ve α alt birimlerini kilitlediği karmaşık bir "kilitleme ve çekme" mekanizması yoluyla işlev gördüğü, alt birimlerin hücre dışı alanlarını (ECD'ler) çekmelerine ve onları saat yönünün tersine döndürmelerine neden olduğu öne sürülmektedir27. Bu hareket, transmembran alanlarını (TMD'ler) bükerek iyon kanalını27 açar. Bu nedenle, kanal aktivitesinin GABAA reseptörleri içindeki yapısal kasetlerle birlikte koordine edildiği görülmektedir. Epilepsi mutasyonlarının bu yapısal kasetlerin bozulması yoluyla kanal aktivitesinde işlev bozukluğuna neden olduğu ortaya çıkmıştır28. Sonuç olarak, çalışmamız, GABAA reseptör alt birimlerinin spesifik yapısal kasetlerinde fonksiyonel olarak tanımlanmış epileptojenik mutasyonlara yakın olarak tahmin edilen patojenik varyantların, bu epileptojenik mutasyonlarda gözlemlendiği gibi, kanal fonksiyonunda benzer elektrofizyolojik veya biyofiziksel bozulma paternleri sergileyebileceği fikrine dayanmaktadır. GABAA reseptör alt birimlerinde28 epileptojenik yapısal kasetlerin varlığı bu görüşü dolaylı olarak desteklerken, çalışmamız epileptojenik mutasyonların biyofiziksel parametrelerini tahmin edilen patojenik mutasyonlarınkilerle ilişkilendirmenin karmaşıklığını ve zorluğunu göstermektedir. Bu karmaşık ilişkilerin maskesini düşürmek için, çerçevemiz DNA'dan protein fonksiyonuna ve epilepsi araştırmaları için kritik olan nöral davranışa kadar çok ölçekli bir yaklaşımı vurguladığı için önemlidir. Bu yaklaşım, hesaplamalı genetiği moleküler modelleme ve nöral simülasyonlarla bütünleştirirken, aynı zamanda mutasyonların kanal yapısı, aktivitesi ve nöral uyarılabilirlik üzerindeki etkilerini yakalayabilecek büyük veri kümeleri üzerinde eğitilmiş makine öğrenimi gibi tamamlayıcı yöntemlerin önemini vurgular. Ek olarak, hipokampal piramidal nöron modeli üzerinde epileptojenik γ2-GABAA reseptör aktivitesinin simülasyonu, GABAA reseptör kanalopatisi ile ilişkili in vitro hücresel fenotipin replikasyonuna ve ağ disfonksiyonunun merkezinde değişmiş tek nöron yanıtlarının gösterilmesine izin verir.
Protokol
1. Patojenik varyantların in silico tahmini
- Varyant veri toplama
- ClinVar veri tabanını29 kullanarak, ilgilenilen genin kodlama bölgesinde belirsiz öneme sahip varyantları (VUS) web sitesi aracılığıyla arayın: https://www.ncbi.nlm.nih.gov/clinvar/. Arama çubuğuna gen sembolünü (örneğin, GABRG2) girin ve sonuçları, yalnızca tek nükleotid, belirsiz öneme sahip yanlış anlamlı varyantlar gibi istenen varyant türlerini içerecek şekilde filtreleyin. Verileri data.xlxs olarak indirin ve kaydedin (Ek Dosya 4: Ek Tablo S1). İndirilen verilerin tarihini kaydedin.
NOT: Mevcut protokolde, GABAA reseptörünün insan γ2 alt birimi, özellikle Homo sapiens gama-aminobütirik asit tip A reseptör alt birimi gama2 (GABRG2), transkript varyantı 1, γ2L olarak da bilinen mRNA (NCBI Ref. sırası: NM_198904.4) analiz edilecektir. Farklı hesaplama yöntemleri farklı tanımlayıcılar gerektirebileceğinden, ilgilenilen genin referans transkriptinin yanı sıra diğer karşılık gelen tanımlayıcıların farklı veritabanlarına (UniProt, ENSEMBL, PDB) kaydedilmesi önemlidir (Ek Dosya 4: Ek Tablo S2). Veritabanının veya hesaplama aracının dizi tanımlayıcılarının sürüm numaralarını tanımaması durumunda, hem sürüm numarasıyla (NM_198904.4) hem de sürüm numarası olmadan (NM_198904) kimliği deneyin. - Referans protein temel bilgileri
- NCBI veritabanı https://www.ncbi.nlm.nih.gov/, arama seçeneklerinde Nucleotide'ı seçin ve ilgilenilen genin NCBI Ref. devamı ID'sini girin (NM_198904.4). Ardından, sağ sütunu aşağı kaydırarak, transkript NM_198904.4 tarafından kodlanan proteini (NP_944494.1) bulmak için İlgili bilgiler kategorisi altındaki Protein'e tıklayın. Protein NP_944494.1 için verilen bilgileri kullanarak, belirli bölgelerin dizi konumlarını bir tablo şeklinde kaydedin (Ek Dosya 4: Ek Tablo S3).
NOT: Protein alanları, fosforilasyon bölgeleri, ligand bağlanma bölgeleri ve moleküler etkileşim arayüzleri gibi işlevsel ve yapısal olarak kritik bölgelerin, motiflerin veya kalıntıların dizi konumu için bilinen ön bilgilerin belirlenmesi önemlidir. Bu, veri tabanı (NCBI, ENSEMBL, UniProt...) ve literatür taramalarının birleştirilmesiyle başarılabilir.
- NCBI veritabanı https://www.ncbi.nlm.nih.gov/, arama seçeneklerinde Nucleotide'ı seçin ve ilgilenilen genin NCBI Ref. devamı ID'sini girin (NM_198904.4). Ardından, sağ sütunu aşağı kaydırarak, transkript NM_198904.4 tarafından kodlanan proteini (NP_944494.1) bulmak için İlgili bilgiler kategorisi altındaki Protein'e tıklayın. Protein NP_944494.1 için verilen bilgileri kullanarak, belirli bölgelerin dizi konumlarını bir tablo şeklinde kaydedin (Ek Dosya 4: Ek Tablo S3).
- ClinVar veri tabanını29 kullanarak, ilgilenilen genin kodlama bölgesinde belirsiz öneme sahip varyantları (VUS) web sitesi aracılığıyla arayın: https://www.ncbi.nlm.nih.gov/clinvar/. Arama çubuğuna gen sembolünü (örneğin, GABRG2) girin ve sonuçları, yalnızca tek nükleotid, belirsiz öneme sahip yanlış anlamlı varyantlar gibi istenen varyant türlerini içerecek şekilde filtreleyin. Verileri data.xlxs olarak indirin ve kaydedin (Ek Dosya 4: Ek Tablo S1). İndirilen verilerin tarihini kaydedin.
- Varyant veri organizasyonu
- Verileri, seçilen tahminciler için giriş gereksinimlerini karşılayacak şekilde düzenleyin. Alınan verilerin biçiminin, dbNSFP sunucu http://database.liulab.science/dbNSFP gereksinimlerine uyacak şekilde düzenlendiğinden emin olun. Bunu yapmak için, data.xlsx dosyasından gereksiz sütunları kaldırın (Ek Dosya 4: Adım 1.1.1'deki Ek Tablo S1), yalnızca aşağıdaki sütunları belirtilen sırada tutun:
"GRCh38Kromozom", "GRCh38Konum", "İsim", "Protein değişimi". - Dosyayı yeni bir dosya adı altında kaydedin: "data1.xlsx" (Ek Tablo S4). Kodu çalıştırarak data1.xlsx dosyasını R'de biçimlendirin (Ek Dosya 1: Data_GABAA. R), biçimlendirilmiş verileri R projesiyle ilgili çalışma dizininde data1_output.xlsx (Ek Dosya 4: Ek Tablo S5) olarak kaydeder.
NOT: Farklı hesaplama yöntemleri, farklı veri türleri ve biçimleri gerektirir. Bir düzine varyant için bile belirli biçim gereksinimlerine göre veri toplamak ve düzenlemek hatalara açık ve zaman alıcı olabilir, bu nedenle varyant havuzu yalnızca birkaç varyanttan oluşmadığı sürece bu adım önemlidir. Ardından, manuel veri organizasyonu mümkün olabilir.
- Verileri, seçilen tahminciler için giriş gereksinimlerini karşılayacak şekilde düzenleyin. Alınan verilerin biçiminin, dbNSFP sunucu http://database.liulab.science/dbNSFP gereksinimlerine uyacak şekilde düzenlendiğinden emin olun. Bunu yapmak için, data.xlsx dosyasından gereksiz sütunları kaldırın (Ek Dosya 4: Adım 1.1.1'deki Ek Tablo S1), yalnızca aşağıdaki sütunları belirtilen sırada tutun:
- Patojenite tahmini
- data1_output.xlsx dosyasının içeriğini, http://database.liulab.science/dbNSFP aracılığıyla erişilen dbNSFP sunucusu 30,31'in akademik sürümüne aktarın. Bunu yapmak için dosyayı kopyalayın/yapıştırın veya doğrudan .txt biçiminde yükleyin.
- Aşağıdaki seçeneklerin sunucuda önceden seçildiğinden ve onaylandığından emin olun: Göndermeden önce HG38 (genom oluşturma), ClinPred32 ve BayesDEL33 . Birkaç dakika içinde sunucu sonuçları oluşturacaktır.
NOT: Mevcut protokolde, yüksek doğruluk34 ve pratiklik için BayesDEL33 ve ClinPred32 olmak üzere iki topluluk tahmincisi seçilmiştir. Bununla birlikte, dbNSFP veritabanında30,31 bulunan AlphaMissense gibi diğer öngörücüler de seçilebilir. In silico araçların seçimi, güçlü tahmin12 için yeterli sayıda hesaplama kanıtının oluşturulması da dahil olmak üzere çeşitli faktörlere bağlıdır. Çoklu tahmine dayalı algoritmaların analizini entegre eden topluluk tahmincileri bu amaca hizmet edebilir. - Çıktı dosyasını (.txt bir formatta) indirin ve data2.xlsx olarak kaydedin (Ek Dosya 4: Ek Tablo S6).
- Menüdeki filtre seçeneğine tıklayarak ve D için filtreleme yaparak her iki sütundaki konsensüs varyantlarını belirleyerek filtreleri data2.xlsx (Ek Dosya 4: Ek Tablo S6) ayarlayın. Bu, en patojenik varyantların listesini verecektir; kaydedin (Ek Tablo S6'daki Konsensüs sekmesine bakın [ Ek Dosya 4]).
- Varyant seçimi
- Konsensüs patojenik tahminleri arasında, literatürden elde edilen epileptojenik mutasyonların yakınlığındaki varyantları belirleyin. İkincisinin nöron modellemesi için uygun yapısal ve biyofiziksel parametrelere sahip olduğundan emin olun.
NOT: Bu adım keşif amaçlıdır ve aynı zamanda ilgilenilen proteinin yapısal, fizikokimyasal ve biyofiziksel parametreleri açısından araştırılmasıyla da ilgilidir. Bu çalışmada bu veriler Brünger ve ark.35 ve Guo ve ark.36'dan elde edilmiş ve epilepsi ile ilişkili mutasyonlar ile ilgili bir anket yapılmıştır. Ayrıca, bir seçenek olarak, AlphaMissense37 puanlarına dbNSFP veri tabanından30,31 tekrar eden adım 1.3'ten erişilmiştir (Ek Dosya 4: Ek Tablo S7). Daha fazla ayrıntı protokol bölümleri 2.1.1 ve 2.1.2'de ve Sonuçlarda verilmiştir (bakınız "Yapısal ve Biyofiziksel Parametreler için Kümeleme Varyantları"). - Temel görselleştirme için, seçilen GABRG2 gen mutasyonları bağlamında önceki adımdaki varyantları incelemek için Protter38 ((https://wlab.ethz.ch/protter/start/) ve HOPE39 (https://www3.cmbi.umcn.nl/hope/) sunucularını kullanın: P302L40 ve K328M (veya 39 kalıntı sinyal peptidi hariç tutulduğunda K289M41).
NOT: Muazzam karmaşıklık nedeniyle, varyant etkilerinin yapısal değerlendirmesi birden fazla analiz düzeyinde yapılmalıdır. Protter38 gibi araçlar, proteinin topolojik özellikleri bağlamında varyantların net bir şekilde görselleştirilmesine izin verecek ve HOPE39 gibi kullanıcı dostu sunucular, moleküler modelleme yoluyla varyant etkisi hakkında fikir verecektir. Ek olarak, ilgilenilen proteinin kapsamlı bir literatür taraması, epilepsi ile ilişkili mutasyonlar hakkındaki bilgileri tanımlamak ve entegre etmek için kritik öneme sahiptir. - Evrimsel koruma ve yapısal içgörülerin analizi
- Proteinlerin düzenlenmesi, görselleştirilmesi ve analizi için açık kaynaklı bir program olan Jalview 42,43,44'ü açın.
- Hizalama için dizileri içe aktarın. Üst menüdeki Dosya'ya tıklayın | Dizileri getirme; iletişim kutusunda veritabanını seçin (UniProt gibi); kimlikleri al sekmesine tıklayın; ve iletişim kutusunda açıklandığı gibi, insan ve diğer omurgalı türlerinden ilgilenilen genin (GABRG2) UniProt erişim kimliklerini girin: P18507, P22723, Q6PW52, A0A2I3TKX0, F1RR72, A0A8I3MDZ2, A0A8M1P4D6. Tamam'a tıklayın.
NOT: GABRG2 tarafından kodlanan proteinlerin UniProt erişim sayıları aşağıdaki gibidir: Homo sapiens için P18507 (P18507-2), Mus musculus için P22723, Pan troglodytes için A0A2I3TKX0, Sus scrofa için F1RR72, Canis familiaris için A0A8I3MDZ2 ve Danio rerio için A0A8M1P4D6. - İlgilenilen gene bağlı olarak, bazı diziler açıklanmayabilir; bu nedenle, daha iyi bir bağlamsal anlayış için ilgili bilgileri ve potansiyel homologları belirlemek için bir BLAST araması yapın. Bu durumda, istenen dizilerin birden çok dizi hizalamasını oluşturmak için Dosya menüsü altındaki Dizi Ekle/Buradan metin kutusu seçeneği aracılığıyla protein dizilerinin FASTA formatını yükleyin.
- Hizalama yüklendikten sonra, çoklu dizi karşılaştırması için görüntülenen dizileri gözlemleyin. Her satır bir diziyi temsil eder ve her sütun hizalamadaki bir konumu temsil eder. En iyi hizalama yöntemini belirlemek için farklı yaklaşımlar kullanın; örneğin, Sıra menüsündeki Web hizmetlerine tıklayın ve optimum hizalamaya izin veren T-Coffee'yi önceden ayarlanmış olarak çalıştır seçeneğini seçin.
- P18507 Homo sapiens (mevcut çalışmadaki referans dizisi) dizisine sağ tıklayın ve bunu referans dizisi olarak ayarlayın. Üst menüden Format'ı seçin ve ekrandaki tam hizalamanın görselleştirilmesi için Wrap'e tıklayın. Aynı Biçim menüsünde, belirli kalıntı numaralarının görselleştirmesini geliştirmek için yukarıdaki ölçeğe tıklayın. Görselleştirmeyi daha da geliştirmek için, Renk'e gidip farklı seçenekler belirleyerek renk şemalarını ayarlayın (örneğin, Renkli Renk, Kimyasal Özellik); Gerekirse yazı tipi boyutunu değiştirin.
- Menü çubuğunda Hesapla'ya tıklayın ve korunan bölgeleri vurgulamak için Konsensüsü otomatik hesapla'yı seçin.
- In-silico tahmin adımında tanımlanan ilgilenilen varyantların konumuna odaklanın ve belirli varyant pozisyonlarını inceleyin. Belirli kalıntılara sağ tıklayarak ve Ek Açıklama Ekle'yi seçerek açıklama ekleyin. Etiketi (ör. varyant kimliği) uygun renk koduyla yazın ve kaydedin.
NOT: Mevcut analizde, P302L (mor) ve A303T (kırmızı), bunları yapısal verilerle birlikte çoklu dizi hizalamasında görselleştirmek için seçilmiştir (bir sonraki bölüme bakın).
- Seçilen korunmuş kalıntıları gösteren tam proteinin üç boyutlu rekonstrüksiyonu
- Önceki adımdan elde edilen dosyada, referans dizisine (GABRG2 insan) sağ tıklayın ve 3B yapı verilerini seçin.
- Açılır menüden uygun yapısal verileri (7QNE, Chain C)26 belirleyin ve Jmol ile yeni yapı görünümü aç'ı seçin.
NOT: Bu, çoklu dizi hizalamasında seçilen kalıntıların, 3B kimyasal yapılar için açık kaynaklı Java tabanlı bir görüntüleyici olan Jmol tarafından yapısal verilere dahil edilmesine izin verecektir.
- Konsensüs patojenik tahminleri arasında, literatürden elde edilen epileptojenik mutasyonların yakınlığındaki varyantları belirleyin. İkincisinin nöron modellemesi için uygun yapısal ve biyofiziksel parametrelere sahip olduğundan emin olun.
2. Parametre seçimi ve biyofiziksel modelleme
- Varyanta özgü meta-analiz ve parametre normalizasyonu
- Elektrofizyolojik veri kanalı iletkenliği (gGABAA), deaktivasyon süresi (τdeaktivasyon), yükselme süresi (τyükselme) ve maksimum akım genliği (Imaks.) ile tanımlanmış alt birim varyantlarını toplamak için mevcut literatürü araştırın. Her durum için alt birim bileşimlerini, hücre tipini ve vahşi tip ölçümlerini sağlayın. Varyantları ve kontrollerini buna göre etiketleyin (örneğin, tanımlanmış biyofiziksel özelliklere sahip varyantlar için bilinir ve her varyant için vahşi tip ölçümler için bilinen kontrol).
- Tanımlanmış biyofiziksel özelliklere sahip varyantlar için AlphaMissense patojenite skorları elde edin.
NOT: Daha fazla ayrıntı için protokol bölüm 1.3'e bakın. - Her varyant için alt birim ve amino asit pozisyonu, orijinal ve değiştirilmiş amino asitler, patojenite skoru ve literatürden elde edilen biyofiziksel parametreleri içeren bir veri çerçevesi oluşturun. Deneysel tutarsızlıkları önlemek için, tanımlanan varyantlar için biyofiziksel parametreleri, vahşi tip ölçümlerde x kat değişiklikleri olarak normalleştirin.
- Yapısal ve fonksiyonel özelliklere göre karşılaştırmalı varyant analizi
- Tahmin edilen varyantları bir veri çerçevesi üzerinde düzenleyin; buna göre etiketleyin (örneğin, biyofiziksel özellikleri hakkında literatür bulunmayan varyantlar için tahmin edilir).
- Varyantları amino asit dizisindeki konumlarına ve üçüncül yapıya göre sınıflandırın. Veri çerçevesine yapısal sınıflandırma parametreleri (örneğin, alfa sarmallarında, bobinlerde, beta tabakalarında, hücre dışı, hücre içi veya transmembran alanlarda, gözenek astarında, agonist bağlanmasında, protein-protein etkileşimlerinde lokalizasyon) ekleyin ve her varyant için amino asit konumlarına göre bilgi sağlayın.
- Varyantları membran merkezine ve gözenek eksenine olan mesafelerine göre sınıflandırın. Veri çerçevesindeki gözenek eksenine olan mesafeyi ve membran merkezi parametrelerine olan mesafeyi ekleyin.
- Bilinen varyantlar üzerinden yapısal ve biyofiziksel parametreler arasındaki korelasyonu analiz edin. Mümkünse, elde edilen korelasyonlara göre tahmin edilen varyantları değerlendirin.
- Sinaps ve nöron modeli yapımı
- Çok bölmeli iletkenlik tabanlı hipokampal piramidal nöron üzerinde GABAerjik sinapsın çok bölmeli bir biyofiziksel modelini oluşturmak için Python'da sivri sinir ağlarını modellemek ve simüle etmek için geliştirilen açık kaynaklı bir sinir simülatörü olan Brian245'i kullanın.
- İyon kanalı geçit kinetiğini, pasif ve aktif parametreleri ve postsinaptik iletkenlikleri tanımlayarak iletkenlik tabanlı modeli tasarlayın. Modelde kullanılan denklemleri açıklayan Ek Dosya 2'de verildiği gibi iletkenlik tabanlı modeli tanımlayın.
- Membran kapasitansını (Cm) 1 μF/cm2 ve hücre içi direnci (Ra) 200 Ω.cm olarak ayarlayın.
- gL= 0.0003 S/cm2, gK= 0.036 S/cm2, EL = -76.5 mV, ENa = 50 mV ve EK = -90 mV olan hipokampal piramidal nöronlar39 için modifiye edilmiş Hodgkin-Huxley tipi iletkenlikleri kullanın.
- NaV kanallarının gNa üzerindeki yoğunluk dağılımını soma için 0.05 S/cm2 , akson başlangıç segmenti (AIS) ve Ranvier (NR) düğümü için 0.5 S/cm2 ve dendritler için 0.005 S/cm2 olarak ayarlayın. Miyelinli segmentlerde gK ve gNa'yı 0 olarak ayarlayın.
- Ek Dosya 2'de açıklandığıgibi Na V ve KV için iyon kanalı geçit kinetiği oluşturun.
- Bir bölmedeki tüm glutamaterjik ve GABAerjik sinapsların toplamı olarak sinaptik akımları (Isyn) tanıtın. Glutamaterjik akımda (I glu) hem hızlı AMPA reseptörü aracılı akımı (IAMPA) hem de yavaş NMDA reseptörü aracılı akımı (INMDA) dahil edin. GABAerjik akımda (IGABA) yalnızca hızlı GABAA reseptör aracılı akımı dahil edin. Her presinaptik sivri uç için sinapsa sabit miktarda glutamat salındığını varsayalım; bu nedenle, reseptörlerin aktivasyonu spike-time'a bağlıdır (sAMPA ve sNMDA) ve toplam reseptör iletkenlikleri (gAMPA ve gNMDA) her olay tarafından salınan glutamat miktarını yansıtır.
- Ek Dosya 2'de açıklandığı gibi sinaptik modeli kullanın.
NOT: Denklemlerin ayrıntılı bir açıklaması için, modelde kullanılan denklemleri açıklayan Ek Dosya 2'ye bakın.
- Soma ve nöritler için deneysel olarak ölçülen çapı ve her bir nörit bölmesinin uzunluğunu ve dallanma modellerini önceki literatürdenelde edin 46,47. Hücreyi birden fazla bölmeye bölerek, ana dallanma yapısını doğru bir şekilde koruyan ve iki taraflı simetriyi koruyan gerçek nöron morfolojisini çok bölmeli bir modele indirgeyin.
- Morfolojik (segment uzunluğu ve çapı; yani d_soma: 30 μm; l_AH: 5 μm; d_AH_i: 1,5 μm; d_AH_f: 1,3 μm; l_AIS: 40 μm; d_axon: 1 μm; l_myseg: 100 μm; l_NR: 2 μm; l_AxTer: 4 μm; d_AxTer: 2 μm; l_approx: 100 μm; l_apmed: 100 μm; l_apdis: 200 μm; d_approx_i: 4 μm; d_approx_f: 3 μm; d_apmed : 2 μm; d_apdis: 2 μm; l_apLM: 70 μm; d_apLM: 2 μm; l_nAcDbasal: 400 μm; d_nAcDbasal: 1,4 μm; l_nAcDbasal_stem: 20 μm; d_nAcDbasal_stem: 1.5 μm) ve piramidal nöron modelinin46,47 her bir bölmesi için biyofiziksel parametreler (bölüm 2.3.2'de verildiği gibi) Python betiğinde de ayrıntılı olarak açıklanmıştır (Ek Dosya 3: GABAAvar.py).
- Adım 2.1.1'de elde edilen vahşi tip kontrol ölçümlerini değerlendirerek GABAerjik sinaps modeli için biyofiziksel parametreleri belirleyin.
- Nöron modelinin topolojisini tasarlayın ve daha önce elde edilen morfolojik ve dallanma bilgilerine dayanarak bölmelerin uzamsal düzenlemesini ve ara bağlantılarını belirlemeyi içeren morfolojik ve biyofiziksel parametreleri atayın. Ek Dosya 3: GABAAvar.py'da belirtildiği gibi, modelin her bir bölmesine uygun morfolojik (örneğin, segment uzunluğu ve çapı) ve biyofiziksel parametreleri (Bölüm 2.3.2) atayın.
- Sinapsların oluşturulması ve akım enjeksiyonu
- "GABAAvar.py" (Ek Dosya 3) bölümünde verildiği gibi SpikeGeneratorGroup (Brian2 kitaplığından bir sınıf) kullanarak presynaptic aktiviteyi oluşturun. Sinaptik bağlantıları modellemek için Synapses sınıfını kullanarak spike üretecini model nöronun hedef bölmesine bağlayın.
- Sürekli bir sabit akımı (Iinj) 0.85 nA olarak ayarlayın ve Ek Dosya 3: GABAAvar.py'da belirtildiği gibi belirli bir zamanda temel iyonik akım yükü tarafından yönlendirilen eşik altı aktiviteyi taklit etmek için soma'ya yerleştirin.
- Kayıt monitörleri oluşturmak için, StateMonitor kullanarak hedef bölmelerden voltaj izlerini kaydedin.
- Ağı oluşturun ve çalıştırın.
- Network'ü kullanarak model nöron, bağlantılar ve monitörlerle ağı oluşturun.
- Simülasyonun zaman adımını defaultclock.dt ile ayarlayın (örneğin, 0,01 ms).
- Örnekte T'nin 1.000 ms olarak ayarlandığı network.run(T*ms) ile simülasyonu ağ üzerinde çalıştırın.
- GABAA reseptörü yanlış algılama mutasyonlarının etkisinin test edilmesi
- Adım 2.1.1'de toplanan biyofiziksel parametreler aracılığıyla her bir yanlış anlamlı mutasyonun kanal kinetiği üzerindeki etkisini tanımlayın.
- Bu parametreleri değiştirerek stimülasyonu çalıştırın ve sonuçları "GABAAvar.py" da verildiği gibi "matplotlib.pyplot" kullanarak çizin (Ek Dosya 3).
- Ateşleme modellerindeki ve hızlarındaki değişiklikleri analiz etmek için parametre kombinasyonlarını test edin. Karşılaştırmalar için sonuçları çizin.
Sonuçlar
Bu çalışma, epilepsinin patofizyolojisinde önemli bir bileşen olan GABAA reseptörünün γ2 alt birimindeki patojenik varyantları tahmin etmek ve karakterize etmek için çok ölçekli bir yaklaşım kullanmaktadır. Öngörücü modeller, moleküler modelleme, evrimsel koruma, yapısal inceleme, korelasyon analizi ve nöral simülasyonların kullanılması yoluyla bu yaklaşım, epilepsi araştırmaları ve muhtemelen klinik kullanım için önemli bir önemi olan varyantların sınıflandırılmasını geliştirir. Metodolojinin genel özeti Şekil 1'de sunulmuştur.
İki bitişik γ2 alt birim mutasyonunun karşılaştırmalı değerlendirmesi
GABAA reseptör alt birimlerindeki epileptojenik mutasyonlara komşu öngörülen patojenik mutasyonların kanal fonksiyonu ve nöral davranış üzerinde benzer elektrofizyolojik etkiler üretebileceği varsayımımıza dayanarak, önce iyi bilinen bir epileptojenik mutasyon ile γ2 alt biriminin proksimal olarak tahmin edilen mutasyonu arasındaki ilişkinin kısa bir incelemesini yaptık.
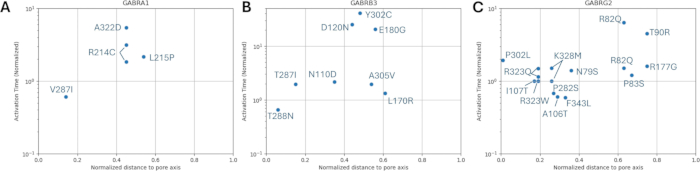
Patojenik olarak tahmin edilen varyantlar arasında (Ek Tablo S6), A303T (rs1581439874, ClinVar Katılımı: VCV000663033.6) örnek olarak seçilmiştir. Topluluk modelleri tarafından yapılan tahmine ek olarak, A303T'nin patojenitesi AlphaMissense skorları ile doğrulandı (Ek Dosya 4: Ek Tablo S7). A303T, GABAA reseptörü γ2 alt biriminin ikinci transmembran alanındadır ve Şekil 2'de gösterildiği gibi epileptojenik mutasyon P302L40'ın yanında bulunur. Moleküler modelleme ile değerlendirildiği gibi, hem γ2P302L hem de γ2A303T ikameleri, Şekil 3'te gösterildiği gibi daha büyük yan zincirlere sahip amino asitlerle sonuçlandı. γ2P302L mutasyonunda hem mutant hem de vahşi tip kalıntılar polar değildir, γ2A303T'de mutant kalıntı polar bir yan zincire sahiptir ve vahşi tip kalıntı polar olmayan bir yan zincire sahiptir. Hem P302 hem de Ala303, β3 alt birimi ile alt birim etkileşim arayüzünde bulunur (sırasıyla 7QNB ve 7QNA'da gözlenir). Hem P302 hem de Ala303, karşılaştırılabilir solvent erişilebilir yüzey alanına (SASA) sahiptir. Ek olarak, her iki kalıntı da omurgalı evrimi boyunca %100 korunur (Şekil 4, üst panel). Her ikisi de, GABAA reseptör proteininin üç boyutlu rekonstrüksiyonunda sarı ile gösterildiği gibi, γ2 alt biriminin ikinci transmembran bölgesinin (TM2 alanı) yakınında bulunur (7QNE26, burada kırmızı ile gösterilen A303, bu alandaki ilk kalıntıdır (Şekil 4). Bu karşılaştırılabilir özelliklere dayanarak ve bir piramidal nöron modeli kullanılarak, γ2 alt birim mutasyonu P302L40 gibi proksimal epileptojenik mutasyonların simülasyonu, tahmin edilen varyantın (γ2A303T) nöral yanıt üzerindeki etkisinin ön karakterizasyonu için kullanılabilir. Bir sonraki adımda, analizimizi GABAA reseptör alt birimleri içindeki daha geniş bir varyant kümesine genişlettik.
Yapısal ve biyofiziksel parametreler için kümeleme varyantları
Önceki bölümde iki bitişik mutasyonun karşılaştırmalı değerlendirmesinin ardından, varyantlar arasındaki ortak moleküler özelliklerin tanımlanıp tanımlanamayacağını değerlendirmek için sistematik bir yaklaşım uyguladık. Bu aşama, amino asitler ve varyantlar arasında yapısal, fizikokimyasal ve biyofiziksel özelliklerde tutarlı modellerin ortaya çıkıp çıkmadığını araştırmayı ve böylece ilk hipotezimiz için daha fazla destek sağlamayı amaçladı.
Bu çalışmada kullanılan veri çerçevesi ve referanslar Ek Dosya 4'te verilmiştir: Ek Tablo S840, 48,49,50,51,52,53,54,55,56,57,58,59,60, 61,62,63, Ek TabloS936 ve Ek TabloS1035. Ek olarak, her bir alt birim için ve alt birim ayrımı olmayan tüm varyantlar için yapısal ve biyofiziksel parametreler arasındaki korelasyonlar belirlenmiştir (Ek Dosya 4: Ek Tablo S11, Ek Tablo S12, Ek Tablo S13, Ek Tablo S14 ve Ek Tablo S15). Yapısal parametreler (alfa sarmalları, bobinler, beta tabakaları üzerindeki lokalizasyon; hücre dışı, hücre içi veya transmembran alanlar; gözenek astarı, agonist/allosterik bağlanma ve protein-protein etkileşimleri) hakkında bilgi Brünger ve ark.35'ten elde edilmiştir. Biyofiziksel parametreler İnsan Embriyonik Böbreği (HEK) 293 hücreleri üzerinde yapılan yama-klems elektrofizyolojisi çalışmalarından elde edildi. Değerler, ilgili vahşi tip reseptör aktivasyon süresi (τr) ve deaktivasyon süresi (τd) sabitleri ile normalize edildi.
Çalışmamız, GABAA reseptör alt birimlerinde fonksiyonel olarak tanımlanmış mutasyonlara bitişik veya yakın amino asit varyantlarının, bu mutasyonlarda gözlemlendiği gibi, kanal fonksiyonunda benzer elektrofizyolojik değişiklik paternleri gösterebileceği fikrine dayandığından, yapısal, fizikokimyasal ve biyofiziksel parametreler arasında bir ilişki olasılığını araştırdık. Varyantların membran merkezine ve gözenek eksenine olan uzaklıklarına göre konumları Şekil 5 ve Şekil 6'da verilmiştir. Bu bağlamda, AlphaMissense37'nin skorlarını da (Ek Dosya 4: Ek Tablo S7); Temel amino asit dizisini girdi olarak kullanabilen son derece hassas protein yapısı tahmin modeli AlphaFold264 tarafından desteklenmektedir. AlphaMissense, tek amino asit ikamelerinin yapısal yönleri için ipuçları sağlayabilir. GABAA reseptör alt birimlerinin (sırasıyla GABRA1, GABRB3, GABRG2 genleri tarafından kodlanan α1, β3, γ2 alt birimleri) varyant konumuna (amino asit konumu, zar merkezine olan mesafe ve gözenek eksenine olan mesafe) göre bilinen (siyah) ve tahmin edilen (kırmızı) varyantlar için AlphaMissense skorlarının dağılımı Şekil 7'de verilmiştir.
Şekil 7A-C, amino asit pozisyonları boyunca AlphaMissense skor dağılımını gösterir, Şekil 7D-F, gözenek ekseninden normalleştirilmiş mesafe boyunca AlphaMissense skor dağılımını gösterir ve Şekil 7G-I, membran merkezinden uzaklık boyunca AlphaMissense skor dağılımını gösterir. Şekil 7'deki korelasyon analizi, yeni tanımlanan varyantlar için sonucu tahmin etmek için yapısal özellikler aracılığıyla altta yatan bir ilişkiyi tespit etmenin zorluğunu göstermiştir. b2 alt birimi (GABRB2 geni tarafından kodlanan) varyantları, daha geniş bir analiz yapabilmek için kümeleme ve korelasyon bölümlerine dahil edildi. Bununla birlikte, yalnızca GABRA1 tarafından kodlanan α1 alt biriminin varyantları (Şekil 7A,D,G), GABRB3 tarafından kodlanan β3 alt birimi (Şekil 7B,E,H) ve GABRBG2 tarafından kodlanan γ2 alt birimi (Şekil 7C,F,I) biyofiziksel modellere dahil edildi, çünkü model bir hipokampal piramidal nöronun işlevine ve GABAA'nın α1β3γ2 kombinasyonuna odaklanmaktadır Reseptör alt birimleri, hipokampus65'teki en yaygın kombinasyondur. Benzer şekilde, bir α1β3γ2 GABAA reseptöründe kanal kinetiği çalışılmayan herhangi bir α1, β3 veya γ2 varyantı da simülasyonlardan çıkarıldı. Mevcut analizdeGABA A reseptör mutasyonlarının (Ek Dosya 4 ve Ek Tablo S8) etkilerinden türetilen AlphaMissense skorları ile biyofiziksel parametreler (normalleştirilmiş aktivasyon ve deaktivasyon süreleri) arasında hafif bir korelasyon (Ek Şekil S1 ve Ek Şekil S2) vardı. Bu, patojenik olduğu tahmin edilen mutasyonların (AlphaMissense skorlarına dayanarak) reseptör kinetiğinde (örneğin, aktivasyon ve deaktivasyon süreleri) ölçülebilir, potansiyel olarak yıkıcı değişikliklere yol açabileceğini düşündürmektedir. Bununla birlikte, Şekil 7'deki konumsal korelasyonlar arasındaki korelasyon eksikliği, bitişik amino asitlerin biyofiziksel özellikler için benzer sonuçlara sahip olacağı fikrine dayanan varsayımımız için AlphaMissense skorlarının kullanılmasını zorlaştırmaktadır.
Bilinen varyantların aktivasyon ve deaktivasyon kinetiğine göre gözenek eksenine normalleştirilmiş mesafenin dağılımları Şekil 8 ve Şekil 9'da gösterilmektedir. γ2 alt birimi için hafif bir korelasyon vardır (Şekil 8C), bu da bitişik amino asitlerin benzer sonuçlara sahip olacağı varsayımına dayanan hipotezimizin, bazı bölgelerde, özellikle reseptör kanalının gözenek alanının, TM2 alanının yakınında geçerli olabileceği olasılığını düşündürmektedir. Bu bölge, referans epileptojenik mutasyonumuza (Şekil 2 ve Şekil 4; γ2P302) bitişiktir, bu da onu nöral simülasyonlar için nispeten iyi bir aday haline getirir. Buna dayanarak, γ2A303T (Şekil 2 ve Şekil 4) gibi bitişik tahmin edilen mutasyonların etkilerinin kabaca bir tahmini yapılabilir. Burada sunulan sonuçlarımız yalnızca α1β3γ2 üzerindeki ölçümleri dikkate alır; bu nedenle, modelimizde değerlendirilen varyantlar, Ek Dosya 4: Ek Tablo S16'da verilen varyantlarla sınırlandırılmıştır.
Mutasyonların GABA A reseptör aracılı CA1 piramidal nöron ateşlemesinin inhibisyonu üzerindeki etkisi
Mutasyonların GABAA reseptör aracılı inhibisyonu üzerindeki etkisi, çok bölmeli iletkenlik tabanlı bir CA1 piramidal nöron modelinde gösterilmiştir. GABAA reseptörü yanlış anlamlı varyantlarının hipokampal piramidal nöron fonksiyonu üzerindeki etkisi, CA3 ve entorinal korteks (EC) III piramidal nöronların projeksiyonlarından nörona apikal girdilerin GABAerjik şantlanması yoluyla araştırılabilir. Başka bir deyişle, GABAA reseptörlerinin aktivitesini simüle etmenin bir yolu, simülasyonun, GABAerjik inhibisyon mekanizmalarından biri olan şant inhibisyonu gibi reseptörlerin fizyolojik önemi hakkında gerçekçi varsayımları temsil ettiği bir bağlamı varsaymaktır. CA1 hipokampal piramidal nöronları, tipik olarak apikal dendritlerinde, CA3 ve EC III'ün nöronlarından gelen çıkıntılar tarafından hedeflenen bu bölgelerde GABAA reseptörlerine sahiptir. Bu nedenle bu düzenleme simülasyon için uygundur. Bu araştırma sorusu, değişen gecikmelere ve yoğunluklara sahip bir girdi tasarımı gerektirir. Bu nedenle Şekil 10'da gösterildiği gibi distal apikal, medial apikal ve bazal dendritler üzerine üç farklı glutamaterjik sinaps (GluS1/2/3) yerleştirildi ve bunlar sırayla aktive edildi. Sinaptik girişlerin etkisini değerlendirmek için, sabit akım genliği minimum ani tetikleme eşiğinin altında kalmalıdır (Iinj < Idk). Vahşi tip veya mutant GABAA reseptörüne sahip piramidal nöron modeli, somada 0.85 nA'lık sabit bir akım enjeksiyonu ile başlatıldı. GABAerjik sinaps daha sonra somaya yerleştirildi. Spike jeneratörü tarafından taklit edilen presinaptik aktivite ilk olarak distal apikal dendritte başlatıldı. Medial apikal ve bazal dendritler üzerindeki sinaptik girişler sırasıyla 25 ms ve 50 ms gecikti. GABAerjik sinaps 40 ms'lik bir gecikmeyle aktive edildi. GABAerjik inhibisyon yoğunluğu, ilk spike hariç tüm spike treni inhibe edilecek şekilde ayarlandı. Daha sonra, varyantların etkisi bu ortamda τartışı, τdeaktivasyonu ve gGABAA'nın değişmesiyle araştırılır.
Vahşi tip ve mutant reseptörler için parametreler, özellikle hipokampal piramidal nöronlarda en bol bulunan alt birim bileşimi olan α1β3γ2'den oluşan reseptörler için protokol adımı 2.1.1'de açıklanan koleksiyondanelde edildi 65. Parametre dağılımı Ek Dosya 4'te verilmiştir: Ek Tablo S16.
Her alt birim mutasyonu tek, ikili ve üçlü glutamaterjik sinaps vakalarında test edildi. Basit bir yaklaşımla, mutasyonların etkisi, ateşleme hızı ve modeli üzerinden değerlendirilebilir. ΔtISI ortalamaları ve standart sapma, ΔtISI'nin ani yükselme aralığındaki değişikliği temsil ettiği ateşleme modelindeki değişiklikleri daha fazla ölçmek için de tahmin edilebilir. Her durum için sonuçlar, Ek Dosya 4'te ateşleme hızları ve ΔtISI (ortalama ve standart sapma) olarak verilmiştir: Ek Tablo S17 ve Ek Şekil S3. Ateşleme modellerini değiştiren varyantlar için sivri uç trenleri ve voltaj izleri Şekil 11, Şekil 12 ve Şekil 13'te verilmiştir.
Tek (GluS1) ve üçlü (GluS1-2-3) glutamaterjik sinaps aktivasyonu için, nöron yanıtını değiştiren mutasyonlar sadece3N110D ve γ2K328M mutasyonu β. Tek glutamaterjik giriş durumunda, β3N110D bozulmuş inhibisyona yol açtı ve ateşleme paterni, kısa bir gecikmeyle 4. presinaptik spike'dan sonra glutamaterjik spike trenine kilitlendi (Şekil 11). γ2K328M ayrıca, sadece 5. presinaptik spike civarında da olsa, inhibisyonu bozdu ve β3N110D'ye kıyasla postsinaptik spike'da daha büyük bir gecikme yarattı (Şekil 11). GluS1-2-3 aktivasyon durumunda (Şekil 13), yanıt β3N110D ve γ2K328M mutasyonları arasında benzerdi. Her iki mutant da, neredeyse tüm kümülatif presinaptik sivri uçların tespit edildiği ve bir yanıtı tetiklediği bir ateşleme modeli verdi. Her iki durumda da, nöron modelleri presinaptik aktiviteye yanıt olarak bir spike çifti ile ateşlendi.
Çift glutamaterjik sinaps aktivasyonu, diğer iki ayara kıyasla farklı sonuçlar vermiştir (Şekil 12). Bu durumda, GABAA reseptörü b3 alt birimindeki (β3N110D ve β3T288N) iki mutasyon ve GABAA reseptörü γ2 alt birimindeki (γ2P302L ve γ2K328M) iki mutasyon GABAerjik inhibisyonunu bozmuştur. γ2P302L mutantlı nöron modeli, GluS2 ile neredeyse senkronize olarak ateşlendi, bu büyük olasılıkla GluS1-2 arasında yaklaşık olarak aynı presinaptik ani yükselme gecikmesi ile GluS1'e gecikmiş bir yanıttı. β3T288N mutasyonu da benzer bir sonuç verdi ve ikinci sivri ucun ayrımı hala GluS2 ile senkronize durumdaydı. β3N110D mutantına sahip nöron modeli, daha kısa bir ΔtISI ile tanıtılan GluS1 / 2'nin ilk iki presinaptik sivri ucu dışında, hemen hemen her kümülatif glutamaterjik girdiye yanıt verdi. γ2K328M için ateşleme modeli yine β3N110D gibiydi ve ikinci ve üçüncü presinaptik sivri uçların ayrımı gözden kaçırıldı.
Bu sonuçlar, b3 alt birimi (GABRB3 geni tarafından kodlanan) ve γ2 alt birimi (GABRG2 tarafından kodlanan) mutasyonlarının hipokampal piramidal nöron aktivitesi üzerindeki çeşitli etkilerini göstermektedir. İlginç β bir şekilde,3L170R, β3A305V, β3E180G, β3D120N, β3Y302C ve γ2R82Q mutasyonları nöral aktivitede herhangi bir değişiklik göstermedi. İnhibisyondaki en ciddi bozulma, her ikisi de önemli ölçüde daha düşük τdeaktivasyonu ve daha yüksek τartışına sahip olan β3N110D ve γ2K328M içindi. Ön analizimiz ayrıca τyükselmesi veya gGABAA'daki değişikliklerin tek başına inhibisyonu bozmak için yeterli olmadığını gösterdi (veriler gösterilmemiştir). Artmış τyükselmesi ile birlikte önemli ölçüde azalmış τdeaktivasyonuna yol açan mutasyonların GABAerjik inhibisyonda daha anlamlı bir bozulmaya yol açtığı söylenebilir.
Tüm uyarıcı girdilerin inhibe edilmesi gerektiği durumda, ateşlemeyle sonuçlanan herhangi bir mutasyon, aynı mutasyonlara sahip nöronlardan oluşan bir nöral devrede abartılma potansiyeline sahip anormal ve spesifik olmayan nöral tepkiler verecektir. Bir sinir ağındaki uyarma/inhibisyon dengesi, herhangi bir ağ aktivitesinin çok önemli bir bileşeni olan sonuçta ortaya çıkan bozulmuş inhibitör geri bildirimden önemli ölçüde etkilenebilir.
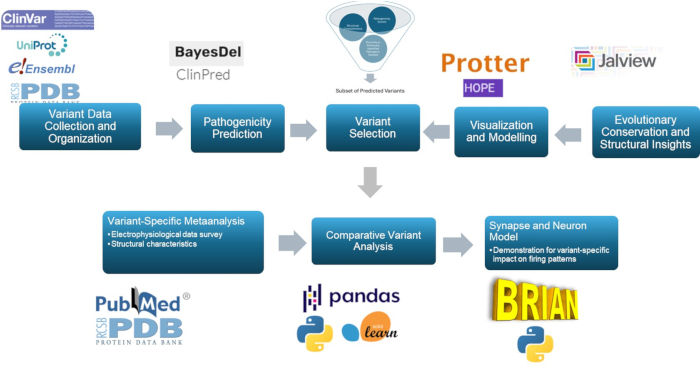
Şekil 1: In silico analiz ve nöral yanıt simülasyonlarına özel olarak odaklanarak, klinik ve araştırma amaçlı varyant etki tahmini ve analizine genel bakış. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: γ2A303T'nin konumu ve seçilmiş hasta mutasyonları;nöral simülasyonlar için γ2P302L ve γ2K328L kullanılır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
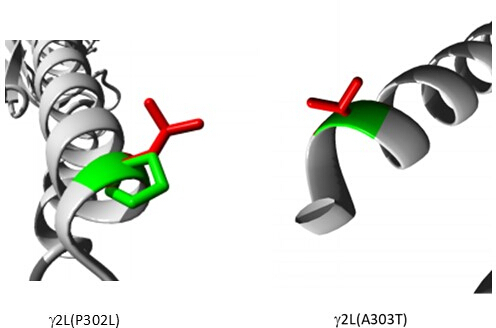
Şekil 3: Patojenik olarak tahmin edilen hasta mutasyonu γ2P302L ve bitişik varyant γ2L(A303T)'nin karşılaştırmalı modellemesi. Her iki modelde de yeşil, vahşi tipi, kırmızı ise mutant kalıntıları temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
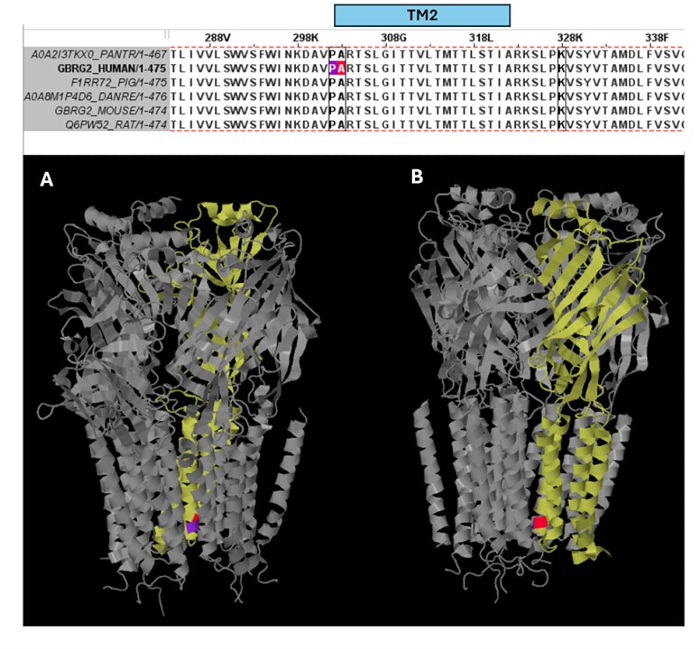
Şekil 4: Çoklu dizi hizalama ve yapısal içgörüler. Üst panel, TM2'nin kenarındaki hasta mutasyonunun (P302L) (mor) ve γ2 alt biriminin TM2'sinin başlangıcındaki patojenik varyant A303T'nin (kırmızı renk) pozisyonundaki kalıntıların evrimsel korunumunu gösterir. Alt panel, γ2 alt biriminin (7QNE'deki Zincir C) sarı olarak ve farklı açılardan (A, B) gösterildiği (A) üç boyutlu GABAA reseptör yapısında (7QNE) bu korunmuş kalıntıların görselleştirilmesini gösterir. Kısaltma: TM2 = ikinci transmembran alanı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: Dahil edilen tüm varyantların yerelleştirilmesi. Bilinen (siyah) ve tahmin edilen (kırmızı) varyantların (A) gözenek eksenine olan normalleştirilmiş uzaklıklarına ve (B) membran merkezine olan mesafelerine göre konumları gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
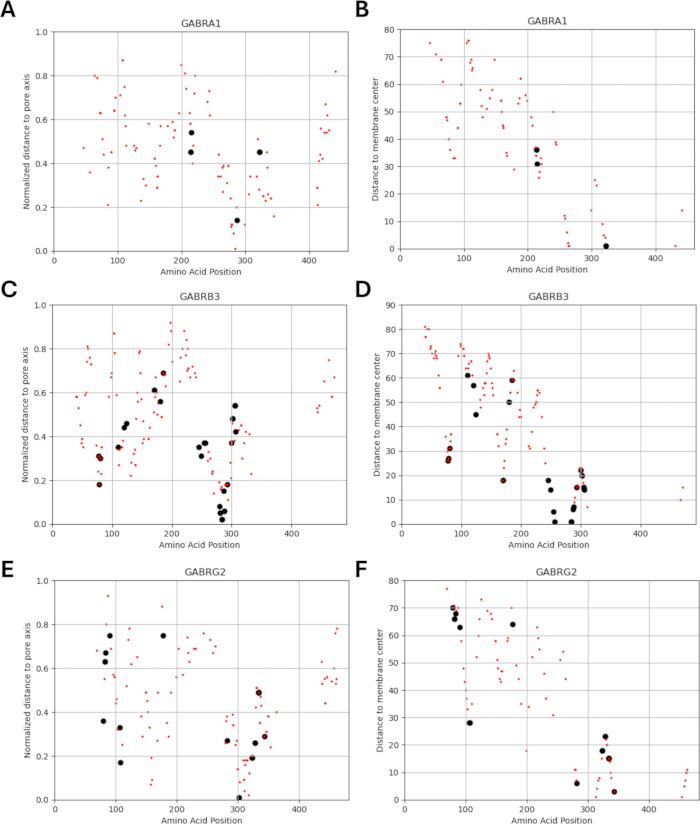
Şekil 6: Her bir alt birim için varyantların yerelleştirilmesi. Bilinen (siyah) ve tahmin edilen (kırmızı) varyantların (A, C, E) gözenek eksenine olan normalize edilmiş uzaklıklarına ve (B, D, F) membran merkezine olan mesafelerine göre konumları gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
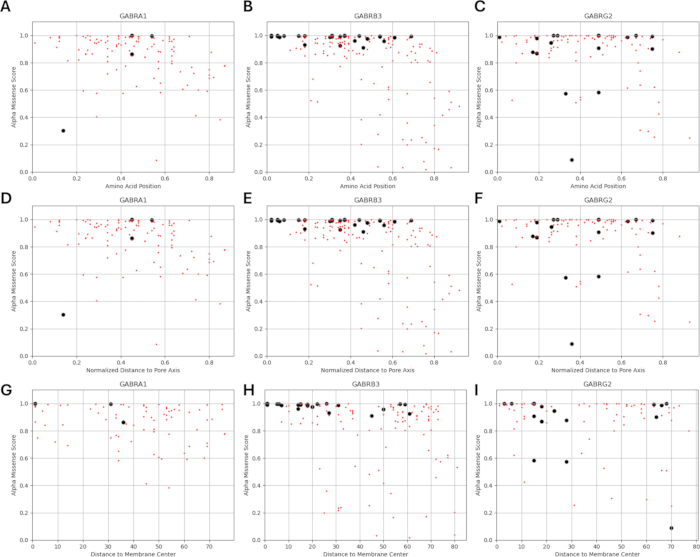
Şekil 7: Varyant konumuna göre AlphaMissense puan dağılımı. (A-C) Amino asit pozisyonu üzerinden AlphaMissense skor dağılımı, gözenek ekseninden (DF) normalize edilmiş uzaklık ve membran merkezinden (GI) uzaklık, bilinen (siyah) ve tahmin edilen (kırmızı) varyantlar için verilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 8: α1 (GABRA1) alt biriminde, β3 (GABRB3) alt biriminde ve γ2 (GABRG2) alt biriminde mutasyonlara sahip GABAA reseptörlerinin normalleştirilmiş aktivasyon süresi.(A) α1, (B) β3 ve (C) γ2 alt birimleri üzerindeki her mutasyon için gözenek ekseninden normalleştirilmiş mesafeye göre deneysel olarak elde edilen aktivasyon zaman sabitleri görüntülenir. Değerler, ilgili vahşi tip reseptör aktivasyon süresi ile normalize edildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 9: GABAA reseptörünün α1 (GABRA1) alt biriminde, GABAA reseptörünün β3 (GABRB3) alt biriminde ve γ2 (GABRG2) alt biriminde mutasyonlarla GABA A reseptörlerinin normalleştirilmiş deaktivasyon süresi.(A) α1, (B) β3 ve (C) γ2 alt birimleri üzerindeki her mutasyon için gözenek ekseninden normalleştirilmiş mesafeye göre deneysel olarak elde edilen devre dışı bırakma zaman sabitleri görüntülenir. Değerler, ilgili vahşi tip reseptör deaktivasyon süresi ile normalize edildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 10: CA1 piramidal nöron modeli. Model nöron şunlardan oluşur: (1) soma, (2) lamina molecularis'te iki dal ile biten proksimal, medial ve distal bölmeleri olan apikal bir dendrit, (3) kısa bir bazal dendrit sapından sonra iki bölüme ayrılan simetrik olarak oluşturulmuş iki bazal dendrit ve (4) konik bir akson tepecikliği ile başlayan ve ardından silindirik akson başlangıç segmenti ile devam eden bir akson, miyelinli segmentler ve Ranvier düğümleri, küresel bir akson terminali ile biter. Yeşil üçgenler glutamaterjik sinapsların yerini gösterir ve kırmızı üçgen somada bulunan GABAerjik sinapsları temsil eder. Ölçek çubuğu = 100 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 11: Sadece GluS1 aktivitesine sahip ateşleme modeli. Presinaptik nöronlar (GluS1 (yeşil üçgen) ve GABAerjik (kırmızı daire)) için spike trenleri ve vahşi tip veya mutant GABAA reseptörlerine (siyah kare) sahip postsinaptik nöronlar üst panelde verilmiştir. GABAerjik inhibisyonu olan veya olmayan vahşi tip GABAA reseptörüne sahip nöronlar ve GABAerjik inhibisyonu olan mutant GABAA reseptörlerine sahip nöronlar için bireysel voltaj izleri alt panellerde görüntülenir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
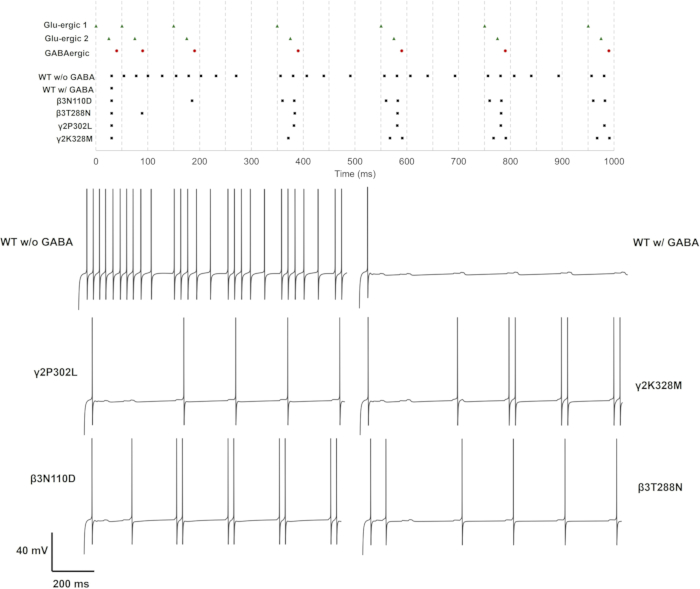
Şekil 12: Sadece GluS1 ve GluS2 aktivitesine sahip ateşleme modeli. Presinaptik nöronlar (GluS1/2 (yeşil üçgen) ve GABAerjik (kırmızı daire)) için spike trenleri ve vahşi tip veya mutant GABAA reseptörlerine (siyah kare) sahip postsinaptik nöronlar üst panelde verilmiştir. GABAerjik inhibisyonu olan veya olmayan vahşi tip GABAA reseptörüne sahip nöronlar ve GABAerjik inhibisyonu olan mutant GABAA reseptörlerine sahip nöronlar için bireysel voltaj izleri alt panellerde görüntülenir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 13: Sadece GluS1, GluS2 ve GluS3 aktivitesine sahip ateşleme modeli. Presinaptik nöronlar (GluS1/2/3 (yeşil üçgen) ve GABAerjik (kırmızı daire) için spike trenleri ve vahşi tip veya mutant GABAA reseptörlerine (siyah kare) sahip postsinaptik nöronlar üst panelde verilmiştir. GABAerjik inhibisyonu olan veya olmayan vahşi tip GABAA reseptörüne sahip nöronlar ve GABAerjik inhibisyonu olan mutant GABAA reseptörlerine sahip nöronlar için bireysel voltaj izleri alt panellerde görüntülenir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil S1: AlphaMissense skorlarının ve biyofiziksel parametrelerin dağılımı (Normalleştirilmiş deaktivasyon süresi; Bu çalışmada seçilen GABAA reseptör alt birimi mutasyonlarının normalleştirilmiş τd). Ayrıca bakınız Ek Dosya 4: Ek Tablo 8. Bu rakamı indirmek için lütfen buraya tıklayın.
Ek Şekil S2: AlphaMissense skorlarının ve biyofiziksel parametrelerin dağılımı (Normalleştirilmiş aktivasyon süresi; Bu çalışmada seçilen GABAA reseptör alt birim mutasyonlarının normalleştirilmiş τr). Ayrıca bakınız Ek Dosya 4: Ek Tablo 8. Bu rakamı indirmek için lütfen buraya tıklayın.
Ek Şekil S3: Vahşi tip ve mutant GABAA reseptörleri ile nöral yanıt için interspike aralıkları. En üstteki grafik, tek glutamaterjik girdi için interspike zaman aralıklarını gösterir. Ortadaki çizim iki tane gösterir ve en alttaki çizim aynı anda aktif olan üç glutamaterjik sinapsı gösterir. Bu rakamı indirmek için lütfen buraya tıklayın.
Ek Dosya 1: "Data_GABAA. R", veri biçimlendirme için R'de çalıştırmak için gereklidir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 2: İletkenlik Tabanlı Model tasarımında kullanılan denklemler. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 3: GABAAvar.py Neural Simulation için Brian2'de çalışması gerekir. Dosya, Brian2 tabanlı çok bölmeli nöron modeli (fonksiyon: CA1_Pyr), iletkenlik tabanlı nöron ve sinaptik modeller için denklemler (fonksiyon: model_eqns, syn_eqns) ve başlangıç parametreleri (fonksiyon: biophys_param, morpho_param, syn_param) için Python kodlarını içerir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 4: Tüm Ek Tabloları içeren bir zip klasörü. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Tablo S1: ClinVar'dan .txt dosya olarak indirilen ve daha sonra "data.xlxs" olarak kaydedilen GABRG2 geninde önemi bilinmeyen yanlış anlamlı varyantlar. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S2: Çalışmada kullanılan dizilerin tanımlayıcıları, ilgilenilen genin referans transkripti (NCBI Ref. devamı) ve farklı veritabanlarındaki diğer karşılık gelen tanımlayıcılar). Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S3: Yapısal ve işlevsel bölgelerin konumları. Referans transkripti (NM_198904.4) tarafından kodlanan γ2 alt birim proteininin (NCBI Referans Dizisi: NP_944494.1) spesifik bölgelerinin konumları Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S4: GABRG2'nin ClinVar verilerini temsil eden ve yalnızca şu sütunları içeren "data1.xlxs" dosyasının içeriği: "GRCh38Chromosome", "GRCh38Location", "Name", "Protein change". Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S5: Varyant etkisi tahmini için dbNSFP sunucusuna yüklenecek GABRG2 verilerinin yanlış anlamlı varyantlarının gerekli biçimlendirmesini içeren "data1_output.xlsx" dosyasının içeriği. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S6: GABRG2'nin bilinmeyen yanlış anlamlı varyantları için varyant etkisi tahmini için dbNSFP sunucusundan alınan çıktıyı içeren "data2.xlsx" dosyasının içeriği. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S7: GABAA reseptör alt birim varyantları için AlphaMissense skorları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S8: Varyantların biyofiziksel özellikleri. Biyofiziksel parametreler için değerler, elektrofizyoloji deneyleri ile yapılan önceki çalışmalardan elde edilmiştir. Varyantlar "S" (ikame) tipi ile etiketlenirken, vahşi tip reseptör parametreleri her ikame için verilir ve "C" (kontrol) ile etiketlenir. τd : Deaktivasyon zaman sabiti, PAçık : Açık olasılık, gGABA: Reseptör iletkenliği, Imaks: Maksimum akım, τr : Aktivasyon zaman sabiti. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S9: Varyantların fizikokimyasal özellikleri. Biyofiziksel parametrelerle daha önce tanımlanmış varyantlar "S" (ikame) tipi ile etiketlenirken, tahmin edilen varyantlar "P" ile temsil edilir. H: Hidrofobiklikteki değişim, VSC: Yan zincirin hacmindeki değişiklik, P1: Polaritedeki değişiklik, P2: Polarizasyondaki değişiklik, SASA: Solventle erişilebilen yüzeydeki değişiklik, NCISC: Net yük indeksindeki değişiklik. Değerler, Guo ve ark.36'dan her bir orijinal amino asit ve varyant için elde edilir ve her bir parametredeki değişiklik verildiği gibi tahmin edilir. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S10: Varyantların yapısal özellikleri. Biyofiziksel parametrelerle daha önce tanımlanmış varyantlar "S" (ikame) tipi ile etiketlenirken, tahmin edilen varyantlar "P" ile temsil edilir. Varyantın bir etki alanındaki yerelleştirmesi 1, aksi takdirde 0 ile temsil edilir. Tüm değerler Brünger ve ark.35'ten elde edilmiştir. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S11: Bilinen tüm varyantlar için yapısal ve biyofiziksel parametre korelasyonları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S12: Bilinen GABRA1 varyantları için yapısal, fizikokimyasal ve biyofiziksel parametre korelasyonları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S13: Bilinen GABRB2 varyantları için yapısal, fizikokimyasal ve biyofiziksel parametre korelasyonları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S14: Bilinen GABRB3 varyantları için yapısal, fizikokimyasal ve biyofiziksel parametre korelasyonları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S15: Bilinen GABRG2 varyantları için yapısal, fizikokimyasal ve biyofiziksel parametre korelasyonları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S16: Yabani tip ve mutant α1β3γ2 GABAA reseptörleri için biyofiziksel parametreler. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo S17: Vahşi tip ve mutant GABAA reseptörleri ile tek, çift veya üçlü glutamaterjik sinapslara yanıt olarak ateşleme hızı ve ani yükselme aralıkları. Bu tabloyu indirmek için lütfen buraya tıklayın.
Tartışmalar
Hesaplamalı genetik, moleküler modelleme ve nöral simülasyonların bir kombinasyonunu uygulayarak, bu yazıda sunulan yaklaşım, hem epilepsi araştırmaları hem de klinik uygulamalar için değerli bilgiler sunan GABAA reseptör varyantlarının sınıflandırılmasını iyileştirme potansiyeline sahiptir. Tahmin edilen patojenik mutasyonların tanımlanması ve önceliklendirilmesi için kapsamlı bir analiz sunulur ve protein ve hücresel fenotip üzerindeki varyant etkileri arasındaki boşluğu potansiyel olarak kapatan bir çerçeveye genişletilir. Epileptojenik GABAA reseptör aktivitesinin hipokampal piramidal nöron simülasyonu üzerindeki etkisinin değerlendirilmesi, GABAA reseptör disfonksiyonu ile ilişkili in vitro fenotipin replikasyonuna ve ağ disfonksiyonunun kökünde tek nöron yanıtı değişikliğinin gösterilmesine izin verir. Epileptojenik mutasyonlar tarafından üretilen bu nöral tepkilerin simülasyonlarına dayanarak, yapısal olarak proksimal olarak tahmin edilen mutasyonların fonksiyonel etkilerinin kaba bir tahmini araştırıldı. Tahmin edilen mutasyonların kanal kinetiği üzerindeki etkisine ilişkin tahminler, bilinen varyant setleri ile kapsamlı bir analiz gerektirir. Bu yazıda sunulanlar gibi karşılaştırmalı analizler ve nöral aktivite simülasyonları, protein fonksiyonu ve nöral patoloji üzerindeki varyant etkisine odaklanan öngörücü modellerin daha fazla oluşturulması ve iyileştirilmesi için kritik bilgiler sağlar. Ek olarak, metodolojimiz, GABAA reseptörü ile ilişkili nörogelişimsel bozukluklarla bağlantılı varyant etkilerini incelemek için bilinmeyen varyantlar arasında en patojenik varyantları seçmek ve önceliklendirmek için kullanılabilir. Örneğin, floresan problar 66,67,68,69,70 ile etiketlenen ve tahmin edilen mutasyonları taşıyan reseptör alt birimleri, trafiklerini, hücre yüzey ekspresyonlarını ve nörofizyolojilerini incelemek için in vitro olarak ifade edilebilir. Ayrıca, C. elegans gibi hayvan modellerinin, tahmin edilen mutasyonların etkilerini doğruladığı düşünülebilir. Örneğin, CRISPR-Cas9 gen düzenlemesi, bir C. elegans GABAA reseptörü olan unc-49'un bir delesyonunu oluşturmak için kullanılmıştır, böylece unc-49 veya insan GABAA reseptörü71'in alt birimlerinde homozigot epilepsi ile ilişkili mutasyonlar üretilmiştir.
Genel olarak, varyantların sınıflandırılması, ACMG-AMP12 tarafından önerildiği gibi, birden fazla hesaplama kanıtı seviyesinin kullanılmasından yararlanır. Bu yaklaşım, farklı öngörücü araçları ve veri kaynaklarını entegre ederek, sonuçta klinik değerlendirmelerin doğruluğunu artırarak ve genomik teşhiste genel karar verme sürecini iyileştirerek varyant sınıflandırmasının güvenilirliğini güçlendirir. Metodolojimizde, birden fazla aracın tahminlerini birleştiren, böylece birden fazla hesaplama kanıtı gereksinimini karşılayan ve farklı araçları ayrı ayrı kullanma ihtiyacını ortadan kaldıran topluluk tahmincilerinin kullanılması bir avantajdır. Bu yaklaşım aynı zamanda tek tek araçlardan farklı çıktıları işleme zorluğunun üstesinden gelir, böylece tahmin sürecini kolaylaştırır ve verimliliği artırır. Bununla birlikte, gen merkezli veya varyanta özgü analizlerin öngörücü doğruluğuna ilişkin bir garanti yoktur. Bu, gen merkezli veya varyanta özgü tahminlerin, belirli bağlamlar ve hedefler için ayarlanmış belirli koşullar altında gerçekleştirilmesi gerektiği sonucuna götürür 15,72,73,74. Klinik müdahaleler için bu, genellikle bireyselleştirilmiş optimizasyon ile belirli bir hastalık bağlamında belirli bir gen veya gen alt kümesi için in silico araçların öngörücü doğruluğunun değerlendirilmesini gerektirecektir75. Bununla birlikte, tahmine dayalı doğruluğun değerlendirilmesi, genellikle doğruluk değerlendirmesinin güvenilirliğini etkileyebilecek yeterli sayıda varyantın olmaması ile sınırlıdır.
Literatürde farklı araçlar mevcuttur ve bunların doğruluğu veri setlerinde test edilmiş ve doğrulanmıştır14. Bununla birlikte, büyük veri kümelerine dayanan bu doğruluk sonuçları, belirli bir gen için birkaç bilinmeyen varyantın tahminine mutlaka yansıtılmaz. Bu bağlamda, biriken literatür, bireysel öngörücülerin sonuçlarını derleyen ve hesaplayan topluluk öngörücülerinin, bireysel öngörücülerin 33,76,77,78 uyumundan daha iyi performans gösterdiğinin bilindiğini ve bu nedenle, bu çalışmada, özellikle üstün performansları için BayesDEL 33 ve ClinPred32 topluluk tahmincilerini kullanmayı seçtik32, 34 BayesDEL, voltaj kapılı sodyum kanalı alfa alt birimi 5 (SCN5A) gibi transmembran proteinleri kodlayan genler de dahil olmak üzere klinik olarak anlamlı genlerdeki 4.094 yanlış anlamlı varyant için karşılaştırmalı olarak değerlendirildi ve üstün performans gösterdi33. Varyant etkisi tahmin protokolümüzde, ilk adım olarak, iki topluluk tahmincisinin (BayesDEL ve ClinPred) fikir birliğini dikkate aldık. Google DeepMind tarafından geliştirilen bir derin öğrenme modeli olan AlphaMissense37, AlphaFold 64,79'un bir uzantısıdır ve böylece yüksek doğrulukta protein yapısı tahmininin gücünden yararlanır. Topluluk modellerinin ilk tahmin sonuçlarını (protokol adımımız 1.3'te açıklandığı gibi BayesDEL ve ClinPred) AlphaMissense'in sonuçlarıyla karşılaştırdığımızda, tahminler kısmen birbiriyle uyumluydu (Ek Dosya 4: Ek Tablo S15) ve patojenik veya hastalıkla ilişkili bir fikir birliğine ulaşan topluluk modellerinin (BayesDEL ve ClinPred) tahminleriyle tam olarak uyumlu değildi. pembe satırlar olarak gösterilir (Ek Dosya 4: Ek Tablo S15). Bununla birlikte, nöron modelimizde kullandığımız ve hem ClinPred hem de BayesDEL tarafından patojenik olarak tahmin edilen GABRG2 mutasyonları R82Q, P302L ve K328M'nin yakınındaki bilinmeyen varyantlar (L81F, A303T ve V329F), sarı vurgularla gösterildiği gibi AlphaMissense tarafından da patojenik olarak tahmin edildi (Ek Dosya 4: Ek Tablo S15).
AlphaMissense29, dizi ve yapısal bağlam tahminini kullandığından, çalışmamızda, AlphaMissense skorları ile GABAA reseptör mutasyon konumları arasında, membran merkezi ve gözenek eksenine olan mesafelerine dayalı olarak herhangi bir ilişki olup olmadığını görmek istedik. Hipotezimiz, GABAA reseptör alt birimlerinin fonksiyonel olarak tanımlanmış mutasyonlarına bitişik veya yakın amino asit varyantlarının fonksiyonel etkisinin, mutasyon durumlarında gözlenen kanal fonksiyonunda benzer fizikokimyasal değişiklik paternleri gösterebileceği fikrine dayanmaktadır. GABAA reseptör alt birim mutasyon pozisyonları ile AlphaMissense skorları arasındaki bir korelasyon, GABAA reseptör alt birimlerindeki yeni yanlış anlamlı varyantların işlevsel sonuçlarının tahmin edilmesine izin veren hipotezimiz için bir çerçeve oluşturmak için kullanılabilir ilişkiyi belirlememize yardımcı olacaktır. Bununla birlikte, AlphaMissense skorları bu biyofiziksel parametrelerdeki değişiklikleri öngörmemiştir (Şekil 7). Analizimizdeki sınırlı örneklem boyutunun kesin sonuçlar çıkarmayı zorlaştırdığına dikkat etmek önemlidir. Bununla birlikte, analizimiz AlphaMissense skorlarının GABAA reseptörlerinin yapısal parametreleri ile korelasyon göstermediğini buldu. Net bir konumsal korelasyonun olmaması (örneğin, mutasyonların pozisyonları ile AlphaMissense skorları arasında) varsayımımızın geçerliliğini sorgulamaktadır. Bitişik kalıntılar gerçekten benzer etkilere sahip olsaydı, daha net bir korelasyon görmeyi beklerdik. Durum böyle olmadığından, AlphaMissense puanlarını varsayımımızı test etmek için güvenilir bir araç olarak kullanma yeteneğimizi zayıflatır.
İlginç bir şekilde, çalışmamızda, GABRG2 gen mutantları için varyantın gözenek eksenine olan mesafesi ile normalleştirilmiş kanal aktivasyon süresi arasında hafif bir korelasyon bulduk. Bu nedenle, bitişik amino asitlerin benzer sonuçlara sahip olacağına dair ön varsayımımız, gözenekteki bölgeler veya geçitle ilgili kilit bölgeler gibi kanalın bazı bölgelerinde geçerli olabilir, ancak diğer bölgelerde o kadar net olmayabilir. Küçük veri seti, bu değişkenliği ayırt etme yeteneğini sınırlar, ancak gelecekteki veriler veya daha ayrıntılı yapısal analizler, hipotezimizin bu yönünü iyileştirmeye yardımcı olabilir. Moleküler dinamik simülasyonları80 , özellikle iki bitişik γ2 alt birim mutasyonunun, yani epileptojenik mutasyon γ2P302L40 ve proksimal tahmin edilen mutasyon γ2A303T'nin (rs1581439874) karşılaştırmalı değerlendirmesi bağlamında, bu ön bulguları daha fazla araştırmak için güçlü bir tamamlayıcı yaklaşım olarak hizmet edebilir. Gelecekte, bu yaklaşım, özellikle çalışmamızda sunulan nöral simülasyonlarla entegre edildiğinde, bilinmeyen varyantın hücresel fenotip üzerindeki etkisinin daha doğru bir şekilde tahmin edilmesini sağlayabilir.
Ek olarak, GABAA reseptör alt birimlerinin yapısal ve fizikokimyasal özelliklerinin, diğer özelliklerle birlikte, kanal, nöron, ağ ve hastalık fenotipi üzerindeki yeni varyant etkilerinin işlevsel tahmini için güçlü makine öğrenimi modellerini eğitmek için kullanılıp kullanılamayacağını araştırmak ilginç olacaktır. Otomatik makine öğrenimi yaklaşımlarının ortaya çıkmasıyla, doktorların ve ıslak laboratuvar bilim adamlarının da daha demokratik bir ortamda kendi modellerini geliştirebilecekleri bir noktaya ulaştık81. Bu nedenle, bu teknolojilerin klinik uygulamaya entegrasyonu, kişiselleştirilmiş tıbbı daha erişilebilir hale getirerek ve fonksiyonel varyant analizi için son derece uzmanlaşmış uzmanlığa olan bağımlılığı azaltarak süreci potansiyel olarak kolaylaştırabilir. Bu bağlamda, yaklaşımımız, reseptörün yapısal ve fonksiyonel dinamikleri hakkında bilgi vererek, varyant etkisinin fonksiyonel tahmini için gelecekteki çalışmalara potansiyel olarak yardımcı olur.
Protein yapısı tahminindeki mevcut ilerlemeye ve AlphaFold64 tarafından temsil edilen atılıma rağmen, mutasyonların ve protein fonksiyonunun etkisinin doğru tahmini, model79'u eğitmek için gereken veri eksikliği nedeniyle bir zorluk olmaya devam etmektedir. Varyant etkisinin tahmini için AlphaMissense, öngörücü modellerin bir alt kümesine kıyasla daha yüksek performans gösterirken, çalışmamızda kullanılan topluluk tahmincileri BayesDEL25 ve ClinPred24 bu karşılaştırmaya dahil edilmemiştir29. Çalışmamızda, in silico araçlar BayesDEL, ClinPred ve AlphaMissense'in farklı amaçlar için kullanıldığını belirtmek önemlidir. Topluluk belirleyicileri, BayesDEL ve ClinPred, öncelikle patojenite tahmini için kullanılırken, AlphaMissense, skorları ile γ2 alt birimindeki mutasyonların etkisi için bilinen veriler arasındaki ilişkiyi araştırmak için özel olarak kullanıldı. Spesifik olarak, hipotezimiz, tahmin edilen patojenik varyantların, özellikle GABAA reseptör alt birimlerinde fonksiyonel olarak tanımlanmış mutasyonların yakınında veya bitişiğinde bulunanların, fonksiyonel olarak karakterize edilmiş mutasyonlarda gözlemlenenlere benzer biyofiziksel parametreler sergileyeceğini varsayar. Bunu araştırmak için, tek amino asit ikamelerinin sonuçlarını tahmin etmek için temel peptit dizisini kullanan son derece hassas AlphaFold264 modeli tarafından desteklendiği için AlphaMissense'i seçtik.
Sonuç olarak, çalışmamızın önemli bir sınırlaması, öncelikle deneysel verilerin sınırlı mevcudiyetinden kaynaklanmaktadır. Örneğin, nöron modelimiz, GABAA reseptörlerinin α1β3γ2 alt birim kombinasyonundan türetilen verilerin ifadesine dayanmaktadır, bu da literatürde incelenen mutasyonları doğal olarak bu spesifik reseptör kombinasyonunun bir parçası olarak ifade edilen alt birimlerle sınırlar. Ek olarak, yalnızca bu alt birimlerin HEK hücrelerinde ekspresyonundan elde edilen elektrofizyolojik verilere güvendik ve literatürdeki mevcut verilerin kapsamını daha da daralttık. Bilinmeyen varyantların etkilerini tahmin etmek için nöral modelleme kullanımımız, bilinen mutasyonlara yakın bulunan bilinmeyen varyantların (iş akışımızda patojenik olarak tahmin edilen) kanal kinetiğinde, parametrelerinde veya literatürde açıklanan mutasyon etkilerinin fizikokimyasal özelliklerinde benzer modeller sergileyeceğini varsayar. Bu varsayım, HEK293 hücrelerindeki spesifik reseptör düzenekleri için elektrofizyolojik verilere duyulan ihtiyaç ile birleştiğinde, modelleme için mevcut deneysel veri miktarını azaltır. Bu kısıtlamaların bir sonucu olarak, mevcut veriler α1β3γ2 alt birimlerinde yalnızca sınırlı sayıda varyantı modellememize izin verdi. Bununla birlikte, nöron modelinin, alt birime özgü hücresel, devre ve ağ düzeyinde etkileri olan α1β2γ2 alt birim kombinasyonları, α1β2δ veya α4β3δ alt birim kombinasyonları gibi farklı alt birim düzenekleri için eğitilmesi, muhtemelen çeşitli epilepsi tiplerine ve nörogelişimsel bozukluklara daha geniş uygulanabilirlik gösterecektir. Gelecekte, mevcut elektrofizyolojik verilerin artması ve iyi tanımlanmış reseptör düzeneklerindeki mutasyonlara ve spesifik hücre tiplerine odaklanan çalışmalarla, yaklaşımımızın genellenebilirliğini ve doğruluğunu artırmak mümkün olabilir.
Çok bölmeli iletkenlik tabanlı nöron modelleri, tek nöron yanıtının reseptör varyantlarının işlevsel önemi için tahminler üretmek için güçlü bir araç sağlar. Bu araç, herhangi bir belirli soruyu test etmek için hem hücresel/sinaptik parametrelerin hem de konumlarının esnek tanımlarını sağlar. Bu protokolde kullanılan basit spike jeneratörleri, mikro devre aktivitesini incelemek için diğer nöron modelleriyle değiştirilebilir. Protokolün kritik adımı aynı zamanda en sınırlayıcı adımdır: değiştirilmiş kanal kinetiği açısından herhangi bir reseptör varyantının tanımı. İhtiyaç duyulan bilgiler ideal olarak yama-klemp elektrofizyoloji çalışmaları ile sağlanır; Bununla birlikte, tahmin edilen klinik öneme sahip amino asit ikamelerinin hesaplamalı analizi ve bilinen ikamelerle karşılaştırmalar da bazı bilgiler sağlayabilir. Çalışmamız ve açıklanan protokol, nöral aktivite simülasyonlarının öngörücü bir araç olarak değil, GABAA reseptörünün değişmiş biyofiziksel özelliklerinin tek nöron aktivitesi üzerindeki sonuçlarına ilişkin daha geniş bir bakış açısını desteklemek için mutasyonların etkilerini araştırmak için bir araç olarak kullanımını içermektedir. Nöral simülasyonlarda deneysel verilere bağımlılık, yapı ve işlev arasındaki boşluğu kapatmak için gelişmiş moleküler modellemeden yararlanabilecek yaklaşımımızda önemli bir sınırlamadır.
Protokolümüzde bazı adımların eleştirel bir şekilde değerlendirilmesi gerekmektedir. İlk olarak, protokolümüzün ilk bölümünde kullanılan tahmine dayalı modelin seçimi kritik olabilir. In silico araçların seçimi, güçlü tahmin12 için yeterli sayıda hesaplama kanıtının oluşturulması da dahil olmak üzere çeşitli faktörlere bağlıdır. Çoklu tahmine dayalı algoritmaların analizini entegre eden topluluk tahmincileri, bu öneriye daha iyi uyar, bu nedenle bireysel tahmincilere kıyasla tercih edilir. Farklı öngörücüler vardır ve bunların doğruluğu genellikle büyük veri kümelerinde test edilir, bu da belirli bir gende bulunan bilinmeyen varyant etkileri için kullanılan öngörücü modelin doğruluğunu garanti etmez. Bu, birden çok tahminciden tahmin sonuçlarını toplayan ve hesaplayan iki topluluk modeli kullanılarak telafi edilir. Ek olarak, amaç öncelikle en patojenik varyantları tanımlamaksa, öngörücü modellerin kesintileri özgüllüğü artırmak için ayarlanabilir. Duyarlılık ve özgüllük arasındaki dengeyi dengelemek ve varyantların doğru bir şekilde sınıflandırılmasını sağlamak için uygun kesimlerin ayarlanması önemlidir. Çalışmamızda varsayılan kesmeleri kullandık. Özellikle, iş akışımızda açıklandığı gibi analizimizin birden fazla düzeyinde incelenecek varyantların kapsamını azaltacağı için, patojenik olma olasılığı daha yüksek olabilecek varyantları yakalamak lehine kesme oranını değiştirmedik. İlgilenilen proteinin üç boyutlu rekonstrüksiyonu için yapısal verileri seçerken, literatürdeki yapısal verilerin ön incelemesine ihtiyaç duyulduğunu belirtmek de önemlidir. GABAA reseptörlerinin yapısal incelemesi, farklı koşullarda farklı reseptör düzeneklerinin üç boyutlu yapısını bildiren sağlam yapısal çalışmalarla son zamanlarda ivme kazanmıştır 26,27,82,83,84,85. Bu verilerin mevcudiyeti göz önüne alındığında, çalışmamızda, yapısal verilerin yeniden yapılandırılması için deneysel olarak belirlenen yapılara odaklandık. Bununla birlikte, AlphaFold64'ün tahmini, deneysel olarak belirlenmiş verilere sahip olmayan diğer proteinlerin analizi için tercih edilebilir. Deneysel çalışmalardan elde edilen yapısal veriler için amino asit numaralandırmasına dikkat etmek önemlidir. Amino asitlerin PDB numaralandırması, UniProt numaralandırmasından farklı olabilir, çünkü birincisi, protein olgunlaşması sırasında çıkarılan sinyal peptidini içermeyebilir. Ayrıca, deney sisteminde ifade edilen kimerik proteinler tutarsızlıklara neden olabilir. Bu durumda, ilgilenilen dizinin yapısal verilerden türetilen dizi ile ikili olarak hizalanması, tutarlılığın korunmasına yardımcı olacaktır. Literatürde, γ2 alt birim proteini için yapısal veriler, elektron mikroskobu (EM) gibi deneysel yöntemler ve AlphaFold'un yüksek doğrulukta tahmin yöntemi dahil olmak üzere farklı yöntemlere dayanmaktadır. Deneysel yöntem, istenen diziyi yüksek çözünürlükle tam olarak kapsamıyorsa, AlphaFold tahminleri kullanılabilir. Bu çalışmada, 7QNE26 yapısı, insan tam uzunlukta sinaptik α1β3γ2 GABAA reseptörünün kriyojenik elektron mikroskobik yapısına karşılık geldiği için seçilmiştir. Bu tam olarak, bu çalışmanın odak noktası olan tam uzunlukta alt birim kombinasyonunu temsil etmektedir.
Ek olarak, karşılaştırmalı analiz için, kanal kinetiğinin normalleştirilmiş parametrelerinin kullanılması tercih edilmelidir, çünkü bu parametrelerin değerleri reseptör alt birim bileşimine ve deneysel ayarlara bağlı olarak değişebilir. Örneğin, τyükselmesi ve τdevre dışı bırakma , vahşi tip kontrol değerleri üzerindeki x kat değişiklikleri üzerinden belirlenmelidir. Protokol adımı 2.5'te, bilinen varyant sayısının az olması durumunda, parametrik veya kategorik korelasyon analizi ve rho ve p değerlerinin belirlenmesi tercih edilebilir. İdeal olarak, temel bileşen analizi gibi yöntemlerin daha doğru ilişkiler sağlaması beklenir, ancak daha fazla sayıda numune gerektirir.
Simülasyon ortamı değiştirilebilir. Bu çalışmada, Brian2 aşağıdaki nedenlerden dolayı tercih edilmiştir: Brian2'deki uzamsal nöron sınıfı, nöral aktiviteyi simüle etmek için değerli bir araç sağlar. Brian2, önceden tanımlanmış "kara kutu" modellerine güvenmek yerine sürekli dinamikleri tanımlamak ve ayrık olaylar için ifadeleri güncellemek için diferansiyel denklemleri kullanmada önemli bir avantaja sahiptir ve bu, modelin her yönü tek bir Python betiğinde açıkça tanımlanabildiğinden, model denklemleri matematiksel gösterimde belirtildiğinden ve Brian'a özgü kelime dağarcığının yalnızca küçük bir miktarı kullanıldığından, mükemmel kod okunabilirliği ve uyarlanabilirliği sağlar37. Model açıkça tanımlandığından, tüm özellikler belgelenir ve Stimberg ve ark.45,86 tarafından yapılan çalışmalarda belirtildiği gibi, daha önce güvenilen "kara kutu" modellerine olan ihtiyacı ortadan kaldıran birincil simülasyon açıklama dosyasında bulunabilir.
Mevcut nöron modeli, Na +, K + ve kaçak akım ile modifiye edilmiş Hodgkin-Huxley tipi iletkenlikler46 kullanır. Bu iletkenlik tabanlı model, Ca2+ kanalları gibi diğer birkaç kanal tipinin dahil edilmesiyle daha da genişletilebilir. Sinaps modelleri için, bu parametrelerin belirli alt birim bileşimleri için elde edilmesi gerektiğine ve yalnızca bu bileşimlerle ölçülen parametrelere sahip varyantların değerlendirilmesi gerektiğine dikkat etmek önemlidir. Bu çalışmada, α1β3γ2 reseptör bileşimi seçildi; bu nedenle, bir α1β3γ2 GABAA reseptörü üzerinde ölçülen kanal kinetik parametreleri ile sadece α1, β3 veya γ2 varyantları dahil edildi.
Hücresel biyofiziğin tahmini, hücreyi, her biri potansiyel olarak değişen iletkenlik özelliklerine sahip olan çok sayıda silindirik bölmeye ayırmayı içerir. Bir nöronun dendritlerindeki düzensizliklere rağmen, bunlar yerel olarak homojen iplikler olarak görülebilir. Bu tür modeller, hücrelerin karmaşık yapısını ve işleyişini anlamada yardımcı olur. Model tasarımı, bu özellikleri yansıtabilecek gerçek morfolojinin basitleştirilmiş bir versiyonuna odaklanır.
Değiştirilmiş amino asidin konumu ve fizikokimyasal özellikleri, kanal kinetiği üzerindeki etkiyi belirler. Örneğin, daha büyük bir yan zincire sahip bir amino asit ile sonuçlanan bir amino asit ikamesi, bu değişiklik kanalın gözeneklerini kaplayan bir amino asit üzerinde meydana gelirse, kanalın iletkenliğini azaltacaktır. Değişiklikler ayrıca kanalın açılmasını/kapanmasını da etkileyebilir. Bu modelde basitlik için, GABA bağlanma kinetiği sadece mevcut reseptörlerin bir oranına indirgenir; Bununla birlikte, ligand bağlanma afinitesini değiştiren ikamelerin olası etkisini incelemek için bu etkileşimi içerecek şekilde daha ayrıntılı modeller tasarlanabilir.
Sonuç olarak, bu çalışma, GABAA reseptör alt birimlerindeki patojenik varyantları tahmin etmek ve hipokampal piramidal nöron modelindeki epilepsi ile ilişkili mutasyonların simülasyonuna dayalı olarak olası hücresel fenotipi kalitatif olarak tahmin etmek için hesaplamalı yöntemler kullanmaktadır. Bildiğimiz kadarıyla, bu, genetik varyantların DNA'dan protein fonksiyonuna ve nöral davranışa kadar çoklu karmaşıklık seviyelerinde GABAA reseptör disfonksiyonuna nasıl katkıda bulunabileceğini değerlendirmek için hesaplamalı genetik, moleküler modelleme ve nöral simülasyonların birleşik uygulamasını araştıran ilk protokoldür. Bu protokol, epilepside potansiyel olarak zararlı varyantların ve ilişkili nöral patolojinin tahminini iyileştirmek için bir temel sağlayabilir. Ek olarak, ilgili nörogelişimsel ve ağ bozukluklarının altında yatan mekanizma hakkında önemli bilgiler veren diğer kanalopatilerin çalışmasında kullanılabilir. Buna dayanarak, varyantların fonksiyonel etkisini ve bunların GABAA reseptörünün kanal kinetiğine entegrasyonunu incelemek için GABAA reseptörünün yapısal ve fizikokimyasal değerlendirmelerini dahil ederek, gelecekte daha doğru bir analiz geliştirilebilir.
Açıklamalar
Tüm yazarlar bu çalışma ile ilgili herhangi bir çıkar çatışması olmadığını beyan eder.
Teşekkürler
Model nöronun yapımındaki yardımları için Çağla Koca'ya teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Brian2 | Sorbonne Université, INSERM, CNRS, Institut de la Vision, France; Imperial College London, United Kingdom | 2.8.0.4 | Stimberg et al., 2019 (https://pypi.org/project/Brian2/ ) |
| dbNSFP server | Genos Bioinformatics LLC, USA | v3.0 | Liu et al., 2020 (http://database.liulab.science/dbNSFP) (https://sites.google.com/site/jpopgen/dbNSFP) |
| HOPE | Centre for Molecular and Biomolecular Informatics CMBI, Radboud University, Netherlands | 1.1.1 | Venselaar et al., 2010 (https://www3.cmbi.umcn.nl/hope/) |
| Jalview | University of Dundee, UK | JV2 | Waterhouse et al., 2009 (https://www.jalview.org/) |
| Jupyter Notebook | Project Jupyter, USA | https://jupyter.org/install | |
| Phyton | Python Software Foundation, USA | 3.13 | https://www.python.org/downloads/ |
| Protter | ETH Zurich, Switzerland | Version 1.0 | Omasits, et al., 2014 (https://wlab.ethz.ch/protter/start/) |
| R | The R Foundation for Statistical Computing, USA | R version 4.3.2 | https://www.r-project.org/ |
| RStudio | Posit software, PBC, USA | RStudio 2023.12.1+402 "Ocean Storm" Release | https://posit.co/downloads/ |
Referanslar
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır