Method Article
位置特异性 GABAA 受体亚基错义变异在海马锥体神经元中的作用的鉴定和分类
摘要
本研究引入了一个多尺度框架,涵盖从 DNA 到蛋白质功能和神经行为。它提出了一种研究 GABAA 受体亚基中预测的致病性突变的新方法,假设预测为致病性的致痫突变和近端突变可能对 CA1 锥体神经元模型产生类似的影响。
摘要
了解癫痫相关基因中功能未知变异的影响对于阐明疾病病理生理学和开发个性化疗法至关重要。在从 DNA 序列到蛋白质功能和神经行为的多尺度框架下,我们描述了一种预测和研究致病突变的新方法,假设 GABAA 受体亚基中的致癫痫突变和附近的预测突变可能对 CA1 锥体神经元模型产生类似影响。通过探讨预测的致病突变与近端致痫突变之间的特征关系,本研究旨在根据致癫痫突变对海马锥体神经元模拟的影响来估计预测突变的影响。
该方法从收集 GABAA 受体 γ2 亚基遗传数据开始,然后使用自定义脚本在 R 中执行数据清理和格式化。接下来,将应用集成预测器来识别和优先考虑 γ2 亚基的致病性错义变体。将说明将特定的致病性变异(预测的)映射到致癫痫突变共享的亚基结构域,并附有其影响的分子建模和进化保守性的考虑。然后,将进行变异特异性荟萃分析和参数归一化,然后进行相关性分析,以确定预测突变和近端致痫突变之间的任何显着关系。使用基于 Python 的神经模拟器,将描述基于多区室电导的神经元模型,反映野生型和致癫痫突变体的作用。将考虑模拟致癫痫性 GABAA 受体亚型产生的神经反应,以粗略估计预测的致病性变异对神经反应的影响。据我们所知,这是第一个探索多尺度框架的方案,用于估计 GABAA 受体变体对神经元行为的影响,这对癫痫研究至关重要。该方案可以作为增强由与癫痫相关的 GABAA 受体的潜在致病性变体引起的细胞表型预测的基础。
引言
对于几乎所有的人类疾病,遗传变异在个体易感性中起着重要作用。因此,了解序列变异与疾病风险的关系为揭示疾病发展中涉及的关键过程并确定预防和治疗的新方法提供了一种有价值的方法1。这也适用于神经发育障碍,神经发育障碍是儿科初级保健中最普遍的慢性疾病之一 2。自闭症谱系障碍、智力障碍和癫痫等疾病说明了遗传变异如何显着影响发育过程中的个体易感性3。
发育中的大脑比成人大脑更容易患癫痫发作,因为在兴奋和抑制之间的关键平衡中,遗传程序性神经发育不匹配4。由于 GABA(γ-氨基丁酸)是成人大脑中的主要抑制性神经递质,在胚胎和出生后早期发育过程中具有兴奋性,因此不利于防止年轻大脑癫痫发作所需的稳定性。这种临时状态是由 K-Cl 协同转运蛋白5 缺乏足够的表达引起的,在存在功能失调的 GABAA 受体的情况下,会导致癫痫发作活动的风险增加。GABAA 受体介导 GABA 的兴奋性和抑制作用,具体取决于 Cl- 离子的细胞内浓度6。因此,随着大脑的成熟,GABAA 受体编码基因以及其他离子通道的突变会扭曲兴奋性,而参与神经元代谢、细胞信号传导和突触形成7 的基因突变会导致儿童失神癫痫8 等疾病。
临床干预越来越多地利用基因分析来提高治疗神经发育障碍的精确度2。小儿癫痫的基因检测为精准医学方法提供了潜在目标9,突出了遗传变异在指导治疗决策中的重要性。此外,~25% 的具有 新发 突变的癫痫患者接受了基因诊断,以确定精准医疗的潜在靶点,这强调了遗传变异在指导治疗决策方面的重要价值10。新一代测序技术的进步推动了这一点,例如靶向基因面板、全外显子组测序和全基因组测序,这些技术极大地加速了基因发现11。然而,当结果产生意义未知的变异 (VUS) 时,新基因发现数量的增加带来了挑战,这种分类反映了关于变异在疾病发病机制中的分子作用的相互矛盾的证据或信息不足。归类为 VUS 的变异对应于美国医学遗传学与基因组学学院 (ACMG) 和分子病理学协会 (AMP) 提出的五级变异分类系统中的一个类别12。
应对功能未知的遗传变异的挑战需要在两个关键维度上做出努力:临床实践和研究。在临床上,围绕 VUS 的不确定性会使患者管理和决策复杂化13。从科学研究的角度来看,在越来越多的意义不明的变异中识别致病性变异并确定它们在疾病病理生理学和表型效应中的作用至关重要1。一种理想的情况是准确预测所有功能上未表征的变体的分子、神经元和网络水平效应,从而最大限度地减少基于实验室的研究所需的资源、时间和精力。这些方面强调了准确分类遗传变异的重要性,以便能够精确诊断遗传性癫痫,支持个性化治疗,并促进潜在药理学靶点的发现。目前的预测工具 14,15,16,17 相对准确,但通常仅提供二元分类(致病性与良性),并且缺乏对分子病理生理学、表型后果和潜在机制的疾病特异性见解。本文专注于所选 GABAA 受体亚基编码基因的未知错义变异,提出了一个框架,旨在通过结合变异的背景因素(如分子、进化和结构方面)以及来自癫痫相关突变的体外生物物理学数据的神经病理模拟来加强研究指导。我们的方法解决了 GABA A 受体 γ2 亚基的未知致病性变异的鉴定,GABAA 受体是参与癫痫病理生理学的关键亚基 18,19,20。随后探索这些预测变异与以结构和电生理数据为特征的癫痫相关突变的位置特异性匹配。然后,这些数据用于估计对表达 GABAA 受体亚型的海马锥体神经元模型的变异影响,该亚型由 γ2、α1 和 β3 亚基(γ2-GABAA 受体)组成,负责快速突触抑制6。需要注意的是,GABAA 受体从一个大的亚基池(α1-α6、β1-β3、γ1-γ3、δ、Ε、θ、π 和 ρ1-ρ3)组装而成,根据亚基组成,GABAA 受体的调节、生物物理特性以及区域、细胞和亚细胞表达模式与特定功能相结合 6,21,22,23,24,25.因此,本研究仅侧重于 γ2-GABAA 受体或含有 γ2 的 GABAA 受体。
GABAA 受体亚基由特征性结构特征组成——一个长的 N 末端细胞外结构域 (ECD)、四个跨膜结构域(TM1 至 TM4)、一个连接 TM1 和 TM2 的细胞内接头、一个连接 TM2 和 TM3 的细胞外接头、TM3 和 TM4 之间的一个大细胞内环(TM3-TM4 环)和一个短的细胞外 C 末端 6,26,27.研究表明 GABAA 受体通过复杂的“锁和拉”机制发挥作用,其中 GABA 结合锁定β和 α 亚基,使它们拉动亚基的细胞外结构域 (ECD),逆时针旋转它们 27。这种运动弯曲了跨膜结构域 (TMD),从而打开了离子通道27。因此,通道活性似乎与 GABAA 受体内的结构盒一起协调。事实证明,癫痫突变通过这些结构盒的扭曲导致通道活动功能障碍28。因此,我们的研究基于这样一种想法,即在 GABAA 受体亚基的特定结构盒中功能鉴定的致癫痫突变附近预测的致病性变异可能表现出类似的通道功能电生理或生物物理扭曲模式,如在这些致癫痫突变的情况下观察到的那样。虽然 GABAA 受体亚基28 中存在致癫痫结构盒间接支持这一观点,但我们的研究证明了将致癫痫突变的生物物理参数与预测的致病突变的生物物理参数相关联的复杂性和挑战。为了揭示这些复杂的关系,我们的框架具有重要意义,因为它强调了一种多尺度方法,范围从 DNA 到蛋白质功能和神经行为,对癫痫研究至关重要。这种方法将计算遗传学与分子建模和神经模拟相结合,同时还强调了互补方法的重要性,例如在大型数据集上训练的机器学习,这些方法可以捕获突变对通道结构、活性和神经兴奋性的影响。此外,在海马锥体神经元模型上模拟致癫痫 γ2-GABAA 受体活性允许复制与 GABAA 受体通道病相关的体外细胞表型,并证明网络功能障碍中心单神经元反应改变。
研究方案
1 . 致 病性变异的计算机预测
- 变体数据收集
- 使用 ClinVar 数据库29,通过网站在目标基因的编码区搜索意义不确定的变异 (VUS):https://www.ncbi.nlm.nih.gov/clinvar/。在搜索栏中输入基因符号(例如 GABRG2)并过滤结果以仅包含所需的变异类型,例如单核苷酸、意义不确定的错义变异。下载数据并将其保存为 data.xlxs (补充文件 4:补充表 S1)。记录下载数据的日期。
注意:在本方案中,将分析 GABAA 受体的人类 γ2 亚基,特别是 智人 γ-氨基丁酸 A 型受体亚基 γ2 (GABRG2),转录变体 1,mRNA(NCBI 参考文献如下:NM_198904.4),也称为 γ2L。记录目标基因的参考转录本以及不同数据库(UniProt、ENSEMBL、PDB)中的其他相应标识符非常重要,因为不同的计算方法可能需要不同的标识符(补充文件 4:补充表 S2)。如果数据库或计算工具无法识别序列标识符的版本号,请尝试使用带有版本号 (NM_198904.4) 和不带版本号 (NM_198904) 的 ID。 - 参比蛋白基本信息
- 在 NCBI 数据库 https://www.ncbi.nlm.nih.gov/ 中,在搜索选项中选择核苷酸,然后输入目标基因的 NCBI 参考文献 ID (NM_198904.4)。然后,向下滚动右列,单击类别 Related information 下的 Protein,以查找由转录本 NM_198904.4 编码的蛋白质 (NP_944494.1)。使用为蛋白质 NP_944494.1 提供的信息,以表格的形式记录特定区域的序列位置(补充文件 4:补充表 S3)。
注:确定功能和结构关键区域、基序或残基(如蛋白质结构域、磷酸化位点、配体结合位点和分子相互作用界面)的序列位置的初步已知信息非常重要。这可以通过结合数据库(NCBI、ENSEMBL、UniProt...)和文献检索来实现。
- 在 NCBI 数据库 https://www.ncbi.nlm.nih.gov/ 中,在搜索选项中选择核苷酸,然后输入目标基因的 NCBI 参考文献 ID (NM_198904.4)。然后,向下滚动右列,单击类别 Related information 下的 Protein,以查找由转录本 NM_198904.4 编码的蛋白质 (NP_944494.1)。使用为蛋白质 NP_944494.1 提供的信息,以表格的形式记录特定区域的序列位置(补充文件 4:补充表 S3)。
- 使用 ClinVar 数据库29,通过网站在目标基因的编码区搜索意义不确定的变异 (VUS):https://www.ncbi.nlm.nih.gov/clinvar/。在搜索栏中输入基因符号(例如 GABRG2)并过滤结果以仅包含所需的变异类型,例如单核苷酸、意义不确定的错义变异。下载数据并将其保存为 data.xlxs (补充文件 4:补充表 S1)。记录下载数据的日期。
- 变体数据组织
- 组织数据以满足所选预测变量的输入要求。确保检索到的数据的格式已组织起来,以符合 dbNSFP 服务器 http://database.liulab.science/dbNSFP 的要求。为此,请从 data.xlsx 文件(补充文件 4:步骤 1.1.1 中的补充表 S1 )中删除不必要的列,仅按指定顺序保留以下列:
“GRCh38染色体”, “GRCh38位置”, “名称”, “蛋白质变化”。 - 以新文件名保存文件:“data1.xlsx”(补充表 S4)。通过运行代码在 R 中格式化 data1.xlsx 文件 (补充文件 1:Data_GABAA。R),它将格式化的数据保存为 data1_output.xlsx (补充文件 4:补充表 S5)在与 R 项目相关的工作目录中。
注意:不同的计算方法需要不同的数据类型和格式。根据特定格式要求收集和组织数据,即使对于十几个变体,也容易出现错误且耗时,因此此步骤非常重要,除非变体池仅由几个变体组成。然后,手动数据组织可能是可能的。
- 组织数据以满足所选预测变量的输入要求。确保检索到的数据的格式已组织起来,以符合 dbNSFP 服务器 http://database.liulab.science/dbNSFP 的要求。为此,请从 data.xlsx 文件(补充文件 4:步骤 1.1.1 中的补充表 S1 )中删除不必要的列,仅按指定顺序保留以下列:
- 致病性预测
- 将 data1_output.xlsx 文件的内容传输到通过 http://database.liulab.science/dbNSFP 访问的 dbNSFP 服务器30,31 的学术版本中。为此,请复制/粘贴或直接上传 .txt 格式的文件。
- 在提交之前,请确保在服务器中预先选择并确认以下选项:HG38(基因组构建)、ClinPred32 和 BayesDEL33 。几分钟后,服务器将生成结果。
注意:在本协议中,选择了两个集成预测因子,即 BayesDEL33 和 ClinPred32,以实现高精度34 和实用性。但是,也可以选择其他预测变量,例如 dbNSFP 数据库30,31 中提供的 AlphaMissense。计算机工具的选择取决于几个因素,包括为强大的预测生成足够的多行计算证据12。集成多个预测算法分析的集成预测器可以达到此目的。 - 下载输出文件(.txt格式)并将其另存为 data2.xlsx(补充文件 4:补充表 S6)。
- 通过单击菜单中的过滤器选项来设置 data2.xlsx(补充文件 4:补充表 S6)中的过滤器,并通过过滤 D 来确定两列中的共识变体。这将给出致病性最强的变体列表;保存它(参见补充表 S6 [ 补充文件 4] 中的共识选项卡)。
- 变体选择
- 在共识致病性预测中,确定从文献中获得的致癫痫突变附近的变异。确保后者具有适合神经元建模的结构和生物物理参数。
注意:此步骤是探索性的,也与研究目标蛋白质的结构、物理化学和生物物理参数有关。在本研究中,这些数据来自 Brünger 等人 35 和 Guo 等人 36 以及癫痫相关突变的调查。此外,作为一种选择,从 dbNSFP 数据库30,31 重复步骤 1.3 访问 AlphaMissense37 分数(补充文件 4:补充表 S7)。更多详细信息在协议第 2.1.1 节和 2.1.2 以及结果中给出(参见“结构和生物物理参数的聚类变体”)。 - 对于基本的可视化,使用 Protter38 ((https://wlab.ethz.ch/protter/start/) 和 HOPE39 (https://www3.cmbi.umcn.nl/hope/) 服务器在选定的 GABRG2 基因突变的背景下检查上一步中的变体:P302L40 和 K328M(或 K289M41,当排除 39 个残基信号肽时)。
注意:由于复杂性巨大,应在多个分析层次上对变异效应进行结构评价。像 Protter38 这样的工具将允许在蛋白质的拓扑特征的背景下清晰地可视化变异,而 HOPE39 等用户友好的服务器将通过分子建模来深入了解变异效应。此外,对目标蛋白质的全面文献综述对于识别和整合与癫痫相关的突变信息至关重要。 - 进化守恒和结构见解分析
- 打开 Jalview 42,43,44,这是一个用于编辑、可视化和分析蛋白质的开源程序。
- 导入序列以进行对齐。点击顶部菜单中的 File |获取序列;在对话框中选择数据库(如 UniProt);单击 Retrieve IDs 选项卡;,如对话框中所述,输入来自人类和其他脊椎动物物种的目标基因 (GABRG2) 的 UniProt 登录 ID:P18507、P22723、Q6PW52、A0A2I3TKX0、F1RR72、A0A8I3MDZ2、A0A8M1P4D6。单击 OK。
注:GABRG2 编码的蛋白质的 UniProt 登录号如下:智人的 P18507 (P18507-2),Mus musculus 的 P22723,Pan troglodytes的 A0A2I3TKX0,Sus scrofa 的 F1RR72,Canis familiaris 的 A0A8I3MDZ2,Danio rerio的 A0A8M1P4D6。 - 根据感兴趣的基因,一些序列可能没有注释;因此,执行 BLAST 搜索以识别相关信息和潜在的同源物,以便更好地理解上下文。在这种情况下,通过 File 菜单下的 Add Sequences/From 文本框选项上传蛋白质序列的 FASTA 格式,以生成所需序列的多个序列比对。
- 加载比对后,观察显示的序列以进行多个序列比较。每行表示一个序列,每列表示比对中的一个位置。要确定最佳对齐方法,请使用不同的方法;例如,单击 Sequence 菜单中的 Web 服务,然后选择 Run T-Coffee with preset 选项,这样就可以实现最佳对齐。
- 右键单击序列 P18507 Homo sapiens (本研究中的参考序列)并将其设置为 参考序列。在上方菜单中选择 Format (格式 ),然后单击 Wrap (换行 ) 以在屏幕上可视化完全对齐。在相同的 Format (格式) 菜单中,单击 上面的刻度 以增强特定残基数的可视化。为了进一步增强可视化效果,请通过转到 Color 并选择不同的选项(例如,Clustal Color、Chemical Property)来调整配色方案;如果需要,请修改字体大小。
- 单击菜单栏中的 Calculate ,然后选择 Autocalculate 一致 以突出显示保守区域。
- 专注于在 计算机模拟 预测步骤中识别出的感兴趣变体的位置,并检查特定的变体位置。通过右键单击特定残基并选择 Add Annotation 来注释它们。使用适当的颜色代码编写标签(例如,变体 ID)并保存。
注:在本分析中,选择了 P302L(紫色)和 A303T(红色),以便在多序列比对中与结构数据一起可视化它们(参见下一节)。
- 完整蛋白质的三维重建,显示选定的保守残基
- 在上一步获得的文件中,右键单击参考序列 (GABRG2 human) 并选择 3D 结构数据。
- 从下拉菜单中确定适当的结构数据 (7QNE, Chain C)26 ,然后选择 Open new structure view with Jmol。
注意:这将允许 Jmol 将多序列比对中选择的残基合并到结构数据中,Jmol 是一种基于 Java 的开源 3D 化学结构查看器。
- 在共识致病性预测中,确定从文献中获得的致癫痫突变附近的变异。确保后者具有适合神经元建模的结构和生物物理参数。
2. 参数选择和生物物理建模
- 变异特异性荟萃分析和参数归一化
- 调查当前文献以收集已鉴定的具有电生理数据通道电导 (gGABAA)、失活时间 (τ失活)、上升时间 (τ上升) 和最大电流振幅 (Imax) 的亚基变体。提供每种情况的亚基组成、细胞类型和野生型测量值。相应地标记变体及其对照(例如,已知具有已确定生物物理特征的变体和已知每个变体的野生型测量对照)。
- 获取具有已识别生物物理特征的变异的 AlphaMissense 致病性评分。
注意:有关更多详细信息,请参阅协议第 1.3 节。 - 创建一个数据框,其中包含每个变体的亚基和氨基酸位置、原始和改变的氨基酸、致病性评分以及从文献中获得的生物物理参数。为避免实验差异,将已鉴定变异的生物物理参数标准化为野生型测量的 x 倍变化。
- 按结构和功能特性进行比较变异分析
- 在数据帧上组织预测的变体;相应地标记(例如,在没有关于其生物物理特征的文献的情况下预测变体)。
- 根据变体在氨基酸序列和三级结构中的位置对变体进行分类。在数据帧上添加结构分类参数(例如,在 α 螺旋、线圈、β 折叠、细胞外、细胞内或跨膜结构域中的定位、孔衬里、激动剂结合、蛋白质-蛋白质相互作用),并为每个变体提供有关其氨基酸位置的信息。
- 根据它们到膜中心和孔轴的距离对变体进行分类。在数据框上添加孔轴的距离和膜中心参数的距离。
- 分析已知变体的结构和生物物理参数之间的相关性。如果可能,请根据获得的相关性评估预测的变体。
- Synapse 和神经元模型构建
- 使用 Brian245(一种用 Python 开发的开源神经模拟器,用于建模和模拟脉冲神经网络),在基于多区室电导的海马锥体神经元上构建 GABA 能突触的多区室生物物理模型。
- 通过定义离子通道门控动力学、被动和主动参数以及突触后电导来设计基于电导的模型。定义基于电导的模型,如 补充文件 2 中给出,它描述了模型中使用的方程。
- 将膜电容(CM) 设置为 1 μF/cm2 ,将细胞内电阻(RA) 设置为 200 Ω.cm。
- 对海马锥体神经元39 使用改良的 Hodgkin-Huxley 型电导,其中 gL = 0.0003 S/cm2,g K = 0.036 S/cm2,E L = -76.5 mV,ENa = 50 mV,EK = -90 mV。
- 将 NaV 通道在 gNa 上的密度分布调整为 0.05 S / cm2 对于体细胞,0.5 S / cm2 用于轴突初始段 (AIS) 和 Ranvier 节点 (NR),以及 0.005 S / cm2 用于树突。在有髓段中将 gK 和 gNa 设置为 0。
- 如补充文件 2 中所述,为 NaV 和 KV 构建离子通道门控动力学。
- 引入突触电流 (Isyn) 作为区室中所有谷氨酸能突触和 GABA 能突触的总和。在谷氨酸能电流 (Iglu) 中包括快速 AMPA 受体介导的电流 (IAMPA) 和慢速 NMDA 受体介导的电流 (INMDA)。在 GABA 能电流 (IGABA) 中仅包括快速 GABAA 受体介导的电流。假设每个突触前尖峰都会向突触释放一定量的谷氨酸;因此,受体的激活是刺突时间依赖性的(sAMPA 和 sNMDA),总受体电导率(gAMPA 和 gNMDA)反映了每个事件释放的谷氨酸量。
- 使用 补充文件 2 中所述的突触模型。
注意:有关方程的详细说明,请参阅描述模型中使用的方程的 补充文件 2 。
- 从以前的文献中获得 soma 和 neurites 的实验测量直径以及每个神经突隔室和分支模式的长度46,47。通过将细胞划分为多个隔室,将真实的神经元形态简化为多区室模型,从而准确保留主要分支结构并保持双侧对称性。
- 设置形态学(段长和直径;即d_soma:30 μm;l_AH:5 μm;d_AH_i:1.5 μm;d_AH_f:1.3 μm;l_AIS:40 μm;d_axon:1 μm;l_myseg:100 μm;l_NR:2 μm;l_AxTer:4 μm;d_AxTer:2 μm;l_approx:100 μm;l_apmed: 100 微米;l_apdis:200 μm;d_approx_i:4 μm;d_approx_f:3 μm;d_apmed : 2 μm;d_apdis:2 μm;l_apLM:70 μm;d_apLM:2 μm;l_nAcDbasal:400 μm;d_nAcDbasal:1.4 μm;l_nAcDbasal_stem:20 μm;d_nAcDbasal_stem:1.5 μm)和生物物理参数(如第 2.3.2 节中给出)对于锥体神经元模型 46,47,Python 脚本(补充文件 3:GABAAvar.py)中也有详细说明。
- 通过评估在步骤 2.1.1 中获得的野生型对照测量值,确定 GABA 能突触模型的生物物理参数。
- 设计神经元模型的拓扑结构并分配形态学和生物物理参数,其中包括根据先前获得的形态学和分支信息指定隔室的空间排列和互连。为模型的每个隔室分配适当的形态学(例如,节段长度和直径)和生物物理参数(第 2.3.2 节),如 补充文件 3:GABAAvar.py 中所述。
- 突触构建和电流注入
- 使用 SpikeGeneratorGroup (Brian2 库中的一个类)创建突触前活动,如 “GABAAvar.py”(补充文件 3) 中给出。使用 Synapses 类将尖峰发生器连接到模型神经元的目标隔室,以对突触连接进行建模。
- 将持续恒定电流 (Iinj) 设置为 0.85 nA 并放置在 soma 上,以模拟在给定时间由基线离子电流负载驱动的亚阈值活动,如 补充文件 3:GABAAvar.py 中所述。
- 要构建记录监视器,请使用 StateMonitor 记录来自目标隔室的电压轨迹。
- 构建并运行网络。
- 使用 Network 使用模型 neuron、connections 和 monitor 构建网络。
- 通过 defaultclock.dt 设置模拟的时间步长(例如,0.01 ms)。
- 使用 network.run(T*ms) 在网络上运行模拟,其中 T 在示例中设置为 1000 毫秒。
- 测试 GABAA 受体错义突变的影响
- 通过步骤 2.1.1 中收集的生物物理参数定义每个错义突变对通道动力学的影响。
- 通过更改这些参数来运行刺激,并使用“GABAAvar.py”(补充文件 3)中给出的“matplotlib.pyplot”绘制结果。
- 测试参数组合以分析射击模式和速率的变化。绘制结果以进行比较。
结果
本研究利用多尺度方法来预测和表征 GABA A 受体 γ2 亚基的致病性变异,GABAA 受体是癫痫病理生理学的关键组成部分。通过使用预测模型、分子建模、进化保守、结构检查、相关性分析和神经模拟,这种方法增强了变异的分类,与癫痫研究和临床应用具有重要相关性。该方法的总体总结如图 1 所示。
两个相邻 γ2 亚基突变的比较评价
基于我们的假设,即 GABAA 受体亚基中与致癫痫突变相邻的预测致病性突变可能会对通道功能和神经行为产生类似的电生理效应,我们首先对众所周知的致癫痫突变与 γ2 亚基的近端预测突变之间的关系进行了简要检查。
在预测为致病性的变体(补充表 S6)中,选择 A303T (rs1581439874,ClinVar 登录信息:VCV000663033.6) 作为示例。除了通过集成模型进行预测外,A303T 的致病性还通过 AlphaMissense 评分得到证实 (补充文件 4:补充表 S7)。A303T 位于 GABAA 受体 γ2 亚基的第二个跨膜结构域,位于致癫痫突变 P302L40 旁边,如图 2 所示。通过分子建模评估,γ2P302L 和 γ2A303T 取代都导致氨基酸具有更大的侧链,如图 3 所示。在 γ2P302L 突变中,突变体残基和野生型残基都是非极性的,而在 γ2A303T 中,突变残基具有极性侧链,野生型残基具有非极性侧链。P302 和 Ala303 均位于与 β3 亚基的亚基相互作用界面中(分别在 7QNB 和 7QNA 中观察到)。P302 和 Ala303 均具有相当的溶剂可及表面积 (SASA)。此外,这两种残留物在脊椎动物进化的整个跨度中都是 100% 保守的(图 4,上图)。它们都位于 γ2 亚基的第二个跨膜区域(TM2 结构域)附近,如 GABAA 受体蛋白的三维重建(7QNE26,其中 A303(红色所示)是该结构域中的第一个残基(图 4)。基于这些可比特征并使用锥体神经元模型,近端致痫突变(例如 γ2 亚基突变 P302L40 )的模拟可用于初步表征预测变体 (γ2A303T) 对神经反应的影响。下一步,我们将分析扩展到 GABAA 受体亚基内更广泛的变体集。
结构和生物物理参数的聚类变体
在上一节中对两个相邻突变进行比较评估之后,我们实施了一种系统方法来评估是否可以识别变体之间的共享分子特征。此阶段旨在探索氨基酸和变体的结构、物理化学和生物物理特征是否出现一致的模式,从而为我们的初始假设提供进一步的支持。
本研究中使用的数据框架和参考文献在补充文件 4 中提供:补充表 S840、48、49、50、51、52、53、54、55、56、57、58、59、60、 61,62,63,补充表S936 和 补充表S1035。此外,还确定了每个亚基和所有没有亚基区别的变体的结构和生物物理参数之间的相关性(补充文件 4:补充表 S11、补充表 S12、补充表 S13、补充表 S14 和补充表 S15)。从 Brünger 等人那里获得了有关结构参数(α 螺旋、线圈、β 折叠的定位;细胞外、细胞内或跨膜结构域;孔衬里、激动剂/变构结合和蛋白质-蛋白质相互作用)的信息35。生物物理参数是通过对人胚胎肾 (HEK) 293 细胞的膜片钳电生理学研究获得的。用相应的野生型受体激活时间 (τr) 和失活时间 (τd) 常数对值进行归一化。
由于我们的研究基于这样一种观点,即 GABAA 受体亚基中功能鉴定的突变附近或附近的氨基酸变体可能表现出类似的通道功能电生理变化模式,正如在这些突变的情况下观察到的那样,我们探索了结构、物理化学和生物物理参数之间关系的可能性。图 5 和图 6 给出了变体相对于它们与膜中心和孔轴的距离的位置。在这种情况下,我们还使用了 AlphaMissense37 的分数(补充文件 4:补充表 S7);由高精度蛋白质结构预测模型 AlphaFold264 提供支持,该模型可以利用碱性氨基酸序列作为输入。AlphaMissense 可以为单个氨基酸取代的结构方面提供线索。图 7 给出了已知(黑色)和预测(红色)变体相对于 GABAA 受体亚基(分别由 GABRA1、GABRB3、GABRG2 基因编码的 α1、β3、γ2 亚基)的变体位置(氨基酸位置、到膜中心的距离和到孔轴的距离)的 AlphaMissense 分数分布。
图 7A-C 显示了跨氨基酸位置的 Alpha 错义分数分布,图 7D-F 显示了距孔轴归一化距离的 AlphaMissense 分数分布,图 7G-I 显示了距膜中心距离的 AlphaMissense 分数分布。图 7 中的相关性分析表明,通过结构特性确定潜在关系以预测新发现的变异的结果是困难的。b2 亚基 (由 GABRB2 基因编码) 变体包含在聚类和相关性部分,以便能够进行更广泛的分析。然而,只有 GABRA1 编码的 α1 亚基的变体(图 7A,D,G),GABRB3 编码的 β3 亚基(图 7B,E,H)和 GABRBG2 编码的 γ2 亚基的变体(图 7C,F,I)包含在生物物理模型中,因为该模型侧重于海马锥体神经元的功能和 GABAA 的 α1β3γ2 组合受体亚基是海马体中最广泛的组合65。同样,在 α1β3γ2 GABAA 受体中未研究通道动力学的任何 α1、β3 或 γ2 变体也被排除在模拟之外。在本分析中,AlphaMissense 评分与源自 GABAA 受体突变影响的生物物理参数 (标准化激活和失活时间) 之间存在轻微的相关性 (补充图 S1 和补充图 S2) (补充文件 4 和补充表 S8)。这表明预测为致病性的突变(基于 AlphaMissense 评分)也可能导致受体动力学(例如,激活和失活时间)发生可测量的、潜在的破坏性变化。然而,图 7 中的位置相关性之间缺乏相关性使得很难将 AlphaMissense 分数用于我们的假设,该假设基于相邻氨基酸对生物物理特性产生类似影响的想法。
相对于已知变体的激活和失活动力学的归一化孔轴距离分布如图 8 和 图 9 所示。γ2 亚基存在轻微的相关性(图 8C),这表明我们的假设(基于相邻氨基酸将具有类似后果的假设)可能在某些区域成立,特别是在受体通道的孔区附近,TM2 结构域。该区域与我们的参考致癫痫突变相邻(图 2 和 图 4; γ2P302),使其成为神经模拟的相对良好的候选者。基于此,可以粗略估计相邻预测突变(如 γ2A303T)的影响(图 2 和 图 4)。我们在这里提供的结果仅考虑了 α1β3γ2 的测量;因此,在我们的模型中评估的变异被限制为 补充文件 4:补充表 S16 中给出的变异。
突变对 GABAA 受体介导的 CA1 锥体神经元放电抑制的影响
突变对 GABAA 受体介导的抑制作用在基于多房传导的 CA1 锥体神经元模型上得到证明。GABAA 受体错义变异对海马锥体神经元功能的影响可以通过 GABA 能分流神经元的顶端输入来探索,来自 CA3 和内嗅皮层 (EC) III 锥体神经元的投射。换句话说,模拟 GABAA 受体活性的一种方法是假设模拟代表了关于受体生理意义的现实假设,例如分流抑制,GABA 能抑制机制之一。CA1 海马锥体神经元,通常位于其顶端树突中,在这些区域具有 GABAA 受体,这些受体是 CA3 和 EC III 神经元投射的靶标。因此,这种布置适用于模拟。这个研究问题需要一个具有不同延迟和强度的输入设计。因此,如图 10 所示,三种不同的谷氨酸能突触 (GluS1/2/3) 被放置在远端顶端、内侧顶端和基底树突上,并且它们依次被激活。为了评估突触输入的影响,恒流幅度应保持在最小尖峰触发阈值 (Iinj < Imin) 以下。具有野生型或突变型 GABAA 受体的锥体神经元模型是通过在体细胞处恒流注射 0.85 nA 开始的。然后将 GABA 能突触放置在体细胞上。由尖峰发生器模拟的突触前活动首先在远端顶端树突处启动。内侧根尖和基底树突上的突触输入分别延迟了 25 ms 和 50 ms。GABA 能突触以 40 ms 的延迟激活。调整 GABA 能抑制强度,使除第一个尖峰外的整个刺突序列都受到抑制。然后,在这种情况下,通过改变 τ上升、τ失活和 gGABAA 来探索变体的影响。
野生型和突变型受体的参数是从方案步骤 2.1.1 中描述的集合中获得的,特别是由 α1β3γ2 组成的受体,α1β3γ2 是海马锥体神经元中最丰富的亚基组成65。参数分布在 补充文件 4:补充表 S16 中给出。
在单、双和三谷氨酸能突触病例上测试每个亚基突变。用一种简单的方法,可以通过放电速率和模式来评估突变的影响。ΔtISI 平均值和标准差也可以估计,以进一步衡量发射模式的变化,其中 ΔtISI 表示尖峰间隔的变化。每种情况的结果在补充文件 4:补充表 S17 和补充图 S3 中以发射速率和 ΔtISI(平均值和标准差)给出。图 11、图 12 和图 13 给出了改变触发模式的变体的尖峰序列和电压轨迹。
对于单 (GluS1) 和三重 (GluS1-2-3) 谷氨酸能突触激活,改变神经元反应的突变仅β3个 N110D 和 γ2K328M 突变。在单个谷氨酸能输入情况下,β3N110D 导致抑制受损,并且在第 4 个 突触前尖峰之后,放电模式被锁定在谷氨酸能尖峰序列上,并有短暂的延迟(图 11)。γ2K328M 也损害了抑制作用,尽管仅在第 5 个 突触前尖峰附近,并且与 β3N110D 相比,突触后尖峰引入了更大的延迟(图 11)。在 GluS1-2-3 激活案例 (图 13) 中,β3N110D 和 γ2K328M 突变之间的反应相似。两种突变体都产生了一种放电模式,其中几乎所有累积的突触前尖峰都被检测到并触发了反应。在这两种情况下,神经元模型都响应突触前活动而触发了刺突对。
与其他两种设置相比,双谷氨酸能突触激活产生了不同的结果(图 12)。在这种情况下,GABAA 受体 b3 亚基上的两个突变 (β3N110D 和 β3T288N) 和 GABAA 受体 γ2 亚基上的两个突变 (γ2P302L 和 γ2K328M) 损害了 GABA 能抑制。具有 γ2P302L 突变体的神经元模型几乎与 GluS2 同步触发,这很可能是对 GluS1 的延迟反应,与 GluS1-2 之间突触前尖峰的延迟大致相同。β3T288N 突变产生了类似的结果,第二个刺突的区别仍然与 GluS2 同步。具有 β3N110D 突变体的神经元模型几乎响应了所有累积的谷氨酸能输入,除了 GluS1/2 的前两个突触前尖峰,它们是以较短的 ΔtISI 引入的。γ2K328M 的放电模式再次类似于 β3N110D,错过了第二和第三突触前尖峰的区别。
这些结果表明 b3 亚基 (由 GABRB3 基因编码) 和 γ2 亚基 (由 GABRG2 编码) 突变对海马锥体神经元活性的不同影响。有趣的是,3L170R、β3A305V、β3E180G、β3D120N、β3Y302C 和 γ2R82Q β的突变没有产生任何神经活动变化。抑制最严重的损害是 β3N110D 和 γ2K328M,两者的 τ失活 率显著降低,τ上升率较高。我们的初步分析还表明,仅 τ升高 或 gGABAA 的变化不足以损害抑制作用(数据未显示)。可以说,导致 τ失活显着降低以及 τ升高增加的突变导致 GABA 能抑制更显着的损害。
在必须抑制所有兴奋性输入的情况下,任何导致放电的突变都会产生异常和非特异性的神经反应,这些反应有可能在由具有相同突变的神经元组成的神经回路中被夸大。神经网络中兴奋/抑制的平衡会受到由此产生的抑制反馈受损的显著影响,这是任何网络活动的关键组成部分。
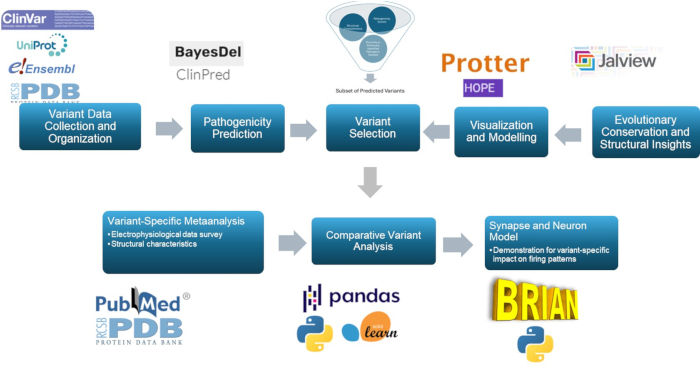
图 1:用于临床和研究目的的变异效应预测和分析概述,特别关注 计算机分析和 神经反应模拟。 请单击此处查看此图的较大版本。

图 2:γ2A303T 的位置和选定的患者突变;γ2P302L 和 γ2K328L 用于神经模拟。 请单击此处查看此图的较大版本。
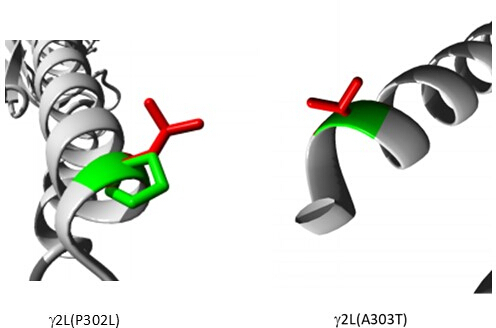
图 3:预测为致病性的患者突变 γ2P302L 和相邻变体 γ2L (A303T) 的比较模型。 在这两个模型中,绿色代表野生型,红色代表突变残基。 请单击此处查看此图的较大版本。
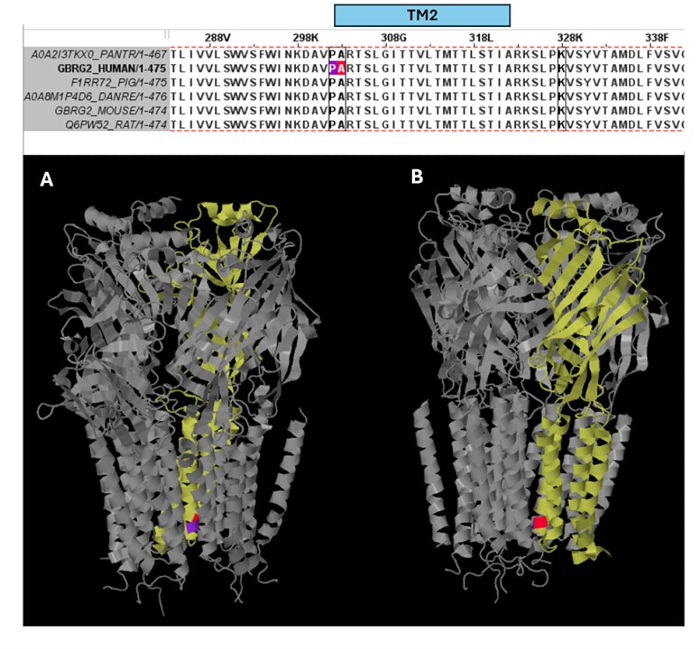
图 4:多序列比对和结构见解。 上图显示了 TM2 边缘患者突变 (P302L)(紫色)和γ2 亚基 TM2 开头的致病性变异 A303T(红色)位置的残基的进化保守性。下图显示了 (A) 三维 GABAA 受体结构 (7QNE) 中这些保守残基的可视化,其中 γ2 亚基(7QNE 中的链 C)从不同角度 (A、B) 显示为黄色。缩写:TM2 = 第二个跨膜结构域。 请单击此处查看此图的较大版本。

图 5:所有包含的变体的本地化。 显示了已知(黑色)和预测(红色)变体相对于其 (A) 距孔轴的归一化距离和 (B) 距膜中心的距离的位置。 请单击此处查看此图的较大版本。
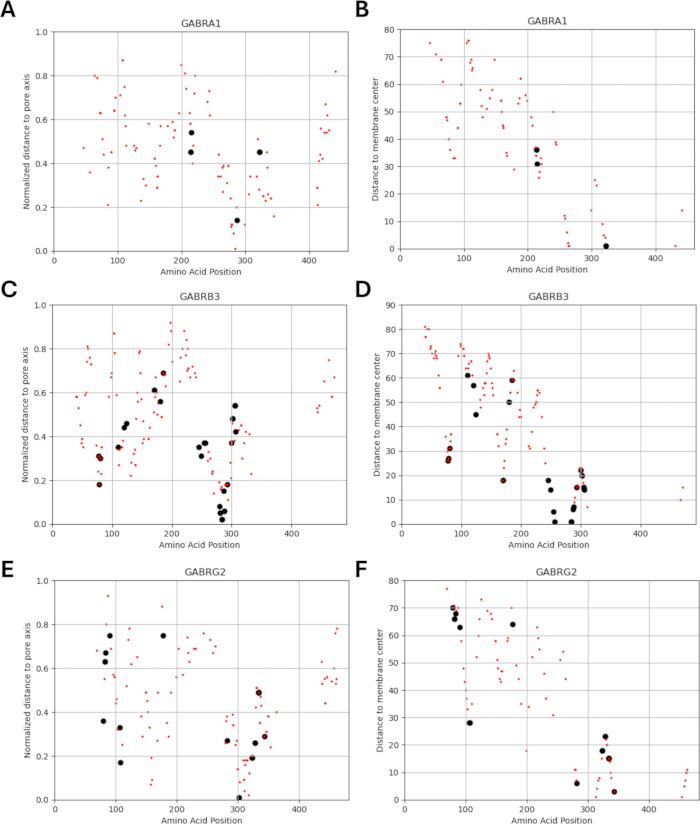
图 6:每个亚基的变体定位。 显示了已知(黑色)和预测(红色)变体相对于其 (A, C, E) 距孔轴的归一化距离和 (B, D, F) 距膜中心的距离的位置。 请单击此处查看此图的较大版本。
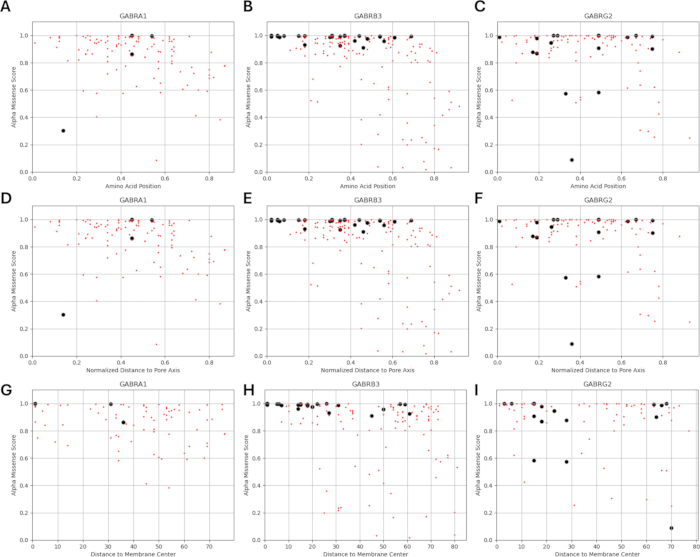
图 7:变体位置的 AlphaMissense 分数分布。 (A-C)对于已知(黑色)和预测的(红色)变体,给出了氨基酸位置的 AlphaMissense 分数分布、(DF) 距孔轴的归一化距离和 (GI) 距膜中心的距离。 请单击此处查看此图的较大版本。
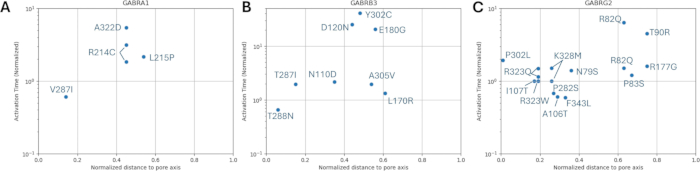
图 8:α1 (GABRA1) 亚基、β3 (GABRB3) 亚基和 γ2 (GABRG2) 亚基突变的 GABAA 受体的标准化激活时间。显示了 (A) α1、(B) β3 和 (C) γ2 亚基上每个突变相对于孔轴的归一化距离的实验获得的激活时间常数。用相应的野生型受体激活时间对值进行归一化。请单击此处查看此图的较大版本。

图 9:GABA A 受体的 α1 (GABRA1) 亚基、GABA A 受体的 β3 (GABRB3) 亚基和 γ2 (GABRG2) 亚基发生突变的 GABAA 受体的标准化失活时间。显示了 (A) α1、(B) β3 和 (C) γ2 亚基上每个突变相对于孔轴归一化距离的实验获得的失活时间常数。用相应的野生型受体失活时间对值进行归一化。请单击此处查看此图的较大版本。

图 10:CA1 锥体神经元模型。 模型神经元由 (1) 体细胞,(2) 具有近端、内侧和远端隔室的顶端树突,在分子层处以两个分支结束,(3) 两个对称组成的基部树突,在短基部树突茎后分支成两部分,以及 (4) 一个轴突,从锥形轴突小丘开始,然后是圆柱形轴突初始段, 有髓节段和 Ranvier 节点,以球形轴突末端结束。绿色三角形表示谷氨酸能突触的位置,红色三角形表示位于体细胞的 GABA 能突触。比例尺 = 100 μm。请点击此处查看此图的较大版本。

图 11:仅具有 GluS1 活性的触发模式。 上图给出了突触前神经元(GluS1(绿色三角形)和 GABA能(红色圆圈))和具有野生型或突变型 GABAA 受体(黑色方块)的突触后神经元的刺突序列。具有野生型 GABAA 受体的神经元具有或不具有 GABA 能抑制的神经元以及具有具有 GABA 能抑制的突变型 GABAA 受体的神经元的单个电压轨迹显示在下面板中。 请单击此处查看此图的较大版本。
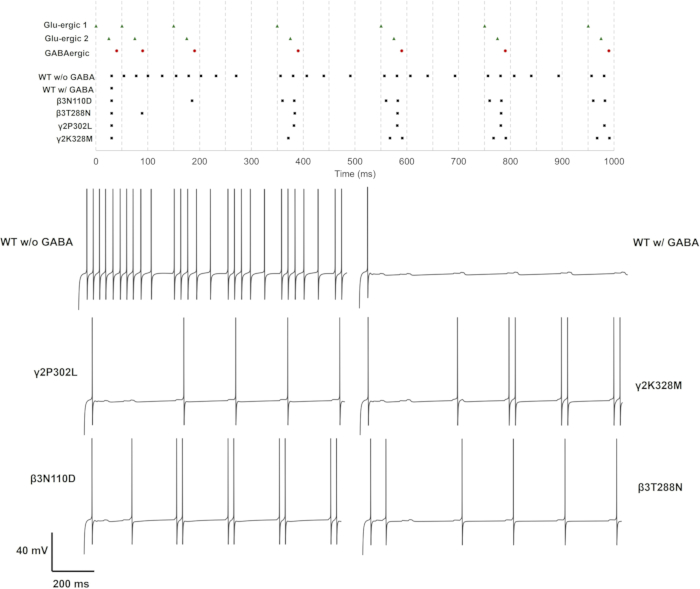
图 12:仅具有 GluS1 和 GluS2 活性的放电模式。 上图给出了突触前神经元(GluS1/2(绿色三角形)和 GABAergic(红色圆圈))和具有野生型或突变型 GABAA 受体(黑色方块)的突触后神经元的刺突序列。具有野生型 GABAA 受体的神经元具有或不具有 GABA 能抑制的神经元以及具有具有 GABA 能抑制的突变型 GABAA 受体的神经元的单个电压轨迹显示在下面板中。 请单击此处查看此图的较大版本。

图 13:仅具有 GluS1、GluS2 和 GluS3 活性的放电模式。 上图给出了突触前神经元(GluS1/2/3(绿色三角形)和 GABA能(红色圆圈))和具有野生型或突变型 GABAA 受体(黑色方块)的突触后神经元的刺突序列。具有野生型 GABAA 受体的神经元具有或不具有 GABA 能抑制的神经元以及具有具有 GABA 能抑制的突变型 GABAA 受体的神经元的单个电压轨迹显示在下面板中。 请单击此处查看此图的较大版本。
补充图 S1:AlphaMissense 分数和生物物理参数的分布(标准化停用时间;归一化 τd) 在本研究中选择的 GABAA 受体亚基突变。另见补充文件 4:补充表 8。请点击此处下载此图表。
补充图 S2:AlphaMissense 分数和生物物理参数的分布(标准化激活时间;本研究中选择的 GABAA 受体亚基突变的标准化 τr)。另见补充文件 4:补充表 8。 请点击此处下载此图表。
补充图 S3:野生型和突变型 GABAA 受体的神经反应的间尖峰区间。 最上面的图表示单个谷氨酸能输入的间尖峰时间间隔。中间的图显示两个,最下面的图显示三个谷氨酸能突触同时活跃。 请点击此处下载此图表。
补充文件 1:文件 “Data_GABAA.R“才能在 R 中运行以进行数据格式化。请点击此处下载此文件。
补充文件 2:基于电导的模型设计中使用的方程。请点击此处下载此文件。
补充文件 3:在 Brian2 中运行神经模拟所需的 GABAAvar.py。 该文件包含基于 Brian2 的多房神经元模型(函数:CA1_Pyr)、基于电导的神经元和突触模型的方程(函数:model_eqns、syn_eqns)和初始参数(函数:biophys_param、morpho_param、syn_param)。 请点击此处下载此文件。
补充文件 4:包含所有补充表的 zip 文件夹。请点击此处下载此文件。
补充表 S1:从 ClinVar 下载的 GABRG2 基因中意义未知的错义变异.txt文件,随后保存为“data.xlxs”。请点击此处下载此表格。
补充表 S2:研究中使用的序列标识符、目标基因的参考转录本(NCBI 参考文献 seq.)和不同数据库中的其他相应标识符)。请点击此处下载此表格。
补充表 S3:结构和功能区域的位置。 由参考转录本 (NM_198904.4) 编码的 γ2 亚基蛋白 (NCBI 参考序列: NP_944494.1) 的特定区域的位置 请点击此处下载此表格。
补充表 S4:文件“data1.xlxs”的内容,它代表 GABRG2 的 ClinVar 数据,仅包括列:“GRCh38Chromosome”、“GRCh38Location”、“Name”、“Protein change”。请点击此处下载此表格。
补充表 S5:“data1_output.xlsx”文件的内容,其中包含要上传到 dbNSFP 服务器以进行变异效应预测的 GABRG2 数据的错义变体所需的格式。请点击此处下载此表格。
补充表 S6:“data2.xlsx”文件的内容,其中包含 dbNSFP 服务器的输出,用于预测 GABRG2 的未知错义变体的变体效应。请点击此处下载此表格。
补充表 S7:GABAA 受体亚基变体的 AlphaMissense 评分。请点击此处下载此表格。
补充表 S8:变体的生物物理特性。 生物物理参数的值是从以前的电生理学实验中获得的。变体用 “S” (替换)类型标记,而野生型受体参数为每个替换给出,并用 “C” (对照)标记。τd :停用时间常数,P开路 :开路概率,gGABA:受体电导,Imax:最大电流,τr :激活时间常数。 请点击此处下载此表格。
补充表 S9:变体的物理化学特性。 先前识别的具有生物物理参数的变体用 “S” (替代)类型标记,而预测的变体用 “P” 表示。H:疏水性的变化,VSC:侧链体积的变化,P1:极性的变化,P2:极化的变化,SASA:溶剂可及表面的变化,NCISC:净电荷指数的变化。从 Guo 等人 36 获得每个原始氨基酸和变体的值,并按给定方式估计每个参数的变化。 请点击此处下载此表格。
补充表 S10:变体的结构特征。 先前识别的具有生物物理参数的变体用 “S” (替代)类型标记,而预测的变体用 “P” 表示。域中变体的本地化用 1 表示,否则用 0 表示。所有值均来自 Brünger 等人35。 请点击此处下载此表格。
补充表 S11:所有已知变体的结构和生物物理参数相关性。请点击此处下载此表格。
补充表 S12:已知 GABRA1 变体的结构、物理化学和生物物理参数相关性。 请单击此处下载此表格。
补充表 S13:已知 GABRB2 变体的结构、物理化学和生物物理参数相关性。 请点击此处下载此表格。
补充表 S14:已知 GABRB3 变体的结构、物理化学和生物物理参数相关性。 请点击此处下载此表格。
补充表 S15:已知 GABRG2 变体的结构、物理化学和生物物理参数相关性。 请点击此处下载此表格。
补充表 S16:野生型和突变型 α1β3γ2 GABAA 受体的生物物理参数。请点击此处下载此表格。
补充表 S17:响应野生型和突变型 GABAA 受体的单、双或三重谷氨酸能突触的放电速率和刺间间隔。 请点击此处下载此表格。
讨论
通过应用计算遗传学、分子建模和神经模拟的结合,本文提出的方法有可能改善 GABAA 受体变体的分类,为癫痫研究和临床应用提供有价值的见解。提出了用于识别和确定预测致病突变优先级的综合分析,并将其扩展到一个框架中,该框架可能弥合变异对蛋白质和细胞表型的影响之间的差距。评估致癫痫性 GABAA 受体活性对海马锥体神经元模拟的影响,可以复制与 GABAA 受体功能障碍相关的体外表型,并证明网络功能障碍根源中的单个神经元反应改变。基于这些对致癫痫突变产生的神经反应的模拟,探索了对结构近端预测突变的功能影响的粗略估计。预测突变对通道动力学影响的预测需要对已知的变异集进行彻底分析。比较分析(例如本文中介绍的分析)和神经活动模拟为进一步生成和改进预测模型提供了关键见解,这些模型侧重于变异对蛋白质功能和神经病理学的影响。此外,我们的方法可用于在未知变异中选择和优先考虑最具致病性的变异,以检查与 GABAA 受体相关的神经发育障碍相关的变异效应。例如,用荧光探针66、67、68、69、70 标记并携带预测突变的受体亚基可以在体外表达,以研究它们的运输、细胞表面表达和神经生理学。此外,可以考虑使用秀丽隐杆线虫等动物模型来验证预测突变的效果。例如,CRISPR-Cas9 基因编辑已被用于产生秀丽隐杆线虫 GABAA 受体 unc-49 的缺失,从而在 unc-49 或人 GABAA 受体71 的亚基中产生纯合癫痫相关突变。
一般来说,正如 ACMG-AMP12 所建议的,变体的分类受益于使用多层次的计算证据。这种方法通过集成不同的预测工具和数据源来增强变异分类的可靠性,最终提高临床评估的准确性并改善基因组诊断的整体决策过程。在我们的方法中,利用集成预测器,它结合了多种工具的预测,从而满足了多行计算证据的要求,并且无需单独使用不同工具,这是一个优势。这种方法还克服了处理来自单个工具的不同输出的挑战,从而简化了预测过程并提高了效率。然而,无法保证以基因为中心或变异特异性分析的预测准确性。这导致结论,以基因为中心或变异特异性的预测应该在针对特定环境和目标进行调整的特定条件下进行 15,72,73,74。对于临床干预,这将需要在给定疾病的背景下评估计算机工具对特定基因或基因子集的预测准确性,通常进行个体化优化75。但是,预测准确性的评估通常受到缺乏足够数量的变体的限制,这可能会影响准确性评估的可靠性。
文献中提供了不同的工具,它们的准确性在数据集14 中得到了测试和验证。然而,这些基于大型数据集的准确性结果不一定反映在对给定基因的一些未知变异的预测上。在这种情况下,积累的文献表明,众所周知,编译和计算单个预测变量结果的集成预测器比单个预测变量的一致性表现更好 33,76,77,78,因此,在本研究中,我们选择使用集成预测器,即 BayesDEL33 和 ClinPred32,特别是因为它们的卓越性能32,34 BayesDEL 对临床相关基因中的 4,094 个错义变异进行了比较评估,包括编码跨膜蛋白的基因,如电压门控钠通道 α 亚基 5 (SCN5A),并显示出优异的性能33。在我们的变体效应预测方案中,作为第一步,我们考虑了两个集成预测因子(BayesDEL 和 ClinPred)的共识。AlphaMissense37 是由 Google DeepMind 开发的深度学习模型,是 AlphaFold 64,79 的扩展,因此利用了高精度蛋白质结构预测的能力。当我们将集成模型(BayesDEL 和 ClinPred,如我们的协议步骤 1.3 中所述)的初始预测结果与 AlphaMissense 的结果进行比较时,预测彼此部分一致(补充文件 4:补充表 S15),与集成模型(BayesDEL 和 ClinPred)的预测不完全一致,后者达成了致病性或疾病相关的共识, 显示为粉红色行(补充文件 4:补充表 S15)。然而,我们在神经元模型中使用的 GABRG2 突变 R82Q、P302L 和 K328M 附近的未知变异(L81F、A303T 和 V329F),并被 ClinPred 和 BayesDEL 预测为致病性,也被 AlphaMissense 预测为致病性,如黄色突出显示所示(补充文件 4:补充表 S15)。
由于 AlphaMissense29 使用序列和结构上下文预测,在我们的研究中,我们还想根据 AlphaMissense 评分和 GABAA 受体突变位置与膜中心和孔轴的距离,查看它们之间是否存在任何关联。我们的假设基于这样一种想法,即与 GABAA 受体亚基的功能鉴定突变相邻或近端的氨基酸变体的功能影响可能显示出在突变情况下观察到的通道功能的类似物理化学变化模式。GABAA 受体亚基突变位置与 AlphaMissense 评分之间的相关性将帮助我们确定可用的关系,为我们的假设建立一个框架,从而允许预测 GABAA 受体亚基中新错义变异的功能后果。然而,AlphaMissense 评分不能预测这些生物物理参数的变化(图 7)。需要注意的是,我们分析中的样本量有限,因此很难得出明确的结论。然而,我们的分析发现 AlphaMissense 评分与 GABAA 受体结构参数无关。缺乏明确的位置相关性(例如,突变的位置和 AlphaMissense 分数之间)挑战了我们假设的有效性。如果相邻残基确实具有相似的效果,我们预计会看到更清晰的相关性。由于情况并非如此,它削弱了使用 AlphaMissense 分数作为测试假设的可靠工具的能力。
有趣的是,在我们的研究中,我们发现 GABRG2 基因突变体的变体到孔轴的距离与归一化通道激活时间之间存在轻微的相关性。因此,我们初步假设相邻氨基酸将产生类似的后果,这可能在通道的某些区域成立,例如孔中区域或涉及门控的关键位点,但在其他区域可能不那么明确。较小的数据集限制了辨别这种可变性的能力,但未来的数据或更详细的结构分析可能有助于完善我们假设的这一方面。分子动力学模拟80 可以作为进一步研究这些初步发现的有力补充方法,尤其是在两个相邻的 γ2 亚基突变的比较评估中,即致癫痫突变 γ2P302L40 和近端预测突变 γ2A303T (rs1581439874),在我们的研究中进行。将来,这种方法可以更准确地估计未知变异对细胞表型的影响,尤其是与我们研究中提出的神经模拟相结合时。
此外,探索 GABAA 受体亚基的结构和物理化学特性以及其他特征是否可用于训练强大的机器学习模型,以对通道、神经元、网络和疾病表型的新变体效应进行功能预测,这将是一件有趣的事情。随着自动化机器学习方法的出现,我们已经达到了医生和湿实验室科学家也可以在更加民主化的环境中开发自己的模型的地步81。因此,将这些技术整合到临床实践中可能会简化流程,使个性化医疗更容易获得,并减少对功能变异分析的高度专业化专业知识的依赖。在这种情况下,我们的方法提供了对受体结构和功能动力学的见解,可能有助于未来研究变异效应的功能预测。
尽管目前蛋白质结构预测取得了进展,并以 AlphaFold64 为代表取得了突破,但由于缺乏训练模型所需的数据,准确预测突变和蛋白质功能的影响仍然是一个挑战79.对于变异效应的预测,与预测模型的子集相比,AlphaMissense 显示出更高的性能,但我们研究中使用的集成预测器 BayesDEL25 和 ClinPred24 未包含在该比较中29。值得注意的是,在我们的研究中,计算机工具 BayesDEL、ClinPred 和 AlphaMissense 被用于不同的目的。集成预测因子 BayesDEL 和 ClinPred 主要用于致病性预测,而 AlphaMissense 专门用于探索其分数与 γ2 亚基突变影响的已知数据之间的关系。具体来说,我们的假设假设预测的致病性变异,特别是那些位于 GABAA 受体亚基中功能鉴定突变附近或相邻的变异,将表现出与在功能特征突变中观察到的相似的生物物理参数。为了研究这一点,我们选择了 AlphaMissense,因为它由高度准确的 AlphaFold264 模型提供支持,该模型利用基本肽序列来预测单个氨基酸取代的后果。
因此,我们研究的一个主要局限性主要是由于实验数据的有限可用性。例如,我们的神经元模型基于来自 GABAA 受体的 α1β3γ2 亚基组合的数据的表达,这本质上将文献中研究的突变限制为作为这种特定受体组合的一部分表达的亚基。此外,我们依赖于完全来自这些亚基在 HEK 细胞中的表达的电生理数据,进一步缩小了文献中可用数据的范围。我们使用神经建模来估计未知变异的影响,假设位于已知突变附近的未知变异(在我们的工作流程中预测为致病性)将在文献中描述的通道动力学参数或突变效应的物理化学特性方面表现出相似的模式。这一假设,再加上需要 HEK293 细胞中特定受体组装体的电生理数据,减少了可用于建模的实验数据量。由于这些限制,现有数据允许我们只对 α1β3γ2 亚基中有限数量的变体进行建模。然而,针对不同的亚基组装(例如 α1β2γ2 亚基组合、α1β2δ 或 α4β3δ 亚基组合)训练神经元模型,这些亚基组合具有亚基特异性细胞、回路和网络水平的影响,可能会显示出对各种癫痫类型和神经发育障碍的更广泛适用性。未来,随着可用电生理数据的增加以及专注于明确定义的受体组装体和特定细胞类型突变的研究,有可能提高我们方法的普遍性和准确性。
基于多室电导的神经元模型为单神经元反应的受体变体的功能意义生成预测提供了强大的工具。该工具可以灵活定义细胞/突触参数及其位置,以测试任何特定问题。该协议中使用的简单尖峰发生器可以替换为其他神经元模型来研究微电路活动。该方案的关键步骤也是最具限制性的步骤:根据改变的通道动力学定义任何受体变体。理想情况下,膜片钳电生理学研究可以提供所需的信息;然而,对具有预测临床意义的氨基酸取代的计算分析以及与已知取代的比较也可以提供一些见解。我们的研究和描述的方案将神经活动模拟的使用纳入其中,而不是作为一种预测工具,而不是作为一种探索突变影响的工具,以支持对 GABAA 受体生物物理特性改变对单神经元活动的影响的更广泛观点。在神经模拟中对实验数据的依赖是我们方法的一个重要限制,它可能受益于先进的分子建模来弥合结构和功能之间的差距。
在我们的协议中,应严格评估某些步骤。首先,我们协议第一部分中使用的预测模型的选择可能很关键。计算机工具的选择取决于几个因素,包括为强大的预测生成足够的多行计算证据12。集成预测变量集成多个预测算法的分析更适合此建议,因此与单个预测变量相比,首选。有不同的预测因子,它们的准确性通常在大型数据集中进行测试,这不一定保证用于位于特定基因中的未知变异效应的预测模型的准确性。这可以通过使用两个集成模型进行补偿,这两个模型收集并计算来自多个预测变量的预测结果。此外,如果主要目的是识别致病性最强的变异,则可以调整预测模型的临界值以提高特异性。设置适当的临界值对于平衡敏感性和特异性之间的权衡以及确保变异的准确分类非常重要。在我们的研究中,我们使用了默认截止值。我们特别没有改变临界值,而是选择捕获可能更具致病性的变异,因为这会减少我们工作流程中描述的在多个分析层次中要检查的变异范围。同样重要的是要注意,在为目标蛋白质的三维重建选择结构数据时,需要对文献中的结构数据进行初步审查。GABAA 受体的结构检查最近势头强劲,稳健的结构研究报告了不同条件下不同受体组装体的三维结构 26,27,82,83,84,85。鉴于这些数据的可用性,在我们的研究中,我们专注于用于重建结构数据的实验确定的结构。然而,AlphaFold64 的预测可能有利于分析缺乏此类实验确定数据的其他蛋白质。对于来自实验研究的结构数据,重要的是要注意氨基酸编号。氨基酸的 PDB 编号可能与 UniProt 编号不同,因为前者可能不包括在蛋白质成熟过程中去除的信号肽。此外,在实验系统中表达的嵌合蛋白可能会导致差异。在这种情况下,目标序列与从结构数据得出的序列成对比对将有助于保持一致性。在文献中,γ2 亚基蛋白的结构数据基于不同的方法,包括电子显微镜 (EM) 等实验方法和 AlphaFold 的高精度预测方法。如果实验方法不能以高分辨率完全覆盖所需的序列,则可以使用 AlphaFold 预测。在本研究中,选择结构 7QNE26 是因为它对应于人类全长突触 α1β3γ2 GABAA 受体的低温电子显微镜结构。这恰好代表了全长亚基组合,这是本研究的重点。
此外,对于比较分析,应首选使用通道动力学的归一化参数,因为这些参数的值可能会根据受体亚基组成和实验设置而变化。例如,τ上升 和 τ停用 应通过野生型对照值的 x 倍变化来确定。在方案步骤 2.5 中,对于少量已知变体,可能首选参数或分类相关性分析以及 rho 和 p 值的确定。理想情况下,主成分分析等方法应产生更准确的关系,但需要更多的样本。
可以更改模拟环境。在这项研究中,Brian2 是首选,原因如下: Brian2 中的 spatialneuron 类为模拟神经活动提供了有价值的工具。Brian2 在使用微分方程来描述连续动力学和更新离散事件的语句方面具有显着优势,而不是依赖预定义的“黑盒”模型,这导致了出色的代码可读性和适应性,因为模型的每个方面都可以在单个 Python 脚本中显式定义,模型方程以数学符号表示,并且只使用了少量的 Brian 特定词汇37.由于模型被明确描述,因此所有特征都被记录在案,并且可以在主要仿真描述文件中找到,这消除了对以前依赖的“黑盒”模型的需求,如 Stimberg 等人的研究中所述45,86。
目前的神经元模型利用改进的 Hodgkin-Huxley 型电导46 具有 Na + 、 K + 和泄漏电流。这种基于电导的模型可以通过包含其他几种通道类型(例如 Ca2+ 通道)来进一步扩展。对于突触模型,重要的是要注意,这些参数应该针对特定的亚基组成获得,并且只应评估具有使用这些组合物测量的参数的变体。在本研究中,选择了 α1β3γ2 受体组成;因此,仅包括在 α1β3γ2 GABAA 受体上测量通道动力学参数的 α1、β3 或 γ2 的变体。
细胞生物物理学的估计涉及将细胞分割成多个圆柱形隔室,每个隔室可能具有不同的电导率特性。尽管神经元的树突不规则,但它们可以被视为局部均匀的链。此类模型有助于了解细胞的复杂结构和功能。模型设计侧重于可以反映这些特征的实际形态的简化版本。
改变的氨基酸的位置和理化特性决定了对通道动力学的影响。例如,如果这种变化发生在通道孔内的氨基酸上,则导致氨基酸具有较大的侧链,则会降低通道的电导。替换也可能影响通道的打开/关闭。为了在该模型中简单起见,GABA 结合动力学仅减少到可用受体的比例;然而,可以设计更详细的模型来包括这种相互作用,以研究改变配体结合亲和力的取代的可能影响。
总之,本研究采用计算方法预测 GABAA 受体亚基中的致病性变异,并根据海马锥体神经元模型中癫痫相关突变的模拟定性估计可能的细胞表型。据我们所知,这是第一个探索计算遗传学、分子建模和神经模拟组合应用的方案,以评估遗传变异如何在从 DNA 到蛋白质功能和神经行为的多个复杂层次上导致 GABAA 受体功能障碍。该协议可以为改进对癫痫中潜在有害变异和相关神经病理学的估计提供基础。此外,它可能用于研究其他离子通道病,从而对相关神经发育和网络疾病的潜在机制产生重要见解。基于此,通过结合 GABAA 受体的结构和物理化学评估来检查变体的功能影响及其整合到 GABAA 受体的通道动力学中,将来可以开发更准确的分析。
披露声明
所有作者声明他们与本作品没有利益冲突。
致谢
我们感谢 Çağla Koca 在构建模型神经元方面的帮助。
材料
| Name | Company | Catalog Number | Comments |
| Brian2 | Sorbonne Université, INSERM, CNRS, Institut de la Vision, France; Imperial College London, United Kingdom | 2.8.0.4 | Stimberg et al., 2019 (https://pypi.org/project/Brian2/ ) |
| dbNSFP server | Genos Bioinformatics LLC, USA | v3.0 | Liu et al., 2020 (http://database.liulab.science/dbNSFP) (https://sites.google.com/site/jpopgen/dbNSFP) |
| HOPE | Centre for Molecular and Biomolecular Informatics CMBI, Radboud University, Netherlands | 1.1.1 | Venselaar et al., 2010 (https://www3.cmbi.umcn.nl/hope/) |
| Jalview | University of Dundee, UK | JV2 | Waterhouse et al., 2009 (https://www.jalview.org/) |
| Jupyter Notebook | Project Jupyter, USA | https://jupyter.org/install | |
| Phyton | Python Software Foundation, USA | 3.13 | https://www.python.org/downloads/ |
| Protter | ETH Zurich, Switzerland | Version 1.0 | Omasits, et al., 2014 (https://wlab.ethz.ch/protter/start/) |
| R | The R Foundation for Statistical Computing, USA | R version 4.3.2 | https://www.r-project.org/ |
| RStudio | Posit software, PBC, USA | RStudio 2023.12.1+402 "Ocean Storm" Release | https://posit.co/downloads/ |
参考文献
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。