Method Article
Identifizierung und Klassifizierung von positionsspezifischen GABAA Rezeptor Subunit Missense Varianten für ihre Rolle in hippokampalen Pyramidenneuronen
In diesem Artikel
Zusammenfassung
Diese Studie stellt ein Multiskalen-Framework vor, das von der DNA über die Proteinfunktion bis hin zum neuronalen Verhalten reicht. Es stellt einen neuartigen Ansatz zur Untersuchung vorhergesagter pathogener Mutationen in der GABA-A-Rezeptor-Untereinheit vor und stellt die Hypothese auf, dass epileptogene Mutationen und proximale Mutationen, die als pathogen vorhergesagt werden, ähnliche Effekte auf das CA1-Pyramidenneuronenmodell hervorrufen können.
Zusammenfassung
Das Verständnis der Auswirkungen funktionell unbekannter Varianten in Epilepsie-assoziierten Genen ist entscheidend für die Aufklärung der Pathophysiologie der Krankheit und die Entwicklung personalisierter Therapeutika. Mit einem Multiskalen-Rahmenwerk, das von der DNA-Sequenz über die Proteinfunktion bis hin zum neuronalen Verhalten reicht, beschreiben wir einen neuartigen Ansatz zur Vorhersage und Untersuchung pathogener Mutationen, wobei wir die Hypothese aufstellen, dass epileptogene Mutationen in der GABA-A-Rezeptor-Untereinheit und nahe gelegenen vorhergesagten Mutationen ähnliche Effekte auf das CA1-Pyramidenneuronenmodell haben könnten. Durch die Erforschung der charakteristischen Beziehungen zwischen vorhergesagten pathogenen Mutationen und proximalen epileptogenen Mutationen zielt die Studie darauf ab, die Auswirkungen vorhergesagter Mutationen auf der Grundlage der Auswirkungen epileptogener Mutationen auf hippokampale pyramidale Neuronensimulationen abzuschätzen.
Die Methodik beginnt mit der Erfassung genetischer Datendes GABA-A-Rezeptors γ2-Untereinheiten, gefolgt von der Datenbereinigung und -formatierung, die in R mit einem benutzerdefinierten Skript durchgeführt wird. Als nächstes werden Ensemble-Prädiktoren angewendet, um die pathogenen Missense-Varianten der γ2-Untereinheit zu identifizieren und zu priorisieren. Es wird die Kartierung einer spezifischen pathogenen Variante (vorhergesagt) auf die strukturellen Domänen der Untereinheiten veranschaulicht, die von epileptogenen Mutationen geteilt werden, begleitet von der molekularen Modellierung ihrer Auswirkungen und der Berücksichtigung der evolutionären Konservierung. Anschließend wird eine variantenspezifische Metaanalyse und Parameternormalisierung durchgeführt, gefolgt von einer Korrelationsanalyse, um signifikante Beziehungen zwischen vorhergesagten Mutationen und proximalen epileptogenen Mutationen zu identifizieren. Unter Verwendung eines Python-basierten neuronalen Simulators wird ein multikompartimentelles Leitfähigkeitsmodell beschrieben, das die Wirkung von Wildtyp- und epileptogenen Mutanten widerspiegelt. Die Simulation neuronaler Reaktionen, die durch den epileptogenen GABA-A-Rezeptor-Subtyp erzeugt werden, wird für die grobe Abschätzung der prognostizierten Wirkung pathogener Varianten auf die neuronale Reaktion berücksichtigt. Unseres Wissens ist dies das erste Protokoll, das ein Multiskalen-Framework zur Abschätzung der Auswirkungen von GABA-A-Rezeptorvarianten auf das neuronale Verhalten untersucht, das für die Epilepsieforschung von entscheidender Bedeutung ist. Dieses Protokoll kann als Grundlage für die Verbesserung der Vorhersage von zellulären Phänotypen dienen, die durch potenziell pathogene Varianten von GABA-A-Rezeptoren verursacht werden, die mit Epilepsie assoziiert sind.
Einleitung
Bei fast allen menschlichen Krankheiten spielt die genetische Variation eine bedeutende Rolle für die individuelle Anfälligkeit. Daher bietet das Verständnis, wie Sequenzvariationen mit dem Krankheitsrisiko zusammenhängen, eine wertvolle Möglichkeit, Schlüsselprozesse bei der Krankheitsentstehung aufzudecken und neue Ansätze für Prävention und Behandlung zu identifizieren1. Dies gilt auch für neurologische Entwicklungsstörungen, die zu den häufigsten chronischen Erkrankungen in der pädiatrischen Primärversorgung gehören2. Erkrankungen wie Autismus-Spektrum-Störung, geistige Behinderung und Epilepsie veranschaulichen, wie genetische Variation die individuelle Anfälligkeit während der Entwicklung signifikant beeinflusst3.
Das sich entwickelnde Gehirn ist anfälliger für epileptische Anfälle als das erwachsene Gehirn, was auf eine genetisch programmierte neurologische Entwicklungsdiskrepanz im kritischen Gleichgewicht zwischen Erregung und Hemmungzurückzuführen ist 4. Da GABA (Gamma-Aminobuttersäure), der primäre hemmende Neurotransmitter im erwachsenen Gehirn, während der embryonalen und frühen postnatalen Entwicklung exzitatorisch ist, ist dies nicht günstig für die Stabilität, die zur Vorbeugung von Anfällen in jungen Gehirnen erforderlich ist. Dieser vorübergehende Zustand, der durch den Mangel an ausreichender Expression von K-Cl-Co-Transportern5 verursacht wird, kann zu einem erhöhten Risiko für Anfallsaktivität in Gegenwart dysfunktionaler GABA-A-Rezeptoren beitragen. GABA-A-Rezeptoren vermitteln exzitatorische und inhibitorische Wirkungen von GABA, abhängig von der intrazellulären Konzentration des Cl-Ions 6. Wenn das Gehirn reift, verzerren Mutationen in den GABA-A-Rezeptor-kodierenden Genen sowie in anderen Ionenkanälen die Erregbarkeit, und Mutationen in Genen, die am neuronalen Stoffwechsel, der Zellsignalgebung und der Synapsenbildung beteiligt sind7, können Erkrankungen wie Absence-Epilepsie im Kindesalter verursachen8.
Klinische Interventionen nutzen zunehmend genetische Analysen, um die Präzision bei der Behandlung von neurologischen Entwicklungsstörungen zu verbessern2. Gentests bei pädiatrischer Epilepsie stellen potenzielle Ziele für Ansätze der Präzisionsmedizindar 9 und unterstreichen die Bedeutung genetischer Varianten für Behandlungsentscheidungen. Darüber hinaus erhalten ~25 % der Epilepsiepatienten mit De-novo-Mutationen genetische Diagnosen, die potenzielle Ziele für die Präzisionsmedizin identifizieren, was den erheblichen Wert genetischer Varianten bei der Orientierung von Behandlungsentscheidungen unterstreicht10. Dies wurde durch Fortschritte bei Sequenzierungstechnologien der nächsten Generation vorangetrieben, wie z. B. gezielte Gen-Panels, die Sequenzierung des gesamten Exoms und die Sequenzierung des gesamten Genoms, die die genetischen Entdeckungen dramatisch beschleunigt haben11. Die zunehmende Zahl neuer Genentdeckungen bringt jedoch eine Herausforderung mit sich, wenn die Ergebnisse eine Variante von unbekannter Signifikanz (VUS) ergeben, eine Klassifikation, die widersprüchliche Beweise oder unzureichende Informationen über die molekulare Rolle der Variante bei der Pathogenese der Krankheit widerspiegelt. Varianten, die als VUS klassifiziert werden, entsprechen einer Kategorie innerhalb des fünfstufigen Variantenklassifikationssystems, das vom American College of Medical Genetics and Genomics (ACMG) und der Association for Molecular Pathology (AMP)12 vorgeschlagen wurde.
Um die Herausforderung funktionell unbekannter genetischer Varianten anzugehen, sind Anstrengungen in zwei Schlüsseldimensionen erforderlich: klinische Praxis und Forschung. Klinisch kann die Unsicherheit im Zusammenhang mit VUS das Patientenmanagement und die Entscheidungsfindung erschweren13. Aus Sicht der wissenschaftlichen Forschung ist es von entscheidender Bedeutung, pathogene Varianten in der zunehmenden Anzahl von Varianten mit ungewisser Signifikanz zu identifizieren und ihre Rolle in der Pathophysiologie der Krankheit und bei phänotypischen Effekten zu bestimmen1. Ein ideales Szenario wäre die genaue Vorhersage der molekularen, neuronalen und netzwerkbasierten Effekte aller funktionell nicht charakterisierten Varianten, um so die Ressourcen, den Zeit- und Arbeitsaufwand für laborbasierte Untersuchungen zu minimieren. Diese Aspekte unterstreichen die Bedeutung einer genauen Klassifizierung genetischer Varianten, um eine präzise Diagnose genetischer Epilepsien zu ermöglichen, eine personalisierte Behandlung zu unterstützen und die Entdeckung potenzieller pharmakologischer Ziele zu erleichtern. Die derzeitigen Vorhersagewerkzeuge 14,15,16,17 sind relativ genau, liefern aber in der Regel nur binäre Klassifikationen (pathogen vs. gutartig) und es fehlen krankheitsspezifische Einblicke in die molekulare Pathophysiologie, phänotypische Konsequenzen und zugrunde liegende Mechanismen. Dieser Artikel konzentriert sich auf die unbekannten Missense-Varianten ausgewählter GABA-A-Rezeptor-Untereinheiten-kodierender Gene und stellt einen Rahmen vor, der darauf abzielt, die Forschungsberatung zu verbessern, indem kontextuelle Faktoren von Varianten wie molekulare, evolutionäre und strukturelle Aspekte sowie Simulationen der neuronalen Pathologie einbezogen werden, die aus in vitro biophysikalischen Daten von Epilepsie-assoziierten Mutationen abgeleitet wurden. Unsere Methodik befasst sich mit der Identifizierung unbekannter pathogener Varianten der γ2-Untereinheit des GABA-A-Rezeptors, einer Schlüsseluntereinheit, die an der Pathophysiologie der Epilepsie beteiligt ist 18,19,20. Es folgt die Untersuchung des positionsspezifischen Matchings dieser vorhergesagten Varianten mit den epilepsieassoziierten Mutationen, die durch strukturelle und elektrophysiologische Daten charakterisiert sind. Diese Daten werden dann verwendet, um den Effekt der Variante auf ein Modell eines hippokampalen Pyramidenneurons abzuschätzen, das einen GABA-A-Rezeptor-Subtyp exprimiert, der aus γ2-, α1- und β3-Untereinheiten (γ2-GABA-A-Rezeptoren) besteht, die für die schnelle synaptische Hemmung verantwortlich sind6. Es ist wichtig zu beachten, dass sich GABA-A-Rezeptoren aus einem großen Pool von Untereinheiten (α1-α6, β1-β3, γ1-γ3, δ, Ε, θ, π und ρ1-ρ3) zusammensetzen und sich je nach Zusammensetzung der Untereinheiten die GABA-A-Rezeptoren in ihrer Modulation, ihren biophysikalischen Eigenschaften sowie in regionalen, zellulären und subzellulären Expressionsmustern unterscheiden, die mit spezifischen Funktionen gekoppelt sind 6,21,22,23, 24,25. Daher konzentriert sich die vorliegende Studie ausschließlich auf die γ2-GABAA-Rezeptoren bzw. γ2-haltigen GABAA Rezeptoren.
GABA-A-Rezeptor-Untereinheiten bestehen aus charakteristischen strukturellen Merkmalen: einer langen N-terminalen extrazellulären Domäne (ECD), vier Transmembran-übergreifenden Domänen (TM1 bis TM4), einem intrazellulären Linker, der TM1 und TM2 verbindet, einem extrazellulären Linker, der TM2 und TM3 verbindet, einer großen intrazellulären Schleife zwischen TM3 und TM4 (TM3-TM4-Schleife) und einem kurzen extrazellulären C-Terminus 6,26, 27. Urheberrecht Es wird vermutet, dass der GABA-A-Rezeptor über einen komplexen "Lock-and-Pull"-Mechanismus funktioniert, bei dem die GABA-Bindung die β und α Untereinheiten sperrt, wodurch sie an den extrazellulären Domänen (ECDs) der Untereinheiten ziehen und sie gegen den Uhrzeigersinn drehen27. Durch diese Bewegung werden die Transmembrandomänen (TMDs) gekrümmt, wodurch der Ionenkanal27 geöffnet wird. Die Kanalaktivität scheint also zusammen mit strukturellen Kassetten innerhalb der GABA-A-Rezeptoren koordiniert zu sein. Es stellt sich heraus, dass Epilepsiemutationen durch Verzerrung dieser strukturellen Kassetten eine Dysfunktion der Kanalaktivität verursachen28. Folglich basiert unsere Studie auf der Idee, dass vorhergesagte pathogene Varianten in der Nähe von funktionell identifizierten epileptogenen Mutationen in den spezifischen Strukturkassetten der GABA-A-Rezeptor-Untereinheiten ähnliche Muster elektrophysiologischer oder biophysikalischer Verzerrungen in der Kanalfunktion aufweisen können, wie sie bei diesen epileptogenen Mutationen beobachtet wurden. Während das Vorhandensein epileptogener Strukturkassetten in den GABA-A-Rezeptor-Untereinheiten28 diese Annahme indirekt unterstützt, zeigt unsere Studie die Komplexität und Herausforderung der Korrelation biophysikalischer Parameter epileptogener Mutationen mit denen der vorhergesagten pathogenen Mutationen. Um diese komplexen Zusammenhänge zu entlarven, ist unser Rahmenwerk von Bedeutung, da es einen mehrskaligen Ansatz hervorhebt, der von der DNA über die Proteinfunktion bis hin zum neuronalen Verhalten reicht und für die Epilepsieforschung entscheidend ist. Dieser Ansatz integriert computergestützte Genetik mit molekularer Modellierung und neuronalen Simulationen und betont gleichzeitig die Bedeutung komplementärer Methoden, wie z. B. maschinelles Lernen, das auf großen Datensätzen trainiert wird und die Auswirkungen von Mutationen auf die Kanalstruktur, die Aktivität und die neuronale Erregbarkeit erfassen können. Darüber hinaus ermöglicht die Simulation der epileptogenen γ2-GABA-A-Rezeptoraktivität im hippokampalen pyramidalen Neuronenmodell die Replikation des in vitro zellulären Phänotyps, der mit der GABA-A-Rezeptorkanalopathie assoziiert ist, und den Nachweis veränderter Einzelneuronenantworten im Zentrum der Netzwerkdysfunktion.
Protokoll
1. In-silico-Vorhersage pathogener Varianten
- Erhebung von Variantendaten
- Suchen Sie mit der ClinVar-Datenbank29 über die Website nach Varianten mit ungewisser Signifikanz (VUS) in der kodierenden Region des interessierenden Gens über die Website: https://www.ncbi.nlm.nih.gov/clinvar/. Geben Sie das Gensymbol (z. B. GABRG2) in die Suchleiste ein und filtern Sie die Ergebnisse so, dass nur die gewünschten Variantentypen enthalten sind, z. B. Einzelnukleotid, Missense-Varianten mit ungewisser Signifikanz. Laden Sie die Daten herunter und speichern Sie sie als data.xlxs (Ergänzungsdatei 4: Ergänzungstabelle S1). Notieren Sie sich das Datum der heruntergeladenen Daten.
ANMERKUNG: Im vorliegenden Protokoll wird die humane γ2-Untereinheit des GABA-A-Rezeptors, insbesondere die Homo sapiens gamma-Aminobuttersäure Typ A-Rezeptor-Untereinheit gamma2 (GABRG2), Transkriptvariante 1, mRNA (NCBI Ref. seq.: NM_198904.4), auch bekannt als γ2L, analysiert. Es ist wichtig, das Referenztranskript des interessierenden Gens sowie andere entsprechende Identifikatoren in verschiedenen Datenbanken (UniProt, ENSEMBL, PDB) aufzuzeichnen, da unterschiedliche Berechnungsmethoden unterschiedliche Identifikatoren erfordern können (Ergänzungsdatei 4: Ergänzende Tabelle S2). Falls die Datenbank oder das Berechnungstool die Versionsnummern der Sequenzkennungen nicht erkennt, versuchen Sie es sowohl mit der ID mit der Versionsnummer (NM_198904.4) als auch ohne die Versionsnummer (NM_198904). - Grundlegende Informationen zum Referenzprotein
- Wählen Sie in der NCBI-Datenbank https://www.ncbi.nlm.nih.gov/ in den Suchoptionen Nukleotid aus und geben Sie die NCBI Ref. seq. ID des interessierenden Gens (NM_198904.4) ein. Wenn Sie dann in der rechten Spalte nach unten scrollen, klicken Sie auf das Protein unter der Kategorie Verwandte Informationen , um das Protein (NP_944494.1) zu finden, das vom Transkript NM_198904.4 kodiert wird. Unter Verwendung der für das Protein NP_944494.1 angegebenen Informationen werden die Sequenzpositionen der spezifischen Regionen in Form einer Tabelle festgehalten (Ergänzungsdatei 4: Ergänzungstabelle S3).
HINWEIS: Es ist wichtig, die vorläufigen bekannten Informationen für die Sequenzposition von funktionell und strukturell kritischen Regionen, Motiven oder Resten wie Proteindomänen, Phosphorylierungsstellen, Ligandenbindungsstellen und molekularen Interaktionsgrenzflächen zu bestimmen. Dies kann durch die Kombination von Datenbank- (NCBI, ENSEMBL, UniProt...) und Literaturrecherchen erreicht werden.
- Wählen Sie in der NCBI-Datenbank https://www.ncbi.nlm.nih.gov/ in den Suchoptionen Nukleotid aus und geben Sie die NCBI Ref. seq. ID des interessierenden Gens (NM_198904.4) ein. Wenn Sie dann in der rechten Spalte nach unten scrollen, klicken Sie auf das Protein unter der Kategorie Verwandte Informationen , um das Protein (NP_944494.1) zu finden, das vom Transkript NM_198904.4 kodiert wird. Unter Verwendung der für das Protein NP_944494.1 angegebenen Informationen werden die Sequenzpositionen der spezifischen Regionen in Form einer Tabelle festgehalten (Ergänzungsdatei 4: Ergänzungstabelle S3).
- Suchen Sie mit der ClinVar-Datenbank29 über die Website nach Varianten mit ungewisser Signifikanz (VUS) in der kodierenden Region des interessierenden Gens über die Website: https://www.ncbi.nlm.nih.gov/clinvar/. Geben Sie das Gensymbol (z. B. GABRG2) in die Suchleiste ein und filtern Sie die Ergebnisse so, dass nur die gewünschten Variantentypen enthalten sind, z. B. Einzelnukleotid, Missense-Varianten mit ungewisser Signifikanz. Laden Sie die Daten herunter und speichern Sie sie als data.xlxs (Ergänzungsdatei 4: Ergänzungstabelle S1). Notieren Sie sich das Datum der heruntergeladenen Daten.
- Organisation von Variantendaten
- Organisieren Sie die Daten so, dass sie die Eingabeanforderungen für die ausgewählten Prädiktoren erfüllen. Stellen Sie sicher, dass das Format der abgerufenen Daten so organisiert ist, dass es den Anforderungen des dbNSFP-Servers http://database.liulab.science/dbNSFP entspricht. Entfernen Sie dazu nicht benötigte Spalten aus der data.xlsx Datei (Zusatzdatei 4: Zusatztabelle S1 aus Schritt 1.1.1) und behalten Sie nur die folgenden Spalten in der angegebenen Reihenfolge bei:
"GRCh38Chromosom", "GRCh38Location", "Name", "Proteinveränderung". - Speichern Sie die Datei unter dem neuen Dateinamen: "data1.xlsx" (Zusatztabelle S4). Formatieren Sie die data1.xlsx Datei in R, indem Sie den Code ausführen (Ergänzende Datei 1: Data_GABAA. R), der die formatierten Daten als data1_output.xlsx (Ergänzungsdatei 4: Ergänzungstabelle S5) in dem für das R-Projekt relevanten Arbeitsverzeichnis speichert.
HINWEIS: Unterschiedliche Berechnungsmethoden erfordern unterschiedliche Datentypen und Formate. Das Sammeln und Organisieren von Daten nach bestimmten Formatanforderungen, selbst für ein Dutzend Varianten, kann fehleranfällig und zeitaufwändig sein, daher ist dieser Schritt wichtig, es sei denn, der Variantenpool besteht nur aus wenigen Varianten. Dann ist möglicherweise eine manuelle Datenorganisation möglich.
- Organisieren Sie die Daten so, dass sie die Eingabeanforderungen für die ausgewählten Prädiktoren erfüllen. Stellen Sie sicher, dass das Format der abgerufenen Daten so organisiert ist, dass es den Anforderungen des dbNSFP-Servers http://database.liulab.science/dbNSFP entspricht. Entfernen Sie dazu nicht benötigte Spalten aus der data.xlsx Datei (Zusatzdatei 4: Zusatztabelle S1 aus Schritt 1.1.1) und behalten Sie nur die folgenden Spalten in der angegebenen Reihenfolge bei:
- Vorhersage der Pathogenität
- Übertragen Sie den Inhalt der data1_output.xlsx Datei in die akademische Version des dbNSFP-Servers30,31, auf die über http://database.liulab.science/dbNSFP zugegriffen wird. Kopieren Sie dazu die Datei oder laden Sie sie direkt in .txt Format hoch.
- Stellen Sie sicher, dass die folgenden Optionen vor der Übermittlung vorausgewählt und auf dem Server bestätigt werden: HG38 (Genom-Build), ClinPred32 und BayesDEL33 . Innerhalb weniger Minuten generiert der Server die Ergebnisse.
ANMERKUNG: In dem vorliegenden Protokoll wurden zwei Ensemble-Prädiktoren, nämlich BayesDEL33 und ClinPred32, für eine hohe Genauigkeit34 und Praktikabilität ausgewählt. Es können aber auch andere Prädiktoren, wie z.B. AlphaMissense, das in der dbNSFP-Datenbank30,31 verfügbar ist, ausgewählt werden. Die Auswahl von In-silico-Werkzeugen hängt von mehreren Faktoren ab, einschließlich der Generierung ausreichender mehrerer Zeilen rechnerischer Beweise für eine aussagekräftige Vorhersage12. Ensemble-Prädiktoren, die die Analyse mehrerer Vorhersagealgorithmen integrieren, können diesem Zweck dienen. - Laden Sie die Ausgabedatei (in einem .txt Format) herunter und speichern Sie sie als data2.xlsx (Ergänzungsdatei 4: Ergänzungstabelle S6).
- Setzen Sie die Filter in data2.xlsx (Ergänzungsdatei 4: Ergänzungstabelle S6), indem Sie auf die Filteroption im Menü klicken und ermitteln Sie die Konsensusvarianten in beiden Spalten durch Filtern nach D. Daraufhin wird die Liste der pathogensten Varianten angezeigt. Speichern Sie es (siehe Registerkarte Konsens in der Ergänzungstabelle S6 [ Ergänzungsdatei 4]).
- Variantenauswahl
- Unter den konsensusartigen pathogenen Vorhersagen bestimmen Sie die Varianten in der Nähe von epileptogenen Mutationen, die aus der Literatur gewonnen wurden. Stellen Sie sicher, dass letztere strukturelle und biophysikalische Parameter aufweisen, die für die Modellierung von Neuronen geeignet sind.
HINWEIS: Dieser Schritt ist explorativ und bezieht sich auch auf die Untersuchung des interessierenden Proteins in Bezug auf seine strukturellen, physikalisch-chemischen und biophysikalischen Parameter. In der vorliegenden Studie wurden diese Daten von Brünger et al.35 und Guo et al.36 zusätzlich zu einer Erhebung von Epilepsie-assoziierten Mutationen gewonnen. Zusätzlich wurde optional auf AlphaMissense37 Scores aus der dbNSFP-Datenbank 30,31 zugegriffen, die Schritt 1.3 wiederholt (Ergänzungsdatei 4: Ergänzungstabelle S7). Näheres dazu finden Sie in den Protokollabschnitten 2.1.1 und 2.1.2 sowie in den Ergebnissen (siehe "Clustering-Varianten für strukturelle und biophysikalische Parameter"). - Verwenden Sie für eine grundlegende Visualisierung die Server Protter38 ((https://wlab.ethz.ch/protter/start/) und HOPE39 (https://www3.cmbi.umcn.nl/hope/), um die Varianten im vorherigen Schritt im Kontext ausgewählter GABRG2-Genmutationen zu untersuchen: P302L40 und K328M (oder K289M41, wenn das Signalpeptid mit 39 Resten ausgeschlossen wird).
HINWEIS: Aufgrund der enormen Komplexität sollte die strukturelle Bewertung von Varianteneffekten auf mehreren Analyseebenen erfolgen. Werkzeuge wie Protter38 werden die klare Visualisierung der Varianten im Kontext topologischer Merkmale des Proteins ermöglichen und benutzerfreundliche Server wie HOPE39 werden durch molekulare Modellierung Einblick in den Varianteneffekt geben. Darüber hinaus ist eine umfassende Literaturrecherche des interessierenden Proteins von entscheidender Bedeutung, um die Informationen über Mutationen, die mit Epilepsie assoziiert sind, zu identifizieren und zu integrieren. - Analyse von evolutionären Erhaltungs- und Strukturerkenntnissen
- Öffnen Sie Jalview 42,43,44, ein Open-Source-Programm zum Bearbeiten, Visualisieren und Analysieren von Proteinen.
- Importieren Sie Sequenzen für die Ausrichtung. Klicken Sie im oberen Menü auf Datei | Abrufen von Sequenzen; Wählen Sie die Datenbank im Dialogfeld aus (z. B. UniProt); Klicken Sie auf die Registerkarte IDs abrufen. und geben Sie, wie im Dialogfeld beschrieben, die UniProt-Zugangs-IDs des interessierenden Gens (GABRG2) von Menschen und anderen Wirbeltierarten ein: P18507, P22723, Q6PW52, A0A2I3TKX0, F1RR72, A0A8I3MDZ2 A0A8M1P4D6. Klicken Sie auf OK.
HINWEIS: Die UniProt-Zugangsnummern der von GABRG2 kodierten Proteine lauten wie folgt: P18507 (P18507-2) für Homo sapiens, P22723 für Mus musculus, A0A2I3TKX0 für Pan troglodytes, F1RR72 für Sus scrofa, A0A8I3MDZ2 für Canis familiaris und A0A8M1P4D6 für Danio rerio. - Abhängig vom interessierenden Gen können einige Sequenzen nicht annotiert werden. Führen Sie daher eine BLAST-Suche durch, um relevante Informationen und potenzielle Homologe für ein besseres kontextuelles Verständnis zu identifizieren. Laden Sie in diesem Fall das FASTA-Format von Proteinsequenzen über die Textfeldoption Sequenzen/Von hinzufügen im Menü Datei hoch, um mehrere Sequenzalignments der gewünschten Sequenzen zu erstellen.
- Nachdem die Achse geladen ist, beobachten Sie die angezeigten Sequenzen für den Vergleich mehrerer Sequenzen. Jede Zeile stellt eine Sequenz dar, und jede Spalte stellt eine Position in der Ausrichtung dar. Um die beste Ausrichtungsmethode zu bestimmen, verwenden Sie verschiedene Ansätze. Klicken Sie beispielsweise im Menü Sequenz auf die Webdienste und wählen Sie die Option T-Coffee mit Voreinstellung ausführen, die eine optimale Ausrichtung ermöglicht.
- Klicken Sie mit der rechten Maustaste auf die Sequenz P18507 Homo sapiens (die Referenzsequenz in der vorliegenden Studie) und legen Sie sie als Referenzsequenz fest. Wählen Sie im oberen Menü Format und klicken Sie auf Wrap , um die vollständige Ausrichtung im Bildschirm zu visualisieren. Klicken Sie im selben Formatmenü auf die obige Skala , um die Visualisierung bestimmter Restnummern zu verbessern. Um die Visualisierung weiter zu verbessern, passen Sie die Farbschemata an, indem Sie zu Farbe gehen und verschiedene Optionen auswählen (z. B. Farbfarbe, Chemische Eigenschaft); Ändern Sie bei Bedarf die Schriftgröße.
- Klicken Sie in der Menüleiste auf Berechnen und wählen Sie Konsens automatisch berechnen , um konservierte Regionen hervorzuheben.
- Konzentrieren Sie sich auf die Position der im In-silico-Vorhersageschritt identifizierten Varianten von Interesse und untersuchen Sie spezifische Variantenpositionen. Beschriften Sie bestimmte Rückstände, indem Sie mit der rechten Maustaste darauf klicken und Anmerkung hinzufügen auswählen. Schreiben Sie das Etikett (z.B. Varianten-ID) mit dem entsprechenden Farbcode und speichern Sie.
HINWEIS: In der vorliegenden Analyse wurden P302L (violett) und A303T (rot) ausgewählt, um sie in der Mehrfachsequenzausrichtung zusammen mit den Strukturdaten zu visualisieren (siehe nächster Abschnitt).
- Dreidimensionale Rekonstruktion des Vollproteins mit den ausgewählten konservierten Resten
- Klicken Sie in der Datei aus dem vorherigen Schritt mit der rechten Maustaste auf die Referenzsequenz (GABRG2 human) und wählen Sie 3D-Strukturdaten.
- Identifizieren Sie die entsprechenden Strukturdaten (7QNE, Kette C)26 aus dem Dropdown-Menü und wählen Sie Neue Strukturansicht mit Jmol öffnen.
HINWEIS: Dies ermöglicht die Einbeziehung der Reste, die in der Mehrfachsequenzausrichtung ausgewählt wurden, in die Strukturdaten durch Jmol, einen Open-Source-Java-basierten Viewer für chemische 3D-Strukturen.
- Unter den konsensusartigen pathogenen Vorhersagen bestimmen Sie die Varianten in der Nähe von epileptogenen Mutationen, die aus der Literatur gewonnen wurden. Stellen Sie sicher, dass letztere strukturelle und biophysikalische Parameter aufweisen, die für die Modellierung von Neuronen geeignet sind.
2. Parameterauswahl und biophysikalische Modellierung
- Variantenspezifische Metaanalyse und Parameternormalisierung
- Überblick über die aktuelle Literatur, um identifizierte Untereinheitenvarianten mit elektrophysiologischer Datenkanalleitfähigkeit (gGABAA), Deaktivierungszeit (τ Deaktivierung), Anstiegszeit (τ Anstieg) und maximaler Stromamplitude (Imax) zu sammeln. Geben Sie die Zusammensetzung der Untereinheiten, den Zelltyp und die Wildtyp-Messungen für jeden Fall an. Kennzeichnen Sie die Varianten und ihre Kontrollen entsprechend (z. B. bekannt für Varianten mit identifizierten biophysikalischen Merkmalen und bekannte Kontrolle für die Wildtyp-Messungen für jede Variante).
- Erhalten Sie AlphaMissense-Pathogenitätsbewertungen für Varianten mit identifizierten biophysikalischen Merkmalen.
HINWEIS: Weitere Informationen finden Sie im Protokollabschnitt 1.3. - Erstellen Sie einen Datenrahmen mit der Position der Untereinheit und der Aminosäure für jede Variante, den ursprünglichen und veränderten Aminosäuren, dem Pathogenitätswert und den biophysikalischen Parametern aus der Literatur. Um experimentelle Diskrepanzen zu vermeiden, normalisieren Sie die biophysikalischen Parameter für identifizierte Varianten als x-fache Veränderungen bei Wildtyp-Messungen.
- Vergleichende Variantenanalyse nach strukturellen und funktionalen Merkmalen
- Organisieren der vorhergesagten Varianten in einem Datenrahmen; entsprechend gekennzeichnet (z. B. für Varianten vorhergesagt, für die keine Literatur zu ihren biophysikalischen Eigenschaften verfügbar ist).
- Klassifizieren Sie die Varianten nach ihrer Position in der Aminosäuresequenz und der Tertiärstruktur. Fügen Sie dem Datenrahmen strukturelle Klassifikationsparameter hinzu (z. B. Lokalisation in Alpha-Helices, Spulen, Beta-Faltblättern, extrazellulären, intrazellulären oder Transmembrandomänen, Porenauskleidung, Agonistenbindung, Protein-Protein-Interaktionen) und geben Sie Informationen für jede Variante in Bezug auf ihre Aminosäureposition an.
- Klassifizieren Sie die Varianten nach ihrem Abstand zum Membranmittelpunkt und zur Porenachse. Fügen Sie den Abstand zur Porenachse und den Abstand zu den Parametern für den Membranmittelpunkt im Datenrahmen hinzu.
- Analysieren Sie die Korrelation zwischen strukturellen und biophysikalischen Parametern über bekannte Varianten. Wenn möglich, bewerten Sie die vorhergesagten Varianten in Bezug auf die erhaltenen Korrelationen.
- Aufbau von Synapsen- und Neuronenmodellen
- Verwenden Sie den Brian245, einen in Python entwickelten Open-Source-Neurosimulator zur Modellierung und Simulation von Spiking Neural Networks, um ein multikompartimentelles biophysikalisches Modell der GABAergen Synapse auf einem auf Multikompartimentellen Leitfähigkeit basierenden Hippocampus-Pyramidenneuron zu erstellen.
- Entwerfen Sie das leitfähigkeitsbasierte Modell, indem Sie die Kinetik des Ionenkanal-Gatings, passive und aktive Parameter sowie die postsynaptische Leitfähigkeit definieren. Definieren Sie das leitwertbasierte Modell wie in der Zusatzdatei 2 angegeben, in der die im Modell verwendeten Gleichungen beschrieben werden.
- Die Membrankapazität (Cm) auf 1 μF/cm2 und den intrazellulären Widerstand (Ra) auf 200 Ω cm einstellen.
- Verwenden Sie die modifizierten Leitwerte vom Hodgkin-Huxley-Typ für hippokampale Pyramidenneuronen39 mit gL = 0,0003 S/cm2, gK = 0,036 S/cm2, EL = -76,5 mV, ENa = 50 mV und EK = -90 mV.
- Passen Sie die Dichteverteilung der NaV-Kanäle über gNa auf 0,05 S/cm2 für Soma, 0,5 S/cm2 für das Axon-Anfangssegment (AIS) und den Knoten von Ranvier (NR) und 0,005 S/cm2 für Dendriten an. Legen Sie gK und gNa in myelinisierten Segmenten auf 0 fest.
- Aufbau einer Ionenkanal-Gating-Kinetik für NaV und KV , wie in der Zusatzdatei 2 beschrieben.
- Führen Sie synaptische Ströme (Isyn) als Summe aller glutamatergen und GABAergen Synapsen in einem Kompartiment ein. Beziehen Sie sowohl den schnellen AMPA-Rezeptor-vermittelten Strom (IAMPA) als auch den langsamen NMDA-Rezeptor-vermittelten Strom (INMDA) in den glutamatergen Strom (Iglu) ein. Beziehen Sie nur schnellen GABA-A-Rezeptor-vermittelten Strom in den GABAergen Strom (IGABA) ein. Nehmen wir an, dass für jeden präsynaptischen Spike eine konstante Menge an Glutamat an die Synapse abgegeben wird; Daher ist die Aktivierung der Rezeptoren spike-zeitabhängig (sAMPA und sNMDA) und die Gesamtrezeptorleitwerte (gAMPA und gNMDA) spiegeln die Menge an Glutamat wider, die bei jedem Ereignis freigesetzt wird.
- Verwenden Sie das synaptische Modell, wie in der Zusatzdatei 2 beschrieben.
HINWEIS: Eine detaillierte Erläuterung der Gleichungen finden Sie in der Ergänzungsdatei 2 , in der die im Modell verwendeten Gleichungen beschrieben werden.
- Ermitteln Sie den experimentell gemessenen Durchmesser für Soma und Neuriten sowie die Länge jedes Neuritenkompartiments und der Verzweigungsmuster aus der früheren Literatur46,47. Reduzieren Sie die reale Neuronenmorphologie in ein Modell mit mehreren Kompartimenten, indem Sie die Zelle in mehrere Kompartimente aufteilen, das die Hauptverzweigungsstruktur genau beibehält und die bilaterale Symmetrie beibehält.
- Stellen Sie die Morphologie (Segmentlänge und -durchmesser; d. h. d_soma: 30 μm; l_AH: 5 μm; d_AH_i: 1,5 μm; d_AH_f: 1,3 μm; l_AIS: 40 μm; d_axon: 1 μm; l_myseg: 100 μm; l_NR: 2 μm; l_AxTer: 4 μm; d_AxTer: 2 μm; l_approx: 100 μm; l_apmed: 100 μm; l_apdis: 200 μm; d_approx_i: 4 μm; d_approx_f: 3 μm; d_apmed : 2 μm; d_apdis: 2 μm; l_apLM: 70 μm; d_apLM: 2 μm; l_nAcDbasal: 400 μm; d_nAcDbasal: 1,4 μm; l_nAcDbasal_stem: 20 μm; d_nAcDbasal_stem: 1,5 μm) und biophysikalische Parameter (wie in Abschnitt 2.3.2 angegeben) für jedes Kompartiment des pyramidalen Neuronenmodells46,47, wie auch im Python-Skript (Ergänzende Datei 3: GABAAvar.py) beschrieben.
- Bestimmen Sie die biophysikalischen Parameter für das GABAerge Synapsenmodell, indem Sie die in Schritt 2.1.1 erhaltenen Wildtyp-Kontrollmessungen auswerten.
- Entwerfen Sie die Topologie des Neuronenmodells und weisen Sie morphologische und biophysikalische Parameter zu, was die Spezifizierung der räumlichen Anordnung und der Verbindungen der Kompartimente auf der Grundlage der zuvor erhaltenen morphologischen und Verzweigungsinformationen umfasst. Weisen Sie jedem Kompartiment des Modells die entsprechenden morphologischen (z. B. Segmentlänge und -durchmesser) und biophysikalischen Parameter (Abschnitt 2.3.2) zu, wie in der Zusatzdatei 3: GABAAvar.py beschrieben.
- Aufbau von Synapsen und Strominjektion
- Erstellen Sie die präsynaptische Aktivität mit SpikeGeneratorGroup (einer Klasse aus der Brian2-Bibliothek), wie in "GABAAvar.py" (Ergänzende Datei 3) angegeben. Verbinden Sie den Spike-Generator mit dem Zielkompartiment des Modellneurons mithilfe der Synapsen-Klasse , um synaptische Verbindungen zu modellieren.
- Stellen Sie einen konstanten Dauerstrom (Iinj) auf 0,85 nA ein und platzieren Sie ihn am Soma, um die unterschwellige Aktivität nachzuahmen, die durch die Ionenstrombelastung zu einem bestimmten Zeitpunkt verursacht wird, wie in der Zusatzdatei 3: GABAAvar.py beschrieben.
- Um Aufzeichnungsmonitore zu erstellen, zeichnen Sie Spannungsspuren aus Zielabteilungen mit StateMonitor auf.
- Erstellen und betreiben Sie das Netzwerk.
- Erstellen Sie das Netzwerk mit dem Modellneuron, den Verbindungen und Monitoren mithilfe des Netzwerks.
- Legen Sie den Zeitschritt der Simulation mit defaultclock.dt fest (z. B. 0,01 ms).
- Führen Sie die Simulation im Netzwerk mit network.run(T*ms) aus, wobei T im Beispiel auf 1.000 ms festgelegt ist.
- Testen der Auswirkungen von GABA-A-Rezeptor-Missense-Mutationen
- Definieren Sie den Einfluss jeder Missense-Mutation auf die Kanalkinetik anhand der in Schritt 2.1.1 gesammelten biophysikalischen Parameter.
- Führen Sie die Stimulation aus, indem Sie diese Parameter ändern, und stellen Sie die Ergebnisse mit "matplotlib.pyplot" dar, wie in "GABAAvar.py" (Ergänzende Datei 3) angegeben.
- Testen Sie Parameterkombinationen, um die Änderungen der Zündmuster und -raten zu analysieren. Plotten Sie die Ergebnisse für Vergleiche.
Ergebnisse
Diese Studie verwendet einen multiskaligen Ansatz, um die pathogenen Varianten in der γ2-Untereinheit des GABA-A-Rezeptors, einer Schlüsselkomponente in der Pathophysiologie der Epilepsie, vorherzusagen und zu charakterisieren. Durch den Einsatz von Vorhersagemodellen, molekularer Modellierung, evolutionärer Konservierung, Strukturuntersuchung, Korrelationsanalyse und neuronalen Simulationen verbessert dieser Ansatz die Klassifizierung von Varianten, mit erheblicher Relevanz für die Epilepsieforschung und möglicherweise für den klinischen Einsatz. Die Gesamtzusammenfassung der Methodik ist in Abbildung 1 dargestellt.
Vergleichende Bewertung von zwei benachbarten γ2-Untereinheitsmutationen
Aufbauend auf unserer Annahme, dass vorhergesagte pathogene Mutationen, die an epileptogene Mutationen in GABA-A-Rezeptor-Untereinheiten angrenzen, ähnliche elektrophysiologische Effekte auf die Kanalfunktion und das neuronale Verhalten hervorrufen können, haben wir zunächst eine kurze Untersuchung der Beziehung zwischen einer bekannten epileptogenen Mutation und einer proximal vorhergesagten Mutation der γ2-Untereinheit durchgeführt.
Unter den Varianten, die als pathogen vorhergesagt werden (Ergänzende Tabelle S6), wird A303T (rs1581439874, ClinVar Accession: VCV000663033.6) als Beispiel ausgewählt. Zusätzlich zur Vorhersage durch Ensemble-Modelle wurde die Pathogenität von A303T durch AlphaMissense-Scores bestätigt (Ergänzungsdatei 4: Ergänzende Tabelle S7). A303T befindet sich in der zweiten Transmembrandomäne der GABAA-Rezeptor γ2-Untereinheit und befindet sich neben der epileptogenen Mutation P302L40, wie in Abbildung 2 gezeigt. Wie durch molekulare Modellierung festgestellt wurde, führten sowohl γ2P302L- als auch γ2A303T-Substitutionen zu Aminosäuren mit größeren Seitenketten, wie in Abbildung 3 gezeigt. Sowohl die Mutante als auch die Wildtyp-Reste sind bei der γ2P302L-Mutation unpolar, während bei der γ2A303T der mutierte Rest eine polare Seitenkette und der Wildtyp-Rest eine unpolare Seitenkette aufweist. Sowohl P302 als auch Ala303 befinden sich in der Wechselwirkungsschnittstelle der Untereinheit mit der β3-Untereinheit (beobachtet in 7QNB bzw. 7QNA). Sowohl P302 als auch Ala303 haben eine vergleichbare lösemittelzugängliche Oberfläche (SASA). Darüber hinaus sind beide Rückstände über die gesamte Spanne der Wirbeltierevolution zu 100 % konserviert (Abbildung 4, obere Tafel). Sie befinden sich beide in der Nähe der zweiten Transmembranregion (TM2-Domäne) der γ2-Untereinheit, wie in der dreidimensionalen Rekonstruktion des GABA-A-Rezeptorproteins (7QNE26) gelb dargestellt, wobei A303, rot dargestellt, der erste Rest in dieser Domäne ist (Abbildung 4). Basierend auf diesen vergleichbaren Merkmalen und unter Verwendung eines pyramidalen Neuronenmodells kann die Simulation proximaler epileptogener Mutation(en) wie der γ2-Untereinheitenmutation P302L40 zur vorläufigen Charakterisierung der Wirkung der vorhergesagten Variante (γ2A303T) auf die neuronale Reaktion verwendet werden. Im nächsten Schritt erweiterten wir unsere Analyse auf eine breitere Palette von Varianten innerhalb der GABA-A-Rezeptor-Untereinheiten.
Clustering-Varianten für strukturelle und biophysikalische Parameter
Nach der vergleichenden Bewertung zweier benachbarter Mutationen im vorherigen Abschnitt haben wir einen systematischen Ansatz implementiert, um zu beurteilen, ob gemeinsame molekulare Merkmale zwischen den Varianten identifiziert werden können. In dieser Phase sollte untersucht werden, ob sich konsistente Muster in strukturellen, physikalisch-chemischen und biophysikalischen Merkmalen über die Aminosäuren und Varianten hinweg ergeben, um unsere ursprüngliche Hypothese weiter zu unterstützen.
Der in dieser Studie verwendete Datenrahmen und die Referenzen sind in der Zusatzdatei 4 enthalten: Ergänzende Tabelle S840, 48,49,50,51,52,53,54,55,56,57,58,59,60, 61,62,63, ErgänzungstabelleS936 und ErgänzungstabelleS1035. Darüber hinaus wurden für jede Untereinheit und für alle Varianten ohne Unterscheidung der Untereinheiten Korrelationen zwischen strukturellen und biophysikalischen Parametern ermittelt (Ergänzungsdatei 4: Ergänzungstabelle S11, Ergänzungstabelle S12, Ergänzungstabelle S13, Ergänzungstabelle S14 und Ergänzungstabelle S15). Informationen zu strukturellen Parametern (Lokalisation auf alpha-Helices, Coils, Beta-Faltblättern; extrazelluläre, intrazelluläre oder Transmembrandomänen; Porenauskleidung, Agonist/allosterische Bindung und Protein-Protein-Interaktionen) wurden von Brünger et al.35 gewonnen. Biophysikalische Parameter wurden aus elektrophysiologischen Patch-Clamp-Studien an 293 Zellen der menschlichen embryonalen Niere (HEK) ermittelt. Die Werte wurden mit den jeweiligen Wildtyp-Rezeptor-Aktivierungszeit- (τr) und Deaktivierungszeitkonstanten (τd) normiert.
Da unsere Studie auf der Idee basiert, dass Aminosäurevarianten, die an oder in der Nähe von funktionell identifizierten Mutationen in GABA-A-Rezeptor-Untereinheiten liegen, ähnliche Muster elektrophysiologischer Veränderungen der Kanalfunktion aufweisen können, wie sie bei diesen Mutationen beobachtet wurden, untersuchten wir die Möglichkeit einer Beziehung zwischen strukturellen, physikalisch-chemischen und biophysikalischen Parametern. Die Positionen der Varianten in Bezug auf ihre Abstände vom Membranzentrum und zur Porenachse sind in Abbildung 5 und Abbildung 6 dargestellt. In diesem Zusammenhang haben wir auch die Scores (Ergänzungsdatei 4: Ergänzungstabelle S7) von AlphaMissense37 verwendet; angetrieben von dem hochpräzisen Proteinstrukturvorhersagemodell AlphaFold264, das die basische Aminosäuresequenz als Input verwenden kann. AlphaMissense kann Hinweise auf die strukturellen Aspekte von Einzelaminosäuresubstitutionen liefern. Die Verteilung der AlphaMissense-Scores für bekannte (schwarz) und vorhergesagte (rot) Varianten in Bezug auf die Variantenposition (Aminosäureposition, Abstand zum Membranzentrum und Abstand zur Porenachse) von GABA-A-Rezeptor-Untereinheiten (α1, β3, γ2 Untereinheiten, die jeweils von den Genen GABRA1, GABRB3 und GABRG2 kodiert werden) ist in Abbildung 7 dargestellt.
Abbildung 7A-C zeigt die AlphaMissense-Score-Verteilung über die Aminosäurepositionen, Abbildung 7D-F zeigt die AlphaMissense-Score-Verteilung über den normalisierten Abstand von der Porenachse und Abbildung 7G-I zeigt die AlphaMissense-Score-Verteilung über die Entfernung vom Membranzentrum. Die Korrelationsanalyse in Abbildung 7 zeigte, wie schwierig es ist, eine zugrunde liegende Beziehung anhand struktureller Eigenschaften zu ermitteln, um das Ergebnis für neu identifizierte Varianten vorherzusagen. Varianten der b2-Untereinheit (kodiert durch das GABRB2-Gen) wurden in die Clustering- und Korrelationsabschnitte aufgenommen, um eine breitere Analyse durchführen zu können. In den biophysikalischen Modellen wurden jedoch nur die Varianten der α1-Untereinheit, die von GABRA1 kodiert wird (Abbildung 7A,D,G), der β3-Untereinheit, die von GABRB3 kodiert wird (Abbildung 7B,E,H), und der γ2-Untereinheit, die von GABRBG2 kodiert wird (Abbildung 7C,F,I), in die biophysikalischen Modelle aufgenommen, da sich das Modell auf die Funktion eines hippokampalen Pyramidenneurons und die α1β3γ2-Kombination von GABAA konzentriert Rezeptor-Untereinheiten ist die am weitesten verbreitete Kombination im Hippocampus65. In ähnlicher Weise wurden alle Varianten von α1, β3 oder γ2, für die die Kanalkinetik in einem α1β3γ2 GABAA-Rezeptor nicht untersucht wurde, ebenfalls von den Simulationen ausgeschlossen. In der vorliegenden Analyse zeigte sich eine leichte Korrelation (Ergänzende Abbildung S1 und Ergänzende Abbildung S2) zwischen den AlphaMissense-Scores und biophysikalischen Parametern (normierte Aktivierungs- und Deaktivierungszeiten), die aus den Effekten der GABA-A-Rezeptor-Mutationen abgeleitet wurden (Ergänzende Datei 4 und Ergänzende Tabelle S8). Dies deutet darauf hin, dass Mutationen, die als pathogen vorhergesagt werden (basierend auf AlphaMissense-Scores), auch zu messbaren, potenziell störenden Veränderungen der Rezeptorkinetik (z. B. Aktivierungs- und Deaktivierungszeiten) führen können. Nichtsdestotrotz macht es die fehlende Korrelation zwischen den Positionskorrelationen in Abbildung 7 schwierig, AlphaMissense-Scores für unsere Annahme zu verwenden, die auf der Idee basiert, dass benachbarte Aminosäuren ähnliche Konsequenzen für biophysikalische Eigenschaften haben.
Die Verteilungen des normierten Abstands zur Porenachse in Bezug auf die Aktivierungs- und Deaktivierungskinetik der bekannten Varianten sind in Abbildung 8 und Abbildung 9 dargestellt. Es gibt eine milde Korrelation für die γ2-Untereinheit (Abbildung 8C), was auf die Möglichkeit hindeutet, dass unsere Hypothese, die auf der Annahme basiert, dass benachbarte Aminosäuren ähnliche Folgen haben, in einigen Regionen zutreffen könnte, insbesondere in der Nähe des Porenbereichs des Rezeptorkanals, der TM2-Domäne. Diese Region grenzt an unsere epileptogene Referenzmutation (Abbildung 2 und Abbildung 4; γ2P302) und ist daher ein relativ guter Kandidat für neuronale Simulationen. Darauf aufbauend kann eine grobe Abschätzung der Auswirkungen benachbarter vorhergesagter Mutationen wie γ2A303T (Abbildung 2 und Abbildung 4) vorgenommen werden. Unsere hier vorgestellten Ergebnisse berücksichtigen nur die Messungen an α1β3γ2; daher wurden die in unserem Modell bewerteten Varianten auf die Varianten beschränkt, die in der Ergänzungsdatei 4:Ergänzende Tabelle S16 angegeben sind.
Wirkung von Mutationen auf die GABA-A-Rezeptor-vermittelte Hemmung des Feuerns von CA1-Pyramidenneuronen
Die Wirkung von Mutationen auf die GABA-A-Rezeptor-vermittelte Hemmung wird an einem auf multikompartimentellen Leitfähigkeiten basierenden CA1-Pyramidenneuronenmodell demonstriert. Der Einfluss von GABA-A-Rezeptor-Missense-Varianten auf die Funktion der hippokampalen Pyramidenneuronen kann durch das GABAerge Shunting von apikalen Eingaben an das Neuron aus den Projektionen von CA3 und entorhinalen Kortex (EC) III Pyramidenneuronen untersucht werden. Mit anderen Worten, eine Möglichkeit, die Aktivität von GABA-A-Rezeptoren zu simulieren, besteht darin, einen Kontext anzunehmen, in dem die Simulation realistische Annahmen über die physiologische Bedeutung der Rezeptoren darstellt, wie z. B. die Shunt-Hemmung, einen der Mechanismen der GABAergen Hemmung. Die Pyramidenneuronen des CA1-Hippocampus, typischerweise in ihren apikalen Dendriten, haben GABA-A-Rezeptoren in diesen Zonen, die von den Projektionen der Neuronen des CA3 und EC III anvisiert werden. Diese Anordnung eignet sich somit für die Simulation. Diese Forschungsfrage erfordert ein Input-Design mit unterschiedlichen Verzögerungen und Intensitäten. Daher wurden drei verschiedene glutamaterge Synapsen (GluS1/2/3) auf den distalen apikalen, medialen apikalen und basalen Dendriten platziert, wie in Abbildung 10 gezeigt, und sie wurden nacheinander aktiviert. Um den Einfluss synaptischer Eingänge zu bewerten, sollte die Konstantstromamplitude unter der minimalen Schwelle für die Spike-Auslösung (Iinj <I min) bleiben. Das pyramidale Neuronenmodell mit entweder Wildtyp- oder mutiertem GABA-A-Rezeptor wurde mit einer Konstantstrominjektion von 0,85 nA am Soma initiiert. Die GABAerge Synapse wurde dann am Soma platziert. Die präsynaptische Aktivität, die vom Spike-Generator nachgeahmt wird, wurde zuerst am distalen apikalen Dendriten initiiert. Die synaptischen Eingänge an den medialen apikalen und basalen Dendriten waren um 25 ms bzw. 50 ms verzögert. Die GABAerge Synapse wurde mit einer Verzögerung von 40 ms aktiviert. Die GABAerge Inhibitionsintensität wurde so eingestellt, dass der gesamte Spike-Zug, mit Ausnahme des ersten Spikes, gehemmt wird. Dann wird der Einfluss von Varianten in diesem Setting untersucht, indem derτ-Anstieg, dieτ-Deaktivierung und gGABAA variiert werden.
Die Parameter für Wildtyp- und mutierte Rezeptoren wurden aus der im Protokollschritt 2.1.1 beschriebenen Sammlung speziell für Rezeptoren erhalten, die aus α1β3γ2 bestehen, was die häufigste Untereinheitenzusammensetzung in hippokampalen Pyramidenneuronendarstellt 65. Die Parameterverteilung ist in der Zusatzdatei 4: Zusatztabelle S16 angegeben.
Jede Untereinheitenmutation wurde an einfachen, doppelten und dreifachen glutamatergen Synapsenfällen getestet. In einem einfachen Ansatz kann der Einfluss von Mutationen über die Feuerrate und das Feuermuster bewertet werden. Die ΔtISI-Mittelwerte und die Standardabweichung können ebenfalls geschätzt werden, um die Änderungen im Brennmuster weiter zu messen, wobei ΔtISI die Änderung des Interspike-Intervalls darstellt. Die Ergebnisse für jeden Fall sind als Feuerraten und ΔtISI (Mittelwert und Standardabweichung) in der Zusatzdatei 4: Zusatztabelle S17 und Zusatzabbildung S3 angegeben. Die Spike-Züge und Spannungskurven für die Varianten, die die Zündmuster veränderten, sind in Abbildung 11, Abbildung 12 und Abbildung 13 dargestellt.
Für die einfache (GluS1) und dreifache (GluS1-2-3) glutamaterge Synapsenaktivierung waren die Mutationen, die die Neuronenantwort veränderten, nur β3N110D- und γ2K328M-Mutationen. Im Fall der einzelnen glutamatergen Eingabe führte β3N110D zu einer beeinträchtigten Hemmung, und das Feuermuster war nach dem 4. präsynaptischen Spike mit einer kurzen Verzögerung auf dem glutamatergen Spike-Zug fixiert (Abbildung 11). γ2K328M beeinträchtigte ebenfalls die Hemmung, wenn auch nur um den 5. präsynaptischen Spike, und führte zu einer größeren Verzögerung des postsynaptischen Spikes im Vergleich zu β3N110D (Abbildung 11). Im Fall der GluS1-2-3-Aktivierung (Abbildung 13) war das Ansprechen zwischen β3N110D- und γ2K328M-Mutationen ähnlich. Beide Mutanten ergaben ein Feuermuster, bei dem fast alle kumulierten präsynaptischen Spikes nachgewiesen wurden und eine Reaktion auslösten. In beiden Fällen feuerten Neuronenmodelle mit einem Spike-Paar als Reaktion auf präsynaptische Aktivität.
Die Aktivierung der doppelten glutamatergen Synapse führte im Vergleich zu den beiden anderen Einstellungen zu unterschiedlichen Ergebnissen (Abbildung 12). In diesem Fall beeinträchtigten zwei Mutationen auf der Untereinheitdes GABA-A-Rezeptors b3 (β3N110D und β3T288N) und zwei Mutationen auf der Untereinheitdes GABA-A-Rezeptors γ2 (γ2P302L und γ2K328M) die GABAerge Hemmung. Das Neuronenmodell mit der γ2P302L-Mutante feuerte fast synchron mit dem GluS2, was höchstwahrscheinlich eine verzögerte Reaktion auf das GluS1 mit ungefähr der gleichen Verzögerung wie präsynaptische Spikes zwischen GluS1-2 war. Die β3T288N-Mutation lieferte ein ähnliches Ergebnis, wobei die Unterscheidung des zweiten Spikes immer noch synchron mit GluS2 war. Das Neuronenmodell mit der β3N110D-Mutante reagierte auf fast jeden kumulierten glutamatergen Input, mit Ausnahme der ersten beiden präsynaptischen Spikes von GluS1/2, die mit einem kürzeren ΔtISI eingeführt wurden. Das Feuermuster für γ2K328M war wieder wie bei β3N110D, wobei die Unterscheidung zwischen dem zweiten und dritten präsynaptischen Spike übersehen wurde.
Diese Ergebnisse zeigen die vielfältigen Auswirkungen von Mutationen der b3-Untereinheit (kodiert durch das GABRB3-Gen ) und der γ2-Untereinheit (kodiert durch GABRG2) auf die Aktivität der pyramidalen Neuronen im Hippocampus. Interessanterweise führten die Mutationen β3L170R, β3A305V, β3E180G, β3D120N, β3Y302C und γ2R82Q zu keiner Veränderung der neuronalen Aktivität. Die schwerwiegendste Beeinträchtigung der Hemmung trat bei β3N110D und γ2K328M auf, die beide eine signifikant geringereτ-Deaktivierung und einen höherenτ-Anstieg aufwiesen. Unsere vorläufige Analyse zeigte auch, dass Veränderungen desτ-Anstiegs oder gGABAA allein nicht ausreichen, um die Hemmung zu beeinträchtigen (Daten nicht gezeigt). Es kann argumentiert werden, dass die Mutationen, die zu einer signifikant verringertenτ-Deaktivierung führen, zusammen mit einem erhöhtenτ-Anstieg zu einer signifikanteren Beeinträchtigung der GABAergen Hemmung führen.
In dem Fall, in dem alle exzitatorischen Eingaben gehemmt werden müssen, führt jede Mutation, die zum Feuern führt, zu abnormalen und unspezifischen neuronalen Reaktionen, die in einem neuronalen Schaltkreis, der aus Neuronen mit den gleichen Mutationen besteht, möglicherweise übertrieben werden. Das Gleichgewicht von Erregung/Hemmung in einem neuronalen Netzwerk kann durch die daraus resultierende beeinträchtigte inhibitorische Rückkopplung, die eine entscheidende Komponente jeder Netzwerkaktivität ist, erheblich beeinflusst werden.
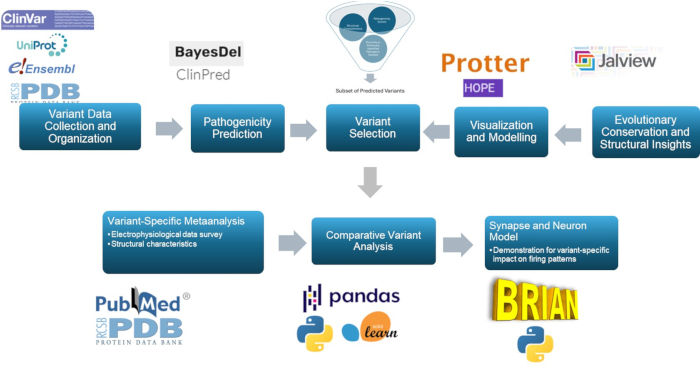
Abbildung 1: Überblick über die Vorhersage und Analyse von Varianteneffekten für klinische und Forschungszwecke, mit besonderem Fokus auf in silico Analyse und neuronale Reaktionssimulationen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Position von γ2A303T und ausgewählte Patientenmutationen;γ2P302L und γ2K328L werden für neuronale Simulationen verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
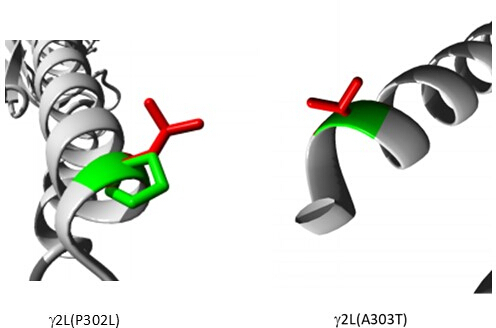
Abbildung 3: Vergleichende Modellierung der Patientenmutation γ2P302L und der benachbarten Variante γ2L (A303T), die als pathogen vorhergesagt wird. In beiden Modellen steht Grün für Wildtyp und Rot für mutierte Reste. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
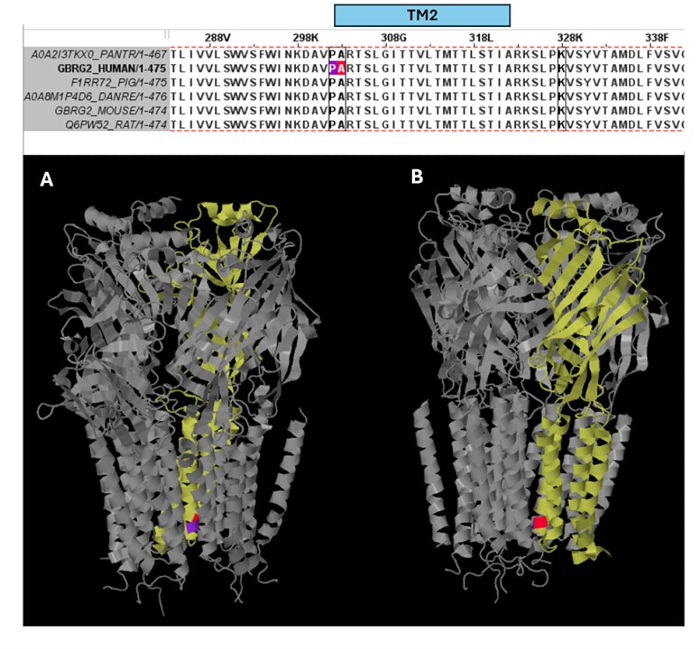
Abbildung 4: Multiple Sequenzausrichtung und strukturelle Einblicke. Das obere Bild zeigt die evolutionäre Konservierung der Reste in der Position der Patientenmutation (P302L) (violett) am Rand des TM2 und der pathogenen Variante A303T (rote Farbe) am Anfang des TM2 der γ2-Untereinheit. Das untere Bild zeigt die Visualisierung dieser konservierten Reste in der (A) dreidimensionalen GABAA Rezeptorstruktur (7QNE), wobei die γ2-Untereinheit (Kette C in der 7QNE) gelb und aus verschiedenen Winkeln (A, B) dargestellt ist. Abkürzung: TM2 = zweite Transmembrandomäne. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Lokalisierung aller enthaltenen Varianten. Es werden die Positionen der bekannten (schwarz) und der vorhergesagten (rot) Varianten in Bezug auf ihren (A) normierten Abstand von der Porenachse und (B) Abstand vom Membranzentrum gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
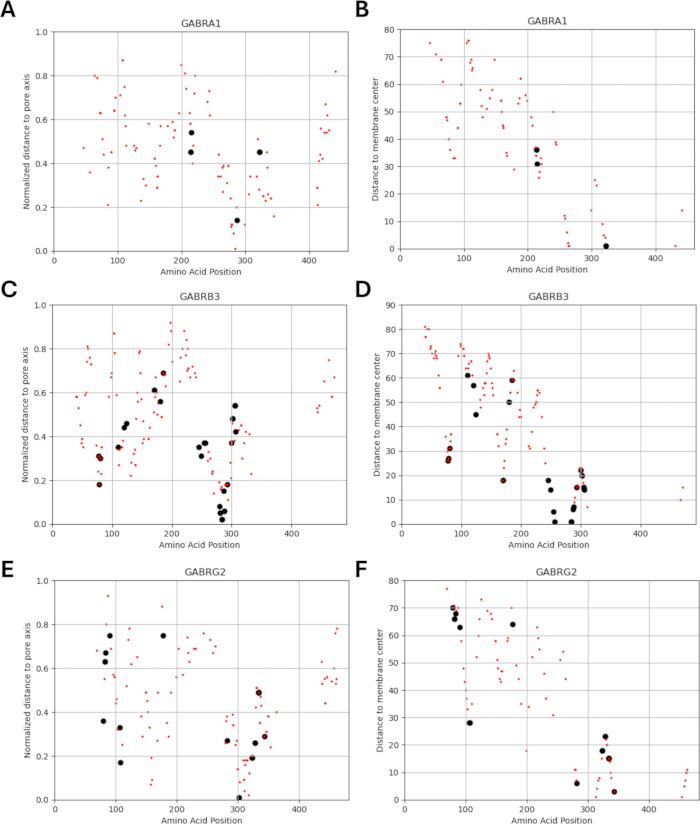
Abbildung 6: Lokalisierung der Varianten für jede Untereinheit. Es werden die Positionen der bekannten (schwarz) und vorhergesagten (rot) Varianten in Bezug auf ihren (A, C, E) normierten Abstand von der Porenachse und (B, D, F) Abstand vom Membranzentrum gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
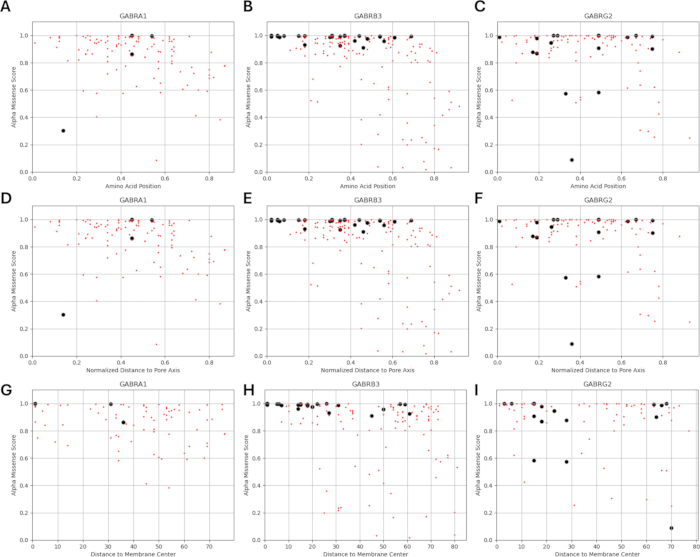
Abbildung 7: Verteilung des AlphaMissense-Scores über den Standort der Varianten. (A-C) Die AlphaMissense-Score-Verteilung über die Aminosäureposition, der (D-F) normalisierte Abstand von der Porenachse und der (G-I) Abstand vom Membranzentrum sind für bekannte (schwarze) und vorhergesagte (rot) Varianten angegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
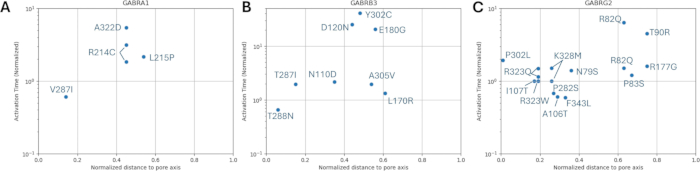
Abbildung 8: Normalisierte Aktivierungszeit von GABA-A-Rezeptoren mit Mutationen in der Untereinheit α1 (GABRA1), der Untereinheit β3 (GABRB3) und der Untereinheit γ2 (GABRG2). Die experimentell erhaltenen Aktivierungszeitkonstanten in Bezug auf den normierten Abstand von der Porenachse für jede Mutation auf (A) α1, (B) β3 und (C) γ2 Untereinheiten sind dargestellt. Die Werte wurden mit der jeweiligen Aktivierungszeit des Wildtyp-Rezeptors normiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 9: Normalisierte Deaktivierungszeit von GABA-A-Rezeptoren mit Mutationen in der α1 (GABRA1)-Untereinheit des GABA-A-Rezeptors, der β3 (GABRB3)-Untereinheit des GABA-A-Rezeptors und der γ2 (GABRG2)-Untereinheit. Die experimentell erhaltenen Deaktivierungszeitkonstanten in Bezug auf den normierten Abstand von der Porenachse für jede Mutation auf (A) α1, (B) β3 und (C) γ2 Untereinheiten sind dargestellt. Die Werte wurden mit der jeweiligen Deaktivierungszeit des Wildtyp-Rezeptors normiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 10: Pyramidenneuronenmodell CA1. Das Modellneuron besteht aus (1) einem Soma, (2) einem apikalen Dendriten mit proximalen, medialen und distalen Kompartimenten, die mit zwei Ästen an der Lamina molecularis enden, (3) zwei symmetrisch zusammengesetzten basalen Dendriten, die sich nach einem kurzen basalen Dendritenstamm in zwei Abschnitte verzweigen, und (4) einem Axon, das mit einem konischen Axonhügel beginnt, gefolgt von einem zylindrischen Axonanfangssegment, myelinisierte Segmente und Knoten von Ranvier, die mit einem kugelförmigen Axonterminal enden. Die grünen Dreiecke zeigen die Position der glutamatergen Synapsen an, und das rote Dreieck stellt die GABAerge Synapse dar, die sich am Soma befindet. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 11: Brennmuster mit nur GluS1-Aktivität. Die Spike-Züge für präsynaptische Neuronen (GluS1 (grünes Dreieck) und GABAergen (roter Kreis)) und die postsynaptischen Neuronen mit entweder Wildtyp- oder mutierten GABA-A-Rezeptoren (schwarzes Quadrat) sind im oberen Feld dargestellt. In den unteren Feldern sind einzelne Spannungskurven für Neuronen mit Wildtyp-GABA-A-Rezeptor mit oder ohne GABAerge Hemmung und für Neuronen mit mutierten GABA-A-Rezeptoren mit GABAerger Hemmung dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
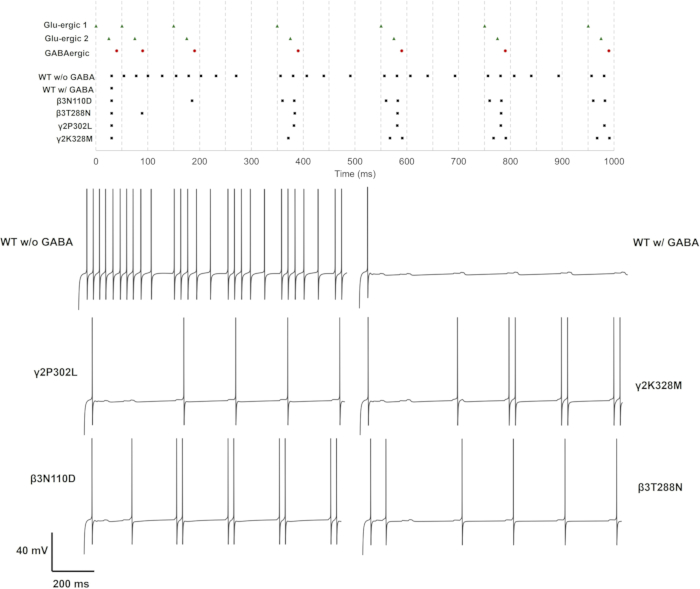
Abbildung 12: Brennmuster mit nur GluS1- und GluS2-Aktivität. Im oberen Bild sind die Spike-Züge für präsynaptische Neuronen (GluS1/2 (grünes Dreieck) und GABAerge (roter Kreis)) und die postsynaptischen Neuronen mit entweder Wildtyp- oder mutierten GABA-A-Rezeptoren (schwarzes Quadrat) dargestellt. In den unteren Feldern sind einzelne Spannungskurven für Neuronen mit Wildtyp-GABA-A-Rezeptor mit oder ohne GABAerge Hemmung und für Neuronen mit mutierten GABA-A-Rezeptoren mit GABAerger Hemmung dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 13: Brennmuster mit nur GluS1-, GluS2- und GluS3-Aktivität. Im oberen Bild sind die Spike-Züge für präsynaptische Neuronen (GluS1/2/3 (grünes Dreieck) und GABAergen (roter Kreis) sowie die postsynaptischen Neuronen mit entweder Wildtyp- oder mutierten GABA-A-Rezeptoren (schwarzes Quadrat) dargestellt. In den unteren Feldern sind einzelne Spannungskurven für Neuronen mit Wildtyp-GABA-A-Rezeptor mit oder ohne GABAerge Hemmung und für Neuronen mit mutierten GABA-A-Rezeptoren mit GABAerger Hemmung dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Verteilung der AlphaMissense-Scores und biophysikalischen Parameter (Normalisierte Deaktivierungszeit; Normalisiertes τd) von Mutationen der GABA-A-Rezeptor-Untereinheiten, die in der vorliegenden Studie ausgewählt wurden. Siehe auch Ergänzungsdatei 4: Ergänzende Tabelle 8. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Abbildung S2: Verteilung der AlphaMissense-Scores und biophysikalischen Parameter (Normalisierte Aktivierungszeit; Normalisiertes τr) der in der vorliegenden Studie ausgewählten Mutationen der GABA-A-Rezeptor-Untereinheit. Siehe auch Ergänzungsdatei 4: Ergänzende Tabelle 8. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Abbildung S3: Die Interspike-Intervalle für die neuronale Reaktion mit Wildtyp- und mutierten GABA-A-Rezeptoren. Das oberste Diagramm zeigt die Zeitintervalle zwischen den Spitzen für eine einzelne glutamaterge Eingabe. Das mittlere Diagramm zeigt zwei, und das unterste Diagramm zeigt drei glutamaterge Synapsen, die gleichzeitig aktiv sind. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzungsdatei 1: Die Datei "Data_GABAA. R" erforderlich, um in R für die Datenformatierung ausgeführt zu werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Gleichungen, die beim Entwurf des leitfähigkeitsbasierten Modells verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: GABAAvar.py erforderlich, um in Brian2 für die neuronale Simulation ausgeführt zu werden. Die Datei enthält die Python-Codes für das Brian2-basierte multikompartimentelle Neuronenmodell (Funktion: CA1_Pyr), Gleichungen für leitfähigkeitsbasierte Neuronen- und synaptische Modelle (Funktion: model_eqns, syn_eqns) und Anfangsparameter (Funktion: biophys_param, morpho_param, syn_param). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: Ein Zip-Ordner, der alle ergänzenden Tabellen enthält. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S1: Missense-Varianten unbekannter Signifikanz im GABRG2-Gen , die von ClinVar als .txt Datei heruntergeladen und anschließend als "data.xlxs" gespeichert wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S2: Identifikatoren der in der Studie verwendeten Sequenzen, das Referenztranskript des interessierenden Gens (NCBI Ref. ff.) und andere entsprechende Identifikatoren in verschiedenen Datenbanken). Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S3: Positionen der strukturellen und funktionalen Regionen. Die Positionen der spezifischen Regionen des γ2-Untereinheitsproteins (NCBI-Referenzsequenz: NP_944494.1), die durch das Referenztranskript kodiert werden (NM_198904.4) Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S4: Der Inhalt der Datei "data1.xlxs", die ClinVar-Daten von GABRG2 darstellt und nur die Spalten "GRCh38Chromosome", "GRCh38Location", "Name", "Protein change" enthält. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S5: Der Inhalt der Datei "data1_output.xlsx", die die erforderliche Formatierung von Missense-Varianten von GABRG2-Daten enthält, die zur Vorhersage des Varianteneffekts auf den dbNSFP-Server hochgeladen werden sollen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S6: Der Inhalt der Datei "data2.xlsx", die die Ausgabe des dbNSFP-Servers für die Vorhersage des Varianteneffekts für unbekannte Missense-Varianten von GABRG2 enthält. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S7: AlphaMissense-Scores für GABA-A-Rezeptor-Untereinheitenvarianten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S8: Biophysikalische Eigenschaften von Varianten. Die Werte für die biophysikalischen Parameter wurden aus früheren Studien mit elektrophysiologischen Experimenten gewonnen. Varianten werden mit dem Typ "S" (Substitution) markiert, während die Wildtyp-Rezeptorparameter für jede Substitution angegeben und mit "C" (Kontrolle) gekennzeichnet sind. τd : Konstante der Deaktivierungszeit, POffen : Öffnungswahrscheinlichkeit, gGABA: Leitfähigkeit des Rezeptors, Imax: Maximaler Strom, τr : Konstante der Aktivierungszeit. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S9: Physikalisch-chemische Eigenschaften der Varianten. Zuvor identifizierte Varianten mit biophysikalischen Parametern werden mit dem Typ "S" (Substitution) gekennzeichnet, während die vorhergesagten Varianten mit "P" dargestellt werden. H: Änderung der Hydrophobizität, VSC: Änderung des Volumens der Seitenkette, P1: Änderung der Polarität, P2: Änderung der Polarisation, SASA: Änderung der lösungsmittelzugänglichen Oberfläche, NCISC: Änderung des Nettoladungsindex. Die Werte werden für jede ursprüngliche Aminosäure und Variante von Guo et al.36 erhalten und die Änderung jedes Parameters wird wie gegeben geschätzt. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S10: Strukturelle Merkmale der Varianten. Zuvor identifizierte Varianten mit biophysikalischen Parametern werden mit dem Typ "S" (Substitution) gekennzeichnet, während die vorhergesagten Varianten mit "P" dargestellt werden. Die Lokalisierung der Variante in einer Domäne wird mit 1 dargestellt, sonst mit 0. Alle Werte stammen von Brünger et al.35. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S11: Strukturelle und biophysikalische Parameterkorrelationen für alle bekannten Varianten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S12: Strukturelle, physikalisch-chemische und biophysikalische Parameterkorrelationen für bekannte GABRA1-Varianten . Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S13: Strukturelle, physikalisch-chemische und biophysikalische Parameterkorrelationen für bekannte GABRB2-Varianten . Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S14: Strukturelle, physikalisch-chemische und biophysikalische Parameterkorrelationen für bekannte GABRB3-Varianten . Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S15: Strukturelle, physikalisch-chemische und biophysikalische Parameterkorrelationen für bekannte GABRG2-Varianten . Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S16: Biophysikalische Parameter für Wildtyp- und mutierte α1β3γ2 GABAA-Rezeptoren. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S17: Feuerungsrate und Interspike-Intervalle als Reaktion auf einfache, doppelte oder dreifache glutamaterge Synapsen mit Wildtyp- und mutierten GABA-A-Rezeptoren.Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Durch die Anwendung einer Kombination aus computergestützter Genetik, molekularer Modellierung und neuronalen Simulationen hat der in dieser Arbeit vorgestellte Ansatz das Potenzial, die Klassifizierung von GABA-A-Rezeptorvarianten zu verbessern und wertvolle Erkenntnisse sowohl für die Epilepsieforschung als auch für klinische Anwendungen zu liefern. Eine umfassende Analyse zur Identifizierung und Priorisierung von vorhergesagten pathogenen Mutationen wird vorgestellt und zu einem Framework erweitert, das möglicherweise die Lücke zwischen varianten Effekten auf Protein und zellulären Phänotyp schließt. Die Bewertung des Einflusses der epileptogenen GABA-A-Rezeptoraktivität auf die Simulation von pyramidalen Neuronen im Hippocampus ermöglicht die Replikation des in vitro-Phänotyps, der mit einer GABA-A-Rezeptor-Dysfunktion assoziiert ist, und den Nachweis einer Veränderung der Einzelneuronenantwort an der Wurzel der Netzwerkdysfunktion. Basierend auf diesen Simulationen neuronaler Reaktionen, die durch epileptogene Mutationen hervorgerufen werden, wurde eine grobe Abschätzung der funktionellen Effekte strukturell proximal vorhergesagter Mutationen untersucht. Die Vorhersagen über den Einfluss vorhergesagter Mutationen auf die Kanalkinetik erfordern eine gründliche Analyse mit bekannten Variantensätzen. Vergleichende Analysen, wie sie in diesem Artikel vorgestellt werden, und neuronale Aktivitätssimulationen liefern wichtige Erkenntnisse für die weitere Generierung und Verbesserung von Vorhersagemodellen, die sich auf den Varianteneffekt auf die Proteinfunktion und die neuronale Pathologie konzentrieren. Darüber hinaus kann unsere Methodik verwendet werden, um die pathogensten Varianten unter den unbekannten Varianten auszuwählen und zu priorisieren, um Varianteneffekte zu untersuchen, die mit GABA-A-Rezeptor-bedingten neurologischen Entwicklungsstörungen verbunden sind. Zum Beispiel können Rezeptoruntereinheiten, die mit den Fluoreszenzsonden 66,67,68,69,70 markiert sind und die vorhergesagten Mutationen tragen, in vitro exprimiert werden, um ihren Transport, ihre Zelloberflächenexpression und ihre Neurophysiologie zu untersuchen. Darüber hinaus können Tiermodelle wie C. elegans in Betracht gezogen werden, um die Auswirkungen vorhergesagter Mutationen zu validieren. Zum Beispiel wurde die CRISPR-Cas9-Geneditierung verwendet, um eine Deletion von unc-49, einem C. elegans-GABA-A-Rezeptor, zu erzeugen, wodurch homozygote Epilepsie-assoziierte Mutationen in unc-49 oder Untereinheiten des humanen GABA-A-Rezeptors71 erzeugt wurden.
Im Allgemeinen profitiert die Klassifizierung von Varianten von der Verwendung mehrerer Ebenen der rechnerischen Evidenz, wie sie von ACMG-AMP12 empfohlen wird. Dieser Ansatz stärkt die Zuverlässigkeit der Variantenklassifikation durch die Integration verschiedener prädiktiver Instrumente und Datenquellen, wodurch letztendlich die Genauigkeit klinischer Bewertungen und der gesamte Entscheidungsprozess in der genomischen Diagnostik verbessert werden. In unserer Methodik ist die Verwendung von Ensemble-Prädiktoren, die die Vorhersagen mehrerer Werkzeuge kombinieren, von Vorteil, wodurch die Anforderung an mehrere Zeilen rechnerischer Evidenz erfüllt wird und die Notwendigkeit entfällt, verschiedene Werkzeuge separat zu verwenden. Dieser Ansatz überwindet auch die Herausforderung, unterschiedliche Ergebnisse einzelner Tools zu verarbeiten, wodurch der Vorhersageprozess rationalisiert und die Effizienz gesteigert wird. Dennoch gibt es keine Garantie für die Vorhersagegenauigkeit genzentrierter oder variantenspezifischer Analysen. Dies führt zu der Schlussfolgerung, dass genzentrierte oder variantenspezifische Vorhersagen unter spezifischen Bedingungen durchgeführt werden sollten, die an die spezifischen Kontexte und Ziele angepasst sind 15,72,73,74. Für klinische Interventionen würde dies die Bewertung der Vorhersagegenauigkeit von In-silico-Werkzeugen für ein bestimmtes Gen oder eine Untergruppe von Genen im Kontext einer gegebenen Krankheit erfordern, oft mit individualisierter Optimierung75. Die Beurteilung der prädiktiven Genauigkeit wird jedoch häufig durch das Fehlen einer ausreichenden Anzahl von Varianten eingeschränkt, was die Zuverlässigkeit der Genauigkeitsbewertung beeinträchtigen kann.
In der Literatur stehen verschiedene Werkzeuge zur Verfügung, deren Genauigkeit in Datensätzen getestet und validiertwird 14. Diese Genauigkeitsergebnisse, die auf großen Datensätzen basieren, spiegeln sich jedoch nicht unbedingt in der Vorhersage einiger weniger unbekannter Varianten für ein bestimmtes Gen wider. In diesem Zusammenhang deutet die zunehmende Literatur darauf hin, dass Ensemble-Prädiktoren, die die Ergebnisse einzelner Prädiktoren zusammenstellen und berechnen, bekanntermaßen besser abschneiden als die Konkordanz einzelner Prädiktoren 33,76,77,78, und daher haben wir uns in der vorliegenden Studie entschieden, Ensemble-Prädiktoren, nämlich BayesDEL 33 und ClinPred 32, speziell für ihre überlegene Leistung32 zu verwenden. 34 BayesDEL wurde vergleichend auf 4.094 Missense-Varianten in klinisch relevanten Genen untersucht, einschließlich Genen, die für Transmembranproteine wie den spannungsgesteuerten Natriumkanal alpha subunit 5 (SCN5A) kodieren, und zeigte eine überlegene Leistung33. In unserem Protokoll zur Vorhersage von Varianteneffekten haben wir in einem ersten Schritt den Konsens von zwei Ensemble-Prädiktoren (BayesDEL und ClinPred) berücksichtigt. AlphaMissense37, ein von Google DeepMind entwickeltes Deep-Learning-Modell, ist eine Erweiterung von AlphaFold64,79 und nutzt somit die Leistungsfähigkeit einer hochpräzisen Vorhersage der Proteinstruktur. Als wir die anfänglichen Vorhersageergebnisse von Ensemble-Modellen (BayesDEL und ClinPred, wie in unserem Protokollschritt 1.3 beschrieben) mit den Ergebnissen von AlphaMissense verglichen, stimmten die Vorhersagen teilweise miteinander überein (Ergänzende Datei 4: Ergänzende Tabelle S15) und stimmten nicht vollständig mit den Vorhersagen von Ensemble-Modellen (BayesDEL und ClinPred) überein, die einen Konsens von pathogenen oder krankheitsassoziierten als rosa Zeilen dargestellt (Zusatzdatei 4: Zusatztabelle S15). Die unbekannten Varianten (L81F, A303T und V329F) in der Nähe der GABRG2-Mutationen R82Q, P302L und K328M, die wir in unserem Neuronenmodell verwendet haben und die sowohl von ClinPred als auch von BayesDEL als pathogen vorhergesagt wurden, wurden jedoch auch von AlphaMissense als pathogen vorhergesagt, wie durch gelbe Hervorhebungen dargestellt (Ergänzungsdatei 4: Ergänzende Tabelle S15).
Da AlphaMissense29 die Vorhersage von Sequenz und strukturellem Kontext verwendet, wollten wir in unserer Studie auch sehen, ob es einen Zusammenhang zwischen den AlphaMissense-Scores und den Mutationsorten des GABA-A-Rezeptors gibt, basierend auf ihren Abständen vom Membranzentrum und der Porenachse. Unsere Hypothese basiert auf der Idee, dass der funktionelle Einfluss von Aminosäurevarianten neben oder in der Nähe der funktionell identifizierten Mutationen der GABA-A-Rezeptor-Untereinheiten ähnliche Muster physikalisch-chemischer Veränderungen in der Kanalfunktion aufweisen könnte, die bei Mutationen beobachtet wurden. Eine Korrelation zwischen den Mutationspositionen der GABA-A-Rezeptor-Untereinheiten und den AlphaMissense-Scores würde uns helfen, eine brauchbare Beziehung zu identifizieren, um einen Rahmen für unsere Hypothese zu schaffen, der die Vorhersage der funktionellen Konsequenzen neuer Missense-Varianten in GABA-A-Rezeptor-Untereinheiten ermöglicht. Die AlphaMissense-Scores waren jedoch nicht prädiktiv für Veränderungen dieser biophysikalischen Parameter (Abbildung 7). Es ist wichtig zu beachten, dass die begrenzte Stichprobengröße in unserer Analyse es schwierig macht, endgültige Schlussfolgerungen zu ziehen. Unsere Analyse ergab jedoch, dass die AlphaMissense-Scores nicht mit den strukturellen Parametern derGABA-A-Rezeptoren korrelierten. Das Fehlen einer klaren Positionskorrelation (z.B. zwischen den Positionen der Mutationen und den AlphaMissense-Scores) stellt die Gültigkeit unserer Annahme in Frage. Wenn benachbarte Reste tatsächlich ähnliche Effekte hätten, würden wir eine klarere Korrelation erwarten. Da dies nicht der Fall ist, schwächt es die Fähigkeit, AlphaMissense-Scores als zuverlässiges Werkzeug zum Testen unserer Annahme zu verwenden.
Interessanterweise haben wir in unserer Studie eine leichte Korrelation zwischen dem Abstand der Variante zur Porenachse und der normalisierten Kanalaktivierungszeit für die GABRG2-Genmutanten gefunden. Daher kann unsere vorläufige Annahme, dass benachbarte Aminosäuren ähnliche Konsequenzen haben, in einigen Regionen des Kanals zutreffen, wie z. B. in Regionen in der Pore oder an Schlüsselstellen, die an der Ansteuerung beteiligt sind, aber in anderen Regionen möglicherweise nicht so eindeutig sein. Der kleine Datensatz schränkt die Fähigkeit ein, diese Variabilität zu erkennen, aber zukünftige Daten oder eine detailliertere Strukturanalyse könnten helfen, diesen Aspekt unserer Hypothese zu verfeinern. Molekulardynamische Simulationen80 könnten als leistungsfähiger ergänzender Ansatz dienen, um diese vorläufigen Ergebnisse weiter zu untersuchen, insbesondere im Rahmen der vergleichenden Bewertung von zwei benachbarten γ2-Untereinheitsmutationen, nämlich der epileptogenen Mutation γ2P302L40 und der proximalen vorhergesagten Mutation γ2A303T (rs1581439874), die in unserer Studie durchgeführt wurde. In Zukunft könnte dieser Ansatz eine genauere Abschätzung des Effekts einer unbekannten Variante auf den zellulären Phänotyp ermöglichen, insbesondere wenn er mit den in unserer Studie vorgestellten neuronalen Simulationen integriert wird.
Darüber hinaus wird es interessant sein zu untersuchen, ob die strukturellen und physikalisch-chemischen Eigenschaften von GABA-A-Rezeptor-Untereinheiten zusammen mit anderen Merkmalen genutzt werden können, um leistungsfähige maschinelle Lernmodelle für die funktionelle Vorhersage neuartiger Varianteneffekte auf den Kanal, das Neuron, das Netzwerk und den Krankheitsphänotyp zu trainieren. Mit dem Aufkommen automatisierter Ansätze des maschinellen Lernens haben wir einen Punkt erreicht, an dem Ärzte und Nasslaborwissenschaftler auch ihre eigenen Modelle in einer stärker demokratisierten Umgebung entwickeln können81. Daher könnte die Integration dieser Technologien in die klinische Praxis den Prozess möglicherweise rationalisieren, die personalisierte Medizin zugänglicher machen und den Rückgriff auf hochspezialisiertes Fachwissen für die Analyse funktioneller Varianten verringern. In diesem Zusammenhang bietet unser Ansatz Einblicke in die strukturelle und funktionelle Dynamik des Rezeptors und hilft möglicherweise bei zukünftigen Studien zur funktionellen Vorhersage von Varianteneffekten.
Trotz der aktuellen Fortschritte in der Vorhersage der Proteinstruktur und des Durchbruchs, den AlphaFold64 darstellt, bleibt eine genaue Vorhersage der Auswirkungen von Mutationen und der Proteinfunktion eine Herausforderung, da für das Training des Modells nur wenige Daten erforderlich sind79. Für die Vorhersage des Varianteneffekts zeigt AlphaMissense eine höhere Leistung im Vergleich zu einer Untergruppe von Vorhersagemodellen, aber die Ensemble-Prädiktoren BayesDEL25 und ClinPred24, die in unserer Studie verwendet wurden, wurden in diesem Vergleich nicht berücksichtigt29. Es ist wichtig zu beachten, dass in unserer Studie die In-silico-Tools BayesDEL, ClinPred und AlphaMissense für unterschiedliche Zwecke eingesetzt wurden. Die Ensemble-Prädiktoren BayesDEL und ClinPred wurden hauptsächlich für die Vorhersage der Pathogenität verwendet, während AlphaMissense speziell verwendet wurde, um die Beziehung zwischen seinen Scores und den bekannten Daten für den Einfluss von Mutationen in der γ2-Untereinheit zu untersuchen. Konkret geht unsere Hypothese davon aus, dass vorhergesagte pathogene Varianten, insbesondere solche, die sich in der Nähe oder in der Nähe von funktionell identifizierten Mutationen in GABA-A-Rezeptor-Untereinheiten befinden, biophysikalische Parameter aufweisen, die denen von funktionell charakterisierten Mutationen ähneln. Um dies zu untersuchen, haben wir AlphaMissense ausgewählt, da es auf dem hochpräzisen AlphaFold264-Modell basiert, das die basische Peptidsequenz verwendet, um die Folgen einzelner Aminosäuresubstitutionen vorherzusagen.
Folglich ist eine wesentliche Einschränkung unserer Studie in erster Linie auf die begrenzte Verfügbarkeit experimenteller Daten zurückzuführen. Zum Beispiel basiert unser Neuronenmodell auf der Expression der Daten, die aus der α1β3γ2-Untereinheitenkombination von GABA-A-Rezeptoren abgeleitet wurden, was die in der Literatur untersuchten Mutationen inhärent auf die Untereinheiten beschränkt, die als Teil dieser spezifischen Rezeptorkombination exprimiert werden. Darüber hinaus stützten wir uns auf elektrophysiologische Daten, die ausschließlich aus der Expression dieser Untereinheiten in HEK-Zellen abgeleitet wurden, was den Umfang der in der Literatur verfügbaren Daten weiter einschränkte. Unsere Verwendung neuronaler Modellierung zur Abschätzung der Auswirkungen unbekannter Varianten geht davon aus, dass unbekannte Varianten (die in unserem Arbeitsablauf als pathogen vorhergesagt werden), die sich in unmittelbarer Nähe bekannter Mutationen befinden, ähnliche Muster in den Parametern der Kanalkinetik oder den physikalisch-chemischen Eigenschaften von Mutationseffekten aufweisen, die in der Literatur beschrieben sind. Diese Annahme, gepaart mit dem Bedarf an elektrophysiologischen Daten für spezifische Rezeptoranordnungen in HEK293-Zellen, reduziert die Menge an experimentellen Daten, die für die Modellierung zur Verfügung stehen. Aufgrund dieser Einschränkungen konnten wir mit den verfügbaren Daten nur eine begrenzte Anzahl von Varianten in α1β3γ2-Untereinheiten modellieren. Das Training des Neuronenmodells für verschiedene Untereinheitenanordnungen wie α1β2γ2 Untereinheitenkombinationen, α1β2δ oder α4β3δ Untereinheitenkombinationen, die untereinheitenspezifische Auswirkungen auf Zell-, Schaltkreis- und Netzwerkebene haben, wird jedoch wahrscheinlich eine breitere Anwendbarkeit auf verschiedene Epilepsietypen und neurologische Entwicklungsstörungen zeigen. In Zukunft könnte es mit der Zunahme verfügbarer elektrophysiologischer Daten und Studien, die sich auf Mutationen in gut definierten Rezeptoranordnungen und spezifischen Zelltypen konzentrieren, möglich werden, die Generalisierbarkeit und Genauigkeit unseres Ansatzes zu verbessern.
Die multikompartimentellen Leitfähigkeits-basierten Neuronenmodelle bieten ein leistungsfähiges Werkzeug, um Vorhersagen für die funktionelle Bedeutung von Rezeptorvarianten der Einzelneuronenantwort zu generieren. Dieses Tool ermöglicht flexible Definitionen sowohl zellulärer/synaptischer Parameter als auch ihrer Positionen, um jede spezifische Frage zu testen. Einfache Spike-Generatoren, die in diesem Protokoll verwendet werden, können durch andere Neuronenmodelle ersetzt werden, um die Aktivität von Mikroschaltkreisen zu untersuchen. Der kritische Schritt des Protokolls ist auch der einschränkendste Schritt: die Definition einer Rezeptorvariante in Bezug auf eine veränderte Kanalkinetik. Die benötigten Informationen werden idealerweise durch Patch-Clamp-Elektrophysiologie-Studien bereitgestellt; Aber auch die computergestützte Analyse von Aminosäuresubstitutionen mit prognostizierter klinischer Signifikanz und die Vergleiche mit bekannten Substitutionen können Aufschluss geben. Unsere Studie und das beschriebene Protokoll beinhalten die Verwendung von neuronalen Aktivitätssimulationen nicht als Vorhersagewerkzeug, sondern als Werkzeug zur Erforschung der Auswirkungen von Mutationen, um einen breiteren Blick auf die Auswirkungen veränderter biophysikalischer Eigenschaften des GABA-A-Rezeptors auf die Aktivität einzelner Neuronen zu ermöglichen. Die Abhängigkeit von experimentellen Daten in neuronalen Simulationen ist eine wichtige Einschränkung in unserem Ansatz, der von fortschrittlicher molekularer Modellierung profitieren könnte, um die Lücke zwischen Struktur und Funktion zu schließen.
In unserem Protokoll sollten bestimmte Schritte kritisch bewertet werden. Erstens kann die Wahl des Vorhersagemodells, das im ersten Teil unseres Protokolls verwendet wird, entscheidend sein. Die Auswahl von In-silico-Werkzeugen hängt von mehreren Faktoren ab, einschließlich der Generierung ausreichender mehrerer Zeilen rechnerischer Beweise für eine aussagekräftige Vorhersage12. Ensemble-Prädiktoren, die die Analyse mehrerer prädiktiver Algorithmen integrieren, passen besser zu dieser Empfehlung und werden daher im Vergleich zu individuellen Prädiktoren bevorzugt. Es gibt verschiedene Prädiktoren, und ihre Genauigkeit wird in der Regel in großen Datensätzen getestet, was nicht unbedingt die Genauigkeit des Vorhersagemodells garantiert, das für unbekannte Varianteneffekte in einem bestimmten Gen verwendet wird. Dies wird durch die Verwendung von zwei Ensemble-Modellen kompensiert, die die Vorhersageergebnisse aus mehreren Prädiktoren sammeln und berechnen. Darüber hinaus können die Cutoffs der Vorhersagemodelle angepasst werden, um die Spezifität zu erhöhen, wenn der Zweck in erster Linie darin besteht, die pathogensten Varianten zu identifizieren. Die Festlegung geeigneter Grenzwerte ist wichtig, um den Kompromiss zwischen Sensitivität und Spezifität auszugleichen und eine genaue Klassifizierung der Varianten zu gewährleisten. In unserer Studie haben wir die Standard-Cutoffs verwendet. Insbesondere haben wir den Cutoff nicht zugunsten der Erfassung von Varianten geändert, die mit größerer Wahrscheinlichkeit pathogen sind, da dies den Umfang der zu untersuchenden Varianten auf mehreren Ebenen unserer Analyse, wie in unserem Workflow beschrieben, verringern würde. Es ist auch wichtig zu beachten, dass bei der Auswahl der Strukturdaten für die dreidimensionale Rekonstruktion des interessierenden Proteins eine vorläufige Überprüfung der Strukturdaten in der Literatur erforderlich ist. Die strukturelle Untersuchung von GABA-A-Rezeptoren hat in letzter Zeit an Dynamik gewonnen, mit robusten Strukturstudien, die über die dreidimensionale Struktur verschiedener Rezeptoranordnungen unter verschiedenen Bedingungen berichten 26,27,82,83,84,85. Angesichts der Verfügbarkeit dieser Daten haben wir uns in unserer Studie auf die experimentell ermittelten Strukturen für die Rekonstruktion von Strukturdaten konzentriert. Die Vorhersage von AlphaFold64 könnte jedoch für die Analyse anderer Proteine bevorzugt werden, für die solche experimentell ermittelten Daten fehlen. Bei Strukturdaten, die aus experimentellen Studien abgeleitet werden, ist es wichtig, auf die Aminosäurenummerierung zu achten. Die PDB-Nummerierung von Aminosäuren kann sich von der UniProt-Nummerierung unterscheiden, da erstere möglicherweise nicht das Signalpeptid enthält, das während der Proteinreifung entfernt wird. Außerdem können chimäre Proteine, die im experimentellen System exprimiert werden, Diskrepanzen verursachen. In diesem Fall trägt die paarweise Ausrichtung der interessierenden Sequenz mit der aus den Strukturdaten abgeleiteten Sequenz dazu bei, die Konsistenz zu wahren. In der Literatur basieren die Strukturdaten für das γ2-Untereinheitsprotein auf unterschiedlichen Methoden, darunter experimentelle Methoden wie die Elektronenmikroskopie (EM) und die hochgenaue Vorhersagemethode von AlphaFold. Wenn die experimentelle Methode die gewünschte Sequenz nicht vollständig mit hoher Auflösung abdeckt, können AlphaFold-Vorhersagen verwendet werden. In der vorliegenden Studie wurde die Struktur 7QNE26 gewählt, da sie der kryogenen elektronenmikroskopischen Struktur des menschlichen synaptischen α1β3γ2 GABA A-Rezeptors in voller Länge entspricht. Dies stellt genau die Kombination von Untereinheiten in voller Länge dar, die im Mittelpunkt der vorliegenden Studie stand.
Darüber hinaus sollte für die vergleichende Analyse die Verwendung normalisierter Parameter der Kanalkinetik bevorzugt werden, da die Werte dieser Parameter je nach Zusammensetzung der Rezeptoruntereinheit und experimentellen Einstellungen variieren können. Zum Beispiel sollten derAnstieg von τ und dieDeaktivierung von τ über x-fache Änderungen über die Wildtyp-Kontrollwerte bestimmt werden. Im Protokollschritt 2.5 kann für eine geringe Anzahl bekannter Varianten eine parametrische oder kategoriale Korrelationsanalyse und die Bestimmung von rho- und p-Werten bevorzugt werden. Im Idealfall wird erwartet, dass Methoden wie die Hauptkomponentenanalyse genauere Beziehungen liefern, aber eine höhere Anzahl von Proben erfordern.
Die Simulationsumgebung kann geändert werden. In dieser Studie wurde Brian2 aus folgenden Gründen bevorzugt: Die Spatialneuronenklasse in Brian2 bietet ein wertvolles Werkzeug zur Simulation neuronaler Aktivität. Brian2 hat einen erheblichen Vorteil bei der Verwendung von Differentialgleichungen zur Beschreibung kontinuierlicher Dynamik und Aktualisierungsanweisungen für diskrete Ereignisse, anstatt sich auf vordefinierte "Black-Box"-Modelle zu verlassen, und dies führt zu einer hervorragenden Lesbarkeit und Anpassungsfähigkeit des Codes, da jeder Aspekt des Modells explizit in einem einzigen Python-Skript definiert werden kann, wobei die Modellgleichungen in mathematischer Schreibweise angegeben werden und nur eine winzige Menge an Brian-spezifischem Vokabular verwendet wird37. Da das Modell explizit beschrieben wird, sind alle Merkmale dokumentiert und können in der primären Simulationsbeschreibungsdatei gefunden werden, wodurch die bisher verwendeten "Black-Box"-Modelle, wie sie in Studien von Stimberg et al.45,86 erwähnt wurden, überflüssig werden.
Das aktuelle Neuronenmodell verwendet modifizierte Leitwerte46 vom Hodgkin-Huxley-Typ mit Na+, K+ und Leckstrom. Dieses leitfähigkeitsbasierte Modell kann durch die Einbeziehung mehrerer anderer Kanaltypen, wie z. B. Ca2+ -Kanäle, weiter erweitert werden. Für Synapsenmodelle ist es wichtig zu beachten, dass diese Parameter für bestimmte Untereinheitenzusammensetzungen erhalten werden sollten und nur die Varianten mit Parametern, die mit diesen Zusammensetzungen gemessen wurden, bewertet werden sollten. In dieser Studie wurde die α1β3γ2-Rezeptorzusammensetzung ausgewählt; Daher wurden nur die Varianten von α1, β3 oder γ2 mit Kanalkinetikparametern, die an einem α1β3γ2 GABAA-Rezeptor gemessen wurden, eingeschlossen.
Die Schätzung der zellulären Biophysik beinhaltet die Segmentierung der Zelle in mehrere zylindrische Kompartimente, von denen jedes möglicherweise unterschiedliche Leitfähigkeitseigenschaften besitzt. Trotz der Unregelmäßigkeiten in den Dendriten eines Neurons können sie als lokal homogene Stränge betrachtet werden. Solche Modelle helfen dabei, die komplexe Struktur und Funktionsweise von Zellen zu verstehen. Das Modelldesign konzentriert sich auf eine vereinfachte Version der tatsächlichen Morphologie, die diese Merkmale widerspiegeln kann.
Der Ort und die physikalisch-chemischen Eigenschaften der veränderten Aminosäure bestimmen den Einfluss auf die Kanalkinetik. Zum Beispiel verringert eine Aminosäuresubstitution, die zu einer Aminosäure mit einer größeren Seitenkette führt, die Leitfähigkeit des Kanals, wenn diese Änderung an einer Aminosäure auftritt, die die Pore des Kanals auskleidet. Die Ersetzungen können sich auch auf das Öffnen/Schließen des Kanals auswirken. Der Einfachheit halber ist in diesem Modell die GABA-Bindungskinetik nur auf ein Verhältnis der verfügbaren Rezeptoren reduziert; Es können jedoch detailliertere Modelle entworfen werden, die diese Wechselwirkung einbeziehen, um die möglichen Auswirkungen von Substitutionen zu untersuchen, die die Bindungsaffinität der Liganden verändern.
Zusammenfassend lässt sich sagen, dass in der vorliegenden Studie computergestützte Methoden eingesetzt werden, um pathogene Varianten in den GABA-A-Rezeptor-Untereinheiten vorherzusagen und den möglichen zellulären Phänotyp qualitativ abzuschätzen, basierend auf der Simulation von Epilepsie-assoziierten Mutationen im hippokampalen pyramidalen Neuronenmodell. Nach unserem besten Wissen ist dies das erste Protokoll, das die kombinierte Anwendung von Computergenetik, molekularer Modellierung und neuronalen Simulationen untersucht, um zu bewerten, wie genetische Varianten zur Dysfunktion von GABA-A-Rezeptoren auf mehreren Komplexitätsebenen beitragen können, von der DNA über die Proteinfunktion bis hin zum neuronalen Verhalten. Dieses Protokoll kann eine Grundlage für eine bessere Abschätzung potenziell schädlicher Varianten und der damit verbundenen neuronalen Pathologie bei Epilepsie bieten. Darüber hinaus kann es bei der Untersuchung anderer Kanalopathien eingesetzt werden, was wichtige Einblicke in den zugrunde liegenden Mechanismus relevanter neurologischer Entwicklungs- und Netzwerkstörungen liefert. Auf dieser Grundlage kann durch die Einbeziehung der strukturellen und physikalisch-chemischen Bewertungen von GABA-A-Rezeptoren zur Untersuchung der funktionellen Auswirkungen von Varianten und ihrer Integration in die Kanalkinetik des GABA-A-Rezeptors in Zukunft eine genauere Analyse entwickelt werden.
Offenlegungen
Alle Autoren erklären, dass sie keine Interessenkonflikte im Zusammenhang mit dieser Arbeit haben.
Danksagungen
Wir danken Çağla Koca für ihre Unterstützung beim Aufbau des Modellneurons.
Materialien
| Name | Company | Catalog Number | Comments |
| Brian2 | Sorbonne Université, INSERM, CNRS, Institut de la Vision, France; Imperial College London, United Kingdom | 2.8.0.4 | Stimberg et al., 2019 (https://pypi.org/project/Brian2/ ) |
| dbNSFP server | Genos Bioinformatics LLC, USA | v3.0 | Liu et al., 2020 (http://database.liulab.science/dbNSFP) (https://sites.google.com/site/jpopgen/dbNSFP) |
| HOPE | Centre for Molecular and Biomolecular Informatics CMBI, Radboud University, Netherlands | 1.1.1 | Venselaar et al., 2010 (https://www3.cmbi.umcn.nl/hope/) |
| Jalview | University of Dundee, UK | JV2 | Waterhouse et al., 2009 (https://www.jalview.org/) |
| Jupyter Notebook | Project Jupyter, USA | https://jupyter.org/install | |
| Phyton | Python Software Foundation, USA | 3.13 | https://www.python.org/downloads/ |
| Protter | ETH Zurich, Switzerland | Version 1.0 | Omasits, et al., 2014 (https://wlab.ethz.ch/protter/start/) |
| R | The R Foundation for Statistical Computing, USA | R version 4.3.2 | https://www.r-project.org/ |
| RStudio | Posit software, PBC, USA | RStudio 2023.12.1+402 "Ocean Storm" Release | https://posit.co/downloads/ |
Referenzen
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten