Method Article
Identificazione e classificazione delle varianti missenso della subunità del recettore GABAA posizionate per il loro ruolo nei neuroni piramidali dell'ippocampo
In questo articolo
Riepilogo
Questo studio introduce un quadro multiscala, che spazia dal DNA alla funzione delle proteine e al comportamento neurale. Presenta un nuovo approccio per lo studio delle mutazioni patogene previste nella subunità del recettore GABAA , ipotizzando che le mutazioni epilettogene e le mutazioni prossimali, previste come patogene, possano produrre effetti simili sul modello di neurone piramidale CA1.
Abstract
Comprendere gli effetti di varianti funzionalmente sconosciute nei geni associati all'epilessia è fondamentale per chiarire la fisiopatologia della malattia e sviluppare terapie personalizzate. Con un quadro multiscala, che spazia dalla sequenza del DNA alla funzione proteica e al comportamento neurale, descriviamo un nuovo approccio per prevedere e studiare le mutazioni patogene, ipotizzando che le mutazioni epilettogene nella subunità del recettore GABAA e le mutazioni predette vicine possano produrre effetti simili sul modello di neurone piramidale CA1. Esplorando le relazioni caratteristiche tra mutazioni patogene previste e mutazioni epilettogene prossimali, lo studio mira a stimare gli effetti delle mutazioni predette sulla base degli effetti delle mutazioni epilettogene sulle simulazioni dei neuroni piramidali dell'ippocampo.
La metodologia inizia con la raccolta dei dati genetici della subunità γ2 del recettore GABAA , seguita dalla pulizia e dalla formattazione dei dati eseguita in R utilizzando uno script personalizzato. Successivamente, verranno applicati predittori d'insieme per identificare e dare priorità alle varianti missenso patogene della subunità γ2 . Verrà illustrata la mappatura di una specifica variante patogenetica (predicted) ai domini strutturali delle subunità condivisi dalle mutazioni epilettogene, accompagnata da modelli molecolari dei loro effetti e dalla considerazione della conservazione evolutiva. Successivamente, verrà eseguita la meta-analisi variant-specifica e la normalizzazione dei parametri, seguita dall'analisi di correlazione per identificare eventuali relazioni significative tra mutazioni previste e mutazioni epilettogene prossimali. Utilizzando un simulatore neurale basato su Python, verrà descritto un modello neuronale multicompartimentale basato sulla conduttanza, che riflette l'effetto dei mutanti wild-type ed epilettogeni. La simulazione delle risposte neurali generate dal sottotipo epilettogeno del recettore GABAA sarà presa in considerazione per la stima approssimativa dell'effetto delle varianti patogene previste sulla risposta neurale. Per quanto ne sappiamo, questo è il primo protocollo che esplora un framework multiscala per stimare gli effetti delle varianti del recettore GABAA sul comportamento neuronale, cruciale per la ricerca sull'epilessia. Questo protocollo può servire come base per migliorare le previsioni dei fenotipi cellulari causati da varianti potenzialmente patogene dei recettori GABAA associate all'epilessia.
Introduzione
Per quasi tutte le malattie umane, la variazione genetica gioca un ruolo significativo nella suscettibilità individuale. Pertanto, comprendere in che modo le variazioni di sequenza sono correlate al rischio di malattia offre un modo prezioso per scoprire i processi chiave coinvolti nello sviluppo della malattia e identificare nuovi approcci per la prevenzione e il trattamento1. Questo vale anche per i disturbi del neurosviluppo, che si collocano tra le condizioni mediche croniche più diffuse nelle cure primarie pediatriche2. Condizioni come il disturbo dello spettro autistico, la disabilità intellettiva e l'epilessia illustrano come la variazione genetica influenzi in modo significativo la suscettibilità individuale durante lo sviluppo3.
Il cervello in via di sviluppo è più suscettibile alle crisi epilettiche rispetto al cervello adulto a causa della mancata corrispondenza dello sviluppo neurologico geneticamente programmata nell'equilibrio critico tra eccitazione e inibizione4. Poiché il GABA (acido gamma-aminobutirrico), il principale neurotrasmettitore inibitorio nel cervello adulto, è eccitatorio durante lo sviluppo embrionale e postnatale precoce, ciò non è favorevole alla stabilità necessaria per prevenire le convulsioni nei cervelli giovani. Questo stato temporaneo, causato dalla mancanza di una sufficiente espressione dei co-trasportatori K-Cl5, può contribuire ad aumentare il rischio di attività convulsiva in presenza di recettori GABAA disfunzionali. I recettori GABAA mediano le azioni eccitatorie e inibitorie del GABA, a seconda della concentrazione intracellulare dello ione Cl- 6. Pertanto, man mano che il cervello matura, le mutazioni nei geni codificanti il recettore GABAA , così come in altri canali ionici, distorcono l'eccitabilità e le mutazioni nei geni coinvolti nel metabolismo neuronale, nella segnalazione cellulare e nella formazione delle sinapsi7, possono causare condizioni come l'epilessia infantile8.
Gli interventi clinici sfruttano sempre più l'analisi genetica per migliorare la precisione nel trattamento dei disturbi del neurosviluppo2. I test genetici nell'epilessia pediatrica presentano potenziali bersagli per gli approcci della medicina di precisione9, evidenziando l'importanza delle varianti genetiche nel guidare le decisioni terapeutiche. Inoltre, ~25% dei pazienti con epilessia con mutazioni de novo riceve diagnosi genetiche che identificano potenziali bersagli per la medicina di precisione, sottolineando il valore significativo delle varianti genetiche nel guidare le decisioni terapeutiche10. Ciò è stato alimentato dai progressi nelle tecnologie di sequenziamento di nuova generazione, come i pannelli genici mirati, il sequenziamento dell'intero esoma e il sequenziamento dell'intero genoma, che hanno notevolmente accelerato le scoperte genetiche11. Tuttavia, il numero crescente di nuove scoperte genetiche comporta una sfida quando i risultati producono una variante di significato sconosciuto (VUS), una classificazione che riflette prove contrastanti o informazioni insufficienti riguardo al ruolo molecolare della variante nella patogenesi della malattia. Le varianti classificate come VUS corrispondono a una categoria all'interno del sistema di classificazione delle varianti a cinque livelli proposto dall'American College of Medical Genetics and Genomics (ACMG) e dall'Association for Molecular Pathology (AMP)12.
Affrontare la sfida delle varianti genetiche funzionalmente sconosciute richiede sforzi in due dimensioni chiave: la pratica clinica e la ricerca. Clinicamente, l'incertezza che circonda la VUS può complicare la gestione del paziente e il processo decisionale13. Dal punto di vista della ricerca scientifica, è fondamentale identificare le varianti patogene tra il crescente numero di varianti di incerta significatività e determinare il loro ruolo nella fisiopatologia della malattia e negli effetti fenotipici1. Uno scenario ideale comporterebbe la previsione accurata degli effetti molecolari, neuronali e a livello di rete di tutte le varianti funzionalmente non caratterizzate, riducendo così al minimo le risorse, il tempo e lo sforzo necessari per le indagini di laboratorio. Questi aspetti sottolineano l'importanza di classificare accuratamente le varianti genetiche per consentire una diagnosi precisa delle epilessie genetiche, supportare un trattamento personalizzato e facilitare la scoperta di potenziali bersagli farmacologici. Gli attuali strumenti predittivi 14,15,16,17 sono relativamente accurati, ma in genere forniscono solo classificazioni binarie (patogeni vs. benigni) e mancano di informazioni specifiche sulla fisiopatologia molecolare, sulle conseguenze fenotipiche e sui meccanismi sottostanti. Concentrandosi sulle varianti missenso sconosciute di geni selezionati che codificano per le subunità del recettore GABAA, questo articolo presenta un quadro volto a migliorare le linee guida della ricerca incorporando fattori contestuali di varianti come aspetti molecolari, evolutivi e strutturali, nonché simulazioni di patologia neurale derivate da dati biofisici in vitro di mutazioni associate all'epilessia. La nostra metodologia affronta l'identificazione di varianti patogene sconosciute della subunità γ2 del recettore GABAA, una subunità chiave coinvolta nella fisiopatologia dell'epilessia 18,19,20. Segue l'esplorazione della corrispondenza posizione-specifica di queste varianti predette con le mutazioni associate all'epilessia caratterizzate da dati strutturali ed elettrofisiologici. Questi dati vengono poi utilizzati per stimare l'effetto variante su un modello di neurone piramidale ippocampale che esprime un sottotipo di recettore GABAA, composto da subunità γ2, α1 e β3 (recettori γ2-GABAA), responsabili dell'inibizione sinaptica veloce6. È importante notare che i recettori GABAA si assemblano da un ampio pool di subunità (α1-α6, β1-β3, γ1-γ3, δ, Ε, θ, π e ρ1-ρ3) e, a seconda della composizione delle subunità, i recettori GABAA differiscono per la loro modulazione, caratteristiche biofisiche, nonché modelli di espressione regionali, cellulari e subcellulari accoppiati con funzioni specifiche 6,21,22,23, 24,25. Pertanto, il presente studio si concentra solo sui recettori γ2-GABAA o sui recettori GABAA contenenti γ2.
Le subunità del recettore GABAA sono composte da caratteristiche strutturali: un lungo dominio extracellulare N-terminale (ECD), quattro domini transmembrana che si estendono (da TM1 a TM4), un linker intracellulare che collega TM1 e TM2, un linker extracellulare che collega TM2 e TM3, un grande loop intracellulare tra TM3 e TM4 (TM3-TM4 loop) e un breve terminale C extracellulare 6,26, 27. Si suggerisce che il recettore GABAA funzioni attraverso un complesso meccanismo di "blocco e trazione", in cui il legame GABA blocca le subunità β e α, inducendole a tirare i domini extracellulari (ECD) delle subunità, ruotandole in senso antiorario27. Questo movimento piega i domini transmembrana (TMD), aprendo così il canale ionico27. Pertanto, l'attività del canale sembra essere coordinata insieme alle cassette strutturali all'interno dei recettori GABAA. Si scopre che le mutazioni dell'epilessia causano disfunzione nell'attività del canale attraverso la distorsione di queste cassette strutturali28. Di conseguenza, il nostro studio si basa sull'idea che varianti patogene predette in prossimità di mutazioni epilettogene funzionalmente identificate nelle specifiche cassette strutturali delle subunità del recettore GABAA possano mostrare modelli simili di distorsione elettrofisiologica o biofisica nella funzione del canale, come osservato nei casi di queste mutazioni epilettogene. Mentre la presenza di cassette strutturali epilettogene nelle subunità28 del recettore GABAA supporta indirettamente questa nozione, il nostro studio dimostra la complessità e la sfida di correlare i parametri biofisici delle mutazioni epilettogene con quelli delle mutazioni patogene previste. Per smascherare queste complesse relazioni, il nostro quadro è significativo in quanto evidenzia un approccio multiscala che va dal DNA alla funzione delle proteine e al comportamento neurale critico per la ricerca sull'epilessia. Questo approccio integra la genetica computazionale con la modellazione molecolare e le simulazioni neurali, sottolineando anche l'importanza di metodi complementari, come l'apprendimento automatico addestrato su grandi set di dati, che potrebbero catturare gli effetti delle mutazioni sulla struttura del canale, sull'attività e sull'eccitabilità neurale. Inoltre, la simulazione dell'attività epilettogena del recettore γ2-GABAA sul modello di neurone piramidale ippocampale consente la replicazione del fenotipo cellulare in vitro associato alla canalopatia del recettore GABAA e la dimostrazione di risposte alterate a singolo neurone al centro della disfunzione di rete.
Protocollo
1. Predizione in silico di varianti patogene
- Raccolta dei dati sulle varianti
- Utilizzando il database ClinVar29, è possibile cercare varianti di significato incerto (VUS) nella regione codificante del gene di interesse tramite il sito web: https://www.ncbi.nlm.nih.gov/clinvar/. Inserisci il simbolo del gene (ad esempio, GABRG2) nella barra di ricerca e filtra i risultati per includere solo i tipi di varianti desiderati, come le varianti missenso a singolo nucleotide con significato incerto. Scaricare e salvare i dati come data.xlxs (File supplementare 4: Tabella supplementare S1). Registra la data dei dati scaricati.
NOTA: Nel presente protocollo, verrà analizzata la subunità γ2 umana del recettore GABAA, in particolare la subunità gamma2 del recettore dell'acido gamma-aminobutirrico A dell'acido gamma A (GABRG2), variante di trascrizione 1, mRNA (NCBI Ref. seq.: NM_198904.4), nota anche come γ2L. È importante registrare il trascritto di riferimento del gene di interesse e altri identificatori corrispondenti in diversi database (UniProt, ENSEMBL, PDB) poiché diversi metodi computazionali possono richiedere identificatori diversi (Supplementary File 4: Supplementary Table S2). Nel caso in cui il database o lo strumento computazionale non riconosca i numeri di versione degli identificatori di sequenza, provare sia l'ID con il numero di versione (NM_198904.4) che senza il numero di versione (NM_198904). - Informazioni di base sulla proteina di riferimento
- Nella https://www.ncbi.nlm.nih.gov/ del database NCBI, selezionare Nucleotide nelle opzioni di ricerca e inserire l'ID NCBI del gene di interesse (NM_198904.4). Quindi, scorrendo verso il basso sulla colonna di destra, fare clic su Proteina sotto la categoria Informazioni correlate per trovare la proteina (NP_944494.1) codificata dalla trascrizione NM_198904.4. Utilizzando le informazioni fornite per la proteina NP_944494.1, registrare le posizioni di sequenza delle regioni specifiche sotto forma di tabella (File supplementare 4: Tabella supplementare S3).
NOTA: È importante determinare le informazioni preliminari note per la posizione della sequenza di regioni, motivi o residui funzionalmente e strutturalmente critici come domini proteici, siti di fosforilazione, siti di legame del ligando e interfacce di interazione molecolare. Ciò può essere ottenuto combinando banche dati (NCBI, ENSEMBL, UniProt...) e ricerche bibliografiche.
- Nella https://www.ncbi.nlm.nih.gov/ del database NCBI, selezionare Nucleotide nelle opzioni di ricerca e inserire l'ID NCBI del gene di interesse (NM_198904.4). Quindi, scorrendo verso il basso sulla colonna di destra, fare clic su Proteina sotto la categoria Informazioni correlate per trovare la proteina (NP_944494.1) codificata dalla trascrizione NM_198904.4. Utilizzando le informazioni fornite per la proteina NP_944494.1, registrare le posizioni di sequenza delle regioni specifiche sotto forma di tabella (File supplementare 4: Tabella supplementare S3).
- Utilizzando il database ClinVar29, è possibile cercare varianti di significato incerto (VUS) nella regione codificante del gene di interesse tramite il sito web: https://www.ncbi.nlm.nih.gov/clinvar/. Inserisci il simbolo del gene (ad esempio, GABRG2) nella barra di ricerca e filtra i risultati per includere solo i tipi di varianti desiderati, come le varianti missenso a singolo nucleotide con significato incerto. Scaricare e salvare i dati come data.xlxs (File supplementare 4: Tabella supplementare S1). Registra la data dei dati scaricati.
- Organizzazione dei dati delle varianti
- Organizzare i dati in modo da soddisfare i requisiti di input per i predittori scelti. Assicurarsi che il formato dei dati recuperati sia organizzato in modo da soddisfare i requisiti del server dbNSFP http://database.liulab.science/dbNSFP. A tale scopo, rimuovere le colonne non necessarie dal file data.xlsx (File supplementare 4: Tabella supplementare S1 dal passaggio 1.1.1), mantenendo solo le colonne seguenti nell'ordine specificato:
"Cromosoma GRCh38", "Posizione GRCh38", "Nome", "Cambiamento proteico". - Salvare il file con un nuovo nome: "data1.xlsx" (Tabella supplementare S4). Formattare il file data1.xlsx in R eseguendo il codice (File supplementare 1: Data_GABAA. R), che salverà i dati formattati come data1_output.xlsx (File supplementare 4: Tabella supplementare S5) nella directory di lavoro relativa al progetto R.
NOTA: Diversi metodi computazionali richiedono diversi tipi e formati di dati. La raccolta e l'organizzazione dei dati in base a requisiti di formato specifici, anche per una dozzina di varianti, può essere soggetta a errori e richiedere molto tempo, quindi questo passaggio è importante a meno che il pool di varianti non sia composto solo da poche varianti. Quindi, potrebbe essere possibile l'organizzazione manuale dei dati.
- Organizzare i dati in modo da soddisfare i requisiti di input per i predittori scelti. Assicurarsi che il formato dei dati recuperati sia organizzato in modo da soddisfare i requisiti del server dbNSFP http://database.liulab.science/dbNSFP. A tale scopo, rimuovere le colonne non necessarie dal file data.xlsx (File supplementare 4: Tabella supplementare S1 dal passaggio 1.1.1), mantenendo solo le colonne seguenti nell'ordine specificato:
- Previsione della patogenicità
- Trasferisci il contenuto del file data1_output.xlsx nella versione accademica del server dbNSFP30,31 accessibile tramite http://database.liulab.science/dbNSFP. Per fare ciò, copia/incolla o carica direttamente il file in formato .txt.
- Assicurati che le seguenti opzioni siano preselezionate e confermate nel server: HG38 (build del genoma), ClinPred32 e BayesDEL33 prima dell'invio. Entro pochi minuti, il server genererà i risultati.
NOTA: Nel presente protocollo, due predittori d'insieme, vale a dire BayesDEL33 e ClinPred32, sono stati selezionati per l'elevata precisione34 e la praticità. Tuttavia, è possibile selezionare anche altri predittori, come AlphaMissense, disponibile nel database dbNSFP 30,31. La selezione di strumenti in silico dipende da diversi fattori, tra cui la generazione di sufficienti linee multiple di evidenza computazionale per una potente previsione12. I predittori d'insieme che integrano l'analisi di più algoritmi predittivi possono servire a questo scopo. - Scaricare il file di output (in formato .txt) e salvarlo come data2.xlsx (File supplementare 4: Tabella supplementare S6).
- Impostare i filtri in data2.xlsx (File supplementare 4: Tabella supplementare S6) facendo clic sull'opzione di filtro nel menu e determinando le varianti di consenso in entrambe le colonne filtrando per D. Questo darà l'elenco delle varianti più patogene; salvarlo (vedere la scheda Consenso nella Tabella Supplementare S6 [ File Supplementare 4]).
- Selezione delle varianti
- Tra le previsioni patogenetiche consensuali, determinare le varianti in prossimità di mutazioni epilettogene ottenute dalla letteratura. Assicurarsi che questi ultimi abbiano parametri strutturali e biofisici adatti alla modellazione dei neuroni.
NOTA: Questa fase è esplorativa ed è anche correlata all'indagine della proteina di interesse in termini di parametri strutturali, fisico-chimici e biofisici. Nel presente studio, questi dati sono stati ottenuti da Brünger et al.35 e Guo et al.36 in aggiunta a un'indagine sulle mutazioni associate all'epilessia. Inoltre, come opzione, i punteggi AlphaMissense37 sono stati accessibili dal database dbNSFP30,31 ripetendo il passaggio 1.3 (File supplementare 4: Tabella supplementare S7). Maggiori dettagli sono forniti nelle sezioni 2.1.1 e 2.1.2 del protocollo e nei risultati (vedere "Varianti di clustering per parametri strutturali e biofisici"). - Per la visualizzazione di base, utilizzare i server Protter38 ((https://wlab.ethz.ch/protter/start/) e HOPE39 (https://www3.cmbi.umcn.nl/hope/) per esaminare le varianti del passaggio precedente nel contesto di mutazioni genetiche GABRG2 selezionate: P302L40 e K328M (o K289M41, se si esclude il peptide segnale 39-residue).
NOTA: A causa dell'enorme complessità, la valutazione strutturale degli effetti delle varianti dovrebbe essere condotta a più livelli di analisi. Strumenti come Protter38 consentiranno una chiara visualizzazione delle varianti nel contesto delle caratteristiche topologiche della proteina e server di facile utilizzo come HOPE39 forniranno informazioni sull'effetto della variante mediante modellazione molecolare. Inoltre, una revisione completa della letteratura sulla proteina di interesse è fondamentale per identificare e integrare le informazioni sulle mutazioni associate all'epilessia. - Analisi della conservazione evolutiva e intuizioni strutturali
- Apri Jalview 42,43,44, un programma open source per l'editing, la visualizzazione e l'analisi delle proteine.
- Importare sequenze per l'allineamento. Clicca su File nel menu in alto | Recupera sequenze; selezionare il database nella finestra di dialogo (ad esempio UniProt); fare clic sulla scheda recupera ID; e come descritto nella finestra di dialogo, immettere gli ID di adesione UniProt del gene di interesse (GABRG2) dall'uomo e da altre specie vertebrate: P18507, P22723, Q6PW52, A0A2I3TKX0, F1RR72, A0A8I3MDZ2, A0A8M1P4D6. Fare clic su OK.
NOTA: I numeri di accesso UniProt delle proteine codificate da GABRG2 sono i seguenti: P18507 (P18507-2) per Homo sapiens, P22723 per Mus musculus, A0A2I3TKX0 per Pan troglodytes, F1RR72 per Sus scrofa, A0A8I3MDZ2 per Canis familiaris e A0A8M1P4D6 per Danio rerio. - A seconda del gene di interesse, alcune sequenze potrebbero non essere annotate; pertanto, eseguire una ricerca BLAST per identificare le informazioni rilevanti e i potenziali omologhi per una migliore comprensione contestuale. In questo caso, caricare il formato FASTA delle sequenze proteiche tramite l'opzione Aggiungi sequenze/Da casella di testo nel menu File per produrre allineamenti multipli delle sequenze desiderate.
- Una volta caricato l'allineamento, osservare le sequenze visualizzate per il confronto di più sequenze. Ogni riga rappresenta una sequenza e ogni colonna rappresenta una posizione nell'allineamento. Per determinare il miglior metodo di allineamento, utilizzare diversi approcci; ad esempio, fare clic su Web services nel menu Sequenza e selezionare l'opzione Esegui T-Coffee con preimpostazione , che consente un allineamento ottimale.
- Fare clic con il pulsante destro del mouse sulla sequenza P18507 Homo sapiens (la sequenza di riferimento nel presente studio) e impostarla come sequenza di riferimento. Scegli Formato nel menu in alto e fai clic su Avvolgi per visualizzare l'allineamento completo sullo schermo. Nello stesso menu Formato, fare clic sulla scala in alto per migliorare la visualizzazione dei numeri di residui specifici. Per migliorare ulteriormente la visualizzazione, regola le combinazioni di colori andando su Colore e selezionando diverse opzioni (ad esempio, Colore Clustal, Proprietà chimica); Se necessario, modificare la dimensione del carattere.
- Fare clic su Calcola nella barra dei menu e selezionare Calcola automaticamente consenso per evidenziare le regioni conservate.
- Concentrarsi sulla posizione delle varianti di interesse identificate nella fase di previsione in-silico ed esaminare le posizioni specifiche delle varianti. Annota residui specifici facendo clic con il pulsante destro del mouse su di essi e selezionando Aggiungi annotazione. Scrivi l'etichetta (ad es. ID variante) con il codice colore appropriato e salva.
NOTA: Nella presente analisi, P302L (viola) e A303T (rosso) sono stati selezionati per visualizzarli nell'allineamento a sequenza multipla insieme ai dati strutturali (vedere la sezione successiva).
- Ricostruzione tridimensionale dell'intera proteina con i residui conservati selezionati
- Nel file ottenuto dal passaggio precedente, fare clic con il pulsante destro del mouse sulla sequenza di riferimento (GABRG2 umano) e selezionare Dati struttura 3D.
- Identificare i dati strutturali appropriati (7QNE, Catena C)26 dal menu a discesa e selezionare Apri nuova vista struttura con Jmol.
NOTA: Ciò consentirà l'incorporazione dei residui selezionati nell'allineamento di sequenze multiple nei dati strutturali di Jmol, un visualizzatore open-source basato su Java per strutture chimiche 3D.
- Tra le previsioni patogenetiche consensuali, determinare le varianti in prossimità di mutazioni epilettogene ottenute dalla letteratura. Assicurarsi che questi ultimi abbiano parametri strutturali e biofisici adatti alla modellazione dei neuroni.
2. Selezione dei parametri e modellazione biofisica
- Meta-analisi variant-specifica e normalizzazione dei parametri
- Esaminare la letteratura attuale per raccogliere varianti di subunità identificate con conduttanza elettrofisiologica del canale dati (gGABAA), tempo di disattivazione (disattivazione τ), tempo di salita (aumentoτ) e ampiezza massima di corrente (Imax). Fornire le composizioni delle subunità, il tipo di cella e le misure wild-type per ogni caso. Etichettare le varianti e i relativi controlli di conseguenza (ad esempio, noti per le varianti con caratteristiche biofisiche identificate e controlli noti per le misurazioni wild-type per ciascuna variante).
- Ottieni i punteggi di patogenicità AlphaMissense per le varianti con caratteristiche biofisiche identificate.
NOTA: Per ulteriori dettagli, vedere la sezione 1.3 del protocollo. - Creare un frame di dati con la posizione delle subunità e degli amminoacidi per ogni variante, gli amminoacidi originali e alterati, il punteggio di patogenicità e i parametri biofisici ottenuti dalla letteratura. Per evitare discrepanze sperimentali, normalizzare i parametri biofisici per le varianti identificate come cambiamenti x-fold sulle misurazioni wild-type.
- Analisi comparativa delle varianti in base alle caratteristiche strutturali e funzionali
- Organizzare le varianti previste su un frame di dati; etichetta di conseguenza (ad esempio, prevista per varianti senza letteratura disponibile sulle loro caratteristiche biofisiche).
- Classificare le varianti in base alla loro posizione nella sequenza di amminoacidi e nella struttura terziaria. Aggiungere parametri di classificazione strutturale (ad esempio, localizzazione in alfa eliche, bobine, fogli beta, domini extracellulari, intracellulari o transmembrana, rivestimento dei pori, legame con agonisti, interazioni proteina-proteina) sul frame di dati e fornire informazioni per ciascuna variante rispetto alla loro posizione amminoacidica.
- Classificare le varianti in base alla loro distanza dal centro della membrana e dall'asse dei pori. Aggiungere la distanza all'asse dei pori e la distanza ai parametri del centro della membrana nel frame di dati.
- Analizzare la correlazione tra parametri strutturali e biofisici rispetto a varianti note. Se possibile, valutare le varianti previste rispetto alle correlazioni ottenute.
- Costruzione di modelli di sinapsi e neuroni
- Usa il Brian245, un simulatore neurale open-source sviluppato in Python per la modellazione e la simulazione di reti neurali spiking, per costruire un modello biofisico multicompartimentale della sinapsi GABAergica su un neurone piramidale ippocampale basato sulla conduttanza multicompartimentale.
- Progetta il modello basato sulla conduttanza definendo la cinetica di gating dei canali ionici, i parametri passivi e attivi e le conduttanze postsinaptiche. Definire il modello basato sulla conduttanza come indicato nel File supplementare 2, che descrive le equazioni utilizzate nel modello.
- Impostare la capacità di membrana (Cm) a 1 μF/cm2 e la resistenza intracellulare (Ra) a 200 Ω,cm.
- Utilizzare le conduttanze modificate di tipo Hodgkin-Huxley per i neuroni piramidali ippocampali39 con gL= 0,0003 S/cm2, gK= 0,036 S/cm2, EL = -76,5 mV, ENa = 50 mV e EK = -90 mV.
- Regolare la distribuzione della densità dei canali NaV su gNa come 0,05 S/cm2 per il soma, 0,5 S/cm2 per il segmento iniziale dell'assone (AIS) e il nodo di Ranvier (NR) e 0,005 S/cm2 per i dendriti. Impostare gK e gNa come 0 nei segmenti mielinizzati.
- Costruire la cinetica di gating dei canali ionici per NaV e KV come descritto nel File supplementare 2.
- Introdurre le correnti sinaptiche (Isyn) come somma di tutte le sinapsi glutammatergiche e GABAergiche in un compartimento. Includere sia la corrente mediata dal recettore AMPA VELOCE (IAMPA) che la corrente mediata dal recettore NMDA lento (INMDA) nella corrente glutammatergica (Iglu). Includere solo la corrente veloce mediata dal recettore GABAA nella corrente GABAergica (IGABA). Supponiamo che una quantità costante di glutammato venga rilasciata alla sinapsi per ogni picco presinaptico; pertanto, l'attivazione dei recettori è spike-tempo-dipendente (sAMPA e sNMDA) e le conduttanze totali dei recettori (gAMPA e gNMDA) riflettono la quantità di glutammato che viene rilasciata da ogni evento.
- Utilizzare il modello sinaptico come descritto nel File supplementare 2.
NOTA: Per una spiegazione dettagliata delle equazioni, vedere il File supplementare 2 che descrive le equazioni utilizzate nel modello.
- Ottenere il diametro misurato sperimentalmente per soma e neuriti e la lunghezza di ciascun compartimento neuritico e i modelli di ramificazione dalla letteratura precedente46,47. Ridurre la morfologia del neurone reale in un modello multicompartimentale, dividendo la cellula in più compartimenti, che preservi accuratamente la struttura ramificata principale e mantenga la simmetria bilaterale.
- Impostare le caratteristiche morfologiche (lunghezza e diametro del segmento; ad esempio, d_soma: 30 μm; l_AH: 5 μm; d_AH_i: 1,5 μm; d_AH_f: 1,3 μm; l_AIS: 40 μm; d_axon: 1 μm; l_myseg: 100 μm; l_NR: 2 μm; l_AxTer: 4 μm; d_AxTer: 2 μm; l_approx: 100 μm; l_apmed: 100 μm; l_apdis: 200 μm; d_approx_i: 4 μm; d_approx_f: 3 μm; d_apmed : 2 μm; d_apdis: 2 μm; l_apLM: 70 μm; d_apLM: 2 μm; l_nAcDbasal: 400 μm; d_nAcDbasal: 1,4 μm; l_nAcDbasal_stem: 20 μm; d_nAcDbasal_stem: 1,5 μm) e parametri biofisici (come indicato nella sezione 2.3.2) per ciascun compartimento del modello di neurone piramidale46,47, come descritto anche nello script Python (File supplementare 3: GABAAvar.py).
- Determinare i parametri biofisici per il modello di sinapsi GABAergica valutando le misure di controllo wild-type ottenute nel passaggio 2.1.1.
- Progettare la topologia del modello neuronale e assegnare i parametri morfologici e biofisici, che includono la specifica della disposizione spaziale e delle interconnessioni dei compartimenti, sulla base delle informazioni morfologiche e di ramificazione precedentemente ottenute. Assegnare i parametri morfologici (ad esempio, lunghezza e diametro del segmento) e biofisici appropriati (Sezione 2.3.2) a ciascun compartimento del modello, come indicato nel File supplementare 3: GABAAvar.py.
- Costruzione delle sinapsi e iniezione di corrente
- Creare l'attività presinaptica utilizzando SpikeGeneratorGroup (una classe della libreria Brian2) come indicato in "GABAAvar.py" (File supplementare 3). Collegare il generatore di spike al compartimento target del neurone modello utilizzando la classe Synapses per modellare le connessioni sinaptiche.
- Impostare una corrente costante sostenuta (Iinj) su 0,85 nA e posizionarla al soma per imitare l'attività sottosoglia guidata dal carico di corrente ionica basale in un dato momento, come indicato nel file supplementare 3: GABAAvar.py.
- Per creare monitor di registrazione, registrare le tracce di tensione dai compartimenti di destinazione utilizzando StateMonitor.
- Crea ed esegui la rete.
- Costruisci la rete con il neurone modello, le connessioni e i monitor utilizzando Network.
- Impostare il passo temporale della simulazione per impostazione predefinitaclock.dt (ad esempio, 0,01 ms).
- Esegui la simulazione sulla rete con network.run(T*ms), dove T è impostato su 1.000 ms nell'esempio.
- Testare l'impatto delle mutazioni missenso del recettore GABAA
- Definire l'impatto di ciascuna mutazione missenso sulla cinetica del canale attraverso i parametri biofisici raccolti nella fase 2.1.1.
- Esegui la stimolazione modificando questi parametri e traccia i risultati utilizzando "matplotlib.pyplot" come indicato in "GABAAvar.py" (File supplementare 3).
- Testare le combinazioni di parametri per analizzare i cambiamenti nei modelli e nelle velocità di sparo. Traccia i risultati per i confronti.
Risultati
Questo studio utilizza un approccio multiscala per prevedere e caratterizzare le varianti patogene nella subunità γ2 del recettore GABAA , un componente chiave nella fisiopatologia dell'epilessia. Attraverso l'uso di modelli predittivi, modellazione molecolare, conservazione evolutiva, esame strutturale, analisi di correlazione e simulazioni neurali, questo approccio migliora la classificazione delle varianti, con una rilevanza significativa per la ricerca sull'epilessia e possibilmente per l'uso clinico. La sintesi generale della metodologia è presentata nella Figura 1.
Valutazione comparativa di due mutazioni adiacenti della subunità γ2
Sulla base della nostra ipotesi che le mutazioni patogene previste adiacenti alle mutazioni epilettogene nelle subunità del recettore GABAA possano produrre effetti elettrofisiologici simili sulla funzione del canale e sul comportamento neurale, abbiamo prima condotto un breve esame della relazione tra una mutazione epilettogena ben nota e una mutazione predetta prossimale della subunità γ2.
Tra le varianti previste come patogene (Tabella supplementare S6), A303T (rs1581439874, ClinVar Accession: VCV000663033.6) è selezionato come esempio. Oltre alla predizione da parte di modelli d'insieme, la patogenicità di A303T è stata confermata dai punteggi AlphaMissense (File supplementare 4: Tabella supplementare S7). A303T si trova nel secondo dominio transmembrana della subunità γ2 del recettore GABAA e si trova accanto alla mutazione epilettogena P302L40, come mostrato nella Figura 2. Come valutato dalla modellazione molecolare, entrambe le sostituzioni γ2P302L e γ2A303T hanno portato ad amminoacidi che hanno catene laterali più grandi, come mostrato nella Figura 3. Sia il residuo mutante che quello wild-type sono apolari nella mutazione γ2P302L, mentre nella γ2A303T, il residuo mutante ha una catena laterale polare e il residuo wild-type ha una catena laterale non polare. Sia P302 che Ala303 si trovano nell'interfaccia di interazione della subunità con la subunità β3 (osservata rispettivamente in 7QNB e 7QNA). Sia P302 che Ala303 hanno un'area superficiale accessibile ai solventi (SASA) comparabile. Inoltre, entrambi i residui sono conservati al 100% durante l'arco dell'evoluzione dei vertebrati (Figura 4, pannello superiore). Entrambi sono localizzati in prossimità della seconda regione transmembrana (dominio TM2) della subunità γ2, come mostrato in giallo nella ricostruzione tridimensionale della proteina del recettore GABAA (7QNE26, dove A303, mostrato in rosso, è il primo residuo in questo dominio (Figura 4). Sulla base di queste caratteristiche comparabili e utilizzando un modello di neurone piramidale, la simulazione di mutazioni epilettogene prossimali come la mutazione della subunità γ2 P302L40 può essere utilizzata per la caratterizzazione preliminare dell'effetto della variante prevista (γ2A303T) sulla risposta neurale. Nella fase successiva, abbiamo esteso la nostra analisi a un insieme più ampio di varianti all'interno delle subunità del recettore GABAA .
Varianti di clustering per parametri strutturali e biofisici
A seguito della valutazione comparativa di due mutazioni adiacenti nella sezione precedente, abbiamo implementato un approccio sistematico per valutare se fosse possibile identificare caratteristiche molecolari condivise tra le varianti. Questa fase mirava a esplorare se emergono modelli coerenti nelle caratteristiche strutturali, fisico-chimiche e biofisiche tra gli amminoacidi e le varianti, fornendo così ulteriore supporto alla nostra ipotesi iniziale.
La cornice di dati utilizzata in questo studio e i riferimenti sono forniti nel file supplementare 4: Tabella supplementare S840, 48,49,50,51,52,53,54,55,56,57,58,59,60, 61,62,63, Tabella supplementareS936 e Tabella supplementareS1035. Inoltre, sono state determinate le correlazioni tra i parametri strutturali e biofisici per ciascuna subunità e per tutte le varianti senza distinzione di subunità (File supplementare 4: Tabella supplementare S11, Tabella supplementare S12, Tabella supplementare S13, Tabella supplementare S14 e Tabella supplementare S15). Le informazioni sui parametri strutturali (localizzazione su alfa eliche, bobine, fogli beta, domini extracellulari, intracellulari o transmembrana, rivestimento dei pori, legame agonista/allosterico e interazioni proteina-proteina) sono state ottenute da Brünger et al.35. I parametri biofisici sono stati ottenuti da studi di elettrofisiologia patch-clamp su cellule del rene embrionale umano (HEK) 293. I valori sono stati normalizzati con le rispettive costanti del tempo di attivazione del recettore wild-type (τr) e del tempo di disattivazione (τd).
Poiché il nostro studio si basa sull'idea che varianti di aminoacidi adiacenti o in prossimità di mutazioni funzionalmente identificate nelle subunità del recettore GABAA possano mostrare modelli simili di cambiamenti elettrofisiologici nella funzione del canale, come osservato nei casi di queste mutazioni, abbiamo esplorato la possibilità di una relazione tra parametri strutturali, fisico-chimici e biofisici. Le posizioni delle varianti rispetto alle loro distanze dal centro della membrana e dall'asse dei pori sono riportate nella Figura 5 e nella Figura 6. In questo contesto, abbiamo utilizzato anche i punteggi (Supplementary File 4: Supplementary Table S7) di AlphaMissense37; alimentato dal modello di previsione della struttura proteica altamente accurato AlphaFold264, che può utilizzare la sequenza di amminoacidi di base come input. AlphaMissense può fornire indizi per gli aspetti strutturali delle sostituzioni di singoli aminoacidi. La distribuzione dei punteggi AlphaMissense per le varianti note (nero) e previste (rosso) rispetto alla posizione della variante (posizione degli amminoacidi, distanza dal centro della membrana e distanza dall'asse dei pori) delle subunità del recettore GABAA (subunità α1, β3, γ2 codificate rispettivamente dai geni GABRA1, GABRB3, GABRG2 ) è riportata nella Figura 7.
La Figura 7A-C mostra la distribuzione del punteggio AlphaMissense tra le posizioni degli amminoacidi, la Figura 7D-F mostra la distribuzione del punteggio AlphaMissense sulla distanza normalizzata dall'asse dei pori e la Figura 7G-I mostra la distribuzione del punteggio AlphaMissense sulla distanza dal centro della membrana. L'analisi di correlazione nella Figura 7 ha indicato la difficoltà di accertare una relazione sottostante attraverso le proprietà strutturali per prevedere l'esito per le varianti appena identificate. Le varianti della subunità b2 (codificate dal gene GABRB2) sono state incluse nelle sezioni di clustering e correlazione per poter condurre un'analisi più ampia. Tuttavia, solo le varianti della subunità α1 codificata da GABRA1 (Figura 7A, D, G), della subunità β3 codificata da GABRB3 (Figura 7B, E, H) e della subunità γ2 codificata da GABRBG2 (Figura 7C, F, I) sono state incluse nei modelli biofisici, poiché il modello si concentra sulla funzione di un neurone piramidale ippocampale e sulla combinazione α1β3γ2 di GABAA subunità recettoriale è la combinazione più diffusa nell'ippocampo65. Allo stesso modo, sono state escluse dalle simulazioni anche tutte le varianti di α1, β3 o γ2 per le quali la cinetica del canale non è stata studiata in un recettore GABAA α1β3γ2. C'è stata una lieve correlazione (Figura supplementare S1 e Figura supplementare S2) tra i punteggi AlphaMissense e i parametri biofisici (tempi di attivazione e disattivazione normalizzati) derivati dagli effetti delle mutazioni del recettore GABAA (File supplementare 4 e Tabella supplementare S8) nella presente analisi. Ciò suggerisce che le mutazioni previste come patogene (sulla base dei punteggi AlphaMissense) potrebbero anche portare a cambiamenti misurabili e potenzialmente dirompenti nella cinetica del recettore (ad esempio, tempi di attivazione e disattivazione). Tuttavia, la mancanza di correlazione tra le correlazioni posizionali nella Figura 7 rende difficile utilizzare i punteggi AlphaMissense per la nostra ipotesi, che si basa sull'idea che gli amminoacidi adiacenti avranno conseguenze simili per le caratteristiche biofisiche.
Le distribuzioni della distanza normalizzata dall'asse dei pori rispetto alla cinetica di attivazione e disattivazione delle varianti note sono mostrate in Figura 8 e Figura 9. C'è una lieve correlazione per la subunità γ2 (Figura 8C), suggerendo la possibilità che la nostra ipotesi, che si basa sull'assunzione che gli amminoacidi adiacenti avranno conseguenze simili, possa essere vera in alcune regioni, in particolare in prossimità dell'area dei pori del canale recettoriale, il dominio TM2. Questa regione è adiacente alla nostra mutazione epilettogena di riferimento (Figura 2 e Figura 4; γ2P302), il che la rende un candidato relativamente buono per le simulazioni neurali. Sulla base di ciò, è possibile effettuare una stima approssimativa degli effetti delle mutazioni predette adiacenti come γ2A303T (Figura 2 e Figura 4). I nostri risultati qui presentati considerano solo le misure su α1β3γ2; pertanto, le varianti valutate nel nostro modello erano vincolate alle varianti fornite nel File supplementare 4: Tabella supplementare S16.
Effetto delle mutazioni sull'inibizione mediata dal recettore GABA A dell'attivazione dei neuroni piramidali CA1
L'effetto delle mutazioni sull'inibizione mediata dal recettore GABAA è dimostrato su un modello di neurone piramidale CA1 basato sulla conduttanza multicompartimentale. L'impatto delle varianti missenso del recettore GABAA sulla funzione del neurone piramidale dell'ippocampo può essere esplorato attraverso lo shunt GABAergico degli input apicali al neurone, dalle proiezioni dei neuroni piramidali CA3 e della corteccia entorinale (EC) III. In altre parole, un modo per simulare l'attività dei recettori GABAA è assumere un contesto in cui la simulazione rappresenta ipotesi realistiche sul significato fisiologico dei recettori, come l'inibizione dello shunt, uno dei meccanismi dell'inibizione GABAergica. I neuroni piramidali dell'ippocampo CA1, tipicamente nei loro dendriti apicali, hanno recettori GABAA in queste zone, che sono bersaglio delle proiezioni dei neuroni del CA3 e dell'EC III. Questa disposizione è quindi adatta per la simulazione. Questa domanda di ricerca richiede un progetto di input con ritardi e intensità variabili. Pertanto, tre diverse sinapsi glutammatergiche (GluS1/2/3) sono state posizionate sui dendriti apicali distali, apicali mediali e basali, come mostrato nella Figura 10, e sono state attivate in sequenza. Per valutare l'impatto degli input sinaptici, l'ampiezza della corrente costante dovrebbe rimanere al di sotto della soglia minima di innesco del picco (Iinj < Imin). Il modello di neurone piramidale con recettore GABAA wild-type o mutante è stato avviato con un'iniezione di corrente costante di 0,85 nA al soma. La sinapsi GABAergica è stata quindi posizionata al soma. L'attività presinaptica, imitata dal generatore di spike, è stata avviata prima al dendrite apicale distale. Gli input sinaptici sui dendriti apicali e basali mediali sono stati ritardati rispettivamente di 25 ms e 50 ms. La sinapsi GABAergica è stata attivata con un ritardo di 40 ms. L'intensità dell'inibizione GABAergica è stata regolata in modo tale che l'intero treno di spike, tranne il primo spike, sia inibito. Quindi, l'impatto delle varianti viene esplorato in questo contesto variandol'aumento di τ, ladisattivazione di τ e ilGABAA g .
I parametri per i recettori wild-type e mutanti sono stati ottenuti dalla collezione descritta nella fase 2.1.1 del protocollo specificamente per i recettori composti da α1β3γ2, che è la composizione di subunità più abbondante nei neuroni piramidali ippocampali65. La distribuzione dei parametri è data nel File Supplementare 4: Tabella Supplementare S16.
Ogni mutazione della subunità è stata testata su casi di sinapsi glutammatergica singola, doppia e tripla. In un approccio semplice, l'impatto delle mutazioni può essere valutato in base alla velocità e al modello di attivazione. Le medie ΔtISI e la deviazione standard possono anche essere stimate per valutare ulteriormente i cambiamenti nel modello di sparo, dove ΔtISI rappresenta la variazione dell'intervallo tra i picchi. I risultati per ciascun caso sono dati come tassi di attivazione e ΔtISI (media e deviazione standard) nel file supplementare 4: Tabella supplementare S17 e Figura supplementare S3. I treni di picchi e le tracce di tensione per le varianti che hanno alterato i modelli di accensione sono riportati in Figura 11, Figura 12 e Figura 13.
Per l'attivazione singola (GluS1) e tripla (GluS1-2-3) delle sinapsi glutammatergiche, le mutazioni che hanno alterato la risposta neuronale erano solo β3mutazioni N110D e γ2K328M. Nel singolo caso di input glutammatergico, β3N110D ha portato a un'inibizione compromessa e il modello di attivazione è stato bloccato sul treno di spike glutammatergico dopo il 4°picco presinaptico con un breve ritardo (Figura 11). γ2K328M ha anche compromesso l'inibizione, anche se solo intorno al 5° picco presinaptico, e ha introdotto un ritardo maggiore nel picco postsinaptico rispetto a β3N110D (Figura 11). Nel caso dell'attivazione di GluS1-2-3 (Figura 13), la risposta è stata simile tra le mutazioni β3N110D e γ2K328M. Entrambi i mutanti hanno prodotto un modello di attivazione in cui quasi tutti i picchi presinaptici cumulati sono stati rilevati e hanno innescato una risposta. In entrambi i casi, i modelli neuronali si sono attivati con una coppia di spike in risposta all'attività presinaptica.
La doppia attivazione delle sinapsi glutammatergiche ha prodotto risultati distinti rispetto alle altre due impostazioni (Figura 12). In questo caso, due mutazioni sulla subunità b3 del recettore GABAA (β3N110D e β3T288N) e due mutazioni sulla subunità γ2 del recettore GABAA (γ2P302L e γ2K328M) hanno compromesso l'inibizione GABAergica. Il modello neuronale con il mutante γ2P302L si è attivato quasi in sincronia con il GluS2, che era molto probabilmente una risposta ritardata al GluS1 con approssimativamente lo stesso ritardo dei picchi presinaptici tra GluS1-2. La mutazione β3T288N ha prodotto un risultato simile, con la distinzione del secondo spike ancora in sincronia con GluS2. Il modello neuronale con il mutante N110D β3ha risposto a quasi tutti gli input glutammatergici cumulativi, ad eccezione dei primi due picchi presinaptici di GluS1/2, che sono stati introdotti con un ΔtISI più corto. Il modello di attivazione per γ2K328M era ancora una volta simile a quello di β3N110D, con la distinzione del secondo e del terzo picco presinaptico che mancavano.
Questi risultati dimostrano i diversi effetti delle mutazioni della subunità b3 (codificata dal gene GABRB3) e della subunità γ2 (codificata da GABRG2) sull'attività dei neuroni piramidali dell'ippocampo. È interessante notare che le mutazioni β3L170R, β3A305V, β3E180G, β3D120N, β3Y302C e γ2R82Q non hanno prodotto alcun cambiamento nell'attività neurale. La compromissione più grave dell'inibizione è stata per β3N110D e γ2K328M, entrambi con unadisattivazione τ significativamente inferiore e unaumento τ più elevato. La nostra analisi preliminare ha anche mostrato che le variazionidell'aumento di τ o gGABA A da sole non sono sufficienti a compromettere l'inibizione (dati non mostrati). Si può sostenere che le mutazioni che portano a una significativa diminuzione delladisattivazione del τ insieme all'aumento delτ portano a una compromissione più significativa dell'inibizione GABAergica.
Nel caso in cui tutti gli input eccitatori debbano essere inibiti, qualsiasi mutazione che provochi l'attivazione produrrà risposte neurali anormali e aspecifiche, che hanno il potenziale per essere esagerate in un circuito neurale composto da neuroni con le stesse mutazioni. L'equilibrio tra eccitazione/inibizione in una rete neurale può essere influenzato in modo significativo dal conseguente feedback inibitorio alterato, che è una componente cruciale di qualsiasi attività di rete.
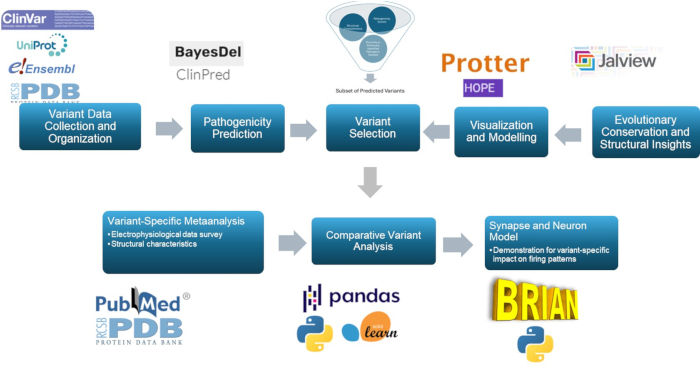
Figura 1: Panoramica della previsione e dell'analisi degli effetti delle varianti per scopi clinici e di ricerca, con particolare attenzione all'analisi in silico e alle simulazioni di risposta neurale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Posizione di γ2A303T e mutazioni selezionate dei pazienti;γ2P302L e γ2K328L utilizzati per le simulazioni neurali. Fare clic qui per visualizzare una versione più grande di questa figura.
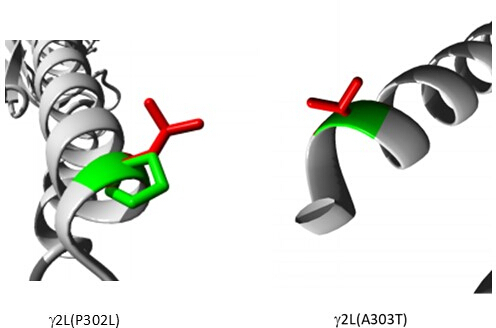
Figura 3: Modellazione comparativa della mutazione del paziente γ2P302L e della variante adiacente γ2L (A303T) predetta come patogena. In entrambi i modelli, il verde rappresenta il wild-type e il rosso i residui mutanti. Clicca qui per visualizzare una versione più grande di questa figura.
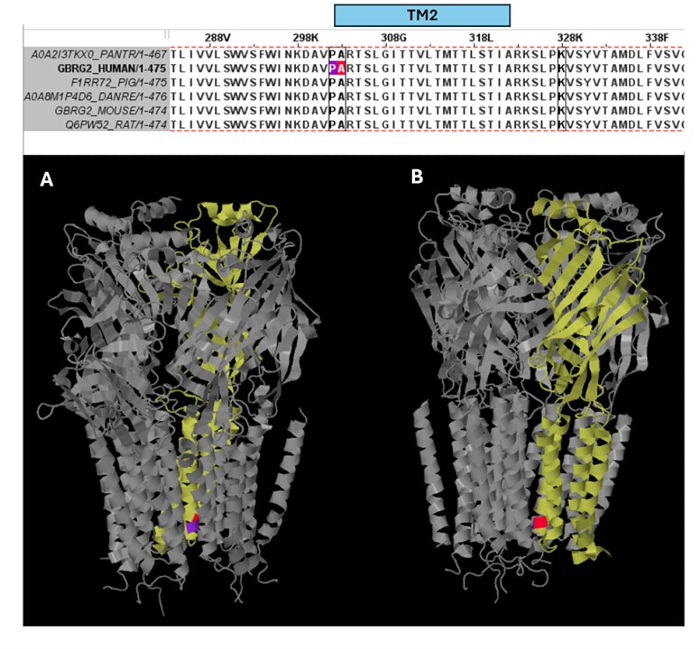
Figura 4: Allineamento di sequenze multiple e approfondimenti strutturali. Il pannello superiore mostra la conservazione evolutiva dei residui nella posizione della mutazione del paziente (P302L) (viola) al bordo del TM2 e della variante patogena A303T (colore rosso) all'inizio del TM2 della subunità γ2. Il pannello inferiore mostra la visualizzazione di questi residui conservati nella struttura tridimensionale del recettore GABAA (7QNE) (A), dove la subunità γ2 (Catena C nel 7QNE) è mostrata in giallo e da diverse angolazioni (A, B). Abbreviazione: TM2 = secondo dominio transmembrana. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Localizzazione di tutte le varianti incluse. Vengono mostrate le posizioni delle varianti note (nere) e previste (rosso) rispetto alla loro (A) distanza normalizzata dall'asse dei pori e (B) distanza dal centro della membrana. Clicca qui per visualizzare una versione più grande di questa figura.
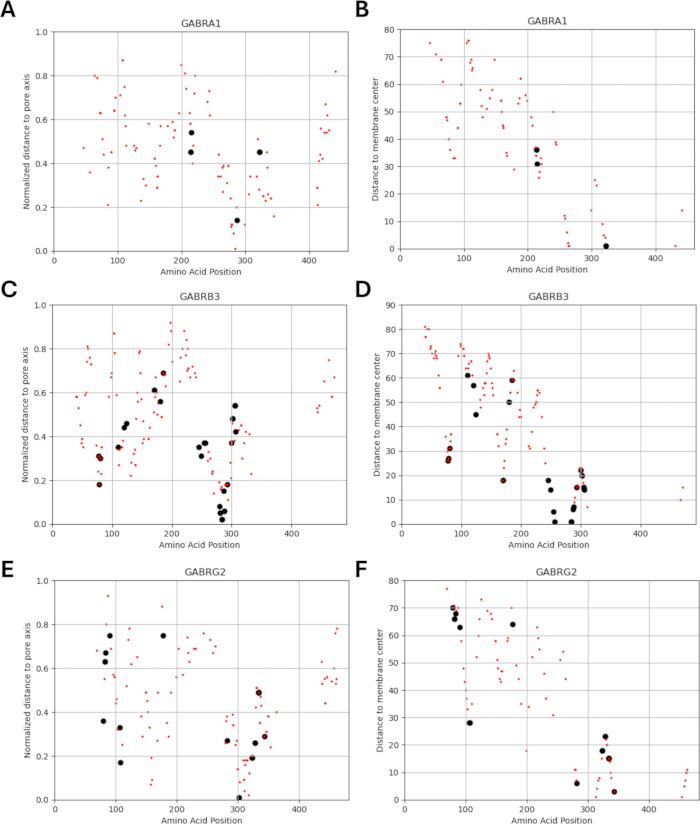
Figura 6: Localizzazione delle varianti per ogni subunità. Vengono mostrate le posizioni delle varianti note (nere) e previste (rosso) rispetto alla loro distanza normalizzata (A, C, E) dall'asse dei pori e alla distanza (B, D, F) dal centro della membrana. Clicca qui per visualizzare una versione più grande di questa figura.
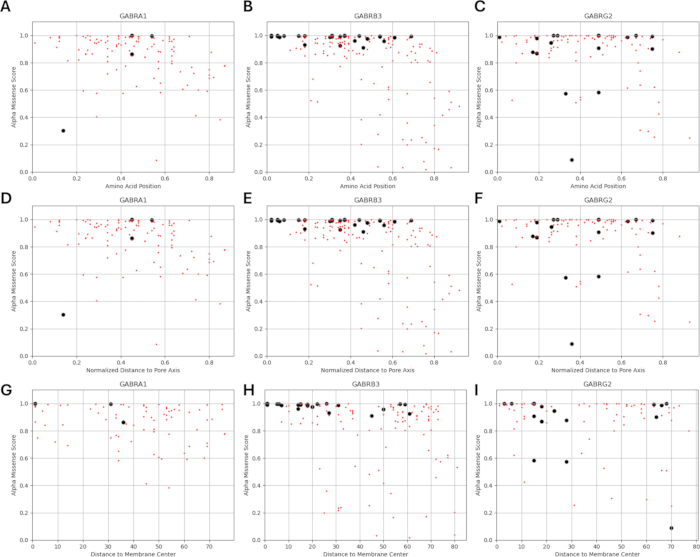
Figura 7: Distribuzione del punteggio AlphaMissense sulla posizione della variante. (A-C) La distribuzione del punteggio AlphaMissense sulla posizione degli amminoacidi, la distanza normalizzata (D-F) dall'asse dei pori e la distanza (G-I) dal centro della membrana sono fornite per le varianti note (nero) e previste (rosso). Clicca qui per visualizzare una versione più grande di questa figura.
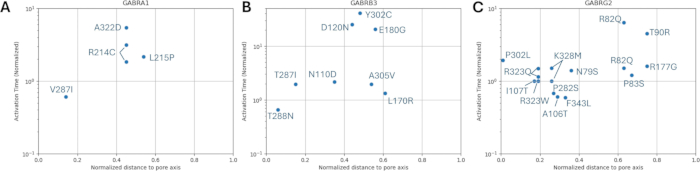
Figura 8: Tempo di attivazione normalizzato dei recettori GABAA con mutazioni nella subunità α1 (GABRA1), nella subunità β3 (GABRB3) e nella subunità γ2 (GABRG2).Vengono visualizzate le costanti di tempo di attivazione ottenute sperimentalmente rispetto alla distanza normalizzata dall'asse dei pori per ciascuna mutazione sulle subunità (A) α1, (B) β3 e (C) γ2. I valori sono stati normalizzati con il rispettivo tempo di attivazione del recettore wild-type. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 9: Tempo di disattivazione normalizzato dei recettori GABAA con mutazioni nella subunità α1 (GABRA1) del recettore GABAA, nella subunità β3 (GABRB3) del recettore GABAA e nella subunità γ2 (GABRG2).Vengono visualizzate le costanti di tempo di disattivazione ottenute sperimentalmente rispetto alla distanza normalizzata dall'asse dei pori per ciascuna mutazione sulle subunità (A) α1, (B) β3 e (C) γ2. I valori sono stati normalizzati con il rispettivo tempo di disattivazione del recettore wild-type. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 10: Modello di neurone piramidale CA1. Il neurone modello è costituito da (1) soma, (2) un dendrite apicale con compartimenti prossimali, mediali e distali, che termina con due rami alla lamina molecolare, (3) due dendriti basali composti simmetricamente che si ramificano in due sezioni dopo un corto gambo di dendrite basale e (4) un assone che inizia con una collinetta assonale conica, seguita dal segmento iniziale dell'assone cilindrico, segmenti mielinizzati e nodi di Ranvier, che terminano con un terminale sferico dell'assone. I triangoli verdi indicano la posizione delle sinapsi glutammatergiche e il triangolo rosso rappresenta la sinapsi GABAergica situata al soma. Barra della scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 11: Schema di cottura con solo attività di GluS1. I treni spike per i neuroni presinaptici (GluS1 (triangolo verde) e GABAergic (cerchio rosso)) e i neuroni postsinaptici con recettori GABAA wild-type o mutanti (quadrato nero) sono riportati nel pannello superiore. Nei pannelli inferiori sono visualizzate le tracce di voltaggio individuali per i neuroni con recettore GABAA wild-type con o senza inibizione GABAergica e per i neuroni con recettori GABAA mutanti con inibizione GABAergica. Clicca qui per visualizzare una versione più grande di questa figura.
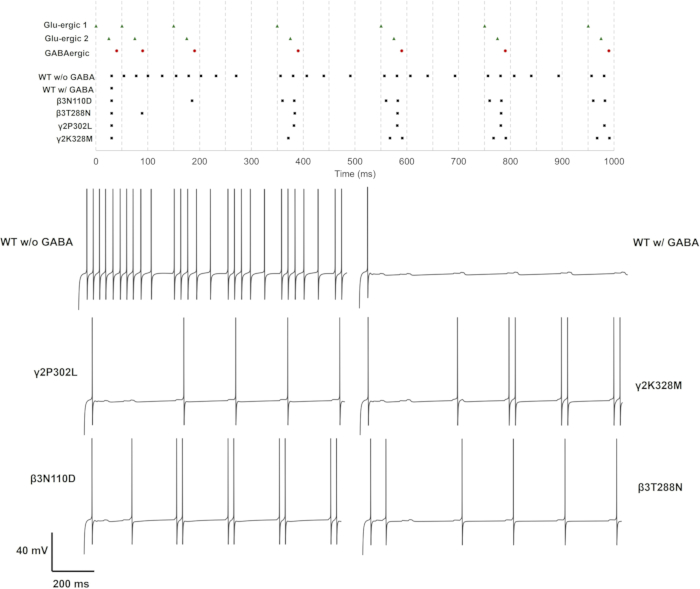
Figura 12: Schema di cottura con solo attività GluS1 e GluS2. I treni spike per i neuroni presinaptici (GluS1/2 (triangolo verde) e GABAergic (cerchio rosso)) e i neuroni postsinaptici con recettori GABAA wild-type o mutanti (quadrato nero) sono riportati nel pannello superiore. Nei pannelli inferiori sono visualizzate le tracce di voltaggio individuali per i neuroni con recettore GABAA wild-type con o senza inibizione GABAergica e per i neuroni con recettori GABAA mutanti con inibizione GABAergica. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 13: Schema di cottura con solo attività di GluS1, GluS2 e GluS3. I treni spike per i neuroni presinaptici (GluS1/2/3 (triangolo verde) e GABAergic (cerchio rosso) e i neuroni postsinaptici con recettori GABAA wild-type o mutanti (quadrato nero) sono riportati nel pannello superiore. Nei pannelli inferiori sono visualizzate le tracce di voltaggio individuali per i neuroni con recettore GABAA wild-type con o senza inibizione GABAergica e per i neuroni con recettori GABAA mutanti con inibizione GABAergica. Clicca qui per visualizzare una versione più grande di questa figura.
Figura supplementare S1: Distribuzione dei punteggi AlphaMissense e dei parametri biofisici (Tempo di disattivazione normalizzato; τd) normalizzato delle mutazioni della subunità del recettore GABAA selezionate nel presente studio. Vedi anche File supplementare 4: Tabella supplementare 8. Clicca qui per scaricare questa figura.
Figura supplementare S2: Distribuzione dei punteggi AlphaMissense e dei parametri biofisici (Tempo di attivazione normalizzato; τr) normalizzato delle mutazioni della subunità del recettore GABAA selezionate nel presente studio. Vedi anche File supplementare 4: Tabella supplementare 8. Clicca qui per scaricare questa figura.
Figura supplementare S3: Gli intervalli interspike per la risposta neurale con recettori GABAA wild-type e mutanti. Il grafico più in alto indica gli intervalli di tempo tra i picchi per un singolo input glutammatergico. Il grafico centrale ne mostra due, mentre il grafico più in basso mostra tre sinapsi glutammatergiche attive contemporaneamente. Clicca qui per scaricare questa figura.
File supplementare 1: Il file "Data_GABAA. R" necessario per l'esecuzione in R per la formattazione dei dati. Clicca qui per scaricare questo file.
File supplementare 2: Equazioni utilizzate nella progettazione del modello basato sulla conduttanza. Clicca qui per scaricare questo file.
File supplementare 3: GABAAvar.py necessario per l'esecuzione in Brian2 per la simulazione neurale. Il file contiene i codici Python per il modello di neurone multicompartimentale basato su Brian2 (funzione: CA1_Pyr), le equazioni per i modelli neuronali e sinaptici basati sulla conduttanza (funzione: model_eqns, syn_eqns) e i parametri iniziali (funzione: biophys_param, morpho_param, syn_param). Clicca qui per scaricare questo file.
File supplementare 4: una cartella zip contenente tutte le tabelle supplementari. Clicca qui per scaricare questo file.
Tabella supplementare S1: Varianti missenso di significato sconosciuto nel gene GABRG2 scaricate da ClinVar come file .txt e successivamente salvate come "data.xlxs". Clicca qui per scaricare questa tabella.
Tabella supplementare S2: Identificatori delle sequenze utilizzate nello studio, il trascritto di riferimento del gene di interesse (NCBI Ref. seq.) e altri identificatori corrispondenti in diversi database). Clicca qui per scaricare questa tabella.
Tabella supplementare S3: Posizioni delle regioni strutturali e funzionali. Le posizioni delle regioni specifiche della proteina della subunità γ2 (sequenza di riferimento NCBI: NP_944494.1) codificate dal trascritto di riferimento (NM_198904.4) Clicca qui per scaricare questa tabella.
Tabella supplementare S4: Il contenuto del file "data1.xlxs", che rappresenta i dati ClinVar di GABRG2 che include solo le colonne: "GRCh38Chromosome", "GRCh38Location", "Name", "Protein change". Clicca qui per scaricare questa tabella.
Tabella supplementare S5: Il contenuto del file "data1_output.xlsx" che contiene la formattazione richiesta delle varianti missenso dei dati GABRG2 da caricare sul server dbNSFP per la previsione dell'effetto variante. Clicca qui per scaricare questa tabella.
Tabella supplementare S6: Il contenuto del file "data2.xlsx" che contiene l'output dal server dbNSFP per la previsione dell'effetto variante per varianti missenso sconosciute di GABRG2. Clicca qui per scaricare questa tabella.
Tabella supplementare S7: Punteggi AlphaMissense per le varianti di subunità del recettore GABAA . Clicca qui per scaricare questa tabella.
Tabella supplementare S8: Caratteristiche biofisiche delle varianti. I valori per i parametri biofisici sono stati ottenuti da studi precedenti con esperimenti di elettrofisiologia. Le varianti sono etichettate con il tipo "S" (sostituzione), mentre i parametri del recettore wild-type sono forniti per ogni sostituzione ed etichettati con "C" (controllo). τd : Costante di tempo di disattivazione, POpen : Probabilità aperta, gGABA: Conduttanza del recettore, Imax: Corrente massima, τr : Costante di tempo di attivazione. Clicca qui per scaricare questa tabella.
Tabella supplementare S9: Caratteristiche fisico-chimiche delle varianti. Le varianti precedentemente identificate con parametri biofisici sono etichettate con il tipo "S" (sostituzione), mentre le varianti previste sono rappresentate con "P". H: Variazione dell'idrofobicità, VSC: Variazione del volume della catena laterale, P1: Variazione della polarità, P2: Variazione della polarizzazione, SASA: Variazione della superficie accessibile al solvente, NCISC: Variazione dell'indice di carica netta. I valori sono ottenuti per ogni amminoacido originale e variante da Guo et al.36 e la variazione di ciascun parametro è stimata come dato. Clicca qui per scaricare questa tabella.
Tabella supplementare S10: Caratteristiche strutturali delle varianti. Le varianti precedentemente identificate con parametri biofisici sono etichettate con il tipo "S" (sostituzione), mentre le varianti previste sono rappresentate con "P". La localizzazione della variante in un dominio è rappresentata con 1, altrimenti 0. Tutti i valori sono ottenuti da Brünger et al.35. Clicca qui per scaricare questa tabella.
Tabella supplementare S11: Correlazioni dei parametri strutturali e biofisici per tutte le varianti note. Clicca qui per scaricare questa tabella.
Tabella supplementare S12: Correlazioni tra parametri strutturali, fisico-chimici e biofisici per varianti note di GABRA1 . Clicca qui per scaricare questa tabella.
Tabella supplementare S13: Correlazioni dei parametri strutturali, fisico-chimici e biofisici per varianti note di GABRB2 . Clicca qui per scaricare questa tabella.
Tabella supplementare S14: Correlazioni dei parametri strutturali, fisico-chimici e biofisici per varianti note di GABRB3 . Clicca qui per scaricare questa tabella.
Tabella supplementare S15: Correlazioni dei parametri strutturali, fisico-chimici e biofisici per varianti note di GABRG2 . Clicca qui per scaricare questa tabella.
Tabella supplementare S16: Parametri biofisici per i recettori GABA A wild-type e mutanti α1β3γ2. Clicca qui per scaricare questa tabella.
Tabella supplementare S17: Velocità di attivazione e intervalli interspike in risposta a sinapsi glutammatergiche singole, doppie o triple con recettori GABAA wild-type e mutanti. Clicca qui per scaricare questa tabella.
Discussione
Applicando una combinazione di genetica computazionale, modellazione molecolare e simulazioni neurali, l'approccio presentato in questo articolo ha il potenziale per migliorare la classificazione delle varianti del recettore GABAA, offrendo preziose informazioni sia per la ricerca sull'epilessia che per le applicazioni cliniche. Un'analisi completa per l'identificazione e la prioritizzazione delle mutazioni patogene previste viene presentata ed estesa in un quadro che potenzialmente colma il divario tra gli effetti delle varianti sulla proteina e il fenotipo cellulare. La valutazione dell'impatto dell'attività epilettogena del recettore GABAA sulla simulazione dei neuroni piramidali dell'ippocampo consente la replicazione del fenotipo in vitro associato alla disfunzione del recettore GABAA e la dimostrazione dell'alterazione della risposta di un singolo neurone alla radice della disfunzione della rete. Sulla base di queste simulazioni di risposte neurali generate da mutazioni epilettogene, è stata esplorata una stima approssimativa degli effetti funzionali delle mutazioni predette strutturalmente prossimali. Le previsioni sull'effetto delle mutazioni previste sulla cinetica del canale richiedono un'analisi approfondita con set di varianti noti. Le analisi comparative, come quelle presentate in questo articolo, e le simulazioni dell'attività neurale forniscono informazioni critiche per l'ulteriore generazione e miglioramento di modelli predittivi incentrati sull'effetto delle varianti sulla funzione della proteina e sulla patologia neurale. Inoltre, la nostra metodologia può essere utilizzata per selezionare e dare priorità alle varianti più patogene tra le varianti sconosciute per esaminare gli effetti delle varianti legate ai disturbi dello sviluppo neurologico correlati al recettore GABAA. Ad esempio, le subunità recettoriali marcate con sonde fluorescenti 66,67,68,69,70 e portatrici delle mutazioni previste possono essere espresse in vitro per studiarne il traffico, l'espressione della superficie cellulare e la neurofisiologia. Inoltre, modelli animali come C. elegans possono essere considerati per convalidare gli effetti delle mutazioni previste. Ad esempio, l'editing genetico CRISPR-Cas9 è stato utilizzato per generare una delezione di unc-49, un recettore GABAA di C. elegans, generando così mutazioni omozigoti associate all'epilessia in unc-49 o subunità del recettore GABAA umano71.
In generale, la classificazione delle varianti beneficia dell'uso di più livelli di evidenza computazionale, come raccomandato da ACMG-AMP12. Questo approccio rafforza l'affidabilità della classificazione delle varianti integrando diversi strumenti predittivi e fonti di dati, migliorando in ultima analisi l'accuratezza delle valutazioni cliniche e migliorando il processo decisionale complessivo nella diagnostica genomica. Nella nostra metodologia, l'utilizzo di predittori d'insieme, che combinano le previsioni di più strumenti, soddisfacendo così il requisito di più linee di evidenza computazionale ed eliminando la necessità di utilizzare strumenti diversi separatamente, è un vantaggio. Questo approccio supera anche la sfida di gestire output disparati dai singoli strumenti, semplificando così il processo di previsione e migliorando l'efficienza. Tuttavia, non vi è alcuna garanzia per quanto riguarda l'accuratezza predittiva delle analisi gene-centriche o specifiche delle varianti. Ciò porta alla conclusione che le previsioni gene-centriche o specifiche per le varianti dovrebbero essere eseguite in condizioni specifiche aggiustate per i contesti e gli obiettivi specifici 15,72,73,74. Per gli interventi clinici, ciò richiederebbe la valutazione dell'accuratezza predittiva degli strumenti in silico per un gene specifico o un sottoinsieme di geni nel contesto di una determinata malattia, spesso con ottimizzazione individualizzata75. Tuttavia, la valutazione dell'accuratezza predittiva è spesso limitata dalla mancanza di un numero sufficiente di varianti, che può influire sull'affidabilità della valutazione dell'accuratezza.
In letteratura sono disponibili diversi strumenti e la loro accuratezza è testata e convalidata nei dataset14. Tuttavia, questi risultati di accuratezza basati su grandi set di dati non si riflettono necessariamente sulla previsione di alcune varianti sconosciute per un determinato gene. In questo contesto, l'accumulazione della letteratura suggerisce che i predittori d'insieme, che compilano e calcolano i risultati dei singoli predittori, sono noti per ottenere prestazioni migliori rispetto alla concordanza dei singoli predittori 33,76,77,78 e quindi, nel presente studio, abbiamo scelto di utilizzare i predittori d'insieme, vale a dire BayesDEL 33 e ClinPred32 specificamente per le loro prestazioni superiori 32, 34 BayesDEL è stato valutato comparativamente per 4.094 varianti missenso in geni clinicamente rilevanti, inclusi geni che codificano per proteine transmembrana come la subunità alfa del canale del sodio voltaggio-dipendente 5 (SCN5A), e ha mostrato prestazioni superiori33. Nel nostro protocollo di previsione dell'effetto variante, come primo passo, abbiamo considerato il consenso di due predittori d'insieme (BayesDEL e ClinPred). AlphaMissense37, un modello di deep learning sviluppato da Google DeepMind, è un'estensione di AlphaFold 64,79, utilizzando così la potenza della previsione della struttura proteica ad alta precisione. Quando abbiamo confrontato i risultati predittivi iniziali dei modelli d'insieme (BayesDEL e ClinPred come descritto nella fase 1.3 del nostro protocollo) con i risultati di AlphaMissense, le previsioni erano parzialmente in accordo tra loro (File supplementare 4: Tabella supplementare S15) e non si allineavano completamente con le previsioni dei modelli d'insieme (BayesDEL e ClinPred), che hanno raggiunto un consenso di patogenicità o malattia associata, mostrate come righe rosa (File supplementare 4: Tabella supplementare S15). Tuttavia, le varianti sconosciute (L81F, A303T e V329F) vicine alle mutazioni GABRG2 R82Q, P302L e K328M, che abbiamo utilizzato nel nostro modello neuronale e previste come patogene sia da ClinPred che da BayesDEL, sono state anche predette come patogene da AlphaMissense, come mostrato dalle evidenziazioni gialle (File supplementare 4: Tabella supplementare S15).
Poiché AlphaMissense29 utilizza la previsione della sequenza e del contesto strutturale, nel nostro studio volevamo anche vedere se c'era qualche associazione tra i punteggi di AlphaMissense e le posizioni delle mutazioni del recettore GABAA in base alle loro distanze dal centro della membrana e dall'asse dei pori. La nostra ipotesi si basa sull'idea che l'impatto funzionale delle varianti di aminoacidi adiacenti o prossimali alle mutazioni funzionalmente identificate delle subunità del recettore GABAA possa mostrare modelli simili di cambiamenti fisico-chimici nella funzione del canale osservati nei casi di mutazioni. Una correlazione tra le posizioni di mutazione della subunità del recettore GABAA e i punteggi di AlphaMissense ci aiuterebbe a identificare una relazione utilizzabile per costruire un quadro per la nostra ipotesi che consenta la previsione delle conseguenze funzionali di nuove varianti missenso nelle subunità del recettore GABAA . Tuttavia, i punteggi AlphaMissense non erano predittivi di cambiamenti in questi parametri biofisici (Figura 7). È importante notare che la dimensione limitata del campione nella nostra analisi rende difficile trarre conclusioni definitive. Tuttavia, la nostra analisi ha rilevato che i punteggi AlphaMissense non erano correlati con i parametri strutturali dei recettori GABAA . La mancanza di una chiara correlazione posizionale (ad esempio, tra le posizioni delle mutazioni e i punteggi di AlphaMissense) mette in discussione la validità della nostra ipotesi. Se i residui adiacenti avessero davvero effetti simili, ci aspetteremmo di vedere una correlazione più chiara. Poiché questo non è il caso, indebolisce la capacità di utilizzare i punteggi AlphaMissense come strumento affidabile per testare la nostra ipotesi.
È interessante notare che, nel nostro studio, abbiamo trovato una lieve correlazione tra la distanza tra la variante e l'asse dei pori e il tempo di attivazione del canale normalizzato per i mutanti del gene GABRG2 . Pertanto, la nostra ipotesi preliminare che gli amminoacidi adiacenti avranno conseguenze simili può essere vera in alcune regioni del canale, come le regioni del poro o nei siti chiave coinvolti nel gating, ma potrebbe non essere così chiara in altre regioni. Il piccolo set di dati limita la capacità di discernere questa variabilità, ma dati futuri o analisi strutturali più dettagliate potrebbero aiutare a perfezionare questo aspetto della nostra ipotesi. Le simulazioni di dinamica molecolare80 potrebbero servire come un potente approccio complementare per indagare ulteriormente questi risultati preliminari, specialmente nel contesto della valutazione comparativa di due mutazioni adiacenti della subunità γ2, vale a dire la mutazione epilettogena γ2P302L40 e la mutazione prossimale predetta γ2A303T (rs1581439874), eseguite nel nostro studio. In futuro, questo approccio potrebbe consentire una stima più accurata dell'effetto di una variante sconosciuta sul fenotipo cellulare, soprattutto se integrato con le simulazioni neurali presentate nel nostro studio.
Inoltre, sarà interessante esplorare se le proprietà strutturali e fisico-chimiche delle subunità del recettore GABAA , insieme ad altre caratteristiche, possono essere utilizzate per addestrare potenti modelli di apprendimento automatico per la previsione funzionale di nuovi effetti di varianti sul canale, sul neurone, sulla rete e sul fenotipo della malattia. Con l'avvento degli approcci automatizzati di apprendimento automatico, abbiamo raggiunto un punto in cui anche i medici e gli scienziati di laboratorio possono sviluppare i propri modelli in un ambiente più democratizzato81. Pertanto, l'integrazione di queste tecnologie nella pratica clinica potrebbe potenzialmente semplificare il processo, rendendo la medicina personalizzata più accessibile e riducendo la dipendenza da competenze altamente specializzate per l'analisi delle varianti funzionali. In questo contesto, il nostro approccio fornisce informazioni sulle dinamiche strutturali e funzionali del recettore, potenzialmente aiutando in studi futuri per la previsione funzionale dell'effetto variante.
Nonostante gli attuali progressi nella predizione della struttura proteica e la svolta rappresentata da AlphaFold64, la previsione accurata dell'effetto delle mutazioni e della funzione proteica rimane una sfida a causa della mancanza di dati necessari per addestrare il modello79. Per la previsione dell'effetto variante, AlphaMissense mostra prestazioni più elevate rispetto a un sottoinsieme di modelli predittivi, ma i predittori d'insieme BayesDEL25 e ClinPred24, che sono stati utilizzati nel nostro studio, non sono stati inclusi in questo confronto29. È importante notare che, nel nostro studio, gli strumenti in silico BayesDEL, ClinPred e AlphaMissense sono stati impiegati per scopi distinti. I predittori dell'ensemble, BayesDEL e ClinPred, sono stati utilizzati principalmente per la previsione della patogenicità, mentre AlphaMissense è stato utilizzato specificamente per esplorare la relazione tra i suoi punteggi e i dati noti per l'impatto delle mutazioni nella subunità γ2. In particolare, la nostra ipotesi assume che le varianti patogene predette, in particolare quelle situate vicino o adiacenti a mutazioni funzionalmente identificate nelle subunità del recettore GABAA , mostreranno parametri biofisici simili a quelli osservati nelle mutazioni funzionalmente caratterizzate. Per indagare su questo, abbiamo scelto AlphaMissense perché è alimentato dal modello altamente accurato AlphaFold264 , che utilizza la sequenza peptidica di base per prevedere le conseguenze delle sostituzioni di singoli aminoacidi.
Di conseguenza, una delle principali limitazioni del nostro studio è principalmente guidata dalla limitata disponibilità di dati sperimentali. Ad esempio, il nostro modello neuronale si basa sull'espressione dei dati derivati dalla combinazione di subunità α1β3γ2 dei recettori GABAA , che limita intrinsecamente le mutazioni studiate in letteratura alle subunità espresse come parte di questa specifica combinazione di recettori. Inoltre, ci siamo basati su dati elettrofisiologici derivati esclusivamente dall'espressione di queste subunità nelle cellule HEK, restringendo ulteriormente la portata dei dati disponibili in letteratura. Il nostro uso della modellazione neurale per stimare gli effetti di varianti sconosciute presuppone che le varianti sconosciute (previste come patogene nel nostro flusso di lavoro) situate in prossimità di mutazioni note mostreranno modelli simili nei parametri cinetici del canale o nelle proprietà fisico-chimiche degli effetti di mutazione descritti in letteratura. Questa ipotesi, unita alla necessità di dati elettrofisiologici per specifici assemblaggi di recettori nelle cellule HEK293, riduce la quantità di dati sperimentali disponibili per la modellazione. Come risultato di questi vincoli, i dati disponibili ci hanno permesso di modellare solo un numero limitato di varianti in subunità α1β3γ2. Tuttavia, l'addestramento del modello neuronale per diversi assemblaggi di subunità come le combinazioni di subunità α1β2γ2, le combinazioni di subunità α1β2δ o α4β3δ, che hanno implicazioni a livello cellulare, circuitale e di rete specifiche per le subunità, mostrerà probabilmente un'applicabilità più ampia a vari tipi di epilessia e disturbi dello sviluppo neurologico. In futuro, con l'aumento dei dati elettrofisiologici disponibili e degli studi incentrati sulle mutazioni in assemblaggi recettoriali ben definiti e tipi cellulari specifici, potrebbe diventare possibile migliorare la generalizzabilità e l'accuratezza del nostro approccio.
I modelli di neuroni basati sulla conduttanza multicompartimentale forniscono un potente strumento per generare previsioni sul significato funzionale delle varianti recettoriali della risposta a singolo neurone. Questo strumento consente definizioni flessibili dei parametri cellulari/sinaptici e della loro posizione per testare qualsiasi domanda specifica. I semplici generatori di spike utilizzati in questo protocollo possono essere sostituiti con altri modelli di neuroni per studiare l'attività dei microcircuiti. La fase critica del protocollo è anche la più limitante: la definizione di qualsiasi variante recettoriale in termini di cinetica alterata del canale. Le informazioni richieste sono idealmente fornite da studi di elettrofisiologia patch-clamp; Tuttavia, l'analisi computazionale delle sostituzioni di aminoacidi con significato clinico previsto e i confronti con sostituzioni note possono anche fornire alcune informazioni. Il nostro studio e il protocollo descritto incorporano l'uso di simulazioni di attività neurale non come strumento predittivo, ma piuttosto come strumento per esplorare gli effetti delle mutazioni per supportare una visione più ampia delle conseguenze delle caratteristiche biofisiche alterate del recettore GABAA sull'attività di un singolo neurone. La dipendenza dai dati sperimentali nelle simulazioni neurali è un limite importante nel nostro approccio, che potrebbe trarre vantaggio dalla modellazione molecolare avanzata per colmare il divario tra struttura e funzione.
Nel nostro protocollo, alcuni passaggi dovrebbero essere valutati criticamente. In primo luogo, la scelta del modello predittivo utilizzato nella prima parte del nostro protocollo può essere fondamentale. La selezione di strumenti in silico dipende da diversi fattori, tra cui la generazione di sufficienti linee multiple di evidenza computazionale per una potente previsione12. I predittori d'insieme che integrano l'analisi di più algoritmi predittivi si adattano meglio a questa raccomandazione, quindi preferiti rispetto ai predittori individuali. Esistono diversi predittori e la loro accuratezza viene solitamente testata in grandi set di dati, il che non garantisce necessariamente l'accuratezza del modello predittivo utilizzato per effetti di varianti sconosciute localizzate in un gene specifico. Ciò viene compensato utilizzando due modelli di ensemble, che raccolgono e calcolano i risultati della previsione da più predittori. Inoltre, i cutoff dei modelli predittivi possono essere regolati per aumentare la specificità se lo scopo è principalmente quello di identificare le varianti più patogene. L'impostazione di cutoff appropriati è importante per bilanciare il compromesso tra sensibilità e specificità e garantire una classificazione accurata delle varianti. Nel nostro studio, abbiamo utilizzato i cutoff predefiniti. In particolare, non abbiamo modificato il cutoff a favore dell'acquisizione di varianti che potrebbero avere maggiori probabilità di essere patogene, poiché ciò ridurrebbe l'ambito delle varianti da esaminare in più livelli della nostra analisi, come descritto nel nostro flusso di lavoro. È anche importante notare che nella scelta dei dati strutturali per la ricostruzione tridimensionale della proteina di interesse, è necessaria la revisione preliminare dei dati strutturali in letteratura. L'esame strutturale dei recettori GABAA ha recentemente guadagnato slancio con robusti studi strutturali che riportano la struttura tridimensionale di diversi gruppi di recettori in diverse condizioni 26,27,82,83,84,85. Data la disponibilità di questi dati, nel nostro studio, ci siamo concentrati sulle strutture determinate sperimentalmente per la ricostruzione dei dati strutturali. Tuttavia, la previsione di AlphaFold64 può essere favorita per l'analisi di altre proteine che mancano di tali dati determinati sperimentalmente. Per i dati strutturali derivati da studi sperimentali, è importante prestare attenzione alla numerazione degli amminoacidi. La numerazione PDB degli amminoacidi può essere diversa dalla numerazione UniProt poiché la prima potrebbe non includere il peptide segnale che viene rimosso durante la maturazione delle proteine. Inoltre, le proteine chimeriche espresse nel sistema sperimentale possono causare discrepanze. In questo caso, l'allineamento a coppie della sequenza di interesse con la sequenza derivata dai dati strutturali aiuterà a preservare la coerenza. In letteratura, i dati strutturali per la proteina della subunità γ2 si basano su diversi metodi, inclusi metodi sperimentali come la microscopia elettronica (EM) e il metodo di previsione ad alta accuratezza di AlphaFold. Se il metodo sperimentale non copre completamente la sequenza desiderata ad alta risoluzione, possono essere utilizzate le previsioni di AlphaFold. Nel presente studio, la struttura 7QNE26 è stata scelta poiché corrisponde alla struttura criogenica al microscopio elettronico del recettore GABAA sinaptico umano a lunghezza intera α1β3γ2. Questo rappresenta esattamente la combinazione di subunità a lunghezza intera, che è stata al centro del presente studio.
Inoltre, per l'analisi comparativa, dovrebbe essere preferito l'uso di parametri normalizzati della cinetica del canale, poiché i valori di questi parametri potrebbero variare a seconda della composizione della subunità del recettore e delle impostazioni sperimentali. Ad esempio,l'aumento e ladisattivazione di τ dovrebbero essere determinati su variazioni di x volte rispetto ai valori di controllo wild-type. Nella fase 2.5 del protocollo, per un numero ridotto di varianti note, potrebbe essere preferibile l'analisi di correlazione parametrica o categoriale e la determinazione dei valori rho e p. Idealmente, ci si aspetta che metodi come l'analisi delle componenti principali producano relazioni più accurate, ma richiedano un numero maggiore di campioni.
L'ambiente di simulazione può essere modificato. In questo studio, Brian2 è stato preferito per i seguenti motivi: la classe di neurone spaziale in Brian2 fornisce uno strumento prezioso per simulare l'attività neurale. Brian2 ha un vantaggio significativo nell'uso di equazioni differenziali per descrivere la dinamica continua e aggiornare le istruzioni per eventi discreti invece di fare affidamento su modelli "black-box" predefiniti, e questo porta a un'eccellente leggibilità e adattabilità del codice poiché ogni aspetto del modello può essere definito esplicitamente in un singolo script Python, con le equazioni del modello dichiarate in notazione matematica e solo una piccola quantità di vocabolario specifico di Brian utilizzato37. Poiché il modello è descritto in modo esplicito, tutte le caratteristiche sono documentate e possono essere trovate nel file di descrizione della simulazione primaria, il che elimina la necessità di modelli "black-box" precedentemente utilizzati, come menzionato negli studi di Stimberg et al.45,86.
L'attuale modello neuronale utilizza conduttanze modificate di tipo Hodgkin-Huxley46 con Na+, K+ e corrente di dispersione. Questo modello basato sulla conduttanza può essere ulteriormente ampliato includendo diversi altri tipi di canali, come i canali Ca2+ . Per i modelli sinaptici, è importante notare che questi parametri dovrebbero essere ottenuti per specifiche composizioni di subunità e dovrebbero essere valutate solo le varianti con parametri misurati con queste composizioni. In questo studio è stata selezionata la composizione del recettore α1β3γ2; pertanto, sono state incluse solo le varianti di α1, β3 o γ2, con parametri cinetici di canale misurati su un recettore GABAA α1β3γ2.
La stima della biofisica cellulare comporta la segmentazione della cellula in più compartimenti cilindrici, ciascuno potenzialmente in possesso di caratteristiche di conducibilità variabili. Nonostante le irregolarità nei dendriti di un neurone, possono essere visti come filamenti localmente omogenei. Tali modelli aiutano a comprendere la complessa struttura e il funzionamento delle cellule. La progettazione del modello si concentra su una versione semplificata della morfologia reale che può riflettere queste caratteristiche.
La posizione e le caratteristiche fisico-chimiche dell'amminoacido alterato determinano l'impatto sulla cinetica del canale. Ad esempio, una sostituzione di amminoacidi risultante in un amminoacido con una catena laterale più grande diminuirà la conduttanza del canale se questo cambiamento si verifica su un amminoacido che riveste il poro del canale. Le sostituzioni potrebbero anche influenzare l'apertura/chiusura del canale. Per semplicità in questo modello, la cinetica di legame del GABA è ridotta solo a un rapporto di recettori disponibili; Tuttavia, modelli più dettagliati possono essere progettati per includere questa interazione per studiare il possibile impatto delle sostituzioni che alterano l'affinità di legame del ligando.
In conclusione, il presente studio impiega metodi computazionali per prevedere varianti patogene nelle subunità del recettore GABAA e stimare qualitativamente il possibile fenotipo cellulare sulla base della simulazione di mutazioni associate all'epilessia nel modello di neurone piramidale ippocampale. Per quanto ne sappiamo, questo è il primo protocollo a esplorare l'applicazione combinata di genetica computazionale, modellazione molecolare e simulazioni neurali per valutare come le varianti genetiche possano contribuire alla disfunzione del recettore GABAA su più livelli di complessità, dal DNA alla funzione proteica e al comportamento neurale. Questo protocollo può fornire una base per migliorare la stima delle varianti potenzialmente deleterie e della patologia neurale associata nell'epilessia. Inoltre, può essere utilizzato nello studio di altre canalopatie, fornendo importanti intuizioni sul meccanismo alla base di rilevanti disturbi dello sviluppo neurologico e della rete. Sulla base di ciò, incorporando le valutazioni strutturali e fisico-chimiche del recettore GABAA per esaminare l'impatto funzionale delle varianti e la loro integrazione nella cinetica del canale del recettore GABAA , in futuro è possibile sviluppare un'analisi più accurata.
Divulgazioni
Tutti gli autori dichiarano di non avere conflitti di interesse relativi a questo lavoro.
Riconoscimenti
Ringraziamo Çağla Koca per la sua assistenza nella costruzione del modello di neurone.
Materiali
| Name | Company | Catalog Number | Comments |
| Brian2 | Sorbonne Université, INSERM, CNRS, Institut de la Vision, France; Imperial College London, United Kingdom | 2.8.0.4 | Stimberg et al., 2019 (https://pypi.org/project/Brian2/ ) |
| dbNSFP server | Genos Bioinformatics LLC, USA | v3.0 | Liu et al., 2020 (http://database.liulab.science/dbNSFP) (https://sites.google.com/site/jpopgen/dbNSFP) |
| HOPE | Centre for Molecular and Biomolecular Informatics CMBI, Radboud University, Netherlands | 1.1.1 | Venselaar et al., 2010 (https://www3.cmbi.umcn.nl/hope/) |
| Jalview | University of Dundee, UK | JV2 | Waterhouse et al., 2009 (https://www.jalview.org/) |
| Jupyter Notebook | Project Jupyter, USA | https://jupyter.org/install | |
| Phyton | Python Software Foundation, USA | 3.13 | https://www.python.org/downloads/ |
| Protter | ETH Zurich, Switzerland | Version 1.0 | Omasits, et al., 2014 (https://wlab.ethz.ch/protter/start/) |
| R | The R Foundation for Statistical Computing, USA | R version 4.3.2 | https://www.r-project.org/ |
| RStudio | Posit software, PBC, USA | RStudio 2023.12.1+402 "Ocean Storm" Release | https://posit.co/downloads/ |
Riferimenti
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon