Method Article
Мультимодальная платформа оптической визуализации для изучения клеточного метаболизма
* Эти авторы внесли равный вклад
В этой статье
Резюме
Используя мультимодальную платформу, сочетающую безмаркерные методы оптической визуализации, мы разработали протокол для визуализации и количественной оценки клеточной динамики и метаболизма. С помощью визуализации с помощью многофотонной флуоресценции, генерации второй гармоники и микроскопии вынужденного комбинационного рассеяния мы можем создать целостный обзор клеточной и молекулярной среды.
Аннотация
Технологии оптической визуализации имеют решающее значение в биомедицинских исследованиях благодаря их способности получать как морфологическую, так и функциональную информацию из биологических образцов с высоким пространственным разрешением. Эти оптические процессы используют различные взаимодействия световых молекул, такие как рассеяние, поглощение, излучение и генерация гармоник, между фотонами и молекулами в клетках, тканях или органах. В то время как традиционная биомедицинская визуализация исторически была сосредоточена на применении одного метода, недавние исследования показали, что эти различные методы обеспечивают взаимодополняющие идеи, а их совместные результаты обеспечивают более полное понимание молекулярных изменений в процессах старения и развитии заболеваний, а также основы клеточной биологии.
В последние десятилетия методы оптической визуализации без меток продвинулись вперед, что позволило детально исследовать клеточную и субклеточную среду. Например, многофотонная флуоресценция (MPF) не только облегчает целевую визуализацию белков, но и количественно измеряет метаболическую активность с помощью автофлуоресцентных коферментов, достигая высокой глубины проникновения и пространственного разрешения. Генерация второй гармоники (ГВГ) используется для визуализации таких структур, как коллаген во внеклеточном матриксе, в то время как стимулированное комбинационное рассеяние (SRS) отображает химические связи и молекулярный состав in situ с субклеточным разрешением.
Мы разработали мультимодальную платформу визуализации, которая сочетает в себе методы MPF, SHG и SRS. Интеграция этих модальностей в единую платформу позволяет получать многогранную информацию из одной и той же локализации в клетках, тканях, органах или даже телах, способствуя более детальному исследованию сложных взаимоотношений между клеточным метаболизмом, структурой внеклеточного матрикса и молекулярным составом. Эта мультимодальная система обеспечивает субклеточное разрешение, глубокое проникновение в ткани, визуализацию живых клеток/тканей in situ, а также обнаружение без меток и мгновенную корегистрацию без необходимости корректировки положения, переключения устройства или выравнивания после анализа. В этой статье мы представляем протокол визуализации без меток с помощью этой мультимодальной платформы и демонстрируем его применение для характеристики клеточного метаболизма и молекулярной гетерогенности в клетках и тканях для изучения старения и заболеваний.
Введение
Оптическая биомедицинская визуализация сыграла ключевую роль в углублении нашего понимания биологической структуры и функций. Изображения генерируются путем модуляции возбуждающего света и детектирования сигналов от взаимодействия света с тканью. Первый составной микроскоп, разработанный Гансом и Захариасом Янссенами около 1590 года, использовал две выпуклые линзы в трубке, обеспечивающие увеличение до 30x1. Современные оптические микроскопы, следуя многовековым достижениям, теперь могут достигать разрешения до 1-3 нм 2,3. В дополнение к высокому разрешению, усовершенствованные системы визуализации теперь обеспечивают более глубокое проникновение в ткани, большую эффективность и минимальное повреждение образцов, что делает их особенно подходящими для визуализации живых клеток и тканей. Визуализация без меток особенно выгодна, поскольку она захватывает информацию, не нарушая внутриклеточные процессы и не нарушая целостность образца.
Многофотонная флуоресцентная микроскопия (МПФ), в частности двухфотонная флуоресцентная микроскопия, широко используется для получения изображений без меток. В отличие от обычной флуоресцентной микроскопии, которая основана на линейном поглощении и излучении одного фотона, возбуждение MPF включает одновременное поглощение нескольких фотонов, объединенная энергия которых возбуждает одну молекулу флуорофора 4,5. Эти фотоны, как правило, в инфракрасном спектре, обладают половиной или меньшим количества энергии, необходимой для возбуждения одиночных фотонов. Более длинные волны и локализованное возбуждение в фокусной точке в этом нелинейном процессе приводят к меньшему рассеянию, более глубокому проникновению в ткани и снижению фототоксичности.
Клеточная метаболическая информация может быть получена с помощью безмаркерной микроскопии MPF путем обнаружения сигналов автофлуоресценции от эндогенных метаболических субстратов, таких как восстановленный никотинамидадениндинуклеотид (NADH) и флавинадениндинуклеотид (FAD). Эти коферменты демонстрируют различные спектры возбуждения и излучения, а их коэффициент интенсивности флуоресценции, известный как окислительно-восстановительный коэффициент (NADH/FAD), отражает окислительное состояние клетки. С тех пор, как Бриттон Чанс впервые представил концепцию окислительно-восстановительного коэффициента в 1979 году,были предложены дополнительные соотношения, включая NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) и FAD/(FAD + NAD(P)H), 6,7,8,9. Количественная оценка этих оптических окислительно-восстановительных соотношений с помощью визуализации MPF дает ценную информацию о метаболической динамике. Например, визуализация MPF может отличать раковые клетки от нормальных на основе их измененного метаболизма, демонстрируя свои возможности для диагностики рака 10,11,12. Однако автофлуоресцентное детектирование на основе MPF имеет ограничения. Другие собственные флуорофоры, такие как кератин, могут влиять на интенсивность флуоресценции, что приводит к спектральным перекрестным помехам и неточной интерпретации сигнала13. Кроме того, окислительно-восстановительный коэффициент отражает только общие клеточные окислительно-восстановительные изменения и не делает различий между НАДН из различных источников (например, цитоплазматических или митохондриальных) или между НАДН и НАД(П)Н, поскольку оба демонстрируют сходные спектральные пики на длине волны 450 нм, что приводит к смешанным сигналам интенсивности14.
Генерация второй гармоники (ГВГ), нелинейный оптический процесс, впервые продемонстрированный в биомедицинской области в 1980-х годах, широко используется для визуализации клеточных структур без меток 15,16. Подобно MPF, ГСП предполагает одновременное поглощение двух фотонов одинаковой энергии от сверхбыстрого импульсного лазера. Эти фотоны рекомбинируются для испускания нового фотона, в два раза превышающего частоту падающего света, что приводит к обнаружению сигнала второй гармоники. Это нелинейное оптическое взаимодействие происходит исключительно в нецентросимметричных материалах, которые проявляют ненулевую восприимчивость второго порядка к индуцированию поляризации для генерации сигнала второй гармоники 17,18. Это делает ГВГ особенно эффективным для визуализации нитчатых белков и фибриллярных структур, таких как коллаген, миозин и тубулин, не требуя экзогенных флуоресцентных красителей 15,17,19,20. Аномалии в обилии, жесткости, выравнивании и структуре фиброза и коллагена преобладают при многих состояниях, таких как воспаление и рак, что делает ГСП многообещающим инструментом для эффективного и неинвазивного выявления определенных заболеваний. Широкое применение визуализации ГВГ в онкологических исследованиях, включая исследования рака молочной железы, яичников и кожи, подчеркнуло ее решающую роль как в фундаментальных исследованиях, так и в потенциальном клиническом применении 24,25,26,27.
Различные молекулы демонстрируют различные уровни энергии колебаний, которые вызывают различную степень неупругого рассеяния при возбуждении падающим светом — явление, впервые описанное К. В. Раманом в 1928 году. С тех пор рамановский эффект широко используется в оптической микроскопии для обнаружения молекулярных и тканевых композиций без экзогенного мечения. Как вынужденное комбинационное рассеяние (SRS), так и когерентное антистоксово комбинационное рассеяние (CARS) когерентно возбуждают молекулярные колебания и используют нелинейное взаимодействие света для получения более сильного сигнала по сравнению с обычной спектроскопии спонтанного комбинационного рассеяния. Феномен SRS был впервые описан в 1962-29 гг. В 2008 году этот механизм был интегрирован в трехмерную многофотонную визуализацию, что позволило селективно обнаруживать химические вещества на основе изменений интенсивности в пучках Пампа и Стокса из-за молекулярных колебательныхпереходов30. Этот метод сводит к минимуму нерезонансные фоновые помехи, генерируя чистый сигнал интенсивности, превосходящий сигнал CARS. Визуализация SRS превосходна в обеспечении мультиплексной и гиперспектральной визуализации, что позволяет одновременно обнаруживать множественные химические связи и позволяет визуализировать молекулярный состав в образцах с высоким разрешением и значительной глубиной проникновения. Несмотря на то, что это относительно новый метод, SRS-визуализация доказала свою эффективность как в клинической диагностике, так и в метаболических исследованиях, in vivo и in vitro 30,31,32,33,34,35,36. Например, SRS может дифференцировать инфильтрированные опухолью ткани головного мозга от коры головного мозга и белого вещества путем количественной оценки соотношения липидов и белков, что позволяет очертить края опухоли неинвазивным способом без меток37,38. Кроме того, метаболические изменения, часто считающиеся признаками заболеваний, связанных со старением и раком, могут быть количественно оценены с помощью SRS, полученных путем обнаружения углерод-дейтериевых связей в образцах, обработанных тяжелой водой (D2O), что позволяет количественно измерить синтез белка, липогенез и другие макромолекулярные метаболические процессы 31,33,34,35,36. Возможность отслеживания метаболитов с высоким временным и пространственным разрешением делает SRS многообещающим инструментом для исследования и диагностики заболеваний с потенциалом для более широкого клинического применения.
Мультимодальная визуализация стала мощным подходом в биомедицинских исследованиях, объединяющим два или более методов визуализации для получения более полного понимания сложных биологических систем в одном образце. В 2018 году был представлен метод безметочной автофлуоресцентной мультигармонической микроскопии (SLAM), объединяющий двухфотонную флуоресценцию (2PF), трехфотонную флуоресценцию (3PF), SHG и генерацию третьей гармоники (THG)39. Такой подход способствует одновременной визуализации клеточных взаимодействий, динамических процессов и отдельных компонентов в микроокружении опухоли. Микроскопия SLAM обеспечивает минимальное возмущение и сниженные требования к мощности лазера для образца, что позволяет проводить глубокое профилирование тканей и обеспечивает более безопасный метод прижизненного мониторинга40. Другой мультимодальный метод, сочетающий собственную флуоресцентную спектроскопию, спектроскопию диффузного отражения и рамановскую спектроскопию, был разработан для обнаружения рака in situ во время хирургических процедур41. Кроме того, недавно разработанная мультимодальная система нелинейной эндоскопии, которая объединяет CARS, SHG и двухфотонную флуоресценцию (TPF), продемонстрировала возможность визуализации биологических образцов с субмикронным и субклеточным пространственным разрешением42. Комбинированная микроскопия 2PF и SRS также использовалась для визуализации тканей, клеток и органелл in vivo с высоким разрешением 42,43,44,45. Эти новые методы мультимодальной визуализации используют сильные стороны отдельных модальностей, что приводит к повышению разрешения, глубины проникновения и эффективности получения изображений, тем самым демонстрируя значительный потенциал для клинического и хирургического применения.
Этот мультимодальный подход становится все более предпочтительным по сравнению с одномодальной визуализацией, поскольку он обеспечивает более широкий диапазон измерений и снижает ограничения, связанные с отдельными методами. Как обсуждалось ранее, MPF измеряет эндогенную флуоресценцию для отражения метаболических изменений, ГСП может визуализировать нецентросимметричные структуры, такие как коллаген, в биологических образцах, а SRS преимущественно обнаруживает белки и липиды из-за высокой плотности химических связей, которые генерируют характерные комбинационные сигналы на основе их колебательных мод. Учитывая их когерентные свойства и общий принцип нелинейных оптических свойств, эти методы визуализации могут быть интегрированы в единую установку микроскопа с использованием ультракоротких импульсных лазеров, что позволяет получать различные биомаркеры в локализованных областях для обеспечения более полного представления о биологических процессах44,45. В этом документе описывается протокол реализации мультимодальной платформы визуализации, которая интегрирует MPF, SHG и SRS для приложений биомедицинских исследований.
протокол
1. Эксперименты по мультимодальной визуализации без меток
ПРИМЕЧАНИЕ: Данный протокол посвящен настройке и процедуре получения данных мультимодальной визуализации без этикеток.
- Настройка мультимодального микроскопа (рис. 1A) и калибровка системы
- Разогрейте лазер и подождите примерно 15-20 минут.
- Включите блоки управления и мониторы в следующей последовательности: Блок управления | Контроллер с сенсорной панелью | Адаптер переменного тока для основного лазерного пульта Адаптер переменного тока для вспомогательного лазерного пульта.
- Питание на детекторе фотодиодов Si и синхронном усилителе.
- Настройте накачку лазерных лучей и пучка Стокса. Настройте лазерную систему с перестраиваемым лучом накачки от 780 нм до 990 нм, длительностью импульса 5 - 6 пс и частотой следования 80 МГц. Лазерный луч Стокса имеет фиксированную длину волны 1031 нм с импульсом 6 пс и частотой повторения 80 МГц . Убедитесь, что оба луча насоса и пучка Стокса имеют низкую мощность (не менее 20 мВт), чтобы их было видно на юстировочной пластине.
- Поместите одну юстировочную пластину в оптическую траекторию (сразу после лазерной коробки), чтобы проверить пространственное перекрытие насосов и лучей Стокса, и отрегулируйте зеркало до тех пор, пока оба пятна не перекрываются в центре юстировочной пластины.
ПРИМЕЧАНИЕ: Несмотря на то, что накачки и пучки Стокса примерно перекрываются, тонкая настройка пространственного перекрытия двух лазерных лучей может обеспечить оптимальный сигнал SRS. - После пространственного перекрытия выполните тонкую настройку с помощью PSD (позиционно-чувствительного детектора), сначала щелкнув по управляющему программному обеспечению OPO . С помощью шестигранной клавиши тщательно отрегулируйте оптическое зеркало 1 (OM1) и наблюдайте за изменением положения PSD для X-Y, чтобы добиться минимального отклонения от центра. Затем, чтобы выполнить более точную настройку, используйте шестигранный ключ на оптическом зеркале 2 (OM2) до тех пор, пока индикатор положения не окажется по центру дисплея PSD.
ПРИМЕЧАНИЕ: OM1 и OM2 - это два зеркала в головке OPO для регулировки юстировки лазера. - Наконец, отцентрируйте конденсор, повернув два винта для центрирования конденсора, чтобы переместить изображение ирисовой диафрагмы в центр поля зрения.
ПРИМЕЧАНИЕ: Полевая ирисовая диафрагма может ограничивать диаметр луча света, попадающего на объектив, и, таким образом, исключать посторонний свет, улучшая контрастность изображения.
- Этапы получения мультимодальной визуализации без меток
- Нанесите масло на масляный конденсатор с высокой числовой апертурой (1,4 NA) и установите предметное стекло микроскопа на смазанный маслом конденсатор. Наконец, поместите большую каплю воды на предметное стекло микроскопа для объектива с 25-кратным увеличением воды. Убедитесь, что микроскоп надежно закреплен и неподвижен, а затем отрегулируйте z-ступень для настройки фокуса до тех пор, пока изображение биологического образца в ярком поле не станет видимым под объективом 25-кратного увеличения.
- Начните процесс визуализации в правильной последовательности: MPF, SHG и SRS, чтобы избежать фотообесцвечивания, которое повлияет на качество изображения и точный анализ MPF. Чтобы быстро переключаться между MPF и SHG, переключитесь с балки насоса на фиксированную балку Стокса.
ПРИМЕЧАНИЕ: Настройка лазера накачки от 800 нм (автофлуоресценция NADH и FAD) до 791,3 нм (CH3 рамановского сдвига) может занять 1-2 минуты. - Выберите разрешение изображения (512 x 512 пикселей) и подходящее время задержки для каждой модальности. Используйте 8 μс/пиксель для MPF и SHG со средним кадром выше 3. Используйте 40 μс/пиксель со средним кадром 2 для модальности SRS.
ПРИМЕЧАНИЕ: Средний кадр означает количество изображений, которые должны быть усреднены перед сохранением. - Чтобы получить автофлуоресценцию с помощью MPF, выключите лазерный луч Стокса и настройте лазер накачки на 800 нм для возбуждения NADH и флавина. Установите фильтрующий куб FVOPT в световой тракт с 460 ± 10 нм и 515 ± 10 нм для NADH и флавина соответственно. Таким образом, мощность образца составляет примерно 15 мВт.
- Получите сигнал коллагенового волокна с помощью ГВГ. Выключите лазерный луч накачки, используйте только лазерный луч Стокса и установите мощность 500 мВт. Приобретайте только канал, относящийся к фильтру 515 нм.
- Получите пространственное распределение белков и липидов с помощью SRS. Держите оба лазерных луча включенными и отрегулируйте частоту лазерного луча в соответствии с конкретной модой колебаний для каждой молекулы.
ПРИМЕЧАНИЕ: Как правило, 791,3 нм используется для CH3, 797 нм - для CH2, 787 нм - для ненасыщенных липидов и 794,6 - для насыщенных липидов. Таким образом, мощность образца составляет примерно 40 мВт. - Чтобы получить наборы гиперспектральных изображений SRS, откройте управляющее программное обеспечение OPO, чтобы выбрать развертку, установите диапазон длин волн от 780 нм до 806,5 нм и выберите номер стека не менее 60. Затем захватите стек гиперспектральных изображений.
ПРИМЕЧАНИЕ: Полученный набор данных содержит 60 изображений, каждое из которых представляет пространственное распределение при определенном сдвиге комбинационного рассеяния света (от 2700 см-1 до 3150 см-1). Время получения гиперспектральных данных зависит от поля зрения (FOV) изображения и времени сканирования, но будет составлять примерно от 5 до 8 минут для изображения 512 x 512. При получении больших изображений следует позаботиться о том, чтобы свести к минимуму продолжительность работы под лазером, чтобы предотвратить фотоповреждение, которое может произойти через 2 часа (в зависимости от толщины образца и размера поля зрения). - Сохраняйте все изображения одних и тех же областей интереса (ROI) в одной папке. Формат изображения - файл Olympus .oir.
2. Анализ изображений
- Используйте программное обеспечение для обработки изображений, чтобы открыть все сохраненные необработанные изображения, чтобы назначить цвет и добавить масштабную линейку для отображения изображения. Также используйте его для создания бинарной маски NADH и изображений насыщенных жирных кислот (SFA, 2,880 см-1) для оптического окислительно-восстановительного отношения и анализа ненасыщения липидами.
ПРИМЕЧАНИЕ: Убедитесь, что интенсивность маски имеет только 0 и 1. - Используйте собственный скрипт Python для выполнения любого нисходящего анализа (код доступен по адресу https://github.com/lingyanshi2020/HSI_Analysis).
ПРИМЕЧАНИЕ: Этот скрипт предназначен для обработки нескольких областей интереса (ROI) как из контрольных, так и из раковых образцов. Основная функция скрипта требует только двух входных данных: пути к файлам для папки control и папки с изображением рака.- Перебирайте папки, сопоставляя изображения с одинаковым базовым именем (например, "roi_1_flavin.tif", "roi_1_nadh.tif", "roi1_mask.tif").
- Для каждого набора совпадающих изображений позвольте скрипту выполнить ратиометрический анализ и создать новые ратиометрические изображения на основе этих вычислений.
Оптическое окислительно-восстановительное отношение рассчитывается как флавин / (NADH + флавин).
Ненасыщенность липидами рассчитывается как: USFAs / (SFA + USFAs), где USFAs представляют собой ненасыщенные жирные кислоты. - Все ратиметрические значения с изображений затем сохраняются в кадре данных pandas для упрощения работы. Выполните одностороннюю ANOVA и используйте matplotlib с styple "ggplot" или seaborn для создания barplot или boxplot для количественной оценки.
- Используйте изображения размером 2 930 см-1 (CH3) и 2 850 см-1 (CH2) для создания изображения с вычетом (CH3 -CH 2). Затем смешайте изображение CH2 CH3 - CH2 в цветовом пространстве RGB с пользовательской таблицей LUT, которая имитирует окрашивание H&E, для создания цифрового гистологического изображения46.
- После получения гиперспектральных изображений SRS используйте собственный скрипт Python для кластеризации k-средних для сегментации изображений на основе спектрального сходства (код доступен по адресу https://github.com/lingyanshi2020/HSI_Analysis).
- Загрузите .tiff стеки изображений с помощью пакета tifffile. Преобразование стека гиперспектральных изображений SRS (x, y, спектры) в 2D-массив (пиксели, спектры).
- Используйте пакет scikit-learn для развертывания алгоритма k-средних в этом 2D-массиве с определенным числом кластеров.
- Измените форму полученных меток кластера обратно в исходные размеры изображения, создав карту сегментации.
- Назначьте уникальные цвета для каждого кластера, создав изображение в ложных цветах, представляющее различные биохимические составы в ткани.
- Используйте matplotlib для построения графиков спектров каждого кластера со средним значением (сплошная линия) и стандартным отклонением (область тени)
- Следуйте протоколу протокола сопоставления штрафных ссылок SRS (PRM-SRS) с использованием MATLAB для создания пространственного безметочного детектирования липидного подтипа с помощью наборов гиперспектральных изображений SRS47.
- Систематизируйте изображения и сохраните их в формате .tif 300 dpi.
Результаты
Изображения на рисунке 2 являются репрезентативными для результатов, полученных в результате следования протоколу получения автофлуоресценции FAD и NADH, а также четырех каналов SRS для белка, общих липидов, ненасыщенных липидов и насыщенных липидов. Здесь мы также генерируем псевдогистологическое изображение, как описано в протоколе, с помощью смешивания цветов RGB. Получение каналов MPF и SRS позволяет получить файлы изображений, которые в дальнейшем будут использоваться в ратиометрическом анализе. Пример такого анализа можно увидеть на рисунке 3 на примере легочной ткани человека. После получения изображений в протоколе наша методология анализа изображений с использованием Python или ImageJ использует соотношение отдельных каналов для предоставления количественной метаболической информации. Как показано на изображениях ненасыщенности липидов и оптического окислительно-восстановительного коэффициента на рисунке 3, ратиометрический анализ позволяет получить цветовую карту распределения относительной метаболической активности и молекулярного состава. Мы использовали эти измерения для проведения наблюдений за изменениями в метаболических путях и содержании липидов в конкретных тканях, патологиях или отдельных биологических факторах. Как видно на рисунке 4, это может обеспечить количественное сравнение здоровой и опухолевой ткани путем сравнения средних значений окислительного стресса и ненасыщения липидами в дополнение к их распределению на изображении 2D-микроскопии.
Для ратиометрического анализа изображений MPF и SRS мы обычно ориентируемся на оптический окислительно-восстановительный потенциал и коэффициент ненасыщенности, как показано в уравнениях (1) и (2).
 (1)
(1)
 (2)
(2)
Где IC — интенсивность пикселей из канала C. Таким образом, мы измеряем отношение на пиксель и получаем пространственное распределение этих метаболических и молекулярных маркеров.
В дополнение к ратиометрическому анализу на рисунке 4 также показано еще одно потенциальное направление применения нашей мультимодальной платформы: гиперспектральный анализ изображений. Как описано в этом протоколе, мы можем получить гиперспектральное изображение SRS, выполнив развертку по нескольким длинам волн лазера с использованием перестраиваемого луча накачки. Это позволяет нам реконструировать CH-область спектра комбинационного сдвига для каждого пикселя в микроскопическом изображении, объединяя как химическую информацию, полученную с помощью спектроскопии комбинационного рассеяния, так и пространственную информацию, полученную с помощью оптической микроскопии. В рабочем процессе анализа изображений мы выделяем два метода, реализованных для анализа этих гиперспектральных изображений: PRM-SRS для обнаружения биомолекул и кластеризацию k-средних. На рисунке 4E показано применение PRM-SRS на изображении опухоли печени мыши. Алгоритм PRM-SRS обеспечивает корреляцию между спектрами для каждого отдельного пикселя, скорректированную штрафом за сдвиги в спектре комбинационного рассеяния света; Затем алгоритм генерирует изображения распределения вероятностей для каждого из липидов.
Кластеризация пиксельных спектров с использованием K-средних является еще одним методом, который мы реализуем для визуализации группировки конкретных спектральных фенотипов, что позволяет разделить их по концентрации связи и молекулярному составу по интенсивности и спектральной форме соответственно. На рисунке 5 представлен пример того, как анализ изображений с помощью кластеризации k-средних может быть применен в нашем протоколе. Мы ожидаем, что кластеры k-средних структурно выровняются с признаками псевдогистологических рисунков в дополнение к дальнейшей кластеризации из признаков, которые не так легко различимы как по каналам SRS, так и по псевдогематоксилину и гистологическому окрашиванию (H&E). PRM-SRS и кластерный анализ k-средних дополняют эту безметочную платформу визуализации, предоставляя химическую и полуцелевую информацию о конкретных аналитах и молекулярных связях без необходимости экзогенного зондирования.
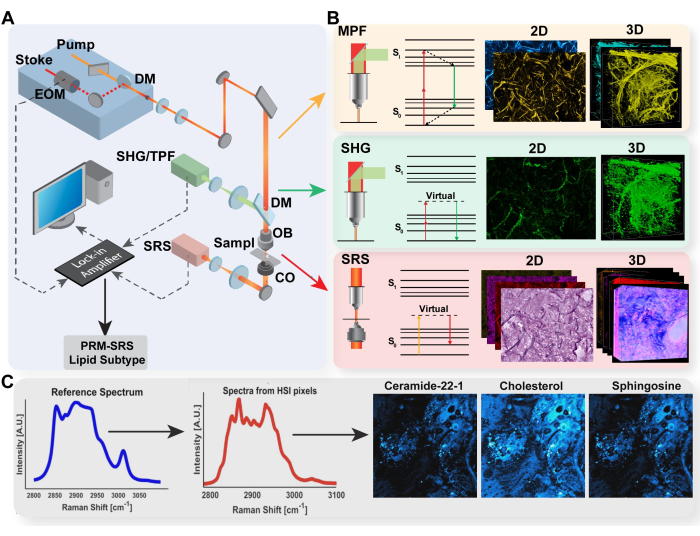
Рисунок 1: Схема мультимодальной платформы визуализации и детектирования биомолекул PRM-SRS. (A) Схема настройки аппаратного обеспечения для систем визуализации MPF/TPF, SHG и SRS. (B) Световой путь и диаграмма Яблонски для TPF, SHG и SRS с репрезентативными полученными изображениями для каждой модальности. (C) Диаграмма рабочего процесса PRM-SRS для согласования спектральных эталонов между спектроскопией спонтанного комбинационного рассеяния света и гиперспектральной визуализацией SRS. Сокращения: MPF = многофотонная флуоресценция; TPF = двухфотонная флуоресценция; SHG = генерация второй гармоники; SRS = вынужденное комбинационное рассеяние; PRM = штрафное сопоставление ссылок. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 2: Каналы MPF и SRS из ткани первичной моторной коры головного мозга человека (M1). (А,Б) Автофлуоресцентные каналы MPF для Flavin/FAD и NADH. (К-Ж) Каналы SRS, захваченные на определенных пиках комбинационного сдвига; пик асимметричного растяженияCH3 (2930 см-1) для белков, пик асимметричного растяженияCH2 (2845 см-1) для липидов, пик 2885 см-1 для насыщенных жирных кислот и пик 3010 см-1 для ненасыщенных жирных кислот. (G) Объединенные белковые (синие) и липидные (зеленые) каналы SRS для определения их соответствующего пространственного распределения. (H) Псевдогистологические изображения (SRH), отражающие окрашивание H&E. Масштабные линейки = 20 μм. Сокращения: MPF = многофотонная флуоресценция; FAD = флавинадениндинуклеотид; NADH = восстановленный никотинамидадениндинуклеотид; SRS = вынужденное комбинационное рассеяние; SRH = гистология SRS. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
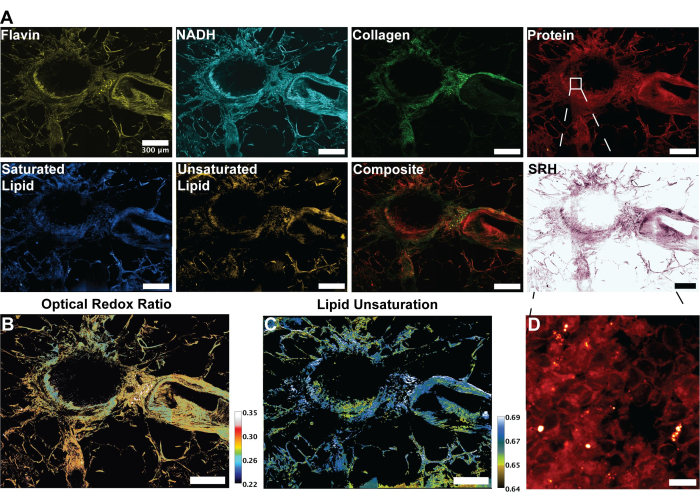
Рисунок 3: Изображения MPF, SHG и SRS и анализ легочной ткани человека. (A) автофлуоресценция MPF для Flavin/FAD и NADH, сигнал SHG для коллагеновых волокон, сигнал SRS для белка (2,930 см-1), липидов (2,845 см-1), насыщенных липидов (2,885 см-1) и ненасыщенных липидов (3,010 см-1), а также составное объединенное изображение множественных модальностей. (B) Ратиометрическое изображение оптического окислительно-восстановительного коэффициента. (В) Ратиометрическое изображение ненасыщения липидами. (D) Увеличенное изображение эпителиальных клеток во внутреннем слое бронхов. Масштабные линейки = 200 мкм. Сокращения: MPF = многофотонная флуоресценция; SHG = генерация второй гармоники; SRS = вынужденное комбинационное рассеяние; FAD = флавинадениндинуклеотид; NADH = восстановленный никотинамидадениндинуклеотид. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 4: Ратиометрический и гиперспектральный анализ опухолей печени мышей. (A) Автофлуоресцентные изображения MPF для NAD[P]H и FAD, SRS-каналы для общего белка, насыщенных и ненасыщенных липидов, а также псевдогистологические изображения здоровой контрольной печени мыши (вверху) и опухолевой (внизу) ткани. (B) Окислительный стресс (желто-зеленый), измеряемый оптическим окислительно-восстановительным коэффициентом, и ненасыщенность липидами (сине-красный), рассчитанный по контрольной (слева) и опухолевой (справа) тканям. Масштабные линейки = 100 мкм. (C,D) Гистограммы, демонстрирующие ратиометрические различия в окислительном стрессе и ненасыщенности липидами между контрольными (синим) и опухолевыми (красными) образцами печени. Статистика значимости: * для значимости p ≤ 0,05, ** для значимости p ≤ 0,01. (E) Анализ подтипов липидов PRM-SRS является результатом анализа гиперспектральных изображений. Слева направо: белковый канал SRS для структурного эталона, масштабная линейка = 100 мкм, изображение распределения вероятностей подтипов липидов (TAG 18:1, холестерин и церамид C24:0). График референсных спектров липидных подтипов и средних спектров пикселей из SRS HSI по области CH. Сокращения: MPF = многофотонная флуоресценция; SRS = вынужденное комбинационное рассеяние; NAD[P]H = восстановленный никотинамидадениндинуклеотидфосфат; FAD = флавинадениндинуклеотид; HSI = гиперспектральные изображения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 5: Гиперспектральная кластеризация k-средних SRS на первичной моторной коре головного мозга человека (M1). (A-F) Шесть репрезентативных областей, показывающих: слева: изображения стимулированной гистологии комбинационного рассеяния света (SRH), обеспечивающие H&E-подобную визуализацию тканей; в центре: соответствующие результаты кластеризации k-средних; Справа: спектральные профили комбинационного рассеяния света со средними интенсивностями (сплошные линии) и стандартными отклонениями (затененные области). Количество центроидов в диапазоне от 4 до 6 средних. Использовалась кластеризация без учителя, поэтому разные цвета на кластеризованных изображениях представляют различные химические составы, идентифицированные алгоритмом k-средних. Каждый кластер (представленный уникальным цветом) соответствует областям со схожими спектральными профилями в области растяжения CH. Масштабная линейка = 20 мкм. Сокращения: SRH = гистология SRS. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обсуждение
Эта мультимодальная система представляет собой мощную платформу визуализации для целостной визуализации молекулярного окружения образцов в широком диапазоне биологического происхождения и патологических состояний. Преимущество использования различных безметочных методов заключается в возможности получения дополнительной информации и нацеливания на конкретные аналиты, что в противном случае было бы затруднительно или невозможно в рамках одного метода визуализации без меток. В частности, три метода нелинейной визуализации (SRS, MPF, SHG), упомянутые в этой статье, позволяют количественно оценить состав макромолекул, динамику энергии с помощью оптического окислительно-восстановительного коэффициента и структурную информацию, включая состав и морфологию внеклеточного матрикса 6,48,49. Кроме того, по сравнению с использованием каждого отдельного метода визуализации по отдельности, визуализация с помощью одной комбинированной системы микроскопа обеспечивает немедленную регистрацию изображений и сокращение времени хранения образцов. Мы даже проводили визуализацию живых клеток с помощью этого подхода, возможно, из-за более низкой фотоэкспозиции по сравнению с конфокальной флуоресцентной микроскопией, используя выбранные временные точки для последовательного получения каждой модальности, как указано в вышеуказанном протоколе. Помимо индивидуальных преимуществ, мультимодальная платформа также выигрывает от множества точек зрения на биологическое состояние за счет измерения различных биомаркеров без меток.
Визуализация автофлуоресцентных белков без меток с помощью MPF в первую очередь направлена на количественное определение оптического окислительно-восстановительного отношения, измеряющего окислительно-восстановительные реакции, чему способствует окисление FAD в FAD+ и восстановление NAD+ до NADH50. Это важный маркер метаболизма в клетках и тканях, поскольку он обеспечивает измерение относительной активности между окислительным фосфорилированием и гликолизом, двумя основными путями для АТФ поколения6. В частности, снижение концентрации NADH и увеличение FAD+ являются маркером повышенного окислительного фосфорилирования в митохондриях; Между тем, для повышенного гликолиза в цитоплазме верно обратное. Тенденция предпочитать один метаболический путь другому в производстве энергии была связана с несколькими патологическими изменениями и активациями путей, связанными с раком, что подчеркивает потенциальное использование ЧОО в качестве маркера раннего обнаружения 6,51.
Аналогичным образом, получение сигнала фиброзного коллагена ГВГ использует нелинейный метод визуализации без меток для количественной оценки и визуализации биологического маркера здоровья. ГСП может эффективно отслеживать распределение коллагена I-III типа благодаря их нецентросимметричной структуре. Волокнистые коллагеновые белки, измеренные с помощью ГВГ, были признаны важными диагностическими маркерами для нескольких заболеваний, включая рак и фиброз 25,26,27,52,53. В дополнение к обнаружению коллагена в качестве маркера заболевания, наше приобретение коллагена через ГВГ может служить сильным индикатором структуры клеток и тканей из-за роли волокнистого коллагена в составе внеклеточного матрикса и структурных границах через ткани54,55. С помощью сигнала коллагена ГВГ мы можем проводить обоснованные наблюдения за отдельными клетками или отдельными тканевыми функциональными единицами без необходимости использования экзогенного маркера для клеточных мембран. В целом, количественное определение коллагеновых фибрилл методом ГСП имеет явное преимущество как для диагностических возможностей, так и для простоты анализа при интеграции с другими методами визуализации без меток.
Используя микроскопию SRS, мы можем захватить несколько маркеров макромолекулярного состава, в частности липидов и жирных кислот. В этом протоколе мы описываем, как получить сигнал SRS для ненасыщенных и насыщенных жирных кислот для ратиометрического анализа ненасыщенности липидов в одном образце. Вертикальный лазерный сканирующий микроскоп с 25-кратным водяным объективом был применен для обеспечения ближней ИК-пропускной способности. Лазерные лучи проходили через образец и собирались масляным конденсатором с большой числовой апертурой (1,4 NA). Для блокировки пучка Стокса был использован фильтр с высоким внешним диаметром (950 нм), в то время как луч накачки достигал только детектора фотодиодов на основе кремния для обнаружения стимулированного сигнала комбинационных потерь. Выходной ток от фотодиода завершался и фильтровался. Впоследствии синхронный усилитель на частоте 20 МГц демодулировал ток в X с нулевым сдвигом фазы, а программный модуль использовал демодулированный сигнал для генерации изображения во время лазерного сканирования.
Исследования показали, что изменения ненасыщения липидами оказывают глубокое влияние на клеточную и органелловую мембрану, а нарушение регуляции липидного обмена может быть сильным маркером для нескольких заболеваний, включая рак и нейродегенеративныезаболевания. Для более полного обзора липидной дисрегуляции мы анализируем состав подтипов с помощью гиперспектральной визуализации SRS. Гиперспектральные изображения SRS (HSI) сочетают пространственную информацию с высоким разрешением, полученную с помощью микроскопии SRS, с химически значимым сигналом рамановской спектроскопии посредством последовательной настройки, как описано в протоколе. Затем эти пиксельные спектры анализируются двумя методами: кластеризация с помощью k-средних и обнаружение биомолекул с помощью PRM-SRS.
Кластеризация спектров пикселей с помощью K-средних приводит к разделению областей в 2D-изображении по молекулярной концентрации и составу из-за линейной зависимости между интенсивностью сигнала SRS и концентрацией молекулярной связи60,61. С помощью этой методологии мы применяем протокол для определения областей, обладающих специфическими молекулярными фенотипами, предписанными пиксельными спектрами и общим спектральным центроидом. Для тщательного изучения различной молекулярной экспрессии в конкретных пикселях мы реализуем PRM-SRS для выполнения спектрального согласования между референсным спектром для конкретной молекулы, полученным с помощью спонтанной рамановской спектроскопии, и пиксельными спектрами, полученными в результате регистрации SRS HSI. Таким образом, мы можем определить вероятность экспрессии для отдельных молекул без использования экзогенных зондов или меток. Этот анализ продемонстрировал способность отслеживать определенные подтипы липидов в тканях и биологических моделях, а также способность выявлять патологические изменения в липидном составе и, следовательно, дислипидемию. Кроме того, последние достижения в улучшении спектрального разрешения за счет спектрального расщепления пикосекундного распространения импульсов через волокна диоксида кремния еще больше стимулируют применение SRS HSI в качестве модальности для биохимической визуализации без меток62. С помощью этих методов, выделенных в протоколе, мы создаем платформу для выполнения комплексного ратиометрического и спектрального анализа с использованием безметочной модальности SRS.
В дополнение к относительной сложности и новизне этих методов, существуют некоторые важные ограничения в нашем подходе, который следует учитывать. Получение изображений MPF с помощью пикосекундного импульсного лазера, в отличие от широко используемых фемтосекундных лазеров, требует более высокой мощности возбуждения флуорофора и риска фотообесцвечивания. Учитывая это, мы оптимизировали вышеуказанный протокол, чтобы уменьшить потенциальное фотоповреждение от нашей платформы за счет уменьшения лазерной мощности, падающей на образец. Кроме того, в то время как предыдущие исследования продемонстрировали возможность оценки оптического окислительно-восстановительного коэффициента по данным автофлуоресцентной визуализации MPF NADH и FAD, флуоресцентная микроскопия времени жизни (FLIM) продемонстрировала более высокую точность в оценке энергетического метаболизма 63,64,65. Это связано со способностью дифференцировать связанные с белками и несвязанные концентрации NADH и FAD на основе их различного времени жизни флуоресценции, По сравнению с возможностями MPF, это позволило использовать FLIM для всесторонних исследований метаболической активности по различным энергетическим путям, включая окислительное фосфорилирование и гликолиз 16,63,64,65,66,67, 68. Несмотря на то, что этот протокол исключает FLIM, мы ожидаем, что в ближайшем будущем этот метод будет интегрирован в эту платформу визуализации, что улучшит анализ энергетического метаболизма, который мы выполняем в настоящее время. Тем не менее, с помощью разработанного здесь протокола мы представляем рабочий процесс и аппаратную настройку для мультимодального подхода к визуализации, который может измерять метаболизм биологической структуры с различных точек зрения.
Раскрытие информации
У авторов нет конфликта интересов, о котором можно было бы заявить.
Благодарности
Мы благодарим доктора Глорию Прихубер и членов ее команды HuBMAP за предоставленные срезы легочной ткани человека. Мы благодарим доктора Кун Чжана за предоставление тканей человеческого мозга. Мы также благодарим доктора Ген-Шен Фэн за предоставленные образцы печени мышей. Мы выражаем признательность за поддержку со стороны NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, фондов стартапов UCSD, премии Sloan Research Fellow Award и премии CZI DAF2023-328667.
Материалы
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Ссылки
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены