Method Article
Piattaforma di imaging ottico multimodale per lo studio del metabolismo cellulare
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Utilizzando una piattaforma multimodale che combina modalità di imaging ottico label-free, abbiamo sviluppato un protocollo per la visualizzazione e la quantificazione delle dinamiche cellulari e del metabolismo. Attraverso l'imaging tramite fluorescenza multifotone, generazione di seconda armonica e microscopia a scattering Raman stimolato, possiamo generare una panoramica olistica dell'ambiente cellulare e molecolare.
Abstract
Le tecnologie di imaging ottico sono fondamentali negli studi biomedici per la loro capacità di ottenere informazioni sia morfologiche che funzionali da campioni biologici ad alta risoluzione spaziale. Questi processi ottici sfruttano varie interazioni luce-molecola, come la diffusione, l'assorbimento, l'emissione e la generazione armonica, tra i fotoni e le molecole all'interno di cellule, tessuti o organi. Mentre l'imaging biomedico convenzionale si è storicamente concentrato sull'applicazione di un'unica modalità, ricerche recenti hanno dimostrato che queste diverse tecniche forniscono intuizioni complementari e i loro risultati combinati offrono una comprensione più completa dei cambiamenti molecolari nei processi di invecchiamento e nello sviluppo della malattia e dei fondamenti della biologia cellulare.
Negli ultimi decenni, i metodi di imaging ottico label-free sono avanzati, consentendo un'esplorazione dettagliata degli ambienti cellulari e subcellulari. Ad esempio, la fluorescenza multifotone (MPF) non solo facilita l'imaging mirato delle proteine, ma quantifica anche l'attività metabolica attraverso coenzimi autofluorescenti, raggiungendo un'elevata profondità di penetrazione e risoluzione spaziale. La generazione di seconda armonica (SHG) viene utilizzata per visualizzare strutture come il collagene nella matrice extracellulare, mentre lo scattering Raman stimolato (SRS) mappa i legami chimici e la composizione molecolare in situ con risoluzione subcellulare.
Abbiamo sviluppato una piattaforma di imaging multimodale che combina le modalità MPF, SHG e SRS. L'integrazione di queste modalità in un'unica piattaforma consente l'acquisizione di informazioni sfaccettate dalla stessa localizzazione all'interno di cellule, tessuti, organi o persino corpi, facilitando un'esplorazione più dettagliata delle intricate relazioni tra metabolismo cellulare, struttura della matrice extracellulare e composizione molecolare. Questo sistema multimodale offre risoluzione subcellulare, penetrazione profonda dei tessuti, imaging in situ di cellule vive/tessuti, nonché rilevamento senza marcatura e coregistrazione istantanea senza la necessità di regolazioni della posizione, commutazione del dispositivo o allineamento post-analisi. Qui, presentiamo un protocollo per l'imaging label-free con questa piattaforma multimodale e dimostriamo la sua applicazione nella caratterizzazione del metabolismo cellulare e dell'eterogeneità molecolare in cellule e tessuti per lo studio dell'invecchiamento e delle malattie.
Introduzione
L'imaging biomedico ottico è stato fondamentale per far progredire la nostra comprensione della struttura e della funzione biologica. Le immagini sono generate modulando la luce di eccitazione e rilevando i segnali dalle interazioni luce-tessuto. Il primo microscopio composto, sviluppato da Hans e Zacharias Janssen intorno al 1590, utilizzava due lenti convesse in un tubo, fornendo un ingrandimento fino a 30x1. I moderni microscopi ottici, dopo secoli di progressi, possono ora raggiungere risoluzioni fino a 1-3 nm 2,3. Oltre a offrire un'alta risoluzione, i sistemi di imaging avanzati offrono ora una penetrazione più profonda nei tessuti, una maggiore efficienza e danni minimi al campione, rendendoli particolarmente adatti per l'imaging di cellule vive e tessuti. L'imaging label-free è particolarmente vantaggioso in quanto acquisisce le informazioni senza interrompere i processi intracellulari o compromettere l'integrità del campione.
La microscopia a fluorescenza multifotone (MPF), in particolare la microscopia a fluorescenza a due fotoni, è stata ampiamente utilizzata per l'imaging label-free. A differenza della microscopia a fluorescenza convenzionale, che si basa sull'assorbimento e l'emissione lineare di un singolo fotone, l'eccitazione MPF comporta l'assorbimento simultaneo di più fotoni, la cui energia combinata eccita una singola molecola di fluoroforo 4,5. Questi fotoni, tipicamente nello spettro infrarosso, possiedono la metà o meno dell'energia richiesta per l'eccitazione di un singolo fotone. Le lunghezze d'onda più lunghe e l'eccitazione localizzata nel punto focale di questo processo non lineare si traducono in una minore diffusione, una penetrazione più profonda nei tessuti e una ridotta fototossicità.
Le informazioni metaboliche cellulari possono essere acquisite mediante microscopia MPF label-free attraverso il rilevamento di segnali di autofluorescenza da substrati metabolici endogeni, come la riduzione della nicotinammide adenina dinucleotide (NADH) e la flavina adenina dinucleotide (FAD). Questi coenzimi mostrano spettri di eccitazione ed emissione distinti e il loro rapporto di intensità della fluorescenza, noto come rapporto redox (NADH/FAD), riflette lo stato ossidativo della cellula. Da quando Britton Chance ha introdotto per la prima volta il concetto di rapporto redox nel 1979, sono stati proposti rapporti aggiuntivi, tra cui NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) e FAD/(FAD + NAD(P)H), 6,7,8,9. La quantificazione di questi rapporti ottici redox tramite l'imaging MPF fornisce preziose informazioni sulle dinamiche metaboliche. Ad esempio, l'imaging MPF può distinguere le cellule tumorali dalle cellule normali in base al loro metabolismo alterato, dimostrando il suo potenziale per la diagnosi del cancro 10,11,12. Tuttavia, il rilevamento dell'autofluorescenza basato su MPF presenta dei limiti. Altri fluorofori intrinseci, come la cheratina, possono contribuire all'intensità della fluorescenza, portando a diafonia spettrale e interpretazione imprecisa del segnale13. Inoltre, il rapporto redox riflette solo i cambiamenti complessivi di ossidoriduzione cellulare e non distingue tra NADH proveniente da fonti diverse (ad esempio, citoplasmatico o mitocondriale) o tra NADH e NAD(P)H, poiché entrambi mostrano picchi spettrali simili a 450 nm, risultando in segnali di intensità miscelati14.
La Second Harmonic Generation (SHG), un processo ottico non lineare dimostrato per la prima volta in campo biomedico negli anni '80, è stato ampiamente utilizzato per l'imaging label-free di strutture cellulari15,16. Simile all'MPF, l'SHG comporta l'assorbimento simultaneo di due fotoni della stessa energia da un laser pulsato ultraveloce. Questi fotoni vengono ricombinati per emettere un nuovo fotone a una frequenza doppia rispetto alla luce incidente, con conseguente rilevamento del segnale della seconda armonica. Questa interazione ottica non lineare si verifica esclusivamente in materiali non centrosimmetrici che mostrano una suscettibilità del secondo ordine diversa da zero per indurre una polarizzazione per generare il segnale della seconda armonica17,18. Ciò rende l'SHG particolarmente efficace per l'imaging delle proteine filamentose e delle strutture fibrillari, come il collagene, la miosina e la tubulina, senza richiedere coloranti a fluorescenza esogena 15,17,19,20. L'anomalia nell'abbondanza, nella rigidità, nell'allineamento e nella struttura della fibrosi e del collagene sono prevalenti in molte condizioni come l'infiammazione e il cancro, rendendo l'SHG uno strumento promettente per il rilevamento efficiente e non invasivo di alcune condizioni patologiche 21,22,23. L'ampia applicazione dell'imaging SHG nella ricerca oncologica, compresi gli studi sui tumori al seno, alle ovaie e alla pelle, ha evidenziato il suo ruolo cruciale sia nella ricerca di base che nelle potenziali applicazioni cliniche 24,25,26,27.
Diverse molecole esibiscono livelli distinti di energia vibrazionale, che inducono vari gradi di dispersione anelastica quando eccitati dalla luce incidente, un fenomeno caratterizzato per la prima volta da C. V. Raman nel 192828. Da allora l'effetto Raman è stato ampiamente utilizzato nella microscopia ottica per la rilevazione di composizioni molecolari e tissutali senza marcatura esogena. Sia lo Stimulated Raman Scattering (SRS) che il coerente anti-Stokes Raman Scattering (CARS) eccitano le vibrazioni molecolari in modo coerente e sfruttano l'interazione non lineare della luce per produrre un segnale più forte rispetto alla convenzionale spettroscopia Raman spontanea. Il fenomeno SRS è stato segnalato per la prima volta nel 196229. Nel 2008, questo meccanismo è stato integrato nell'imaging multifotone tridimensionale, consentendo il rilevamento selettivo di sostanze chimiche in base alle variazioni di intensità nei fasci di Pump e Stokes dovute alle transizioni vibrazionali molecolari30. Questo metodo riduce al minimo l'interferenza di fondo non risonante, generando un segnale di intensità pulita che supera quello delle CARS. L'imaging SRS eccelle nel fornire immagini multiplex e iperspettrali, consentendo il rilevamento simultaneo di più legami chimici e consentendo la visualizzazione ad alta risoluzione della composizione molecolare in campioni con una notevole profondità di penetrazione. Sebbene sia una tecnica relativamente nuova, l'imaging SRS si è dimostrato efficace sia nella diagnostica clinica che nella ricerca metabolica, in vivo e in vitro 30,31,32,33,34,35,36. Ad esempio, la SRS può differenziare i tessuti infiltrati nel tumore cerebrale dalla corteccia e dalla sostanza bianca quantificando il rapporto lipidi/proteine, consentendo la delineazione dei margini tumorali in modo non invasivo e senza marcature37,38. Inoltre, le alterazioni metaboliche, spesso considerate segni distintivi di malattie legate all'invecchiamento e associate al cancro, possono essere valutate quantitativamente utilizzando la SRS ottenuta rilevando i legami carbonio-deuterio in campioni trattati con acqua pesante (D2O), consentendo la misurazione quantitativa della sintesi proteica, della lipogenesi e di altri processi metabolici macromolecolari 31,33,34,35,36. La capacità di tracciare i metaboliti con un'elevata risoluzione temporale e spaziale posiziona la SRS come uno strumento promettente per l'indagine e la diagnosi delle malattie, con un potenziale per applicazioni cliniche più ampie.
L'imaging multimodale è emerso come un approccio potente nella ricerca biomedica, integrando due o più modalità di imaging per ottenere una comprensione più completa di sistemi biologici complessi all'interno dello stesso campione. Nel 2018 è stata introdotta una tecnica di microscopia multiarmonica autofluorescenza senza marcatura (SLAM), che integra la fluorescenza a due fotoni (2PF), la fluorescenza a tre fotoni (3PF), l'SHG e la terza generazione armonica (THG)39. Questo approccio facilita la visualizzazione simultanea delle interazioni cellulari, dei processi dinamici e dei singoli componenti all'interno del microambiente tumorale. La microscopia SLAM offre una perturbazione minima e requisiti di potenza laser ridotti per il campione, consentendo la profilazione profonda dei tessuti e fornendo un metodo più sicuro per il monitoraggio intravitale40. Un'altra modalità multimodale, che combina la spettroscopia di fluorescenza intrinseca, la spettroscopia di riflettanza diffusa e la spettroscopia Raman, è stata sviluppata per il rilevamento del cancro in situ durante le procedure chirurgiche41. Inoltre, un sistema di endoscopia non lineare multimodale di recente progettazione, che integra CARS, SHG e fluorescenza a due fotoni (TPF), ha dimostrato la capacità di visualizzare campioni biologici con risoluzione spaziale sub-micronica e sub-cellulare42. La microscopia combinata 2PF e SRS è stata utilizzata in modo simile per l'imaging in vivo ad alta risoluzione di tessuti, cellule e organelli 42,43,44,45. Queste tecniche di imaging multimodale emergenti sfruttano i punti di forza delle singole modalità, portando a una migliore risoluzione, profondità di penetrazione ed efficienza di acquisizione delle immagini, mostrando così un notevole potenziale per applicazioni cliniche e chirurgiche.
Questo approccio multimodale è sempre più favorito rispetto all'imaging monomodale perché fornisce una gamma più ampia di misurazioni mitigando al contempo le limitazioni associate alle singole tecniche. Come discusso in precedenza, l'MPF misura la fluorescenza endogena per riflettere i cambiamenti metabolici, l'SHG può visualizzare strutture non centrosimmetriche come il collagene in campioni biologici e l'SRS rileva prevalentemente proteine e lipidi a causa dell'alta densità di legami chimici che generano segnali Raman distintivi in base alle loro modalità vibrazionali. Date le loro proprietà coerenti e il principio condiviso delle proprietà ottiche non lineari, queste modalità di imaging possono essere integrate in un'unica configurazione di microscopio utilizzando laser pulsati ultracorti, consentendo l'acquisizione di vari biomarcatori in regioni localizzate per fornire una visione più completa dei processi biologici44,45. Questo documento delinea un protocollo per l'implementazione di una piattaforma di imaging multimodale che integra MPF, SHG e SRS per applicazioni di ricerca biomedica.
Protocollo
1. Esperimenti di imaging multimodale label-free
NOTA: Questo protocollo si concentra sulla procedura di configurazione e acquisizione dell'imaging multimodale label-free.
- Configurazione del microscopio multimodale (Figura 1A) e calibrazione del sistema
- Riscalda il laser e attendi circa 15-20 minuti.
- Accendere le unità di controllo e i monitor nella seguente sequenza: Scatola di controllo | Controller con pannello a sfioramento | Adattatore CA per il telecomando laser principale Adattatore CA per il telecomando laser secondario.
- Accendere il rilevatore di fotodiodi Si e l'amplificatore di blocco.
- Configurare i raggi laser della pompa e il raggio Stokes. Configura il sistema laser con un raggio di pompa sintonizzabile da 780 nm a 990 nm, un'ampiezza dell'impulso di 5 - 6 ps e una frequenza di ripetizione di 80 MHz. Il raggio laser Stokes ha una lunghezza d'onda fissa di 1.031 nm con un impulso di 6 ps e una frequenza di ripetizione di 80 MHz . Assicurarsi che sia la pompa che i raggi Stokes siano a bassa potenza (almeno 20 mW) per essere visibili sulla piastra di allineamento.
- Posizionare una piastra di allineamento nel percorso ottico (subito dopo la scatola laser) per verificare la sovrapposizione spaziale delle pompe e dei raggi di Stokes e regolare lo specchio fino a quando entrambi i punti non si sovrappongono al centro della piastra di allineamento.
NOTA: Sebbene le pompe e i raggi di Stokes siano approssimativamente sovrapposti, la regolazione fine della sovrapposizione spaziale di due raggi laser può ottenere il segnale SRS ottimale. - Dopo la sovrapposizione spaziale, sintonizzarsi con il PSD (Position Sensitive Detector) facendo prima clic sul software di controllo OPO . Utilizzare il tasto esagonale per regolare con attenzione lo specchio ottico 1 (OM1) e osservare il display PSD per il cambio di posizione XY per mirare alla deviazione minima dal centro. Quindi, per effettuare regolazioni più precise, utilizzare il tasto esagonale sullo specchio ottico 2 (OM2) fino a quando l'indicatore di posizione non è centrato sul display PSD.
NOTA: OM1 e OM2 sono due specchi nella testa OPO per regolare l'allineamento del laser. - Infine, centrare il condensatore ruotando le due viti di centraggio del condensatore per spostare l'immagine del diaframma a iride al centro del campo visivo.
NOTA: Il diaframma dell'iride di campo può limitare il diametro del raggio di luce che entra nell'obiettivo e quindi escludere la luce estranea, migliorando il contrasto dell'immagine.
- Fasi di acquisizione dell'imaging multimodale senza marcatura
- Applicare l'olio sul condensatore d'olio ad alta apertura numerica (1,4 NA) e montare il vetrino del microscopio sul condensatore oliato. Infine, posizionare una grossa goccia d'acqua sul vetrino del microscopio per l'obiettivo ad acqua 25x. Assicurarsi che il microscopio sia montato in modo sicuro e inamovibile, quindi regolare il tavolino z per regolare la messa a fuoco fino a quando l'immagine in campo chiaro del campione biologico può essere vista sotto l'obiettivo dell'acqua 25x.
- Inizia il processo di imaging nella sequenza corretta: MPF, SHG e SRS per evitare il fotosbiancamento, che influirà sulla qualità dell'immagine e sull'analisi precisa dell'MPF. Per passare rapidamente da MPF a SHG, passare dal raggio della pompa al raggio fisso di Stokes.
NOTA: La regolazione del laser della pompa da 800 nm (autofluorescenza NADH e FAD) a 791,3 nm (spostamento CH3 Raman) può richiedere 1-2 minuti. - Seleziona la risoluzione dell'immagine (512 x 512 pixel) e il tempo di permanenza adatto per ciascuna modalità. Utilizzare 8 μs/pixel per MPF e SHG con fotogrammi medi superiori a 3. Utilizzare 40 μs/pixel con fotogramma medio 2 per la modalità SRS.
NOTA: Il fotogramma medio indica il numero di immagini di cui fare la media prima del salvataggio. - Per acquisire l'autofluorescenza con MPF, spegnere il raggio laser di Stokes e regolare il laser della pompa a 800 nm per eccitare NADH e flavina. Installare il cubo filtrante FVOPT nel percorso della luce con 460 ± 10 nm e 515 ± 10 nm rispettivamente per il NADH e la flavina. La potenza del campione è quindi di circa 15 mW.
- Acquisisci il segnale della fibra di collagene usando SHG. Spegnere il raggio laser della pompa, utilizzare solo il raggio laser Stokes e impostare la potenza su 500 mW. Acquisire solo il canale relativo al filtro a 515 nm.
- Ottenere la distribuzione spaziale di proteine e lipidi utilizzando SRS. Mantieni accesi entrambi i raggi laser e regola la frequenza del raggio laser in modo che corrisponda alla modalità vibrazionale specifica per ciascuna molecola.
NOTA: Tipicamente, 791,3 nm viene utilizzato per CH3, 797 nm viene utilizzato per CH2, 787 nm è per i lipidi insaturi e 794,6 è per i lipidi saturi. La potenza del campione è quindi di circa 40 mW. - Per acquisire i set di dati dell'immagine iperspettrale SRS, aprire il software di controllo OPO per selezionare lo sweep, impostare l'intervallo di lunghezze d'onda da 780 nm a 806,5 nm e scegliere un numero di stack di almeno 60. Quindi, acquisisci lo stack di immagini iperspettrali.
NOTA: Il set di dati risultante contiene 60 immagini, ognuna delle quali rappresenta la distribuzione spaziale a uno specifico spostamento Raman (da 2.700 cm-1 a 3.150 cm-1). Il tempo per l'acquisizione iperspettrale dipende dal campo visivo (FOV) dell'immagine e dal tempo di scansione, ma sarà approssimativamente compreso tra 5 minuti e 8 minuti per un'immagine 512 x 512. Per immagini più grandi, è necessario prestare attenzione a ridurre al minimo la durata sotto il laser per evitare fotodanni, che possono verificarsi dopo 2 ore (a seconda dello spessore del campione e delle dimensioni del FOV). - Salva tutte le immagini delle stesse aree di interesse (ROI) nella stessa cartella. Il formato dell'immagine è il file .oir Olympus.
2. Analisi delle immagini
- Utilizzare il software di elaborazione delle immagini per aprire tutte le immagini raw salvate per assegnare il colore e aggiungere la barra della scala per la visualizzazione dell'immagine. Usalo anche per generare la maschera binaria di NADH e immagini degli acidi grassi di saturazione (SFA, 2.880 cm-1) per l'analisi del rapporto redox ottico e dell'insaturazione lipidica.
NOTA: Assicurati che l'intensità della maschera abbia solo 0 e 1. - Usa lo script python costruito in casa per eseguire qualsiasi analisi a valle (il codice è disponibile all'indirizzo https://github.com/lingyanshi2020/HSI_Analysis).
NOTA: questo script è progettato per elaborare più regioni di interesse (ROI) sia da campioni di controllo che da campioni di cancro. La funzione principale dello script richiede solo due input: i percorsi dei file per le cartelle di controllo e le cartelle delle immagini del cancro.- Scorri le cartelle, abbinando le immagini con lo stesso nome di base (ad esempio, "roi_1_flavin.tif", "roi_1_nadh.tif", "roi1_mask.tif").
- Per ogni set di immagini corrispondenti, lasciare che lo script esegua l'analisi raziometrica e generi nuove immagini raziometriche in base a questi calcoli.
Il rapporto redox ottico è calcolato come flavina / (NADH + flavina).
L'insaturazione lipidica è calcolata come: USFA / (SFA + USFA) dove gli USFA rappresentano acidi grassi insaturi. - Tutti i valori raziometrici delle immagini vengono quindi memorizzati in un DataFrame pandas per una facile manipolazione. Esegui l'ANOVA unidirezionale e usa matplotlib con styple "ggplot" o seaborn per eseguire il barplot o il boxplot per la quantificazione.
- Utilizzare le immagini da 2.930 cm-1 (CH3) e 2.850 cm-1 (CH2) per generare un'immagine sottratta (CH3 - CH2). Quindi, fondere l'immagine CH2 CH3 - CH2 nello spazio colore RGB con una LUT personalizzata che imita la colorazione H&E per generare l'immagine istologia digitale46.
- Dopo l'acquisizione di immagini SRS iperspettrali, utilizzare lo script python costruito in casa per eseguire il clustering k-means per segmentare le immagini in base alle somiglianze spettrali (il codice è disponibile all'indirizzo https://github.com/lingyanshi2020/HSI_Analysis).
- Carica le pile di immagini .tiff con il pacchetto tifffile. Rimodellare lo stack di immagini iperspettrali SRS (x, y, spettri) in un array 2D (pixel, spettri).
- Utilizzare il pacchetto scikit-learn per distribuire l'algoritmo k-means in questa matrice 2D, con il numero definito di cluster.
- Rimodella le etichette del cluster risultanti riportandole alle dimensioni originali dell'immagine, creando una mappa di segmentazione.
- Assegna colori univoci a ciascun cluster, generando un'immagine in falsi colori che rappresenta diverse composizioni biochimiche all'interno del tessuto.
- Usa matplotlib per tracciare gli spettri di ogni cluster con il valore medio (linea continua) e la deviazione standard (area d'ombra)
- Seguire il protocollo del protocollo PRM-SRS (Penalized Reference Matching SRS) utilizzando MATLAB per creare il rilevamento spaziale del sottotipo lipidico senza etichetta con set di dati di immagini iperspettrali SRS47.
- Organizza le immagini e salvale in un formato .tif a 300 dpi.
Risultati
Le immagini in Figura 2 sono rappresentative dei risultati ottenuti seguendo il protocollo per l'acquisizione dell'autofluorescenza di FAD e NADH, nonché dei quattro canali SRS per proteine, lipidi totali, lipidi insaturi e lipidi saturi. Qui generiamo anche un'immagine pseudo-istologica come descritto nel protocollo attraverso la fusione dei colori RGB. L'acquisizione dei canali MPF e SRS fornisce i file di immagine che verranno successivamente utilizzati nell'analisi raziometrica. Un esempio di questa analisi può essere visto nella Figura 3 sul tessuto polmonare umano. Dopo l'acquisizione delle immagini nel protocollo, la nostra metodologia di analisi delle immagini che utilizza Python o ImageJ sfrutta il rapporto tra canali distinti per fornire informazioni metaboliche quantitative. Come mostrato nelle immagini dell'insaturazione lipidica e del rapporto redox ottico della Figura 3, l'analisi raziometrica fornisce una mappa a colori della distribuzione dell'attività metabolica relativa e della composizione molecolare. Abbiamo sfruttato queste misurazioni per fare osservazioni riguardanti i cambiamenti nelle vie metaboliche e nel contenuto lipidico di tessuti specifici, patologie o fattori biologici distinti. Come si vede nella Figura 4, questo può fornire un confronto quantitativo del tessuto sano e tumorale confrontando i valori medi di stress ossidativo e insaturazione lipidica oltre alla sua distribuzione in un'immagine di microscopia 2D.
Per l'analisi raziometrica delle immagini MPF e SRS, miriamo comunemente al rapporto di ossidoriduzione ottica e di insaturazione, come mostrato dall'Eq (1) e (2).
 (1)
(1)
 (2)
(2)
Dove IC è l'intensità dei pixel del canale C. Pertanto, misuriamo il rapporto per pixel e acquisiamo la distribuzione spaziale di questi marcatori metabolici e molecolari.
Oltre all'analisi raziometrica, la Figura 4 mostra anche un'altra potenziale via di applicazione per la nostra piattaforma multimodale: l'analisi iperspettrale delle immagini. Come descritto in questo protocollo, possiamo acquisire un'immagine iperspettrale SRS eseguendo uno sweep su più lunghezze d'onda del laser utilizzando un fascio di pompa sintonizzabile. Questo ci permette di ricostruire la regione CH dello spettro di spostamento Raman per ogni pixel all'interno dell'immagine microscopica, unendo sia le informazioni chimiche fornite dalla spettroscopia Raman che le informazioni spaziali fornite dalla microscopia ottica. Nel flusso di lavoro per l'analisi delle immagini, evidenziamo due tecniche implementate per l'analisi di queste immagini iperspettrali: PRM-SRS per la rivelazione di biomolecole e clustering k-means. La Figura 4E mostra l'applicazione di PRM-SRS su un'immagine di un tumore epatico di topo. L'algoritmo PRM-SRS fornisce la correlazione tra gli spettri per ogni singolo pixel, regolata da una penalità per gli spostamenti nello spettro Raman; L'algoritmo genera quindi immagini di distribuzione di probabilità per ciascuno dei lipidi.
Il clustering K-means degli spettri dei pixel è un'altra tecnica che implementiamo per visualizzare il raggruppamento di specifici fenotipi spettrali, consentendo la separazione per concentrazione di legame e composizione molecolare attraverso l'intensità e la forma spettrale rispettivamente. La Figura 5 fornisce un esempio di come l'analisi delle immagini tramite il clustering k-means possa essere applicata nel nostro protocollo. Ci aspettiamo che i cluster k-media si allineino strutturalmente con le caratteristiche delle figure pseudo-istologiche, oltre a fornire ulteriori raggruppamenti da caratteristiche che non sono così facilmente distinguibili né dai canali SRS né dalla colorazione istologica pseudo-ematossilina ed eosina (H&E). L'analisi di clustering PRM-SRS e k-means completa questa piattaforma di imaging label-free fornendo informazioni chimiche e semi-targettizzabili relative ad analiti specifici e legami molecolari senza la necessità di sondaggi esogeni.
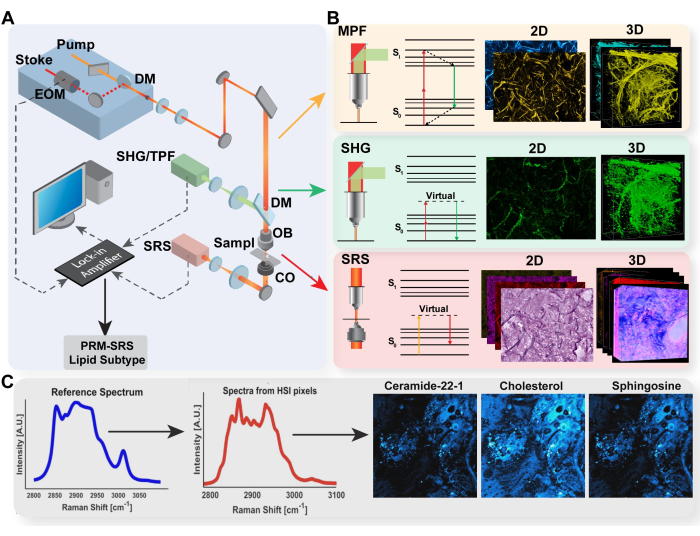
Figura 1: Diagramma della piattaforma di imaging multimodale e del rilevamento di biomolecole PRM-SRS. (A) Schema della configurazione hardware per i sistemi di imaging MPF/TPF, SHG e SRS. (B) Percorso luminoso e diagramma di Jablonski per TPF, SHG e SRS con immagini acquisite rappresentative per ciascuna modalità. (C) Diagramma del flusso di lavoro PRM-SRS per la corrispondenza del riferimento spettrale tra spettroscopia Raman spontanea e imaging iperspettrale SRS. Abbreviazioni: MPF = fluorescenza multifotonica; TPF = fluorescenza a due fotoni; SHG = generazione di seconda armonica; SRS = scattering Raman stimolato; PRM = corrispondenza dei riferimenti penalizzata. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Canali MPF e SRS dal tessuto della corteccia motoria primaria umana (M1). (A,B) Canali di autofluorescenza MPF per Flavina/FAD e NADH. (C-F) Canali SRS catturati in corrispondenza di specifici picchi di spostamento Raman; il picco di stretching asimmetrico CH3 (2.930 cm-1) per le proteine, il picco di stretching asimmetrico CH2 (2.845 cm-1) per i lipidi, il picco di 2.885 cm-1 per gli acidi grassi saturi e il picco di 3.010 cm-1 per gli acidi grassi insaturi. (G) Fusione dei canali proteici SRS (blu) e lipidici (verdi) per delineare la loro rispettiva distribuzione spaziale. (H) Immagini pseudo-istologiche SRS (SRH) che rispecchiano la colorazione H&E. Barre di scala = 20 μm. Abbreviazioni: MPF = fluorescenza multifotonica; FAD = flavina adenina dinucleotide; NADH = nicotinammide adenina dinucleotide ridotto; SRS = scattering Raman stimolato; SRH = Istologia SRS. Clicca qui per visualizzare una versione più grande di questa figura.
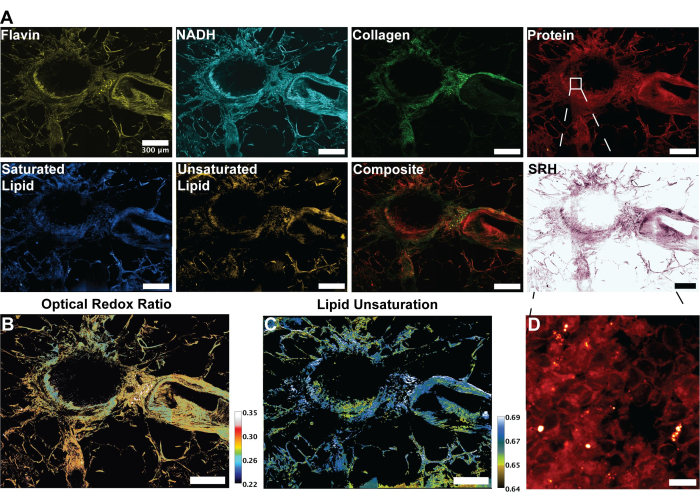
Figura 3: Immagini MPF, SHG e SRS e analisi del tessuto polmonare umano. (A) Picchi di autofluorescenza MPF per flavina/FAD e NADH, segnale SHG per fibre di collagene, segnale SRS per proteine (2.930 cm-1), lipidi (2.845 cm-1), lipidi saturi (2.885 cm-1) e lipidi insaturi (3.010 cm-1) e un'immagine composita unita delle molteplici modalità. (B) Immagine raziometrica del rapporto redox ottico. (C) Immagine raziometrica dell'insaturazione lipidica. (D) Immagine ingrandita delle cellule epiteliali nello strato interno dei bronchi. Barre di scala = 200 μm. Abbreviazioni: MPF = fluorescenza multifotonica; SHG = generazione di seconda armonica; SRS = scattering Raman stimolato; FAD = flavina adenina dinucleotide; NADH = nicotinammide adenina dinucleotide ridotta. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Analisi raziometrica e iperspettrale di tumori epatici di topo. (A) Immagini in autofluorescenza MPF per NAD[P]H e FAD, canali SRS per proteine totali, lipidi saturi e insaturi e immagini pseudo-istologiche per fegato di topo di controllo sano (in alto) e tessuto tumorale (in basso). (B) Stress ossidativo (giallo-verde), misurato dal rapporto redox ottico, e insaturazione lipidica (blu-rosso) calcolata dai tessuti di controllo (a sinistra) e tumorali (a destra). Barre della scala = 100 μm. (C,D) Grafici a barre che dimostrano le differenze raziometriche nello stress ossidativo e nell'insaturazione lipidica tra campioni di fegato di controllo (blu) e tumorali (rosso). Statistiche di significatività: * per una significatività di p ≤ 0,05, ** per una significatività di p ≤ 0,01. (E) Risultati dell'analisi dei sottotipi lipidici PRM-SRS da immagini iperspettrali. Da sinistra a destra: canale proteico SRS per riferimento strutturale, barra della scala = 100 μm, immagine della distribuzione di probabilità dei sottotipi lipidici (TAG 18:1, colesterolo e ceramide C24:0). Grafico degli spettri di riferimento del sottotipo lipidico e degli spettri medi dei pixel da SRS HSI nella regione CH. Abbreviazioni: MPF = fluorescenza multifotonica; SRS = scattering Raman stimolato; NAD[P]H = nicotinammide adenina dinucleotide fosfato ridotto; FAD = flavina adenina dinucleotide; HSI = immagini iperspettrali. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Clustering iperspettrale k-means SRS sulla corteccia motoria primaria umana (M1). (A-F) Sei regioni rappresentative che mostrano: a sinistra: immagini di istologia Raman stimolata (SRH) che forniscono una visualizzazione tissutale simile a H&E; al centro: corrispondenti risultati del clustering k-means; A destra: profili spettrali Raman specifici dell'ammasso con intensità medie (linee continue) e deviazioni standard (aree ombreggiate). Numero di baridi che varia da 4 a 6 medie. È stato utilizzato il clustering non supervisionato, quindi colori diversi nelle immagini raggruppate rappresentano composizioni chimiche distinte identificate dall'algoritmo k-means. Ogni cluster (rappresentato da un colore univoco) corrisponde a regioni con profili spettrali simili nella regione di allungamento CH. Barra della scala = 20 μm. Abbreviazioni: SRH = Istologia SRS. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo sistema multimodale è una potente piattaforma di imaging per l'acquisizione di una visualizzazione olistica dell'ambiente molecolare dei campioni in un'ampia gamma di origini biologiche e condizioni patologiche. Il vantaggio di sfruttare diverse modalità label-free risiede nella capacità di acquisire informazioni complementari e indirizzare analiti specifici che altrimenti potrebbero essere difficili o impossibili in un'unica tecnica di imaging label-free. In particolare, le tre tecniche di imaging non lineare (SRS, MPF, SHG) menzionate in questo articolo consentono la quantificazione della composizione delle macromolecole, la dinamica dell'energia tramite il rapporto redox ottico e le informazioni strutturali, tra cui la composizione e la morfologia della matrice extracellulare 6,48,49. Inoltre, rispetto all'utilizzo separato di ogni singola modalità di imaging, l'imaging con un unico sistema di microscopio combinato consente la registrazione immediata delle immagini e tempi di conservazione dei campioni più brevi. Abbiamo anche condotto l'imaging di cellule vive con questo approccio, probabilmente a causa della minore fotoesposizione rispetto alla microscopia a fluorescenza confocale, utilizzando punti temporali selezionati per acquisire ciascuna modalità in sequenza come stabilito nel protocollo di cui sopra. Oltre ai vantaggi individuali, la piattaforma multimodale beneficia anche delle molteplici prospettive sulle condizioni biologiche attraverso la misurazione senza marcatura di diversi biomarcatori.
L'imaging label-free di proteine autofluorescenti tramite MPF è stato principalmente diretto alla quantificazione del rapporto redox ottico, una misura delle reazioni redox facilitate dall'ossidazione di FAD in FAD+ e dalla riduzione di NAD+ in NADH50. Questo è un marcatore significativo del metabolismo all'interno delle cellule e dei tessuti in quanto fornisce una misura dell'attività relativa tra fosforilazione ossidativa e glicolisi, le due principali vie perla generazione 6 di ATP. In particolare, le diminuzioni della concentrazione di NADH e gli aumenti di FAD+ sono un marker per l'aumento della fosforilazione ossidativa nei mitocondri; Nel frattempo, è vero il contrario per l'aumento della glicolisi nel citoplasma. La tendenza a preferire una via metabolica rispetto all'altra nella produzione di energia è stata collegata a diversi cambiamenti patologici e attivazioni di vie legate al cancro, evidenziando il potenziale uso dell'ORR come marcatore di diagnosi precoce 6,51.
Allo stesso modo, l'acquisizione del segnale del collagene fibroso SHG sfrutta una modalità di imaging non lineare label-free per quantificare e visualizzare un marcatore biologico per la salute. L'SHG è in grado di tracciare efficacemente la distribuzione del collagene di tipo I-III grazie alla sua struttura non centrosimmetrica. Le proteine fibrose del collagene misurate da SHG sono state riconosciute come importanti marcatori diagnostici per diverse malattie, tra cui il cancro e la fibrosi 25,26,27,52,53. Oltre al suo rilevamento come marcatore di malattia, la nostra acquisizione di collagene tramite SHG può fungere da forte indicatore della struttura nelle cellule e nei tessuti a causa del ruolo del collagene fibroso nella composizione della matrice extracellulare e dei confini strutturali tra i tessuti54,55. Attraverso il segnale del collagene SHG, possiamo quindi effettuare osservazioni informate di cellule separate o unità funzionali tissutali distinte senza la necessità di un marcatore esogeno per le membrane cellulari. Nel complesso, la quantificazione SHG delle fibrille di collagene presenta un chiaro vantaggio sia per la capacità diagnostica che per la facilità di analisi quando integrata con altre tecniche di imaging label-free.
Utilizzando la microscopia SRS, possiamo catturare diversi marcatori per la composizione delle macromolecole, in particolare per i lipidi e gli acidi grassi. In questo protocollo, descriviamo come acquisire il segnale SRS per acidi grassi insaturi e saturi per l'analisi raziometrica dell'insaturazione lipidica all'interno dello stesso campione. È stato applicato un microscopio a scansione laser verticale con un obiettivo ad acqua 25x per una produttività nel vicino infrarosso. I raggi laser sono passati attraverso il campione e sono stati raccolti da un condensatore d'olio ad alta apertura numerica (1,4 NA). Un filtro passa-corto ad alto diametro esterno (950 nm) è stato utilizzato per bloccare il fascio di Stokes, consentendo solo al raggio della pompa di raggiungere un rivelatore a fotodiodo Si per rilevare il segnale di perdita Raman stimolato. La corrente di uscita dal fotodiodo è stata terminata e filtrata. Successivamente, un amplificatore lock-in a 20 MHz ha demodulato la corrente in X con uno sfasamento zero, e un modulo software ha utilizzato il segnale demodulato per generare l'immagine durante la scansione laser.
Gli studi hanno dimostrato che i cambiamenti nell'insaturazione lipidica hanno un profondo effetto sulla membrana cellulare e degli organelli e una disregolazione nel metabolismo dei lipidi può essere un forte marcatore per diverse malattie, tra cui il cancro e le malattie neurodegenerative 56,57,58,59. Per una panoramica più completa della disregolazione lipidica, analizziamo la composizione del sottotipo utilizzando l'imaging iperspettrale SRS. Le immagini iperspettrali SRS (HSI) combinano le informazioni spaziali ad alta risoluzione acquisite dalla microscopia SRS con il segnale chimicamente significativo della spettroscopia Raman attraverso la sintonizzazione sequenziale come descritto nel protocollo. Questi spettri di pixel vengono quindi analizzati in due metodi, clustering tramite k-means e rilevamento di biomolecole con PRM-SRS.
Il clustering K-means degli spettri dei pixel determina la separazione delle regioni all'interno di un'immagine 2D in base alla concentrazione e alla composizione molecolare a causa della relazione lineare tra l'intensità del segnale SRS e la concentrazione del legame molecolare60,61. Attraverso questa metodologia, applichiamo il protocollo per accertare le regioni che possiedono fenotipi molecolari specifici come prescritto dagli spettri dei pixel e dal centroide spettrale condiviso. Per un esame approfondito dell'espressione molecolare distinta in pixel specifici, implementiamo PRM-SRS per eseguire la corrispondenza spettrale tra uno spettro di riferimento per una molecola specifica catturata con la spettroscopia Raman spontanea e gli spettri dei pixel dall'acquisizione SRS HSI. In tal modo, possiamo determinare la probabilità di espressione per molecole distinte senza utilizzare sonde o etichette esogene. Questa analisi ha dimostrato la capacità di tracciare alcuni sottotipi lipidici attraverso tessuti e modelli biologici e la capacità di identificare cambiamenti patologici nei cambiamenti di composizione lipidica e quindi dislipidemia47. Inoltre, i recenti progressi nel miglioramento della risoluzione spettrale attraverso la scissione spettrale della propagazione dell'impulso di picosecondi attraverso le fibre di silice incentivano ulteriormente l'applicazione di SRS HSI come modalità per la visualizzazione biochimica senza marcatura62. Attraverso questi metodi evidenziati nel protocollo, stabiliamo una piattaforma per eseguire analisi raziometriche e spettrali complete utilizzando una modalità SRS label-free.
Oltre alla relativa complessità e novità di queste tecniche, ci sono alcuni importanti limiti nel nostro approccio da considerare. L'acquisizione di immagini MPF con un laser pulsato a picosecondi, a differenza dei laser a femtosecondi comunemente utilizzati, richiede una maggiore potenza di eccitazione del fluoroforo e rischia il fotosbiancamento. Con questo in mente, abbiamo ottimizzato il protocollo di cui sopra per mitigare il potenziale fotodanno dalla nostra piattaforma riducendo l'incidente di potenza laser sul campione. Inoltre, mentre studi precedenti hanno dimostrato la capacità di valutare il rapporto redox ottico dall'imaging in autofluorescenza MPF di NADH e FAD, la microscopia per imaging a fluorescenza (FLIM) ha dimostrato una maggiore accuratezza nella valutazione del metabolismo energetico 63,64,65. Ciò è dovuto alla capacità di differenziare le concentrazioni legate alle proteine e non legate di NADH e FAD in base ai loro distinti tempi di vita in fluorescenza, Rispetto alle capacità di MPF, ciò ha permesso a FLIM di essere utilizzato per studi completi sull'attività metabolica da diverse vie energetiche, tra cui la fosforilazione ossidativa e la glicolisi 16,63,64,65,66,67, 68. Sebbene questo protocollo escluda FLIM, ci aspettiamo che la modalità venga integrata in questa piattaforma di imaging nel prossimo futuro, migliorando l'analisi del metabolismo energetico che attualmente eseguiamo. Tuttavia, con il protocollo qui stabilito, presentiamo un flusso di lavoro e una configurazione hardware per un approccio di imaging multimodale in grado di misurare il metabolismo della struttura biologica da più prospettive.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Ringraziamo la dottoressa Gloria Pryhuber e i membri del suo team HuBMAP per aver fornito fette di tessuto polmonare umano. Ringraziamo il Dr. Kun Zhang per aver fornito tessuti cerebrali umani. Ringraziamo anche il Dr. Gen-Sheng Feng per aver fornito campioni di fegato di topo. Ringraziamo il supporto di NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, UCSD Startup funds, Sloan Research Fellow Award e CZI DAF2023-328667 Award.
Materiali
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Riferimenti
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon