Method Article
Multimodale optische Bildgebungsplattform zur Untersuchung des zellulären Stoffwechsels
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Unter Verwendung einer multimodalen Plattform, die markierungsfreie optische Bildgebungsmodalitäten kombiniert, haben wir ein Protokoll zur Visualisierung und Quantifizierung der zellulären Dynamik und des Stoffwechsels entwickelt. Durch Bildgebung mittels Multiphotonenfluoreszenz, Erzeugung der zweiten Harmonischen und stimulierte Raman-Streumikroskopie können wir einen ganzheitlichen Überblick über die zelluläre und molekulare Umgebung erhalten.
Zusammenfassung
Optische Bildgebungstechnologien sind in biomedizinischen Studien von entscheidender Bedeutung, da sie sowohl morphologische als auch funktionelle Informationen aus biologischen Proben mit hoher räumlicher Auflösung erhalten können. Diese optischen Prozesse nutzen verschiedene Licht-Molekül-Wechselwirkungen wie Streuung, Absorption, Emission und harmonische Erzeugung zwischen Photonen und den Molekülen in Zellen, Geweben oder Organen. Während sich die konventionelle biomedizinische Bildgebung in der Vergangenheit auf die Anwendung einer einzigen Modalität konzentriert hat, haben neuere Forschungen gezeigt, dass diese verschiedenen Techniken komplementäre Erkenntnisse liefern und ihre kombinierten Ergebnisse ein umfassenderes Verständnis der molekularen Veränderungen in Alterungsprozessen und der Krankheitsentwicklung sowie der Grundlagen der Zellbiologie bieten.
In den letzten Jahrzehnten haben sich markierungsfreie optische Bildgebungsverfahren weiterentwickelt, die eine detaillierte Erforschung zellulärer und subzellulärer Umgebungen ermöglichen. So ermöglicht die Multiphotonenfluoreszenz (MPF) nicht nur die gezielte Bildgebung von Proteinen, sondern quantifiziert auch die Stoffwechselaktivität durch autofluoreszierende Coenzyme, wodurch eine hohe Eindringtiefe und räumliche Auflösung erreicht wird. Die zweite harmonische Erzeugung (SHG) wird verwendet, um Strukturen wie Kollagen in der extrazellulären Matrix abzubilden, während die stimulierte Raman-Streuung (SRS) chemische Bindungen und molekulare Zusammensetzung in situ mit subzellulärer Auflösung abbildet.
Wir haben eine multimodale Bildgebungsplattform entwickelt, die MPF-, SHG- und SRS-Modalitäten kombiniert. Die Integration dieser Modalitäten in eine einzige Plattform ermöglicht die Erfassung facettenreicher Informationen aus derselben Lokalisation in Zellen, Geweben, Organen oder sogar Körpern und ermöglicht so eine detailliertere Erforschung der komplizierten Beziehungen zwischen dem Zellstoffwechsel, der extrazellulären Matrixstruktur und der molekularen Zusammensetzung. Dieses multimodale System bietet subzelluläre Auflösung, tiefe Gewebepenetration, In-situ-Bildgebung von lebenden Zellen/Geweben sowie markierungsfreie Detektion und sofortige Koregistrierung, ohne dass Positionsanpassungen, Gerätewechsel oder Ausrichtung nach der Analyse erforderlich sind. Hier stellen wir ein Protokoll für die markierungsfreie Bildgebung mit dieser multimodalen Plattform vor und demonstrieren seine Anwendung bei der Charakterisierung des zellulären Stoffwechsels und der molekularen Heterogenität in Zellen und Geweben zur Untersuchung von Alterung und Krankheiten.
Einleitung
Die optische biomedizinische Bildgebung hat entscheidend dazu beigetragen, unser Verständnis der biologischen Struktur und Funktion zu verbessern. Die Bilder werden durch die Modulation von Anregungslicht und die Detektion von Signalen aus Licht-Gewebe-Wechselwirkungen erzeugt. Das erste zusammengesetzte Mikroskop, das um 1590 von Hans und Zacharias Janssen entwickelt wurde, verwendete zwei konvexe Linsen in einem Tubus, die eine Vergrößerung von bis zu 30x1 ermöglichten. Moderne optische Mikroskope können nach jahrhundertelangen Fortschritten heute Auflösungen von bis zu 1-3 nmerreichen 2,3. Fortschrittliche Bildgebungssysteme bieten nicht nur eine hohe Auflösung, sondern bieten jetzt auch eine tiefere Gewebepenetration, eine höhere Effizienz und minimale Probenschäden, wodurch sie sich besonders für die Bildgebung von lebenden Zellen und Geweben eignen. Die markierungsfreie Bildgebung ist besonders vorteilhaft, da sie Informationen erfasst, ohne intrazelluläre Prozesse zu stören oder die Probenintegrität zu beeinträchtigen.
Die Multiphotonen-Fluoreszenzmikroskopie (MPF), insbesondere die Zwei-Photonen-Fluoreszenzmikroskopie, wird in großem Umfang für die markierungsfreie Bildgebung eingesetzt. Im Gegensatz zur herkömmlichen Fluoreszenzmikroskopie, die auf linearer Einzelphotonenabsorption und -emission beruht, beinhaltet die MPF-Anregung die gleichzeitige Absorption mehrerer Photonen, deren kombinierte Energie ein einzelnes Fluorophormolekülanregt 4,5. Diese Photonen, typischerweise im Infrarotspektrum, besitzen die Hälfte oder weniger der Energie, die für die Einzelphotonenanregung erforderlich ist. Die längeren Wellenlängen und die lokalisierte Anregung im Brennpunkt dieses nichtlinearen Prozesses führen zu einer geringeren Streuung, einer tieferen Gewebepenetration und einer verringerten Phototoxizität.
Zelluläre Stoffwechselinformationen können durch markierungsfreie MPF-Mikroskopie durch den Nachweis von Autofluoreszenzsignalen von endogenen Stoffwechselsubstraten wie reduziertem Nicotinamid-Adenin-Dinukleotid (NADH) und Flavinadenin-Dinukleotid (FAD) erfasst werden. Diese Coenzyme weisen unterschiedliche Anregungs- und Emissionsspektren auf, und ihr Fluoreszenzintensitätsverhältnis, das als Redoxverhältnis (NADH/FAD) bekannt ist, spiegelt den oxidativen Zustand der Zelle wider. Seit Britton Chance 1979 das Konzept des Redox-Verhältnisses erstmals eingeführt hat, wurden zusätzliche Verhältnisse, darunter NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) und FAD/(FAD + NAD(P)H), vorgeschlagen 6,7,8,9. Die Quantifizierung dieser optischen Redoxverhältnisse mittels MPF-Bildgebung liefert wertvolle Einblicke in die Stoffwechseldynamik. Zum Beispiel kann die MPF-Bildgebung Krebszellen von normalen Zellen anhand ihres veränderten Stoffwechsels unterscheiden, was ihr Potenzial für die Krebsdiagnose demonstriert 10,11,12. Die MPF-basierte Autofluoreszenzdetektion hat jedoch Grenzen. Andere intrinsische Fluorophore, wie z. B. Keratin, können zur Fluoreszenzintensität beitragen, was zu spektralem Übersprechen und ungenauer Signalinterpretation führt13. Darüber hinaus spiegelt das Redoxverhältnis nur die gesamten zellulären Oxidations-Reduktionsänderungen wider und unterscheidet nicht zwischen NADH aus verschiedenen Quellen (z. B. zytoplasmatisch oder mitochondrial) oder zwischen NADH und NAD(P)H, da beide ähnliche spektrale Peaks bei 450 nm aufweisen, was zu gemischten Intensitätssignalen führt14.
Second Harmonic Generation (SHG), ein nichtlineares optisches Verfahren, das erstmals in den 1980er Jahren im biomedizinischen Bereich demonstriert wurde, wurde in großem Umfang für die markierungsfreie Abbildung zellulärer Strukturen eingesetzt15,16. Ähnlich wie bei der MPF werden bei der SHG zwei Photonen gleicher Energie von einem ultraschnell gepulsten Laser absorbiert. Diese Photonen werden rekombiniert, um ein neues Photon mit der doppelten Frequenz des einfallenden Lichts zu emittieren, was zur Detektion des Signals der zweiten Harmonischen führt. Diese nichtlineare optische Wechselwirkung tritt ausschließlich in nicht-zentrosymmetrischen Materialien auf, die eine Suszeptibilität ungleich Null zweiter Ordnung aufweisen, eine Polarisation zur Erzeugung des zweiten harmonischen Signals zu induzieren17,18. Dies macht SHG besonders effektiv für die Abbildung von filamentösen Proteinen und fibrillären Strukturen, wie Kollagen, Myosin und Tubulin, ohne dass exogene Fluoreszenzfarbstoffe erforderlich sind 15,17,19,20. Die Anomalie in Bezug auf Abundanz, Steifigkeit, Ausrichtung und Struktur von Fibrose und Kollagen ist bei vielen Erkrankungen wie Entzündungen und Krebs weit verbreitet, was SHG zu einem vielversprechenden Werkzeug für die effiziente und nicht-invasive Erkennung bestimmter Krankheitszustände macht 21,22,23. Die weit verbreitete Anwendung der SHG-Bildgebung in der onkologischen Forschung, einschließlich Studien zu Brust-, Eierstock- und Hautkrebs, hat ihre entscheidende Rolle sowohl in der Grundlagenforschung als auch in potenziellen klinischen Anwendungen hervorgehoben 24,25,26,27.
Verschiedene Moleküle weisen unterschiedliche Schwingungsenergieniveaus auf, die bei Anregung durch einfallendes Licht unterschiedliche Grade an inelastischer Streuung hervorrufen - ein Phänomen, das erstmals 1928 von C. V. Raman charakterisiertwurde. Der Raman-Effekt wird seither in der optischen Mikroskopie ausgiebig genutzt, um Molekül- und Gewebezusammensetzungen ohne exogene Markierung nachzuweisen. Sowohl die stimulierte Raman-Streuung (SRS) als auch die kohärente Anti-Stokes-Raman-Streuung (CARS) regen molekulare Schwingungen kohärent an und nutzen die nichtlineare Wechselwirkung des Lichts, um ein stärkeres Signal im Vergleich zur herkömmlichen spontanen Raman-Spektroskopie zu erzeugen. Das SRS-Phänomen wurde erstmals 1962 berichtet29. Im Jahr 2008 wurde dieser Mechanismus in die dreidimensionale Multiphotonen-Bildgebung integriert, was eine selektive Detektion von Chemikalien auf der Grundlage von Intensitätsänderungen in den Pump- und Stokes-Strahlen aufgrund molekularer Schwingungsübergängeermöglicht 30. Diese Methode minimiert nicht-resonante Hintergrundinterferenzen und erzeugt ein sauberes Intensitätssignal, das das von CARS übertrifft. Die SRS-Bildgebung zeichnet sich durch Multiplex- und Hyperspektralbildgebung aus, die den gleichzeitigen Nachweis mehrerer chemischer Bindungen ermöglicht und eine hochauflösende Visualisierung der molekularen Zusammensetzung in Proben mit beträchtlicher Eindringtiefe ermöglicht. Obwohl es sich um eine relativ neue Technik handelt, hat sich die SRS-Bildgebung sowohl in der klinischen Diagnostik als auch in der Stoffwechselforschung sowohl in vivo als auch in vitro als wirksam erwiesen 30,31,32,33,34,35,36. Zum Beispiel kann SRS Hirntumor-infiltriertes Gewebe von der Hirnrinde und der weißen Substanz unterscheiden, indem es das Lipid-Protein-Verhältnis quantifiziert, was die Abgrenzung von Tumorrändern auf markierungsfreie, nicht-invasive Weise ermöglicht37,38. Darüber hinaus können metabolische Veränderungen, die oft als Kennzeichen altersbedingter und krebsassoziierter Krankheiten angesehen werden, mit Hilfe von SRS quantitativ bewertet werden, indem Kohlenstoff-Deuterium-Bindungen in Proben nachgewiesen werden, die mit schwerem Wasser (D2O) behandelt wurden, was eine quantitative Messung der Proteinsynthese, der Lipogenese und anderer makromolekularer Stoffwechselprozesse ermöglicht 31,33,34,35,36. Die Fähigkeit, Metaboliten mit hoher zeitlicher und räumlicher Auflösung zu verfolgen, positioniert SRS als vielversprechendes Werkzeug für die Erforschung und Diagnose von Krankheiten mit Potenzial für breitere klinische Anwendungen.
Die multimodale Bildgebung hat sich zu einem leistungsstarken Ansatz in der biomedizinischen Forschung entwickelt, bei dem zwei oder mehr Bildgebungsmodalitäten integriert werden, um ein umfassenderes Verständnis komplexer biologischer Systeme innerhalb derselben Probe zu erlangen. Im Jahr 2018 wurde eine markierungsfreie Autofluoreszenz-Multiharmonische (SLAM)-Mikroskopietechnik eingeführt, die Zwei-Photonen-Fluoreszenz (2PF), Drei-Photonen-Fluoreszenz (3PF), SHG und dritte Harmonische (THG) integriert39. Dieser Ansatz ermöglicht die gleichzeitige Visualisierung von zellulären Interaktionen, dynamischen Prozessen und einzelnen Komponenten innerhalb der Tumormikroumgebung. Die SLAM-Mikroskopie bietet minimale Störungen und reduzierte Anforderungen an die Laserleistung der Probe, ermöglicht eine tiefe Gewebeprofilierung und bietet eine sicherere Methode für das intravitale Monitoring40. Eine weitere multimodale Modalität, die intrinsische Fluoreszenzspektroskopie, diffuse Reflexionsspektroskopie und Raman-Spektroskopie kombiniert, wurde für die In-situ-Krebserkennung bei chirurgischen Eingriffen entwickelt41. Darüber hinaus hat ein kürzlich entwickeltes multimodales nichtlineares Endoskopiesystem, das CARS, SHG und Zwei-Photonen-Fluoreszenz (TPF) integriert, die Fähigkeit demonstriert, biologische Proben mit räumlicher Auflösung im Submikrometer- und subzellulären Bereich abzubilden42. Die kombinierte 2PF- und SRS-Mikroskopie wurde in ähnlicher Weise für die hochauflösende In-vivo-Bildgebung von Geweben, Zellen und Organellen eingesetzt 42,43,44,45. Diese aufkommenden multimodalen Bildgebungsverfahren nutzen die Stärken einzelner Modalitäten, was zu einer verbesserten Auflösung, Eindringtiefe und Bildaufnahmeeffizienz führt und somit ein erhebliches Potenzial für klinische und chirurgische Anwendungen aufweist.
Dieser multimodale Ansatz wird zunehmend gegenüber der einzelmodalen Bildgebung bevorzugt, da er ein breiteres Spektrum an Messungen bietet und gleichzeitig die mit einzelnen Techniken verbundenen Einschränkungen abschwächt. Wie bereits erwähnt, misst MPF die endogene Fluoreszenz, um metabolische Veränderungen widerzuspiegeln, SHG kann nicht-zentrosymmetrische Strukturen wie Kollagen in biologischen Proben abbilden, und SRS erkennt aufgrund der hohen Dichte chemischer Bindungen, die aufgrund ihrer Schwingungsmoden unterschiedliche Raman-Signale erzeugen, überwiegend Proteine und Lipide. Aufgrund ihrer kohärenten Eigenschaften und des gemeinsamen Prinzips nichtlinearer optischer Eigenschaften können diese Bildgebungsmodalitäten unter Verwendung von ultrakurzgepulsten Lasern in einen einzigen Mikroskopaufbau integriert werden, was die Erfassung verschiedener Biomarker in lokalisierten Regionen ermöglicht, um ein vollständigeres Bild der biologischen Prozesse zu erhalten44,45. In diesem Artikel wird ein Protokoll für die Implementierung einer multimodalen Bildgebungsplattform vorgestellt, die MPF, SHG und SRS für biomedizinische Forschungsanwendungen integriert.
Protokoll
1. Markierungsfreie multimodale Bildgebungsexperimente
HINWEIS: Dieses Protokoll konzentriert sich auf die Einrichtung und das Erfassungsverfahren der markierungsfreien multimodalen Bildgebung.
- Aufbau des multimodalen Mikroskops (Abbildung 1A) und Systemkalibrierung
- Den Laser aufwärmen und ca. 15-20 Minuten warten.
- Schalten Sie die Steuergeräte und Monitore in der folgenden Reihenfolge ein: Schaltkasten | Touchpanel-Steuerung | Netzteil für die Haupt-Laser-Fernbedienung Netzteil für die Sub-Laser-Fernbedienung.
- Schalten Sie den Si-Fotodiodendetektor und den Lock-in-Verstärker ein.
- Konfigurieren Sie die Pumplaserstrahlen und den Stokes-Strahl. Richten Sie das Lasersystem mit einem Pumpstrahl ein, der von 780 nm bis 990 nm abstimmbar ist, einer Pulsbreite von 5 bis 6 ps und einer Wiederholrate von 80 MHz. Der Stokes-Laserstrahl hat eine feste Wellenlänge von 1.031 nm mit einem Puls von 6 ps und einer Wiederholrate von 80 MHz . Stellen Sie sicher, dass sowohl die Pumpen- als auch die Stokes-Strahlen eine geringe Leistung (mindestens 20 mW) haben, damit sie auf der Ausrichtungsplatte sichtbar sind.
- Platzieren Sie eine Ausrichtungsplatte im Strahlengang (direkt nach der Laserbox), um die räumliche Überlappung der Pumpen und Stokes-Strahlen zu überprüfen, und stellen Sie den Spiegel so ein, dass sich beide Punkte in der Mitte der Ausrichtungsplatte überlappen.
HINWEIS: Obwohl sich die Pumpen und Stokes-Strahlen grob überlappen, kann durch Feinabstimmung der räumlichen Überlappung zweier Laserstrahlen das optimale SRS-Signal erreicht werden. - Nach der räumlichen Überlappung nehmen Sie die Feinabstimmung mit dem PSD (Position Sensitive Detector) vor, indem Sie zuerst auf die OPO-Steuerungssoftware klicken. Verwenden Sie die Hexadezimaltaste , um den optischen Spiegel 1 (OM1) vorsichtig einzustellen, und beobachten Sie die PSD-Anzeige auf die Änderung der X-Y-Position, um die minimale Abweichung von der Mitte zu erreichen. Um genauere Einstellungen vorzunehmen, verwenden Sie dann den Inbusschlüssel am optischen Spiegel 2 (OM2), bis die Positionsanzeige auf dem PSD-Display zentriert ist.
HINWEIS: OM1 und OM2 sind zwei Spiegel im OPO-Kopf, um die Laserausrichtung einzustellen. - Zentrieren Sie abschließend den Kondensor, indem Sie die beiden Zentrierschrauben des Kondensors drehen, um das Bild der Irisblende in die Mitte des Sichtfelds zu verschieben.
HINWEIS: Die Feldblende kann den Durchmesser des in das Objektiv eintretenden Lichtstrahls begrenzen und so Fremdlicht ausschließen, wodurch der Bildkontrast verbessert wird.
- Markierungsfreie multimodale Bildgebungsschritte
- Tragen Sie Öl auf den Ölkondensator mit hoher numerischer Apertur (1,4 NA) auf und montieren Sie den Objektträger auf den geölten Kondensor. Zum Schluss legen Sie einen großen Wassertropfen auf den Objektträger für das 25x Wasserobjektiv. Stellen Sie sicher, dass das Mikroskop sicher montiert und unbeweglich ist, und stellen Sie dann den Z-Tisch so ein, dass der Fokus eingestellt wird, bis das Hellfeldbild der biologischen Probe unter dem 25-fach-Wasserobjektiv zu sehen ist.
- Beginnen Sie den Bildgebungsprozess in der richtigen Reihenfolge: MPF, SHG und SRS, um Photobleaching zu vermeiden, das sich auf die Bildqualität und die präzise Analyse von MPF auswirkt. Um schnell zwischen MPF und SHG zu wechseln, wechseln Sie vom Pumpbalken auf den festen Stokes-Strahl.
HINWEIS: Das Einstellen des Pumplasers von 800 nm (NADH- und FAD-Autofluoreszenz) auf 791,3 nm (CH3 Raman-Verschiebung) kann 1-2 Minuten dauern. - Wählen Sie für jede Modalität die Bildauflösung (512 x 512 Pixel) und die passende Verweilzeit aus. Verwenden Sie 8 μs/Pixel für MPF und SHG mit einem durchschnittlichen Frame über 3. Verwenden Sie 40 μs/Pixel mit einem durchschnittlichen Frame 2 für die SRS-Modalität.
HINWEIS: Der durchschnittliche Rahmen bezeichnet die Anzahl der Bilder, die vor dem Speichern gemittelt werden sollen. - Um Autofluoreszenz mit MPF zu erhalten, schalten Sie den Stokes-Laserstrahl aus und stimmen Sie den Pumplaser auf 800 nm ab, um NADH und Flavin anzuregen. Bauen Sie den FVOPT-Filterwürfel mit 460 ± 10 nm bzw. 515 ± 10 nm für NADH bzw. Flavin in den Strahlengang ein. Die Leistung der Probe beträgt somit ca. 15 mW.
- Erfassen Sie das Kollagenfasersignal mit SHG. Schalten Sie den Pumplaserstrahl aus, verwenden Sie nur den Stokes-Laserstrahl und stellen Sie die Leistung auf 500 mW ein. Erfassen Sie nur den Kanal, der sich auf den 515-nm-Filter bezieht.
- Erhalten Sie die räumliche Verteilung von Proteinen und Lipiden mit SRS. Lassen Sie beide Laserstrahlen eingeschaltet und passen Sie die Frequenz des Laserstrahls an den spezifischen Schwingungsmodus für jedes Molekül an.
HINWEIS: Typischerweise wird 791,3 nm für CH3, 797 nm für CH2, 787 nm für ungesättigte Lipide und 794,6 nm für gesättigte Lipide verwendet. Die Leistung der Probe beträgt somit ca. 40 mW. - Um die hyperspektralen SRS-Bilddatensätze zu erfassen, öffnen Sie die OPO-Steuerungssoftware, um Sweep auszuwählen, den Wellenlängenbereich von 780 nm bis 806,5 nm einzustellen und eine Stack-Nummer von mindestens 60 auszuwählen. Erfassen Sie dann den hyperspektralen Bildstapel.
HINWEIS: Der resultierende Datensatz enthält 60 Bilder, die jeweils die räumliche Verteilung bei einer bestimmten Raman-Verschiebung (von 2.700 cm-1 bis 3.150 cm-1) darstellen. Die Zeit für die hyperspektrale Erfassung hängt vom Sichtfeld (FOV) des Bildes und der Scanzeit ab, liegt jedoch bei einem Bild mit 512 x 512 Pixeln etwa zwischen 5 und 8 Minuten. Bei größeren Bildern sollte darauf geachtet werden, die Dauer unter dem Laser zu minimieren, um Lichtschäden zu vermeiden, die nach 2 Stunden auftreten können (abhängig von der Dicke der Probe und der FOV-Größe). - Speichern Sie alle Bilder derselben Regions of Interest (ROIs) im selben Ordner. Das Bildformat ist die Olympus .oir-Datei.
2. Bildanalyse
- Verwenden Sie eine Bildbearbeitungssoftware, um alle gespeicherten Rohbilder zu öffnen, die Farbe zuzuweisen und die Maßstabsleiste für die Bildanzeige hinzuzufügen. Verwenden Sie es auch, um die binäre Maske von NADH- und Sättigungsfettsäuren (SFAs, 2.880 cm-1) für die Analyse des optischen Redoxverhältnisses und der Lipidunsättigung zu erstellen.
HINWEIS: Stellen Sie sicher, dass die Maskenintensität nur 0 und 1 hat. - Verwenden Sie das selbst erstellte Python-Skript, um nachgelagerte Analysen durchzuführen (der Code ist unter https://github.com/lingyanshi2020/HSI_Analysis verfügbar).
HINWEIS: Dieses Skript wurde entwickelt, um mehrere Regionen von Interesse (Regions of Interest, ROIs) sowohl aus Kontroll- als auch aus Krebsproben zu verarbeiten. Die Hauptfunktion des Skripts benötigt nur zwei Eingaben: die Dateipfade für die Ordner control und cancer image.- Durchlaufen Sie die Ordner und ordnen Sie Bilder mit demselben Basisnamen zu (z. B. "roi_1_flavin.tif", "roi_1_nadh.tif", "roi1_mask.tif").
- Lassen Sie das Skript für jeden Satz übereinstimmender Bilder eine ratiometrische Analyse durchführen und basierend auf diesen Berechnungen neue ratiometrische Bilder generieren.
Das optische Redoxverhältnis wird als Flavin / (NADH + Flavin) berechnet.
Die Lipidunsättigung wird wie folgt berechnet: USFAs / (SFAs + USFAs), wobei USFAs ungesättigte Fettsäuren darstellen. - Alle ratiometrischen Werte aus den Bildern werden dann in einem Pandas DataFrame gespeichert, um sie leicht bearbeiten zu können. Führen Sie eine unidirektionale ANOVA durch und verwenden Sie matplotlib mit styple "ggplot" oder seaborn, um den Barplot oder Boxplot zur Quantifizierung durchzuführen.
- Verwenden Sie die Bilder 2.930 cm-1 (3CH) und 2.850 cm-1 (2CH), um ein subtrahiertes Bild (3 bis 2CH) zu erzeugen. Mischen Sie dann das CH2-Bild CH3 - CH2-Bild im RGB-Farbraum mit einer benutzerdefinierten LUT, die die H&E-Färbung nachahmt, um das digitale Histologiebild46 zu erzeugen.
- Verwenden Sie nach der Erfassung von hyperspektralen SRS-Bildern das selbst erstellte Python-Skript, um das k-Means-Clustering durchzuführen und die Bilder basierend auf spektralen Ähnlichkeiten zu segmentieren (der Code ist unter https://github.com/lingyanshi2020/HSI_Analysis verfügbar).
- Laden Sie die .tiff Bildstapel mit dem tifffile-Paket. Formen Sie den hyperspektralen SRS-Bildstapel (x, y, Spektren) in ein 2D-Array (Pixel, Spektren) um.
- Verwenden Sie das scikit-learn-Paket, um den k-means-Algorithmus mit der definierten Anzahl von Clustern für dieses 2D-Array bereitzustellen.
- Passen Sie die resultierenden Cluster-Beschriftungen wieder in die ursprünglichen Bildabmessungen an, und erstellen Sie eine Segmentierungskarte.
- Weisen Sie jedem Cluster eindeutige Farben zu und erzeugen Sie ein Falschfarbenbild, das unterschiedliche biochemische Zusammensetzungen im Gewebe darstellt.
- Verwenden Sie matplotlib, um die Spektren jedes Clusters mit Mittelwert (durchgezogene Linie) und Standardabweichung (Schattenbereich) darzustellen
- Befolgen Sie das Protokoll des PRM-SRS-Protokolls (Penalized Reference Matching SRS) unter Verwendung von MATLAB, um die räumliche markierungsfreie Lipid-Subtyp-Detektion mit SRS-hyperspektralen Bilddatensätzenzu erstellen 47.
- Organisieren Sie die Bilder und speichern Sie sie in einem .tif-Format mit 300 dpi.
Ergebnisse
Die Bilder in Abbildung 2 sind repräsentativ für die Ergebnisse, die aus der Befolgung des Protokolls zur Erfassung der Autofluoreszenz von FAD und NADH sowie der vier SRS-Kanäle für Protein, Gesamtlipide, ungesättigte Lipide und gesättigte Lipide erzielt wurden. Auch hier erzeugen wir ein pseudo-histologisches Bild, wie es im Protokoll durch RGB-Farbmischung beschrieben ist. Die Erfassung der MPF- und SRS-Kanäle liefert die Bilddateien, die später in der ratiometrischen Analyse verwendet werden. Ein Beispiel für diese Analyse ist in Abbildung 3 am menschlichen Lungengewebe zu sehen. Nach der Bildaufnahme im Protokoll nutzt unsere Bildanalysemethodik unter Verwendung von Python oder ImageJ das Verhältnis verschiedener Kanäle, um quantitative Stoffwechselinformationen bereitzustellen. Wie in den Bildern der Lipidunsättigung und des optischen Redoxverhältnisses in Abbildung 3 gezeigt, liefert die ratiometrische Analyse eine Farbkarte der Verteilung der relativen Stoffwechselaktivität und der molekularen Zusammensetzung. Wir haben diese Messungen genutzt, um Beobachtungen über die Veränderungen der Stoffwechselwege und des Lipidgehalts bestimmter Gewebe, Pathologien oder verschiedener biologischer Faktoren zu machen. Wie in Abbildung 4 zu sehen ist, kann dies einen quantitativen Vergleich von gesundem und Tumorgewebe ermöglichen, indem die Durchschnittswerte von oxidativem Stress und Lipidunsättigung zusätzlich zu seiner Verteilung in einem 2D-Mikroskopiebild verglichen werden.
Für die ratiometrische Analyse von MPF- und SRS-Bildern zielen wir üblicherweise auf das optische Redox- und Unsättigungsverhältnis ab, wie in Gl (1) und (2) gezeigt.
 (1)
(1)
 (2)
(2)
Dabei ist IC die Intensität der Pixel von Kanal C. So messen wir das Verhältnis pro Pixel und erfassen die räumliche Verteilung dieser metabolischen und molekularen Marker.
Neben der ratiometrischen Analyse zeigt Abbildung 4 auch einen weiteren potenziellen Anwendungsbereich für unsere multimodale Plattform: die hyperspektrale Bildanalyse. Wie in diesem Protokoll beschrieben, können wir ein SRS-Hyperspektralbild aufnehmen, indem wir mit Hilfe eines abstimmbaren Pumpstrahls einen Sweep über mehrere Laserwellenlängen durchführen. Dies ermöglicht es uns, den CH-Bereich des Raman-Verschiebungsspektrums für jedes Pixel innerhalb des Mikroskopiebildes zu rekonstruieren und sowohl die chemischen Informationen der Raman-Spektroskopie als auch die räumlichen Informationen der optischen Mikroskopie zu verbinden. Im Workflow für die Bildanalyse heben wir zwei Techniken hervor, die für die Analyse dieser hyperspektralen Bilder implementiert wurden: PRM-SRS für die Detektion von Biomolekülen und k-Means-Clustering. Abbildung 4E zeigt die Anwendung von PRM-SRS auf einem Bild eines Lebertumors der Maus. Der PRM-SRS-Algorithmus liefert die Korrelation zwischen den Spektren für jedes einzelne Pixel, bereinigt um eine Strafe für Verschiebungen im Raman-Spektrum; Der Algorithmus generiert dann Wahrscheinlichkeitsverteilungsbilder für jedes der Lipide.
Das K-Means-Clustering der Pixelspektren ist eine weitere Technik, die wir einsetzen, um die Gruppierung spezifischer spektraler Phänotypen zu visualisieren und eine Trennung nach Bindungskonzentration und molekularer Zusammensetzung durch Intensität bzw. spektrale Form zu ermöglichen. Abbildung 5 zeigt ein Beispiel dafür, wie die Bildanalyse über k-Means-Clustering in unserem Protokoll angewendet werden kann. Wir gehen davon aus, dass k-Mean-Cluster strukturell mit Merkmalen aus den pseudohistologischen Abbildungen übereinstimmen und eine weitere Clusterbildung von Merkmalen ermöglichen, die weder in den SRS-Kanälen noch in der histologischen Pseudo-Hämatoxylin- und Eosin-Färbung (H&E) so leicht zu erkennen sind. Die PRM-SRS- und k-Means-Clustering-Analyse ergänzen diese markierungsfreie Bildgebungsplattform, indem sie chemische und semi-targetable Informationen zu spezifischen Analyten und molekularen Bindungen liefern, ohne dass exogene Sondierungen erforderlich sind.
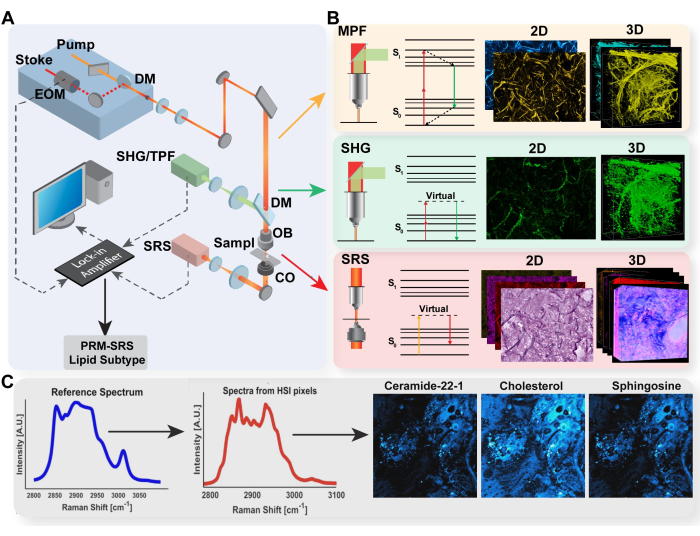
Abbildung 1: Diagramm der multimodalen Bildgebungsplattform und der PRM-SRS-Biomoleküldetektion. (A) Diagramm der Hardware-Einrichtung für MPF/TPF-, SHG- und SRS-Bildgebungssysteme. (B) Lichtpfad und Jablonski-Diagramm für TPF, SHG und SRS mit repräsentativen aufgenommenen Bildern für jede Modalität. (C) PRM-SRS-Workflow-Diagramm für den spektralen Referenzabgleich zwischen spontaner Raman-Spektroskopie und SRS-Hyperspektralbildgebung. Abkürzungen: MPF = Multiphotonenfluoreszenz; TPF = Zwei-Photonen-Fluoreszenz; SHG = Erzeugung der zweiten Harmonischen; SRS = stimulierte Raman-Streuung; PRM = bestrafter Referenzabgleich. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: MPF- und SRS-Kanäle aus humanem primärem motorischem Kortexgewebe (M1). (A,B) MPF-Autofluoreszenzkanäle für Flavin/FAD und NADH. (C-F) SRS-Kanäle, die bei bestimmten Raman-Verschiebungsspitzen erfasst werden; der asymmetrische Dehnungspeak CH3 (2.930 cm-1) für Proteine, der asymmetrische Dehnungspeak CH2 (2.845 cm-1) für Lipide, der 2.885 cm-1 Peak für gesättigte Fettsäuren und der 3.010 cm-1 Peak für ungesättigte Fettsäuren. (G) Zusammengeführte SRS-Protein- (blau) und Lipidkanäle (grün), um ihre jeweilige räumliche Verteilung zu skizzieren. (H) SRS-pseudohistologische Bilder (SRH), die die H&E-Färbung widerspiegeln. Maßstabsleisten = 20 μm. Abkürzungen: MPF = Multiphotonenfluoreszenz; FAD = Flavinadenin-Dinukleotid; NADH = reduziertes Nicotinamid-Adenin-Dinukleotid; SRS = stimulierte Raman-Streuung; SRH = SRS-Histologie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
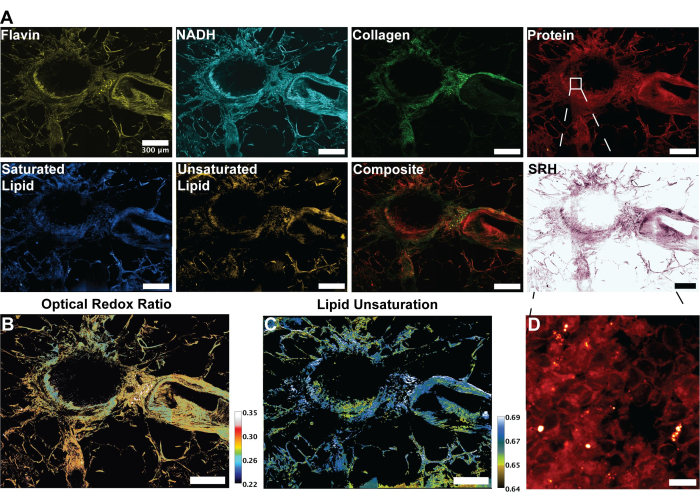
Abbildung 3: MPF-, SHG- und SRS-Bilder und Analyse von menschlichem Lungengewebe. (A) MPF-Autofluoreszenz für Flavin/FAD und NADH, SHG-Signal für Kollagenfasern, SRS-Signal für Protein (2.930 cm-1), Lipid (2.845 cm-1), gesättigte Lipide (2.885 cm-1) und ungesättigte Lipide (3.010 cm-1) Peaks sowie ein zusammengesetztes zusammengeführtes Bild der verschiedenen Modalitäten. (B) Ratiometrisches Bild des optischen Redoxverhältnisses. (C) Ratiometrisches Bild der Lipidunsättigung. (D) Vergrößertes Bild von Epithelzellen in der inneren Schicht der Bronchien. Maßstabsleisten = 200 μm. Abkürzungen: MPF = Multiphotonenfluoreszenz; SHG = Erzeugung der zweiten Harmonischen; SRS = stimulierte Raman-Streuung; FAD = Flavinadenin-Dinukleotid; NADH = reduziertes Nicotinamid-Adenin-Dinukleotid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Ratiometrische und hyperspektrale Analyse von Lebertumoren der Maus. (A) MPF-Autofluoreszenzbilder für NAD[P]H und FAD, SRS-Kanäle für Gesamtproteine, gesättigte und ungesättigte Lipide und pseudohistologische Bilder für gesunde Kontrollleber von Mäusen (oben) und tumoröses (unten) Gewebe. (B) Oxidativer Stress (gelb-grün), gemessen anhand des optischen Redoxverhältnisses, und Lipidunsättigung (blau-rot), berechnet aus dem Kontrollgewebe (links) und dem Tumorgewebe (rechts). Maßstabsbalken = 100 μm. (C,D) Balkendiagramme, die ratiometrische Unterschiede im oxidativen Stress und der Lipidunsättigung zwischen Kontroll- (blau) und Tumor- (rot) Leberproben zeigen. Signifikanzstatistik: * für eine Signifikanz von p ≤ 0,05, ** für eine Signifikanz von p ≤ 0,01. (E) Ergebnisse der PRM-SRS-Lipidsubtyp-Analyse aus Hyperspektralbildern. Von links nach rechts: SRS-Proteinkanal als strukturelle Referenz, Maßstabsbalken = 100 μm, Wahrscheinlichkeitsverteilungsbild von Lipid-Subtypen (TAG 18:1, Cholesterin und C24:0 Ceramid). Grafik der Referenzspektren des Lipidsubtyps und der mittleren Pixelspektren von SRS HSI über die CH-Region. Abkürzungen: MPF = Multiphotonenfluoreszenz; SRS = stimulierte Raman-Streuung; NAD[P]H = reduziertes Nicotinamid-Adenin-Dinukleotidphosphat; FAD = Flavinadenin-Dinukleotid; HSI = hyperspektrale Bilder. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: SRS Hyperspektrales k-Means-Clustering auf dem primären motorischen Kortex (M1) des Menschen. (A-F) Sechs repräsentative Regionen, die zeigen: links: Stimulierte Raman Histology (SRH)-Bilder mit H&E-ähnlicher Gewebevisualisierung; Mitte: entsprechende k-Means-Clustering-Ergebnisse; Rechts: clusterspezifische Raman-Spektralprofile mit mittleren Intensitäten (durchgezogene Linien) und Standardabweichungen (schattierte Bereiche). Anzahl der Schwerpunkte im Bereich von 4 bis 6 Mittelwerten. Es wurde unüberwachtes Clustering verwendet, so dass verschiedene Farben in den geclusterten Bildern unterschiedliche chemische Zusammensetzungen darstellen, die durch den k-Means-Algorithmus identifiziert wurden. Jeder Cluster (dargestellt durch eine eindeutige Farbe) entspricht Regionen mit ähnlichen spektralen Profilen in der CH-Streckungsregion. Maßstabsleiste = 20 μm. Abkürzungen: SRH = SRS-Histologie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Dieses multimodale System ist eine leistungsstarke Bildgebungsplattform für die Erfassung einer ganzheitlichen Visualisierung der molekularen Umgebung von Proben über ein breites Spektrum biologischer Herkunft und pathologischer Zustände hinweg. Der Vorteil der Nutzung verschiedener markierungsfreier Modalitäten liegt in der Fähigkeit, komplementäre Informationen zu erhalten und spezifische Analyten zu adressieren, die sonst mit einer einzigen markierungsfreien Bildgebungstechnik schwierig oder unmöglich wären. Insbesondere ermöglichen die drei in dieser Arbeit erwähnten nichtlinearen bildgebenden Verfahren (SRS, MPF, SHG) die Quantifizierung der Makromolekülzusammensetzung, der Energiedynamik über das optische Redoxverhältnis und der strukturellen Informationen, einschließlich der Zusammensetzung und Morphologie der extrazellulären Matrix 6,48,49. Darüber hinaus ermöglicht die Bildgebung mit einem kombinierten Mikroskopsystem im Vergleich zur separaten Verwendung jeder einzelnen Bildgebungsmodalität eine sofortige Bildregistrierung und kürzere Probenlagerzeiten. Wir haben sogar Live-Zell-Imaging mit diesem Ansatz durchgeführt, möglicherweise aufgrund der geringeren Lichtexposition im Vergleich zur konfokalen Fluoreszenzmikroskopie, wobei ausgewählte Zeitpunkte verwendet wurden, um jede Modalität nacheinander zu erfassen, wie im obigen Protokoll vorgeschrieben. Neben den individuellen Vorteilen profitiert die multimodale Plattform auch von den vielfältigen Perspektiven auf den biologischen Zustand durch die markierungsfreie Messung verschiedener Biomarker.
Die markierungsfreie Bildgebung von autofluoreszierenden Proteinen mittels MPF war in erster Linie auf die Quantifizierung des optischen Redoxverhältnisses ausgerichtet, ein Maß für Redoxreaktionen, das durch die Oxidation von FAD zu FAD+ und die Reduktion von NAD+ zu NADH50 erleichtert wird. Dies ist ein wichtiger Marker für den Stoffwechsel in Zellen und Geweben, da er ein Maß für die relative Aktivität zwischen oxidativer Phosphorylierung und Glykolyse liefert, den beiden Hauptwegen für die ATP-Generation6. Insbesondere eine Abnahme der NADH-Konzentration und ein Anstieg von FAD+ sind ein Marker für eine erhöhte oxidative Phosphorylierung in Mitochondrien; Das Gegenteil gilt für eine erhöhte Glykolyse im Zytoplasma. Die Tendenz, bei der Energieerzeugung einen Stoffwechselweg dem anderen vorzuziehen, wurde mit mehreren pathologischen Veränderungen und Signalwegaktivierungen im Zusammenhang mit Krebs in Verbindung gebracht, was die potenzielle Verwendung der ORR als Früherkennungsmarker unterstreicht 6,51.
In ähnlicher Weise nutzt die Erfassung des SHG-Signals für fibröses Kollagen eine nichtlineare markierungsfreie Bildgebungsmodalität, um einen biologischen Marker für die Gesundheit zu quantifizieren und zu visualisieren. SHG kann die Verteilung von Kollagen vom Typ I-III aufgrund seiner nicht-zentrosymmetrischen Struktur effektiv verfolgen. Fibröse Kollagenproteine, die mit SHG gemessen werden, wurden als wichtige diagnostische Marker für verschiedene Krankheiten wie Krebs und Fibrose anerkannt 25,26,27,52,53. Zusätzlich zu seinem Nachweis als Marker für Krankheiten kann unsere Gewinnung von Kollagen über SHG aufgrund der Rolle von fibrösem Kollagen bei der Zusammensetzung der extrazellulären Matrix und der strukturellen Grenzen zwischen Geweben als starker Indikator für die Struktur in Zellen und Geweben dienen54,55. Durch das SHG-Kollagensignal können wir dann fundierte Beobachtungen von separaten Zellen oder unterschiedlichen funktionellen Einheiten des Gewebes machen, ohne dass ein exogener Marker für Zellmembranen erforderlich ist. Insgesamt stellt die SHG-Quantifizierung von Kollagenfibrillen einen klaren Vorteil sowohl für die diagnostische Fähigkeit als auch für die einfache Analyse dar, wenn sie mit anderen markierungsfreien Bildgebungsverfahren integriert wird.
Mit Hilfe der SRS-Mikroskopie können wir mehrere Marker für die Zusammensetzung von Makromolekülen erfassen, insbesondere für Lipide und Fettsäuren. In diesem Protokoll beschreiben wir, wie das SRS-Signal für ungesättigte und gesättigte Fettsäuren für die ratiometrische Analyse der Lipidunsättigung innerhalb derselben Probe erfasst werden kann. Für den Nah-IR-Durchsatz wurde ein aufrechtes Laser-Scanning-Mikroskop mit einem 25-fachen Wasserobjektiv eingesetzt. Die Laserstrahlen durchliefen die Probe und wurden von einem Ölkondensator mit hoher numerischer Apertur (1,4 NA) gesammelt. Ein Kurzpassfilter mit hohem Außendurchmesser (950 nm) wurde verwendet, um den Stokes-Strahl zu blockieren, während der Pumpstrahl nur einen Si-Photodiodendetektor erreichen konnte, um das stimulierte Raman-Verlustsignal zu detektieren. Der Ausgangsstrom der Fotodiode wurde terminiert und gefiltert. Anschließend demodulierte ein Lock-in-Verstärker bei 20 MHz den Strom in X mit einer Nullphasenverschiebung, und ein Softwaremodul nutzte das demodulierte Signal, um das Bild während des Laserscannings zu erzeugen.
Studien haben gezeigt, dass Veränderungen der Lipidunsättigung einen tiefgreifenden Einfluss auf die Zell- und Organellenmembran haben und eine Dysregulation des Fettstoffwechsels ein starker Marker für verschiedene Krankheiten sein kann, darunter Krebs und neurodegenerative Erkrankungen 56,57,58,59. Für einen umfassenderen Überblick über die Lipiddysregulation analysieren wir die Zusammensetzung des Subtyps mit Hilfe der hyperspektralen SRS-Bildgebung. SRS-Hyperspektralbilder (HSIs) kombinieren die hochauflösenden räumlichen Informationen, die durch die SRS-Mikroskopie erfasst werden, mit dem chemisch signifikanten Signal der Raman-Spektroskopie durch sequentielle Abstimmung, wie im Protokoll beschrieben. Diese Pixelspektren werden dann in zwei Methoden analysiert: Clustering über k-Means und Biomoleküldetektion mit PRM-SRS.
Die K-Means-Clusterung der Pixelspektren führt aufgrund der linearen Beziehung zwischen der SRS-Signalintensität und der molekularen Bindungskonzentration zu einer Trennung der Regionen innerhalb eines 2D-Bildes nach molekularer Konzentrationund Zusammensetzung 60,61. Mit dieser Methodik wenden wir das Protokoll an, um Regionen zu bestimmen, die spezifische molekulare Phänotypen besitzen, wie sie durch die Pixelspektren und den gemeinsamen spektralen Schwerpunkt vorgeschrieben sind. Für eine gründliche Untersuchung der ausgeprägten molekularen Expression in bestimmten Pixeln implementieren wir PRM-SRS, um eine spektrale Anpassung zwischen einem Referenzspektrum für ein bestimmtes Molekül, das mit spontaner Raman-Spektroskopie erfasst wurde, und den Pixelspektren aus der SRS-HSI-Erfassung durchzuführen. Auf diese Weise können wir die Expressionswahrscheinlichkeit für unterschiedliche Moleküle bestimmen, ohne exogene Sonden oder Markierungen zu verwenden. Diese Analyse hat gezeigt, dass bestimmte Lipidsubtypen über Gewebe und biologische Modelle hinweg verfolgt werden können und dass pathologische Veränderungen in der Lipidzusammensetzung und damit Dyslipidämie identifiziert werdenkönnen 47. Darüber hinaus bieten die jüngsten Fortschritte bei der Verbesserung der spektralen Auflösung durch spektrale Aufspaltung der Pikosekunden-Pulsausbreitung durch Siliziumdioxidfasern einen weiteren Anreiz für die Anwendung von SRS HSI als Modalität für die markierungsfreie biochemische Visualisierung62. Durch diese Methoden, die im Protokoll hervorgehoben werden, etablieren wir eine Plattform zur Durchführung umfassender ratiometrischer und spektraler Analysen unter Verwendung einer markierungsfreien SRS-Modalität.
Neben der relativen Komplexität und Neuartigkeit dieser Techniken gibt es einige wichtige Einschränkungen in unserem Ansatz zu berücksichtigen. Die Aufnahme von MPF-Bildern mit einem Pikosekunden-gepulsten Laser erfordert im Gegensatz zu den üblicherweise verwendeten Femtosekundenlasern eine höhere Fluorophor-Anregungsleistung und birgt das Risiko von Photobleichung. Vor diesem Hintergrund haben wir das obige Protokoll optimiert, um den potenziellen Lichtschaden durch unsere Plattform zu mindern, indem wir die auf die Probe einwirkende Laserleistung reduzieren. Während frühere Studien die Fähigkeit gezeigt haben, das optische Redoxverhältnis aus der MPF-Autofluoreszenzbildgebung von NADH und FAD zu bewerten, hat die Fluoreszenzlebensdauer-Bildgebungsmikroskopie (FLIM) eine höhere Genauigkeit bei der Bewertung des Energiestoffwechsels gezeigt 63,64,65. Dies ist auf die Fähigkeit zurückzuführen, proteingebundene und ungebundene Konzentrationen von NADH und FAD auf der Grundlage ihrer unterschiedlichen Fluoreszenzlebensdauer zu unterscheiden. Im Vergleich zu den Fähigkeiten von MPF konnte FLIM für umfassende Studien der Stoffwechselaktivität von verschiedenen Energiewegen verwendet werden, einschließlich oxidativer Phosphorylierung und Glykolyse 16,63,64,65,66,67, 68. Urheberrecht Obwohl dieses Protokoll FLIM ausschließt, gehen wir davon aus, dass die Modalität in naher Zukunft in diese Bildgebungsplattform integriert wird, um die von uns derzeit durchgeführte Analyse des Energiestoffwechsels zu verbessern. Nichtsdestotrotz stellen wir mit dem hier etablierten Protokoll einen Workflow und ein Hardware-Setup für einen multimodalen Bildgebungsansatz vor, der den Stoffwechsel der biologischen Struktur aus mehreren Perspektiven messen kann.
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Wir danken Dr. Gloria Pryhuber und ihren HuBMAP-Teammitgliedern für die Bereitstellung von menschlichen Lungengewebeschnitten. Wir danken Dr. Kun Zhang für die Bereitstellung von menschlichem Gehirngewebe. Wir danken auch Dr. Gen-Sheng Feng für die Bereitstellung von Leberproben von Mäusen. Wir danken der Unterstützung durch NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, UCSD Startup Funds, Sloan Research Fellow Award und CZI DAF2023-328667 Award.
Materialien
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Referenzen
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten