Method Article
Plataforma de imagem óptica multimodal para estudar o metabolismo celular
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Utilizando uma plataforma multimodal que combina modalidades de imagem óptica sem rótulo, desenvolvemos um protocolo para visualizar e quantificar a dinâmica e o metabolismo celular. Por meio de imagens via fluorescência multifotônica, geração de segundo harmônico e microscopia de espalhamento Raman estimulada, podemos gerar uma visão holística do ambiente celular e molecular.
Resumo
As tecnologias de imagem óptica são críticas em estudos biomédicos por sua capacidade de obter informações morfológicas e funcionais de espécimes biológicos em alta resolução espacial. Esses processos ópticos exploram várias interações luz-molécula, como espalhamento, absorção, emissão e geração de harmônicos, entre fótons e as moléculas dentro de células, tecidos ou órgãos. Embora a imagem biomédica convencional tenha historicamente se concentrado na aplicação de uma única modalidade, pesquisas recentes mostraram que essas diversas técnicas fornecem insights complementares e seus resultados combinados oferecem uma compreensão mais abrangente das mudanças moleculares nos processos de envelhecimento e desenvolvimento de doenças e fundamentos na biologia celular.
Nas últimas décadas, os métodos de imagem óptica sem rótulos avançaram, permitindo a exploração detalhada de ambientes celulares e subcelulares. Por exemplo, a fluorescência multifotônica (MPF) não apenas facilita a imagem de proteínas direcionadas, mas também quantifica a atividade metabólica por meio de coenzimas autofluorescentes, alcançando alta profundidade de penetração e resolução espacial. A Geração de Segundo Harmônico (SHG) é usada para obter imagens de estruturas como colágeno na matriz extracelular, enquanto o Espalhamento Raman Estimulado (SRS) mapeia ligações químicas e composição molecular in situ com resolução subcelular.
Desenvolvemos uma plataforma de imagem multimodal que combina as modalidades MPF, SHG e SRS. A integração dessas modalidades em uma única plataforma permite a aquisição de informações multifacetadas da mesma localização dentro de células, tecidos, órgãos ou mesmo corpos, facilitando uma exploração mais detalhada das intrincadas relações entre metabolismo celular, estrutura da matriz extracelular e composição molecular. Este sistema multimodal oferece resolução subcelular, penetração profunda de tecidos, imagens in situ de células vivas/tecidos, bem como detecção sem marcação e co-registro instantâneo sem a necessidade de ajustes de posição, troca de dispositivos ou alinhamento pós-análise. Aqui, apresentamos um protocolo para imagens sem marcação com esta plataforma multimodal e demonstramos sua aplicação na caracterização do metabolismo celular e heterogeneidade molecular em células e tecidos para o estudo do envelhecimento e doenças.
Introdução
A imagem biomédica óptica tem sido fundamental para o avanço de nossa compreensão da estrutura e função biológicas. As imagens são geradas modulando a luz de excitação e detectando sinais de interações luz-tecido. O primeiro microscópio composto, desenvolvido por Hans e Zacharias Janssen por volta de 1590, utilizou duas lentes convexas em um tubo, proporcionando ampliação de até 30x1. Os microscópios ópticos modernos, após séculos de avanços, agora podem atingir resoluções tão finas quanto 1-3 nm 2,3. Além de oferecer alta resolução, os sistemas avançados de imagem agora fornecem penetração mais profunda do tecido, maior eficiência e danos mínimos à amostra, tornando-os especialmente adequados para imagens de células vivas e tecidos. A imagem sem marcação é particularmente vantajosa, pois captura informações sem interromper os processos intracelulares ou comprometer a integridade da amostra.
A microscopia de fluorescência multifotônica (MPF), particularmente a microscopia de fluorescência de dois fótons, tem sido amplamente utilizada para imagens sem marcadores. Ao contrário da microscopia de fluorescência convencional, que depende da absorção e emissão linear de um único fóton, a excitação do MPF envolve a absorção simultânea de vários fótons, cuja energia combinada excita uma única molécula de fluoróforo 4,5. Esses fótons, normalmente no espectro infravermelho, possuem metade ou menos da energia necessária para a excitação de um único fóton. Os comprimentos de onda mais longos e a excitação localizada no ponto focal neste processo não linear resultam em menor espalhamento, penetração mais profunda do tecido e fototoxicidade reduzida.
As informações metabólicas celulares podem ser capturadas por microscopia MPF livre de marcação por meio da detecção de sinais de autofluorescência de substratos metabólicos endógenos, como dinucleotídeo de nicotinamida adenina reduzido (NADH) e dinucleotídeo de flavina adenina (FAD). Essas coenzimas exibem espectros distintos de excitação e emissão, e sua razão de intensidade de fluorescência, conhecida como razão redox (NADH / FAD), reflete o estado oxidativo da célula. Desde que Britton Chance introduziu pela primeira vez o conceito de razão redox em 1979, razões adicionais, incluindo NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) e FAD/(FAD + NAD(P)H), foram propostas 6,7,8,9. A quantificação dessas proporções redox ópticas por meio de imagens MPF fornece informações valiosas sobre a dinâmica metabólica. Por exemplo, a imagem MPF pode distinguir células cancerígenas de células normais com base em seu metabolismo alterado, demonstrando seu potencial para o diagnóstico de câncer 10,11,12. No entanto, a detecção de autofluorescência baseada em MPF tem limitações. Outros fluoróforos intrínsecos, como a queratina, podem contribuir para a intensidade da fluorescência, levando a diafonia espectral e interpretação imprecisa do sinal13. Além disso, a razão redox reflete apenas as alterações gerais de oxidação-redução celular e não distingue entre NADH de diferentes fontes (por exemplo, citoplasmáticas ou mitocondriais) ou entre NADH e NAD (P) H, pois ambos exibem picos espectrais semelhantes em 450 nm, resultando em sinais de intensidade combinados14.
A Geração de Segundo Harmônico (SHG), um processo óptico não linear demonstrado pela primeira vez no campo biomédico na década de 1980, tem sido amplamente utilizado para imagens sem marcação de estruturas celulares 15,16. Semelhante ao MPF, o SHG envolve a absorção simultânea de dois fótons da mesma energia de um laser pulsado ultrarrápido. Esses fótons são recombinados para emitir um novo fóton com o dobro da frequência da luz incidente, resultando na detecção do sinal do segundo harmônico. Essa interação óptica não linear ocorre exclusivamente em materiais não centrossimétricos que exibem uma suscetibilidade de segunda ordem diferente de zero para induzir uma polarização para gerar o sinal de segundo harmônico 17,18. Isso torna o SHG particularmente eficaz para imagens de proteínas filamentosas e estruturas fibrilares, como colágeno, miosina e tubulina, sem a necessidade de corantes de fluorescência exógenos 15,17,19,20. A anormalidade na abundância, rigidez, alinhamento e estrutura da fibrose e do colágeno são prevalentes em muitas condições, como inflamação e câncer, tornando o SHG uma ferramenta promissora para detecção eficiente e não invasiva de certas condições de doença 21,22,23. A aplicação generalizada da imagem SHG em pesquisas oncológicas, incluindo estudos sobre câncer de mama, ovário e pele, destacou seu papel crucial tanto na pesquisa fundamental quanto em possíveis aplicações clínicas 24,25,26,27.
Diferentes moléculas exibem níveis distintos de energia vibracional, que induzem vários graus de espalhamento inelástico quando excitados pela luz incidente - um fenômeno caracterizado pela primeira vez por CV Raman em 192828. Desde então, o efeito Raman tem sido amplamente utilizado em microscopia óptica para a detecção de composições moleculares e teciduais sem marcação exógena. Tanto o Espalhamento Raman Estimulado (SRS) quanto o Espalhamento Raman anti-Stokes coerente (CARS) excitam vibrações moleculares de forma coerente e alavancam a interação não linear da luz para produzir um sinal mais forte em comparação com a Espectroscopia Raman Espontânea convencional. O fenômeno SRS foi relatado pela primeira vez em 196229. Em 2008, esse mecanismo foi integrado à imagem multifotônica tridimensional, permitindo a detecção seletiva de produtos químicos com base nas mudanças de intensidade nos feixes Pump e Stokes devido a transições vibracionais moleculares30. Este método minimiza a interferência de fundo não ressonante, gerando um sinal de intensidade limpa que supera o do CARS. A imagem SRS se destaca por fornecer imagens multiplexadas e hiperespectrais, permitindo a detecção simultânea de múltiplas ligações químicas e permitindo a visualização de alta resolução da composição molecular em amostras com profundidade de penetração considerável. Embora seja uma técnica relativamente nova, a imagem SRS provou ser eficaz tanto no diagnóstico clínico quanto na pesquisa metabólica, in vivo e in vitro 30,31,32,33,34,35,36. Por exemplo, a SRS pode diferenciar tecidos infiltrados por tumores cerebrais do córtex e da substância branca, quantificando a proporção lipídio-proteína, permitindo o delineamento das margens tumorais de maneira não invasiva e sem rótulos37,38. Além disso, as alterações metabólicas, muitas vezes consideradas características de doenças relacionadas ao envelhecimento e ao câncer, podem ser avaliadas quantitativamente usando o SRS obtido pela detecção de ligações carbono-deutério em amostras tratadas com água pesada (D2O), permitindo a medição quantitativa da síntese de proteínas, lipogênese e outros processos metabólicos macromoleculares31 , 33 , 34 , 35 , 36. A capacidade de rastrear metabólitos com alta resolução temporal e espacial posiciona a SRS como uma ferramenta promissora para investigação e diagnóstico de doenças, com potencial para aplicações clínicas mais amplas.
A imagem multimodal surgiu como uma abordagem poderosa na pesquisa biomédica, integrando duas ou mais modalidades de imagem para obter uma compreensão mais abrangente de sistemas biológicos complexos dentro do mesmo espécime. Em 2018, foi introduzida uma técnica de microscopia de autofluorescência multiharmônica (SLAM) sem marcadores, integrando fluorescência de dois fótons (2PF), fluorescência de três fótons (3PF), SHG e geração de terceiro harmônico (THG)39. Essa abordagem facilita a visualização simultânea de interações celulares, processos dinâmicos e componentes individuais dentro do microambiente tumoral. A microscopia SLAM oferece perturbação mínima e requisitos reduzidos de potência do laser para a amostra, permitindo o perfil de tecido profundo e fornecendo um método mais seguro para monitoramento intravital40. Outra modalidade multimodal, combinando espectroscopia de fluorescência intrínseca, espectroscopia de reflectância difusa e espectroscopia Raman, foi desenvolvida para detecção de câncer in situ durante procedimentos cirúrgicos41. Além disso, um sistema de endoscopia não linear multimodal recentemente projetado, que integra CARS, SHG e fluorescência de dois fótons (TPF), demonstrou a capacidade de obter imagens de amostras biológicas em resolução espacial submicrométrica e subcelular42. A microscopia combinada de 2PF e SRS tem sido utilizada de forma semelhante para imagens in vivo de alta resolução de tecidos, células e organelas 42,43,44,45. Essas técnicas emergentes de imagem multimodal aproveitam os pontos fortes de modalidades individuais, levando a uma melhor resolução, profundidade de penetração e eficiência de aquisição de imagem, mostrando assim um potencial considerável para aplicações clínicas e cirúrgicas.
Essa abordagem multimodal é cada vez mais favorecida em relação à imagem de modalidade única porque fornece uma gama mais ampla de medições enquanto mitiga as limitações associadas a técnicas individuais. Como discutido anteriormente, o MPF mede a fluorescência endógena para refletir as alterações metabólicas, o SHG pode obter imagens de estruturas não centrossimétricas, como colágeno em amostras biológicas, e o SRS detecta predominantemente proteínas e lipídios devido à alta densidade de ligações químicas que geram sinais Raman distintos com base em seus modos vibracionais. Dadas suas propriedades coerentes e o princípio compartilhado de propriedades ópticas não lineares, essas modalidades de imagem podem ser integradas em uma única configuração de microscópio utilizando lasers pulsados ultracurtos, permitindo a aquisição de vários biomarcadores em regiões localizadas para fornecer uma visão mais completa dos processos biológicos44,45. Este artigo descreve um protocolo para implementar uma plataforma de imagem multimodal que integra MPF, SHG e SRS para aplicações de pesquisa biomédica.
Protocolo
1. Experimentos de imagem multimodal sem rótulos
NOTA: Este protocolo se concentra no procedimento de configuração e aquisição da imagem multimodal sem marcador.
- Configuração do microscópio multimodal (Figura 1A) e calibração do sistema
- Aqueça o laser e aguarde aproximadamente 15-20 min.
- Ligue as unidades de controle e monitores na seguinte sequência: Caixa de controle | Controlador de painel de toque | Adaptador AC para controle remoto a laser principal Adaptador AC para controle remoto sub laser.
- Ligue o detector de fotodiodo Si e o amplificador de bloqueio.
- Configure os feixes de laser da bomba e o feixe de Stokes. Configure o sistema laser com um feixe de bomba ajustável de 780 nm a 990 nm, largura de pulso de 5 a 6 ps e taxa de repetição de 80 MHz. O feixe de laser Stokes tem um comprimento de onda fixo de 1.031 nm com um pulso de 6 ps e taxa de repetição de 80 MHz . Certifique-se de que a bomba e os feixes Stokes estejam em baixa potência (pelo menos 20 mW) para serem visíveis na placa de alinhamento.
- Coloque uma placa de alinhamento no caminho óptico (logo após a caixa de laser) para verificar a sobreposição espacial das bombas e dos feixes de Stokes e ajuste o espelho até que ambos os pontos se sobreponham no centro da placa de alinhamento.
NOTA: Embora as bombas e os feixes de Stokes estejam aproximadamente sobrepostos, o ajuste fino da sobreposição espacial de dois feixes de laser pode obter o sinal SRS ideal. - Após a sobreposição espacial, ajuste com o PSD (Detector Sensível à Posição) clicando primeiro no software de controle OPO . Use a tecla sextavada para ajustar cuidadosamente o espelho óptico 1 (OM1) e observe a exibição PSD para a mudança de posição XY para apontar para o desvio mínimo do centro. Em seguida, para fazer ajustes mais precisos, use a chave sextavada no espelho óptico 2 (OM2) até que o indicador de posição esteja centralizado no visor PSD.
NOTA: OM1 e OM2 são dois espelhos no cabeçote OPO para ajustar o alinhamento do laser. - Por fim, centralize o condensador girando os dois parafusos de centralização do condensador para mover a imagem do diafragma da íris para o centro do campo de visão.
NOTA: O diafragma de íris de campo pode restringir o diâmetro do feixe de luz que entra na objetiva e, assim, excluir a luz estranha, melhorando o contraste da imagem.
- Etapas de aquisição de imagens multimodais sem rótulos
- Aplique óleo no condensador de óleo de alta abertura numérica (1.4 NA) e monte a lâmina do microscópio no condensador oleado. Por fim, coloque uma gota de água grande na lâmina do microscópio para a objetiva de água 25x. Certifique-se de que o microscópio esteja montado com segurança e imóvel e, em seguida, ajuste o estágio z para ajustar o foco até que a imagem de campo claro da amostra biológica possa ser vista sob a objetiva de água 25x.
- Inicie o processo de imagem na sequência correta: MPF, SHG e SRS para evitar o fotobranqueamento, o que afetará a qualidade da imagem e a análise precisa do MPF. Para alternar rapidamente entre MPF e SHG, mude do feixe da bomba para o feixe fixo de Stokes.
NOTA: Ajustar o laser da bomba de 800 nm (autofluorescência NADH e FAD) a 791.3 nm (CH3 Raman shift) pode levar de 1 a 2 minutos. - Selecione a resolução da imagem (512 x 512 pixels) e o tempo de permanência adequado para cada modalidade. Use 8 μs/pixel para MPF e SHG com quadro médio acima de 3. Use 40 μs/pixel com quadro médio 2 para a modalidade SRS.
NOTA: O quadro médio significa o número de imagens a serem calculadas antes de salvar. - Para adquirir autofluorescência com MPF, desligue o feixe de laser Stokes e ajuste o laser da bomba para 800 nm para excitar NADH e flavina. Instale o cubo do filtro FVOPT no caminho da luz com 460 ± 10 nm e 515 ± 10 nm para NADH e flavina, respectivamente. A potência na amostra é, portanto, de aproximadamente 15 mW.
- Adquira o sinal da fibra de colágeno usando SHG. Desligue o feixe de laser da bomba, use apenas o feixe de laser de combustível e defina a potência para 500 mW. Adquira apenas o canal relacionado ao filtro de 515 nm.
- Obtenha a distribuição espacial de proteínas e lipídios usando SRS. Mantenha os dois feixes de laser ligados e ajuste a frequência do feixe de laser para corresponder ao modo vibracional específico de cada molécula.
NOTA: Normalmente, 791,3 nm é usado para CH3, 797 nm é usado para CH2, 787 nm é para lipídios insaturados e 794,6 é para lipídios saturados. A potência na amostra é, portanto, de aproximadamente 40 mW. - Para adquirir os conjuntos de dados de imagem hiperespectral SRS, abra o software de controle OPO para selecionar varredura, defina a faixa de comprimento de onda de 780 nm a 806,5 nm e escolha um número de pilha de pelo menos 60. Em seguida, capture a pilha de imagens hiperespectrais.
NOTA: O conjunto de dados resultante contém 60 imagens, cada uma representando a distribuição espacial em um deslocamento Raman específico (de 2.700 cm-1 a 3.150 cm-1). O tempo para aquisição hiperespectral depende do campo de visão (FOV) da imagem e do tempo de varredura, mas será aproximadamente entre 5 min e 8 min para uma imagem 512 x 512. Para imagens maiores, deve-se tomar cuidado para minimizar a duração sob o laser para evitar qualquer fotodano, que pode ocorrer após 2 h (dependendo da espessura da amostra e do tamanho do FOV). - Salve todas as imagens das mesmas regiões de interesse (ROIs) na mesma pasta. O formato da imagem é o arquivo .oir da Olympus.
2. Análise de imagem
- Use o software de processamento de imagem para abrir todas as imagens raw salvas para atribuir a cor e adicionar a barra de escala para a exibição da imagem. Use-o também para gerar a máscara binária de imagens de NADH e ácidos graxos de saturação (SFAs, 2.880 cm-1) para análise de razão redox óptica e insaturação lipídica.
NOTA: Certifique-se de que a intensidade da máscara tenha apenas 0 e 1. - Use o script python caseiro para fazer qualquer análise downstream (o código está disponível em https://github.com/lingyanshi2020/HSI_Analysis).
NOTA: Este script foi projetado para processar várias regiões de interesse (ROIs) de amostras de controle e câncer. A função principal do script requer apenas duas entradas: os caminhos de arquivo para as pastas de controle e imagem de câncer.- Itere pelas pastas, combinando imagens com o mesmo nome base (por exemplo, "roi_1_flavin.tif", "roi_1_nadh.tif", "roi1_mask.tif").
- Para cada conjunto de imagens correspondentes, deixe o script executar a análise raciométrica e gerar novas imagens raciométricas com base nesses cálculos.
A razão redox óptica é calculada como flavina / (NADH + flavina).
A insaturação lipídica é calculada como: USFAs / (SFAs + USFAs) onde USFAs representam ácidos graxos insaturados. - Todos os valores ratiométricos das imagens são então armazenados em um DataFrame do pandas para facilitar a manipulação. Execute ANOVA unidirecional e use matplotlib com styple "ggplot" ou seaborn para fazer o barplot ou boxplot para quantificação.
- Use as imagens de 2.930 cm-1 (CH3) e 2.850 cm-1 (CH2) para gerar uma imagem subtraída (CH3 - CH2). Em seguida, misture a imagem CH2 CH3 - CH2 no espaço de cores RGB com uma LUT personalizada que imita a coloração H & E para gerar a imagem histológica digital46.
- Após a aquisição de imagens SRS hiperespectrais, empregue o script python construído em casa para fazer o agrupamento k-means para segmentar as imagens com base em semelhanças espectrais (o código está disponível em https://github.com/lingyanshi2020/HSI_Analysis).
- Carregue as pilhas de imagens .tiff com o pacote tifffile. Remodele a pilha de imagens hiperespectrais SRS (x, y, espectros) em uma matriz 2D (pixels, espectros).
- Use o pacote scikit-learn para implantar o algoritmo k-means nessa matriz 2D, com o número definido de clusters.
- Remodele os rótulos de cluster resultantes de volta para as dimensões originais da imagem, criando um mapa de segmentação.
- Atribua cores exclusivas a cada cluster, gerando uma imagem de cor falsa que representa diferentes composições bioquímicas dentro do tecido.
- Use matplotlib para plotar os espectros de cada aglomerado com valor médio (linha sólida) e desvio padrão (área de sombra)
- Siga o protocolo do protocolo SRS de correspondência de referência penalizada (PRM-SRS) usando MATLAB para criar a detecção de subtipo lipídico livre de marcação espacial com conjuntos de dados de imagem hiperespectral SRS47.
- Organize as imagens e salve-as em um formato de .tif de 300 dpi.
Resultados
As imagens da Figura 2 são representativas dos resultados obtidos ao seguir o protocolo de aquisição de autofluorescência de FAD e NADH, bem como os quatro canais SRS para proteínas, lipídios totais, lipídios insaturados e lipídios saturados. Aqui também geramos uma imagem pseudo-histológica conforme descrito no protocolo por meio da mistura de cores RGB. A aquisição dos canais MPF e SRS fornece os arquivos de imagem que serão posteriormente utilizados na análise raciométrica. Um exemplo dessa análise pode ser visto na Figura 3 sobre tecido pulmonar humano. Após a aquisição de imagens no protocolo, nossa metodologia de análise de imagens utilizando Python ou ImageJ aproveita a proporção de canais distintos para fornecer informações metabólicas quantitativas. Conforme mostrado nas imagens de insaturação lipídica e razão redox óptica da Figura 3, a análise raciométrica fornece um mapa colorido da distribuição da atividade metabólica relativa e da composição molecular. Aproveitamos essas medições para fazer observações sobre as mudanças nas vias metabólicas e no conteúdo lipídico de tecidos específicos, patologias ou fatores biológicos distintos. Como visto na Figura 4, isso pode fornecer uma comparação quantitativa de tecido saudável e tumoral, comparando os valores médios de estresse oxidativo e insaturação lipídica, além de sua distribuição em uma imagem de microscopia 2D.
Para análise raciométrica de imagens MPF e SRS, geralmente visamos o redox óptico e a taxa de insaturação, conforme mostrado pela Eq (1) e (2).
 Características (1)
Características (1)
 (2)
(2)
Onde IC é a intensidade dos pixels do canal C. Assim, medimos a razão por pixel e adquirimos a distribuição espacial desses marcadores metabólicos e moleculares.
Além da análise raciométrica, a Figura 4 também demonstra outra via potencial de aplicação para nossa plataforma multimodal: análise de imagem hiperespectral. Conforme descrito neste protocolo, podemos adquirir uma imagem hiperespectral SRS realizando uma varredura em vários comprimentos de onda do laser, utilizando um feixe de bomba ajustável. Isso nos permite reconstruir a região CH do espectro de deslocamento Raman para cada pixel dentro da imagem de microscopia, unindo as informações químicas fornecidas pela espectroscopia Raman e as informações espaciais fornecidas pela microscopia óptica. No fluxo de trabalho para análise de imagens, destacamos duas técnicas implementadas para análise dessas imagens hiperespectrais: PRM-SRS para detecção de biomoléculas e agrupamento de k-means. A Figura 4E exibe a aplicação de PRM-SRS em uma imagem de um tumor hepático de camundongo. O algoritmo PRM-SRS fornece a correlação entre os espectros para cada pixel individual, ajustado por uma penalidade para mudanças no espectro Raman; O algoritmo então gera imagens de distribuição de probabilidade para cada um dos lipídios.
O agrupamento K-means dos espectros de pixels é outra técnica que implementamos para visualizar o agrupamento de fenótipos espectrais específicos, permitindo a separação por concentração de ligação e composição molecular por meio de intensidade e forma espectral, respectivamente. A Figura 5 fornece um exemplo de como a análise de imagens via clustering k-means pode ser aplicada em nosso protocolo. Esperamos que os clusters k-mean se alinhem estruturalmente com as características das figuras de pseudo-histologia, além de fornecer mais agrupamento de características que não são tão facilmente discernidas dos canais SRS ou da coloração histológica pseudo-hematoxilina e eosina (H & E). PRM-SRS e análise de agrupamento k-means complementam esta plataforma de imagem sem marcadores, fornecendo informações químicas e semi-direcionáveis sobre analitos específicos e ligações moleculares sem a necessidade de sondagem exógena.
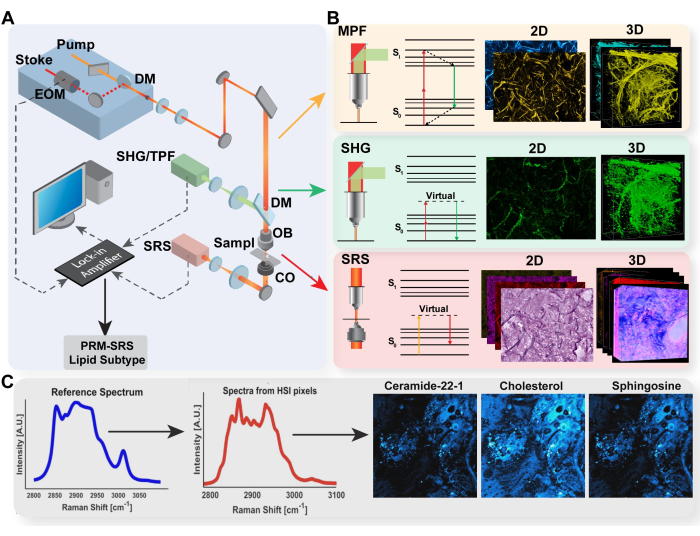
Figura 1: Diagrama da plataforma de imagem multimodal e detecção de biomoléculas PRM-SRS. (A) Diagrama de configuração de hardware para MPF/TPF, SHG e sistema de imagem SRS. (B) Caminho de luz e diagrama de Jablonski para TPF, SHG e SRS com imagens adquiridas representativas para cada modalidade. (C) Diagrama de fluxo de trabalho PRM-SRS para correspondência de referência espectral entre espectroscopia Raman espontânea e imagem hiperespectral SRS. Abreviaturas: MPF = fluorescência multifotônica; TPF = fluorescência de dois fótons; SHG = segunda geração harmônica; SRS = espalhamento Raman estimulado; PRM = correspondência de referência penalizada. Clique aqui para ver uma versão maior desta figura.

Figura 2: Canais MPF e SRS do tecido do córtex motor primário humano (M1). (A, B) Canais de autofluorescência MPF para Flavin/FAD e NADH. (C-F) Canais SRS capturados em picos de deslocamento Raman específicos; o pico de estiramento assimétrico CH3 (2.930 cm-1) para proteínas, o pico de estiramento assimétrico CH2 (2.845 cm-1) para lipídios, o pico de 2.885 cm-1 para ácidos graxos saturados e o pico de 3.010 cm-1 para ácidos graxos insaturados. (G) Canais de proteína SRS (azul) e lipídios (verde) mesclados para delinear suas respectivas distribuições espaciais. (H) Imagens pseudo-histológicas SRS (SRH) espelhando a coloração H & E. Barras de escala = 20 μm. Abreviaturas: MPF = fluorescência multifotônica; FAD = dinucleótido de flavina adenina; NADH = dinucleotídeo de nicotinamida adenina reduzido; SRS = espalhamento Raman estimulado; SRH = histologia SRS. Clique aqui para ver uma versão maior desta figura.
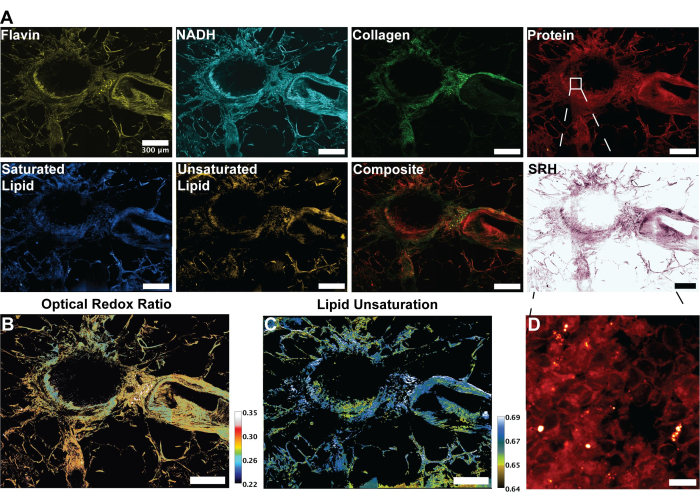
Figura 3: Imagens de MPF, SHG e SRS e análise do tecido pulmonar humano. (A) Autofluorescência MPF para picos de Flavina/FAD e NADH, sinal SHG para fibras colágenas, sinal SRS para picos de proteína (2.930 cm-1), lipídios (2.845 cm-1), lipídios saturados (2.885 cm-1) e lipídios insaturados (3.010 cm-1) e uma imagem mesclada composta das múltiplas modalidades. (B) Imagem raciométrica da razão redox óptica. (C) Imagem raciométrica da insaturação lipídica. (D) Imagem ampliada de células epiteliais na camada interna dos brônquios. Barras de escala = 200 μm. Abreviaturas: MPF = fluorescência multifotônica; SHG = segunda geração harmônica; SRS = espalhamento Raman estimulado; FAD = dinucleótido de flavina adenina; NADH = dinucleotídeo nicotinamida adenina reduzido. Clique aqui para ver uma versão maior desta figura.

Figura 4: Análise raciométrica e hiperespectral de tumores hepáticos de camundongos. (A) Imagens de autofluorescência MPF para NAD [P] H e FAD, canais SRS para proteínas totais, lipídios saturados e insaturados e imagens pseudo-histológicas para fígado de camundongo de controle saudável (superior) e tecido tumoral (inferior). (B) Estresse oxidativo (amarelo-verde), medido pela razão redox óptica, e insaturação lipídica (azul-vermelho) calculada a partir dos tecidos controle (esquerdo) e tumoral (direito). Barras de escala = 100 μm. (C,D) Gráficos de barras demonstrando diferenças raciométricas no estresse oxidativo e insaturação lipídica entre amostras de fígado controle (azul) e tumor (vermelho). Estatística de significância: * para significância de p ≤ 0,05, ** para significância de p ≤ 0,01. (E) Resultados da análise do subtipo lipídico PRM-SRS de imagens hiperespectrais. Da esquerda para a direita: canal de proteína SRS para referência estrutural, barra de escala = 100 μm, imagem de distribuição de probabilidade dos subtipos lipídicos (TAG 18:1, Colesterol e C24:0 Ceramida). Gráfico de espectros de referência de subtipo lipídico e espectros médios de pixels de SRS HSI em toda a região CH. Abreviaturas: MPF = fluorescência multifotônica; SRS = espalhamento Raman estimulado; NAD[P]H = nicotinamida adenina dinucleotídeo fosfato reduzido; FAD = dinucleótido de flavina adenina; HSI = imagens hiperespectrais. Clique aqui para ver uma versão maior desta figura.

Figura 5: Agrupamento de k-means hiperespectrais de SRS no córtex motor primário humano (M1). (A-F) Seis regiões representativas mostrando: esquerda: imagens de histologia Raman estimulada (SRH) fornecendo visualização de tecido semelhante a H&E; meio: resultados de agrupamento de k-means correspondentes; À direita: perfis espectrais Raman específicos do cluster com intensidades médias (linhas sólidas) e desvios padrão (áreas sombreadas). Número de centróides variando de 4 a 6 médias. O agrupamento não supervisionado foi usado, de modo que cores diferentes nas imagens agrupadas representam composições químicas distintas identificadas pelo algoritmo k-means. Cada aglomerado (representado por uma cor única) corresponde a regiões com perfis espectrais semelhantes na região de alongamento do CH. Barra de escala = 20 μm. Abreviaturas: SRH = histologia SRS. Clique aqui para ver uma versão maior desta figura.
Discussão
Este sistema multimodal é uma poderosa plataforma de imagem para capturar uma visualização holística do ambiente molecular de amostras em uma ampla gama de origens biológicas e condições patológicas. A vantagem de alavancar diferentes modalidades sem marcação reside na capacidade de adquirir informações complementares e direcionar analitos específicos que, de outra forma, seriam difíceis ou impossíveis em uma única técnica de imagem sem marcação. Especificamente, as três técnicas de imagem não linear (SRS, MPF, SHG) mencionadas neste artigo permitem a quantificação da composição de macromoléculas, dinâmica de energia por meio da razão redox óptica e informações estruturais, incluindo composição e morfologia da matriz extracelular 6,48,49. Além disso, em comparação com a utilização de cada modalidade de imagem separadamente, a aquisição de imagens com um sistema de microscópio combinado permite o registro imediato da imagem e tempos de armazenamento de amostras mais curtos. Realizamos até imagens de células vivas com essa abordagem, possivelmente devido à menor fotoexposição em comparação com a microscopia de fluorescência confocal, usando pontos de tempo selecionados para adquirir cada modalidade sequencialmente, conforme estipulado no protocolo acima. Além de suas vantagens individuais, a plataforma multimodal também se beneficia das múltiplas perspectivas sobre a condição biológica por meio da medição sem rótulos de diferentes biomarcadores.
A imagem sem marcação de proteínas autofluorescentes via MPF tem sido direcionada principalmente para a quantificação da razão redox óptica, uma medida de reações redox facilitadas pela oxidação de FAD em FAD + e redução de NAD + em NADH50. Este é um marcador significativo do metabolismo dentro das células e tecidos, pois fornece uma medida da atividade relativa entre a fosforilação oxidativa e a glicólise, as duas principais vias para a geração de ATP6. Especificamente, diminuições na concentração de NADH e aumentos no FAD+ são um marcador para o aumento da fosforilação oxidativa nas mitocôndrias; Enquanto isso, o inverso é verdadeiro para o aumento da glicólise no citoplasma. A tendência de preferir uma via metabólica à outra na produção de energia tem sido associada a várias alterações patológicas e ativações de vias ligadas ao câncer, destacando o potencial uso da ORR como marcador de detecção precoce 6,51.
Da mesma forma, a aquisição do sinal de colágeno fibroso SHG aproveita uma modalidade de imagem não linear sem marcação para quantificar e visualizar um marcador biológico para a saúde. O SHG pode rastrear efetivamente a distribuição do colágeno tipo I-III devido à sua estrutura não centrossimétrica. As proteínas fibrosas do colágeno medidas por SHG têm sido reconhecidas como importantes marcadores diagnósticos para várias doenças, incluindo câncer e fibrose 25,26,27,52,53. Além de sua detecção como marcador de doença, nossa aquisição de colágeno via SHG pode servir como um forte indicador de estrutura em células e tecidos devido ao papel do colágeno fibroso na composição da matriz extracelular e nos limites estruturais dos tecidos54,55. Por meio do sinal de colágeno SHG, podemos fazer observações informadas de células separadas ou unidades funcionais de tecido distintas sem a necessidade de um marcador exógeno para membranas celulares. No geral, a quantificação de SHG de fibrilas de colágeno apresenta um claro benefício tanto para a capacidade de diagnóstico quanto para a facilidade de análise quando integrada a outras técnicas de imagem sem marcadores.
Utilizando a microscopia SRS, podemos capturar vários marcadores para a composição de macromoléculas, particularmente para lipídios e ácidos graxos. Neste protocolo, descrevemos como adquirir o sinal SRS para ácidos graxos insaturados e saturados para análise raciométrica da insaturação lipídica dentro da mesma amostra. Um microscópio de varredura a laser vertical com uma objetiva de água de 25x foi aplicado para rendimento de infravermelho próximo. Os feixes de laser passaram pela amostra e foram coletados por um condensador de óleo de alta abertura numérica (1,4 NA). Um filtro passa-curto de alto diâmetro externo (950 nm) foi usado para bloquear o feixe de Stokes, permitindo apenas que o feixe da bomba atingisse um detector de fotodiodo de Si para detectar o sinal de perda Raman estimulado. A corrente de saída do fotodiodo foi terminada e filtrada. Posteriormente, um amplificador lock-in a 20 MHz demodulou a corrente em X com um deslocamento de fase zero, e um módulo de software usou o sinal demodulado para gerar a imagem durante a varredura a laser.
Estudos têm demonstrado que alterações na insaturação lipídica têm um efeito profundo na membrana celular e organela e uma desregulação no metabolismo lipídico pode ser um forte marcador para várias doenças, incluindo câncer e doenças neurodegenerativas 56,57,58,59. Para uma visão geral mais abrangente da desregulação lipídica, analisamos a composição do subtipo usando imagens hiperespectrais SRS. As imagens hiperespectrais SRS (HSIs) combinam as informações espaciais de alta resolução adquiridas pela microscopia SRS com o sinal quimicamente significativo da espectroscopia Raman por meio de sintonia sequencial, conforme descrito no protocolo. Esses espectros de pixels são então analisados em dois métodos, agrupamento via k-means e detecção de biomoléculas com PRM-SRS.
O agrupamento de K-means dos espectros de pixels resulta na separação de regiões dentro de uma imagem 2D por concentração e composição molecular devido à relação linear entre a intensidade do sinal SRS e a concentração da ligação molecular60,61. Por meio dessa metodologia, aplicamos o protocolo para determinar regiões que possuem fenótipos moleculares específicos, conforme prescrito pelos espectros de pixels e centróide espectral compartilhado. Para um exame completo da expressão molecular distinta em pixels específicos, implementamos o PRM-SRS para realizar a correspondência espectral entre um espectro de referência para uma molécula específica capturada com espectroscopia Raman espontânea e os espectros de pixel da aquisição de SRS HSI. Ao fazer isso, podemos determinar a probabilidade de expressão de moléculas distintas sem utilizar sondas ou rótulos exógenos. Essa análise demonstrou a capacidade de rastrear certos subtipos lipídicos em tecidos e modelos biológicos e a capacidade de identificar alterações patológicas nas alterações da composição lipídica e, portanto, dislipidemia47. Além disso, avanços recentes na melhoria da resolução espectral por meio da divisão espectral da propagação de pulso de picossegundos através de fibras de sílica incentivam ainda mais a aplicação de SRS HSI como uma modalidade para visualização bioquímica sem marcação62. Por meio desses métodos destacados no protocolo, estabelecemos uma plataforma para realizar análises raciométricas e espectrais abrangentes utilizando uma modalidade SRS sem rótulos.
Além da relativa complexidade e novidade dessas técnicas, existem algumas limitações importantes em nossa abordagem a serem consideradas. A aquisição de imagens MPF com um laser pulsado de picossegundo, ao contrário dos lasers de femtossegundo comumente utilizados, requer maior poder de excitação do fluoróforo e corre o risco de fotobranqueamento. Com isso em mente, otimizamos o protocolo acima para mitigar o potencial fotodano de nossa plataforma, reduzindo a potência do laser incidente na amostra. Além disso, embora estudos anteriores tenham demonstrado a capacidade de avaliar a razão redox óptica a partir de imagens de autofluorescência MPF de NADH e FAD, a microscopia de imagem de fluorescência ao longo da vida (FLIM) demonstrou maior precisão na avaliação do metabolismo energético 63,64,65. Isso se deve à capacidade de diferenciar concentrações ligadas e não ligadas a proteínas de NADH e FAD com base em seus tempos de vida de fluorescência distintos, Em comparação com as capacidades do MPF, isso permitiu que o FLIM fosse usado para estudos abrangentes de atividade metabólica de vias de energia distintas, incluindo fosforilação oxidativa e glicólise 16,63,64,65,66,67, 68. Embora este protocolo exclua o FLIM, esperamos que a modalidade seja integrada a esta plataforma de imagem em um futuro próximo, aprimorando a análise do metabolismo energético que realizamos atualmente. No entanto, com o protocolo estabelecido aqui, apresentamos um fluxo de trabalho e configuração de hardware para uma abordagem de imagem multimodal que pode medir o metabolismo da estrutura biológica de várias perspectivas.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Agradecemos à Dra. Gloria Pryhuber e aos membros de sua equipe HuBMAP por fornecerem fatias de tecido pulmonar humano. Agradecemos ao Dr. Kun Zhang por fornecer tecidos cerebrais humanos. Também agradecemos ao Dr. Gen-Sheng Feng por fornecer amostras de fígado de camundongo. Reconhecemos o apoio dos fundos NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, UCSD Startup, Sloan Research Fellow Award e CZI DAF2023-328667 Award.
Materiais
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Referências
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados