Method Article
用于研究细胞代谢的多模态光学成像平台
* 这些作者具有相同的贡献
摘要
利用结合无标记光学成像模式的多模式平台,我们开发了一种用于可视化和量化细胞动力学和代谢的方案。通过多光子荧光、二次谐波产生和受激拉曼散射显微镜成像,我们可以生成细胞和分子环境的整体概览。
摘要
光学成像技术在生物医学研究中至关重要,因为它们能够以高空间分辨率从生物标本中获取形态和功能信息。这些光学过程利用光子与细胞、组织或器官内的分子之间的各种光分子相互作用,例如散射、吸收、发射和谐波产生。虽然传统的生物医学成像历来专注于应用单一模式,但最近的研究表明,这些不同的技术提供了互补的见解,它们的综合输出提供了对衰老过程和疾病发展中的分子变化以及细胞生物学基础知识的更全面理解。
在过去的几十年里,无标记光学成像方法取得了进步,能够详细探索细胞和亚细胞环境。例如,多光子荧光 (MPF) 不仅有助于靶向蛋白质成像,还可以通过自发荧光辅酶量化代谢活性,从而实现高穿透深度和空间分辨率。二次谐波产生 (SHG) 用于对细胞外基质中的胶原蛋白等结构进行成像,而受激拉曼散射 (SRS) 则以亚细胞分辨率原位绘制化学键和分子组成。
我们开发了一个结合了 MPF、SHG 和 SRS 模式的多模态成像平台。将这些模式整合到一个平台中,可以从细胞、组织、器官甚至体内的相同定位获取多方面信息,从而有助于更详细地探索细胞代谢、细胞外基质结构和分子组成之间的复杂关系。这种多模式系统提供亚细胞分辨率、深层组织渗透、 原位 活细胞/组织成像以及无标记检测和瞬时共配准,无需位置调整、设备切换或分析后对准。在这里,我们提出了一种使用这种多模式平台进行无标记成像的方案,并展示了其在表征细胞代谢以及细胞和组织中的分子异质性以研究衰老和疾病方面的应用。
引言
光学生物医学成像对于促进我们对生物结构和功能的理解至关重要。图像是通过调制激发光和检测来自光-组织相互作用的信号生成的。第一台复合显微镜由 Hans 和 Zacharias Janssen 于 1590 年左右开发,在管中利用两个凸透镜,可提供高达 30x1 的放大倍率。经过几个世纪的进步,现代光学显微镜现在可以达到 1-3 nm的分辨率 2,3。除了提供高分辨率外,先进的成像系统现在还提供更深的组织穿透、更高的效率和最小的样品损伤,使其特别适用于活细胞和组织成像。无标记成像特别有利,因为它可以在不破坏细胞内过程或损害样品完整性的情况下捕获信息。
多光子荧光 (MPF) 显微镜,尤其是双光子荧光显微镜,已广泛用于无标记成像。与依赖于线性单光子吸收和发射的传统荧光显微镜不同,MPF 激发涉及同时吸收多个光子,其综合能量激发单个荧光团分子 4,5。这些光子通常在红外光谱中,具有单光子激发所需能量的一半或更少。在这个非线性过程中,较长的波长和焦点的局部激发导致较低的散射、更深的组织穿透和更低的光毒性。
通过检测来自内源性代谢底物(如还原烟酰胺腺嘌呤二核苷酸 (NADH) 和黄素腺嘌呤二核苷酸 (FAD))的自发荧光信号,可以通过无标记 MPF 显微镜捕获细胞代谢信息。这些辅酶表现出不同的激发和发射光谱,它们的荧光强度比,称为氧化还原比 (NADH/FAD),反映了细胞的氧化状态。自 Britton Chance 于 1979 年首次引入氧化还原比率的概念以来,已经提出了其他比率,包括 NAD(P)H/FAD、NAD(P)H/(FAD + NAD(P)H) 和 FAD/(FAD + NAD(P)H) 6,7,8,9。通过 MPF 成像量化这些光学氧化还原比率可提供对代谢动力学的宝贵见解。例如,MPF 成像可以根据癌细胞改变的新陈代谢来区分癌细胞和正常细胞,证明其用于癌症诊断的潜力 10,11,12。然而,基于 MPF 的自发荧光检测存在局限性。其他内在荧光团(如角蛋白)可能会导致荧光强度增加,从而导致光谱串扰和信号解释不准确13。此外,氧化还原比仅反映整体细胞氧化还原变化,不区分来自不同来源(例如细胞质或线粒体)的 NADH 或 NADH 和 NAD(P)H,因为两者都在 450 nm 处表现出相似的光谱峰,导致混合强度信号14。
二次谐波产生 (SHG) 是一种非线性光学过程,于 1980 年代首次在生物医学领域得到验证,已广泛用于细胞结构的无标记成像15,16。与 MPF 类似,二次谐波涉及从超快脉冲激光器同时吸收两个相同能量的光子。这些光子重新组合以发射出两倍于入射光频率的新光子,从而检测到二次谐波信号。这种非线性光学相互作用仅发生在非中心对称材料中,这些材料表现出非零二阶敏感性,以诱导极化以产生二次谐波信号17,18。这使得 SHG 对丝状蛋白质和纤维结构(如胶原蛋白、肌球蛋白和微管蛋白)的成像特别有效,而无需外源性荧光染料 15,17,19,20。纤维化和胶原蛋白的丰度、刚度、排列和结构异常在炎症和癌症等许多情况下普遍存在,这使得 SHG 成为对某些疾病状况进行高效和非侵入性检测的有前途的工具 21,22,23。SHG 成像在肿瘤学研究中的广泛应用,包括乳腺癌、卵巢癌和皮肤癌的研究,凸显了其在基础研究和潜在临床应用中的关键作用 24,25,26,27。
不同的分子表现出不同的振动能级,当被入射光激发时,会引起不同程度的非弹性散射——这种现象最早由 C. V. Raman 在 1928 年表征28。此后,拉曼效应已广泛用于光学显微镜中,用于检测分子和组织成分,而无需外源性标记。与传统的自发拉曼光谱相比,受激拉曼散射 (SRS) 和相干反斯托克斯拉曼散射 (CARS) 都一致地激发分子振动,并利用光的非线性相互作用来产生更强的信号。SRS 现象于 1962 年首次报道29。2008 年,这种机制被集成到三维多光子成像中,允许根据分子振动跃迁引起的泵和斯托克斯光束的强度变化对化学物质进行选择性检测30。这种方法最大限度地减少了非谐振背景干扰,产生了超过 CARS 的干净强度信号。SRS 成像擅长提供多重和高光谱成像,能够同时检测多个化学键,并允许对具有相当穿透深度的样品中的分子组成进行高分辨率可视化。虽然是一项相对较新的技术,但 SRS 成像已被证明在临床诊断和代谢研究中都有效,体内和体外30、31、32、33、34、35、36。例如,SRS 可以通过量化脂质与蛋白质的比率来区分脑肿瘤浸润的组织与皮层和白质,从而能够以无标记、无创的方式描绘肿瘤边缘37,38。此外,代谢改变通常被认为是衰老相关和癌症相关疾病的标志,可以使用 SRS 进行定量评估,方法是通过检测重水 (D2O) 处理的样品中的碳氘键来实现,从而允许定量测量蛋白质合成、脂肪生成和其他大分子代谢过程 31,33,34,35,36.SRS 能够以高时间和空间分辨率跟踪代谢物,使其成为一种很有前途的疾病调查和诊断工具,具有更广泛的临床应用潜力。
多模态成像已成为生物医学研究中一种强大的方法,它集成了两种或多种成像模式,以更全面地了解同一标本中的复杂生物系统。2018 年,引入了一种无标记自荧光多谐波 (SLAM) 显微镜技术,集成了双光子荧光 (2PF)、三光子荧光 (3PF)、SHG 和三次谐波产生 (THG)39。这种方法有助于同时可视化肿瘤微环境中的细胞相互作用、动态过程和单个成分。SLAM 显微镜对样品的扰动最小,激光功率要求更低,可实现深层组织分析,并为活体监测提供更安全的方法40。另一种多模式模式,结合了本征荧光光谱、漫反射光谱和拉曼光谱,已被开发用于外科手术过程中的原位癌症检测41。此外,最近设计的多模态非线性内窥镜系统集成了 CARS、SHG 和双光子荧光 (TPF),已证明能够以亚微米和亚细胞空间分辨率对生物样品进行成像42。2PF 和 SRS 显微镜的组合同样用于组织、细胞和细胞器的高分辨率体内成像 42,43,44,45。这些新兴的多模态成像技术利用了单个模态的优势,从而提高了分辨率、穿透深度和图像采集效率,从而在临床和外科应用中显示出巨大的潜力。
这种多模态方法比单模态成像越来越受到青睐,因为它提供了更广泛的测量范围,同时减轻了与单个技术相关的局限性。如前所述,MPF 测量内源性荧光以反映代谢变化,SHG 可以对生物样品中的胶原蛋白等非中心对称结构进行成像,而 SRS 主要检测蛋白质和脂质,因为高密度的化学键会根据其振动模式产生独特的拉曼信号。鉴于它们的相干特性和非线性光学特性的共同原理,这些成像模式可以集成到利用超短脉冲激光器的单个显微镜设置中,从而允许在局部区域采集各种生物标志物,从而提供更完整的生物过程视图44,45.本文概述了一种实现多模态成像平台的协议,该平台集成了 MPF、SHG 和 SRS,用于生物医学研究应用。
研究方案
1. 无标记多模态成像实验
注意:该协议侧重于无标记多模态成像的设置和采集过程。
- 多模式显微镜的设置(图 1A)和系统校准
- 预热激光并等待大约 15-20 分钟。
- 按以下顺序打开控制单元和监视器的电源:控制箱 |触摸屏控制器 |用于主激光遥控器的 AC 适配器 用于副激光遥控器的 AC 适配器。
- 打开 Si 光电二极管检测器和锁相放大器的电源。
- 配置泵浦激光束和斯托克斯光束。使用可在 780 nm 至 990 nm 范围内调谐的泵浦光束、5 - 6 ps 脉冲宽度和 80 MHz 重复频率来设置激光系统。斯托克斯激光束的固定波长为 1,031 nm,脉冲为 6 ps,重复频率为 80 MHz。确保泵和斯托克斯光束都处于低功率(至少 20 mW),以便在对准板上可见。
- 在光路中放置一个对准板(就在激光盒之后)以检查泵浦和斯托克斯光束的空间重叠,并调整镜子,直到两个点在对准板的中心重叠。
注意:尽管泵浦和斯托克斯光束大致重叠,但微调两束激光束的空间重叠可以获得最佳的 SRS 信号。 - 空间重叠后,首先单击 OPO 控制 软件,使用 PSD (位置敏感探测器) 进行微调。使用 十六进制键 仔细调整光学反射镜 1 (OM1) 并观察 PSD 显示以了解 X-Y 位置的变化,以争取与中心的最小偏差。然后,要进行更精确的调整,请使用光学反射镜 2 (OM2) 上的 十六进制键 ,直到位置指示器位于 PSD 显示屏的中心。
注意:OM1 和 OM2 是 OPO 头中的两个镜子,用于调整激光对准。 - 最后,通过转动两个聚光镜居中螺钉将聚光镜居中,将光圈图像移动到视场中心。
注意:场光阑可以限制进入物镜的光束直径,从而排除外来光,从而提高图像对比度。
- 无标记多模态成像采集步骤
- 将油涂抹在高数值孔径 (1.4 NA) 油聚光镜上,然后将显微镜载玻片安装到涂油的聚光镜上。最后,将一个大水滴放在 25 倍水物镜的显微镜载玻片上。确保显微镜安装牢固且不可移动,然后调整 z 载物台以调整焦距,直到在 25 倍水物镜下可以看到生物样品的明场图像。
- 以正确的顺序开始成像过程:MPF、SHG 和 SRS,以避免光漂白,这将影响 MPF 的图像质量和精确分析。要在 MPF 和 SHG 之间快速切换,请从泵梁切换到固定的 Stokes 梁。
注意:将泵浦激光器从 800 nm(NADH 和 FAD 自发荧光)调整到 791.3 nm(CH3 拉曼偏移)可能需要 1-2 分钟。 - 为每种模态选择图像分辨率(512 x 512 像素)和合适的停留时间。对 MPF 和 SHG 使用 8 μs/像素,平均帧数高于 3。使用 40 μs/像素,平均第 2 帧用于 SRS 模式。
注意:平均帧是指在保存之前要平均的图像数量。 - 要使用 MPF 获得自发荧光,请关闭 Stokes 激光束并将泵浦激光调谐到 800 nm,以激发 NADH 和黄素。将 FVOPT 滤光片立方体安装到光路中,NADH 和黄素分别为 460 ± 10 nm 和 515 ± 10 nm。因此,样品的功率约为 15 mW。
- 使用 SHG 获取胶原纤维信号。关闭泵浦激光束,仅使用 stokes 激光束,并将功率设置为 500 mW。仅获取与 515 nm 滤光片相关的通道。
- 使用 SRS 获得蛋白质和脂质的空间分布。保持两个激光束都打开并调整激光束频率以匹配每个分子的特定振动模式。
注:通常,791.3 nm 用于 CH 3,797 nm 用于 CH2,787 nm 用于不饱和脂质,794.6 nm 用于饱和脂质。因此,样品的功率约为 40 mW。 - 要获取 SRS 高光谱图像数据集,请打开 OPO 控制软件以选择扫描,将波长范围设置为 780 nm 至 806.5 nm,然后选择至少 60 的堆栈编号。然后,捕获高光谱图像堆栈。
注:生成的数据集包含 60 张图像,每张图像代表特定拉曼位移(从 2,700 cm-1 到 3,150 cm-1)的空间分布。高光谱采集的时间取决于图像的视场 (FOV) 和扫描时间,但对于 512 x 512 图像,大约在 5 分钟到 8 分钟之间。对于较大的图像,应注意尽量减少激光下的持续时间,以防止 2 小时后可能发生任何光损伤(取决于样品的厚度和 FOV 大小)。 - 将相同感兴趣区域 (ROI) 的所有图像保存在同一文件夹中。图像格式为 Olympus .oir 文件。
2. 图像分析
- 使用图像处理软件打开所有保存的原始图像以分配颜色并添加比例尺用于图像显示。还使用它来生成 NADH 和饱和脂肪酸 (SFA,2,880 cm-1) 图像的二元掩码,用于光学氧化还原比率和脂质不饱和度分析。
注意:确保蒙版强度只有 0 和 1。 - 使用自建的 python 脚本进行任何下游分析(代码可在 https://github.com/lingyanshi2020/HSI_Analysis 获取)。
注:此脚本旨在处理来自对照和癌症样本的多个感兴趣区域 (ROI)。该脚本的主要功能只需要两个输入:control 和 cancer image 文件夹的文件路径。- 遍历文件夹,匹配具有相同基本名称的图像(例如,“roi_1_flavin.tif”、“roi_1_nadh.tif”、“roi1_mask.tif”)。
- 对于每组匹配的图像,让脚本执行比率分析,并根据这些计算生成新的比率图像。
光学氧化还原比计算为黄素 / (NADH + 黄素)。
脂质不饱和度的计算公式为:USFAs / (SFAs + USFAs),其中 USFAs 代表不饱和脂肪酸。 - 然后,图像中的所有比率值都存储在 pandas DataFrame 中,以便于作。执行单因素方差分析,并使用带有 styple “ggplot” 或 seaborn 的 matplotlib 进行条形图或箱线图进行量化。
- 使用 2,930 cm-1 (CH3) 和 2,850 cm-1 (CH2) 图像生成减去的 (CH3 - CH2) 图像。然后,将 CH2 图像 CH3 - CH2 图像在 RGB 色彩空间中与模拟 H&E 染色的自定义 LUT 混合,以生成数字组织学图像46。
- 采集高光谱 SRS 图像后,使用自制的 python 脚本进行 k-means 聚类,以根据光谱相似性对图像进行分割(代码可在 https://github.com/lingyanshi2020/HSI_Analysis 获得)。
- 使用 tifffile 包加载 .tiff 图像堆栈。将 SRS 高光谱图像堆栈(x、y、光谱)重塑为 2D 数组(像素、光谱)。
- 使用 scikit-learn 软件包将 k-means 算法部署到具有定义数量的集群的 2D 数组。
- 将生成的群集标签重塑回原始图像尺寸,从而创建分段图。
- 为每个簇分配唯一的颜色,生成代表组织内不同生化成分的伪彩色图像。
- 使用 matplotlib 用平均值(实线)和标准差(阴影面积)绘制每个簇的光谱
- 使用 MATLAB 遵循惩罚参考匹配 SRS (PRM-SRS) 协议,使用 SRS 高光谱图像数据集创建空间无标记脂质亚型检测47。
- 组织图像并将其保存为 300 dpi .tif格式。
结果
图 2 中的图像代表了遵循获取 FAD 和 NADH 自发荧光的方案以及蛋白质、总脂质、不饱和脂质和饱和脂质的四个 SRS 通道获得的结果。在这里,我们还通过 RGB 颜色混合生成协议中描述的伪组织学图像。采集 MPF 和 SRS 通道可提供图像文件,稍后将用于比率分析。这种分析的一个例子可以在图 3 中看到对人类肺组织的分析。在协议中采集图像后,我们利用 Python 或 ImageJ 的图像分析方法利用不同通道的比率来提供定量代谢信息。如图 3 的脂质不饱和度和光学氧化还原比率图像所示,比率分析提供了相对代谢活性和分子组成分布的彩色图。我们利用这些测量来观察特定组织的代谢途径和脂质含量的变化、病理或不同的生物因素。如图 4 所示,这可以通过比较氧化应激和脂质不饱和度的平均值以及其在 2D 显微镜图像中的分布来提供健康和肿瘤组织的定量比较。
对于 MPF 和 SRS 图像的比率分析,我们通常以光学氧化还原和不饱和度比为目标,如方程 (1) 和 (2) 所示。
 (1)
(1)
 (2)
(2)
其中 IC 是来自通道 C 的像素强度。因此,我们测量每个像素的比率并获得这些代谢和分子标志物的空间分布。
除了比率分析之外, 图 4 还展示了我们的多模态平台的另一个潜在应用途径:高光谱图像分析。如该协议所述,我们可以通过利用可调谐泵浦光束在多个激光波长上执行扫描来获取 SRS 高光谱图像。这使我们能够重建显微镜图像中每个像素的拉曼位移光谱的 CH 区域,将拉曼光谱提供的化学信息和光学显微镜提供的空间信息结合起来。在图像分析工作流程中,我们重点介绍了为分析这些高光谱图像而实施的两种技术:用于生物分子检测的 PRM-SRS 和 k-means 聚类。 图 4E 显示了 PRM-SRS 在小鼠肝肿瘤图像上的应用。PRM-SRS 算法提供每个像素的光谱之间的相关性,通过对拉曼光谱偏移的惩罚进行调整;然后,该算法为每种脂质生成概率分布图像。
像素光谱的 K-means 聚类是我们实施的另一种技术,用于可视化特定光谱表型的分组,从而能够分别通过强度和光谱形状按键浓度和分子组成进行分离。 图 5 提供了一个示例,说明如何在我们的协议中应用通过 k-means 聚类进行的图像分析。我们预计 k 均值聚类在结构上与假组织学数字中的特征保持一致,此外还从不容易从 SRS 通道或假苏木精和伊红组织学染色 (H&E) 中区分的特征中提供进一步的聚类。PRM-SRS 和 k-means 聚类分析通过提供有关特定分析物和分子键的化学和半靶向信息,无需外源性探测,补充了这种无标记成像平台。
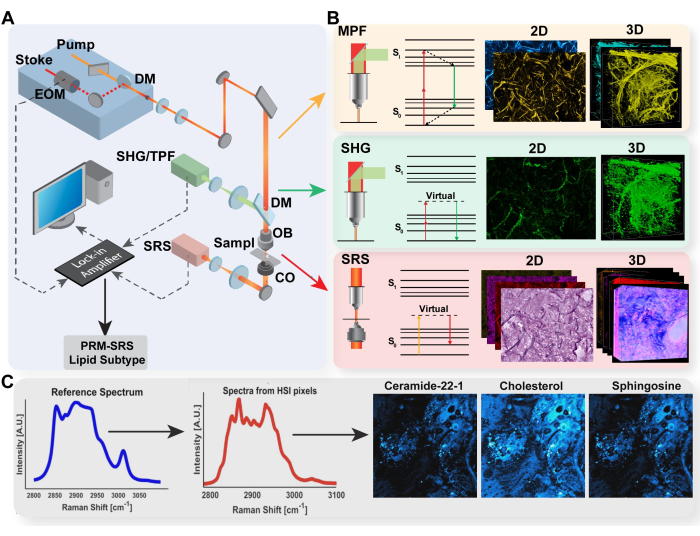
图 1:多模态成像平台和 PRM-SRS 生物分子检测图。 (A) MPF/TPF、SHG 和 SRS 成像系统的硬件设置示意图。(B) TPF 、 SHG 和 SRS 的光程和 Jablonski 图,以及每种模态的代表性采集图像。(C) 自发拉曼光谱和 SRS 高光谱成像之间光谱参考匹配的 PRM-SRS 工作流程图。缩写:MPF = 多光子荧光;TPF = 双光子荧光;SHG = 二次谐波产生;SRS = 受激拉曼散射;PRM = 惩罚参考匹配。 请单击此处查看此图的较大版本。

图 2:来自人类初级运动皮层组织 (M1) 的 MPF 和 SRS 通道。 (A,B)用于 Flavin/FAD 和 NADH 的 MPF 自发荧光通道。(C-F)在特定拉曼位移峰处捕获的 SRS 通道;蛋白质的 CH3 不对称拉伸峰 (2,930 cm-1),脂质的 CH2 不对称拉伸峰 (2,845 cm-1),饱和脂肪酸的 2,885 cm-1 峰和不饱和脂肪酸的 3,010 cm-1 峰。(G) 合并的 SRS 蛋白(蓝色)和脂质(绿色)通道,以勾勒出它们各自的空间分布。(H) 镜像 H&E 染色的 SRS 伪组织学图像 (SRH)。比例尺 = 20 μm。缩写:MPF = 多光子荧光;FAD = 黄素腺嘌呤二核苷酸;NADH = 还原烟酰胺腺嘌呤二核苷酸;SRS = 受激拉曼散射;SRH = SRS 组织学。 请单击此处查看此图的较大版本。
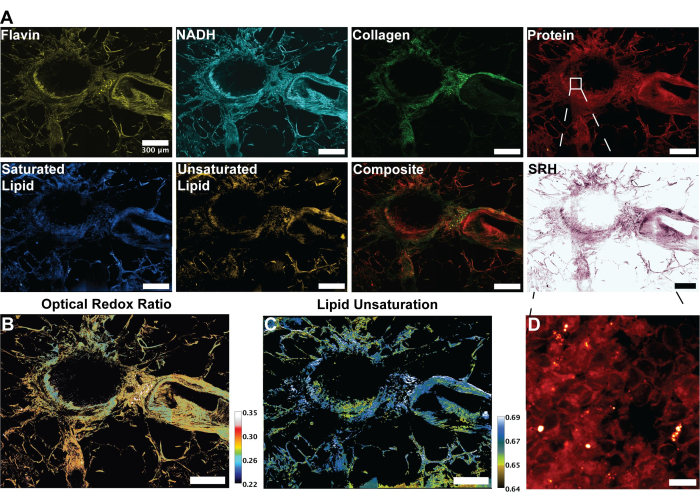
图 3:MPF、SHG 和 SRS 图像以及人体肺组织分析。 (A) 黄素/FAD 和 NADH 的 MPF 自发荧光、胶原纤维的 SHG 信号、蛋白质 (2,930 cm-1)、脂质 (2,845 cm-1)、饱和脂质 (2,885 cm-1) 和不饱和脂质 (3,010 cm-1) 峰的 SRS 信号,以及多种模式的复合合并图像。(B) 光氧化还原比的比率图像。(C) 脂质不饱和度的比率图像。(D) 支气管内层上皮细胞的放大图像。比例尺 = 200 μm。缩写:MPF = 多光子荧光;SHG = 二次谐波产生;SRS = 受激拉曼散射;FAD = 黄素腺嘌呤二核苷酸;NADH = 还原的烟酰胺腺嘌呤二核苷酸。 请单击此处查看此图的较大版本。

图 4:小鼠肝肿瘤的比率和高光谱分析。 (A) NAD[P]H 和 FAD 的 MPF 自发荧光图像,总蛋白、饱和和不饱和脂质的 SRS 通道,以及健康对照小鼠肝脏(上图)和肿瘤(下图)组织的假组织学图像。(B) 氧化应激(黄-绿),通过光学氧化还原比测量,以及从对照(左)和肿瘤(右)组织计算的脂质不饱和度(蓝-红)。比例尺 = 100 μm。(C,D) 条形图显示对照(蓝色)和肿瘤(红色)肝脏样品之间氧化应激和脂质不饱和度的比率差异。显著性统计量:* 对于 p ≤ 0.05 的显著性,** 对于 p 的显著性 ≤ 0.01。(E) 高光谱图像的 PRM-SRS 脂质亚型分析结果。从左到右:用于结构参考的 SRS 蛋白通道,比例尺 = 100 μm,脂质亚型(TAG 18:1、胆固醇和 C24:0 神经酰胺)的概率分布图像。来自 SRS HSI 的脂质亚型参考光谱和整个 CH 区域的平均像素光谱图。缩写:MPF = 多光子荧光;SRS = 受激拉曼散射;NAD[P]H = 还原的烟酰胺腺嘌呤二核苷酸磷酸;FAD = 黄素腺嘌呤二核苷酸;HSI = 高光谱图像。 请单击此处查看此图的较大版本。

图 5:SRS 高光谱 k-means 在人类初级运动皮层 (M1) 上的聚类。 (A-F) 六个代表性区域显示:左:刺激拉曼组织学 (SRH) 图像,提供类似 H&E 的组织可视化;中:相应的 k-means 聚类结果;右:具有平均强度(实线)和标准差(阴影区域)的簇特定拉曼光谱图。质心数范围为 4 到 6 表示。使用了无监督聚类,因此聚类图像中的不同颜色表示由 k-means 算法识别的不同化学成分。每个聚类(由唯一颜色表示)对应于 CH 拉伸区域中具有相似光谱图的区域。比例尺 = 20 μm。缩写:SRH = SRS 组织学。 请单击此处查看此图的较大版本。
讨论
这种多模态系统是一个强大的成像平台,用于捕获各种生物来源和病理条件下样品分子环境的整体可视化。利用不同无标记模式的优势在于能够获取互补信息并靶向特定分析物,否则在单一无标记成像技术中可能难以或不可能。具体来说,本文中提到的三种非线性成像技术(SRS、MPF、SHG)允许量化大分子组成、通过光学氧化还原比的能量动力学和结构信息,包括细胞外基质组成和形态 6,48,49。此外,与单独使用每种单一成像模式相比,使用一个组合显微镜系统进行成像可以立即进行图像配准并缩短样品储存时间。我们甚至用这种方法进行了活细胞成像,可能是由于与共聚焦荧光显微镜相比,光曝光较低,按照上述协议的规定,使用选定的时间点按顺序获取每种模式。除了各自的优势外,多模式平台还通过对不同生物标志物的无标记测量,从多个角度了解生物状况。
通过 MPF 对自发荧光蛋白进行无标记成像主要针对光学氧化还原比率的定量,光学氧化还原比率是通过将 FAD 氧化成 FAD+ 和将 NAD+ 还原成 NADH50 来促进氧化还原反应的量度。这是细胞和组织内代谢的重要标志物,因为它提供了氧化磷酸化和糖酵解之间相对活性的测量,这是 ATP 第6 代的两个主要途径。具体来说,NADH 浓度的降低和 FAD+ 的增加是线粒体中氧化磷酸化增加的标志物;同时,细胞质中糖酵解的增加则相反。在能量产生中偏爱一种代谢途径而不是另一种代谢途径的倾向与与癌症相关的多种病理变化和途径激活有关,突出了 ORR 作为早期检测标志物的潜在用途 6,51。
同样,SHG 纤维胶原信号的采集利用非线性无标记成像模式来量化和可视化健康的生物标志物。由于其非中心对称结构,SHG 可以有效地跟踪 I-III 型胶原蛋白的分布。通过 SHG 测量的纤维胶原蛋白已被公认为包括癌症和纤维化在内的多种疾病的重要诊断标志物 25,26,27,52,53。除了作为疾病标志物的检测外,由于纤维胶原在细胞外基质的组成和跨组织结构边界中的作用,我们通过 SHG 获得胶原蛋白还可以作为细胞和组织结构的有力指标54,55。通过 SHG 胶原信号,我们可以对单独的细胞或不同的组织功能单元进行明智的观察,而无需细胞膜的外源性标记物。总体而言,当与其他无标记成像技术结合使用时,胶原纤维的 SHG 定量在诊断能力和易于分析方面具有明显优势。
利用 SRS 显微镜,我们可以捕获大分子组成的多个标记物,特别是脂质和脂肪酸的标记物。在该协议中,我们描述了如何获取不饱和脂肪酸和饱和脂肪酸的 SRS 信号,以便对同一样品中的脂质不饱和度进行比率分析。使用带有 25 倍水物镜的正置激光扫描显微镜进行近红外通量。激光束穿过样品,由高数值孔径油聚光镜 (1.4 NA) 收集。使用高外径短波通滤光片 (950 nm) 来阻挡斯托克斯光束,同时只允许泵浦光束到达 Si 光电二极管检测器以检测受激拉曼损耗信号。光电二极管的输出电流被端接并过滤。随后,一个 20 MHz 的锁相放大器用零相移解调 X 轴上的电流,软件模块在激光扫描过程中使用解调信号生成图像。
研究表明,脂质不饱和度的变化对细胞和细胞器膜有深远的影响,脂质代谢失调可能是多种疾病的强烈标志,包括癌症和神经退行性疾病 56,57,58,59。为了更全面地概述脂质失调,我们使用 SRS 高光谱成像分析亚型组成。SRS 高光谱图像 (HSI) 通过协议中描述的顺序调谐,将 SRS 显微镜获取的高分辨率空间信息与拉曼光谱的化学重要信号相结合。然后以两种方法分析这些像素光谱,通过 k-means 进行聚类和使用 PRM-SRS 进行生物分子检测。
由于 SRS 信号强度和分子键浓度之间的线性关系,像素光谱的 K 均值聚类导致 2D 图像中的区域按分子浓度和组成分离60,61。通过这种方法,我们应用该协议来确定具有像素光谱和共享光谱质心规定的特定分子表型的区域。为了彻底检查特定像素中的不同分子表达,我们实施了 PRM-SRS,以在自发拉曼光谱捕获的特定分子的参考光谱与 SRS HSI 采集的像素光谱之间进行光谱匹配。在此过程中,我们可以确定不同分子的表达概率,而无需使用外源探针或标记。该分析证明了跨组织和生物模型跟踪某些脂质亚型的能力,以及识别脂质成分变化的病理变化从而识别血脂异常的能力47。此外,通过二氧化硅纤维对皮秒脉冲传播进行光谱分割来提高光谱分辨率的最新进展进一步激励了 SRS HSI 作为无标记生化可视化模式的应用62。通过协议中强调的这些方法,我们建立了一个平台,利用无标记 SRS 模式进行全面的比率和光谱分析。
除了这些技术的相对复杂性和新颖性之外,我们的方法还存在一些重要的局限性需要考虑。与常用的飞秒激光器相反,使用皮秒脉冲激光器获取 MPF 图像需要更高的荧光团激发功率,并且存在光漂白的风险。考虑到这一点,我们优化了上述方案,通过减少入射到标本上的激光功率来减轻我们平台的潜在光损伤。此外,虽然以前的研究已经证明了从 NADH 和 FAD 的 MPF 自荧光成像中评估光学氧化还原比的能力,但荧光寿命成像显微镜 (FLIM) 在评估能量代谢方面表现出更高的准确性 63,64,65。这是由于能够根据它们不同的荧光寿命区分蛋白质结合和未结合浓度的 NADH 和 FAD,与 MPF 的能力相比,这使得 FLIM 可用于来自不同能量途径的代谢活动的综合研究,包括氧化磷酸化和糖酵解 16,63,64,65,66,67,68.虽然该协议不包括 FLIM,但我们预计该模式将在不久的将来集成到该成像平台中,从而增强我们目前进行的能量代谢分析。尽管如此,通过这里建立的协议,我们提出了一种多模态成像方法的工作流程和硬件设置,该方法可以从多个角度测量生物结构代谢。
披露声明
作者没有需要声明的利益冲突。
致谢
我们感谢 Gloria Pryhuber 博士和她的 HuBMAP 团队成员提供人肺组织切片。我们感谢 Kun Zhang 博士提供人类脑组织。我们还感谢 Gen-Sheng Feng 博士提供小鼠肝脏样本。我们感谢 NIHU54DK134301、NIH R01GM149976、NIH U01AI167892、NIH R01HL170107、NIH 5R01NS111039、NIH R21NS125395、NIH U54CA132378、UCSD 创业基金、斯隆研究员奖和 CZI DAF2023-328667 奖的支持。
材料
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
参考文献
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。