Method Article
Uso de montagem in vivo para construção de plasmídeo de alta eficiência
Neste Artigo
Resumo
A montagem in vivo é um método de clonagem independente de ligação que depende de enzimas intrínsecas de reparo de DNA em bactérias para montar fragmentos de DNA por recombinação homóloga. Este protocolo é eficaz em termos de tempo e custo, uma vez que são necessários poucos reagentes e a eficiência da clonagem pode chegar aos 99%.
Resumo
A montagem in vivo (IVA) é um método de clonagem molecular que utiliza enzimas intrínsecas presentes em bactérias que promovem a recombinação intermolecular de fragmentos de DNA para montar plasmídeos. Este método funciona transformando fragmentos de DNA com regiões de 15-50 pb de homologia em cepas de Escherichia coli de laboratório comumente usadas e as bactérias usam a via de reparo independente de RecA para montar os fragmentos de DNA em um plasmídeo. Este método é mais rápido e econômico do que muitos métodos de clonagem molecular que dependem da montagem in vitro de plasmídeos antes da transformação em cepas de E. coli . Isso ocorre porque os métodos in vitro requerem a compra de enzimas especializadas e a realização de reações enzimáticas sequenciais que requerem incubações. No entanto, ao contrário dos métodos in vitro , o IVA não demonstrou experimentalmente montar plasmídeos lineares. Aqui compartilhamos o protocolo IVA usado por nosso laboratório para montar rapidamente plasmídeos e subclonar fragmentos de DNA entre plasmídeos com diferentes origens de replicação e marcadores de resistência a antibióticos.
Introdução
A clonagem molecular engloba uma série de técnicas laboratoriais necessárias para produzir plasmídeos contendo DNA recombinante específico1. Essas técnicas onipresentes geralmente atuam como um gargalo no fluxo de trabalho experimental2. Muitas técnicas de clonagem molecular dependem da montagem de fragmentos de DNA in vitro usando uma série de reações enzimáticas antes da transformação em uma cepa hospedeira (por exemplo, Escherichia coli DH5a) para amplificação 3,4,5,6,7. Como os métodos de montagem de plasmídeos in vitro dependem de enzimas, a compra ou purificação de enzimas pode ser cara e demorada.
A montagem in vivo (IVA) é um método de clonagem molecular que se baseia na recombinação intermolecular de fragmentos de DNA em um hospedeiro adequado 8,9,10,11,12. A base deste método foi a observação de que cepas de clonagem de E. coli comumente usadas poderiam mediar a recombinação intermolecular de um primer de fita simples com um plasmídeo clivado por enzimas de restrição13. O uso de PCR para gerar extremidades de DNA homólogas para montagem de plasmídeos em E. coli foi descrito na década de 1990 e esse método foi denominado PCR de recombinação 3,14. A eficiência da PCR de recombinação foi relatada em aproximadamente 50%14. No entanto, esse método não foi amplamente adotado, provavelmente devido ao alto custo dos primers e ao risco de introdução de mutações indesejadas usando polimerases de baixa fidelidade para amplificar grandes fragmentos de DNA. Essas desvantagens foram significativas na década de 1990 e poderiam ser evitadas usando métodos de clonagem in vitro, como a clonagem mediada por enzimas de restrição.
Nos últimos anos, o custo dos primers diminuiu e novas polimerases comerciais aumentaram a fidelidade da amplificação por PCR. Como resultado, a PCR de recombinação foi revisitada como uma técnica de clonagem molecular rápida e econômica e renomeada como IVA 2,9,15,16. Com o aumento da viabilidade, o IVA foi explorado e o método foi otimizado para atingir eficiências de clonagem de até 99%16. As otimizações identificaram características do DNA homólogo, como número de pares de bases e temperatura de fusão, que maximizam a eficiência da recombinação in vivo 2,16. Uma análise mais aprofundada mostrou que até 6 fragmentos de DNA variando de 150 bp a 7 kbp poderiam ser montados de forma eficiente por IVA15. Além disso, nosso grupo recentemente usou o IVA para montar uma série de plasmídeos com diferentes de resistência a antibióticos e origens de replicação17. Neste estudo, a clonagem IVA foi altamente eficiente (71-100%) com um pequeno número de clones testados para cada plasmídeo (n = 2)17. Aqui descrevemos o protocolo IVA usado por nosso laboratório.
Protocolo
1. Projete primers ou fragmentos de DNA com extremidades homólogas
- Use um software de processamento de texto ou software especializado de análise de DNA para montar artificialmente o plasmídeo desejado.
NOTA: Muitas vezes é útil codificar por cores fragmentos de DNA de diferentes fontes e destacar regiões homólogas que serão usadas nas etapas de montagem a jusante. Além disso, também é útil codificar por cores o DNA homólogo para garantir a montagem direcional. - Projete primers que se liguem a cada fragmento de DNA e contenham 15-50 pb da sequência de DNA homóloga (Figura 1)2,15.
- Para aumentar a eficiência do IVA, certifique-se de que os fragmentos de DNA homólogos tenham temperaturas de fusão de 47-52 °C2.
NOTA: A temperatura de fusão pode ser determinada usando o software online18 ou a fórmula: Temperatura de fusão = 64,9 + 41 * (nG + nC - 16,4)/(nA + nT + nG + nC), onde n = número de G, C, A ou T na sequência 6,19. - Evite primers com sequências altamente repetitivas como CATCATCATCATCATCAT que codificam para uma etiqueta HIS, pois podem introduzir instabilidade20. Se uma sequência final de aminoácidos repetitivos for necessária para aplicações como inserir uma etiqueta poli-HIS, use códons alternativos como CATCACCATCATCACCAC para diminuir a repetição da sequência de ácidos nucleicos.
NOTA: Os primers podem ser encomendados de qualquer fornecedor preferido. Se os fragmentos de DNA forem sintetizados, as extremidades homólogas devem ser incorporadas ao fragmento de DNA sintetizado.
- Para aumentar a eficiência do IVA, certifique-se de que os fragmentos de DNA homólogos tenham temperaturas de fusão de 47-52 °C2.
2. Amplificação de produtos de DNA contendo extremidades homólogas
- Isole o DNA do plasmídeo ou os modelos de DNA genômico usando kits de isolamento de DNA padrão disponíveis comercialmente ou lise alcalina 6,17.
- Utilizar ADN molde (do passo 2.1) e iniciadores contendo regiões homólogas (passo 1.2) numa reação de PCR com uma polimerase de alta fidelidade disponível no mercado. Para limitar o transporte do molde de DNA em reações a jusante, use uma quantidade baixa de DNA molde (20 pg-1 ng) em cada reação. Consulte as recomendações do fabricante para componentes de mistura de reação de PCR específicos da enzima e condições recomendadas de ciclagem de PCR. Prossiga com a reação de PCR.
- Configure as reações de PCR (50 μL) para amplificar os fragmentos de DNA mostrados na Figura 2 para conter 100 μM de dNTPs, 0,2 μM de cada primer de DNA, DNA molde (50 pg de pSU19 e 1 ng de pSU18mCherry), 1x mistura de tampão de reação de PCR contendo Mg2+ (concentração final de 2 mM) e 1 U de DNA polimerase de alta fidelidade.
- Use um programa de ciclagem de PCR de touchdown para amplificar pSU19 e mCherry (Figura 2). Defina a etapa inicial de desnaturação em 95 ° C por 5 min, seguida por 10 ciclos de 95 ° C por 15 s, 55 ° C-0.5 ° C por ciclo por 15 s e 72 ° C por 1 min e, em seguida, 20 ciclos adicionais de 95 ° C por 15 s, 50 ° C por 15 s e 72 ° C por 1 min. Complete o ciclo de PCR com uma etapa final de extensão a 72 °C por 10 min.
NOTA: Se um esqueleto de plasmídeo for amplificado, ajuste a quantidade de molde de DNA com base no peso molecular: com ~ 20 pg / 1 kbp de DNA de plasmídeo por 50 μL de reação de PCR (50 pg de DNA para pSU19 (mostrado)). Além disso, ajuste os ciclos de PCR para minimizar o número de mutações introduzidas durante a amplificação: para amplificação de esqueletos de plasmídeo, apenas 20-25 ciclos de PCR são suficientes.
- Quando a reação de PCR estiver completa, remova uma alíquota (2 μL) da reação de PCR, combine com o corante de carga (até a concentração de 1x) e separe os fragmentos de DNA por eletroforese em gel de agarose21. Use um transiluminador ultravioleta (UV) ou LED para visualizar fragmentos de DNA que migraram no gel de agarose. Determine se os produtos de PCR são únicos e compare os produtos com a escada de DNA para verificar se eles têm o peso molecular esperado (Figura 2). Se nenhum produto de PCR com o peso molecular correto for visualizado, consulte as recomendações do fabricante para otimizar a reação de PCR.
NOTA: Quando os produtos de PCR não são únicos, mas há um fragmento de DNA abundante correspondente ao peso molecular esperado, uma reação de PCR adicional pode ser evitada. - Se houver vários fragmentos de DNA presentes no gel de agarose, excisar o fragmento de DNA correspondente ao peso molecular esperado do gel de agarose e usar um kit de extração de gel de agarose para recuperar o fragmento de DNA.
- Para remover o DNA molde metilado das reações de PCR, adicione 1 μL de DpnI diretamente ao tubo de reação. Incube as reações a 37 ° C por 15 minutos durante a noite.
NOTA: Esta etapa opcional é altamente recomendada para limitar a quantidade de DNA molde que é transportada nas reações a jusante. No entanto, esta etapa pode ser ignorada se concentrações muito baixas de DNA molde forem usadas na reação de PCR. - Use um kit de purificação de ácido nucleico para remover enzimas residuais, sais, dímeros de primer e outros produtos indesejados de DNA de baixo peso molecular resultantes de ensaios enzimáticos.
NOTA: As etapas 2.1-2.6 podem ser ignoradas se fragmentos de DNA forem sintetizados. - Quantificar a concentração de ADN dos fragmentos de ADN purificados por espectrofotometria ou estimativa em gel.
3. IVA (Figura 3)
- Calcule a quantidade de DNA necessária para cada reação IVA (recomenda-se 25-50 ng de DNA plasmidial). Use quantidades maiores de DNA de plasmídeo para plasmídeos com maior peso molecular (por exemplo, para plasmídeos de 2,3 kbp, use 25 ng de plasmídeo, mas quando os plasmídeos forem de 9 kbp, use 50 ng em cada reação). Calcule o volume de DNA inserido necessário para a reação; use uma proporção molar de 1:3 ou 1:5 de plasmídeo: insira o DNA.
NOTA: Calculadoras biológicas on-line ou as fórmulas fornecidas abaixo podem ser usadas para calcular a quantidade molar de um fragmento de DNA de fita dupla e determinar a proporção molar de cada fragmento de DNA.
Usamos a seguinte fórmula para calcular a quantidade molar de um fragmento de DNA de fita dupla: pmol = (peso em ng) × 1.000 / (pares de bases × 650 daltons). Além disso, as razões molares podem ser calculadas por: inserção de massa (g) = razão molar desejada entre inserto/plasmídeo × massa de plasmídeo (g) × razão entre inserção e comprimentos de plasmídeo6. - Combine o volume calculado de plasmídeo e insira o DNA em um tubo de microcentrífuga pré-resfriado de 1,5 mL e mantenha no gelo.
- Transferir o plasmídeo e inserir a mistura de ADN do passo 3.2 para uma alíquota (25-100 μL) de E. coli descongelada e quimicamente competente (por exemplo, E. coli DH5a;22).
NOTA: O volume de células competentes utilizadas depende do volume do plasmídeo e da mistura de ADN inserido; Esta mistura não deve exceder 10% do volume das células competentes. - Prossiga com uma transformação de choque térmico23.
- Incubar a mistura de E. coli quimicamente competente e DNA em gelo por 30 min.
- Transfira para um banho-maria a 42 °C por 1 min e, em seguida, transfira o tubo para gelo por 2 min.
- Transfira assepticamente o LB para o tubo para completar o volume para 1 mL.
- Transferir o volume de 1 ml para um tubo de cultura de vidro e colocá-lo numa incubadora agitada a 37 °C, 220 rpm ou à temperatura recomendada para a recuperação da estirpe bacteriana e/ou plasmídeo (intervalo típico de 25 °C-37 °C) e permitir a produção do marcador de selecção de antibióticos codificado por plasmídeo.
- Após as células terem se recuperado por 30 min a 1 h, deposite uma alíquota (100 μL de 1.000 μL) da reação de transformação em meio de seleção sólido e use um espalhador de células estéril para dispersar a alíquota pela placa de cultura de ágar. Colete o restante das células transformadas por centrifugação (13.000 × g por 1 min), descarte o sobrenadante, ressuspenda o pellet celular em 100 μL de meio estéril e deposite em meio de seleção sólido como acima. Deixe o líquido absorver em meio sólido por 30 min, inverta as placas de cultura. e coloque-os em uma incubadora durante a noite na temperatura recomendada para a cepa bacteriana e/ou plasmídeo.
4. Triagem para montagem correta de plasmídeo
- Após a incubação (etapa 3.5), remova as placas de cultura da incubadora e enumere as colônias por contagem direta se houver várias colônias bacterianas isoladas em cada placa de cultura. Selecione várias colônias (1-10) para triagem para determinar se elas contêm o plasmídeo montado desejado.
- Transferir meios de cultura estéreis suplementados com antibióticos apropriados para tubos de cultura estéreis (vidro ou plástico). Inocule o meio de cultura tocando 1 colônia com uma agulha de transferência estéril ou alça e transfira as células para o meio do tubo de cultura com a agulha de transferência. Coloque os tubos de cultura em uma incubadora agitável e incube por 16-18 h na temperatura recomendada para a cepa bacteriana e / ou plasmídeo (faixa típica de 25 ° C a 37 ° C).
- No dia seguinte, isole o DNA do plasmídeo das culturas bacterianas por lise alcalina6 ou usando um kit de isolamento de plasmídeo disponível comercialmente de acordo com as instruções do fabricante.
- Determine se os plasmídeos estão montados corretamente.
- Quantificar a concentração de DNA dos plasmídeos isolados por espectrofotometria ou estimativa de gel.
- Utilizar uma alíquota (50-150 ng) de ADN plasmidial para uma reacção diagnóstica de digestão por restrição (10 μL de volume final). Selecione enzimas de restrição com base em onde se espera que clivem o DNA do plasmídeo montado.
NOTA: Recomendamos duas reações separadas: (1) tratar o DNA do plasmídeo com uma enzima de restrição que deve clivar o plasmídeo em um único local e (2) tratar o DNA do plasmídeo com duas enzimas de restrição que devem extirpar todo ou parte do fragmento de DNA inserido. As enzimas de restrição estão disponíveis comercialmente e devem ser usadas seguindo as instruções do fabricante. - Quando a reação da enzima de restrição estiver completa, combine com o corante de carregamento (até a concentração de 1x), carregue toda a mistura em um gel de agarose e separe os fragmentos de DNA por eletroforese em gel de agarose21. Use um transiluminador UV ou LED para visualizar os fragmentos de DNA que migraram no gel de agarose. Compare fragmentos de DNA restritos com a escada de peso molecular para determinar se eles são o(s) peso(s) molecular(is) esperado(s) (Figura 4). Tome nota dos plasmídeos com os padrões de restrição enzimática esperados (chamados clones positivos); Descarte os tubos contendo plasmídeos que não têm o padrão de restrição esperado (chamados clones negativos).
- Para garantir que os clones positivos tenham a sequência de nucleotídeos esperada, envie uma alíquota de clones de plasmídeo positivos para sequenciamento.
NOTA: Recomenda-se o sequenciamento de plasmídeo completo (por exemplo, sequenciamento de nanoporos).
Resultados
Neste manuscrito, como exemplo de uso do IVA, seguimos o fluxo de trabalho do IVA fornecido para reclonar o quadro de leitura aberto mCherry no local de clonagem múltipla do plasmídeo pSU19 para gerar um plasmídeo idêntico ao pSU19mCherry (Figura 3)17. Os primers adequados para IVA foram projetados com base nas etapas 1.1 e 1.2 do protocolo. Em seguida, um kit de isolamento de plasmídeo foi usado para isolar pSU19 e pSU18mCherry, que serviram como modelos para reações de PCR para amplificar a estrutura do plasmídeo e inserir DNA, respectivamente (etapa 2.1 do protocolo). O esqueleto do plasmídeo pSU19 foi amplificado por PCR (pares de primers: pSU19_F: TAGGGTACCGAGCTCGAATTC e pSU19_R CCCGGGGATCCTCTAGAGTC) e o quadro de leitura aberto mCherry foi amplificado por PCR a partir de pSU18mCherry (pares de primers mCherry_F: ctctagaggatccccgggATGGTATCAAAAGGAGAGGAAG e mCherry_R: gaattcgagctcggtaccctaTTATCATTACTTGTACAGTTC; sequência de nucleotídeos minúsculos nos primers mCherry indicam nucleotídeos que são homólogos aos primers usados para amplificar o backbone pSU19; etapa do protocolo 2.2). A amplificação específica da PCR foi verificada por eletroforese em gel de agarose (etapa 2.3 do protocolo; Figura 2) e os produtos de PCR foram submetidos ao restante do fluxo de trabalho do dia 1 (etapas de protocolo 2.5-3.5; Figura 3).
No dia seguinte, as colônias na placa de transformação foram enumeradas (etapa 4.1 do protocolo; Figura 3, dia 2). Neste experimento, seis colônias eram visíveis; o número de colónias pode ser aumentado através da plaqueagem de uma alíquota maior da cultura recuperada ou do aumento da quantidade de ADN que é transformada nas células competentes. Se não houver colônias presentes nas placas de transformação, é importante primeiro verificar se as células competentes têm uma alta eficiência de transformação. Tivemos sucesso usando células competentes com eficiências de transformação variando de 10,7 a 10,9 unidades formadoras de colônias/μg de DNA pUC19 transformado. Se a eficiência da transformação não for uma preocupação, pode ser necessário aumentar a quantidade de DNA transformada nas células competentes e/ou aumentar o volume de células competentes ao realizar IVA com plasmídeos de maior peso molecular ou ao montar muitos fragmentos de DNA.
Selecionamos duas colônias para rastrear a montagem correta do plasmídeo (etapas do protocolo 4.1-4.3.3). No dia seguinte, seguimos o fluxo de trabalho para o dia 3 (Figura 3). Após o isolamento do plasmídeo, trate cada plasmídeo com XbaI ou XbaI e EcoRI, que devem clivar o plasmídeo montado corretamente uma ou duas vezes (Figura 4). Como pode ser visto na Figura 4, ambas as reações enzimáticas resultaram em um único produto de DNA correspondente a 2,3 kbp para o clone plasmidial 1, indicando que este é apenas pSU19. Os tratamentos enzimáticos do clone plasmidial 2 resultaram em um único produto de DNA de ~ 3 kbp após o tratamento com XbaI e dois produtos de DNA (2,3 kbp e 770 pb) após o tratamento com XbaI e EcoRI. A montagem correta do clone plasmidial 2 foi então verificada por análise de sequenciamento de plasmídeo completo (etapa 4.4 do protocolo; Figura 3, dia 3). Neste exemplo, a eficiência correta de montagem do plasmídeo foi de 50%. Raramente selecionamos mais de dois clones para a montagem correta do plasmídeo e normalmente temos 50-100% de eficiência de montagem do plasmídeo. Em nossa experiência, os clones negativos normalmente contêm (i) esqueleto plasmidial que é transportado do DNA molde que se recircularizou ou (ii) um produto de DNA híbrido que consiste em DNA molde que se recombinou em um plasmídeo híbrido. DpnI (etapa 2.5 do protocolo) limita o número de clones negativos. Se isso não melhorar a eficiência do IVA, recomenda-se verificar o design do primer de PCR e reamplificar os esqueletos do plasmídeo e os produtos de PCR.

Figura 1: Estratégia de design do primer IVA. O primeiro passo (Etapa 1) no projeto do primer IVA é usar um software para construir um arquivo de sequência do plasmídeo montado desejado in silico. A segunda etapa (Etapa 2) é projetar primers que amplificem a estrutura do plasmídeo perto da junção do plasmídeo e da sequência de DNA inserida. Os primers para a sequência de inserção devem ter nucleotídeos suficientes para ligar e amplificar especificamente o produto de DNA desejado e conter 15-50 pb da estrutura do plasmídeo adjacente para recombinação homóloga. As regiões de homologia no projeto do plasmídeo são codificadas por cores. Abreviatura: IVA = montagem in vivo . Clique aqui para ver uma versão maior desta figura.
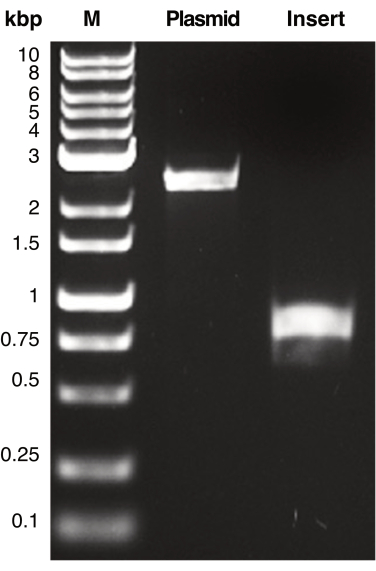
Figura 2: Amplificação por PCR do plasmídeo e do DNA inserido. É mostrada uma imagem representativa de fragmentos de DNA amplificados por PCR correspondentes a plasmídeo e DNA inserido contendo regiões de homologia. Os fragmentos de DNA foram separados por eletroforese em gel e visualizados por transiluminação UV. Os produtos de DNA foram comparados com o marcador de peso molecular (M) para determinar o peso molecular aparente de cada produto de PCR. O peso molecular esperado do plasmídeo (pSU19) e do DNA inserido (mCherry) é de 2,3 kbp e 770 pb, respectivamente. Clique aqui para ver uma versão maior desta figura.

Figura 3: Esquema do fluxo de trabalho de montagem in vivo. O DNA é amplificado por PCR para criar regiões homólogas de 5 'e 3'. DpnO tratamento I é usado para clivar o DNA modelo. Um kit de purificação de nucleotídeos é usado para remover sais e enzimas do DNA amplificado por PCR. A espectrofotometria UV é usada para determinar a concentração de DNA, e uma proporção de 3:1 entre insertos e molares de plasmídeo é combinada em um tubo de microcentrífuga pré-resfriado. O DNA combinado é transferido para um tubo de microcentrífuga gelada contendo uma alíquota de células competentes descongeladas de E. coli DH5α. O DNA é transformado em E. coli DH5α por choque térmico, espalhado em meio de seleção sólido e incubado a 37 ° C por 18 h. Após a incubação, colônias individuais contendo clones de plasmídeo são transferidas para tubos de cultura contendo meio líquido estéril e antibiótico e, em seguida, incubadas a 37 ° C com agitação por 18 h. No dia seguinte, o DNA do plasmídeo é isolado das células e o tratamento com enzimas de restrição é usado para rastrear clones positivos. Os clones positivos são então enviados para análise de sequenciamento para confirmar a sequência de DNA dos plasmídeos. Clique aqui para ver uma versão maior desta figura.

Figura 4: Eletroforese em gel para triagem de clones de plasmídeo positivos. Dois clones de plasmídeos diferentes foram isolados e tratados com enzimas de restrição que devem clivar clones positivos uma vez para linearizar o plasmídeo ou duas vezes para liberar o DNA inserido. Após o tratamento com enzima de restrição, as amostras foram separadas por eletroforese em gel e visualizadas por transiluminação UV. O peso molecular foi estimado comparando-se com o marcador de peso molecular. O peso molecular esperado de um clone positivo linearizado (plasmídeo + inserto) foi de 3,07 kbp, e os pesos moleculares esperados de fragmentos de DNA após um clone positivo ser clivado duas vezes foi de 2,3 kbp (esqueleto plasmidial) e 770 bp (inserto). O clone de plasmídeo 1 é negativo (plasmídeo vazio) e o clone 2 é um clone positivo. Abreviaturas: SC = clivagem simples; DC = clivagem dupla; M = marcador de peso molecular. Clique aqui para ver uma versão maior desta figura.
Discussão
A etapa mais crítica para a clonagem bem-sucedida do IVA é o design do fragmento de DNA e do primer. A eficiência do IVA é muito melhorada quando os fragmentos homólogos são projetados para ter pelo menos 15 pb de comprimento com uma temperatura de fusão de aproximadamente 47-52 °C. Um estudo detalhado explorando a otimização do design do fragmento IVA foi publicado16. Outro passo importante para ter alta eficiência de clonagem é usar o mínimo possível de DNA molde nas etapas de amplificação por PCR. Para reduzir ainda mais o transporte de DNA molde, tratamos nossas reações de PCR pós-ciclo com DpnI para clivar o DNA molde metilado. Isso pode diminuir significativamente a quantidade de triagem necessária para isolar um clone positivo. Em nossa experiência, com o tratamento DpnI, precisamos apenas rastrear 1-2 colônias por transformação; sem o tratamento com DpnI, precisamos rastrear de 5 a 10 colônias por transformação.
Embora nós e outros tenhamos relatado que a clonagem de IVA produz muitas unidades formadoras de colônias16,17, é possível que nenhuma colônia esteja presente nas placas de transformação. Em nossa experiência, aumentar a quantidade de DNA transformado em células competentes (especialmente quando se trabalha com um grande esqueleto de plasmídeo) e/ou aumentar o volume de células competentes usadas pode melhorar a eficiência da transformação. Se nenhuma colônia for visível após o ajuste da quantidade de DNA ou células competentes, confirmamos experimentalmente a eficiência de transformação dos estoques de células competentes e sequenciamos nossos fragmentos lineares de DNA para confirmar que eles contêm as regiões homólogas desejadas. Seguir essas etapas de solução de problemas normalmente melhora a eficiência do IVA.
O método de clonagem do IVA tem várias limitações. Estes estão relacionados ao uso de PCR para amplificar fragmentos específicos de DNA. Por exemplo, os esqueletos do plasmídeo podem variar em tamanho, com alguns se aproximando ou ultrapassando 10 kbp de comprimento (por exemplo, pMMB67HE24, pFUSE25). Se a estrutura do plasmídeo desejada for muito grande ou difícil de amplificar com uma polimerase de revisão de alta fidelidade, ainda há a opção de linearizar o plasmídeo com enzimas de restrição (por exemplo, SmaI ou EcoRV) e usar o plasmídeo linearizado em vez de um produto de PCR para a reação IVA16. Em nossa experiência, a eficiência do IVA é significativamente diminuída se a linearização do plasmídeo próximo a 100% não for alcançada pelo tratamento com enzimas de restrição.
Outra limitação potencial do IVA é o custo associado à compra de novos primers contendo saliências de DNA homólogas para clonar cada fragmento de DNA único. Esse custo pode ser parcialmente aliviado se um laboratório usar um número limitado de esqueletos de plasmídeo e puder usar primers universais para gerar um grande lote de esqueleto de plasmídeo amplificado para futura clonagem de IVA. Além disso, se todos os plasmídeos usados no laboratório tiverem locais de clonagem múltiplos idênticos, os mesmos primers universais podem ser usados para todos os plasmídeos. Outra limitação do método IVA é que, até onde sabemos, ele não foi testado para a montagem de plasmídeos lineares que são normalmente usados para clonagem de DNA repetitivo ou instável26.
O IVA é um método de clonagem com boa relação custo-benefício e tempo que apresenta várias vantagens em comparação com outros métodos de clonagem molecular que dependem da montagem de fragmentos de DNA in vitro. Geralmente é econômico, pois a montagem do plasmídeo não depende de enzimas ou tampões adquiridos comercialmente para montar o DNA 4,5,27. Isso economiza tempo e reduz custos, pois as etapas típicas da reação enzimática são evitadas, o que pode economizar até 72 h de manipulações laboratoriais 2,15. Além disso, o equipamento e os reagentes necessários para a IVA normalmente já estão presentes ou disponíveis em laboratórios que realizam rotineiramente a clonagem molecular.
Nosso grupo recentemente usou o IVA para gerar uma série de 24 plasmídeos com múltiplos locais de clonagem idênticos, três opções para o número de cópias de plasmídeo e cinco opções para marcadores de seleção de antibióticos17. Demonstramos como os primers universais podem ser usados para clonar genes em cada plasmídeo17. Neste estudo, a eficiência da clonagem foi de 71-100%, pois muito poucas colônias foram rastreadas para identificar clones positivos (sete rastreadas para cinco clones positivos)17. Outro estudo relatou taxas de eficiência de clonagem de IVA atingindo um máximo de 97% ± 1,9%, a taxa mais alta pode ser explicada pelos autores testando sistematicamente um grande número de clones para a montagem correta do plasmídeo16. O IVA também pode ser usado para aplicações como mutagênese dirigida ao local ou integração de primers contendo sequências de tags27. O IVA é uma técnica de clonagem versátil e eficiente que pode ser adaptada para muitas aplicações de clonagem molecular de rotina em laboratório.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
O HGB é financiado por um programa de Bolsas de Pós-Graduação do Canadá - Mestrado do Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá (NSERC) e um Prêmio de Doutorado do Reitor (Universidade de Saskatchewan). Este trabalho foi apoiado por um NSERC Discovery Grant (RGPIN-2021-03066), fundos iniciais (para JLT) da Universidade de Saskatchewan e Canada Foundation for Innovation John R. Evans Leaders Fund (Grant número 42269 para JLT). Os autores agradecem ao Sr. Eric Toombs por fornecer a fotografia da quantificação do DNA por espectroscopia UV.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 kb Plus DNA ladder | FroggaBio | DM015-R500 | DNA molecular weight marker |
| AccuReady M Single Channel 0.5-10 µL | Bulldog Bio | BPP020 | Pipettes for transferring liquid |
| AccuReady M Single Channel Pipettor Kit | Bulldog Bio | BPK100 | Pipettes for transferring liquid |
| Agar | BioShop Canada | AGR003.1 | Solidifying agent for growth medium |
| Agarose | Biobasic | D0012 | Make agarose gels for DNA electrophoresis |
| Biometra TOne | Analytikjena | 846-2-070-301 | Thermocycler |
| Caps for glass culture tubes | Fisher Scientific | 14-957-91 | Reusable caps for glass culture tubes |
| DNA primers | Integrated DNA Technologies | N/A | Bind and amplify template DNA in PCR reaction |
| DpnI | New England Biolabs | R0176S | Cleave methylated template DNA following PCR amplification |
| EcoRI | New England Biolabs | R3101L | Restriction enzyme, comes with appropriate buffer |
| Eppendorf microtubes 1.5 mL | Sarstedt | 72.690.300 | Microtube, 1.5 mL, conical base, PP, attached flat cap, molded graduations and frosted writing space |
| Ethidium bromide | Fisher Scientific | AAL0748203 | DNA visualization/intercalating agent, toxic |
| EZ-10 Spin Column Plasmid DNA Miniprep Kit | Biobasic | BS614 | Isolate plasmid DNA from overnight bacterial cultures |
| GelDoc Go-Gel system | Bio-Rad | 12009077 | Imaging DNA gels |
| Glass culture tubes | Fisher Scientific | 14-925E | Glass culture tubes |
| myGel Mini Electrophoresis System | Sigma-Aldrich | Z742288 | System used for gel electrophoresis |
| NanoDrop One | Thermo Fisher | ND-ONE-W | Determine DNA concentration |
| PCR Clean Up for DNA Sequencing | Biobasic | BT5100 | Purify PCR products |
| Petri dish | Sarstedt | 82.1473.011 | Petri dish 92 x 16 mm, PS, transparent, with ventilation cams |
| Pipette tip, 1000 µL | Sarstedt | 70.305 | Pipette tips for 100-1000 µL |
| Pipette tip, 2.5 µL | Sarstedt | 70.3010.265 | Pipette tips for up to 2.5 µL |
| Pipette tip, 20 µL | Sarstedt | 70.3020.200 | Pipette tips for up to 20 µL |
| Pipette tip, 200 µL | Sarstedt | 70.3030.020 | Pipette tips for 1-200 µL |
| Salt (NaCl) | Fisher Scientific | S271-10 | Component of bacterial growth medium (10 g/L) |
| Single 0.2 mL PCR tubes with flat cap | FroggaBio | TF-1000 | PCR tubes |
| Tryptone (Bacteriological) | BioShop Canada | TRP402.5 | Component of bacterial growth medium (10 g/L) |
| VeriFi DNA polymerase | PCR Biosystems | PB10.42-01 | High fidelity polymerase, the PCR buffer containing Mg2+ and dNTPs is provided with purchase |
| Xba I | New England Biolabs | R0145S | Restriction enzyme, comes with appropriate buffer |
| Yeast extract | BioShop Canada | YEX401.205 | Component of bacterial growth medium (5 g/L) |
Referências
- Bertero, A., Brown, S., Vallier, L. Methods of cloning. Basic Science Methods for Clinical Researchers. Chapter 2, 19-39 (2017).
- Garcia-Nafria, J., Watson, J. F., Greger, I. H. IVA cloning: A single-tube universal cloning system exploiting bacterial in vivo assembly. Sci Rep. 6, 27459 (2016).
- Bubeck, P., Winkler, M., Bautsch, W. Rapid cloning by homologous recombination in vivo. Nucleic Acids Res. 21 (15), 3601-3602 (1993).
- Avilan, L. Assembling multiple fragments: the Gibson assembly. Methods Mol Biol. 2633, 45-53 (2023).
- Stukenberg, D., et al. The Marburg Collection: A Golden Gate DNA assembly framework for synthetic biology applications in Vibrio natriegens. ACS Synth Biol. 10 (8), 1904-1919 (2021).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Engler, C., Marillonnet, S. Golden Gate cloning. Methods Mol Biol. 1116, 119-131 (2014).
- Ma, H., Kunes, S., Schatz, P. J., Botstein, D. Plasmid construction by homologous recombination in yeast. Gene. 58 (2-3), 201-216 (1987).
- Cao, P., Wang, L., Zhou, G., Wang, Y., Chen, Y. Rapid assembly of multiple DNA fragments through direct transformation of PCR products into E. coli and Lactobacillus. Plasmid. 76, 40-46 (2014).
- Conley, E. C., Saunders, V. A., Saunders, J. R. Deletion and rearrangement of plasmid DNA during transformation of Escherichia coli with linear plasmid molecules. Nucleic Acids Res. 14 (22), 8905-8917 (1986).
- Conley, E. C., Saunders, V. A., Jackson, V., Saunders, J. R. Mechanism of intramolecular recyclization and deletion formation following transformation of Escherichia coli with linearized plasmid DNA. Nucleic Acids Res. 14 (22), 8919-8932 (1986).
- Sung, W. L., Zahab, D. M. Site-specific recombination directed by single-stranded crossover linkers: specific deletion of the amino-terminal region of the beta-galactosidase gene in pUC plasmids. DNA. 6 (4), 373-379 (1987).
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci USA. 83 (19), 7177-7181 (1986).
- Jones, D. H., Howard, B. H. A rapid method for recombination and site-specific mutagenesis by placing homologous ends on DNA using polymerase chain reaction. Biotechniques. 10 (1), 62-66 (1991).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5alpha-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), e0137466 (2015).
- Chen, F., et al. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 21 (1), 24 (2021).
- Braun, H. G., Kanwal, N., Rivera Lopez, L. F., Thomassin, J. L. Generation of a plasmid series for rapid sub-cloning and use in various Enterobacteriaceae. J Biosci Bioeng. 138 (6), 478-487 (2024).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Res. 35 (Web Server issue), W43-W46 (2007).
- Wallace, R. B., et al. Hybridization of synthetic oligodeoxyribonucleotides to phi chi 174 DNA: the effect of single base pair mismatch. Nucleic Acids Res. 6 (11), 3543-3557 (1979).
- Bzymek, M., Lovett, S. T. Instability of repetitive DNA sequences: The role of replication in multiple mechanisms. Proc Natl Acad Sci USA. 98 (15), 8319-8325 (2001).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Sambrook, J., Russell, D. W. The inoue method for preparation and transformation of competent E. coli: "ultra-competent" cells. CSH Protoc. 2006 (1), (2006).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Fürste, J. P., et al. Molecular cloning of the plasmid RP4 primase region in a multi-host-range tacP expression vector. Gene. 48 (1), 119-131 (1986).
- Bäumler, A. J., et al. Identification of a new iron regulated locus of Salmonella typhi. Gene. 183 (1-2), 207-213 (1996).
- Godiska, R., et al. Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli. Nucleic Acids Res. 38 (6), e88 (2010).
- Watson, J. F., Garcia-Nafria, J. In vivo DNA assembly using common laboratory bacteria: A re-emerging tool to simplify molecular cloning. J Biol Chem. 294 (42), 15271-15281 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados