Method Article
高効率プラスミド構築のための In vivo アセンブリの利用
要約
In vivo assemblyは、ライゲーションに依存しないクローニング法で、細菌の内因性DNA修復酵素を利用して相同組換えによりDNA断片をアセンブルします。このプロトコルは、必要な試薬が少なく、クローニング効率が99 %にもなるため、時間と費用対効果の両方が高いです。
要約
In vivo assembly(IVA)は、細菌に存在する内因性酵素を利用してDNA断片の分子間組換えを促進し、プラスミドを組み立てる分子クローニング法です。この方法は、相同性が15〜50 bpの領域を持つDNA断片を一般的に使用される実験室の大腸菌株に変換し、細菌がRecA非依存的な修復経路を使用してDNA断片をプラスミドに組み立てることによって機能します。この方法は、大腸菌株に形質転換する前にプラスミドをin vitroで組み立てる多くの分子クローニング法よりも迅速で費用対効果が高いです。これは、in vitro法では特殊な酵素の購入と、インキュベーションを必要とする連続的な酵素反応の性能が必要であるためです。ただし、in vitro法とは異なり、IVAは線状プラスミドを組み立てることが実験的に示されていません。ここでは、私たちの研究室がプラスミドを迅速に組み立て、複製の起源と抗生物質耐性マーカーが異なるプラスミド間でDNAフラグメントをサブクローニングするために使用したIVAプロトコルを共有します。
概要
分子クローニングには、特定の組換えDNAを含むプラスミドを作製するために必要な一連の実験技術が含まれます1。これらのユビキタスな手法は、多くの場合、実験ワークフロー2のボトルネックとして機能します。多くの分子クローニング技術は、増幅のための宿主株(例:大腸菌DH5a)への変換に先立って、in vitroでDNA断片を組み立てることに依存しています3,4,5,6,7。in vitroプラスミドアセンブリ法は酵素に依存しているため、酵素の購入や精製にはコストと時間がかかる場合があります。
In vivo assembly(IVA)は、適切な宿主8,9,10,11,12におけるDNA断片の分子間組換えに依存する分子クローニング法である。この方法の基礎は、一般的に使用される大腸菌のrecA-クローニング株が、制限酵素13によって切断されたプラスミドと一本鎖プライマーの分子間組換えを媒介できるという観察であった。PCRを使用して大腸菌のプラスミドアセンブリの相同DNA末端を生成することは1990年代に報告され、この方法は組換えPCRと名付けられました3,14。組換えPCRの効率は約50%であると報告されました14。しかし、この方法は広く採用されていませんでした。これは、プライマーのコストが高いことや、低忠実度のポリメラーゼを使用して大きなDNA断片を増幅する望ましくない突然変異を導入するリスクがあるためです。これらの欠点は1990年代には重要であり、制限酵素媒介クローニングなどのin vitroクローニング法を使用することで回避できました。
近年、プライマーのコストは低下し、新しい市販のポリメラーゼによりPCR増幅の忠実度が向上しています。その結果、組換えPCRは迅速で費用対効果の高い分子クローニング技術として再検討され、IVA 2,9,15,16として再ブランド化されました。実現可能性が高まるにつれ、IVA をさらに検討し、分析法を最適化して最大 99% のクローニング効率を達成しました16。最適化により、塩基対の数や融解温度など、in vivoでの組換え効率を最大化する相同DNAの特徴が特定されました2,16。さらなる分析により、150 bpから7 kbpの範囲の最大6つのDNAフラグメントがIVA15によって効率的に組み立てられることが示されました。さらに、私たちのグループは最近、IVAを使用して、異なる抗生物質耐性カセットと複製の起源を持つプラスミドシリーズを組み立てました17。この研究では、IVAクローニングは非常に効率的(71-100%)であり、各プラスミド(n = 2)に対して少数のクローンが試験された17。ここでは、当研究室で使用しているIVAプロトコールについてご説明します。
プロトコル
1. 相同末端を持つプライマーまたはDNA断片の設計
- ワードプロセッシングソフトウェアまたは専用のDNA分析ソフトウェアを使用して、目的のプラスミドを人工的に組み立てます。
注:多くの場合、さまざまなソースからのDNA断片を色分けし、下流のアセンブリステップで使用される相同領域を強調すると便利です。さらに、相同DNAを色分けして方向性のある組み立てを確保することも役立ちます。 - 各DNA断片に結合し、15-50 bpの相同DNA配列を含むプライマーを設計します(図1)2,15。
- IVAの効率を高めるには、相同DNAフラグメントの融解温度が47〜52°C2であることを確認してください。
注:融解温度は、オンラインソフトウェア18または式を使用して決定することができる:融解温度= 64.9 + 41 *(nG + nC - 16.4)/(nA + nT + nG + nC)、ここで、n =シーケンス6,19のG、C、A、またはTの数。 - HISタグをエンコードするCATCATCATCATCATCATのような反復性の高い配列を持つプライマーは、不安定性20を引き起こす可能性があるため避けてください。poly-HISタグの挿入などのアプリケーションで最終的な反復アミノ酸配列が必要な場合は、CATCACCATCATCACCACなどの代替コドンを使用して、核酸配列の繰り返しを減らします。
注:プライマーは、任意の優先ベンダーから注文できます。DNA断片を合成する場合は、合成されたDNA断片に相同末端を組み込む必要があります。
- IVAの効率を高めるには、相同DNAフラグメントの融解温度が47〜52°C2であることを確認してください。
2. 相同末端を含むDNA産物の増幅
- プラスミドDNAまたはゲノムDNAテンプレートを、標準的な市販のDNA単離キットまたはアルカリ溶解6,17を使用して単離します。
- テンプレートDNA(ステップ2.1から)と相同領域を含むプライマー(ステップ1.2)を、市販の高忠実度ポリメラーゼを使用したPCR反応で使用します。ダウンストリーム反応でのDNAテンプレートのキャリーオーバーを制限するには、各反応で少量のテンプレートDNA(20 pg-1 ng)を使用します。酵素特異的PCR反応混合物の成分および推奨されるPCRサイクリング条件については、メーカーの推奨を参照してください。PCR反応を進めます。
- 図 2 に示す DNA フラグメントを増幅するように PCR 反応液 (50 μL) をセットアップし、100 μM の dNTP、0.2 μM の各 DNA プライマー、テンプレート DNA(50 pg の pSU19 と 1 ng の pSU18mCherry)、Mg2+ を含む 1x PCR 反応バッファー混合物 (2 mM 最終濃度)、および 1 U のハイフィデリティ DNA ポリメラーゼを含めます。
- タッチダウンPCRサイクリングプログラムを使用して、pSU19とmCherryを増幅します(図2)。最初の変性ステップを95°Cで5分間設定し、その後、95°Cで15秒間、1サイクルあたり55°C-0.5°Cで15秒間、72°Cで1分間のサイクルを10サイクル行い、その後、95°Cで15秒、50°Cで15秒、72°Cで1分間のサイクルを20回繰り返します。72°Cで10分間の最終延長ステップでPCRサイクルを完了します。
注:プラスミド骨格を増幅する場合は、分子量に基づいてDNAテンプレートの量を調整します:PCR反応50μLあたり~20 pg/1 kbpのプラスミドDNA(pSU19のDNA50 pg(図))を使用。さらに、増幅中に導入される突然変異の数を最小限に抑えるためにPCRサイクルを調整します:プラスミド骨格の増幅には、わずか20〜25回のPCRサイクルで十分です。
- PCR反応が完了したら、PCR反応のアリコート(2μL)を除去し、ローディング色素と結合し(1x濃度まで)、アガロースゲル電気泳動21によりDNA断片を分離する。紫外線(UV)またはLEDトランスイルミネーターを使用して、アガロースゲル内で移動したDNA断片を可視化します。PCR産物が一意性があるかどうかを判断し、DNAラダーと製品を比較して、予想される分子量であるかどうかを確認します(図2)。正しい分子量のPCR産物が可視化されない場合は、PCR反応の最適化に関するメーカーの推奨事項を参照してください。
注:PCR産物が一意ではないが、予想される分子量に対応する豊富なDNA断片がある場合、追加のPCR反応を回避できます。 - アガロースゲルに複数のDNA断片が存在する場合は、アガロースゲルから予想される分子量に対応するDNA断片を取り出し、アガロースゲル抽出キットを使用してDNA断片を回収します。
- PCR反応からメチル化テンプレートDNAを除去するには、1 μLの DpnIを反応チューブに直接添加します。反応液を37°Cで15分間から一晩インキュベートします。
注:このオプションのステップは、ダウンストリーム反応で繰り越されるテンプレートDNAの量を制限するために強く推奨されます。ただし、PCR反応で非常に低濃度のテンプレートDNAを使用する場合は、このステップをスキップできます。 - 核酸精製キットを使用して、酵素アッセイから生じる残留酵素、塩、プライマーダイマー、およびその他の望ましくない低分子DNA製品を除去します。
注:DNA断片が合成される場合、ステップ2.1-2.6はスキップできます。 - 精製されたDNA断片のDNA濃度を分光光度法またはゲル推定により定量します。
3. IVA(図3)
- 各IVA反応に必要なDNAの量を計算します(25〜50 ngのプラスミドDNAを推奨)。分子量の多いプラスミドには、より多くのプラスミドDNAを使用します(例:2.3 kbpのプラスミドの場合、25 ngのプラスミドを使用しますが、プラスミドが9 kbpの場合は、各反応で50 ngを使用します)。反応に必要なインサートDNAの量を計算します。1:3または1:5のモル比のプラスミド:インサートDNAを使用してください。
注:オンラインバイオ計算機または以下に示す式を使用して、二本鎖DNAフラグメントのモル量を計算し、各DNAフラグメントのモル比を決定できます。
二本鎖DNA断片のモル量を計算するために、次の式を使用します:pmol =(ngの重量)× 1,000 /(塩基対×650ダルトン)。さらに、モル比は、インサートの質量(g)=目的のインサート/プラスミドのモル比×プラスミドの質量(g)×インサートとプラスミドの長さの比率6によって計算できます。 - 計算した容量のプラスミドを組み合わせ、あらかじめ冷却した1.5 mLの微量遠心チューブにDNAを挿入し、氷上に保管します。
- プラスミドを移し、ステップ3.2のDNA混合物を解凍した再薬品大腸菌(例:大腸菌DH5a;22)。
注:使用されるコンピテントセルの量は、プラスミドとインサートDNA混合物の量によって異なります。この混合物は、コンピテントセルの体積の10%を超えてはなりません。 - ヒートショック変形23で進行します。
- 化学的に有能な 大腸菌 とDNAの混合物を氷上で30分間インキュベートします。
- 42°Cのウォーターバスに1分間移し、次にチューブを氷に2分間移します。
- LBをチューブに無菌的に移し替えて、容量を1mLにします。
- 1 mLの容量をガラス製培養チューブに移し、37 °C、220 rpm、または細菌株および/またはプラスミドが回復するための推奨温度(通常25 °C〜37 °C)に設定された振とうインキュベーターに入れ、プラスミドにコードされた抗生物質選択マーカーを産生します。
- 細胞が30分から1時間回復した後、形質転換反応のアリコート(1,000μLの100μL)を固体選択培地に沈着させ、滅菌細胞スプレッダーを使用してアリコートを寒天培地全体に分散させます。残りの形質転換細胞を遠心分離(13,000 g× g で1分間)で回収し、上清を廃棄し、細胞ペレットを100 μLの滅菌培地に再懸濁し、上記のように固体選択培地に沈着します。液体を固体培地に30分間吸収させ、培養プレートを反転させます。そして、細菌株および/またはプラスミドの推奨温度でインキュベーターに一晩置きます。
4. 正しいプラスミドアセンブリのスクリーニング
- インキュベーション後(ステップ3.5)、インキュベーターから培養プレートを取り出し、各培養プレートにいくつかの分離された細菌コロニーがある場合は、直接カウントしてコロニーを列挙します。スクリーニングのためにいくつかのコロニー(1-10)を選択し、それらに目的のアセンブルプラスミドが含まれているかどうかを判断します。
- 適切な抗生物質を添加した無菌増殖培地を無菌培養チューブ(ガラスまたはプラスチック)に移します。滅菌転写針またはループで1コロニーに触れて培地に接種し、転写針で細胞を培養チューブ培地に移します。培養チューブを振とうインキュベーターに入れ、細菌株および/またはプラスミドの推奨温度(一般的な範囲は25°C〜37°C)で16〜18時間インキュベートします。
- 翌日、アルカリ溶解6 により細菌培養物からプラスミドDNAを単離するか、製造元の指示に従って市販のプラスミド単離キットを使用します。
- プラスミドが正しく組み立てられているか確認します。
- 単離されたプラスミドのDNA濃度を分光光度法またはゲル推定により定量します。
- 診断用制限消化反応(最終容量10 μL)には、プラスミドDNAのアリコート(50-150 ng)を使用してください。アセンブルされたプラスミドDNAを切断すると予想される場所に基づいて、制限酵素を選択します。
注:(1)プラスミドを1つの部位で切断すると予想される制限酵素でプラスミドDNAを処理すること、および(2)挿入されたDNA断片の全部または一部を切除すると予想される2つの制限酵素でプラスミドDNAを処理することの2つの別々の反応を推奨します。制限酵素は市販されており、製造元の指示に従って使用する必要があります。 - 制限酵素反応が完了したら、ローディング色素と結合し(1x濃度まで)、混合物全体をアガロースゲルにロードし、アガロースゲル電気泳動21によりDNA断片を分離する。UVまたはLEDトランスイルミネーターを使用して、アガロースゲル内で移動したDNA断片を可視化します。制限されたDNA断片を分子量ラダーと比較して、それらが予想される分子量であるかどうかを判断します(図4)。予想される酵素制限パターンを持つプラスミド( ポジティブクローンと呼ばれる)に注意してください。予想される制限パターンを持たないプラスミドを含むチューブ( ネガティブクローンと呼ばれる)は廃棄します。
- ポジティブクローンが期待通りのヌクレオチド配列を持つように、シーケンシングのためにポジティブプラスミドクローンのアリコートを送ってください。
注:全プラスミドシーケンシングが推奨されます(例:ナノポアシーケンシング)。
結果
この原稿では、IVAの使用例として、提供されたIVAワークフローに従って、プラスミドpSU19のマルチクローニング部位にmCherryオープンリーディングフレームを再クローニングし、pSU19mCherryと同一のプラスミドを生成しました(図3)17。IVAに適したプライマーは、プロトコールのステップ1.1および1.2に基づいて設計されました。次に、プラスミド単離キットを使用してpSU19およびpSU18mCherryを単離し、これはそれぞれプラスミド骨格を増幅し、DNAを挿入するためのPCR反応のテンプレートとして機能しました(プロトコルステップ2.1)。pSU19プラスミド骨格をPCR増幅し(プライマーペア:pSU19_F:TAGGGTACCGAGCTCGAATTCおよびpSU19_R CCCGGGGATCCTCTAGAGTC)、mCherryオープンリーディングフレームをpSU18mCherryからPCR増幅しました(プライマーペアmCherry_F:ctctagaggatccccggggATGGTATCAAAAGGAGGAGGAAGおよびmCherry_R:gaattcgagctcggtaccctaTTATCATTACTTGTACAGTTC;mCherryプライマーの小文字のヌクレオチド配列は、pSU19バックボーンを増幅するために使用されるプライマーと相同なヌクレオチドを示しています;プロトコルステップ2.2)。特異的PCR増幅は、アガロースゲル電気泳動(プロトコルステップ2.3; 図2)PCR産物は、残りの1日目のワークフロー(プロトコルステップ2.5-3.5; 図3)。
翌日、形質転換プレート上のコロニーを列挙しました(プロトコルステップ4.1; 図3、2日目)。この実験では、6つのコロニーが見えました。コロニーの数は、回収された培養物のより大きなアリコートをプレーティングするか、またはコンピテントセルに変換されるDNAの量を増やすことによって増やすことができる。形質転換プレート上にコロニーが存在しない場合は、まずコンピテントセルの形質転換効率が高いことを確認することが重要です。私たちは、形質転換pUC19 DNAの107 から 109 コロニー形成単位/μgの範囲の形質転換効率を持つコンピテントセルを使用することに成功しています。形質転換効率が問題にならない場合は、高分子量プラスミドでIVAを行う場合や、多くのDNA断片を組み立てる場合に、コンピテントセルに形質転換するDNAの量を増やすか、コンピテントセルの量を増やす必要があるかもしれません。
正しいプラスミドアセンブリをスクリーニングするために2つのコロニーを選択しました(プロトコルステップ4.1-4.3.3)。翌日は、3日目のワークフローに従いました(図3)。プラスミドの単離後、各プラスミドを XbaIまたは XbaIと EcoRIで処理し、正しくアセンブルされたプラスミドを1回または2回切断することが期待されます(図4)。 図4に見られるように、両方の酵素反応により、プラスミドクローン1の2.3 kbpに対応する単一のDNA産物が得られ、これがpSU19のみであることを示しています。プラスミドクローン2の酵素処理では、 XbaIで処理した後に1~3kbpのDNA産物が得られ、 XbaIおよび EcoRIでの処理後に2つのDNA産物(2.3 kbpおよび770 bp)が得られました。次に、プラスミドクローン2の正しいアセンブリを、全プラスミドシーケンシング分析(プロトコルステップ4.4; 図3、3日目)。この例では、正しいプラスミドの組み立て効率は50%でした。正しいプラスミドアセンブリのために2つ以上のクローンをスクリーニングすることはめったになく、通常、プラスミドアセンブリ効率は50〜100%です。私たちの経験では、ネガティブクローンには通常、(i)再環状化したテンプレートDNAから引き継がれたプラスミド骨格、または(ii)ハイブリッドプラスミドに再結合したテンプレートDNAからなるハイブリッドDNA製品が含まれています。 DPNのI処理(プロトコールステップ2.5)は、ネガティブクローンの数を制限します。それでもIVAの効率が改善されない場合は、PCRプライマーの設計を検証し、プラスミド骨格とPCR産物を再増幅することをお勧めします。

図1:IVAプライマーの設計戦略。 IVAプライマー設計の最初のステップ(ステップ1)は、ソフトウェアを使用して、目的のアセンブルされたプラス ミドin silicoの配列ファイルを作成することです。第2ステップ(ステップ2)では、プラスミドと挿入DNA配列の接合部付近でプラスミド骨格を増幅するプライマーを設計します。インサート配列のプライマーは、目的のDNA産物に特異的に結合および増幅するのに十分なヌクレオチドを持ち、相同組換えのために15〜50 bpの隣接するプラスミド骨格を含有する必要があります。プラスミド設計における相同性の領域は色分けされています。略語:IVA = in vivo assembly。 この図の拡大版を表示するには、ここをクリックしてください。
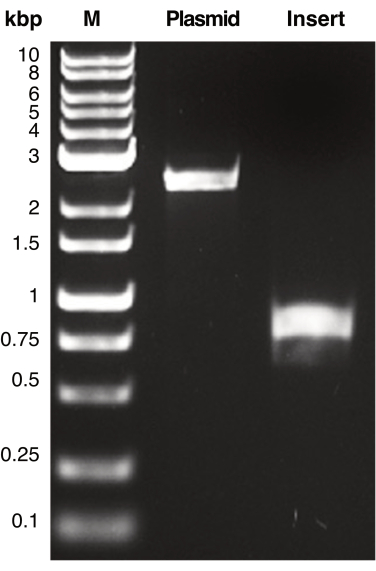
図2:プラスミドとインサートDNAのPCR増幅。 プラスミドに対応するPCR増幅されたDNA断片と、相同性領域を含む挿入DNAの代表的な画像を示す。DNA断片をゲル電気泳動によって分離し、UV透過照明によって可視化しました。DNA産物を分子量マーカー(M)と比較し、各PCR産物の見かけの分子量を決定しました。プラスミド(pSU19)とインサートDNA(mCherry)の予想分子量は、それぞれ2.3 kbpと770 bpです。 この図の拡大版を表示するには、ここをクリックしてください。

図3:in vivoアセンブリワークフローの概略図。 DNAをPCR増幅して、5'および3'相同領域を作成します。 DPNのI処理は、テンプレートDNAを切断するために使用されます。ヌクレオチド精製キットは、PCR増幅されたDNAから塩と酵素を除去するために使用されます。UV分光光度法を使用してDNA濃度を決定し、3:1のインサートとプラスミドのモル比を予め冷却したマイクロフュージチューブに組み合わせます。結合したDNAを、解凍した 大腸菌 DH5αコンピテントセルのアリコートが入った氷冷マイクロフュージチューブに移します。DNAをヒートショックにより 大腸菌 DH5αに形質転換し、固体選択培地に広げて、37°Cで18時間インキュベートします。インキュベーション後、プラスミドクローンを含む単一コロニーを滅菌液体培地と抗生物質を含む培養チューブに移し、次いで37°Cで18時間攪拌しながらインキュベートします。翌日、プラスミドDNAを細胞から単離し、制限酵素処理を使用して陽性クローンをスクリーニングします。その後、ポジティブクローンをシーケンシング解析に送り、プラスミドのDNA配列を確認します。 この図の拡大版を表示するには、ここをクリックしてください。

図4:プラスミドクローン陽性をスクリーニングするためのゲル電気泳動。 2つの異なるプラスミドクローンを単離し、プラスミドを線状化するために1回、または挿入されたDNAを放出するために2回、陽性クローンを切断することが期待される制限酵素で処理しました。制限酵素処理後、サンプルをゲル電気泳動で分離し、UV透過照明で可視化しました。分子量マーカーと比較することにより、分子量を推定しました。直鎖状化されたポジティブクローン(プラスミド+インサート)の予想分子量は3.07 kbp、ポジティブクローンを2回切断した後のDNA断片の予想分子量は2.3 kbp(プラスミドバックボーン)と770 bp(インサート)でした。プラスミドクローン1は陰性(空のプラスミド)であり、クローン2はポジティブクローンです。略語:SC =単一の切断。DC =二重劈開;M =分子量マーカー。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
IVAクローニングを成功させるための最も重要なステップは、DNAフラグメントとプライマーの設計です。相同フラグメントが約47〜52°Cの融解温度で少なくとも15 bpの長さになるように設計されている場合、IVA効率は大幅に改善されます。 IVAフラグメントデザインの最適化を調査する詳細な研究が発表されました16。クローニング効率を高めるためのもう一つの重要なステップは、PCR増幅ステップで使用するテンプレートDNAをできるだけ少なくすることです。テンプレートDNAのキャリーオーバーをさらに低減するために、ポストサイクルPCR反応を DpnIで処理し、メチル化テンプレートDNAを切断します。これにより、陽性クローンの単離に必要なスクリーニングの量を大幅に減らすことができます。私たちの経験では、 DpnI治療では、形質転換ごとに1〜2個のコロニーをスクリーニングするだけで済みます。 DpnI処理を使用しない場合、形質転換ごとに5〜10個のコロニーをスクリーニングする必要があります。
我々や他の人々は、IVAクローニングが多くのコロニー形成ユニット16,17を生じると報告しているが、形質転換プレート上にコロニーが存在しない可能性がある。私たちの経験では、コンピテントセルに形質転換されるDNAの量を増やす(特に大きなプラスミドバックボーンを扱う場合)および/または使用するコンピテントセルの量を増やすことで、形質転換効率を向上させることができます。DNAやコンピテントセルの量を調整してもコロニーが見えない場合は、コンピテントセルストックの形質転換効率を実験的に確認し、直鎖状のDNA断片をシーケンシングして、目的の相同領域が含まれていることを確認します。これらのトラブルシューティング手順に従うと、通常、IVA の効率が向上します。
IVA クローニング方法には、いくつかの制限があります。これらは、特定のDNA断片を増幅するためのPCRの使用に関連しています。例えば、プラスミド骨格はサイズに幅があり、中には10 kbpに近い、またはそれを超えるものもあります(例:pMMB67HE24、pFUSE25)。所望のプラスミド骨格が大きすぎるか、または高忠実度のプルーフリーディングポリメラーゼで増幅するのが難しい場合でも、制限酵素( SmaIまたは EcoRVなど)でプラスミドを直鎖化し、IVA反応にPCR産物の代わりに直鎖化プラスミドを使用する選択肢がまだあります16。私たちの経験では、制限酵素処理によってほぼ100%のプラスミド線形化が達成されない場合、IVAの効率は大幅に低下します。
IVAのもう一つの潜在的な制限は、各ユニークなDNA断片をクローニングするために、相同なDNAオーバーハングを含む新しいプライマーを購入することに伴うコストです。このコストは、ラボが限られた数のプラスミドバックボーンを使用し、ユニバーサルプライマーを使用して、将来のIVAクローニングのための増幅プラスミドバックボーンの大規模なバッチを生成できれば、部分的に軽減できます。さらに、ラボで使用されるすべてのプラスミドに同一の複数のクローニング部位がある場合、すべてのプラスミドに同じユニバーサルプライマーを使用することができます。IVA法の別の制限は、我々の知る限り、反復的または不安定なDNA26のクローニングに典型的に使用される線状プラスミドの集合について試験されていないことである。
IVAは、時間と費用対効果の高いクローニング法であり、in vitro DNAフラグメントアセンブリに依存する他の分子クローニング法と比較して、いくつかの利点があります。プラスミドの集合体は、市販の酵素や緩衝液に依存してDNAを組み立てないため、一般的に費用対効果が高いです4,5,27。これにより、典型的な酵素反応ステップが回避されるため、時間とコストが削減され、実験室での操作を最大72時間節約できます2,15。さらに、IVAに必要な機器や試薬は、通常、分子クローニングを日常的に実施する研究室にすでに存在するか、利用可能です。
私たちのグループは最近、IVAを使用して、同一の複数のクローニング部位、プラスミドコピー数の3つのオプション、および抗生物質選択マーカー17の5つのオプションを持つ一連の24プラスミドを生成しました。我々は、ユニバーサルプライマーを使用して各プラスミド17に遺伝子をクローニングする方法を示しました。この研究では、クローニング効率は71〜100%であったが、これは陽性クローンを同定するためにスクリーニングされたコロニーが非常に少なかった(5つの陽性クローンに対して7つのコロニーがスクリーニングされた)17。別の研究では、IVAクローニングの効率が最大97%±1.9%に達すると報告されており、この高い率は、著者らが正しいプラスミドアセンブリのために多数のクローンを体系的にテストすることで説明できる16。IVAは、部位特異的突然変異誘発やタグ配列27を含むプライマーの組み込みなどのアプリケーションにも使用できる。IVAは、研究室での多くの日常的な分子クローニングアプリケーションに適応できる、汎用性が高く効率的なクローニング技術です。
開示事項
著者は、宣言する利益相反を持っていません。
謝辞
HGBは、カナダ自然科学工学研究評議会(NSERC)のカナダ大学院奨学金-修士プログラムとサスカチュワン大学(サスカチュワン大学)の学部長博士賞によって資金提供されています。この研究は、NSERC Discovery Grant (RGPIN-2021-03066)、サスカチュワン大学からのスタートアップ資金 (JLT へ)、Canada Foundation for Innovation John R. Evans Leaders Fund (Grant number 42269 to JLT) によって支援されました。著者らは、UV分光法によるDNA定量の写真を提供してくださったEric Toombs氏に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 1 kb Plus DNA ladder | FroggaBio | DM015-R500 | DNA molecular weight marker |
| AccuReady M Single Channel 0.5-10 µL | Bulldog Bio | BPP020 | Pipettes for transferring liquid |
| AccuReady M Single Channel Pipettor Kit | Bulldog Bio | BPK100 | Pipettes for transferring liquid |
| Agar | BioShop Canada | AGR003.1 | Solidifying agent for growth medium |
| Agarose | Biobasic | D0012 | Make agarose gels for DNA electrophoresis |
| Biometra TOne | Analytikjena | 846-2-070-301 | Thermocycler |
| Caps for glass culture tubes | Fisher Scientific | 14-957-91 | Reusable caps for glass culture tubes |
| DNA primers | Integrated DNA Technologies | N/A | Bind and amplify template DNA in PCR reaction |
| DpnI | New England Biolabs | R0176S | Cleave methylated template DNA following PCR amplification |
| EcoRI | New England Biolabs | R3101L | Restriction enzyme, comes with appropriate buffer |
| Eppendorf microtubes 1.5 mL | Sarstedt | 72.690.300 | Microtube, 1.5 mL, conical base, PP, attached flat cap, molded graduations and frosted writing space |
| Ethidium bromide | Fisher Scientific | AAL0748203 | DNA visualization/intercalating agent, toxic |
| EZ-10 Spin Column Plasmid DNA Miniprep Kit | Biobasic | BS614 | Isolate plasmid DNA from overnight bacterial cultures |
| GelDoc Go-Gel system | Bio-Rad | 12009077 | Imaging DNA gels |
| Glass culture tubes | Fisher Scientific | 14-925E | Glass culture tubes |
| myGel Mini Electrophoresis System | Sigma-Aldrich | Z742288 | System used for gel electrophoresis |
| NanoDrop One | Thermo Fisher | ND-ONE-W | Determine DNA concentration |
| PCR Clean Up for DNA Sequencing | Biobasic | BT5100 | Purify PCR products |
| Petri dish | Sarstedt | 82.1473.011 | Petri dish 92 x 16 mm, PS, transparent, with ventilation cams |
| Pipette tip, 1000 µL | Sarstedt | 70.305 | Pipette tips for 100-1000 µL |
| Pipette tip, 2.5 µL | Sarstedt | 70.3010.265 | Pipette tips for up to 2.5 µL |
| Pipette tip, 20 µL | Sarstedt | 70.3020.200 | Pipette tips for up to 20 µL |
| Pipette tip, 200 µL | Sarstedt | 70.3030.020 | Pipette tips for 1-200 µL |
| Salt (NaCl) | Fisher Scientific | S271-10 | Component of bacterial growth medium (10 g/L) |
| Single 0.2 mL PCR tubes with flat cap | FroggaBio | TF-1000 | PCR tubes |
| Tryptone (Bacteriological) | BioShop Canada | TRP402.5 | Component of bacterial growth medium (10 g/L) |
| VeriFi DNA polymerase | PCR Biosystems | PB10.42-01 | High fidelity polymerase, the PCR buffer containing Mg2+ and dNTPs is provided with purchase |
| Xba I | New England Biolabs | R0145S | Restriction enzyme, comes with appropriate buffer |
| Yeast extract | BioShop Canada | YEX401.205 | Component of bacterial growth medium (5 g/L) |
参考文献
- Bertero, A., Brown, S., Vallier, L. Methods of cloning. Basic Science Methods for Clinical Researchers. Chapter 2, 19-39 (2017).
- Garcia-Nafria, J., Watson, J. F., Greger, I. H. IVA cloning: A single-tube universal cloning system exploiting bacterial in vivo assembly. Sci Rep. 6, 27459 (2016).
- Bubeck, P., Winkler, M., Bautsch, W. Rapid cloning by homologous recombination in vivo. Nucleic Acids Res. 21 (15), 3601-3602 (1993).
- Avilan, L. Assembling multiple fragments: the Gibson assembly. Methods Mol Biol. 2633, 45-53 (2023).
- Stukenberg, D., et al. The Marburg Collection: A Golden Gate DNA assembly framework for synthetic biology applications in Vibrio natriegens. ACS Synth Biol. 10 (8), 1904-1919 (2021).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Engler, C., Marillonnet, S. Golden Gate cloning. Methods Mol Biol. 1116, 119-131 (2014).
- Ma, H., Kunes, S., Schatz, P. J., Botstein, D. Plasmid construction by homologous recombination in yeast. Gene. 58 (2-3), 201-216 (1987).
- Cao, P., Wang, L., Zhou, G., Wang, Y., Chen, Y. Rapid assembly of multiple DNA fragments through direct transformation of PCR products into E. coli and Lactobacillus. Plasmid. 76, 40-46 (2014).
- Conley, E. C., Saunders, V. A., Saunders, J. R. Deletion and rearrangement of plasmid DNA during transformation of Escherichia coli with linear plasmid molecules. Nucleic Acids Res. 14 (22), 8905-8917 (1986).
- Conley, E. C., Saunders, V. A., Jackson, V., Saunders, J. R. Mechanism of intramolecular recyclization and deletion formation following transformation of Escherichia coli with linearized plasmid DNA. Nucleic Acids Res. 14 (22), 8919-8932 (1986).
- Sung, W. L., Zahab, D. M. Site-specific recombination directed by single-stranded crossover linkers: specific deletion of the amino-terminal region of the beta-galactosidase gene in pUC plasmids. DNA. 6 (4), 373-379 (1987).
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci USA. 83 (19), 7177-7181 (1986).
- Jones, D. H., Howard, B. H. A rapid method for recombination and site-specific mutagenesis by placing homologous ends on DNA using polymerase chain reaction. Biotechniques. 10 (1), 62-66 (1991).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5alpha-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), e0137466 (2015).
- Chen, F., et al. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 21 (1), 24 (2021).
- Braun, H. G., Kanwal, N., Rivera Lopez, L. F., Thomassin, J. L. Generation of a plasmid series for rapid sub-cloning and use in various Enterobacteriaceae. J Biosci Bioeng. 138 (6), 478-487 (2024).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Res. 35 (Web Server issue), W43-W46 (2007).
- Wallace, R. B., et al. Hybridization of synthetic oligodeoxyribonucleotides to phi chi 174 DNA: the effect of single base pair mismatch. Nucleic Acids Res. 6 (11), 3543-3557 (1979).
- Bzymek, M., Lovett, S. T. Instability of repetitive DNA sequences: The role of replication in multiple mechanisms. Proc Natl Acad Sci USA. 98 (15), 8319-8325 (2001).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Sambrook, J., Russell, D. W. The inoue method for preparation and transformation of competent E. coli: "ultra-competent" cells. CSH Protoc. 2006 (1), (2006).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Fürste, J. P., et al. Molecular cloning of the plasmid RP4 primase region in a multi-host-range tacP expression vector. Gene. 48 (1), 119-131 (1986).
- Bäumler, A. J., et al. Identification of a new iron regulated locus of Salmonella typhi. Gene. 183 (1-2), 207-213 (1996).
- Godiska, R., et al. Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli. Nucleic Acids Res. 38 (6), e88 (2010).
- Watson, J. F., Garcia-Nafria, J. In vivo DNA assembly using common laboratory bacteria: A re-emerging tool to simplify molecular cloning. J Biol Chem. 294 (42), 15271-15281 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved