Method Article
Utilizzo dell'assemblaggio in vivo per la costruzione di plasmidi ad alta efficienza
In questo articolo
Riepilogo
L'assemblaggio in vivo è un metodo di clonazione indipendente dalla legatura che si basa su enzimi intrinseci di riparazione del DNA nei batteri per assemblare frammenti di DNA mediante ricombinazione omologa. Questo protocollo è efficiente in termini di tempo e costi, poiché sono necessari pochi reagenti e l'efficienza di clonazione può raggiungere il 99%.
Abstract
L'assemblaggio in vivo (IVA) è un metodo di clonazione molecolare che utilizza enzimi intrinseci presenti nei batteri che promuovono la ricombinazione intermolecolare di frammenti di DNA per assemblare plasmidi. Questo metodo funziona trasformando frammenti di DNA con regioni di 15-50 bp di omologia in ceppi di Escherichia coli di laboratorio comunemente usati e i batteri utilizzano la via di riparazione indipendente da RecA per assemblare i frammenti di DNA in un plasmide. Questo metodo è più rapido ed economico di molti metodi di clonazione molecolare che si basano sull'assemblaggio in vitro di plasmidi prima della trasformazione in ceppi di E. coli . Questo perché i metodi in vitro richiedono l'acquisto di enzimi specializzati e l'esecuzione di reazioni enzimatiche sequenziali che richiedono incubazioni. Tuttavia, a differenza dei metodi in vitro , l'IVA non ha dimostrato sperimentalmente di assemblare plasmidi lineari. Qui condividiamo il protocollo IVA utilizzato dal nostro laboratorio per assemblare rapidamente plasmidi e subclonare frammenti di DNA tra plasmidi con diverse origini di replicazione e marcatori di resistenza agli antibiotici.
Introduzione
Il clonaggio molecolare comprende una serie di tecniche di laboratorio necessarie per produrre plasmidi contenenti DNA ricombinante specifico1. Queste tecniche onnipresenti spesso agiscono come un collo di bottiglia nel flusso di lavoro sperimentale2. Molte tecniche di clonaggio molecolare si basano sull'assemblaggio di frammenti di DNA in vitro utilizzando una serie di reazioni enzimatiche prima della trasformazione in un ceppo ospite (ad esempio, Escherichia coli DH5a) per l'amplificazione 3,4,5,6,7. Poiché i metodi di assemblaggio dei plasmidi in vitro si basano sugli enzimi, l'acquisto o la purificazione degli enzimi può essere costoso e dispendioso in termini di tempo.
L'assemblaggio in vivo (IVA) è un metodo di clonaggio molecolare che si basa sulla ricombinazione intermolecolare di frammenti di DNA in un ospite adatto 8,9,10,11,12. La base di questo metodo è stata l'osservazione che i ceppi di clonaggio recA comunemente usati di E. coli potrebbero mediare la ricombinazione intermolecolare di un primer a singolo filamento con un plasmide scisso da enzimi di restrizione13. L'uso della PCR per generare estremità omologhe di DNA per l'assemblaggio di plasmidi in E. coli è stato descritto negli anni '90 e questo metodo è stato chiamato PCR di ricombinazione 3,14. L'efficienza della PCR a ricombinazione è stata riportata essere di circa il 50%14. Tuttavia, questo metodo non è stato ampiamente adottato, probabilmente a causa dell'alto costo dei primer e del rischio di introdurre mutazioni indesiderate utilizzando polimerasi a bassa fedeltà per amplificare grandi frammenti di DNA. Questi inconvenienti erano significativi negli anni '90 e potevano essere evitati utilizzando metodi di clonazione in vitro come la clonazione mediata da enzimi di restrizione.
Negli ultimi anni, il costo dei primer è diminuito e le nuove polimerasi commerciali hanno aumentato la fedeltà dell'amplificazione PCR. Di conseguenza, la PCR a ricombinazione è stata rivisitata come tecnica di clonaggio molecolare rapida ed economica e rinominata IVA 2,9,15,16. Con una maggiore fattibilità, l'IVA è stata ulteriormente esplorata e il metodo è stato ottimizzato per raggiungere efficienze di clonazione fino al 99%16. Le ottimizzazioni hanno identificato caratteristiche del DNA omologo, come il numero di coppie di basi e la temperatura di fusione, che massimizzano l'efficienza di ricombinazione in vivo 2,16. Ulteriori analisi hanno dimostrato che fino a 6 frammenti di DNA che vanno da 150 bp a 7 kbp potrebbero essere assemblati in modo efficiente con IVA15. Inoltre, il nostro gruppo ha recentemente utilizzato l'IVA per assemblare una serie di plasmidi con diverse cassette di resistenza agli antibiotici e origini di replicazione17. In questo studio, il clonaggio dell'IVA è risultato altamente efficiente (71-100%) con un piccolo numero di cloni testati per ciascun plasmide (n = 2)17. Qui descriviamo il protocollo IVA utilizzato dal nostro laboratorio.
Protocollo
1. Progettare primer o frammenti di DNA con estremità omologhe
- Utilizza un software di elaborazione testi o un software specializzato per l'analisi del DNA per assemblare artificialmente il plasmide desiderato.
NOTA: Spesso è utile codificare a colori frammenti di DNA provenienti da fonti diverse ed evidenziare le regioni omologhe che verranno utilizzate nelle fasi di assemblaggio a valle. Inoltre, è anche utile codificare a colori il DNA omologo per garantire l'assemblaggio direzionale. - Progettare primer che si legano a ciascun frammento di DNA e contengono 15-50 bp della sequenza di DNA omologa (Figura 1)2,15.
- Per una maggiore efficienza IVA, assicurarsi che i frammenti di DNA omologhi abbiano temperature di fusione di 47-52 °C2.
NOTA: La temperatura di fusione può essere determinata utilizzando il software online18 o la formula: Temperatura di fusione = 64,9 + 41 * (nG + nC - 16,4)/(nA + nT + nG + nC), dove n = numero di G, C, A o T nella sequenza 6,19. - Evita i primer con sequenze altamente ripetitive come CATCATCATCATCAT che codificano per un tag HIS in quanto possono introdurre instabilità20. Se è necessaria una sequenza finale ripetitiva di amminoacidi per applicazioni come l'inserimento di un tag poly-HIS, utilizzare codoni alternativi come CATCACCATCATCACCAC per ridurre la ripetizione della sequenza di acidi nucleici.
NOTA: I primer possono essere ordinati da qualsiasi fornitore preferito. Se i frammenti di DNA devono essere sintetizzati, le estremità omologhe dovrebbero essere incorporate nel frammento di DNA sintetizzato.
- Per una maggiore efficienza IVA, assicurarsi che i frammenti di DNA omologhi abbiano temperature di fusione di 47-52 °C2.
2. Amplificazione di prodotti a base di DNA contenenti estremità omologhe
- Isolare il DNA plasmidico o i modelli di DNA genomico utilizzando i kit standard di isolamento del DNA disponibili in commercio o la lisi alcalina 6,17.
- Utilizzare DNA stampo (dal passaggio 2.1) e primer contenenti regioni omologhe (passaggio 1.2) in una reazione PCR con una polimerasi ad alta fedeltà disponibile in commercio. Per limitare il carryover del DNA stampo nelle reazioni a valle, utilizzare una bassa quantità di DNA stampo (20 pg-1 ng) in ciascuna reazione. Fare riferimento alle raccomandazioni del produttore per i componenti della miscela di reazioni PCR specifiche per l'enzima e le condizioni consigliate per il ciclo PCR. Procedere con la reazione PCR.
- Impostare le reazioni PCR (50 μL) per amplificare i frammenti di DNA mostrati nella Figura 2 in modo che contengano 100 μM di dNTP, 0,2 μM di ciascun primer di DNA, DNA stampo (50 pg di pSU19 e 1 ng di pSU18mCherry), 1x miscela di tampone di reazione PCR contenente Mg2+ (concentrazione finale di 2 mM) e 1 U di DNA polimerasi ad alta fedeltà.
- Utilizzare un programma di ciclismo PCR touchdown per amplificare pSU19 e mCherry (Figura 2). Impostare la fase iniziale di denaturazione a 95 °C per 5 minuti, seguita da 10 cicli di 95 °C per 15 secondi, 55 °C-0,5 °C per ciclo per 15 secondi e 72 °C per 1 minuto, quindi 20 cicli aggiuntivi di 95 °C per 15 secondi, 50 °C per 15 secondi e 72 °C per 1 minuto. Completare il ciclo di PCR con una fase finale di estensione a 72 °C per 10 minuti.
NOTA: Se una spina dorsale plasmidica deve essere amplificata, regolare la quantità di DNA stampo in base al peso molecolare: con ~20 pg/1 kbp di DNA plasmidico per 50 μL di reazione PCR (50 pg di DNA per pSU19 (mostrato)). Inoltre, regolare i cicli di PCR per ridurre al minimo il numero di mutazioni introdotte durante l'amplificazione: per l'amplificazione delle dorsali plasmidiche sono sufficienti solo 20-25 cicli di PCR.
- Una volta completata la reazione di PCR, rimuovere un'aliquota (2 μL) della reazione di PCR, combinarla con il colorante di caricamento (fino a 1x la concentrazione) e separare i frammenti di DNA mediante elettroforesi su gel di agarosio21. Utilizzare un transilluminatore a raggi ultravioletti (UV) o LED per visualizzare i frammenti di DNA che sono migrati nel gel di agarosio. Determinare se i prodotti PCR sono unici e confrontare i prodotti con la scala del DNA per verificare se hanno il peso molecolare previsto (Figura 2). Se non vengono visualizzati prodotti per PCR con il peso molecolare corretto, fare riferimento alle raccomandazioni del produttore per ottimizzare la reazione PCR.
NOTA: Quando i prodotti PCR non sono unici ma c'è un abbondante frammento di DNA corrispondente al peso molecolare previsto, è possibile evitare un'ulteriore reazione PCR. - Se nel gel di agarosio sono presenti più frammenti di DNA, asportare il frammento di DNA corrispondente al peso molecolare previsto dal gel di agarosio e utilizzare un kit di estrazione del gel di agarosio per recuperare il frammento di DNA.
- Per rimuovere il DNA stampo metilato dalle reazioni di PCR, aggiungere 1 μL di DpnI direttamente alla provetta di reazione. Incubare le reazioni a 37°C per 15 minuti o tutta la notte.
NOTA: Questo passaggio facoltativo è altamente raccomandato per limitare la quantità di DNA stampo che viene trasportata nelle reazioni a valle. Tuttavia, questo passaggio può essere saltato se nella reazione PCR vengono utilizzate concentrazioni molto basse di DNA stampo. - Utilizzare un kit di purificazione degli acidi nucleici per rimuovere gli enzimi residui, i sali, i dimeri di primer e altri prodotti indesiderati a basso peso molecolare del DNA derivanti da saggi enzimatici.
NOTA: I passaggi 2.1-2.6 possono essere saltati se vengono sintetizzati frammenti di DNA. - Quantificare la concentrazione di DNA dei frammenti di DNA purificati mediante spettrofotometria o stima su gel.
3. IVA (Figura 3)
- Calcolare la quantità di DNA necessaria per ogni reazione IVA (si consigliano 25-50 ng di DNA plasmidico). Utilizzare quantità maggiori di DNA plasmidico per plasmidi con peso molecolare più elevato (ad esempio, per plasmidi di 2,3 kbp, utilizzare 25 ng di plasmide, ma quando i plasmidi sono 9 kbp, utilizzare 50 ng in ogni reazione). Calcolare il volume di DNA dell'inserto necessario per la reazione; utilizzare un rapporto molare 1:3 o 1:5 di plasmide:inserire DNA.
NOTA: I biocalcolatori online o le formule fornite di seguito possono essere utilizzati per calcolare la quantità molare di un frammento di DNA a doppio filamento e determinare il rapporto molare di ciascun frammento di DNA.
Utilizziamo la seguente formula per calcolare la quantità molare di un frammento di DNA a doppio filamento: pmol = (peso in ng) × 1.000 / (coppie di basi × 650 dalton). Inoltre, i rapporti molari possono essere calcolati da: inserto di massa (g) = rapporto molare inserto/plasmide desiderato × massa del plasmide (g) × rapporto tra l'inserto e le lunghezze del plasmide6. - Combinare il volume calcolato di plasmide e inserire il DNA in una provetta da microcentrifuga preraffreddata da 1,5 ml e conservare in ghiaccio.
- Trasferire il plasmide e inserire la miscela di DNA dal passaggio 3.2 in un'aliquota (25-100 μL) di E. coli chimicamente competente (ad es. E. coli DH5a;22).
NOTA: Il volume delle cellule competenti utilizzate dipende dal volume del plasmide e della miscela di DNA da inserire; Questa miscela non deve superare il 10% del volume delle cellule competenti. - Procedere con una trasformazione per shock termico23.
- Incubare la miscela di E. coli chimicamente competente e DNA su ghiaccio per 30 minuti.
- Trasferire a bagnomaria a 42 °C per 1 minuto, quindi trasferire il tubo nel ghiaccio per 2 minuti.
- Trasferire asetticamente LB nella provetta per portare il volume a 1 mL.
- Trasferire il volume di 1 mL in una provetta di vetro e metterlo in un incubatore con agitazione impostato a 37 °C, 220 giri/min o alla temperatura consigliata per il recupero del ceppo batterico e/o del plasmide (intervallo tipico da 25 °C a 37 °C) e consentire la produzione del marcatore di selezione antibiotica codificato dal plasmide.
- Dopo che le cellule si sono riprese per 30 minuti a 1 ora, depositare un'aliquota (100 μL di 1.000 μL) della reazione di trasformazione su un terreno di selezione solido e utilizzare uno spandiconcime di cellule sterile per disperdere l'aliquota sulla piastra di coltura dell'agar. Raccogliere il resto delle cellule trasformate mediante centrifugazione (13.000 × g per 1 minuto), scartare il surnatante, risospendere il pellet cellulare in 100 μL di terreno sterile e depositare su un terreno di selezione solido come sopra. Lasciare assorbire il liquido in un terreno solido per 30 minuti, quindi capovolgere le piastre di coltura. e metterli in un'incubatrice per una notte alla temperatura consigliata per il ceppo batterico e/o plasmide.
4. Screening per il corretto assemblaggio dei plasmidi
- Dopo l'incubazione (passaggio 3.5), rimuovere le piastre di coltura dall'incubatrice ed enumerare le colonie contando direttamente se ci sono diverse colonie batteriche isolate su ciascuna piastra di coltura. Selezionare diverse colonie (1-10) per lo screening per determinare se contengono il plasmide assemblato desiderato.
- Trasferire terreni di coltura sterili integrati con antibiotici appropriati in provette di coltura sterili (vetro o plastica). Inoculare il terreno di coltura toccando 1 colonia con un ago o un'ansa di trasferimento sterile e trasferire le cellule nel terreno della provetta di coltura con l'ago di trasferimento. Collocare le provette di coltura in un incubatore vibrante e incubare per 16-18 ore alla temperatura consigliata per il ceppo batterico e/o plasmide (intervallo tipico da 25 °C a 37 °C).
- Il giorno successivo, isolare il DNA plasmidico dalle colture batteriche mediante lisi alcalina6 o utilizzando un kit di isolamento plasmidico disponibile in commercio secondo le istruzioni del produttore.
- Determina se i plasmidi sono assemblati correttamente.
- Quantificare la concentrazione di DNA dei plasmidi isolati mediante spettrofotometria o stima su gel.
- Utilizzare un'aliquota (50-150 ng) di DNA plasmidico per una reazione di digestione di restrizione diagnostica (10 μL di volume finale). Selezionare gli enzimi di restrizione in base a dove si prevede che scinderanno il DNA plasmidico assemblato.
NOTA: Raccomandiamo due reazioni separate: (1) trattare il DNA plasmidico con un enzima di restrizione che dovrebbe scindere il plasmide in un singolo sito e (2) trattare il DNA plasmidico con due enzimi di restrizione che dovrebbero asportare tutto o parte del frammento di DNA inserito. Gli enzimi di restrizione sono disponibili in commercio e devono essere utilizzati seguendo le istruzioni del produttore. - Una volta completata la reazione dell'enzima di restrizione, combinare con il colorante di caricamento (fino a 1x la concentrazione), caricare l'intera miscela su un gel di agarosio e separare i frammenti di DNA mediante elettroforesi su gel di agarosio21. Utilizzare un transilluminatore UV o LED per visualizzare i frammenti di DNA che sono migrati nel gel di agarosio. Confrontare i frammenti di DNA ristretti con la scala del peso molecolare per determinare se sono i pesi molecolari previsti (Figura 4). Prendere nota dei plasmidi con i modelli di restrizione enzimatica previsti (chiamati cloni positivi); Scartare le provette contenenti plasmidi che non hanno il pattern di restrizione previsto (chiamati cloni negativi).
- Per garantire che i cloni positivi abbiano la sequenza nucleotidica prevista, inviare un'aliquota di cloni plasmidici positivi per il sequenziamento.
NOTA: Si raccomanda il sequenziamento dell'intero plasmide (ad esempio, il sequenziamento dei nanopori).
Risultati
In questo manoscritto, come esempio di utilizzo dell'IVA, abbiamo seguito il flusso di lavoro IVA fornito per riclonare il frame di lettura aperto mCherry nel sito di clonazione multipla del plasmide pSU19 per generare un plasmide identico a pSU19mCherry (Figura 3)17. I primer adatti per l'IVA sono stati progettati sulla base delle fasi del protocollo 1.1 e 1.2. Quindi, è stato utilizzato un kit di isolamento plasmidico per isolare pSU19 e pSU18mCherry, che sono serviti come modelli per le reazioni PCR per amplificare la spina dorsale del plasmide e inserire il DNA, rispettivamente (fase 2.1 del protocollo). La spina dorsale del plasmide pSU19 è stata amplificata con PCR (coppie di primer: pSU19_F: TAGGGTACCGAGCTCGAATTC e pSU19_R CCCGGGGATCCTCTAGAGTC) e il frame di lettura aperto mCherry è stato amplificato con PCR da pSU18mCherry (coppie di primer mCherry_F: ctctagaggatccccgggATGGTATCAAAAGGAGGAAG e mCherry_R: gaattcgagctcggtaccctaTTATCATTACTTGTACAGTTC; la sequenza nucleotidica minuscola nei primer mCherry indica nucleotidi omologhi ai primer utilizzati per amplificare la spina dorsale di pSU19; fase del protocollo 2.2). L'amplificazione specifica della PCR è stata verificata mediante elettroforesi su gel di agarosio (fase 2.3 del protocollo; Figura 2) e i prodotti PCR sono stati sottoposti al resto del flusso di lavoro del giorno 1 (fasi del protocollo 2.5-3.5; Figura 3).
Il giorno successivo, sono state enumerate le colonie sulla piastra di trasformazione (fase del protocollo 4.1; Figura 3, giorno 2). In questo esperimento, erano visibili sei colonie; il numero di colonie può essere aumentato impiattando un'aliquota più grande della coltura recuperata o aumentando la quantità di DNA che viene trasformata nelle cellule competenti. Se non sono presenti colonie sulle piastre di trasformazione, è importante verificare prima che le cellule competenti abbiano un'elevata efficienza di trasformazione. Abbiamo avuto successo utilizzando cellule competenti con efficienze di trasformazione che vanno da 107 a 109 unità formanti colonie/μg di DNA pUC19 trasformato. Se l'efficienza della trasformazione non è un problema, potrebbe essere necessario aumentare la quantità di DNA trasformato nelle cellule competenti e/o aumentare il volume delle cellule competenti quando si esegue l'IVA con plasmidi a peso molecolare più elevato o quando si assemblano molti frammenti di DNA.
Abbiamo selezionato due colonie per lo screening del corretto assemblaggio del plasmide (fasi del protocollo 4.1-4.3.3). Il giorno successivo, abbiamo seguito il flusso di lavoro per il giorno 3 (Figura 3). Dopo l'isolamento del plasmide, trattare ciascun plasmide con XbaI o XbaI ed EcoRI, che dovrebbero scindere il plasmide correttamente assemblato una o due volte (Figura 4). Come si può vedere nella Figura 4, entrambe le reazioni enzimatiche hanno portato a un singolo prodotto di DNA corrispondente a 2,3 kbp per il clone plasmidico 1, indicando che questo è solo pSU19. I trattamenti enzimatici del clone plasmidico 2 hanno portato a un singolo prodotto di DNA di ~3 kbp dopo il trattamento con XbaI e due prodotti di DNA (2,3 kbp e 770 bp) dopo il trattamento con XbaI ed EcoRI. Il corretto assemblaggio del clone plasmidico 2 è stato quindi verificato mediante analisi di sequenziamento dell'intero plasmide (fase 4.4 del protocollo; Figura 3, giorno 3). In questo esempio, l'efficienza corretta dell'assemblaggio del plasmide è stata del 50%. Raramente controlliamo più di due cloni per il corretto assemblaggio dei plasmidi e in genere abbiamo un'efficienza di assemblaggio dei plasmidi del 50-100%. Nella nostra esperienza, i cloni negativi contengono tipicamente (i) una spina dorsale plasmidica che viene riportata dal DNA stampo che si è ricircolato o (ii) un prodotto di DNA ibrido costituito da DNA stampo che si è ricombinato in un plasmide ibrido. DpnI (fase del protocollo 2.5) limita il numero di cloni negativi. Se ciò non migliora l'efficienza dell'IVA, si consiglia di verificare il design del primer PCR e di riamplificare le dorsali plasmidiche e i prodotti PCR.

Figura 1: Strategia di progettazione del primer IVA. Il primo passo (Fase 1) nella progettazione del primer IVA consiste nell'utilizzare un software per costruire un file di sequenza del plasmide assemblato desiderato in silico. Il secondo passo (Fase 2) consiste nel progettare primer che amplifichino la spina dorsale del plasmide vicino alla giunzione del plasmide e della sequenza di DNA inserita. I primer per la sequenza di inserimento dovrebbero avere abbastanza nucleotidi per legare e amplificare in modo specifico il prodotto di DNA desiderato e contenere 15-50 bp della spina dorsale plasmidica adiacente per la ricombinazione omologa. Le regioni di omologia nella progettazione dei plasmidi sono codificate a colori. Abbreviazione: IVA = assemblaggio in vivo . Clicca qui per visualizzare una versione più grande di questa figura.
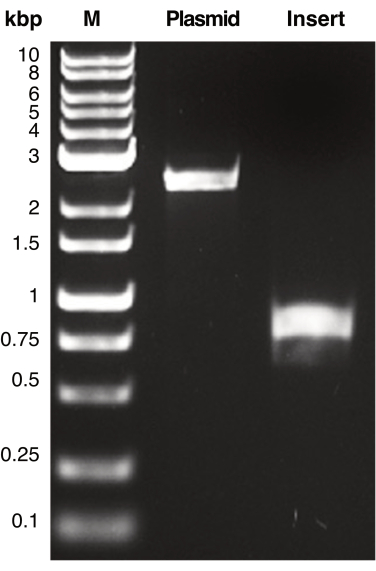
Figura 2: Amplificazione PCR del plasmide e del DNA da inserire. Viene mostrata un'immagine rappresentativa di frammenti di DNA amplificati con PCR corrispondenti a plasmidi e DNA inserito contenenti regioni di omologia. I frammenti di DNA sono stati separati mediante elettroforesi su gel e visualizzati mediante transilluminazione UV. I prodotti del DNA sono stati confrontati con il marcatore del peso molecolare (M) per determinare il peso molecolare apparente di ciascun prodotto della PCR. Il peso molecolare atteso del plasmide (pSU19) e del DNA di inserimento (mCherry) è rispettivamente di 2,3 kbp e 770 bp. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Schema del flusso di lavoro dell'assemblaggio in vivo. Il DNA viene amplificato con PCR per creare regioni omologhe 5' e 3'. DpnIl trattamento I viene utilizzato per scindere il DNA stampo. Un kit di purificazione nucleotidica viene utilizzato per rimuovere sali ed enzimi dal DNA amplificato con PCR. La spettrofotometria UV viene utilizzata per determinare la concentrazione di DNA e un rapporto molare tra inserto e plasmide di 3:1 viene combinato in un tubo microfuge preraffreddato. Il DNA combinato viene trasferito in una provetta per microfuge ghiacciata contenente un'aliquota di cellule scongelate di E. coli DH5α. Il DNA viene trasformato in E. coli DH5α mediante shock termico, steso su terreno di selezione solido e incubato a 37 °C per 18 ore. Dopo l'incubazione, singole colonie contenenti cloni plasmidici vengono trasferite in provette di coltura contenenti terreno liquido sterile e antibiotico, quindi incubate a 37 °C con agitazione per 18 ore. Il giorno successivo, il DNA plasmidico viene isolato dalle cellule e il trattamento con enzimi di restrizione viene utilizzato per lo screening dei cloni positivi. I cloni positivi vengono quindi inviati per l'analisi di sequenziamento per confermare la sequenza di DNA dei plasmidi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Elettroforesi su gel per lo screening di cloni plasmidici positivi. Due diversi cloni plasmidici sono stati isolati e trattati con enzimi di restrizione che dovrebbero scindere i cloni positivi una volta per linearizzare il plasmide o due volte per rilasciare il DNA inserito. Dopo il trattamento con enzima di restrizione, i campioni sono stati separati mediante elettroforesi su gel e visualizzati mediante transilluminazione UV. Il peso molecolare è stato stimato confrontandolo con il marcatore del peso molecolare. Il peso molecolare atteso di un clone positivo linearizzato (plasmide + inserto) era di 3,07 kbp e il peso molecolare atteso dei frammenti di DNA dopo che un clone positivo è stato scisso due volte era di 2,3 kbp (spina dorsale plasmidica) e 770 bp (inserto). Il clone 1 del plasmide è negativo (plasmide vuoto) e il clone 2 è un clone positivo. Abbreviazioni: SC = scollatura singola; DC = doppia scissione; M = marcatore di peso molecolare. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La fase più critica per il successo della clonazione dell'IVA è la progettazione del frammento di DNA e del primer. L'efficienza dell'IVA è notevolmente migliorata quando i frammenti omologhi sono progettati per essere lunghi almeno 15 bp con una temperatura di fusione di circa 47-52 °C. È stato pubblicato uno studio dettagliato che esplora l'ottimizzazione della progettazione di frammenti IVA16. Un altro passo importante per avere un'elevata efficienza di clonazione è utilizzare la minor quantità possibile di DNA stampo nelle fasi di amplificazione della PCR. Per ridurre ulteriormente il carryover del DNA stampo, trattiamo le nostre reazioni di PCR post-ciclo con DpnI per scindere il DNA stampo metilato. Ciò può ridurre significativamente la quantità di screening necessaria per isolare un clone positivo. Nella nostra esperienza, con il trattamento DpnI, abbiamo solo bisogno di esaminare 1-2 colonie per trasformazione; senza il trattamento con DpnI, dobbiamo esaminare 5-10 colonie per trasformazione.
Sebbene noi e altri abbiamo riportato che la clonazione IVA produce molte unità formanti colonie16,17, è possibile che non siano presenti colonie sulle piastre di trasformazione. Nella nostra esperienza, aumentare la quantità di DNA trasformato in cellule competenti (specialmente quando si lavora con una grande spina dorsale plasmidica) e/o aumentare il volume di cellule competenti utilizzate può migliorare l'efficienza della trasformazione. Se non sono visibili colonie dopo aver regolato la quantità di DNA o cellule competenti, confermiamo sperimentalmente l'efficienza di trasformazione degli stock cellulari competenti e sequenziamo i nostri frammenti di DNA lineari per confermare che contengono le regioni omologhe desiderate. Seguendo questi passaggi per la risoluzione dei problemi, in genere si migliora l'efficienza IVA.
Il metodo di clonazione IVA presenta diverse limitazioni. Questi sono legati all'uso della PCR per amplificare specifici frammenti di DNA. Ad esempio, le dorsali plasmidiche possono variare in dimensioni, con alcune che si avvicinano o superano i 10 kbp di lunghezza (ad esempio, pMMB67HE24, pFUSE25). Se la spina dorsale del plasmide desiderata è troppo grande o difficile da amplificare con una polimerasi di correzione di bozze ad alta fedeltà, c'è ancora la possibilità di linearizzare il plasmide con enzimi di restrizione (ad esempio, SmaI o EcoRV) e utilizzare il plasmide linearizzato invece di un prodotto PCR per la reazione IVA16. Nella nostra esperienza, l'efficienza dell'IVA è significativamente ridotta se non si ottiene una linearizzazione del plasmide vicina al 100% mediante trattamento enzimatico di restrizione.
Un'altra potenziale limitazione dell'IVA è il costo associato all'acquisto di nuovi primer contenenti sporgenze di DNA omologhe per clonare ogni singolo frammento di DNA. Questo costo può essere parzialmente alleviato se un laboratorio utilizza un numero limitato di dorsali plasmidiche e può utilizzare primer universali per generare un grande lotto di dorsale plasmidico amplificata per il futuro clonaggio IVA. Inoltre, se tutti i plasmidi utilizzati in laboratorio hanno siti di clonazione multipli identici, gli stessi primer universali possono essere utilizzati per tutti i plasmidi. Un altro limite del metodo IVA è che, per quanto ne sappiamo, non è stato testato per l'assemblaggio di plasmidi lineari che vengono tipicamente utilizzati per la clonazione di DNA26 ripetitivo o instabile.
L'IVA è un metodo di clonazione efficiente in termini di tempo e costi che presenta diversi vantaggi rispetto ad altri metodi di clonazione molecolare che si basano sull'assemblaggio di frammenti di DNA in vitro. È generalmente efficiente in termini di costi, poiché l'assemblaggio dei plasmidi non si basa su enzimi o tamponi acquistati in commercio per assemblare il DNA 4,5,27. Ciò consente di risparmiare tempo e ridurre i costi perché si evitano le tipiche fasi di reazione enzimatica, il che può far risparmiare fino a 72 ore di manipolazioni di laboratorio 2,15. Inoltre, le attrezzature e i reagenti necessari per l'IVA sono in genere già presenti o disponibili nei laboratori che eseguono abitualmente la clonazione molecolare.
Il nostro gruppo ha recentemente utilizzato l'IVA per generare una serie di 24 plasmidi con siti di clonazione multipli identici, tre opzioni per il numero di copie del plasmide e cinque opzioni per i marcatori di selezione degli antibiotici17. Abbiamo dimostrato come i primer universali potrebbero essere utilizzati per clonare i geni in ciascun plasmide17. In questo studio, l'efficienza di clonazione è stata del 71-100%, perché pochissime colonie sono state sottoposte a screening per identificare cloni positivi (sette sono state sottoposte a screening per cinque cloni positivi)17. Un altro studio ha riportato che i tassi di efficienza del clonaggio dell'IVA raggiungono un massimo del 97% ± dell'1,9%, il tasso più alto può essere spiegato dagli autori che testano sistematicamente un gran numero di cloni per il corretto assemblaggio del plasmide16. L'IVA può essere utilizzata anche per applicazioni come la mutagenesi sito-specifica o l'integrazione di primer contenenti sequenze di tag27. L'IVA è una tecnica di clonaggio versatile ed efficiente che può essere adattata a molte applicazioni di clonazione molecolare di routine in laboratorio.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
HGB è finanziato da un programma di master del Consiglio di ricerca sulle scienze naturali e l'ingegneria del Canada (NSERC) e da un premio di dottorato del preside (Università del Saskatchewan). Questo lavoro è stato sostenuto da un NSERC Discovery Grant (RGPIN-2021-03066), fondi di avviamento (a JLT) dell'Università del Saskatchewan e dal Canada Foundation for Innovation John R. Evans Leaders Fund (numero di sovvenzione 42269 a JLT). Gli autori ringraziano Eric Toombs per aver fornito la fotografia della quantificazione del DNA mediante spettroscopia UV.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 kb Plus DNA ladder | FroggaBio | DM015-R500 | DNA molecular weight marker |
| AccuReady M Single Channel 0.5-10 µL | Bulldog Bio | BPP020 | Pipettes for transferring liquid |
| AccuReady M Single Channel Pipettor Kit | Bulldog Bio | BPK100 | Pipettes for transferring liquid |
| Agar | BioShop Canada | AGR003.1 | Solidifying agent for growth medium |
| Agarose | Biobasic | D0012 | Make agarose gels for DNA electrophoresis |
| Biometra TOne | Analytikjena | 846-2-070-301 | Thermocycler |
| Caps for glass culture tubes | Fisher Scientific | 14-957-91 | Reusable caps for glass culture tubes |
| DNA primers | Integrated DNA Technologies | N/A | Bind and amplify template DNA in PCR reaction |
| DpnI | New England Biolabs | R0176S | Cleave methylated template DNA following PCR amplification |
| EcoRI | New England Biolabs | R3101L | Restriction enzyme, comes with appropriate buffer |
| Eppendorf microtubes 1.5 mL | Sarstedt | 72.690.300 | Microtube, 1.5 mL, conical base, PP, attached flat cap, molded graduations and frosted writing space |
| Ethidium bromide | Fisher Scientific | AAL0748203 | DNA visualization/intercalating agent, toxic |
| EZ-10 Spin Column Plasmid DNA Miniprep Kit | Biobasic | BS614 | Isolate plasmid DNA from overnight bacterial cultures |
| GelDoc Go-Gel system | Bio-Rad | 12009077 | Imaging DNA gels |
| Glass culture tubes | Fisher Scientific | 14-925E | Glass culture tubes |
| myGel Mini Electrophoresis System | Sigma-Aldrich | Z742288 | System used for gel electrophoresis |
| NanoDrop One | Thermo Fisher | ND-ONE-W | Determine DNA concentration |
| PCR Clean Up for DNA Sequencing | Biobasic | BT5100 | Purify PCR products |
| Petri dish | Sarstedt | 82.1473.011 | Petri dish 92 x 16 mm, PS, transparent, with ventilation cams |
| Pipette tip, 1000 µL | Sarstedt | 70.305 | Pipette tips for 100-1000 µL |
| Pipette tip, 2.5 µL | Sarstedt | 70.3010.265 | Pipette tips for up to 2.5 µL |
| Pipette tip, 20 µL | Sarstedt | 70.3020.200 | Pipette tips for up to 20 µL |
| Pipette tip, 200 µL | Sarstedt | 70.3030.020 | Pipette tips for 1-200 µL |
| Salt (NaCl) | Fisher Scientific | S271-10 | Component of bacterial growth medium (10 g/L) |
| Single 0.2 mL PCR tubes with flat cap | FroggaBio | TF-1000 | PCR tubes |
| Tryptone (Bacteriological) | BioShop Canada | TRP402.5 | Component of bacterial growth medium (10 g/L) |
| VeriFi DNA polymerase | PCR Biosystems | PB10.42-01 | High fidelity polymerase, the PCR buffer containing Mg2+ and dNTPs is provided with purchase |
| Xba I | New England Biolabs | R0145S | Restriction enzyme, comes with appropriate buffer |
| Yeast extract | BioShop Canada | YEX401.205 | Component of bacterial growth medium (5 g/L) |
Riferimenti
- Bertero, A., Brown, S., Vallier, L. Methods of cloning. Basic Science Methods for Clinical Researchers. Chapter 2, 19-39 (2017).
- Garcia-Nafria, J., Watson, J. F., Greger, I. H. IVA cloning: A single-tube universal cloning system exploiting bacterial in vivo assembly. Sci Rep. 6, 27459 (2016).
- Bubeck, P., Winkler, M., Bautsch, W. Rapid cloning by homologous recombination in vivo. Nucleic Acids Res. 21 (15), 3601-3602 (1993).
- Avilan, L. Assembling multiple fragments: the Gibson assembly. Methods Mol Biol. 2633, 45-53 (2023).
- Stukenberg, D., et al. The Marburg Collection: A Golden Gate DNA assembly framework for synthetic biology applications in Vibrio natriegens. ACS Synth Biol. 10 (8), 1904-1919 (2021).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Engler, C., Marillonnet, S. Golden Gate cloning. Methods Mol Biol. 1116, 119-131 (2014).
- Ma, H., Kunes, S., Schatz, P. J., Botstein, D. Plasmid construction by homologous recombination in yeast. Gene. 58 (2-3), 201-216 (1987).
- Cao, P., Wang, L., Zhou, G., Wang, Y., Chen, Y. Rapid assembly of multiple DNA fragments through direct transformation of PCR products into E. coli and Lactobacillus. Plasmid. 76, 40-46 (2014).
- Conley, E. C., Saunders, V. A., Saunders, J. R. Deletion and rearrangement of plasmid DNA during transformation of Escherichia coli with linear plasmid molecules. Nucleic Acids Res. 14 (22), 8905-8917 (1986).
- Conley, E. C., Saunders, V. A., Jackson, V., Saunders, J. R. Mechanism of intramolecular recyclization and deletion formation following transformation of Escherichia coli with linearized plasmid DNA. Nucleic Acids Res. 14 (22), 8919-8932 (1986).
- Sung, W. L., Zahab, D. M. Site-specific recombination directed by single-stranded crossover linkers: specific deletion of the amino-terminal region of the beta-galactosidase gene in pUC plasmids. DNA. 6 (4), 373-379 (1987).
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci USA. 83 (19), 7177-7181 (1986).
- Jones, D. H., Howard, B. H. A rapid method for recombination and site-specific mutagenesis by placing homologous ends on DNA using polymerase chain reaction. Biotechniques. 10 (1), 62-66 (1991).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5alpha-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), e0137466 (2015).
- Chen, F., et al. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 21 (1), 24 (2021).
- Braun, H. G., Kanwal, N., Rivera Lopez, L. F., Thomassin, J. L. Generation of a plasmid series for rapid sub-cloning and use in various Enterobacteriaceae. J Biosci Bioeng. 138 (6), 478-487 (2024).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Res. 35 (Web Server issue), W43-W46 (2007).
- Wallace, R. B., et al. Hybridization of synthetic oligodeoxyribonucleotides to phi chi 174 DNA: the effect of single base pair mismatch. Nucleic Acids Res. 6 (11), 3543-3557 (1979).
- Bzymek, M., Lovett, S. T. Instability of repetitive DNA sequences: The role of replication in multiple mechanisms. Proc Natl Acad Sci USA. 98 (15), 8319-8325 (2001).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Sambrook, J., Russell, D. W. The inoue method for preparation and transformation of competent E. coli: "ultra-competent" cells. CSH Protoc. 2006 (1), (2006).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Fürste, J. P., et al. Molecular cloning of the plasmid RP4 primase region in a multi-host-range tacP expression vector. Gene. 48 (1), 119-131 (1986).
- Bäumler, A. J., et al. Identification of a new iron regulated locus of Salmonella typhi. Gene. 183 (1-2), 207-213 (1996).
- Godiska, R., et al. Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli. Nucleic Acids Res. 38 (6), e88 (2010).
- Watson, J. F., Garcia-Nafria, J. In vivo DNA assembly using common laboratory bacteria: A re-emerging tool to simplify molecular cloning. J Biol Chem. 294 (42), 15271-15281 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon