Method Article
Utilisation de l’assemblage in vivo pour la construction de plasmides à haut rendement
Dans cet article
Résumé
L’assemblage in vivo est une méthode de clonage indépendante de la ligature qui s’appuie sur des enzymes de réparation de l’ADN intrinsèques aux bactéries pour assembler des fragments d’ADN par recombinaison homologue. Ce protocole est à la fois rapide et économique, car peu de réactifs sont nécessaires et l’efficacité du clonage peut atteindre 99 %.
Résumé
L’assemblage in vivo (IVA) est une méthode de clonage moléculaire qui utilise des enzymes intrinsèques présentes dans les bactéries qui favorisent la recombinaison intermoléculaire de fragments d’ADN pour assembler des plasmides. Cette méthode fonctionne en transformant des fragments d’ADN avec des régions d’homologie de 15 à 50 pb en souches d’Escherichia coli de laboratoire couramment utilisées et les bactéries utilisent la voie de réparation indépendante de RecA pour assembler les fragments d’ADN en un plasmide. Cette méthode est plus rapide et plus rentable que de nombreuses méthodes de clonage moléculaire qui reposent sur l’assemblage in vitro de plasmides avant leur transformation en souches d’E. coli . En effet, les méthodes in vitro nécessitent l’achat d’enzymes spécialisées et la réalisation de réactions enzymatiques séquentielles nécessitant des incubations. Cependant, contrairement aux méthodes in vitro , il n’a pas été démontré expérimentalement que l’IVA assemble des plasmides linéaires. Ici, nous partageons le protocole IVA utilisé par notre laboratoire pour assembler rapidement des plasmides et sous-cloner des fragments d’ADN entre des plasmides ayant différentes origines de réplication et marqueurs de résistance aux antibiotiques.
Introduction
Le clonage moléculaire englobe une série de techniques de laboratoire nécessaires pour produire des plasmides contenant de l’ADN recombinant spécifique1. Ces techniques omniprésentes agissent souvent comme un goulot d’étranglement dans le flux de travail expérimental2. De nombreuses techniques de clonage moléculaire reposent sur l’assemblage de fragments d’ADN in vitro à l’aide d’une série de réactions enzymatiques avant la transformation en souche hôte (par exemple, Escherichia coli DH5a) pour l’amplification 3,4,5,6,7. Comme les méthodes d’assemblage de plasmides in vitro reposent sur des enzymes, l’achat ou la purification d’enzymes peut être à la fois coûteux et chronophage.
L’assemblage in vivo (IVA) est une méthode de clonage moléculaire qui repose sur la recombinaison intermoléculaire de fragments d’ADN dans un hôte approprié 8,9,10,11,12. La base de cette méthode était l’observation que les souches de clonage recA couramment utilisées d’E. coli pouvaient médier la recombinaison intermoléculaire d’une amorce simple brin avec un plasmide clivé par des enzymes de restriction13. L’utilisation de la PCR pour générer des extrémités d’ADN homologues pour l’assemblage de plasmides chez E. coli a été décrite dans les années 1990 et cette méthode a été nommée PCR de recombinaison 3,14. L’efficacité de la PCR de recombinaison était d’environ 50 %14. Cependant, cette méthode n’a pas été largement adoptée, probablement en raison du coût élevé des amorces et du risque d’introduire des mutations indésirables à l’aide de polymérases basse fidélité pour amplifier de gros fragments d’ADN. Ces inconvénients étaient importants dans les années 1990 et pouvaient être évités en utilisant des méthodes de clonage in vitro telles que le clonage médié par des enzymes de restriction.
Ces dernières années, le coût des amorces a diminué et de nouvelles polymérases commerciales ont augmenté la fidélité de l’amplification par PCR. En conséquence, la PCR de recombinaison a été revisitée en tant que technique de clonage moléculaire rapide et rentable et rebaptisée IVA 2,9,15,16. Avec une faisabilité accrue, l’IVA a été explorée plus avant et la méthode a été optimisée pour atteindre des efficacités de clonage allant jusqu’à 99 %16. Les optimisations ont permis d’identifier des caractéristiques de l’ADN homologue, telles que le nombre de paires de bases et la température de fusion, qui maximisent l’efficacité de la recombinaison in vivo 2,16. Une analyse plus poussée a montré que jusqu’à 6 fragments d’ADN allant de 150 pb à 7 kbp pouvaient être assemblés efficacement par IVA15. De plus, notre groupe a récemment utilisé l’IVA pour assembler une série de plasmides avec différentes cassettes de résistance aux antibiotiques et les origines de la réplication17. Dans cette étude, le clonage IVA s’est avéré très efficace (71-100 %) avec un petit nombre de clones testés pour chaque plasmide (n = 2)17. Nous décrivons ici le protocole IVA utilisé par notre laboratoire.
Protocole
1. Concevez des amorces ou des fragments d’ADN avec des extrémités homologues
- Utilisez un logiciel de traitement de texte ou un logiciel d’analyse d’ADN spécialisé pour assembler artificiellement le plasmide souhaité.
REMARQUE : Il est souvent utile d’attribuer un code couleur aux fragments d’ADN provenant de différentes sources et de mettre en évidence les régions homologues qui seront utilisées dans les étapes d’assemblage en aval. De plus, il est également utile de coder par couleur l’ADN homologue pour assurer l’assemblage directionnel. - Concevez des amorces qui se lient à chaque fragment d’ADN et contiennent 15 à 50 pb de la séquence d’ADN homologue (Figure 1)2,15.
- Pour une efficacité accrue de l’IVA, assurez-vous que les fragments d’ADN homologues ont des températures de fusion de 47 à 52 °C2.
REMARQUE : La température de fusion peut être déterminée à l’aide du logiciel en ligne18 ou de la formule : Température de fusion = 64,9 + 41 * (nG + nC - 16,4)/(nA + nT + nG + nC), où n = nombre de G, C, A ou T dans la séquence 6,19. - Évitez les amorces avec des séquences très répétitives comme CATCATCATCATCATCAT qui codent pour une balise HIS, car elles peuvent introduire une instabilité20. Si une séquence finale répétitive d’acides aminés est nécessaire pour des applications telles que l’insertion d’une étiquette poly-HIS, utilisez des codons alternatifs comme CATCACCATCATCACCAC pour diminuer la répétition de la séquence d’acides nucléiques.
REMARQUE : Les apprêts peuvent être commandés auprès de n’importe quel fournisseur préféré. Si des fragments d’ADN doivent être synthétisés, des extrémités homologues doivent être incorporées dans le fragment d’ADN synthétisé.
- Pour une efficacité accrue de l’IVA, assurez-vous que les fragments d’ADN homologues ont des températures de fusion de 47 à 52 °C2.
2. Amplification de produits d’ADN contenant des extrémités homologues
- Isolez l’ADN plasmidique ou les matrices d’ADN génomique à l’aide de kits d’isolement d’ADN standard disponibles dans le commerce ou de lyse alcaline 6,17.
- Utilisez de l’ADN matrice (à l’étape 2.1) et des amorces contenant des régions homologues (étape 1.2) dans une réaction PCR avec une polymérase haute fidélité disponible dans le commerce. Pour limiter le transfert de la matrice d’ADN dans les réactions en aval, utilisez une faible quantité d’ADN matrice (20 pg-1 ng) dans chaque réaction. Reportez-vous aux recommandations du fabricant pour les composants du mélange de réaction PCR spécifiques aux enzymes et les conditions de cycle de PCR recommandées. Procédez à la réaction PCR.
- Configurez les réactions de PCR (50 μL) pour amplifier les fragments d’ADN montrant que la figure 2 contient 100 μM de dNTP, 0,2 μM de chaque amorce d’ADN, de l’ADN matrice (50 pg de pSU19 et 1 ng de pSU18mCherry), 1 mélange de tampon de réaction PCR contenant du Mg2+ (concentration finale de 2 mM) et 1 U d’ADN polymérase haute fidélité.
- Utilisez un programme de cycle PCR pour amplifier pSU19 et mCherry (Figure 2). Réglez l’étape initiale de dénaturation à 95 °C pendant 5 min, suivie de 10 cycles de 95 °C pendant 15 s, de 55 °C à 0,5 °C par cycle pendant 15 s et de 72 °C pendant 1 min, puis de 20 cycles supplémentaires de 95 °C pendant 15 s, 50 °C pendant 15 s et 72 °C pendant 1 min. Terminez le cycle de PCR par une dernière étape d’extension à 72 °C pendant 10 min.
REMARQUE : Si un squelette plasmidique doit être amplifié, ajustez la quantité de matrice d’ADN en fonction du poids moléculaire : avec ~20 pg/1 kbp d’ADN plasmidique par 50 μL de réaction PCR (50 pg d’ADN pour pSU19 (illustré)). De plus, ajustez les cycles de PCR pour minimiser le nombre de mutations introduites lors de l’amplification : pour l’amplification des squelettes plasmidiques, aussi peu que 20 à 25 cycles de PCR suffisent.
- Une fois la réaction de PCR terminée, prélever une aliquote (2 μL) de la réaction de PCR, combiner avec un colorant de charge (jusqu’à une concentration de 1x) et séparer les fragments d’ADN par électrophorèse sur gel d’agarose21. Utilisez un transilluminateur ultraviolet (UV) ou LED pour visualiser les fragments d’ADN qui ont migré dans le gel d’agarose. Déterminez si les produits PCR sont uniques et comparez-les avec l’échelle d’ADN pour vérifier s’ils ont la masse moléculaire attendue (figure 2). Si aucun produit PCR de poids moléculaire correct n’est visualisé, reportez-vous aux recommandations du fabricant pour optimiser la réaction PCR.
REMARQUE : Lorsque les produits de PCR ne sont pas uniques mais qu’il y a un fragment d’ADN abondant correspondant au poids moléculaire attendu, une réaction PCR supplémentaire peut être évitée. - S’il y a plusieurs fragments d’ADN présents dans le gel d’agarose, excisez le fragment d’ADN correspondant au poids moléculaire attendu du gel d’agarose et utilisez un kit d’extraction de gel d’agarose pour récupérer le fragment d’ADN.
- Pour éliminer l’ADN matrice méthylé des réactions de PCR, ajoutez 1 μL de DpnI directement dans le tube de réaction. Incuber les réactions à 37°C pendant 15 min à toute la nuit.
REMARQUE : Cette étape facultative est fortement recommandée pour limiter la quantité d’ADN matrice qui est transportée dans les réactions en aval. Cependant, cette étape peut être sautée si de très faibles concentrations d’ADN matrice sont utilisées dans la réaction PCR. - Utilisez un kit de purification d’acide nucléique pour éliminer les enzymes résiduelles, les sels, les dimères d’amorce et autres produits indésirables de faible poids moléculaire résultant d’essais enzymatiques.
REMARQUE : Les étapes 2.1 à 2.6 peuvent être sautées si des fragments d’ADN sont synthétisés. - Quantifiez la concentration d’ADN des fragments d’ADN purifiés par spectrophotométrie ou estimation sur gel.
3. IVA (Figure 3)
- Calculez la quantité d’ADN nécessaire pour chaque réaction IVA (25 à 50 ng d’ADN plasmidique sont recommandés). Utilisez des quantités plus élevées d’ADN plasmidique pour les plasmides de poids moléculaire plus élevé (par exemple, pour les plasmides de 2,3 kbp, utilisez 25 ng de plasmide, mais lorsque les plasmides sont de 9 kbp, utilisez 50 ng dans chaque réaction). Calculer le volume d’ADN d’insertion nécessaire à la réaction ; utilisez un rapport molaire de 1:3 ou 1:5 de l’ADN plasmide :insert.
REMARQUE : Les calculateurs biographiques en ligne ou les formules fournies ci-dessous peuvent être utilisés pour calculer la quantité molaire d’un fragment d’ADN double brin et déterminer le rapport molaire de chaque fragment d’ADN.
Nous utilisons la formule suivante pour calculer la quantité molaire d’un fragment d’ADN double brin : pmol = (poids en ng) × 1 000 / (paires de bases × 650 daltons). De plus, les rapports molaires peuvent être calculés comme suit : masse insert (g) = rapport insert/plasmide molaire souhaité × masse du plasmide (g) × rapport entre l’insert et les longueurs du plasmide6. - Combinez le volume calculé de plasmide et insérez l’ADN dans un tube de microcentrifugation prérefroidi de 1,5 ml et conservez-le sur de la glace.
- Transvaser le plasmide et insérer le mélange d’ADN de l’étape 3.2 dans une aliquote (25-100 μL) d’E. coli chimiquement compétent (p. ex. E. coli DH5a ;22).
REMARQUE : Le volume de cellules compétentes utilisé dépend du volume du mélange de plasmide et d’ADN d’insert ; Ce mélange ne doit pas dépasser 10 % du volume des cellules compétentes. - Procéder à une transformation par choc thermique23.
- Incuber le mélange d’E. coli chimiquement compétent et d’ADN sur de la glace pendant 30 min.
- Transvasez dans un bain-marie à 42 °C pendant 1 min, puis transférez le tube sur de la glace pendant 2 min.
- Transférer aseptiquement LB dans le tube pour porter le volume à 1 mL.
- Transvaser le volume de 1 mL dans un tube de culture en verre et le placer dans un incubateur à agitation réglé à 37 °C, 220 tr/min ou à la température recommandée pour que la souche bactérienne et/ou le plasmide se rétablisse (plage typique de 25 °C à 37 °C) et permettre la production du marqueur de sélection d’antibiotiques codé par le plasmide.
- Une fois que les cellules ont récupéré pendant 30 min à 1 h, déposer une aliquote (100 μL de 1 000 μL) de la réaction de transformation sur un milieu de sélection solide et utiliser un épandeur de cellules stériles pour disperser l’aliquote sur la plaque de culture gélosée. Recueillir le reste des cellules transformées par centrifugation (13 000 × g pendant 1 min), jeter le surnageant, remettre en suspension la pastille cellulaire dans 100 μL de milieu stérile et déposer sur un milieu de sélection solide comme ci-dessus. Laisser le liquide absorber dans un milieu solide pendant 30 min, retourner les plaques de culture. et placez-les dans un incubateur pendant la nuit à la température recommandée pour la souche bactérienne et/ou le plasmide.
4. Criblage pour un assemblage correct du plasmide
- Après l’incubation (étape 3.5), retirer les plaques de culture de l’incubateur et dénombrer les colonies par comptage direct s’il y a plusieurs colonies bactériennes isolées sur chaque plaque de culture. Sélectionner plusieurs colonies (1 à 10) pour le criblage afin de déterminer si elles contiennent le plasmide assemblé souhaité.
- Transférez les milieux de croissance stériles complétés par des antibiotiques appropriés dans des tubes de culture stériles (verre ou plastique). Inoculer le milieu de culture en touchant 1 colonie avec une aiguille ou une boucle de transfert stérile et transférer les cellules dans le milieu du tube de culture avec l’aiguille de transfert. Placez les tubes de culture dans un incubateur agitateur et incubez pendant 16 à 18 h à la température recommandée pour la souche bactérienne et/ou le plasmide (plage typique de 25 °C à 37 °C).
- Le lendemain, isolez l’ADN plasmidique des cultures bactériennes par lyse alcaline6 ou à l’aide d’un kit d’isolement de plasmide disponible dans le commerce selon les instructions du fabricant.
- Déterminez si les plasmides sont correctement assemblés.
- Quantifier la concentration en ADN des plasmides isolés par spectrophotométrie ou estimation sur gel.
- Utiliser une aliquote (50-150 ng) d’ADN plasmidique pour une réaction de digestion de restriction diagnostique (volume final de 10 μL). Sélectionnez les enzymes de restriction en fonction de l’endroit où elles sont censées cliver l’ADN plasmidique assemblé.
REMARQUE : Nous recommandons deux réactions distinctes : (1) traiter l’ADN plasmidique avec une enzyme de restriction censée cliver le plasmide à un seul endroit et (2) traiter l’ADN plasmidique avec deux enzymes de restriction censées exciser tout ou partie du fragment d’ADN inséré. Les enzymes de restriction sont disponibles dans le commerce et doivent être utilisées conformément aux instructions du fabricant. - Une fois la réaction enzymatique de restriction terminée, combinez avec un colorant de charge (à une concentration de 1x), chargez le mélange entier sur un gel d’agarose et séparez les fragments d’ADN par électrophorèsesur gel d’agarose 21. Utilisez un transilluminateur UV ou LED pour visualiser les fragments d’ADN qui ont migré dans le gel d’agarose. Comparez les fragments d’ADN restreints avec l’échelle de masse moléculaire pour déterminer s’ils correspondent à la ou aux masses moléculaires attendues (figure 4). Prenez note des plasmides avec les modèles de restriction enzymatique attendus (appelés clones positifs) ; Jetez les tubes contenant des plasmides qui n’ont pas le motif de restriction attendu (appelés clones négatifs).
- Pour vous assurer que les clones positifs ont la séquence nucléotidique attendue, envoyez une aliquote de clones de plasmides positifs pour le séquençage.
REMARQUE : Le séquençage du plasmide entier est recommandé (par exemple, le séquençage à nanopores).
Résultats
Dans ce manuscrit, à titre d’exemple d’utilisation de l’IVA, nous avons suivi le flux de travail IVA fourni pour recloner le cadre de lecture ouvert mCherry dans le site de clonage multiple du plasmide pSU19 afin de générer un plasmide identique à pSU19mCherry (Figure 3)17. Les amorces adaptées à l’IVA ont été conçues sur la base des étapes de protocole 1.1 et 1.2. Ensuite, un kit d’isolement plasmidique a été utilisé pour isoler pSU19 et pSU18mCherry, qui ont servi de modèles pour les réactions PCR visant à amplifier le squelette plasmidique et à insérer l’ADN, respectivement (étape 2.1 du protocole). Le squelette plasmidique pSU19 a été amplifié par PCR (paires d’amorces : pSU19_F : TAGGGTACCGAGCTCGAATTC et pSU19_R CCCGGGGATCCTCTAGAGTC) et le cadre de lecture ouvert mCherry a été amplifié par PCR à partir de pSU18mCherry (paires d’amorces mCherry_F : ctctagaggatccccggATGGTATCAAAAGGAGAGGAG et mCherry_R : gaattcgagctcggtaccctaTTATCATTACGTACAGTTC ; la séquence nucléotidique minuscule dans les amorces mCherry indique des nucléotides homologues aux amorces utilisées pour amplifier le squelette pSU19 ; étape de protocole 2.2). L’amplification spécifique de la PCR a été vérifiée par électrophorèse sur gel d’agarose (étape 2.3 du protocole ; Graphique 2) et les produits PCR ont été soumis au reste du flux de travail du jour 1 (étapes de protocole 2.5 à 3.5 ; Figure 3).
Le lendemain, les colonies sur la plaque de transformation ont été dénombrées (étape 4.1 du protocole ; Figure 3, jour 2). Dans cette expérience, six colonies étaient visibles ; le nombre de colonies peut être augmenté en plaquant une plus grande aliquote de la culture récupérée ou en augmentant la quantité d’ADN qui est transformée en cellules compétentes. Si aucune colonie n’est présente sur les plaques de transformation, il est important de vérifier au préalable que les cellules compétentes ont une efficacité de transformation élevée. Nous avons réussi à utiliser des cellules compétentes avec des efficacités de transformation allant de 107 à 109 unités formant des colonies/μg d’ADN pUC19 transformé. Si l’efficacité de la transformation n’est pas une préoccupation, il peut être nécessaire d’augmenter la quantité d’ADN transformée dans les cellules compétentes et/ou d’augmenter le volume de cellules compétentes lors de la réalisation d’IVA avec des plasmides de poids moléculaire plus élevé ou lors de l’assemblage de nombreux fragments d’ADN.
Nous avons sélectionné deux colonies pour vérifier l’assemblage correct du plasmide (étapes 4.1-4.3.3 du protocole). Le lendemain, nous avons suivi le flux de travail du jour 3 (Figure 3). Après l’isolement du plasmide, traitez chaque plasmide avec XbaI ou XbaI et EcoRI, qui devraient cliver le plasmide correctement assemblé une ou deux fois (Figure 4). Comme on peut le voir sur la figure 4, les deux réactions enzymatiques ont abouti à un seul produit d’ADN correspondant à 2,3 kbp pour le clone plasmidique 1, indiquant qu’il s’agit de pSU19 seul. Les traitements enzymatiques du clone plasmidique 2 ont permis d’obtenir un seul produit d’ADN de ~3 kbp après traitement avec XbaI et deux produits d’ADN (2,3 kbp et 770 pb) après traitement avec XbaI et EcoRI. L’assemblage correct du clone plasmidique 2 a ensuite été vérifié par une analyse de séquençage du plasmide entier (étape 4.4 du protocole ; Figure 3, jour 3). Dans cet exemple, l’efficacité correcte de l’assemblage du plasmide était de 50 %. Nous criblons rarement plus de deux clones pour un assemblage correct des plasmides et avons généralement une efficacité d’assemblage des plasmides de 50 à 100 %. D’après notre expérience, les clones négatifs contiennent généralement soit (i) un squelette plasmidique qui est repris de l’ADN matrice qui s’est recircularisé, soit (ii) un produit d’ADN hybride composé d’ADN matrice qui s’est recombiné en un plasmide hybride. DpnLe traitement I (étape 2.5 du protocole) limite le nombre de clones négatifs. Si cela n’améliore pas l’efficacité de l’IVA, il est recommandé de vérifier la conception de l’amorce PCR et de réamplifier les squelettes plasmidiques et les produits PCR.

Figure 1 : Stratégie de conception de l’amorce IVA. La première étape (étape 1) de la conception d’une amorce IVA consiste à utiliser un logiciel pour construire un fichier de séquence du plasmide assemblé souhaité in silico. La deuxième étape (étape 2) consiste à concevoir des amorces qui amplifient le squelette plasmidique près de la jonction du plasmide et de la séquence d’ADN insérée. Les amorces de la séquence d’insertion doivent avoir suffisamment de nucléotides pour se lier et amplifier spécifiquement le produit d’ADN souhaité et contenir 15 à 50 pb du squelette plasmidique adjacent pour la recombinaison homologue. Les régions d’homologie dans la conception des plasmides sont codées par couleur. Abréviation : IVA = assemblage in vivo. Veuillez cliquer ici pour voir une version agrandie de cette figure.
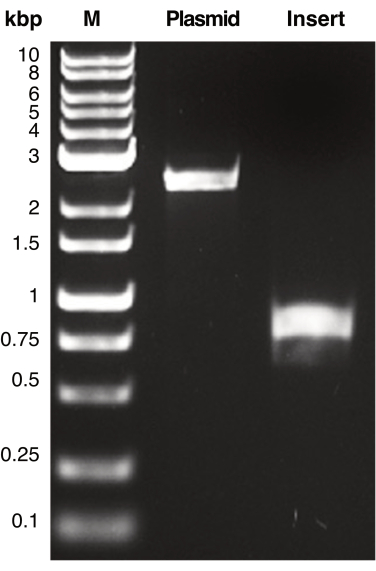
Figure 2 : Amplification par PCR du plasmide et de l’ADN de l’insert. L’image est présentée comme représentative de fragments d’ADN amplifiés par PCR correspondant à des régions d’homologie contenant de l’ADN plasmidique et inséré. Des fragments d’ADN ont été séparés par électrophorèse sur gel et visualisés par transillumination UV. Les produits d’ADN ont été comparés au marqueur de poids moléculaire (M) pour déterminer le poids moléculaire apparent de chaque produit de PCR. Le poids moléculaire attendu du plasmide (pSU19) et de l’ADN inséré (mCherry) est de 2,3 kbp et 770 pb, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Schéma du flux de travail d’assemblage in vivo. L’ADN est amplifié par PCR pour créer des régions homologues 5' et 3'. DpnLe traitement I est utilisé pour cliver l’ADN modèle. Un kit de purification des nucléotides est utilisé pour éliminer les sels et les enzymes de l’ADN amplifié par PCR. La spectrophotométrie UV est utilisée pour déterminer la concentration de l’ADN, et un rapport insert/molaire plasmidique de 3:1 est combiné dans un tube de microfuge prérefroidi. L’ADN combiné est transféré dans un tube microfuge glacé contenant une aliquote de cellules compétentes DH5α d’E. coli décongelées. L’ADN est transformé en E. coli DH5α par choc thermique, étalé sur un milieu de sélection solide et incubé à 37 °C pendant 18 h. Après l’incubation, des colonies individuelles contenant des clones plasmidiques sont transférées dans des tubes de culture contenant un milieu liquide stérile et un antibiotique, puis incubées à 37 °C avec agitation pendant 18 h. Le lendemain, l’ADN plasmidique est isolé des cellules et un traitement enzymatique de restriction est utilisé pour dépister les clones positifs. Les clones positifs sont ensuite envoyés pour une analyse de séquençage afin de confirmer la séquence d’ADN des plasmides. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Électrophorèse sur gel pour dépister les clones de plasmides positifs. Deux clones de plasmide différents ont été isolés et traités avec des enzymes de restriction censées cliver les clones positifs une fois pour linéariser le plasmide ou deux fois pour libérer l’ADN inséré. Après un traitement enzymatique de restriction, les échantillons ont été séparés par électrophorèse sur gel et visualisés par transillumination UV. La masse moléculaire a été estimée en comparant avec le marqueur de la masse moléculaire. La masse moléculaire attendue d’un clone positif linéarisé (plasmide + insert) était de 3,07 kbp, et la masse moléculaire attendue des fragments d’ADN après qu’un clone positif ait été clivé deux fois était de 2,3 kbp (squelette plasmidique) et de 770 pb (insert). Le clone 1 est négatif (plasmide vide) et le clone 2 est positif. Abréviations : SC = décolleté simple ; DC = double clivage ; M = marqueur de poids moléculaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
L’étape la plus critique pour un clonage IVA réussi est la conception du fragment d’ADN et de l’amorce. L’efficacité de l’IVA est considérablement améliorée lorsque les fragments homologues sont conçus pour avoir une longueur d’au moins 15 pb avec une température de fusion d’environ 47-52 °C. Une étude détaillée explorant l’optimisation de la conception des fragments d’IVA a été publiée16. Une autre étape importante pour avoir une efficacité de clonage élevée est d’utiliser le moins d’ADN matrice possible dans les étapes d’amplification PCR. Pour réduire davantage le transfert d’ADN matrice, nous traitons nos réactions PCR post-cycle avec DpnI pour cliver l’ADN matrice méthylé. Cela peut réduire considérablement le nombre de dépistages nécessaires pour isoler un clone positif. D’après notre expérience, avec le traitement DpnI, nous n’avons besoin de cribler que 1 à 2 colonies par transformation ; sans traitement DpnI, nous devons cribler 5 à 10 colonies par transformation.
Bien que nous et d’autres ayons signalé que le clonage IVA produisait de nombreuses unités formant des colonies16,17, il est possible qu’aucune colonie ne soit présente sur les plaques de transformation. D’après notre expérience, l’augmentation de la quantité d’ADN transformée en cellules compétentes (en particulier lorsque l’on travaille avec un grand squelette plasmidique) et/ou l’augmentation du volume de cellules compétentes utilisées peuvent améliorer l’efficacité de la transformation. Si aucune colonie n’est visible après avoir ajusté la quantité d’ADN ou de cellules compétentes, nous confirmons expérimentalement l’efficacité de transformation des stocks de cellules compétentes et séquençons nos fragments d’ADN linéaires pour confirmer qu’ils contiennent les régions homologues souhaitées. Le fait de suivre ces étapes de dépannage améliore généralement l’efficacité de l’IVA.
La méthode de clonage IVA présente plusieurs limites. Ceux-ci sont liés à l’utilisation de la PCR pour amplifier des fragments d’ADN spécifiques. Par exemple, les squelettes plasmidiques peuvent varier en taille, certains approchant ou dépassant 10 kbp de longueur (par exemple, pMMB67HE24, pFUSE25). Si le squelette plasmidique souhaité est trop grand ou difficile à amplifier avec une polymérase de relecture haute fidélité, il est toujours possible de linéariser le plasmide avec des enzymes de restriction (par exemple, SmaI ou EcoRV) et d’utiliser le plasmide linéarisé au lieu d’un produit PCR pour la réaction IVA16. D’après notre expérience, l’efficacité de l’IVA est considérablement diminuée si une linéarisation plasmidique proche de 100 % n’est pas obtenue par un traitement enzymatique de restriction.
Une autre limitation potentielle de l’IVA est le coût associé à l’achat de nouvelles amorces contenant des surplombs d’ADN homologues pour cloner chaque fragment d’ADN unique. Ce coût peut être partiellement atténué si un laboratoire utilise un nombre limité de squelettes plasmidiques et peut utiliser des amorces universelles pour générer un grand lot de squelette plasmidique amplifié pour un futur clonage IVA. De plus, si tous les plasmides utilisés en laboratoire ont plusieurs sites de clonage identiques, les mêmes amorces universelles peuvent être utilisées pour tous les plasmides. Une autre limitation de la méthode IVA est que, à notre connaissance, elle n’a pas été testée pour l’assemblage de plasmides linéaires qui sont généralement utilisés pour le clonage d’ADN26 répétitif ou instable.
L’IVA est une méthode de clonage rapide et économique qui présente plusieurs avantages par rapport aux autres méthodes de clonage moléculaire qui reposent sur l’assemblage de fragments d’ADN in vitro. Il est généralement rentable, car l’assemblage des plasmides ne repose pas sur des enzymes ou des tampons achetés dans le commerce pour assembler l’ADN 4,5,27. Cela permet de gagner du temps et de réduire les coûts, car les étapes typiques de la réaction enzymatique sont évitées, ce qui permet d’économiser jusqu’à 72 h de manipulations en laboratoire 2,15. De plus, l’équipement et les réactifs nécessaires à l’IVA sont généralement déjà présents ou disponibles dans les laboratoires qui effectuent régulièrement du clonage moléculaire.
Notre groupe a récemment utilisé l’IVA pour générer une série de 24 plasmides avec plusieurs sites de clonage identiques, trois options pour le nombre de copies du plasmide et cinq options pour les marqueurs de sélection d’antibiotiques17. Nous avons démontré comment des amorces universelles pouvaient être utilisées pour cloner des gènes dans chaque plasmide17. Dans cette étude, l’efficacité du clonage était de 71 à 100 %, car très peu de colonies ont été dépistées pour identifier les clones positifs (sept ont été dépistées pour cinq clones positifs)17. Une autre étude a rapporté que les taux d’efficacité du clonage IVA atteignaient un maximum de 97 % ± 1,9 %, le taux plus élevé peut s’expliquer par le fait que les auteurs testent systématiquement un grand nombre de clones pour un assemblage correct du plasmide16. L’IVA peut également être utilisée pour des applications telles que la mutagénèse dirigée ou l’intégration d’amorces contenant des séquences de marqueurs27. L’IVA est une technique de clonage polyvalente et efficace qui peut être adaptée à de nombreuses applications de clonage moléculaire de routine en laboratoire.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Le programme HGB est financé par un programme de bourses d’études supérieures du Canada au niveau de la maîtrise du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG) et une bourse de doctorat du doyen (Université de la Saskatchewan). Ces travaux ont été soutenus par une subvention à la découverte du CRSNG (RGPIN-2021-03066), des fonds de démarrage (à JLT) de l’Université de la Saskatchewan et le Fonds des leaders John R. Evans de la Fondation canadienne pour l’innovation (subvention numéro 42269 à JLT). Les auteurs remercient M. Eric Toombs d’avoir fourni la photographie de la quantification de l’ADN par spectroscopie UV.
matériels
| Name | Company | Catalog Number | Comments |
| 1 kb Plus DNA ladder | FroggaBio | DM015-R500 | DNA molecular weight marker |
| AccuReady M Single Channel 0.5-10 µL | Bulldog Bio | BPP020 | Pipettes for transferring liquid |
| AccuReady M Single Channel Pipettor Kit | Bulldog Bio | BPK100 | Pipettes for transferring liquid |
| Agar | BioShop Canada | AGR003.1 | Solidifying agent for growth medium |
| Agarose | Biobasic | D0012 | Make agarose gels for DNA electrophoresis |
| Biometra TOne | Analytikjena | 846-2-070-301 | Thermocycler |
| Caps for glass culture tubes | Fisher Scientific | 14-957-91 | Reusable caps for glass culture tubes |
| DNA primers | Integrated DNA Technologies | N/A | Bind and amplify template DNA in PCR reaction |
| DpnI | New England Biolabs | R0176S | Cleave methylated template DNA following PCR amplification |
| EcoRI | New England Biolabs | R3101L | Restriction enzyme, comes with appropriate buffer |
| Eppendorf microtubes 1.5 mL | Sarstedt | 72.690.300 | Microtube, 1.5 mL, conical base, PP, attached flat cap, molded graduations and frosted writing space |
| Ethidium bromide | Fisher Scientific | AAL0748203 | DNA visualization/intercalating agent, toxic |
| EZ-10 Spin Column Plasmid DNA Miniprep Kit | Biobasic | BS614 | Isolate plasmid DNA from overnight bacterial cultures |
| GelDoc Go-Gel system | Bio-Rad | 12009077 | Imaging DNA gels |
| Glass culture tubes | Fisher Scientific | 14-925E | Glass culture tubes |
| myGel Mini Electrophoresis System | Sigma-Aldrich | Z742288 | System used for gel electrophoresis |
| NanoDrop One | Thermo Fisher | ND-ONE-W | Determine DNA concentration |
| PCR Clean Up for DNA Sequencing | Biobasic | BT5100 | Purify PCR products |
| Petri dish | Sarstedt | 82.1473.011 | Petri dish 92 x 16 mm, PS, transparent, with ventilation cams |
| Pipette tip, 1000 µL | Sarstedt | 70.305 | Pipette tips for 100-1000 µL |
| Pipette tip, 2.5 µL | Sarstedt | 70.3010.265 | Pipette tips for up to 2.5 µL |
| Pipette tip, 20 µL | Sarstedt | 70.3020.200 | Pipette tips for up to 20 µL |
| Pipette tip, 200 µL | Sarstedt | 70.3030.020 | Pipette tips for 1-200 µL |
| Salt (NaCl) | Fisher Scientific | S271-10 | Component of bacterial growth medium (10 g/L) |
| Single 0.2 mL PCR tubes with flat cap | FroggaBio | TF-1000 | PCR tubes |
| Tryptone (Bacteriological) | BioShop Canada | TRP402.5 | Component of bacterial growth medium (10 g/L) |
| VeriFi DNA polymerase | PCR Biosystems | PB10.42-01 | High fidelity polymerase, the PCR buffer containing Mg2+ and dNTPs is provided with purchase |
| Xba I | New England Biolabs | R0145S | Restriction enzyme, comes with appropriate buffer |
| Yeast extract | BioShop Canada | YEX401.205 | Component of bacterial growth medium (5 g/L) |
Références
- Bertero, A., Brown, S., Vallier, L. Methods of cloning. Basic Science Methods for Clinical Researchers. Chapter 2, 19-39 (2017).
- Garcia-Nafria, J., Watson, J. F., Greger, I. H. IVA cloning: A single-tube universal cloning system exploiting bacterial in vivo assembly. Sci Rep. 6, 27459 (2016).
- Bubeck, P., Winkler, M., Bautsch, W. Rapid cloning by homologous recombination in vivo. Nucleic Acids Res. 21 (15), 3601-3602 (1993).
- Avilan, L. Assembling multiple fragments: the Gibson assembly. Methods Mol Biol. 2633, 45-53 (2023).
- Stukenberg, D., et al. The Marburg Collection: A Golden Gate DNA assembly framework for synthetic biology applications in Vibrio natriegens. ACS Synth Biol. 10 (8), 1904-1919 (2021).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Engler, C., Marillonnet, S. Golden Gate cloning. Methods Mol Biol. 1116, 119-131 (2014).
- Ma, H., Kunes, S., Schatz, P. J., Botstein, D. Plasmid construction by homologous recombination in yeast. Gene. 58 (2-3), 201-216 (1987).
- Cao, P., Wang, L., Zhou, G., Wang, Y., Chen, Y. Rapid assembly of multiple DNA fragments through direct transformation of PCR products into E. coli and Lactobacillus. Plasmid. 76, 40-46 (2014).
- Conley, E. C., Saunders, V. A., Saunders, J. R. Deletion and rearrangement of plasmid DNA during transformation of Escherichia coli with linear plasmid molecules. Nucleic Acids Res. 14 (22), 8905-8917 (1986).
- Conley, E. C., Saunders, V. A., Jackson, V., Saunders, J. R. Mechanism of intramolecular recyclization and deletion formation following transformation of Escherichia coli with linearized plasmid DNA. Nucleic Acids Res. 14 (22), 8919-8932 (1986).
- Sung, W. L., Zahab, D. M. Site-specific recombination directed by single-stranded crossover linkers: specific deletion of the amino-terminal region of the beta-galactosidase gene in pUC plasmids. DNA. 6 (4), 373-379 (1987).
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci USA. 83 (19), 7177-7181 (1986).
- Jones, D. H., Howard, B. H. A rapid method for recombination and site-specific mutagenesis by placing homologous ends on DNA using polymerase chain reaction. Biotechniques. 10 (1), 62-66 (1991).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5alpha-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), e0137466 (2015).
- Chen, F., et al. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 21 (1), 24 (2021).
- Braun, H. G., Kanwal, N., Rivera Lopez, L. F., Thomassin, J. L. Generation of a plasmid series for rapid sub-cloning and use in various Enterobacteriaceae. J Biosci Bioeng. 138 (6), 478-487 (2024).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Res. 35 (Web Server issue), W43-W46 (2007).
- Wallace, R. B., et al. Hybridization of synthetic oligodeoxyribonucleotides to phi chi 174 DNA: the effect of single base pair mismatch. Nucleic Acids Res. 6 (11), 3543-3557 (1979).
- Bzymek, M., Lovett, S. T. Instability of repetitive DNA sequences: The role of replication in multiple mechanisms. Proc Natl Acad Sci USA. 98 (15), 8319-8325 (2001).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Sambrook, J., Russell, D. W. The inoue method for preparation and transformation of competent E. coli: "ultra-competent" cells. CSH Protoc. 2006 (1), (2006).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Fürste, J. P., et al. Molecular cloning of the plasmid RP4 primase region in a multi-host-range tacP expression vector. Gene. 48 (1), 119-131 (1986).
- Bäumler, A. J., et al. Identification of a new iron regulated locus of Salmonella typhi. Gene. 183 (1-2), 207-213 (1996).
- Godiska, R., et al. Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli. Nucleic Acids Res. 38 (6), e88 (2010).
- Watson, J. F., Garcia-Nafria, J. In vivo DNA assembly using common laboratory bacteria: A re-emerging tool to simplify molecular cloning. J Biol Chem. 294 (42), 15271-15281 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon