Method Article
Co-cultura de contato direto de astrócitos e células de glioblastoma padronizadas usando modelos multicamadas de polieletrólitos
Neste Artigo
Resumo
Este protocolo descreve uma co-cultura padronizada de glioma-astrócitos de contato direto utilizando impressão de microcontato em multicamadas de polieletrólitos (PEMs) para padronizar células U87 ou A172 GBM e astrócitos primários.
Resumo
O glioblastoma multiforme (GBM) é o câncer cerebral maligno mais abundante e fatal. Existem mais de 13.000 casos projetados nos Estados Unidos em 2020 e 2021. Os tumores GBM geralmente surgem de astrócitos e são caracterizados por sua natureza invasiva, muitas vezes recrutando tecidos saudáveis para o tecido tumoral. Compreender a comunicação entre astrócitos e células de glioblastoma é vital para a compreensão molecular da progressão tumoral. Este protocolo demonstra um novo método de co-cultura padronizado para investigar os efeitos mediados por contato de astrócitos no GBM empregando montagem camada por camada e padronização acionada por força microcapilar. As vantagens incluem um ambiente de cultura de células livre de proteínas e controle preciso da interação celular ditada pelas dimensões do padrão. Esta técnica fornece um protocolo versátil, econômico e reprodutível para imitar a interação celular entre glioma e astrócitos em tumores de glioma. Este modelo pode ainda ser usado para separar mudanças na biologia molecular do GBM devido ao contato físico com astrócitos ou com comunicação de cofatores solúveis mediada por não contato.
Introdução
O glioblastoma multiforme (GBM) é o câncer cerebral mais prolífico e mortal nos Estados Unidos, com um tempo médio de sobrevida de cerca de 15meses. Menos de 7% dos pacientes com GBM sobrevivem mais de 5 anos após o diagnóstico 1,2. Em 10 anos, esse número cai para menos de 1%1,2. Embora outros tipos de câncer tenham feito melhorias marcantes na sobrevida nas últimas décadas, o sucesso dos pacientes com GBM é insuficiente. Para desenvolver intervenções terapêuticas bem-sucedidas, um modelo in situ apropriado deve ser utilizado para desenvolver uma compreensão mais completa da biologia do tumor de GBM. Esse entendimento é crucial para melhorar os resultados clínicos para pacientes com GBM.
O cérebro contém uma grande variedade de tipos de células, cada uma preenchendo nichos específicos para promover a função e a sobrevivência do organismo. Além dos neurônios, há uma variedade de células gliais, incluindo astrócitos, oligodendrócitos e microglia. Os astrócitos, em particular, têm sido implicados no crescimento e invasão do tumor GBM através da secreção de fatores solúveis pró-migratórios3. Além disso, existem alguns relatos de que o contato físico é a força motriz da migração e invasão de células de glioma mediadas por astrócitos 4,5. No entanto, a base molecular que impulsiona essa mudança permanece amplamente desconhecida.
A fim de estudar os efeitos mediados por contato dos astrócitos no crescimento tumoral, este protocolo relata o desenvolvimento de um método reprodutível e livre de proteínas para padronização celular in vitro. Neste método, as multicamadas de polieletrólitos (PEMs) são sistematicamente montadas para formar uma superfície uniforme livre de proteínas. Os PEMs são construídos usando um sistema de policátion-poliânion composto por poli(cloreto de dialilmetilamônio) (PDAC) e poli(estério) sulfonado (SPS), respectivamente. Esses polímeros foram escolhidos com base em estudos anteriores que relataram ligação preferencial de células ao SPS sobre PDAC 6,7,8,9,10. Nessas superfícies PEM, este protocolo utiliza litografia de força microcapilar para projetar modelos padronizados de co-cultura de astrócitos primários e células GBM.
As técnicas aqui apresentadas permitem a engenharia precisa de interações célula-célula específicas por meio do controle da padronização de superfície, apoiando assim a investigação altamente reprodutível da comunicação celular. Além disso, a superfície biomimética inerente a esta plataforma facilita o estudo da comunicação direta célula-célula, que é crucial para o aprofundamento da compreensão mecanicista da comunicação entre diferentes tipos de células. Além disso, o método é de baixo custo, proporcionando uma vantagem significativa para a realização de estudos in vitro para explorar a comunicação celular. Especificamente, este protocolo explora a ligação diferencial de GBM em SPS (-) sobre PDAC (+) para criar co-culturas padronizadas de linhagens celulares GBM e astrócitos primários.
Protocolo
Este estudo foi realizado em estrita conformidade com as recomendações do Guia para o Cuidado e Uso de Animais de Laboratório do National Institutes of Health. O protocolo foi aprovado pelo Comitê de Ética em Experimentos com Animais da Universidade de Nebraska-Lincoln (ID do Projeto: 1046). Os astrócitos primários foram preparados a partir de filhotes de ratos Sprague-Dawley de 1 a 3 dias de idade, em conformidade com o protocolo IACUC 1046 da UNL e de acordo com o protocolo com pequenas modificações 7,11.
1. Preparativos
- Obtenha um padrão mestre da geometria desejada antes de iniciar este protocolo.
NOTA: Wafers de silício disponíveis comercialmente preparados usando técnicas de fotolitografia padrão são usados neste protocolo. O padrão de partida recomendado é de linhas de 100-200 μm. - Prepare buffers e mídia.
- Prepare soluções de polímero de poli (dialilmetilamônio) (PDAC) e poli (estérino) sulfonado (SPS) para revestir placas.
- A solução de revestimento PDAC é 0,3% em peso de PDAC e 0,1 M NaCl em ddH2O.
- A solução de revestimento SPS é 30 μM SPS e 0,1 M NaCl em ddH2O.
NOTA: 1 L de solução de revestimento PDAC e 1 L de solução de revestimento SPS é suficiente para revestir seis placas de 6 poços. As soluções podem ser reutilizadas de 2 a 5 vezes. Substitua as soluções quando elas ficarem turvas.
- Preparar meios de cultura de células padrão, se necessário.
- Os meios típicos de astrócitos e GBM são, respectivamente, 10% de soro fetal bovino (FBS) e 1% de penicilina-estreptomicina no meio de Eagle modificado (DMEM) de Dulbecco.
- Prepare soluções de polímero de poli (dialilmetilamônio) (PDAC) e poli (estérino) sulfonado (SPS) para revestir placas.
- Obtenha, prepare ou descongele células.
NOTA: Este protocolo é descrito para astrócitos primários de ratos isolados de ratos neonatais de 1-3 dias e células de glioma U87MG ou A172-MG. Este protocolo pode ser expandido para linhagens celulares semelhantes após a confirmação da ligação diferencial de linhagens celulares de interesse em superfícies poliméricas.- Devido à perda de células durante a coloração, use um mínimo de 200.000 células GBM no Dia 0 e 400.000 astrócitos primários vivos de ratos no Dia 1 por poço em uma placa de 6 poços. Ajuste os números das células se estiver usando um poço ou placa de tamanho diferente.
NOTA: Astrócitos primários das passagens 2-4 preparados conforme descrito por Wilson e colaboradores12 são recomendados para este protocolo. As células de glioma foram obtidas comercialmente (ver Tabela de Materiais).
- Devido à perda de células durante a coloração, use um mínimo de 200.000 células GBM no Dia 0 e 400.000 astrócitos primários vivos de ratos no Dia 1 por poço em uma placa de 6 poços. Ajuste os números das células se estiver usando um poço ou placa de tamanho diferente.
2. Moldagem de carimbo de polidimetilsiloxano (PDMS)
- Em um recipiente descartável, pesar 12 partes (em peso) de pré-polímero PDMS e 1 parte (em peso) de agente de cura. Misture vigorosamente por 2-4 min até que toda a mistura esteja cheia de bolhas.
NOTA: 26 g da mistura são suficientes para uma placa de Petri de 100 mm. - Coloque o fluorosilano no dessecador de fluorosilano. Coloque o recipiente descartável com a mistura de polímeros no dessecador para desgaseificar por 20 min.
NOTA: Isso permite que as bolhas saiam da mistura. Pode ser necessário mais tempo se ainda houver bolhas grandes após 20 minutos. - Coloque o mestre de estrutura pré-padronizado em uma placa de Petri. Despeje a mistura PDMS lentamente na placa de Petri sobre o mestre de estrutura pré-padronizado, garantindo cobrir completamente o mestre. A profundidade ideal da camada PDMS é de cerca de 2-3 mm de espessura.
NOTA: Evite criar bolhas de ar despejando lentamente a mistura. Certifique-se de que o mestre de silicone esteja deitado na placa de Petri. - Mantenha a placa de Petri no dessecador de fluorosilano até que todas as bolhas sejam removidas. Cure a placa de Petri em um forno a 60 °C por 1 h. Se não estiver disponível 60 °C, catalise a placa de Petri a 37 °C durante a noite.
- Usando um bisturi afiado, corte uniforme e suavemente ao redor do mestre.
CUIDADO: Tenha cuidado ao usar um bisturi. Nunca deixe a lâmina exposta sem vigilância. Os bisturis são afiados. Use as precauções de segurança apropriadas para evitar ferimentos. - Remova o carimbo usando uma pinça e coloque-o em uma superfície de corte, como uma tábua de cortar. Usando um bisturi, corte os selos em um tamanho pequeno o suficiente para caber em placas de 6 poços (aproximadamente 2,5 cm x 2,5 cm).
3. Construção de multicamadas de polieletrólitos (PEMs)
NOTA: Este protocolo descreve o revestimento de plasma com um tanque de oxigênio pressurizado anexado e um acessório de mistura de gás (consulte a Tabela de Materiais). Qualquer método de limpeza uniforme do plasma da superfície do poliestireno da cultura de tecidos (TCPS) é adequado. O tempo e a intensidade podem precisar ser otimizados pelo usuário ao usar um método diferente de limpeza a plasma.
- Limpe com plasma uma placa de 6 poços por 7 min.
- Sangre a câmara girando a válvula de três vias na frente da porta para a direita até que o chiado da liberação de ar possa ser ouvido. Gire a válvula de três vias de volta para a posição vertical quando a leitura da pressão estiver acima de 1800 mTorr.
- Abra a porta e coloque a placa de 6 poços na câmara. Feche a porta e ligue a bomba de vácuo. Evacuar a câmara até que a pressão esteja estável em torno de 100 mTorr.
NOTA: Enquanto a câmara está evacuando, confirme se o tanque de oxigênio está aberto e se a pressão de saída do oxigênio é de 10 psi. - Abra a válvula de três vias para a esquerda para alinhar a linha com a mangueira de entrada de oxigênio. Deixe o gás oxigênio sangrar para a câmara até que a pressão se estabilize entre 400 e 450 mTorr.
NOTA: Confirme se a pressão de entrada no medidor de vazão é de 10 mm e ajuste se necessário. - Ligue a alimentação de RF. Aguarde a formação do plasma (~15 s) e, em seguida, inicie o cronômetro por 7 min.
- Após 7 min, desligue a alimentação de RF e gire a válvula de três vias de volta à sua posição vertical. Deixe a câmara evacuar para 150 mTorr.
- Desligue a bomba de vácuo e espere que a pressão suba para 1500 mTorr. Ventile a câmara girando a válvula de três vias para a direita até que o chiado da liberação de ar possa ser ouvido. Quando a leitura da pressão estiver acima de 1800 mTorr, gire a válvula de três vias de volta à sua posição vertical, abra a porta e recupere a amostra.
- Coloque as placas revestidas de plasma em banhos de temperatura ambiente da seguinte forma: PDAC por 20 min, água DI por 5 min, água DI por 5 min, SPS por 20 min, água DI por 5 min e água DI por 5 min. Certifique-se de que toda a superfície da placa esteja submersa.
NOTA: Opcionalmente, um corador de lâmina automatizado pode ser usado para reduzir o tempo ativo. Use agitação suave, se disponível. - Repita a etapa anterior um total de 10 vezes. Deixe as placas secarem ao ar.
NOTA: As placas secas podem ser armazenadas em temperatura ambiente por várias semanas antes do uso.
4. Padronização PEM via micromoldagem em capilares
- Lave os carimbos PDMS com sabão de laboratório suave seguido de água DI. Seque os carimbos usando um compressor de ar e coloque-os em uma superfície plana e móvel, como uma tampa de placa de 6 poços não utilizada.
OPCIONAL: Os carimbos também podem ser secos ao ar. Os selos devem ser cobertos e protegidos contra poeira durante a secagem. - Cubra os carimbos com plasma, conforme descrito na etapa 3, por 1 min. Remova imediatamente os selos do limpador de plasma e coloque-os voltados para baixo nas placas de 6 poços preparadas.
- Pipetar 10 μL de solução de PDAC ao longo da borda inferior do carimbo, dispensando continuamente o polímero ao longo do comprimento do carimbo. Certifique-se de adicionar o polímero na lateral do carimbo com aberturas dos padrões de linha para permitir que a ação capilar ocorra.
- Coloque um peso de 350 g em cima de cada carimbo por 20 s para ajudar a reforçar o padrão. Deixe os carimbos descansarem nas placas por 20 min.
- Mergulhe a placa em água DI e retire os carimbos na direção dos padrões de linha. Lave a placa duas vezes por 5 min cada em água DI.
- Lave os carimbos com sabão e água DI e seque com um compressor de ar. Deixe a placa secar ao ar.
- Se estiver pronto para cultura de células, esterilize as placas sob luz ultravioleta em uma capa de biossegurança Classe II por um mínimo de 8 h imediatamente antes do uso. A dose total mínima recomendada de UV-C é de 400 mJ/cm2 a 265 nm. Se estiver visualizando padrões, pule a etapa de esterilização UV e execute a etapa 5. Se estiver pronto para a cultura de células, execute a etapa 6 após a esterilização UV.
5. Visualização de padrões estampados com carboxifluoresceína (CFSE)
NOTA: Execute este procedimento para visualizar padrões carimbados ou ignore esta etapa e, em vez disso, prepare padrões para cultura de células. Uma vez que os padrões são corados, eles não podem ser usados para cultura de células. Uma vez que a confiança suficiente na estampagem é adquirida, os padrões podem ser usados para cultura de células. A carboxifluoresceína é sensível à luz. Mancha e transporte no escuro.
- Coloque os padrões secos e estampados na solução de coloração do padrão CFSE (0,1 μM CFSE em 0,1 M NaOH) por 60-90 min. Lave os padrões estampados por 5 min em DI H2O duas vezes.
NOTA: Se estiver usando lâminas de vidro, coloque a lâmina inteira em um tubo de 50 mL contendo a solução de coloração. Se estiver usando placas de poço, adicione solução de coloração suficiente para cobrir todo o padrão. - Visualize padrões usando um microscópio de fluorescência apropriado.
- Ligue a lâmpada fluorescente para permitir que ela aqueça por 10 min. Como os padrões são corados com CFSE, use o filtro azul de interferência (IB) para visualizar os padrões verdes. Veja os padrões e tire fotos.
6. Coloração e semeadura de células de glioblastoma com carboxifluoresceína
NOTA: Todo o trabalho de cultura de células deve ser realizado em um gabinete de biossegurança Classe II adequado.
- Separar as células da superfície de cultura com 0,25% de tripsina-EDTA, centrifugar a 200 x g durante 4 min a 4 °C e remover o sobrenadante.
- Ressuspenda as células em 1 mL de meio de cultura celular sem soro (DMEM e penicilina-estreptomicina a 1%), centrifugue a 200 x g por 4 min a 4 ° C e remova o sobrenadante.
- Ressuspenda as células em 1 mL de PBS e transfira para um tubo estéril de 1,5 mL. Adicione 10 μL de 10 μg / mL CFSE e misture imediatamente pipetando para cima e para baixo. Incube em temperatura ambiente por 10 min e transfira para um tubo estéril de 15 mL.
- Adicione um mínimo de 1 mL de meio de cultura celular (consulte a etapa 1.2.2.1) para extinguir a reação do corante CFSE. Centrifugar a 400 x g durante 10 min a 4 °C e remover o sobrenadante. Ressuspenda as células em 5 ml de meio de cultura celular e transfira para um novo tubo estéril de 15 ml.
- Centrifugue a 400 x g durante 5 min a 4 °C, remova o sobrenadante e ressuspenda as células em 5 ml de meio de cultura celular.
- Repita a etapa de lavagem anterior duas vezes.
- Semeie as células de glioma coradas (100 células/mm2 ou 100.000 células/poço) na placa de cultura de tecidos padronizada de 6 poços da etapa 4 com meio de cultura de astrócitos. Cultivar as células numa incubadora (37 °C, 5% CO2) durante 24 h antes de adicionar astrócitos corados no passo 7.
7. Coloração e semeadura de astrócitos primários com PKH26
NOTA: Todo o trabalho de cultura de células deve ser realizado em um gabinete de biossegurança Classe II adequado.
- Separar as células da superfície de cultura com 0,25% de tripsina-EDTA, centrifugar a 200 x g durante 4 min a 4 °C e remover o sobrenadante.
- Ressuspenda as células em 1 mL de meio de cultura celular sem soro (DMEM e penicilina-estreptomicina a 1%), centrifugue a 200 x g por 4 min a 4 ° C e remova o sobrenadante.
- Prepare uma suspensão de 2 células ressuspendendo o pellet celular em 0,5 mL de Diluente C em temperatura ambiente do kit PKH26.
- Prepare uma solução de corante 2x (4 x 10-6 M) adicionando 2 μL de PKH26 a 0,5 mL de Diluente C em um tubo estéril de 1,5 mL. Prepare a solução de corante antes de usar.
- Adicione 2x suspensão celular a 2x solução de corante e misture imediatamente pipetando para cima e para baixo.
- Incube a suspensão celular por 1-5 min.
NOTA: A coloração acontece rapidamente e o Diluente C é prejudicial às células. Incubação mais longa não significa melhor tingimento. - Extinguir a coloração adicionando um volume mínimo de 5x do meio de cultura de células e incubando por 1 min para ligar o excesso de corante.
- Centrifugue a 200 x g durante 10 min a 4 °C e retire cuidadosamente o sobrenadante. Ressuspenda as células em 5 mL de meio de cultura celular e transfira para um novo tubo estéril de 15 mL.
- Centrifugar a 200 x g durante 5 min a 4 °C, retirar o sobrenadante e ressuspender em 5 ml de PBS.
- Centrifugar a 200 x g durante 5 min a 4 °C, remover o sobrenadante e ressuspender em 5 ml de meios de cultura celular.
- Repita as etapas 7.9 e 7.10 para um total de três lavagens.
- Semeie os astrócitos corados (200 células/mm2 ou 200.000 células/poço) com meio de astrócitos (consulte a etapa 1.2.2.1) na placa de cultura de tecidos padronizada de 6 poços pré-semeada com células de glioma da etapa 6. Cultive as células em incubadora de CO2 a 37 °C, 5% durante o tempo em que os experimentos são realizados.
8. Imagem de fluorescência de monocultura e cocultura padronizadas
- Ligue a lâmpada fluorescente para permitir que ela aqueça por 10 min.
- Remova a cultura de células da incubadora e coloque-a no palco de um microscópio invertido capaz de imagens de fluorescência.
- Insira ou coloque o filtro apropriado. Se as células que estão sendo visualizadas estiverem coradas com CFSE, use o filtro azul de interferência (IB) para visualizar as células verdes. Se as células que estão sendo visualizadas estiverem coradas com PKH26, use um filtro de rodamina para visualizar os glóbulos vermelhos.
- Veja o espécime e tire fotos.
NOTA: As células agora podem ser separadas usando citometria de fluxo para analisar os efeitos biológicos da comunicação mediada por contato.
Resultados
O protocolo aqui descreve a engenharia de co-cultura padronizada de contato direto de células de glioma e astrócitos. Esta plataforma fornece um modelo multicelular biomimético para estudar o papel do contato direto na comunicação entre astrócitos e células de glioma na progressão do glioblastoma multiforme (GBM). A Figura 1 fornece um esquema da modificação passo a passo da superfície e da introdução celular descrita acima. O primeiro passo é obter uma plataforma de cultura (lamínula de vidro, placa de cultura de tecidos, etc.) revestida com um revestimento PEM auto-montado de PDAC / SPS em 10 bicamadas e um carimbo PDMS do padrão espacial desejado para a co-cultura. Dez bicamadas de PDAC / SPS foram estabelecidas anteriormente por Kidambi et al. para fornecer um revestimento PEM uniforme útil para o controle espacial de fibroblastos, neurônios e células de câncer de mama 6,7,8. O selo PDMS formado a partir de um mestre de silício fornece uma ferramenta versátil e reutilizável para aplicar uma variedade de padrões para estudar várias interações celulares, incluindo aquelas que requerem contato físico para explorar a importância de métodos de comunicação específicos na progressão do tumor.

Figura 1: Visão geral esquemática para empregar PEMs e micromoldagem em capilares para criar uma plataforma de co-cultura padronizada de células de glioma e astrócitos do cerebelo. (A) O pré-polímero PDMS e o agente de cura são misturados 12: 1 e derramados sobre wafers de silício com nano-características desejadas, preparados comercialmente usando técnicas de fotolitografia padrão (1). (B) O substrato da cultura de células é tratado com plasma de oxigênio para se tornar carregado negativamente (2). O substrato é mergulhado sequencialmente em poli (cloreto de dialdimetilamônio) (PDAC) por 20 min, deionizado (DI) H2O por 5 min, DI H2O por 5 min, poli (sulfonato de estireno) (SPS) por 20 min, DI H2O por 5 min e DI H2O por 5 min. Este processo de seis banhos é repetido 10 vezes. Isso pode ser feito manualmente ou com a ajuda de um revestidor de imersão programável (3). (C) O substrato agora tem 10 bicamadas estáveis de PDAC e SPS. Os selos PDMS (de A) são cortados em tamanhos utilizáveis, tratados com plasma de oxigênio e colocados (com o lado do padrão voltado para baixo) em placas de 6 poços (4). (D) Uma pipeta é usada para dispensar ~ 10 μL de PDAC ao longo da borda do carimbo PDMS, mostrado em vermelho (5). O movimento lento e constante ao longo de toda a borda do carimbo garantirá uma cobertura uniforme. Os padrões podem ser verificados usando corantes carregados (6) ou podem ser preparados para cultura de células. (E) As superfícies padronizadas do substrato são esterilizadas por UV durante a noite. (F) As células de glioma são coradas com CFSE e são semeadas no substrato padronizado. As células de glioma têm 1-2 dias para se ligarem às superfícies SPS dentro dos padrões no substrato. (G) Os astrócitos primários do cerebelo são corados com PKH26 e são preenchidos no substrato padronizado. Imagens e análises podem ser feitas assim que a co-cultura amadurecer no dia 4. Criado com BioRender.com Clique aqui para ver uma versão maior desta figura.
Vários padrões podem ser utilizados nesta plataforma. A Figura 2 mostra micropadrões visualizados com carboxifluoresceína demonstrando a versatilidade de forma e tamanho alcançada com o uso de micromoldagem nos capilares. Por exemplo, a interação física heterogênea de células ocorrerá apenas na interface entre PDAC e SPS dos padrões. Portanto, o padrão quadrado no canto inferior esquerdo da Figura 2 demonstra a capacidade de controlar a organização espacial dos padrões, que pode ser estendida para controlar a interação célula-célula específica com base nos posicionamentos das células dentro desses padrões. A comparação de co-culturas nesses padrões permitiria investigar a influência geral da comunicação direta em oposição aos fatores solúveis e da comunicação não direta, por meio da sinalização parácrina, com maior precisão13. Além disso, essa plataforma permite que o investigador projete padrões para controlar o número de interações celulares heterogêneas, imitando os vários estágios de crescimento do tumor GBM e as posições dentro do tumor para imitar de maneira ideal as observações in vivo 14.

Figura 2: PEMs com diferentes padrões corados com carboxifluoresceína. Os PEMs permitem que o investigador ajuste as condições da superfície (química, forma e tamanho) para otimizar a cultura. Barra de escala 500 μm. Clique aqui para ver uma versão maior desta figura.
A Figura 3 mostra monoculturas de células de glioma seletivamente ligadas às regiões SPS na superfície PDAC / SPS padronizada após 24 h. As imagens representativas demonstram a capacidade de A172 e U87 de aderir seletivamente a regiões específicas, deixando espaço para o preenchimento de astrócitos. A linhagem celular U87MG é comumente usada para estudos de glioblastoma, pois é uma linha celular de glioma invasiva e altamente tumorigênica. A172 também foi escolhido para este estudo como uma linha celular de glioblastoma invasiva e não tumorigênica. Apesar das diferenças nessas linhagens celulares, elas parecem se comportar de forma semelhante quando introduzidas na superfície padronizada do PDAC/SPS, sendo distribuídas espacialmente de acordo com o padrão disponível. Isso destaca a versatilidade dessa plataforma no estudo de estágios variados do glioblastoma15. Como as células de glioma têm uma taxa de fixação e crescimento relativamente rápida em comparação com os astrócitos, a introdução de astrócitos na cultura é realizada um dia após a semeadura do glioma. Isso é para evitar o crescimento excessivo de glioma em regiões especificamente destinadas à fixação de astrócitos.
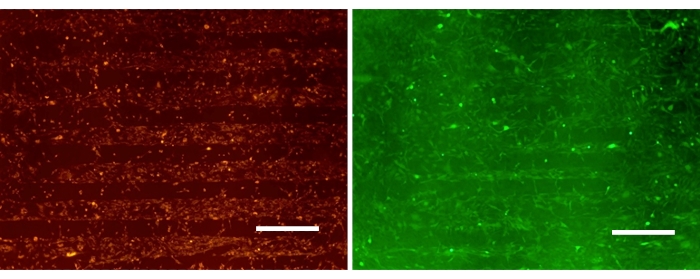
Figura 3: Monoculturas de glioma padronizadas coradas com carboxifluoresceína (verde) e PKH26 (vermelho) em superfícies padronizadas PEM. Barra de escala: 500 μm. Clique aqui para ver uma versão maior desta figura.
A Figura 4 é representativa da co-cultura madura realizada 4 dias após a semeadura dos astrócitos. Os astrócitos são semeados em alta densidade para garantir a fixação total no espaço desocupado pelo glioma. Após a adição de astrócitos, a cultura amadurece 4 dias antes da análise. O tempo de maturação (dia 1-4 da co-cultura) permite que as células do glioma preencham as regiões às quais se ligam e sufoquem os astrócitos que podem ter tentado se ligar nas regiões em que as células do glioma estão estabelecidas. Além disso, o período de maturação permite o estabelecimento de interações físicas, como junções comunicantes, que são comumente vistas como mediadoras da interação celular de astrócitos e células de glioma in vivo e in vitro 16,17,18.

Figura 4: Imagens fluorescentes no dia 4 de co-cultura. (A) Astrócitos co-cultivados com células de glioma U87 e (B) astrócitos co-cultivados com células de glioma A172 no dia 4 de co-cultura demonstrando a capacidade seletiva dessas superfícies PEM. Barra de escala: 500 μm. Clique aqui para ver uma versão maior desta figura.
Discussão
As etapas críticas para garantir a montagem bem-sucedida de uma co-cultura padronizada reproduzível incluem: 1) a padronização bem-sucedida da superfície por micromoldagem em capilares, 2) a lavagem bem-sucedida de células coradas e 3) a análise da co-cultura na janela de "cultura madura". Primeiro, a reprodução bem-sucedida de padrões com micromoldagem em capilares é fundamental para a reprodutibilidade da interação, pois é isso que diferencia a co-cultura padronizada da co-cultura aleatória. Para garantir essa reprodutibilidade, é importante que o usuário desenvolva a habilidade de padronizar por meio da prática ou obtenha um sistema automatizado que possa recriar padrões com precisão. Além disso, embora os carimbos PDMS sejam reutilizáveis, a modificação e o uso repetidos do carimbo podem desgastar a integridade do padrão ao longo do tempo. Portanto, é aconselhável testar periodicamente a integridade do padrão com um microscópio ou visualizar padrões com carboxifluoresceína. Em segundo lugar, a lavagem bem-sucedida de células coradas com PKH26 e carboxifluoresceína é crucial para a diferenciação dos tipos de células ao estabelecer o método pela primeira vez e seguir os diferentes tipos de células. Além disso, a coloração cruzada devido à lavagem malsucedida pode inibir a capacidade futura de separar as células da co-cultura por citometria de fluxo. Este protocolo inclui etapas de lavagem otimizadas para o equipamento de citometria de fluxo e criação bem-sucedida da co-cultura; no entanto, pode ser necessário aumentar o número de etapas de lavagem se o usuário encontrar consistentemente o cruzamento de manchas durante a co-cultura. Finalmente, a janela de tempo para analisar uma cultura madura é limitada, pois as células de glioma acabarão preenchendo o padrão e começarão a invadir regiões ricas em astrócitos do padrão. Portanto, é aconselhável analisar a co-cultura em intervalos de tempo consistentes para garantir a reprodutibilidade dos resultados.
Os problemas potenciais que os usuários podem encontrar incluem problemas com a saturação CFSE. Se as imagens estiverem saturadas com fluorescência, reduza o tempo de coloração do padrão de 60-90 min para 30-60 min. Se o problema persistir, diminua a concentração da mancha para 0,075 μM em NaOH 0,1 M. Alternativamente, se as imagens não mostrarem fluorescência, primeiro confirme se as configurações do microscópio e o prisma são apropriados para comprimentos de onda de excitação e emissão de 492 nm e 517 nm, respectivamente (ou seja, filtro azul de interferência (IB)) e verifique uma faixa de tempos de exposição de 100 ms a 10.000 ms. Se ainda não houver fluorescência, aumente o tempo de coloração do padrão de 60-90 min para 90-120 min. Se o problema persistir, aumente a concentração da coloração para 0,15 μM em NaOH 0,1 M. Problemas adicionais que os usuários podem encontrar com padrões visualizados por CFSE são bordas borradas ou difusas em vez de geometrias nítidas desejadas, o que pode ser um sinal de carimbos PDMS usados em excesso. Nesse caso, faça novos carimbos PDMS usando os wafers mestres de silício. Imagens borradas ou difusas também podem ser um sinal de solução de coloração CFSE contaminada e, neste caso, preparar uma nova solução de coloração CFSE.
Este sistema tem certas limitações inerentes. Em primeiro lugar, é uma monocultura bidimensional que, embora útil para estudos preliminares de alto rendimento 6,7,8 e comparável aos modelos de interação atualmente disponíveis, fica aquém do mimetismo ideal do tecido tridimensional in vivo 19. Além disso, a interação física é otimizada neste modelo de co-cultura, mas a interação por meio de fatores solúveis não é completamente eliminada devido à natureza estática deste sistema. O meio estático pode ser uma vantagem, pois a análise dos fatores solúveis liberados pode ser perspicaz para a compreensão da resposta celular20. A compreensão da interação puramente física requer interação minimizada por meio de fatores solúveis, o que pode ser alcançado por meio da substituição frequente de meios e manutenção de um grande volume de meios para difusão de fatores.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado, no todo ou em parte, por subsídios do NIH 1R01AA027189-01A1 (para SK), P20 GM104320 (para o Centro de Nebraska para a Prevenção de Doenças da Obesidade Subsídio Piloto para SK), P20 GM113126 (para o Centro de Nebraska para Comunicação Biomolecular Integrada - Líder do Projeto SK); Subsídio de Sementes Biomédicas do Escritório de Pesquisa e Desenvolvimento da UNL e Subsídio de Sistemas de Iniciativa de Pesquisa de Nebraska (para SK). O K.M.S. foi financiado por meio do T32GM107001, uma bolsa de treinamento.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Fisher Scientific | 25200056 | |
| 15 ml Nunc Conical Sterile Polypropylene Centrifuge Tubes | Fisher Scientific | 12-565-268 | |
| 5(6)-Carboxyfluorescein diacetate N-succinimidyl ester | Millipore Sigma | Cat#21888 | |
| 50 ml Nunc Conical Sterile Polypropylene Centrifuge Tubes | Fisher Scientific | 12-565-270 | |
| A172-MG GBM cell line | ATCC | CRL-1620 | |
| Bright-Line Hemacytometer | Sigma | Z359629 | Or other suitable cell counting device |
| Cell Incubator | N/A | N/A | |
| Cooled tabletop centrifuge for 15 mL tubes | N/A | N/A | |
| Dulbecco's Modified Eagle Medium (DMEM) | MP Biomedicals | ICN 1033120 | |
| Expanded Plasma Cleaner | Plasma Harrick | PDC-001-HP | With attached pressurized oxygen tank and PlasmaFlo Gas Mixer (PDC-FMG) accessory |
| Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11550H | |
| Fluorosilane | Sigma Aldrich | 667420 | Full chemical name: 1H,1H,2H,2H-Perfluorooctyltriethoxysilane |
| Inverted Tabletop Microscope | N/A | N/A | Microscope capable of fluorescent imaging with λex = 551 nm; λem 567 nm [e.g. Rhodamine filter] (PKH26 dye) and λex 492 nm; λem 517 nm [e.g. interference blue filter (IB)] (CFSE dye) |
| NaCl | Sigma Aldrich | S7653 | |
| NaOH | Sigma Aldrich | 567530 | |
| Penicillin-Streptomicen | Fisher Scientific | 15140122 | |
| PKH26 Red Fluorescent Cell Linker Mini Kit | Millipore Sigma | Cat#MINI26-1KT | |
| Poly(diallyldimethylammonium chloride) solution (PDAC) | Sigma Aldrich | 409014 | 20 wt. % in H2O |
| Poly(sodium 4-styrenesulfonate) (SPS) | Sigma Aldrich | 243051 | average MW ~70,000 |
| Primary Astrocytes, isolated from Srague Dawley rats | Charles River | Crl:SD | Rats from Charles River; Lab isolated Cells |
| Scalpel | N/A | N/A | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Chemical | Cat#2646340 | |
| Trypan Blue Stain | Fisher Scientific | 15-250-061 | |
| TryplE | Fisher Scientific | Gibco TrypLE | |
| U87-MG GBM cell line | ATCC | HTB-14 | |
| Vacuum Desiccator | N/A | N/A |
Referências
- Ostrom, Q. T., et al. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2012-2016. Neuro Oncology. 21, 1 (2019).
- Ostrom, Q. T., et al. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2010-2014. Neuro Oncology. 19, 1-88 (2017).
- Roos, A., Ding, Z., Loftus, J. C., Tran, N. L. Molecular and microenvironmental determinants of glioma stem-like cell survival and invasion. Frontiers in Oncology. 7, 120 (2017).
- Rath, B. H., Fair, J. M., Jamal, M., Camphausen, K., Tofilon, P. J. Astrocytes enhance the invasion potential of glioblastoma stem-like cells. PLoS One. 8 (1), 54752 (2013).
- Le, D. M., et al. Exploitation of astrocytes by glioma cells to facilitate invasiveness: a mechanism involving matrix metalloproteinase-2 and the urokinase-type plasminogen activator-plasmin cascade. The Journal of Neuroscience. 23 (10), 4034-4043 (2003).
- Daverey, A., Brown, K. M., Kidambi, S. Breast cancer/stromal cells coculture on polyelectrolyte films emulates tumor stages and miRNA profiles of clinical samples. Langmuir. 31 (36), 9991-10001 (2015).
- Kidambi, S., Lee, I., Chan, C. Primary neuron/astrocyte co-culture on polyelectrolyte multilayer films: A template for studying astrocyte-mediated oxidative stress in neurons. Advanced Functional Materials. 18 (2), 294-301 (2008).
- Kidambi, S., et al. Patterned co-culture of primary hepatocytes and fibroblasts using polyelectrolyte multilayer templates. Macromolecular Bioscience. 7 (3), 344-353 (2007).
- Kidambi, S., Lee, I., Chan, C. Controlling primary hepatocyte adhesion and spreading on protein-free polyelectrolyte multilayer films. Journal of the American Chemical Society. 126 (50), 16286-16287 (2004).
- Mohammed, J. S., DeCoster, M. A., McShane, M. J. Micropatterning of nanoengineered surfaces to study neuronal cell attachment in vitro. Biomacromolecules. 5 (5), 1745-1755 (2004).
- Beaudoin, G. M., et al. Culturing pyramidal neurons from the early postnatal mouse hippocampus and cortex. Nature Protocols. 7 (9), 1741-1754 (2012).
- Wilson, C. L., Hayward, S. L., Kidambi, S. Astrogliosis in a dish: substrate stiffness induces astrogliosis in primary rat astrocytes. Rsc Advances. 6 (41), 34447-34457 (2016).
- Bogdanowicz, D. R., Lu, H. H. Studying cell-cell communication in co-culture. Biotechnololgy Journal. 8 (4), 395-396 (2013).
- Chen, W., et al. Glioma cells escaped from cytotoxicity of temozolomide and vincristine by communicating with human astrocytes. Medical Oncology. 32 (3), 43 (2015).
- Belot, N., et al. Molecular characterization of cell substratum attachments in human glial tumors relates to prognostic features. Glia. 36 (3), 375-390 (2001).
- Sin, W. C., et al. Astrocytes promote glioma invasion via the gap junction protein connexin43. Oncogene. 35 (12), 1504-1516 (2016).
- Sin, W. C., Crespin, S., Mesnil, M. Opposing roles of connexin43 in glioma progression. Biochimica et Biophysica Acta. 1818 (8), 2058-2067 (2012).
- Oliveira, R., et al. Contribution of gap junctional communication between tumor cells and astroglia to the invasion of the brain parenchyma by human glioblastomas. BMC Cell Biology. 6 (1), 7 (2005).
- Duval, K., et al. Modeling physiological events in 2D vs. 3D cell culture. Physiology (Bethesda). 32 (4), 266-277 (2017).
- Rape, A., Ananthanarayanan, B., Kumar, S. Engineering strategies to mimic the glioblastoma microenvironment). Advanced Drug Delivery Reviews. 79-80, 172-183 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados