Method Article
Co-coltura a contatto diretto di astrociti e cellule di glioblastoma modellate utilizzando modelli multistrato di polielettrolita
In questo articolo
Riepilogo
Questo protocollo descrive una co-coltura di glioma-astrocita a contatto diretto modellata che utilizza la stampa a microcontatto su multistrati di polielettroliti (PEM) per modellare le cellule GBM U87 o A172 e gli astrociti primari.
Abstract
Il glioblastoma multiforme (GBM) è il tumore maligno cerebrale più abbondante e fatale. Ci sono più di 13.000 casi previsti negli Stati Uniti nel 2020 e nel 2021. I tumori GBM derivano più spesso dagli astrociti e sono caratterizzati dalla loro natura invasiva, spesso reclutando tessuti sani nel tessuto tumorale. Comprendere la comunicazione tra astrociti e cellule di glioblastoma è fondamentale per la comprensione molecolare della progressione tumorale. Questo protocollo dimostra un nuovo metodo di co-coltura patterned per studiare gli effetti mediati dal contatto degli astrociti sul GBM impiegando l'assemblaggio strato per strato e il patterning guidato dalla forza microcapillare. I vantaggi includono un ambiente di coltura cellulare privo di proteine e un controllo preciso dell'interazione cellulare dettato dalle dimensioni del modello. Questa tecnica fornisce un protocollo versatile, economico e riproducibile per imitare l'interazione cellulare tra glioma e astrociti nei tumori del glioma. Questo modello può essere ulteriormente utilizzato per individuare i cambiamenti nella biologia molecolare del GBM dovuti al contatto fisico con gli astrociti o con la comunicazione di cofattori solubili mediati senza contatto.
Introduzione
Il glioblastoma multiforme (GBM) è il tumore cerebrale più prolifico e mortale negli Stati Uniti, con un tempo di sopravvivenza mediano di circa 15 mesi. Meno del 7% dei pazienti con GBM sopravvive più di 5 anni dopo la diagnosi 1,2. A 10 anni, questa cifra scende a meno dell'1%1,2. Sebbene altri tipi di cancro abbiano apportato notevoli miglioramenti nella sopravvivenza negli ultimi decenni, il successo dei pazienti con GBM non è all'altezza. Per sviluppare interventi terapeutici di successo, è necessario utilizzare un modello in situ appropriato per sviluppare una comprensione più approfondita della biologia del tumore del GBM. Questa comprensione è fondamentale per migliorare i risultati clinici per i pazienti con GBM.
Il cervello contiene una grande varietà di tipi di cellule, ognuna delle quali riempie nicchie specifiche per promuovere la funzione e la sopravvivenza dell'organismo. Oltre ai neuroni, ci sono una varietà di cellule gliali, tra cui astrociti, oligodendrociti e microglia. Gli astrociti, in particolare, sono stati implicati nella crescita e nell'invasione tumorale del GBM attraverso la secrezione di fattori solubili pro-migratori3. Inoltre, ci sono alcuni rapporti secondo cui il contatto fisico è la forza trainante della migrazione e dell'invasione delle cellule di glioma mediata dagli astrociti 4,5. Tuttavia, le basi molecolari che guidano questo cambiamento rimangono in gran parte sconosciute.
Al fine di studiare gli effetti mediati dal contatto degli astrociti sulla crescita tumorale, questo protocollo riporta lo sviluppo di un metodo riproducibile e privo di proteine per il patterning cellulare in vitro. In questo metodo, i multistrati di polielettroliti (PEM) vengono sistematicamente assemblati per formare una superficie uniforme priva di proteine. I PEM sono costruiti utilizzando un sistema policatione-polianione che comprende rispettivamente poli(diallilmetilammonio cloruro) (PDAC) e poli(sterene) solfonato (SPS). Questi polimeri sono stati scelti sulla base di studi precedenti che hanno riportato un legame preferenziale delle cellule a SPS rispetto a PDAC 6,7,8,9,10. Su queste superfici PEM, questo protocollo utilizza la litografia a forza microcapillare per progettare modelli di co-coltura modellati di astrociti primari e cellule GBM.
Le tecniche qui presentate consentono l'ingegnerizzazione precisa di specifiche interazioni cellula-cellula attraverso il controllo del pattern di superficie, supportando così lo studio altamente riproducibile della comunicazione cellulare. Inoltre, la superficie biomimetica inerente a questa piattaforma facilita lo studio della comunicazione diretta cellula-cellula che è cruciale per approfondire la comprensione meccanicistica della comunicazione tra diversi tipi di cellule. Oltre a questo, il metodo è a basso costo e offre un vantaggio significativo per la conduzione di studi in vitro per esplorare la comunicazione cellulare. In particolare, questo protocollo sfrutta l'attaccamento differenziale di GBM su SPS (-) su PDAC (+) per creare co-colture modellate di linee cellulari di GBM e astrociti primari.
Protocollo
Questo studio è stato condotto in stretta conformità con le raccomandazioni della Guida per la cura e l'uso degli animali da laboratorio del National Institutes of Health. Il protocollo è stato approvato dal Comitato per l'Etica degli Esperimenti Animali dell'Università del Nebraska-Lincoln (ID Progetto: 1046). Gli astrociti primari sono stati preparati da cuccioli di ratto Sprague-Dawley di 1-3 giorni in conformità con il protocollo IACUC 1046 dell'UNL e secondo il protocollo con lievi modifiche 7,11.
1. Preparativi
- Ottenere un modello principale della geometria desiderata prima di iniziare questo protocollo.
NOTA: In questo protocollo vengono utilizzati wafer di silicio disponibili in commercio preparati utilizzando tecniche di fotolitografia standard. Lo schema di partenza consigliato è di linee di 100-200 μm. - Preparare buffer e supporti.
- Preparare soluzioni polimeriche di poli(diallilmetilammonio-cloruro) (PDAC) e poli(sterene) solfonato (SPS) per rivestire le lastre.
- La soluzione di rivestimento PDAC è 0,3% in peso di PDAC e 0,1 M NaCl in ddH2O.
- La soluzione di rivestimento SPS è 30 μM SPS e 0,1 M NaCl in ddH2O.
NOTA: 1 L di soluzione di rivestimento PDAC e 1 L di soluzione di rivestimento SPS sono sufficienti per rivestire sei piastre a 6 pozzetti. Le soluzioni possono essere riutilizzate da 2 a 5 volte. Sostituire le soluzioni quando diventano torbide.
- Preparare terreni di coltura cellulare standard, se necessario.
- I tipici terreni astrocitari e GBM sono, rispettivamente, il 10% di siero fetale bovino (FBS) e l'1% di penicillina-streptomicina nel Modified Eagle Medium (DMEM) di Dulbecco.
- Preparare soluzioni polimeriche di poli(diallilmetilammonio-cloruro) (PDAC) e poli(sterene) solfonato (SPS) per rivestire le lastre.
- Ottenere, preparare o scongelare le cellule.
NOTA: Questo protocollo è descritto per gli astrociti primari di ratto isolati da ratti neonati di 1-3 giorni e per le cellule di glioma U87MG o A172-MG. Questo protocollo può essere esteso a linee cellulari simili dopo la conferma dell'adesione differenziale di linee cellulari di interesse su superfici polimeriche.- A causa della perdita di cellule durante la colorazione, utilizzare un minimo di 200.000 cellule GBM il giorno 0 e 400.000 astrociti primari di ratto vivi il giorno 1 per pozzetto in una piastra a 6 pozzetti. Regolare il numero di celle se si utilizza un pozzetto o una piastra di dimensioni diverse.
NOTA: Per questo protocollo si raccomandano astrociti primari dai passaggi 2-4 preparati come descritto da Wilson e collaboratori12. Le cellule di glioma sono state ottenute commercialmente (vedi Tabella dei materiali).
- A causa della perdita di cellule durante la colorazione, utilizzare un minimo di 200.000 cellule GBM il giorno 0 e 400.000 astrociti primari di ratto vivi il giorno 1 per pozzetto in una piastra a 6 pozzetti. Regolare il numero di celle se si utilizza un pozzetto o una piastra di dimensioni diverse.
2. Stampaggio di timbri in polidimetilsilossano (PDMS)
- In un contenitore monouso, pesare 12 parti (in peso) di prepolimero PDMS e 1 parte (in peso) di agente indurente. Mescolare energicamente per 2-4 minuti fino a riempire l'intero composto di bolle.
NOTA: 26 g di miscela sono sufficienti per una capsula di Petri da 100 mm. - Posizionare il fluorosilano nell'essiccatore fluorosilano. Posizionare il contenitore usa e getta con la miscela di polimeri nell'essiccatore per degassare per 20 minuti.
NOTA: Ciò consente alle bolle di fuoriuscire dalla miscela. Potrebbe essere necessario più tempo se dopo 20 minuti sono ancora presenti bolle di grandi dimensioni. - Posizionare il master di struttura pre-modellato in una capsula di Petri. Versare lentamente la miscela PDMS nella capsula di Petri sopra il master della struttura pre-modellata, assicurandosi di coprire completamente il master. La profondità ottimale dello strato PDMS è di circa 2-3 mm di spessore.
NOTA: Evitare di creare bolle d'aria versando lentamente il composto. Assicurarsi che il master in silicone sia disteso nella piastra di Petri. - Conservare la capsula di Petri nell'essiccatore al fluorosilano fino a quando tutte le bolle non sono state rimosse. Polimerizzare la capsula di Petri in forno a 60 °C per 1 ora. Se non sono disponibili 60 °C, polimerizzare la capsula di Petri a 37 °C per una notte.
- Usando un bisturi affilato, taglia uniformemente e delicatamente attorno al master.
ATTENZIONE: Prestare attenzione quando si utilizza un bisturi. Non lasciare mai incustodita la lama esposta. I bisturi sono affilati. Utilizzare le precauzioni di sicurezza appropriate per evitare lesioni. - Rimuovere il timbro con una pinzetta e posizionarlo su una superficie di taglio come un tagliere. Usando un bisturi, taglia i timbri in una dimensione abbastanza piccola da entrare in piastre a 6 pozzetti (circa 2,5 cm x 2,5 cm).
3. Costruzione di multistrati di polielettrolita (PEM)
NOTA: Questo protocollo descrive il rivestimento al plasma con un serbatoio di ossigeno pressurizzato collegato e un accessorio per la miscelazione del gas (vedere la Tabella dei materiali). È adatto qualsiasi metodo per pulire uniformemente al plasma la superficie del polistirene per colture tissutali (TCPS). Potrebbe essere necessario ottimizzare il tempo e l'intensità dall'utente quando si utilizza un diverso metodo di pulizia al plasma.
- Pulire al plasma una piastra a 6 pozzetti per 7 minuti.
- Spurgare la camera ruotando verso destra la valvola a tre vie sulla parte anteriore della porta fino a quando non si sente il sibilo del rilascio dell'aria. Riportare la valvola a tre vie in posizione verticale una volta che la lettura della pressione è superiore a 1800 mTorr.
- Aprire lo sportello e inserire la piastra a 6 pozzetti nella camera. Chiudere lo sportello e accendere la pompa del vuoto. Evacuare la camera fino a quando la pressione è stabile intorno a 100 mTorr.
NOTA: Durante l'evacuazione della camera, verificare che il serbatoio dell'ossigeno sia aperto e che la pressione di uscita dell'ossigeno sia di 10 psi. - Aprire la valvola a tre vie a sinistra per allineare la linea con il tubo di ingresso dell'ossigeno. Lasciare che l'ossigeno gassoso penetri nella camera fino a quando la pressione non si stabilizza tra 400 e 450 mTorr.
NOTA: Verificare che la pressione di ingresso sul flussometro sia di 10 mm e regolarla se necessario. - Attiva l'alimentazione RF. Attendere che si formi il plasma (~15 s), quindi avviare il timer per 7 minuti.
- Dopo 7 minuti, spegnere l'alimentazione RF e riportare la valvola a tre vie in posizione verticale. Consentire l'evacuazione della camera a 150 mTorr.
- Spegnere la pompa del vuoto e attendere che la pressione salga a 1500 mTorr. Sfiatare la camera ruotando la valvola a tre vie verso destra fino a quando non si sente il sibilo del rilascio dell'aria. Quando la lettura della pressione è superiore a 1800 mTorr, riportare la valvola a tre vie in posizione verticale, aprire lo sportello e recuperare il campione.
- Posizionare le piastre rivestite al plasma in bagni a temperatura ambiente come segue: PDAC per 20 minuti, acqua deionizzata per 5 minuti, acqua deionizzata per 5 minuti, SPS per 20 minuti, acqua deionizzata per 5 minuti e acqua deionizzata per 5 minuti. Assicurarsi che l'intera superficie della piastra sia sommersa.
NOTA: Opzionalmente, è possibile utilizzare un coloratore automatico per vetrini per ridurre il tempo attivo. Usa un'agitazione delicata, se disponibile. - Ripetere il passaggio precedente per un totale di 10 volte. Lasciare asciugare le piastre all'aria.
NOTA: Le piastre essiccate possono essere conservate a temperatura ambiente fino a diverse settimane prima dell'uso.
4. Modellazione PEM tramite microstampaggio nei capillari
- Lavare i timbri PDMS con sapone da laboratorio delicato seguito da acqua deionizzata. Asciugare i timbri utilizzando un compressore d'aria e posizionarli su una superficie piana e mobile, come un coperchio per piastre a 6 pozzetti inutilizzato.
OPZIONALE: I timbri possono anche essere asciugati all'aria. I timbri devono essere coperti e protetti dalla polvere durante l'asciugatura. - Rivestire i timbri al plasma, come descritto al punto 3, per 1 minuto. Rimuovere prontamente i timbri dal pulitore al plasma e posizionarli a faccia in giù sulle piastre a 6 pozzetti preparate.
- Pipettare 10 μl di soluzione di PDAC lungo il bordo inferiore del timbro, erogando costantemente il polimero lungo la lunghezza del timbro. Assicurarsi di aggiungere il polimero sul lato del timbro con le aperture dei modelli di linea per consentire l'azione capillare.
- Metti un peso di 350 g sopra ogni timbro per 20 s per aiutare a rafforzare il motivo. Lasciare riposare i timbri sui piatti per 20 minuti.
- Immergi la piastra in acqua DI e stacca i timbri nella direzione delle linee. Lavare la piastra due volte per 5 minuti ciascuna in acqua deionizzata.
- Lavare i timbri con sapone e acqua deionizzata e asciugarli con un compressore d'aria. Lasciare asciugare la piastra all'aria.
- Se sono pronte per la coltura cellulare, sterilizzare le piastre sotto la luce UV in una cappa di biosicurezza di Classe II per un minimo di 8 ore immediatamente prima dell'uso. La dose totale minima raccomandata di UV-C è di 400 mJ/cm2 a 265 nm. Se si visualizzano i modelli, saltare la fase di sterilizzazione UV ed eseguire il passaggio 5. Se si è pronti per la coltura cellulare, eseguire il passaggio 6 dopo la sterilizzazione UV.
5. Visualizzazione di modelli stampati con carbossifluoresceina (CFSE)
NOTA: Eseguire questa procedura per visualizzare i modelli stampati o saltare questo passaggio e preparare invece i modelli per la coltura cellulare. Una volta che i modelli sono stati colorati, non possono essere utilizzati per la coltura cellulare. Una volta acquisita sufficiente fiducia nella timbratura, i modelli possono essere utilizzati per la coltura cellulare. La carbossifluoresceina è sensibile alla luce. Macchiare e trasportare al buio.
- Mettere i modelli essiccati e stampati nella soluzione di colorazione del modello CFSE (0,1 μM di CFSE in 0,1 M di NaOH) per 60-90 minuti. Lavare i modelli stampati per 5 minuti in DI H2O due volte.
NOTA: Se si utilizzano vetrini, inserire l'intero vetrino in una provetta da 50 ml contenente la soluzione colorante. Se si utilizzano piastre a pozzetti, aggiungere una quantità sufficiente di soluzione colorante per coprire l'intero modello. - Visualizzare i modelli utilizzando un microscopio a fluorescenza appropriato.
- Accendi la lampadina fluorescente per farla riscaldare per 10 minuti. Poiché i modelli sono colorati con CFSE, utilizzare il filtro blu di interferenza (IB) per visualizzare i modelli verdi. Visualizza i modelli e scatta foto.
6. Colorazione e semina di cellule di glioblastoma con carbossifluoresceina
NOTA: Tutte le operazioni di coltura cellulare devono essere eseguite in un'adeguata cappa di biosicurezza di Classe II.
- Staccare le cellule dalla superficie di coltura con tripsina-EDTA allo 0,25%, centrifugare a 200 x g per 4 minuti a 4 °C e rimuovere il surnatante.
- Risospendere le cellule in 1 mL di terreno di coltura cellulare senza siero (DMEM e 1% penicillina-streptomicina), centrifugare a 200 x g per 4 minuti a 4 °C e rimuovere il surnatante.
- Risospendere le cellule in 1 mL di PBS e trasferirle in una provetta sterile da 1,5 mL. Aggiungere 10 μL di 10 μg/mL di CFSE e miscelare immediatamente accuratamente pipettando su e giù. Incubare a temperatura ambiente per 10 minuti e trasferire in una provetta sterile da 15 mL.
- Aggiungere un minimo di 1 mL di terreno di coltura cellulare (vedere il passaggio 1.2.2.1) per estinguere la reazione del colorante CFSE. Centrifugare a 400 x g per 10 min a 4 °C e rimuovere il surnatante. Risospendere le cellule in 5 mL di terreno di coltura cellulare e trasferirle in una provetta sterile fresca da 15 mL.
- Centrifugare a 400 x g per 5 minuti a 4 °C, rimuovere il surnatante e risospendere le cellule in 5 mL di terreno di coltura cellulare.
- Ripetere due volte la fase di lavaggio precedente.
- Seminare le cellule di glioma colorate (100 cellule/mm2 o 100.000 cellule/pozzetto) nella piastra di coltura tissutale a 6 pozzetti modellata dalla fase 4 con terreni di coltura per astrociti. Coltivare le cellule in un incubatore (37 °C, 5% CO2) per 24 ore prima di aggiungere gli astrociti colorati nella fase 7.
7. Colorazione e semina di astrociti primari con PKH26
NOTA: Tutto il lavoro di coltura cellulare deve essere eseguito in una cappa di biosicurezza di Classe II adeguata.
- Staccare le cellule dalla superficie di coltura con tripsina-EDTA allo 0,25%, centrifugare a 200 x g per 4 minuti a 4 °C e rimuovere il surnatante.
- Risospendere le cellule in 1 mL di terreno di coltura cellulare senza siero (DMEM e 1% penicillina-streptomicina), centrifugare a 200 x g per 4 minuti a 4 °C e rimuovere il surnatante.
- Preparare una sospensione cellulare 2x risospendendo il pellet cellulare in 0,5 mL di diluente C a temperatura ambiente del kit PKH26.
- Preparare una soluzione colorante 2x (4 x 10-6 M) aggiungendo 2 μL di PKH26 a 0,5 mL di Diluente C in una provetta sterile da 1,5 mL. Preparare la soluzione colorante subito prima dell'uso.
- Aggiungere 2 sospensioni cellulari a 2 soluzioni coloranti e mescolare immediatamente pipettando su e giù.
- Incubare la sospensione cellulare per 1-5 minuti.
NOTA: La colorazione avviene rapidamente e il diluente C è dannoso per le cellule. Un'incubazione più lunga non significa una tintura migliore. - Estinguere la colorazione aggiungendo un volume minimo di 5 volte il terreno di coltura cellulare e incubando per 1 minuto per legare il colorante in eccesso.
- Centrifugare a 200 x g per 10 minuti a 4 °C e rimuovere con cura il surnatante. Risospendere le cellule in 5 mL di terreno di coltura cellulare e trasferirle in una nuova provetta sterile da 15 mL.
- Centrifugare a 200 x g per 5 minuti a 4 °C, rimuovere il surnatante e risospendere in 5 mL di PBS.
- Centrifugare a 200 x g per 5 minuti a 4 °C, rimuovere il surnatante e risospendere in 5 mL di terreno di coltura cellulare.
- Ripetere i passaggi 7.9 e 7.10 per un totale di tre lavaggi.
- Seminare gli astrociti colorati (200 cellule/mm2 o 200.000 cellule/pozzetto) con terreni astrocitari (vedere il passaggio 1.2.2.1) nella piastra di coltura tissutale a 6 pozzetti pre-seminata con cellule di glioma del passaggio 6. Coltivare le cellule in un incubatore a 37 °C, 5% CO2 per il tempo in cui vengono eseguiti gli esperimenti.
8. Imaging a fluorescenza di monocolture e cocolture patternizzate
- Accendi la lampadina fluorescente per farla riscaldare per 10 minuti.
- Estrarre la coltura cellulare dall'incubatore e posizionarla sul tavolino di un microscopio invertito in grado di eseguire l'imaging a fluorescenza.
- Inserire o collegare il filtro appropriato. Se le cellule di cui si esegue l'imaging sono colorate con CFSE, utilizzare il filtro blu di interferenza (IB) per visualizzare le celle verdi. Se le cellule oggetto dell'imaging sono colorate con PKH26, utilizzare un filtro alla rodamina per visualizzare i globuli rossi.
- Visualizza l'esemplare e scatta foto.
NOTA: Le cellule possono ora essere separate utilizzando la citometria a flusso per analizzare gli effetti biologici della comunicazione mediata dal contatto.
Risultati
Il protocollo qui descritto descrive l'ingegnerizzazione della co-coltura con pattern di contatto diretto di cellule di glioma e astrociti. Questa piattaforma fornisce un modello biomimetico multicellulare per studiare il ruolo del contatto diretto nella comunicazione tra astrociti e cellule di glioma nella progressione del glioblastoma multiforme (GBM). La Figura 1 fornisce uno schema della modifica graduale della superficie e dell'introduzione cellulare delineata sopra. La prima fase consiste nell'ottenere una piattaforma di coltura (vetrino coprioggetti, piastra di coltura tissutale, ecc.) rivestita con un rivestimento PEM autoassemblato di PDAC/SPS in 10 doppi strati e un timbro PDMS del modello spaziale desiderato per la co-coltura. Dieci doppi strati di PDAC/SPS sono stati precedentemente stabiliti da Kidambi et al. per fornire un rivestimento PEM uniforme utile per il controllo spaziale di fibroblasti, neuroni e cellule di cancro al seno 6,7,8. Il timbro PDMS formato da un master in silicio fornisce uno strumento versatile e riutilizzabile per applicare una varietà di modelli per studiare varie interazioni cellulari, comprese quelle che richiedono il contatto fisico per esplorare l'importanza di metodi di comunicazione specifici nella progressione del tumore.

Figura 1: Panoramica schematica per l'impiego di PEM e micro-stampaggio nei capillari per creare una piattaforma di co-coltura modellata di cellule di glioma e astrociti del cervelletto. (A) Il pre-polimero PDMS e l'agente indurente vengono miscelati 12:1 e versati su wafer di silicio con le nano-caratteristiche desiderate, preparati commercialmente utilizzando tecniche di fotolitografia standard (1). (B) Il substrato della coltura cellulare viene trattato con plasma di ossigeno per caricarsi negativamente (2). Il substrato viene immerso in sequenza in poli(diallimetilammoniocloruro) (PDAC) per 20 minuti, deionizzato (DI) H2O per 5 minuti, DI H2O per 5 minuti, poli(stirene solfonato) (SPS) per 20 minuti, DI H2O per 5 minuti e DI H2O per 5 minuti. Questo processo di sei bagni viene ripetuto 10 volte. Questa operazione può essere eseguita manualmente o con l'assistenza di un dip coater programmabile (3). (C) Il substrato ha ora 10 doppi strati stabili di PDAC e SPS. I timbri PDMS (da A) vengono tagliati a pezzi in dimensioni utilizzabili, trattati con plasma di ossigeno e posizionati (con il lato del modello rivolto verso il basso) su piastre a 6 pozzetti (4). (D) Una pipetta viene utilizzata per erogare ~10 μL di PDAC lungo il bordo del timbro PDMS, mostrato in rosso (5). Un movimento lento e costante lungo tutto il bordo del timbro garantirà una copertura uniforme. I modelli possono essere verificati utilizzando coloranti caricati (6) o possono essere preparati per la coltura cellulare. (E) Le superfici del substrato modellato vengono sterilizzate ai raggi UV durante la notte. (F) Le cellule di glioma vengono colorate con CFSE e seminate sul substrato modellato. Alle cellule di glioma vengono concessi 1-2 giorni per attaccarsi alle superfici SPS all'interno dei modelli sul substrato. (G) Gli astrociti del cervelletto primario sono colorati con PKH26 e vengono riempiti sul substrato modellato. Le immagini e l'analisi possono essere scattate una volta che la co-coltura matura il giorno 4. Creato con BioRender.com Clicca qui per visualizzare una versione più grande di questa figura.
In questa piattaforma è possibile utilizzare vari modelli. La Figura 2 mostra i micro-pattern visualizzati con carbossifluoresceina che dimostrano la versatilità di forma e dimensioni ottenuta utilizzando il microstampaggio nei capillari. Ad esempio, l'interazione fisica eterogenea delle cellule si verificherà solo all'interfaccia tra PDAC e SPS dei modelli. Pertanto, il modello quadrato nell'angolo in basso a sinistra della Figura 2 dimostra la capacità di controllare l'organizzazione spaziale dei modelli, che può essere ulteriormente estesa al controllo della specifica interazione cellula-cellula in base al posizionamento delle cellule all'interno di questi modelli. Il confronto delle co-colture in questi modelli consentirebbe di studiare l'influenza complessiva della comunicazione diretta rispetto ai fattori solubili e della comunicazione non diretta, attraverso la segnalazione paracrina, con maggiore precisione13. Inoltre, questa piattaforma consente allo sperimentatore di progettare modelli per il controllo del numero di interazioni cellulari eterogenee che imitano le varie fasi di crescita del tumore GBM e le posizioni all'interno del tumore per imitare in modo ottimale le osservazioni in vivo 14.

Figura 2: PEM con diversi pattern colorati con carbossifluoresceina. I PEM consentono allo sperimentatore di regolare le condizioni della superficie (chimica, forma e dimensioni) per ottimizzare la coltura. Barra graduata 500 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
La Figura 3 mostra le monocolture di cellule di glioma attaccate selettivamente alle regioni SPS nella superficie PDAC/SPS modellata dopo 24 ore. Le immagini rappresentative dimostrano la capacità di A172 e U87 di aderire selettivamente a regioni specifiche, lasciando spazio per il riempimento degli astrociti. La linea cellulare U87MG è comunemente utilizzata per gli studi sul glioblastoma in quanto è una linea cellulare di glioma invasiva e altamente tumorigenica. A172 è stato scelto per questo studio anche come linea cellulare di glioblastoma invasiva e non tumorigenica. Nonostante le differenze in queste linee cellulari, esse sembrano comportarsi in modo simile quando vengono introdotte sulla superficie PDAC/SPS modellata, essendo distribuite spazialmente in base al modello disponibile. Ciò evidenzia la versatilità di questa piattaforma nello studio di vari stadi del glioblastoma15. Poiché le cellule di glioma hanno un tasso di attaccamento e di crescita relativamente rapido rispetto agli astrociti, l'introduzione degli astrociti nella coltura viene eseguita un giorno dopo la semina del glioma. Questo per prevenire la crescita eccessiva di glioma in regioni specificamente destinate all'attacco degli astrociti.
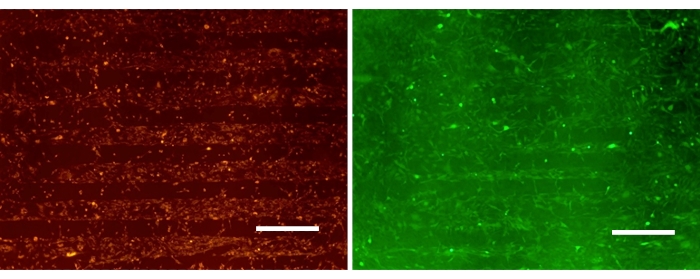
Figura 3: Monocolture di glioma patterned colorate con carbossifluoresceina (verde) e PKH26 (rosso) su superfici patterned PEM. Barra della scala: 500 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
La Figura 4 è rappresentativa della co-coltura matura effettuata 4 giorni dopo la semina degli astrociti. Gli astrociti sono seminati ad alta densità per garantire il pieno attaccamento nello spazio non occupato dal glioma. Dopo l'aggiunta di astrociti, la coltura viene lasciata maturare 4 giorni prima dell'analisi. Il tempo di maturazione (giorno 1-4 di co-coltura) consente alle cellule di glioma di riempire le regioni a cui si attaccano e di soffocare gli astrociti che potrebbero aver tentato di attaccarsi nelle regioni in cui si sono stabilite le cellule di glioma. Inoltre, il periodo di maturazione consente l'instaurazione di interazioni fisiche, come le giunzioni gap, che sono comunemente viste mediare l'interazione cellulare di astrociti e cellule di glioma in vivo e in vitro 16,17,18.

Figura 4: Immagini fluorescenti al 4° giorno di co-coltura. (A) Astrociti co-coltivati con cellule di glioma U87 e (B) astrociti co-coltivati con cellule di glioma A172 al 4° giorno di co-coltura che dimostrano la capacità selettiva di queste superfici PEM. Barra della scala: 500 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
I passaggi critici per garantire il successo dell'assemblaggio di una co-coltura patterned riproducibile includono: 1) il successo del patterning della superficie mediante microstampaggio nei capillari, 2) il successo del lavaggio delle cellule colorate e 3) l'analisi della co-coltura nella finestra di "coltura matura". In primo luogo, la riproduzione di successo dei pattern con microstampaggio nei capillari è fondamentale per la riproducibilità dell'interazione, poiché questo è ciò che distingue la co-coltura patterned dalla co-coltura casuale. Per garantire questa riproducibilità, è importante che l'utente sviluppi l'abilità di modellare attraverso la pratica o ottenga un sistema automatizzato in grado di ricreare modelli con precisione. Inoltre, sebbene i timbri PDMS siano riutilizzabili, la modifica e l'uso ripetuti del timbro possono logorare l'integrità del modello nel tempo. Pertanto, si consiglia di testare periodicamente l'integrità del modello con un microscopio o di visualizzare i modelli con carbossifluoresceina. In secondo luogo, il successo del lavaggio delle cellule colorate con PKH26 e carbossifluoresceina è fondamentale per la differenziazione dei tipi di cellule quando si stabilisce per la prima volta il metodo e si seguono i diversi tipi di cellule. Inoltre, la colorazione incrociata dovuta a un lavaggio non riuscito può inibire la futura capacità di separare le cellule dalla co-coltura tramite citometria a flusso. Questo protocollo include fasi di lavaggio ottimizzate per l'apparecchiatura di citometria a flusso e la creazione di successo della co-coltura; Tuttavia, potrebbe essere necessario aumentare il numero di fasi di lavaggio se l'utente trova costantemente un cross-over di macchie durante la co-coltura. Infine, la finestra temporale per l'analisi di una coltura matura è limitata poiché le cellule di glioma alla fine riempiranno il modello e inizieranno a invadere le regioni ricche di astrociti del modello. Pertanto, è consigliabile analizzare la co-coltura a intervalli di tempo coerenti per garantire la riproducibilità dei risultati.
I potenziali problemi che gli utenti potrebbero incontrare includono problemi con la saturazione CFSE. Se le immagini sono sature di fluorescenza, ridurre il tempo di colorazione del modello da 60-90 minuti a 30-60 minuti. Se il problema persiste, ridurre la concentrazione della macchia a 0,075 μM in 0,1 M NaOH. In alternativa, se le immagini non mostrano fluorescenza, verificare innanzitutto che le impostazioni del microscopio e del prisma siano appropriate per lunghezze d'onda di eccitazione ed emissione rispettivamente di 492 nm e 517 nm (ad esempio, filtro blu di interferenza (IB)) e controllare un intervallo di tempi di esposizione da 100 ms a 10.000 ms. Se non c'è ancora fluorescenza, aumentare il tempo di colorazione del modello da 60-90 minuti a 90-120 minuti. Se il problema persiste, aumentare la concentrazione del colorante a 0,15 μM in 0,1 M NaOH. Ulteriori problemi che gli utenti possono incontrare con i modelli visualizzati da CFSE sono i bordi sfocati o sfocati invece delle geometrie nitide desiderate, che possono essere un segno di timbri PDMS abusati. In questo caso, crea nuovi timbri PDMS utilizzando i wafer master in silicio. Le immagini sfocate o sfocate possono anche essere un segno di una soluzione colorante CFSE contaminata e, in questo caso, preparare una nuova soluzione colorante CFSE.
Questo sistema ha alcune limitazioni intrinseche. In primo luogo, si tratta di una monocoltura bidimensionale che, sebbene utile per studi preliminari ad alto rendimento 6,7,8 e paragonabile ai modelli di interazione attualmente disponibili, non raggiunge il mimetismo ideale del tessuto tridimensionale in vivo 19. Inoltre, l'interazione fisica è ottimizzata in questo modello di co-coltura, ma l'interazione attraverso fattori solubili non è completamente eliminata a causa della natura statica di questo sistema. I terreni statici possono essere un vantaggio in quanto l'analisi dei fattori solubili rilasciati può essere utile per comprendere la risposta cellulare20. La comprensione dell'interazione puramente fisica richiede un'interazione ridotta al minimo attraverso fattori solubili, che può essere ottenuta attraverso la sostituzione frequente dei terreni e il mantenimento di un grande volume di terreni per la diffusione dei fattori.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto, in tutto o in parte, dalle sovvenzioni NIH 1R01AA027189-01A1 (a S.K.), P20 GM104320 (al Nebraska Center for the Prevention of Obesity Diseases Pilot Grant a S.K.), P20 GM113126 (al Nebraska Center for Integrated Biomolecular Communication-Project Leader S.K.); UNL Office of Research and Development Biomedical Seed Grant e Nebraska Research Initiative-Systems Grant (a S.K.). K.M.S. è stato finanziato attraverso T32GM107001, una borsa di studio.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Fisher Scientific | 25200056 | |
| 15 ml Nunc Conical Sterile Polypropylene Centrifuge Tubes | Fisher Scientific | 12-565-268 | |
| 5(6)-Carboxyfluorescein diacetate N-succinimidyl ester | Millipore Sigma | Cat#21888 | |
| 50 ml Nunc Conical Sterile Polypropylene Centrifuge Tubes | Fisher Scientific | 12-565-270 | |
| A172-MG GBM cell line | ATCC | CRL-1620 | |
| Bright-Line Hemacytometer | Sigma | Z359629 | Or other suitable cell counting device |
| Cell Incubator | N/A | N/A | |
| Cooled tabletop centrifuge for 15 mL tubes | N/A | N/A | |
| Dulbecco's Modified Eagle Medium (DMEM) | MP Biomedicals | ICN 1033120 | |
| Expanded Plasma Cleaner | Plasma Harrick | PDC-001-HP | With attached pressurized oxygen tank and PlasmaFlo Gas Mixer (PDC-FMG) accessory |
| Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11550H | |
| Fluorosilane | Sigma Aldrich | 667420 | Full chemical name: 1H,1H,2H,2H-Perfluorooctyltriethoxysilane |
| Inverted Tabletop Microscope | N/A | N/A | Microscope capable of fluorescent imaging with λex = 551 nm; λem 567 nm [e.g. Rhodamine filter] (PKH26 dye) and λex 492 nm; λem 517 nm [e.g. interference blue filter (IB)] (CFSE dye) |
| NaCl | Sigma Aldrich | S7653 | |
| NaOH | Sigma Aldrich | 567530 | |
| Penicillin-Streptomicen | Fisher Scientific | 15140122 | |
| PKH26 Red Fluorescent Cell Linker Mini Kit | Millipore Sigma | Cat#MINI26-1KT | |
| Poly(diallyldimethylammonium chloride) solution (PDAC) | Sigma Aldrich | 409014 | 20 wt. % in H2O |
| Poly(sodium 4-styrenesulfonate) (SPS) | Sigma Aldrich | 243051 | average MW ~70,000 |
| Primary Astrocytes, isolated from Srague Dawley rats | Charles River | Crl:SD | Rats from Charles River; Lab isolated Cells |
| Scalpel | N/A | N/A | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Chemical | Cat#2646340 | |
| Trypan Blue Stain | Fisher Scientific | 15-250-061 | |
| TryplE | Fisher Scientific | Gibco TrypLE | |
| U87-MG GBM cell line | ATCC | HTB-14 | |
| Vacuum Desiccator | N/A | N/A |
Riferimenti
- Ostrom, Q. T., et al. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2012-2016. Neuro Oncology. 21, 1 (2019).
- Ostrom, Q. T., et al. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2010-2014. Neuro Oncology. 19, 1-88 (2017).
- Roos, A., Ding, Z., Loftus, J. C., Tran, N. L. Molecular and microenvironmental determinants of glioma stem-like cell survival and invasion. Frontiers in Oncology. 7, 120 (2017).
- Rath, B. H., Fair, J. M., Jamal, M., Camphausen, K., Tofilon, P. J. Astrocytes enhance the invasion potential of glioblastoma stem-like cells. PLoS One. 8 (1), 54752 (2013).
- Le, D. M., et al. Exploitation of astrocytes by glioma cells to facilitate invasiveness: a mechanism involving matrix metalloproteinase-2 and the urokinase-type plasminogen activator-plasmin cascade. The Journal of Neuroscience. 23 (10), 4034-4043 (2003).
- Daverey, A., Brown, K. M., Kidambi, S. Breast cancer/stromal cells coculture on polyelectrolyte films emulates tumor stages and miRNA profiles of clinical samples. Langmuir. 31 (36), 9991-10001 (2015).
- Kidambi, S., Lee, I., Chan, C. Primary neuron/astrocyte co-culture on polyelectrolyte multilayer films: A template for studying astrocyte-mediated oxidative stress in neurons. Advanced Functional Materials. 18 (2), 294-301 (2008).
- Kidambi, S., et al. Patterned co-culture of primary hepatocytes and fibroblasts using polyelectrolyte multilayer templates. Macromolecular Bioscience. 7 (3), 344-353 (2007).
- Kidambi, S., Lee, I., Chan, C. Controlling primary hepatocyte adhesion and spreading on protein-free polyelectrolyte multilayer films. Journal of the American Chemical Society. 126 (50), 16286-16287 (2004).
- Mohammed, J. S., DeCoster, M. A., McShane, M. J. Micropatterning of nanoengineered surfaces to study neuronal cell attachment in vitro. Biomacromolecules. 5 (5), 1745-1755 (2004).
- Beaudoin, G. M., et al. Culturing pyramidal neurons from the early postnatal mouse hippocampus and cortex. Nature Protocols. 7 (9), 1741-1754 (2012).
- Wilson, C. L., Hayward, S. L., Kidambi, S. Astrogliosis in a dish: substrate stiffness induces astrogliosis in primary rat astrocytes. Rsc Advances. 6 (41), 34447-34457 (2016).
- Bogdanowicz, D. R., Lu, H. H. Studying cell-cell communication in co-culture. Biotechnololgy Journal. 8 (4), 395-396 (2013).
- Chen, W., et al. Glioma cells escaped from cytotoxicity of temozolomide and vincristine by communicating with human astrocytes. Medical Oncology. 32 (3), 43 (2015).
- Belot, N., et al. Molecular characterization of cell substratum attachments in human glial tumors relates to prognostic features. Glia. 36 (3), 375-390 (2001).
- Sin, W. C., et al. Astrocytes promote glioma invasion via the gap junction protein connexin43. Oncogene. 35 (12), 1504-1516 (2016).
- Sin, W. C., Crespin, S., Mesnil, M. Opposing roles of connexin43 in glioma progression. Biochimica et Biophysica Acta. 1818 (8), 2058-2067 (2012).
- Oliveira, R., et al. Contribution of gap junctional communication between tumor cells and astroglia to the invasion of the brain parenchyma by human glioblastomas. BMC Cell Biology. 6 (1), 7 (2005).
- Duval, K., et al. Modeling physiological events in 2D vs. 3D cell culture. Physiology (Bethesda). 32 (4), 266-277 (2017).
- Rape, A., Ananthanarayanan, B., Kumar, S. Engineering strategies to mimic the glioblastoma microenvironment). Advanced Drug Delivery Reviews. 79-80, 172-183 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon