Method Article
Perfil do Repertório de Receptores-Ligantes de Células Natural Killer Humanas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, projetamos dois painéis complementares de citometria de massa (CyTOF) e otimizamos um protocolo de coloração CyTOF com o objetivo de traçar o perfil do receptor de células assassinas naturais e do repertório de ligantes no cenário de infecções virais.
Resumo
As células natural killer (NK) estão entre as primeiras a responder a infecções virais. A capacidade das células NK de reconhecer e matar rapidamente células infectadas por vírus é regulada por sua expressão de receptores inibitórios e ativadores codificados pela linha germinativa. O envolvimento desses receptores por seus ligantes cognatos nas células-alvo determina se a interação intercelular resultará na morte das células NK. Este protocolo detalha o projeto e a otimização de dois painéis complementares de citometria de massa (CyTOF). Um painel foi projetado para fenotipar células NK com base na expressão do receptor. O outro painel foi projetado para interrogar a expressão de ligantes conhecidos para receptores de células NK em vários subconjuntos de células imunes. Juntos, esses dois painéis permitem o perfil do repertório de receptores-ligantes de células NK humanas. Além disso, este protocolo também detalha o processo pelo qual coramos amostras para CyTOF. Este processo foi otimizado para melhorar a reprodutibilidade e padronização. Uma vantagem do CyTOF é sua capacidade de medir mais de 40 marcadores em cada painel, com sobreposição mínima de sinal, permitindo que os pesquisadores capturem a amplitude do repertório de receptores-ligantes de células NK. O código de barras de paládio também reduz a variação entre amostras, bem como o consumo de reagentes, facilitando a coloração de amostras com cada painel em paralelo. As limitações deste protocolo incluem o rendimento relativamente baixo do CyTOF e a incapacidade de recuperar células após a análise. Esses painéis foram projetados para a análise de amostras clínicas de pacientes que sofrem de infecções virais agudas e crônicas, incluindo o vírus da dengue, o vírus da imunodeficiência humana (HIV) e a gripe. No entanto, eles podem ser utilizados em qualquer ambiente para investigar o repertório de ligante do receptor de células NK humanas. É importante ressaltar que esses métodos podem ser aplicados amplamente ao projeto e execução de futuros painéis CyTOF.
Introdução
As células natural killer (NK) são células imunes inatas cujo papel principal é atingir e matar células malignas, infectadas ou estressadas. Por meio de sua secreção de citocinas como IFNγ e TNFα, bem como sua atividade citotóxica, as células NK também podem moldar a resposta imune adaptativa a patógenos e malignidades. A resposta NK é mediada em parte pela sinalização combinatória de receptores inibitórios e ativadores codificados na linha germinativa, que se ligam a uma miríade de ligantes expressos em células-alvo potenciais. Vários receptores de células NK têm mais de um ligante, com novos pares receptor-ligante sendo identificados regularmente.
Há um interesse particular em estudar as células NK no contexto de infecções virais, onde sua capacidade de responder rapidamente a células estressadas pode limitar a disseminação viral ou promover o desenvolvimento de estratégias de evasão de células NK. Esse interesse na biologia celular NK se estende ao campo da imunoterapia contra o câncer, onde os pesquisadores estão investigando o papel das células NK na imunovigilância tumoral e no microambiente tumoral1. No entanto, a capacidade de traçar o perfil das interações célula-célula-alvo NK é complicada pelo fato de que as células NK humanas podem expressar mais de 30 receptores que, por sua vez, podem interagir com mais de 30 ligantes conhecidos2. A detecção simultânea de múltiplos receptores de células NK e seus ligantes cognatos é, portanto, necessária para capturar a complexidade das interações receptor-ligante que controlam a função NK. Consequentemente, recorremos à citometria de massa (CyTOF), que permite a detecção simultânea de mais de 40 marcadores no nível de uma única célula. Nosso objetivo era criar dois painéis CyTOF para traçar o perfil do repertório de ligantes receptores de células NK. Também queríamos projetar um protocolo para processamento e coloração eficazes de amostras clínicas. Amostras clínicas humanas fornecem uma riqueza de informações sobre como o corpo responde à infecção viral. Portanto, desenvolvemos este protocolo para investigar a expressão de receptores de células NK e seus ligantes cognatos em paralelo para melhor padronização, melhor recuperação, consumo reduzido de reagentes e efeitos de lote limitados.
Vários painéis de citometria de fluxo projetados para caracterizar o fenótipo de células NK humanas foram publicados anteriormente 3,4,5,6,7,8. A maioria desses painéis é limitada em sua capacidade de capturar a amplitude do repertório receptor-ligante, permitindo apenas a detecção de uma seleção limitada de marcadores. Além disso, esses painéis são limitados pela sobreposição de sinal entre fluorocromos. O CyTOF usa anticorpos conjugados a isótopos metálicos, que são lidos por espectrometria de massa por tempo de voo, reduzindo drasticamente o transbordamento entre os canais.
Como nós, outros pesquisadores recorreram ao CyTOF para estudar as células NK 9,10,11,12,13,14, embora geralmente com menos marcadores de células NK, o que reduz a profundidade da fenotipagem. Embora os protocolos gerais de coloração usados por esses grupos sejam semelhantes aos nossos, existem algumas diferenças importantes. Outros protocolos não envolvem o isolamento de células NK antes da coloração, embora os pesquisadores estejam interessados apenas nesse subconjunto13,14. Dado que as células NK representam apenas 5-20% das células mononucleares do sangue periférico (PBMCs), a coloração de PBMCs inteiras em vez de células NK isoladas significa que a maioria dos eventos coletados não serão células NK. Isso reduz a quantidade de dados gerados no subconjunto de interesse e resulta em uso ineficiente do tempo da máquina. Além disso, embora muitos desses painéis interroguem a expressão de receptores de células NK, como receptores assassinos semelhantes a Ig (KIRs), NKG2A/C/D e os receptores naturais de citotoxicidade (NKp30, NKp44 e NKp46), a expressão desses marcadores não é colocada em um contexto mais amplo devido à ausência de dados sobre a expressão de seus respectivos ligantes. Consequentemente, embora esses métodos publicados anteriormente para investigar células NK via CyTOF sejam suficientes para a ampla fenotipagem de células NK, usados isoladamente, eles não podem fornecer uma imagem abrangente da atividade das células NK. Isso nos leva à principal vantagem dos métodos descritos aqui, que é que até este ponto não há citometria de fluxo publicada ou painéis CyTOF focados em explorar a expressão de ligantes para receptores de células NK. É importante ressaltar que nosso painel de ligantes possui vários canais abertos para permitir a adição de marcadores para atender às necessidades exclusivas de cada experimento.
Considerando que uma das principais limitações do CyTOF é a incapacidade de recuperar a amostra após a análise, esse método pode não ser apropriado para pesquisadores que possuem amostras limitadas com as quais estão interessados em realizar experimentos adicionais. Além disso, a natureza de baixa taxa de transferência do CyTOF significa que os dados gerados serão de baixa qualidade se o número inicial de células for baixo. Exceto por essas duas limitações, este método terá um bom desempenho em qualquer ambiente para investigar as interações receptor-ligante entre células NK e células-alvo.
Protocolo
PBMCs adultos saudáveis anonimizados foram obtidos de câmaras do sistema de leucorredução adquiridas do Stanford Blood Center. PBMCs de doadores pediátricos saudáveis não identificados e pacientes pediátricos com dengue aguda foram obtidos do Instituto Memorial de Estudos de Saúde Gorgas na Cidade do Panamá, Panamá e hospitais pertencentes ao Ministério da Saúde, ao Sistema de Previdência Social da Cidade do Panamá e áreas suburbanas. O protocolo do estudo da dengue foi aprovado pelo CEP do Hospital del Niño (CBIHN-M-0634), depois aprovado pelos comitês do ICGES, CSS, Hospital Santo Tomas e Universidade de Stanford. PBMCs de pacientes infectados pelo HIV em tratamento antirretroviral foram obtidos do estudo ACTG A5321.
1. Rotulagem de anticorpos, preparação do painel e armazenamento
- Marcação de anticorpos com isótopos metálicos
NOTA: Para aumentar a padronização interexperimental da coloração, recomenda-se realizar várias conjugações para cada anticorpo e, em seguida, combinar os produtos em uma única mistura principal para armazenamento de longo prazo, conforme descrito abaixo.- Determinar a concentração de cada anticorpo medindo a absorvância a 280 nm antes da conjugação. Os anticorpos usados para este protocolo estão disponíveis comercialmente e foram adquiridos dos fornecedores listados na Tabela de Materiais.
- Rotule anticorpos com isótopos metálicos usando kits de rotulagem de anticorpos disponíveis comercialmente de acordo com as instruções do fabricante. Use 100 μg de anticorpo para cada reação.
- Determinar a concentração final do anticorpo recuperado medindo a absorvância a 280 nm. Armazene anticorpos para curto prazo a 4 °C.
- Titulações de anticorpos
NOTA: A tecnologia CyTOF é muito sensível a possíveis sinais de contaminação de metais ambientais. Portanto, todos os tampões/reagentes utilizados devem ser preparados com água ultrapura e armazenados em recipientes de plástico ou vidro que nunca tenham sido lavados com sabão.- Prepare tubos de centrífuga para cada doador contendo 9 mL de RPMI quente e completo (RPMI 1640, 10% FBS, 1% de L-glutamina, 1% de penicilina / estreptomicina) e 20 μL de benzonase por frasco de PBMCs a serem descongelados. Descongele os PBMCs em banho-maria e adicione aos tubos.
NOTA: A benzonase diminui a viscosidade e o fundo do DNA livre das células lisadas. - Centrifugue a 300 x g em temperatura ambiente por 5 min. Ressuspenda os PBMCs em 5 mL de mídia RPMI completa e conte.
- Para cada titulação do painel, coloque 2-4 milhões de PBMCs/poço em 6 poços de uma placa de fundo redondo de 96 poços (um poço para cada título e um para não corado). Centrifugue a placa a 600 x g à temperatura ambiente durante 3 min. Agite a placa para remover o sobrenadante. Ressuspenda cada poço em 200 μL de CyPBS.
- Realize a coloração de viabilidade da cisplatina conforme descrito abaixo.
NOTA: A cisplatina é usada para discriminar células vivas de mortas em citometria de massa.- Ressuspenda as células em 100 μL de estoque de cisplatina 25 μM. Incube em temperatura ambiente por 1 min.
- Extinguir a reação de cisplatina adicionando 100 μL de FBS a cada poço e misturando. Centrifugue e sacuda a placa.
NOTA: Execute todas as etapas subsequentes da centrífuga a 4 °C. - Lave as células duas vezes com 200 μL de CyFACS (1x PBS sem contaminantes de metais pesados em água ultrapura com 0,1% de BSA, 0,05% de azida de sódio). Centrifugue e agite a placa a cada vez.
- Titular o painel de anticorpos de superfície conforme descrito abaixo.
NOTA: Misturas master separadas devem ser feitas para o painel de superfície NK e o painel de ligantes.- Faça uma mistura principal de todos os anticorpos de superfície a uma concentração de 10 μg/mL usando CyFACS. Apontar para um volume final de 150 μL. Fazer diluições seriadas 1:2 usando CyFACS, para obter as seguintes concentrações: 10, 5, 2,5, 1,25 e 0,625 μg/mL.
- Filtre os coquetéis de anticorpos através de uma unidade de filtro centrífugo (tamanho de poro de 0,1 μm) a 10.600 x g por 3 min antes da coloração.
- Ressuspenda as células plaqueadas em 50 μL do coquetel de anticorpos de superfície no respectivo título. Ressuspenda o poço não corado no CyFACS. Incubar a 4 °C durante 30 min.
- Lave as células com 150 μL de CyFACS. Centrifugue e sacuda a placa.
- Lave as células com 200 μL de CyFACS. Centrifugue e sacuda a placa.
- Realize a fixação das células ressuspendendo cada poço em 100 μL de paraformaldeído (PFA) a 2% em CyPBS. Incube a placa em temperatura ambiente no escuro por 20 min. Lave as células com 100 μL de CyFACS. Centrifugue a 700 x g por 5 min.
CUIDADO: O PFA é suspeito de causar defeitos genéticos, bem como câncer. Além disso, é prejudicial se entrar em contato com os olhos, pele ou for inalado. Manuseie adequadamente, garantindo uma boa ventilação, abrindo o recipiente com cuidado e evitando a formação de aerossóis.
NOTA: Todas as centrifugações subsequentes da centrífuga são realizadas a 700 x g por 5 min a 4 °C. - Permeabilize as células ressuspendendo em 200 μL de tampão de permeabilização 1x (tampão Perm) diluído em água ultrapura. Centrifugue e sacuda a placa. Lave as células com 200 μL de tampão Perm. Centrifugue e sacuda a placa.
NOTA: A incubação no tampão Perm não é necessária. - Titulação do painel de anticorpos intracelulares
- Faça uma mistura principal de todos os anticorpos intracelulares a uma concentração de 10 μg/mL usando o Tampão Perm. Apontar para um volume final de 150 μL. Faça diluições seriadas 1:2 usando o Perm Buffer para obter as seguintes concentrações: 10, 5, 2,5, 1,25 e 0,625 μg/mL.
- Filtre os coquetéis de anticorpos através de uma unidade de filtro centrífugo (tamanho de poro de 0,1 μm) a 10.600 x g por 3 min antes da coloração.
- Ressuspenda as células plaqueadas em 50 μL do coquetel de anticorpos intracelulares no respectivo título. Ressuspenda o poço não corado no Perm Buffer. Incubar a 4 °C durante 45 min.
NOTA: Se um painel intracelular não for titulado, ressuspenda os poços em 50 μL do tampão Perm. - Lave as células com 150 μL de tampão Perm. Centrifugue e sacuda a placa.
- Lave as células com 200 μL de tampão Perm. Centrifugue e sacuda a placa.
- Lave as células duas vezes com 200 μL de CyFACS. Centrifugue e sacuda a placa.
- Coloração do intercalador de DNA
NOTA: O intercalador se liga ao ácido nucléico celular e é usado para identificar células nucleadas na citometria de massa.- Ressuspender as células em 200 μL de intercalador diluído 1:10.000 em CyPBS e 2% de PFA. Incubar a placa durante a noite a 4 °C.
- Armazene as placas, se necessário, a 4 °C cobertas com filme de parafina por até uma semana.
NOTA: Execute todas as etapas subsequentes da centrífuga a 700 x g por 5 min a 4 °C. - Antes de executar as amostras em CyTOF, remova o filme de parafina da placa e centrifugue a 700 x g por 5 min a 4 °C. Sacuda o prato. Lave as células uma vez com 200 μL de CyFACS. Centrifugue e sacuda a placa.
- Lave as células três vezes com 200 μL de água ultrapura. Centrifugue e sacuda a placa. Ressuspenda as células em 200 μL de água ultrapura. Imediatamente antes de executar a amostra, ajuste a concentração para aproximadamente 6 x 105 células/mL em grânulos de normalização diluídos a uma concentração de 1x em água ultrapura.
- Execute os exemplos no CyTOF.
- Analise os dados e escolha os títulos apropriados para cada anticorpo, selecionando o título de anticorpo mais baixo, o que resulta na maior intensidade de sinal e na melhor separação entre populações positivas e negativas com base na avaliação visual.
NOTA: As titulações para os painéis NK e ligante são mostradas na Figura 1 e na Figura 2 , respectivamente. Se não for identificada uma distinção clara entre populações positivas e negativas, os títulos podem ser avaliados em vários tipos de células ou em linhagens celulares, para permitir a identificação de populações de células positivas e negativas. - Armazenamento do painel de anticorpos
- Combine anticorpos titulados em uma mistura principal e filtre através de uma unidade de filtro de seringa estéril de 0,1 μm. Misturas principais separadas devem ser feitas para o painel de superfície NK, o painel intracelular NK e o painel ligante.
- Para o armazenamento de painéis a longo prazo, siga uma das duas opções aceitáveis:
- Envie master mix para uma empresa terceirizada para liofilização. Este método é usado para o painel NK. Anticorpos não conjugados internamente não podem ser liofilizados, devido à presença de estabilizador de anticorpos, que interfere no processo de liofilização. Esses anticorpos são adicionados ao painel no dia da coloração.
- Use uma pipeta repetidora para fazer alíquotas de 50 μL de cada master mix e armazene-as a -80 °C.
- Prepare tubos de centrífuga para cada doador contendo 9 mL de RPMI quente e completo (RPMI 1640, 10% FBS, 1% de L-glutamina, 1% de penicilina / estreptomicina) e 20 μL de benzonase por frasco de PBMCs a serem descongelados. Descongele os PBMCs em banho-maria e adicione aos tubos.
2. Protocolo de coloração
- Descongelar as células mononucleares do sangue periférico (PBMCs) conforme descrito nas etapas 1.2.1 e 1.2.2. Separe pelo menos 1 milhão de PBMCs para coloração do painel de ligantes em um tubo de centrífuga de 15 mL. Mantenha esses PBMCs no gelo durante o isolamento da célula NK.
- Isolamento de células NK
NOTA: As etapas de isolamento de células NK a seguir são uma versão modificada do protocolo de um fornecedor específico. No entanto, qualquer kit ou protocolo que realize a seleção negativa de células NK com base magnética seria uma alternativa adequada. Além disso, esta etapa é opcional, pois este protocolo também é adequado para a fenotipagem de células NK de PBMCs inteiros.- Gire os PBMCs restantes a 450 x g por 5 min. Ressuspenda o pellet celular em 40 μL de tampão MACS (PBS, 0,5% BSA, 2 mM EDTA) por 107 células totais.
- Adicione 10 μL de coquetel de biotina-anticorpo de células NK por 107 células totais. Misture bem e incube por 5 min no gelo.
- Adicione 30 μL de tampão MACS por 107 células totais e 20 μL de célula NK MicroBead Cocktail por 107 células totais. Misture bem e incube por 10 min no gelo.
- Prepare as colunas de eluição enxaguando com 500 μL de tampão MACS. Adicione 2 mL de RPMI completo frio a tubos de coleta de 15 mL.
- Adicione tampão MACS aos tubos contendo células para aumentar o volume até 500 μL. Pipete todo o volume de 500 μL na coluna de eluição preparada. Enxágue o tubo com mais 500 μL de tampão MACS e transfira para a coluna.
- Após a interrupção do fluxo, enxágue a coluna de eluição com tampão MACS de 500 μL duas vezes. Depois que o fluxo for interrompido, conte as células NK.
- Placas e células de lavagem.
- Centrifugar células NK isoladas e PBMCs a 300 x g à temperatura ambiente por 10 min. Ressuspenda as células a uma concentração de 5 milhões de células/mL em CyPBS (1x PBS sem contaminantes de metais pesados em água ultrapura). Células de placa em uma placa de 96 poços com fundo em U.
NOTA: Cada alíquota do painel de células NK pode corar até 3 milhões de células. Se seis amostras individuais de células NK estiverem sendo codificadas com código de barras e agrupadas antes da coloração, o número total combinado de células NK não deve exceder 3 milhões. O painel de ligantes pode corar até 6 milhões de PBMCs por amostra. Se seis amostras individuais estiverem sendo codificadas com código de barras e agrupadas antes da coloração, o número total combinado de PBMCs não deve exceder 6 milhões. - Placa centrífuga a 600 x g à temperatura ambiente por 3 min. Agite a placa para remover o sobrenadante.
NOTA: Execute todas as centrifugações subsequentes da centrífuga a 600 x g por 3 min até a etapa 2.7. - Ressuspenda as células em 200 μL de CyPBS. Centrifugue e sacuda a placa.
- Centrifugar células NK isoladas e PBMCs a 300 x g à temperatura ambiente por 10 min. Ressuspenda as células a uma concentração de 5 milhões de células/mL em CyPBS (1x PBS sem contaminantes de metais pesados em água ultrapura). Células de placa em uma placa de 96 poços com fundo em U.
- Realize a coloração de viabilidade da cisplatina conforme descrito na etapa 1.2.4.
- Coloração de código de barras
NOTA: Esses painéis são usados em conjunto com um método de código de barras modificado baseado em paládio de dois de quatro em células vivas não fixadas para minimizar os efeitos do lote e maximizar a recuperação celular15. No entanto, esta etapa é opcional, pois o código de barras não é necessário para obter dados de qualidade.- Ressuspenda cada poço em 50 μL do respectivo código de barras pré-misturado e incube a 4 °C por 30 min. Lave as células com 150 μL de CyFACS. Centrifugue e sacuda a placa.
- Lave as células duas vezes com 200 μL de CyFACS. Centrifugue e sacuda a placa. Ressuspenda todos os poços em 30 μL de CyFACS. Combine até seis poços de células coradas com códigos de barras exclusivos em um poço e execute a centrifugação e o movimento da placa.
- Coloração de superfície
- Dissolva a liosfera do painel NK de superfície em 50 μL de CyFACS com anticorpos de superfície adicionais enriquecidos (anti-CD16, anti-HLA-DR, anti-LILRB1). Descongele o painel de ligantes armazenado a -80 °C e gire o tubo usando uma minicentrífuga. Pico em marcadores adicionais de superfície do painel de ligantes (anti-CD16, anti-CD19).
NOTA: Qualquer coquetel de anticorpos que não tenha sido filtrado anteriormente (ou seja, antes da liofilização ou congelamento) deve ser filtrado através de uma unidade de filtro centrífugo (tamanho de poro de 0,1 μm) a 10.600 x g por 3 min antes da coloração. - Ressuspenda cada poço em 50 μL do respectivo painel. Incubar a 4 °C durante 30 min. Lave as células com 150 μL de CyFACS. Centrifugue e sacuda a placa. Lave as células novamente com 200 μL de CyFACS. Centrifugue e sacuda a placa.
- Dissolva a liosfera do painel NK de superfície em 50 μL de CyFACS com anticorpos de superfície adicionais enriquecidos (anti-CD16, anti-HLA-DR, anti-LILRB1). Descongele o painel de ligantes armazenado a -80 °C e gire o tubo usando uma minicentrífuga. Pico em marcadores adicionais de superfície do painel de ligantes (anti-CD16, anti-CD19).
- Corrija as células conforme descrito na etapa 1.2.6.
NOTA: Execute todas as centrifugações subsequentes da centrífuga a 700 x g por 5 min a 4 °C. - Permeabilize as células conforme descrito na etapa 1.2.7.
- Coloração intracelular
- Dissolva a liosfera do painel NK intracelular em 50 μL de tampão Perm. Prepare um coquetel de anticorpos intracelulares para amostras de PBMC, se desejar.
NOTA: Qualquer coquetel de anticorpos que não tenha sido filtrado anteriormente (ou seja, antes da liofilização ou congelamento) deve ser filtrado através de uma unidade de filtro centrífugo (tamanho de poro de 0,1 μm) a 10.600 x g por 3 min antes da coloração. - Ressuspender os poços em 50 μL dos respectivos painéis intracelulares. Se um painel intracelular não for usado em conjunto com o painel de superfície do ligante, ressuspenda os poços PBMC em 50 μL do tampão Perm. Incubar a 4 °C durante 45 min.
- Lave as células com 150 μL de tampão Perm. Centrifugue e mova a placa. Lave as células com 200 μL de tampão Perm. Centrifugue e sacuda a placa.
- Lave as células duas vezes com 200 μL de CyFACS. Centrifugue e sacuda a placa.
- Dissolva a liosfera do painel NK intracelular em 50 μL de tampão Perm. Prepare um coquetel de anticorpos intracelulares para amostras de PBMC, se desejar.
- Coloração intercaladora de DNA. Realize a coloração do intercalador de DNA conforme descrito na etapa 1.2.9.1. Incubar a placa durante a noite a 4 °C.
NOTA: O intercalador se liga ao ácido nucléico celular e é usado para identificar células nucleadas na citometria de massa. As placas podem ser armazenadas cobertas com filme de parafina por até uma semana a 4 °C. - Antes de executar as amostras no CyTOF, lave as células conforme descrito nas etapas 1.2.9.3 e 1.2.9.4.
- Execute amostras no CyTOF.
Resultados
Os anticorpos foram conjugados a isótopos metálicos usando kits de rotulagem disponíveis comercialmente, de acordo com as instruções do fabricante. Os clones de anticorpos foram validados por citometria de fluxo e citometria de massa antes do uso neste painel. Uma lista inicial de clones foi selecionada com base na revisão da literatura e na disponibilidade de anticorpos. Os níveis de expressão de alguns ligantes para receptores de células NK são baixos ou indetectáveis em PBMCs saudáveis. Portanto, a coloração positiva para alguns anticorpos foi validada pela coloração de PBMCs saudáveis, células K562 de leucemia mielóide crônica, células NALM6 de leucemia linfoblástica aguda ou células B de leucemia linfoblástica aguda 697 (Figura Suplementar 1). Os clones selecionados para o painel de células NK que não produziram uma coloração adequada ou eram muito caros foram substituídos por diferentes, conforme detalhado na Tabela Suplementar 1 e mostrado na Figura Suplementar 2.
O emparelhamento metal-isótopo com anticorpos para esses painéis foi realizado usando os princípios descritos por Takahashi et al.16. Os marcadores de linhagem foram de média a alta intensidade. Consequentemente, eles foram conjugados principalmente a massas de baixa e média sensibilidade, deixando massas de alta sensibilidade disponíveis para conjugação a anticorpos contra marcadores mais fracamente expressos. Um software de design de painel disponível publicamente foi usado para detectar problemas de sensibilidade à abundância (sangramento M ± 1) ou oxidação (sangramento M + 16) e os pares anticorpo-metal foram reatribuídos de acordo. Além disso, vários marcadores foram conjugados em diferentes metais com diferenças mínimas observadas nas intensidades de sinal (Tabela Suplementar 2 e Figura Suplementar 3). Os pares anticorpo-metal e as informações de clone para os painéis NK e ligante estão listados na Tabela 1 e na Tabela 2 , respectivamente.
Os anticorpos conjugados in house foram titulados em PBMCs em cinco títulos diferentes: 0,625, 1,25, 2,5, 5 e 10 μg/mL. O menor título de anticorpos que resultou na maior intensidade de sinal e na melhor separação entre populações positivas e negativas foi selecionado com base na avaliação visual. As titulações para os painéis NK e ligante são mostradas na Figura 1 e na Figura 2, respectivamente. Para certos marcadores, não foi identificada uma distinção clara entre populações positivas e negativas, devido ao fato de o marcador ser fracamente expresso ou universalmente positivo. Para determinar a diluição de trabalho mais precisa para esses anticorpos, os títulos foram avaliados em vários tipos de células (PBMCs, células T, células B ou células NK) ou em linhagens celulares, para permitir a identificação de populações de células positivas e negativas (Figura Suplementar 4). O índice de coloração (IS) para cada marcador não foi calculado, pois essa métrica não é aplicável aos dados do CyTOF17,18.
Os painéis descritos aqui foram projetados para serem compatíveis com o código de barras de amostra. Existem vários métodos de código de barras disponíveis para CyTOF. Os mais comumente usados são um kit à base de Paládio disponível comercialmente, que requer fixação antes do código de barras, e o método de código de barras baseado em CD45 descrito por Mei et al.15, que permite o código de barras de células vivas. Para avaliar qual método de código de barras melhor atende às nossas necessidades, testamos a estabilidade da coloração do marcador de células NK após a fixação em uma versão inicial do painel de células NK (Figura Suplementar 5). Descobrimos que a expressão da maioria dos marcadores de células NK foi afetada pela fixação. Consequentemente, decidimos usar um método de código de barras modificado baseado em CD45 em células vivas15. Este método de código de barras usa 102Pd, 104Pd, 106Pd e 108Pd e difere do método de três de seis originalmente descrito por Mei et al., que usava 104Pd, 106Pd, 108Pd, 110Pd, 113In e 115In. Os canais de índio não foram incluídos em nosso esquema de código de barras, pois interferiam no sinal do 115In-CD3. 110Pd não foi incluído, pois interferiu no sinal do HLA-DR Qdot e do CD19 Qdot na célula NK e nos painéis ligantes, respectivamente.
Embora recomendemos a purificação das células NK antes da coloração, o painel de células NK foi projetado para permitir a fenotipagem de células NK de PBMCs inteiros. Um exemplo de nossa estratégia de gating de células NK é mostrado na Figura 3A usando PBMCs de um doador saudável. A coloração e os portões para cada um dos marcadores NK são mostrados em células NK saudáveis e isoladas na Figura 3B. O painel de ligantes é projetado para detectar a expressão de ligantes de células NK em PBMCs inteiros. A Figura 4A ilustra a estratégia de gating usada para identificar células T CD4+ , células T CD8+ , células NK, monócitos e células B CD19+ em PBMCs de um doador saudável. Exemplos de coloração representativos para cada ligante são mostrados na Figura 4B usando PBMCs de pacientes com dengue aguda e indivíduos infectados pelo HIV que foram virologicamente suprimidos.
Para garantir a estabilidade do painel ao longo do tempo, nosso protocolo inclui duas opções possíveis: liofilização por meio de uma empresa terceirizada em esferas de uso único ou congelamento de alíquotas pré-fabricadas a -80 °C. Para este protocolo, o painel NK foi liofilizado e o painel ligante foi congelado. Ambos os métodos foram validados antes do uso de cada painel em amostras clínicas.
Produzimos mais de 700 reações do painel NK a partir de uma mistura master, realizando várias conjugações de cada anticorpo no painel. Após a validação e titulação de cada anticorpo conjugado, os anticorpos foram combinados em uma mistura master, filtrados através de uma unidade de filtro de seringa estéril de 0,1 μm e enviados a uma empresa terceirizada para liofilização. Dois conjuntos de liosferas de coloração única foram feitos, um para coloração superficial e outro para coloração intracelular. Anticorpos não conjugados internamente (HLA-DR e CD16) não puderam ser adicionados à liosfera, devido à presença de estabilizador de anticorpos, que interfere no processo de liofilização. Esses anticorpos são adicionados ao painel no dia da coloração. Uma comparação entre as colorações obtidas pré e pós-liofilização é mostrada na Figura Suplementar 6. O clone do anticorpo LILRB1 inicialmente usado nas liosferas não produziu uma coloração suficientemente forte (Tabela Suplementar 1 e Figura Suplementar 2). Posteriormente, um clone melhor foi identificado, conjugado e adicionado ao painel no dia da coloração (Tabela 1). Observou-se que o anticorpo policlonal KIR2DS2 usado nas lioferas produz uma coloração inespecífica após a liofilização e não recomendamos seu uso para análises subsequentes (Tabela Suplementar 1 e Figura Suplementar 2). A maioria das colorações intracelulares aumentou ligeiramente de intensidade após a liofilização (Figura Suplementar 6).
Antes do armazenamento da mistura mestre do painel de ligante a -80 °C, testamos duas condições de armazenamento diferentes. Preparamos uma mistura master menor deste painel e armazenamos alíquotas a -80 °C e em nitrogênio líquido por aproximadamente dois meses. Após dois meses, coramos PBMCs inteiros com as alíquotas congeladas. Comparamos a coloração com a de PBMCs do mesmo doador corado com o painel recém-preparado (Figura Suplementar 7). Encontramos armazenamento a -80 °C e em nitrogênio líquido não altera a intensidade do sinal para a maioria dos marcadores. De fato, a intensidade do sinal de anti-pan HLA classe I, anti-CD7, anti-CD4, anti-HLA-Bw4, anti-CD14, anti-CD11b e anti-LFA-3 é maior após o congelamento, particularmente no caso de amostras armazenadas a -80 °C. Não foi possível determinar se as intensidades de sinal de anti-LLT-1, anti-Nectina-1, anti-MICA/B, anti-DR4/5, anti-ULBP-1,2,5,6, anti-Nectina-2, anti-CD155 e anti-B7-H6 foram afetadas pelo congelamento, devido ao fato de que PBMCs saudáveis não expressam altos níveis desses marcadores. No entanto, a validação desses marcadores em linhagens celulares (Figura Suplementar 1) foi realizada usando anticorpos conjugados armazenados a -80 °C. Consequentemente, estávamos confiantes de que o congelamento não resultou em uma perda significativa de sinal. A intensidade do sinal diminuiu após o congelamento para cinco marcadores: anti-CD8, anti-ICAM-1, anti-CCR2, anti-CD33 e anti-CD56. No entanto, em todos esses casos, a separação clara entre as populações positivas e negativas permaneceu. Dado que os anticorpos conjugados com metal não são estáveis a 4 ° C por longos períodos de tempo, o congelamento foi necessário para preservar a estabilidade do painel a longo prazo e, apesar de uma diminuição na intensidade da coloração em um subconjunto de marcadores, fomos capazes de manter a separação de coloração suficiente. É importante ressaltar que a perda de intensidade de sinal de anti-CD8, anti-CCR2 e anti-CD56 foi maior nas amostras armazenadas em nitrogênio líquido em comparação com aquelas armazenadas a -80 °C. Com base nesses dados, decidimos armazenar o painel a -80 °C.
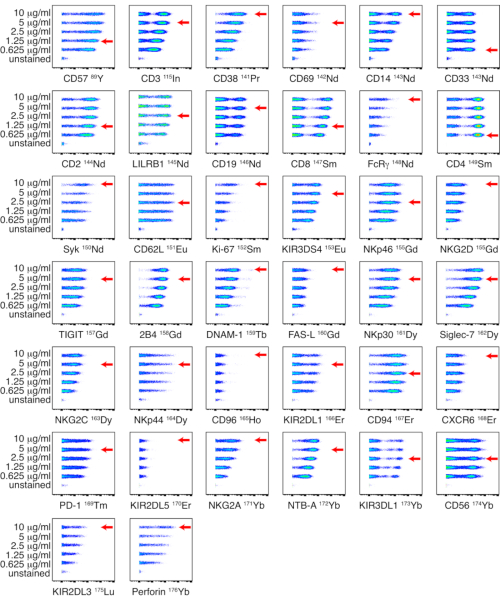
Figura 1: Titulação de conjugados internos de anticorpo-metal conjugados para painel NK. Titulações de anticorpos conjugados in house foram realizadas em PBMCs de um doador saudável usando cinco concentrações diferentes: 0,625, 1,25, 2,5, 5 e 10 μg/mL. Os títulos de anti-CD3, anti-CD14, anti-CD33, anti-CD19, anti-PD-1 e anti-CD56 foram determinados por gating em células vivas. Os títulos de anti-CD4 e anti-CD8 foram determinados por gating em células T. Os títulos para os anticorpos restantes foram determinados por gating em células NK. Como o NKp44 não é expresso em células NK em repouso, os títulos foram determinados em PBMCs estimuladas com IL-2 e mostradas em células NK. As setas vermelhas indicam o título selecionado para cada anticorpo. Clique aqui para ver uma versão maior desta figura.

Figura 2: Titulação de anticorpos de painel de ligante conjugado internos. Titulações de anticorpos conjugados in house foram realizadas em PBMCs de um doador saudável usando cinco concentrações diferentes: 0,625, 1,25, 2,5, 5 e 10 μg/mL. Os títulos de anti-HLA-DR, anti-ICAM-1, anti-CCR2, anti-CD14, anti-CD11b e anti-LFA-3 foram determinados por gating em células CD3-CD7-. Os títulos de anti-CD3, anti-pan HLA classe I, anti-CD7, anti-CD48, anti-LLT-1, anti-HLA-C,E, anti-HLA-E, anti-FasR, anti-Nectina-1, anti-MICA/MICB, anti-DR4/DR5, anti-ULBP-1,2,5,6, anti-Nectina-2, anti-CD155, anti-HLA-Bw4, anti-HLA-Bw6, anti-CD33, anti-CD56 e anti-B7-H6 foram determinados por gating em células vivas. Os títulos de anti-CD4 e anti-CD8 foram determinados por gating em células CD3+. As setas vermelhas indicam o título selecionado para cada anticorpo. Clique aqui para ver uma versão maior desta figura.
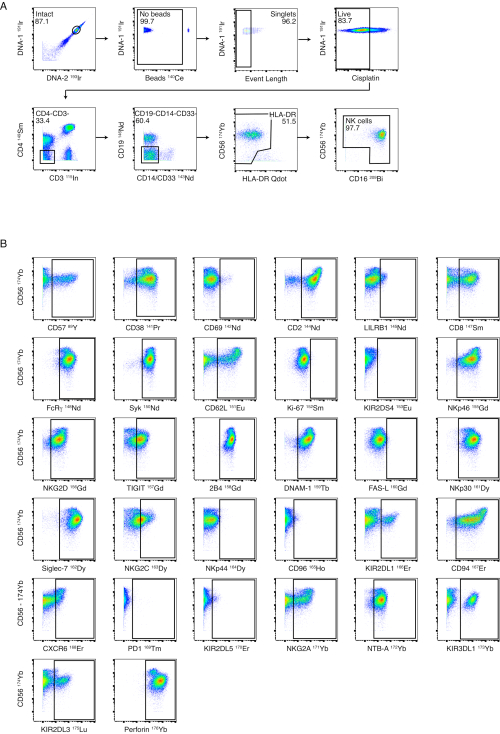
Figura 3: Estratégia e desempenho do painel NK. (A) O gating negativo serial de PBMCs inteiros para células NK é mostrado em um doador saudável. Portões intactos, de cordão e de comprimento de evento garantem um gating bem-sucedido para células individuais. A coloração com cisplatina foi realizada como uma coloração viva/morta. Células T e células B foram excluídas usando CD3 e CD19. Os monócitos foram excluídos por gating negativo em CD4 e CD14 / CD33 e por gating negativo adicional de célulasbrilhantes CD56- / HLA-DR. CD56 e CD16 foram usados para identificar diferentes subconjuntos de células NK (CD56brilhante, CD56escuro e CD56-). (B) Exemplos de expressão de receptores de células NK em células NK de um doador saudável purificado por isolamento de esferas magnéticas. Clique aqui para ver uma versão maior desta figura.

Figura 4: Bloqueio e desempenho do painel de ligantes. (A) Bloqueio dos principais subconjuntos de células imunes de PBMCs derivados de um doador saudável após normalização, remoção de esferas de calibração e debarcoding. (B) Expressão de ligantes para receptores de células NK, bem como vários marcadores mieloides em PBMCs vivos. A coloração para todos os ligantes, exceto Nectina-1 e B7-H6, é mostrada em PBMCs de pacientes com dengue aguda. A coloração para Nectina-1 e B7-H6 é mostrada em PBMCs de indivíduos infectados pelo HIV que foram virologicamente suprimidos. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Verificação de clones de anticorpos para painel de ligantes em linhagens celulares. Anticorpos contra ligantes para receptores de células NK que são expressos em níveis baixos em PBMCs saudáveis foram validados por coloração de linhagens celulares. As células K562 da leucemia mieloide crônica foram coradas com anti-ICAM-1, anti-MICA/MICB, anti-DR4/DR5, anti-ULBP-1,2,5,6, anti-Nectina-2, anti-CD155 e anti-B7-H6. As células NALM6 da leucemia linfoblástica aguda foram coradas com anti-LLT-1 e as células da leucemia linfoblástica aguda 697 foram coradas com anti-Nectina-1. Gráficos de pontos e histogramas mostrando coloração em PBMCs saudáveis estão em azul. Os gráficos de pontos e histogramas que mostram a coloração na respectiva linha celular estão em vermelho. A porcentagem de células da respectiva linha celular que são positivas para um determinado marcador é fornecida. Clique aqui para baixar este arquivo.
Figura suplementar 2: Validação de clones de anticorpos. (A) Diferentes clones de anticorpos foram testados em doadores saudáveis para identificar o clone com a melhor especificidade. 2B4, CXCR6, KIR2DS4, NKG2A e TIGIT são mostrados em células NK. CD56 e LILRB1 são mostrados em células vivas. (B) O clone de anticorpo KIR2DS2 apresentou coloração inespecífica após liofilização. Um exemplo de coloração no mesmo doador é fornecido antes e depois da liofilização. Clique aqui para baixar este arquivo.
Figura suplementar 3: Otimização de pares de anticorpo/metal. A coloração de PBMCs de doadores saudáveis é mostrada. (A) Pares de anticorpos/metais testados para o painel. (B) Pares de anticorpos/metais usados no painel. LILRB1 e PD1 são mostrados em células vivas. Todos os outros marcadores são mostrados nas células NK. Clique aqui para baixar este arquivo.
Figura suplementar 4: Titulação de marcadores de células NK fracamente expressos e principalmente positivos. Os títulos de anticorpos contra marcadores de células NK que não mostraram uma população positiva e negativa clara foram avaliados tanto em células NK (vermelho) quanto em células B (azul) ou PBMCs (cinza) de doadores saudáveis. As setas indicam o título selecionado para cada anticorpo. Clique aqui para baixar este arquivo.
Figura suplementar 5: Otimização do protocolo de código de barras. A estabilidade do epítopo foi testada antes e após a fixação com paraformaldeído a 2% em PBMCs de um doador saudável. (A) A coloração de CD3, CD14 e CD56 foi semelhante antes (vermelho) e após (azul) da fixação, a coloração de CD4 e CD16 foi significativamente afetada pela fixação. (B) Muitos marcadores de células NK foram afetados pela fixação, incluindo CD2, CD38, KIR3DL2, CD62L, KIR2DS4, NKp46, NKG2C, NKp30, NKG2D, KIR3DL1, TIGIT, KIR2DL1, KIR2DL3 e NTB-A. Clique aqui para baixar este arquivo.
Figura suplementar 6: Confirmação da estabilidade do painel após liofilização. A estabilidade dos conjugados de anticorpos in house foi confirmada pela coloração de PBMCs do mesmo doador de banco de sangue pré-liofilização (azul) e pós-liofilização (vermelho). As colorações anti-CD3, anti-CD14, anti-CD33, anti-CD19, anti-PD-1, anti-CD56 são mostradas nas células vivas. Anti-CD4 e anti-CD8 são mostrados em células CD3+ . Os títulos para os anticorpos restantes são mostrados nas células NK, controladas de acordo com o esquema de gating mostrado na Figura 1. Notavelmente, o anti-KIR2DS2 corado de forma não específica após a liofilização e, portanto, não foi usado para análises subsequentes. Clique aqui para baixar este arquivo.
Figura suplementar 7: Confirmação da estabilidade do painel de ligantes a -80 °C. A estabilidade dos conjugados de anticorpos internos foi confirmada pela coloração de PBMCs saudáveis do mesmo doador com uma mistura master recém-preparada, bem como a mesma mistura após armazenamento a -80 ° C ou em nitrogênio líquido. A coloração anti-HLA-DR, anti-ICAM-1 e anti-LFA-3 é mostrada em células CD3-CD7-. A coloração anti-CD3, anti-pan HLA classe I, anti-CD7, anti-CD48, anti-LLT-1, anti-HLA-E, anti-FasR, anti-Nectina-1, anti-MICA/B, anti-DR4/5, anti-ULBP-1,2,5,6, anti-Nectina-2, anti-CD155, anti-HLA-Bw4, anti-HLA-Bw6 e anti-B7-H6 é mostrada em células vivas. A coloração anti-CD8 e anti-CD4 é mostrada em células CD3+. A coloração anti-HLA-C, E, anti-CCR2, anti-CD11b e anti-CD33 é mostrada em células CD3-CD7-CD14+. A coloração anti-CD14 é mostrada em células CD3-CD7-CD33+ e a coloração anti-CD56 é mostrada em células CD3-CD7+CD14-HLA-DR-. Os histogramas que mostram a coloração com o painel recém-preparado estão em vermelho. Os histogramas que mostram a coloração com o painel após o armazenamento a -80 ° C por aproximadamente dois meses estão em azul. Os histogramas que mostram a coloração com o painel após armazenamento em nitrogênio líquido (LN2) por aproximadamente dois meses estão em verde. As amostras foram coradas e executadas em dias diferentes. Os arquivos foram normalizados e as contas foram removidas usando o pacote premessa. Clique aqui para baixar este arquivo.
| Especificidade | Clone | Isótopo | Propósito | Superfície/Intracelular |
| CD57 | HCD57 | 89Y | Maturidade/Memória | Superfície |
| CD45 | HI30 | 102Pd, 104Pd, 106Pd, 108Pd | Código de barras | Superfície |
| HLA-DR | Tü36 | Qdot 655 (112 Cd-114Cd) | Ativação/Linhagem | Superfície |
| CD3 | UCHT | 115Em | Linhagem de células T | Superfície |
| CD38 | HIT2 | 141Pr | Marcador de ativação | Superfície |
| CD69 | FN50 | 142Nd | Marcador de ativação | Superfície |
| CD33 | WM53 | 143Nd | Linhagem de monócitos | Superfície |
| CD14 | M5E5 | 143Nd | Linhagem mieloide | Superfície |
| CD2 | RPA-2.10 | 144Nd | Ativação/Maturidade | Superfície |
| LILRB1 | MAB20172 | 145Nd | Receptor Inibitório | Superfície |
| CD19 | HIB19 | 146Nd | Linhagem de células B | Superfície |
| CD8 | SK1 | 147Sm | Linhagem de células T e ativação/maturidade de células NK | Superfície |
| FcRg | policlonal | 148Nd | Maturidade/Adaptativo | intracelular |
| CD4 | SK3 | 149Sm | Linhagem de células T | Superfície |
| Syk | 4D10.2 | 150Nd | Sinalização | intracelular |
| CD62L | DREG-56 | 151UE | Ativação | Superfície |
| KI-67 | Ki-67 | 152Sm | Proliferação | intracelular |
| KIR2DS4 | 179315 | 153UE | Ativando o Receptor | Superfície |
| NKp46 | 9.00E+02 | 155Gd | Ativando o Receptor | Superfície |
| NKG2D | 1D11 | 156Gd | Ativando o Receptor | Superfície |
| TIGIT | 741182 | 157Gd | Receptor Inibitório | Superfície |
| 2B4 | C1.7 | 158Gd | Ativando o Receptor | Superfície |
| DNAM-1 | DX11 | 159Tuberculose | Ativando o Receptor | Superfície |
| FAS-L | NOK-1 | 160Gd | Apoptose | Superfície |
| NKp30 | Pág. 30-15 | 161Dy | Ativando o Receptor | Superfície |
| Siglec-7 | S7.7 | 162Dy | Receptor Inibitório | Superfície |
| NKG2C | 134522 | 163Dy | Maturidade/Memória | Superfície |
| NKp44 | Pág. 44-8 | 164Dy | Ativando o Receptor | Superfície |
| CD96 | NK92.39 | 165HO | NKG2 Co-receptor | Superfície |
| KIR2DL1/KIR2DS5 | 143211 | 166Er | Receptor Inibitório | Superfície |
| CD94 | DX22 | 167Er | Ativando o Receptor | Superfície |
| CXCR6 | K041E5 | 168Er | Memória | Superfície |
| PD1 | EH12.2H7 | 169Tm | Receptor Inibitório | Superfície |
| KIR2DL5 | UP-R1 | 170Er | Receptor Inibitório | Superfície |
| NKG2A | 131411 | 171Yb | Receptor Inibitório | Superfície |
| NTB-A | NT-7 | 172Tuberculose | Ativando o Receptor | Superfície |
| KIR3DL1 | DX-9 | 173Yb | Receptor Inibitório | Superfície |
| CD56 | NCAM16.2 | 174Yb | Linhagem de células NK | Superfície |
| KIR2DL3 | 180701 | 175Lu | Receptor Inibitório | Superfície |
| Perforina | B-D48 | 176Yb | Proteína citolítica | intracelular |
| DNA-1/DNA-2 | NA | 191Ir / 193Ir | Células nucleadas | Superfície |
| Cisplatina | NA | 194Pt/195Pt | Viabilidade | Superfície |
| CD16 | 3G8 | 209Bi | Receptor FcgRIII | Superfície |
Tabela 1: Painel NK. Os marcadores são ordenados de acordo com a massa isotópica do metal ao qual foram conjugados. 191Ir/193Ir é a abundância natural do intercalador de ácidos nucleicos. 194Pt/195Pt é a abundância natural de cisplatina.
| Especificidade | Clone | Isótopo | Propósito | Superfície/Intracelular |
| HLA-DR | L243 | 89Y | Células apresentadoras de antígeno, marcador de ativação | Superfície |
| CD45 | HI30 | 102Pd, 104Pd, 106Pd, 108Pd | Código de barras | Superfície |
| CD19 | SJ25-C1 | Qdot 655 (112 Cd-114Cd) | Linhagem | Superfície |
| CD3 | UCHT1 | 115Em | Linhagem | Superfície |
| Pan HLA classe I | W6/32 | 143Nd | Ligantes KIR | Superfície |
| CD7 | CD7-6B7 | 144Nd | Linhagem | Superfície |
| CD8 | SK1 | 145Nd | Linhagem | Superfície |
| CD48 | BJ40 | 146Nd | Ligante 2B4 | Superfície |
| ICAM-1 | HA58 | 148Nd | Ligante LFA-1 | Superfície |
| LLT-1 | 402659 | 149Sm | Ligante CD161 | Superfície |
| CD4 | OKT4 | 151UE | Linhagem | Superfície |
| HLA-C,E | DT9 | 153UE | Ligantes KIR | Superfície |
| CCR2 | K036C2 | 154Sm | Marcador funcional de monócitos | Superfície |
| HLA-E | 3D12 | 155Gd | Ligante NKG2A/CD94 e NKG2C/CD94 | Superfície |
| Fas (CD95) | DX2 | 156Gd | Receptor FasL | Superfície |
| Nectina-1 | R1.302 | 157Gd | Ligante CD96 | Superfície |
| MICA/B | 159227/236511 | 158Gd | Ligantes NKG2D | Superfície |
| DR4/5 | DJR1/DJR2-2 | 159Tuberculose | Receptores TRAIL | Superfície |
| ULBP-1/2,5,6 | 170818/165903 | 161Dy | Ligantes NKG2D | Superfície |
| Nectina-2 | TX31 | 164Dy | Ligante DNAM-1, TIGIT e CD96 | Superfície |
| CD155 | ESQUIVOS.4 | 165HO | Ligante DNAM-1, TIGIT e CD96 | Superfície |
| HLA-Bw4 | REA274 | 166Er | KIR3DL1 ligante | Superfície |
| HLA-Bw6 | REA143 | 168Er | Alelo nulo KIR | Superfície |
| CD14 | M5E2 | 169Tm | Linhagem | Superfície |
| CD11b | ICRF44 | 170Er | Linhagem | Superfície |
| LFA-3 | TS2/9 | 171Yb | Ligante CD2 | Superfície |
| CD33 | WM53 | 172Yb | Linhagem | Superfície |
| CD56 | NCAM16.2 | 174Yb | Linhagem | Superfície |
| B7-H6 | 875001 | 176Yb | Ligante NKp30 | Superfície |
| DNA-1/DNA-2 | NA | 191Ir / 193Ir | Células nucleadas | Superfície |
| Cisplatina | NA | 194Pt/195Pt | Viabilidade | Superfície |
| CD16 | 3G8 | 209Bi | Linhagem | Superfície |
Tabela 2: Painel de ligantes. Os marcadores são ordenados de acordo com a massa isotópica do metal ao qual foram conjugados. 191Ir/193Ir é a abundância natural do intercalador de ácidos nucleicos. 194Pt/195Pt é a abundância natural de cisplatina.
| Especificidade | Clone | Fornecedor | Número de catálogo | Superfície/Intracelular | Anotações |
| 2B4 | 2-69 | BD Biociências | 550814 | Superfície | Novo clone validado com coloração melhorada |
| CD56 | N901 | Beckman Coulter | 6602705 | Superfície | Novo clone validado com coloração melhorada |
| CXCR6 | 56811 | Sistemas de P&D | MAB699 | Superfície | Novo clone validado com menor custo |
| KIR2DS2 | policlonal | Abcam | AB175486 | Superfície | coloração inespecífica observada após liofilização - não usada para análises |
| KIR2DS4 | FES172 | Beckman Coulter | Rolamento A60796 | Superfície | Novo clone validado com coloração aprimorada/menor custo |
| LILRB1 | GHI/75 | Biolenda | 333702 | Superfície | Novo clone validado com coloração melhorada |
| NKG2A | Z199 | Beckman Coulter | IM2750 | Superfície | Novo clone validado com menor custo |
| TIGIT | MBSA43 | Thermo Fisher Científico | 16-9500-82 | Superfície | Novo clone validado |
Tabela Suplementar 1: Anticorpos para painel de células NK que foram testados, mas não usados.
| Isótopo | Especificidade | Clone | Fornecedor | Número de catálogo | Superfície/Intracelular |
| 143Nd | NKG2C | 134522 | Sistemas de P&D | MAB1381 | Superfície |
| 145Nd | CD38 | HIT2 | Biolenda | 303502 | Superfície |
| 146Nd | CD8 | SK1 | Biolenda | 344702 | Superfície |
| 149Sm | CD2 | RPA-2.10 | Biolenda | 300202 | Superfície |
| 151UE | Siglec-7 | S7.7 | Biolenda | 347702 | Superfície |
| 154Sm | LILRB1 | 292319 | Sistemas de P&D | MAB20172 | Superfície |
| 163Dy | KIR3DL1 | DX-9 | BD biociências | 555964 | Superfície |
| 168Er | CD62L | DREG-56 | Biolenda | 304802 | Superfície |
| 171Yb | PD1 | EH12.2H7 | Biolenda | 329902 | Superfície |
| 176Yb | CD69 | EH12.2H7 | Biolenda | 329902 | Superfície |
Tabela Suplementar 2: Anticorpos para painel de células NK que foram testados com um par anticorpo/metal diferente.
Discussão
Aqui, descrevemos o projeto e a aplicação de dois painéis CyTOF complementares destinados a traçar o perfil do repertório de ligantes receptores de células NK. Este protocolo inclui várias etapas que são críticas para a obtenção de dados de qualidade. O CyTOF usa íons de metais pesados, em vez de fluorocromos, como sondas de marcação para anticorpos19. Esta tecnologia está, portanto, sujeita a potenciais sinais de contaminação de metais ambientais20. Fontes potenciais de impurezas metálicas incluem detergente de laboratório (bário) e tampões de laboratório (mercúrio, chumbo, estanho). Por esse motivo, é aconselhável que todos os tampões sejam preparados com água ultrapura e que todos os reagentes sejam armazenados em recipientes de plástico ou vidro que nunca tenham sido lavados com sabão. Outra etapa crítica nesse protocolo é a coloração de viabilidade, que utiliza um método à base de cisplatina, conforme descrito por Fienberg et al.21. Este método inclui uma etapa de incubação de um minuto, durante a qual a cisplatina rotula preferencialmente as células não viáveis. A coloração com cisplatina deve ser realizada na ausência de FBS. Consequentemente, as células devem ser cuidadosamente lavadas com CyPBS antes da coloração. Além disso, para evitar a coloração fora do alvo de células viáveis, a coloração de cisplatina precisa ser extinta com FBS após precisamente um minuto. Este protocolo foi otimizado para máxima recuperação celular e desempenho de coloração. Portanto, as etapas de fixação e permeabilização também são significativas. Vários reagentes de fixação e permeabilização são compatíveis com CyTOF. No entanto, observamos que a fixação com 2% de PFA, seguida de permeabilização com um tampão de permeabilização específico detalhado na Tabela de Materiais, resultou em recuperação celular máxima. Como este é um método de permeabilização transitória, a coloração intracelular precisa ser realizada no tampão de permeabilização para garantir a penetrância adequada do anticorpo. As células também precisam ser lavadas completamente com o tampão de permeabilização após a coloração intracelular para remover os anticorpos não ligados.
Este protocolo permite várias modificações possíveis. Os painéis CyTOF detalhados aqui podem ser personalizados para incluir marcadores adicionais ou substituir os existentes. Em particular, o painel de ligantes foi projetado com vários canais abertos para permitir flexibilidade no design do painel. Qualquer alteração ou adição ao painel pode exigir solução de problemas adicional. Em particular, qualquer par de isótopos anticorpo/metal deve ser completamente validado conforme descrito acima, para evitar qualquer problema de transbordamento de sinal para os canais existentes. Esses painéis também foram projetados para serem compatíveis com o código de barras de amostra. O código de barras diminui a possibilidade de efeitos de lote e transferência de amostra para amostra, minimizando o consumo de reagentes22. Embora o código de barras normalmente resulte em maior qualidade geral dos dados, esta etapa não é necessária para a aquisição de dados CyTOF de boa qualidade e pode ser totalmente ignorada. Da mesma forma, embora recomendemos a purificação das células NK antes da coloração, o painel de células NK é compatível com a fenotipagem de células NK de PBMCs inteiros.
Este método tem algumas limitações. Devido à natureza inerentemente baixa do CyTOF, este método não é adequado para amostras com baixa contagem de células. É improvável que essas amostras produzam dados de qualidade suficiente para análise. Além disso, dado que esses painéis foram projetados especificamente para interrogar as interações entre células NK-células-alvo, eles são limitados em sua capacidade de avaliar interações entre outros tipos de células, como células T CD8+ e células mieloides. Da mesma forma, esses painéis foram projetados para imunofenotipagem ex vivo direta e não foram testados ou validados para uso em condições de ativação, como estimulação de citocinas. Além disso, embora esses painéis cubram uma lista abrangente de receptores e ligantes de células NK, eles não são totalmente exaustivos e vários marcadores potencialmente importantes não foram incluídos devido a limitações de espaço. Alguns desses marcadores incluem, mas não estão limitados a KLRG1, CRACC, TIM-3, LAIR-1 no painel NK e PD-L1 no painel de ligantes. Finalmente, este método não é adequado para uso em amostras fixas, uma vez que a maioria dos epítopos dos marcadores de células NK são afetados pela fixação.
O protocolo descrito aqui tem benefícios significativos em comparação com outros métodos. Outros grupos descreveram painéis de citometria de fluxo voltados para o estudo de células NK 3,4,5,6,7,8,23. Em comparação com a citometria de fluxo, o uso do CyTOF elimina problemas relacionados à compensação de fluoróforos, permitindo a detecção simultânea de um grande número de marcadores. Embora outros também tenham desenvolvido painéis CyTOF para estudar células NK 9,10,11,12,13,14, aqui descrevemos o uso de dois painéis CyTOF complementares, que interrogam a expressão de ambos os receptores de células NK e seus ligantes, fornecendo assim uma imagem mais detalhada da função das células NK.
Nosso grupo usou este protocolo e um ou ambos os painéis para caracterizar a resposta das células NK humanas em doadores saudáveis e em uma variedade de contextos de doenças, incluindo infecção por HIV e infecção pelo vírus da dengue 24,25,26,27,28. Apesar de serem projetados com o objetivo de estudar infecções virais, esses painéis se prestam ao estudo de células NK em outras condições, dada a respiração de proteínas que cobrem. De fato, nosso grupo também utilizou esses painéis para caracterizar o repertório de receptores-ligantes de células NK em pacientes com imunodeficiências e esclerose múltipla 25,27,29,30, bem como em camundongos humanizados31,32. Como tal, o uso desses painéis pode ser estendido a outros contextos. Por exemplo, muitos receptores de células NK e ligantes cognatos implicados no cenário do câncer estão incluídos nesses painéis, tornando-os excelentes ferramentas para estudos futuros sobre o papel das células NK na resposta antitumoral. De forma mais ampla, nosso protocolo para produção e armazenamento em massa de painéis CyTOF, bem como processamento paralelo de amostras, pode ser aplicado à execução e aplicação de qualquer painel CyTOF.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer a todos os membros atuais e antigos do Laboratório Blish que contribuíram para este painel. Obrigado ao Grupo de Ensaios Clínicos da AIDS e à equipe do ACTG A5321, bem como à Dra. Sandra López-Vergès e Davis Beltrán do Gorgas Memorial Institute for Health Studies pela curadoria da amostra. Por fim, obrigado a Michael Leipold, Holden Maecker e ao Centro de Monitoramento Imunológico Humano de Stanford pelo uso de suas máquinas Helios. Este trabalho foi apoiado pelo NIH U19AI057229, NIH R21 AI135287, NIH R21 AI130532, NIH DP1 DA046089 e Burroughs Wellcome Fund Investigators in the Pathogenesis of Infectious Diseases # 1016687 to CB, NIH Ruth L. Kirschstein Institutional National Research Service Award T32 AI007502, TL1 TR001084 e NIH / NIAID K08 AI138640 to EV, National Science Foundation Graduate Research Fellowship DGE-1656518 para JM e bolsa de treinamento NIH T32-AI-007290 (PI Olivia Martinez). O estudo ACTG recebeu apoio financeiro do AI-68634 (Centro de Gerenciamento Estatístico e de Dados), UM1-A1-26617, AI-131798 e AI-68636 (ACTG). CB é o Tashia and John Morgridge Faculty Scholar em Medicina Translacional Pediátrica do Stanford Maternal Child Health Research Institute e investigador do Chan Zuckerberg Biohub.
Materiais
| Name | Company | Catalog Number | Comments |
| 89Y | Sigma-Aldrich | 204919 | |
| 102-Palladium nitrate | Trace Sciences International | Special Order | |
| 104-Palladium nitrate | Trace Sciences International | Special Order | |
| 106-Palladium nitrate | Trace Sciences International | Special Order | |
| 108-Palladium nitrate | Trace Sciences International | Special Order | |
| 115In | Trace Sciences International | Special Order | |
| 141Pr | Fluidigm | 201141A | |
| 142Nd | Fluidigm | 201142A | |
| 143Nd | Fluidigm | 201143A | |
| 144Nd | Fluidigm | 201144A | |
| 145Nd | Fluidigm | 201145A | |
| 146Nd | Fluidigm | 201146A | |
| 147Sm | Fluidigm | 201147A | |
| 148Nd | Fluidigm | 201148A | |
| 149Sm | Fluidigm | 201149A | |
| 150Nd | Fluidigm | 201150A | |
| 151Eu | Fluidigm | 201151A | |
| 152Sm | Fluidigm | 201152A | |
| 153Eu | Fluidigm | 201153A | |
| 154Sm | Fluidigm | 201154A | |
| 155Gd | Fluidigm | 201155A | |
| 156Gd | Fluidigm | 201156A | |
| 157Gd | Trace Sciences International | N/A | |
| 158Gd | Fluidigm | 201158A | |
| 159Tb | Fluidigm | 201159A | |
| 160Gd | Fluidigm | 201160A | |
| 161Dy | Fluidigm | 201161A | |
| 162Dy | Fluidigm | 201162A | |
| 163Dy | Fluidigm | 201163A | |
| 164Dy | Fluidigm | 201164A | |
| 165Ho | Fluidigm | 201165A | |
| 166Er | Fluidigm | 201166A | |
| 167Er | Fluidigm | 201167A | |
| 168Er | Fluidigm | 201168A | |
| 169Tm | Fluidigm | 201169A | |
| 170Er | Fluidigm | 201170A | |
| 171Yb | Fluidigm | 201171A | |
| 172Yb | Fluidigm | 201172A | |
| 173Yb | Fluidigm | 201173A | |
| 174Yb | Fluidigm | 201174A | |
| 175Lu | Fluidigm | 201175A | |
| 176Yb | Fluidigm | 201176A | |
| 209Bi anti-CD16 | Fluidigm | 3209002B | Clone 3G8. Used at a 1:50 dilution. |
| 697 cells | Creative Bioarray | CSC-C0217 | |
| Amicon Ultra Centrifugal Filter Units 0.5 with Ultracel-30 Membrane, 30 kDa | Millipore | UFC503096 | |
| Anhydrous acetonitrile | Fisher Scientific | BP1165-50 | |
| anti-2B4 | Biolegend | 329502 | Clone C1.7. |
| anti-B7-H6 | R&D Systems | MAB7144 | Clone 875001. |
| anti-CCR2 | Biolegend | 357202 | Clone K036C2. |
| anti-CD2 | Biolegend | 300202 | Clone RPA-2.10. |
| anti-CD3 | Biolegend | 300402 | Clone UCHT1. |
| anti-CD4 | Biolegend | 317402 | Clone OKT4. |
| anti-CD4 | Biolegend | 344602 | Clone SK3. |
| anti-CD7 | Biolegend | 343102 | Clone CD7-6B7. |
| anti-CD8 | Biolegend | 344702 | Clone SK1. |
| anti-CD11b | Biolegend | 301302 | Clone ICRF44. |
| anti-CD14 | Biolegend | 301802 | Clone M5E2. |
| anti-CD19 | Biolegend | 302202 | Clone HIB19. |
| anti-CD33 | Biolegend | 303402 | Clone WM53. |
| anti-CD38 | Biolegend | 303502 | Clone HIT2. |
| anti-CD48 | Biolegend | 336702 | Clone BJ40. |
| anti-CD56 | BD Pharmingen | 559043 | Clone NCAM16.2. |
| anti-CD57 | Biolegend | 322302 | Clone HCD57. |
| anti-CD62L | Biolegend | 304802 | Clone DREG-56. |
| anti-CD69 | Biolegend | 310902 | Clone FN50. |
| anti-CD94 | Biolegend | 305502 | Clone DX22. |
| anti-CD95 | Biolegend | 305602 | Clone DX2. |
| anti-CD155 | Biolegend | 337602 | Clone SKII.4. |
| anti-CXCR6 | Biolegend | 356002 | Clone K041E5. |
| anti-DNAM-1 | BD Biosciences | 559787 | Clone DX11. |
| anti-DR4 | Biolegend | 307202 | Clone DJR1. |
| anti-DR5 | Biolegend | 307302 | Clone DJR2-2. |
| anti-FAS-L | Biolegend | 306402 | Clone NOK-1. |
| anti-FcRg | Millipore | 06-727 | Polyclonal antibody. |
| anti-HLA-C,E | Millipore | MABF233 | Clone DT9. |
| anti-HLA-Bw4 | Miltenyi Biotec | Special Order | Clone REA274. |
| anti-HLA-Bw6 | Miltenyi Biotec | 130-124-530 | Clone REA143. |
| anti-HLA-DR | Biolegend | 307602 | Clone L243. |
| anti-HLA-E | Biolegend | 342602 | Clone 3D12. |
| anti-ICAM-1 | Biolegend | 353102 | Clone HA58. |
| anti-Ki-67 | Biolegend | 350502 | Clone Ki-67. |
| anti-KIR2DL1/KIR2DS5 | R&D Systems | MAB1844 | Clone 143211. |
| anti-KIR2DL3 | R&D Systems | MAB2014 | Clone 180701. |
| anti-KIR2DL5 | Miltenyi Biotec | 130-096-200 | Clone UP-R1. |
| anti-KIR2DS4 | R&D Systems | MAB1847 | Clone 179315. |
| anti-KIR3DL1 | BD Biosciences | 555964 | Clone DX-9. |
| anti-LFA-3 | Biolegend | 330902 | Clone TS2/9. |
| anti-LILRB1 | R&D Systems | 292319 | Clone MAB20172. |
| anti-LLT-1 | R&D Systems | AF3480 | Clone 402659. |
| anti-MICA | R&D Systems | MAB1300-100 | Clone 159227. |
| anti-MICB | R&D Systems | MAB1599-100 | Clone 236511. |
| anti-Nectin-1 | Biolegend | 340402 | Clone R1.302. |
| anti-Nectin-2 | Biolegend | 337402 | Clone TX31. |
| anti-NKG2A | R&D Systems | MAB1059 | Clone 131411. |
| anti-NKG2C | R&D Systems | MAB1381 | Clone 134522. |
| anti-NKG2D | Biolegend | 320802 | Clone 1D11. |
| anti-NKp30 | Biolegend | 325202 | Clone P30-15. |
| anti-NKp44 | Biolegend | 325102 | Clone P44-8. |
| anti-NKp46 | Biolegend | 331902 | Clone 9E2. |
| anti-NTB-A | Biolegend | 317202 | Clone NT-7. |
| anti-Pan HLA class I | Biolegend | 311402 | Clone W6/32. |
| anti-PD1 | Biolegend | 329902 | Clone EH12.2H7. |
| anti-Perforin | Abcam | ab47225 | Clone B-D48. |
| anti-Siglec-7 | Biolegend | 347702 | Clone S7.7. |
| anti-Syk | Biolegend | 644302 | Clone 4D10.2. |
| anti-TACTILE | Biolegend | 338402 | Clone NK92.39. |
| anti-TIGIT | R&D Systems | MAB7898 | Clone 741182. |
| anti-ULBP-1 | R&D Systems | MAB1380-100 | Clone 170818. |
| anti-ULBP-2, 5, 6 | R&D Systems | MAB1298-100 | Clone 165903. |
| Antibody Stabilizer | Candor Bioscience | 131 050 | |
| Benzonase Nuclease | Millipore | 70664 | |
| Bond-Breaker TCEP Solution | Thermo Fisher Scientific | 77720 | |
| Bovine Serum Albumin solution | Sigma-Aldrich | A9576 | |
| Calcium chloride dihydrate (CaCl2+2H2O) | Sigma-Aldrich | 223506-25G | |
| Cis-Platinum(II)diamine dichloride (cisplatin) | Enzo Life Sciences | ALX-400-040-M250 | A 100 mM stock solution was prepared in DMSO and divided into 25 µL aliquots. Used at a 25 µM dilution for live/dead stain. Signal appears in 194Pt and 195Pt channels. |
| DMSO | Sigma-Aldrich | D2650 | |
| eBioscience Permeabilization Buffer | Thermo Fisher Scientific | 00-8333-56 | |
| EDTA (0.5 M) | Hoefer | GR123-100 | A double-concentrated HEPES buffer with EDTA was made according to the following recipe: 1.3 g NaCl (Thermo Fisher Scientific), 27 mg CaCl2+2H2O (Sigma-Aldrich), 23 mg MgCl2 (Sigma-Aldrich), 83.6 mg KH2PO4 (Thermo Fisher Scientific), 4 mL of 1M HEPES (Thermo Fisher Scientific), 2 mL of 0.5M EDTA (Hoefer, Holliston, MA, USA), and 100mL H2O. The pH of this double-concentrated HEPES buffer was adjusted to a pH of 7.3 using 1M HCl and 1M NaOH. |
| EQ Four Element Calibration Beads | Fluidigm | 201078 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | N/A | |
| Helios mass cytometer | Fluidigm | N/A | |
| HEPES (1M) | Thermo Fisher Scientific | 15630080 | |
| HyClone Antibiotic/Antimycotic Solution (Pen/Strep/Fungiezone) solution | Fisher Scientific | SV3007901 | |
| Iridium - 191Ir/193Ir intercalator | DVS Sciences (Fluidigm) | 201192B | Used at a 1:10000 dilution. |
| Isothiocyanobenzyl-EDTA (ITCB-EDTA) | Dojindo Molecular Technologies, Inc. | M030-10 | Diluted to 1.25 mg/mL in anhydrous acetonitrile. |
| K562 cells | American Type Culture Collection (ATCC) | ATCC CCL-243 | |
| L-Glutamine (200 mM) | Thermo Fisher Scientific | SH30034 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 208337-100G | |
| Maxpar X8 Antibody Labeling Kits | Fluidigm | N/A | No catalog number as kits come with metals. |
| Millex-VV Syringe Filter Unit, 0.1 µm | Millipore | SLVV033RS | |
| Milli-Q Advantage A10 Water Purification System | Millipore | Z00Q0V0WW | |
| MS Columns | Miltenyi Biotec | ||
| NALM6 cells | American Type Culture Collection (ATCC) | ATCC CRL-3273 | |
| Nanosep Centrifugal Devices with Omega Membrane 3K | Pall Corporation | OD003C35 | |
| NK Cell Isolation Kit, human | Miltenyi Biotec | 130-092-657 | |
| Paraformaldehyde (16%) | Electron Microscopy Sciences | 15710 | |
| PBS | Thermo Fisher Scientific | 10010023 | |
| Potassium Phosphate Monobasic (KH2PO4) | Fisher Scientific | MP021954531 | |
| Qdot 655 anti-CD19 | Thermo Fisher Scientific | Q10179 | Clone SJ25-C1. Used at a 1:50 dilution. Signal appears in 112Cd-114Cd channels. |
| Qdot 655 anti-HLA-DR | Thermo Fisher Scientific | Q22158 | Clone Tü36. Used at a 1:200 dilution. |
| Rockland PBS | Rockland Immunochemicals, Inc. | MB-008 | Used to make CyPBS (10X Rockland PBS diluted to 1X in Milli-Q water) and CyFACS buffers (10X Rockland PBS diluted to 1X in Milli-Q water with 0.1% BSA and 0.05% sodium azide). Buffers were sterile-filtered through a 0.22 µM filter and sotred at 4°C in Stericup bottles. |
| RPMI 1640 | Thermo Fisher Scientific | 21870092 | |
| Sodium azide (NaN3) | Sigma-Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stericup Quick Release-GP Sterile Vacuum Filtration System | Millipore Sigma | S2GPU10RE | |
| Tuning solution | Fluidigm | 201072 | |
| Washing solution | Fluidigm | 201070 |
Referências
- Shimasaki, N., Jain, A., Campana, D. NK cells for cancer immunotherapy. Nature Reviews. Drug Discovery. 19 (3), 200-218 (2020).
- Vivier, E., Tomasello, E., Baratin, M., Walzer, T., Ugolini, S. Functions of natural killer cells. Nature Immunology. 9 (5), 503-510 (2008).
- Eller, M. A., Currier, J. R. OMIP-007: phenotypic analysis of human natural killer cells. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 81 (6), 447-449 (2012).
- Mahnke, Y. D., Beddall, M. H., Roederer, M. OMIP-029: Human NK-cell phenotypization. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 87 (11), 986-988 (2015).
- Hammer, Q., Romagnani, C. OMIP-039: Detection and analysis of human adaptive NKG2C + natural killer cells : Detection of Human Adaptive NKG2C + NK Cells. Cytometry. 91 (10), 997-1000 (2017).
- Liechti, T., Roederer, M. OMIP-058: 30-Parameter flow cytometry panel to characterize iNKT, NK, unconventional and conventional T cells. Cytometry. 95 (9), 946-951 (2019).
- Béziat, V., et al. NK cell responses to cytomegalovirus infection lead to stable imprints in the human KIR repertoire and involve activating KIRs. Blood. 121 (14), 2678-2688 (2013).
- Pfefferle, A., et al. Intra-lineage plasticity and functional reprogramming maintain natural killer cell repertoire diversity. Cell Reports. 29 (8), 2284-2294 (2019).
- Barcenilla, H., Åkerman, L., Pihl, M., Ludvigsson, J., Casas, R. Mass cytometry identifies distinct subsets of regulatory T cells and Natural killer cells associated with high risk for Type 1 diabetes. Frontiers in Immunology. 10, 982(2019).
- Kurioka, A., et al. CD161 defines a functionally distinct subset of pro-inflammatory Natural killer cells. Frontiers in Immunology. 9, 486(2018).
- Romee, R., et al. Cytokine-induced memory-like natural killer cells exhibit enhanced responses against myeloid leukemia. Science Translational Medicine. 8 (357), (2016).
- Shinko, D., et al. Mass cytometry reveals a sustained reduction in CD16+ Natural killer cells following chemotherapy in colorectal cancer patients. Frontiers in Immunology. 10, 2584(2019).
- Pohlmeyer, C. W., et al. Identification of NK cell subpopulations that differentiate HIV-infected subject cohorts with diverse levels of virus control. Journal of Virology. 93 (7), 01790(2019).
- Palgen, J. -L., et al. NK cell immune responses differ after prime and boost vaccination. Journal of Leukocyte Biology. 105 (5), 1055-1073 (2019).
- Mei, H. E., Leipold, M. D., Schulz, A. R. Barcoding of live human peripheral blood mononuclear cells for multiplexed mass cytometry. The Journal of Immunology. 194 (4), 2022-2031 (2015).
- Takahashi, C., et al. Mass cytometry panel optimization through the designed distribution of signal interference. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 91 (1), 39-47 (2017).
- Baumgart, S., Peddinghaus, A., Schulte-Wrede, U., Mei, H. E., Grützkau, A. OMIP-034: Comprehensive immune phenotyping of human peripheral leukocytes by mass cytometry for monitoring immunomodulatory therapies. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 91 (1), 34-38 (2017).
- Leipold, M. D. Another step on the path to mass cytometry standardization. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 87 (5), 380-382 (2015).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Leipold, M. D., Newell, E. W., Maecker, H. T. Multiparameter phenotyping of human PBMCs using mass cytometry. Methods in Molecular Biology. 1343, 81-95 (2015).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 81 (6), 467-475 (2012).
- Zivanovic, N., Jacobs, A., Bodenmiller, B. A practical guide to multiplexed mass cytometry. Current Topics in Microbiology and Immunology. 377, 95-109 (2014).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150 (3), 248-264 (2017).
- Wilk, A. J., et al. Charge-altering releasable transporters enable specific phenotypic manipulation of resting primary natural killer cells. BioRxiv. , 970491(2020).
- Vendrame, E., et al. TIGIT is upregulated by HIV-1 infection and marks a highly functional adaptive and mature subset of natural killer cells. AIDS. 34 (6), 801-813 (2020).
- Zhao, N. Q., et al. Natural killer cell phenotype is altered in HIV-exposed seronegative women. PloS One. 15 (9), 0238347(2020).
- McKechnie, J. L., et al. HLA upregulation during dengue virus infection suppresses the natural killer cell response. Frontiers in Cellular and Infection Microbiology. 9, 268(2019).
- McKechnie, J. L., et al. Mass cytometry analysis of the NK cell receptor-ligand repertoire reveals unique differences between dengue-infected children and adults. ImmunoHorizons. 4 (10), 634-647 (2020).
- Ranganath, T., et al. Characterization of the impact of daclizumab beta on circulating natural killer cells by mass cytometry. Frontiers in immunology. 11, 714(2020).
- Fernandez, I. Z., et al. A novel human IL2RB mutation results in T and NK cell--driven immune dysregulation. The Journal of Experimental Medicine. 216 (6), 1255-1267 (2019).
- Herndler-Brandstetter, D., et al. Humanized mouse model supports development, function, and tissue residency of human natural killer cells. Proceedings of the National Academy of Sciences. 114 (45), 9626-9634 (2017).
- Nikzad, R., et al. Human natural killer cells mediate adaptive immunity to viral antigens. Science Immunology. 4 (35), (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados