Method Article
Profilage du répertoire des récepteurs et ligands des cellules tueuses naturelles humaines
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous concevons deux panels de cytométrie de masse complémentaire (CyTOF) et optimisons un protocole de coloration CyTOF dans le but de profiler le récepteur des cellules tueuses naturelles et le répertoire de ligands dans le cadre d’infections virales.
Résumé
Les cellules tueuses naturelles (NK) sont parmi les premières à répondre aux infections virales. La capacité des cellules NK à reconnaître et à tuer rapidement les cellules infectées par le virus est régulée par l’expression de récepteurs inhibiteurs et activateurs codés par la lignée germinale. L’engagement de ces récepteurs par leurs ligands apparentés sur les cellules cibles détermine si l’interaction intercellulaire entraînera la destruction des cellules NK. Ce protocole détaille la conception et l’optimisation de deux panels de cytométrie de masse complémentaire (CyTOF). Un panel a été conçu pour phénotyper les cellules NK en fonction de l’expression des récepteurs. L’autre panel a été conçu pour interroger l’expression de ligands connus pour les récepteurs des cellules NK sur plusieurs sous-ensembles de cellules immunitaires. Ensemble, ces deux panels permettent de profiler le répertoire récepteur-ligand des cellules NK humaines. De plus, ce protocole détaille également le processus par lequel nous colorons les échantillons pour le CyTOF. Ce processus a été optimisé pour une meilleure reproductibilité et une meilleure standardisation. L’un des avantages de CyTOF est sa capacité à mesurer plus de 40 marqueurs dans chaque panneau, avec un chevauchement minimal des signaux, ce qui permet aux chercheurs de capturer l’étendue du répertoire récepteur-ligand des cellules NK. Le code-barres au palladium réduit également les variations entre les échantillons, ainsi que la consommation de réactifs, ce qui facilite la coloration des échantillons avec chaque panneau en parallèle. Les limites de ce protocole comprennent le débit relativement faible de CyTOF et l’incapacité de récupérer les cellules après l’analyse. Ces panels ont été conçus pour l’analyse d’échantillons cliniques de patients souffrant d’infections virales aiguës et chroniques, notamment le virus de la dengue, le virus de l’immunodéficience humaine (VIH) et la grippe. Cependant, ils peuvent être utilisés dans n’importe quel contexte pour étudier le répertoire des récepteurs et ligands des cellules NK humaines. Il est important de noter que ces méthodes peuvent être largement appliquées à la conception et à l’exécution des futurs panneaux CyTOF.
Introduction
Les cellules tueuses naturelles (NK) sont des cellules immunitaires innées dont le rôle principal est de cibler et de tuer les cellules malignes, infectées ou autrement stressées. Grâce à leur sécrétion de cytokines telles que l’IFNγ et le TNFα, ainsi qu’à leur activité cytotoxique, les cellules NK peuvent également façonner la réponse immunitaire adaptative aux agents pathogènes et aux tumeurs malignes. La réponse NK est médiée en partie par la signalisation combinatoire des récepteurs inhibiteurs et activateurs codés par la lignée germinale, qui se lient à une myriade de ligands exprimés sur des cellules cibles potentielles. Plusieurs récepteurs des cellules NK ont plus d’un ligand, de nouvelles paires récepteur-ligand étant régulièrement identifiées.
On s’intéresse particulièrement à l’étude des cellules NK dans le contexte des infections virales, où leur capacité à répondre rapidement aux cellules stressées peut limiter la propagation virale ou favoriser le développement de stratégies d’évasion des cellules NK. Cet intérêt pour la biologie des cellules NK s’étend au domaine de l’immunothérapie du cancer, où les chercheurs étudient le rôle des cellules NK dans l’immunosurveillance tumorale et dans le microenvironnement tumoral1. Cependant, la capacité de profiler les interactions cellule-cellule cible NK est compliquée par le fait que les cellules NK humaines peuvent exprimer plus de 30 récepteurs qui peuvent à leur tour interagir avec plus de 30 ligands connus2. La détection simultanée de plusieurs récepteurs de cellules NK et de leurs ligands apparentés est donc nécessaire pour saisir la complexité des interactions récepteur-ligand qui contrôlent la fonction NK. Nous nous sommes donc tournés vers la cytométrie de masse (CyTOF), qui permet de détecter simultanément plus de 40 marqueurs au niveau de la cellule unique. Notre objectif était de créer deux panels CyTOF pour profiler le répertoire récepteur-ligand des cellules NK. Nous voulions également concevoir un protocole pour un traitement et une coloration efficaces des échantillons cliniques. Les échantillons humains cliniques fournissent une mine d’informations sur la façon dont le corps réagit à l’infection virale. Par conséquent, nous avons développé ce protocole pour étudier l’expression des récepteurs des cellules NK et de leurs ligands apparentés en parallèle afin une meilleure standardisation, une meilleure récupération, une consommation réduite de réactifs et des effets de lot limités.
Plusieurs panels de cytométrie en flux conçus pour caractériser le phénotype des cellules NK humaines ont été publiés précédemment 3,4,5,6,7,8. La plupart de ces panels sont limités dans leur capacité à saisir l’étendue du répertoire récepteur-ligand, ne permettant la détection que d’une sélection limitée de marqueurs. De plus, ces panneaux sont limités par le chevauchement des signaux entre les fluorochromes. CyTOF utilise des anticorps conjugués à des isotopes métalliques, qui sont lus par spectrométrie de masse à temps de vol, réduisant ainsi considérablement le débordement entre les canaux.
Comme nous, d’autres chercheurs se sont tournés vers CyTOF pour étudier les cellules NK9, 10, 11, 12, 13, 14, bien qu’avec généralement moins de marqueurs de cellules NK, ce qui réduit la profondeur du phénotypage. Bien que les protocoles généraux de coloration utilisés par ces groupes soient similaires aux nôtres, il existe quelques différences clés. D’autres protocoles n’impliquent pas d’isoler les cellules NK avant la coloration, même si les chercheurs ne s’intéressent qu’à ce sous-ensemble13,14. Étant donné que les cellules NK ne représentent que 5 à 20 % des cellules mononucléées du sang périphérique (PBMC), la coloration de PBMC entiers plutôt que de cellules NK isolées signifie que la plupart des événements collectés ne seront pas des cellules NK. Cela réduit la quantité de données générées sur le sous-ensemble d’intérêt et entraîne une utilisation inefficace du temps machine. De plus, alors que bon nombre de ces panels interrogent l’expression des récepteurs des cellules NK tels que les récepteurs Ig-like tueurs (KIR), les NKG2A/C/D et les récepteurs naturels de cytotoxicité (NKp30, NKp44 et NKp46), l’expression de ces marqueurs n’est pas placée dans un contexte plus large en raison de l’absence de données sur l’expression de leurs ligands respectifs. Par conséquent, bien que ces méthodes précédemment publiées pour étudier les cellules NK via CyTOF soient suffisantes pour le phénotypage large des cellules NK, utilisées isolément, elles ne peuvent pas fournir une image complète de l’activité des cellules NK. Cela nous amène au principal avantage des méthodes décrites ici, à savoir qu’à ce jour, il n’existe pas de cytométrie en flux publiée ou de panels CyTOF axés sur l’exploration de l’expression des ligands des récepteurs des cellules NK. Il est important de noter que notre panel de ligands dispose de plusieurs canaux ouverts pour permettre l’ajout de marqueurs afin de répondre aux besoins uniques de chaque expérience.
Étant donné que l’une des principales limites de CyTOF est l’incapacité de récupérer l’échantillon après l’analyse, cette méthode peut ne pas convenir aux chercheurs qui ont des échantillons limités avec lesquels ils souhaitent effectuer des expériences supplémentaires. De plus, la nature à faible débit de CyTOF signifie que les données générées seront de mauvaise qualité si le nombre de cellules de départ est faible. À l’exception de ces deux limitations, cette méthode fonctionnera bien dans n’importe quel contexte pour étudier les interactions récepteur-ligand entre les cellules NK et les cellules cibles.
Protocole
Des PBMC adultes sains anonymisés ont été obtenus à partir de chambres du système de leucoréduction achetées au Stanford Blood Center. Des PBMC provenant de donneurs pédiatriques sains anonymisés et de patients pédiatriques atteints de dengue aiguë ont été obtenus auprès de l’Institut d’études sur la santé Gorgas Memorial à Panama City, au Panama, et dans des hôpitaux appartenant au ministère de la Santé, au système de sécurité sociale de Panama City et dans les zones suburbaines. Le protocole d’étude sur la dengue a été approuvé par l’IRB de l’Hospital del Niño (CBIHN-M-0634), puis approuvé par les comités de l’ICGES, du CSS, de l’hôpital Santo Tomas et de l’Université de Stanford. Les PBMC de patients infectés par le VIH sous traitement antirétroviral ont été obtenus à partir de l’étude ACTG A5321.
1. Marquage des anticorps, préparation du panel et stockage
- Marquage des anticorps avec des isotopes métalliques
REMARQUE : Pour augmenter la standardisation inter-expérimentale de la coloration, il est recommandé d’effectuer plusieurs conjugaisons pour chaque anticorps, puis de combiner les produits en un seul mélange maître pour un stockage à long terme, comme décrit ci-dessous.- Déterminez la concentration de chaque anticorps en mesurant l’absorbance à 280 nm avant la conjugaison. Les anticorps utilisés pour ce protocole sont disponibles dans le commerce et ont été achetés auprès des fournisseurs énumérés dans la table des matières.
- Étiquetez les anticorps avec des isotopes métalliques à l’aide de trousses d’étiquetage d’anticorps disponibles dans le commerce, conformément aux instructions du fabricant. Utilisez 100 μg d’anticorps pour chaque réaction.
- Déterminez la concentration finale de l’anticorps récupéré en mesurant l’absorbance à 280 nm. Stocker les anticorps à court terme à 4 °C.
- Titrage des anticorps
REMARQUE : La technologie CyTOF est très sensible aux signaux de contamination potentiels des métaux environnementaux. Par conséquent, tous les tampons/réactifs utilisés doivent être préparés avec de l’eau ultrapure et stockés dans des récipients en plastique ou en verre qui n’ont jamais été lavés avec du savon.- Préparez pour chaque donneur des tubes à centrifuger contenant 9 mL de RPMI complet et chaud (RPMI 1640, 10 % FBS, 1 % L-glutamine, 1 % pénicilline/streptomycine) et 20 μL de benzonase par flacon de PBMC à décongeler. Décongelez les PBMC dans un bain-marie et ajoutez-les dans des tubes.
REMARQUE : La benzonase diminue la viscosité et le bruit de fond de l’ADN libre des cellules lysées. - Centrifugeuse à 300 x g à température ambiante pendant 5 min. Remettez les PBMC en suspension dans 5 ml de support RPMI complet et comptez.
- Pour chaque titrage de panneau, plaquez 2 à 4 millions de PBMC/puits dans 6 puits d’une plaque à fond rond de 96 puits (un puits pour chaque titre et un pour non coloré). Centrifuger la plaque à 600 x g à température ambiante pendant 3 min. Retournez la plaque pour retirer le surnageant. Remettre chaque puits en suspension dans 200 μL de CyPBS.
- Effectuez la coloration de viabilité au cisplatine comme décrit ci-dessous.
REMARQUE : Le cisplatine est utilisé pour distinguer les cellules vivantes des cellules mortes en cytométrie de masse.- Remettre les cellules en suspension dans 100 μL de stock de cisplatine 25 μM. Incuber à température ambiante pendant 1 min.
- Tremper la réaction de cisplatine en ajoutant 100 μL de FBS dans chaque puits et en mélangeant. Centrifugez et agissez la plaque.
REMARQUE : Effectuez toutes les étapes de centrifugation suivantes à 4 °C. - Laver les cellules deux fois avec 200 μL de CyFACS (1x PBS sans contaminants de métaux lourds dans une eau ultrapure avec 0,1 % de BSA, 0,05 % d’azoture de sodium). Centrifugez et retournez la plaque à chaque fois.
- Titrez le panel d’anticorps de surface comme décrit ci-dessous.
REMARQUE : Des mélanges principaux séparés doivent être réalisés pour le panneau de surface NK et le panneau de ligand.- Réaliser un mélange magistral de tous les anticorps de surface à une concentration de 10 μg/mL à l’aide de CyFACS. Visez un volume final de 150 μL. Faites des dilutions en série de 1:2 à l’aide de CyFACS, pour obtenir les concentrations suivantes : 10, 5, 2,5, 1,25 et 0,625 μg/mL.
- Filtrer les cocktails d’anticorps à travers une unité de filtration centrifuge (pores de 0,1 μm) à 10 600 x g pendant 3 min avant la coloration.
- Remettre les cellules en suspension dans 50 μL du cocktail d’anticorps de surface au titre respectif. Remettez en suspension le puits non taché dans CyFACS. Incuber à 4 °C pendant 30 min.
- Laver les cellules avec 150 μL de CyFACS. Centrifugez et agissez la plaque.
- Laver les cellules avec 200 μL de CyFACS. Centrifugez et agissez la plaque.
- Effectuer la fixation des cellules en remettant en suspension chaque puits dans 100 μL de paraformaldéhyde (PFA) à 2 % dans du CyPBS. Incuber la plaque à température ambiante dans l’obscurité pendant 20 min. Laver les cellules avec 100 μL de CyFACS. Centrifugeuse à 700 x g pendant 5 min.
ATTENTION : Le PFA est soupçonné de provoquer des anomalies génétiques ainsi que le cancer. De plus, il est nocif s’il entre en contact avec les yeux, la peau ou est inhalé. Manipulez correctement en assurant une bonne ventilation, en ouvrant le récipient avec précaution et en empêchant la formation d’aérosols.
REMARQUE : Tous les centrifugations ultérieures sont effectuées à 700 x g pendant 5 min à 4 °C. - Perméabiliser les cellules en les remettant en suspension dans 200 μL de 1x tampon de perméabilisation (tampon Perm) dilué dans de l’eau ultrapure. Centrifugez et agissez la plaque. Laver les cellules avec 200 μL de tampon Perm. Centrifugez et agissez la plaque.
REMARQUE : L’incubation dans le tampon de Perm n’est pas nécessaire. - Titrage du panel d’anticorps intracellulaires
- Faites un mélange maître de tous les anticorps intracellulaires à une concentration de 10 μg/mL à l’aide du tampon Perm. Visez un volume final de 150 μL. Faites des dilutions en série de 1:2 à l’aide du tampon Perm pour obtenir les concentrations suivantes : 10, 5, 2,5, 1,25 et 0,625 μg/mL.
- Filtrer les cocktails d’anticorps à travers une unité de filtration centrifuge (pores de 0,1 μm) à 10 600 x g pendant 3 min avant la coloration.
- Remettre les cellules en suspension dans 50 μL du cocktail d’anticorps intracellulaires au titre respectif. Remettez en suspension le puits non taché dans le tampon permanent. Incuber à 4 °C pendant 45 min.
REMARQUE : Si un panneau intracellulaire ne doit pas être titré, remettre les puits en suspension dans 50 μL du tampon Perm. - Laver les cellules avec 150 μL de tampon Perm. Centrifugez et agissez la plaque.
- Laver les cellules avec 200 μL de tampon Perm. Centrifugez et agissez la plaque.
- Lavez les cellules deux fois avec 200 μL de CyFACS. Centrifugez et agissez la plaque.
- Coloration de l’intercalateur d’ADN
REMARQUE : L’intercalateur se lie à l’acide nucléique cellulaire et est utilisé pour identifier les cellules nucléées en cytométrie de masse.- Remettre en suspension les cellules dans 200 μL d’intercalateur dilué 1:10 000 dans du CyPBS et 2 % de PFA. Incuber la plaque pendant une nuit à 4 °C.
- Conservez les plaques, si nécessaire, à 4 °C recouvertes d’un film de paraffine jusqu’à une semaine.
REMARQUE : Effectuez toutes les étapes de centrifugation suivantes à 700 x g pendant 5 min à 4 °C. - Avant d’analyser les échantillons sur CyTOF, retirez le film de paraffine de la plaque et centrifugez-le à 700 x g pendant 5 min à 4 °C. Retournez l’assiette. Lavez les cellules une fois avec 200 μL de CyFACS. Centrifugez et agissez la plaque.
- Laver les cellules trois fois avec 200 μL d’eau ultrapure. Centrifugez et agissez la plaque. Remettez les cellules en suspension dans 200 μL d’eau ultrapure. Immédiatement avant de prélever l’échantillon, ajuster la concentration à environ 6 x 105 cellules/mL dans des billes de normalisation diluées à une concentration de 1 fois dans de l’eau ultrapure.
- Exécutez les échantillons sur CyTOF.
- Analysez les données et choisissez les titres appropriés pour chaque anticorps en sélectionnant le titre d’anticorps le plus bas, ce qui se traduit par l’intensité du signal la plus élevée et la meilleure séparation entre les populations positives et négatives sur la base d’une évaluation visuelle.
REMARQUE : Les titrages pour les panneaux NK et ligand sont illustrés respectivement à la figure 1 et à la figure 2 . Si une distinction claire entre les populations positives et négatives n’est pas identifiée, les titres peuvent être évalués sur plusieurs types de cellules ou sur des lignées cellulaires, afin de permettre l’identification des populations cellulaires positives et négatives. - Stockage du panneau d’anticorps
- Combinez les anticorps titrés dans un mélange maître et filtrez à travers une unité de filtre à seringue stérile de 0,1 μm. Des mélanges maîtres séparés doivent être réalisés pour le panneau de surface NK, le panneau intracellulaire NK et le panneau de ligand.
- Pour l’entreposage à long terme des panneaux, suivez l’une des deux options acceptables :
- Envoyez le master mix à une société tierce pour la lyophilisation. Cette méthode est utilisée pour le panneau NK. Les anticorps non conjugués en interne ne peuvent pas être lyophilisés, en raison de la présence d’un stabilisateur d’anticorps, qui interfère avec le processus de lyophilisation. Ces anticorps sont ajoutés au panel le jour de la coloration.
- À l’aide d’une pipette à répéteur, faites des aliquotes de 50 μL de chaque mélange maître et conservez-les à -80 °C.
- Préparez pour chaque donneur des tubes à centrifuger contenant 9 mL de RPMI complet et chaud (RPMI 1640, 10 % FBS, 1 % L-glutamine, 1 % pénicilline/streptomycine) et 20 μL de benzonase par flacon de PBMC à décongeler. Décongelez les PBMC dans un bain-marie et ajoutez-les dans des tubes.
2. Protocole de coloration
- Décongeler les cellules mononucléées du sang périphérique (PBMC) comme décrit aux étapes 1.2.1 et 1.2.2. Mettez de côté au moins 1 million de PBMC pour la coloration du panneau de ligand dans un tube à centrifuger de 15 ml. Gardez ces PBMC sur la glace pendant l’isolement des cellules NK.
- Isolement des cellules NK
REMARQUE : Les étapes d’isolement de cellule NK suivantes sont une version modifiée du protocole d’un fournisseur spécifique. Cependant, tout kit ou protocole qui effectue une sélection négative magnétique des cellules NK serait une alternative appropriée. De plus, cette étape est facultative car ce protocole est également adapté au phénotypage des cellules NK à partir de PBMC entiers.- Faites tourner les PBMC restants à 450 x g pendant 5 min. Mettre en suspension la pastille de cellule dans 40 μL de tampon MACS (PBS, 0,5 % BSA, 2 mM EDTA) par 10à 7 cellules au total.
- Ajouter 10 μL de cocktail biotine-anticorps de cellules NK pour 10à 7 cellules au total. Bien mélanger et incuber 5 min sur glace.
- Ajouter 30 μL de tampon MACS pour 107 cellules au total et 20 μL de cocktail MicroBead de cellules NK pour 107 cellules au total. Bien mélanger et incuber 10 min sur glace.
- Préparez les colonnes d’élution en les rinçant avec 500 μL de tampon MACS. Ajouter 2 mL de RPMI complet froid dans des tubes de prélèvement de 15 mL.
- Ajoutez un tampon MACS aux tubes contenant des cellules pour porter le volume à 500 μL. Pipetez tout le volume de 500 μL sur la colonne d’élution préparée. Rincez le tube avec 500 μL supplémentaires de tampon MACS et transférez-le dans la colonne.
- Une fois le débit arrêté, rincez deux fois la colonne d’élution avec un tampon MACS de 500 μL. Une fois le flux arrêté, comptez les cellules NK.
- Plaques et cellules de lavage.
- La centrifugeuse a isolé les cellules NK et les PBMC à 300 x g à température ambiante pendant 10 min. Remettre en suspension les cellules à une concentration de 5 millions de cellules/mL dans le CyPBS (1x PBS sans contaminants de métaux lourds dans l’eau ultrapure). Cellules en U à 96 puits.
REMARQUE : Chaque aliquote du panneau de cellules NK peut colorer jusqu’à 3 millions de cellules. Si six échantillons individuels de cellules NK sont codés à barres et regroupés avant la coloration, le nombre total combiné de cellules NK ne doit pas dépasser 3 millions. Le panel de ligands peut colorer jusqu’à 6 millions de PBMC par échantillon. Si six échantillons individuels sont codés à barres et regroupés avant la coloration, le nombre total combiné de PBMC ne devrait pas dépasser 6 millions. - Plaque de centrifugation à 600 x g à température ambiante pendant 3 min. Agissez la plaque pour retirer le surnageant.
REMARQUE : Effectuez tous les essorages ultérieurs de la centrifugeuse à 600 x g pendant 3 min jusqu’à l’étape 2.7. - Remettre les cellules en suspension dans 200 μL de CyPBS. Centrifugez et agissez la plaque.
- La centrifugeuse a isolé les cellules NK et les PBMC à 300 x g à température ambiante pendant 10 min. Remettre en suspension les cellules à une concentration de 5 millions de cellules/mL dans le CyPBS (1x PBS sans contaminants de métaux lourds dans l’eau ultrapure). Cellules en U à 96 puits.
- Effectuez la coloration de viabilité au cisplatine comme décrit à l’étape 1.2.4.
- Coloration par code-barres
REMARQUE : Ces panneaux sont utilisés conjointement avec une méthode modifiée de codage à barres à base de palladium sur des cellules vivantes non fixées afin de minimiser les effets de lot et de maximiser la récupération des cellules15. Cependant, cette étape est facultative car le code-barres n’est pas nécessaire pour obtenir des données de qualité.- Remettre chaque puits en suspension dans 50 μL de code-barres prémélangé respectif et incuber à 4 °C pendant 30 min. Laver les cellules avec 150 μL de CyFACS. Centrifugez et agissez la plaque.
- Lavez les cellules deux fois avec 200 μL de CyFACS. Centrifugez et agissez la plaque. Remettre tous les puits en suspension dans 30 μL de CyFACS. Combinez jusqu’à six puits de cellules colorées avec des codes-barres uniques en un seul puits et effectuez la centrifugation et le basculement de la plaque.
- Coloration de surface
- Dissoudre la lyosphère de surface du panneau NK dans 50 μL de CyFACS avec des anticorps de surface supplémentaires ajoutés (anti-CD16, anti-HLA-DR, anti-LILRB1). Décongeler le panneau de ligand stocké à -80 °C et faire tourner le tube à l’aide d’une mini-centrifugeuse. Ajout de marqueurs de surface de ligand supplémentaires (anti-CD16, anti-CD19).
REMARQUE : Tout cocktail d’anticorps qui n’a pas été préalablement filtré (c’est-à-dire avant la lyophilisation ou la congélation) doit être filtré à travers une unité de filtre centrifuge (pores de 0,1 μm) à 10 600 x g pendant 3 minutes avant la coloration. - Remettez chaque puits en suspension dans 50 μL du panneau respectif. Incuber à 4 °C pendant 30 min. Laver les cellules avec 150 μL de CyFACS. Centrifugez et agissez la plaque. Laver à nouveau les cellules avec 200 μL de CyFACS. Centrifugez et agissez la plaque.
- Dissoudre la lyosphère de surface du panneau NK dans 50 μL de CyFACS avec des anticorps de surface supplémentaires ajoutés (anti-CD16, anti-HLA-DR, anti-LILRB1). Décongeler le panneau de ligand stocké à -80 °C et faire tourner le tube à l’aide d’une mini-centrifugeuse. Ajout de marqueurs de surface de ligand supplémentaires (anti-CD16, anti-CD19).
- Fixez les cellules comme décrit à l’étape 1.2.6.
REMARQUE : Effectuez tous les essorages ultérieurs de la centrifugeuse à 700 x g pendant 5 min à 4 °C. - Perméabiliser les cellules comme décrit à l’étape 1.2.7.
- Coloration intracellulaire
- Dissoudre la lyosphère intracellulaire du panneau NK dans 50 μL de tampon Perm. Préparez un cocktail d’anticorps intracellulaires pour les échantillons de PBMC si vous le souhaitez.
REMARQUE : Tout cocktail d’anticorps qui n’a pas été préalablement filtré (c’est-à-dire avant la lyophilisation ou la congélation) doit être filtré à travers une unité de filtre centrifuge (pores de 0,1 μm) à 10 600 x g pendant 3 minutes avant la coloration. - Remettre les puits en suspension dans 50 μL des panneaux intracellulaires respectifs. Si un panneau intracellulaire n’est pas utilisé en conjonction avec le panneau de surface du ligand, remettre en suspension les puits PBMC dans 50 μL du tampon Perm. Incuber à 4 °C pendant 45 min.
- Laver les cellules avec 150 μL de tampon Perm. Centrifugeuse et plaque de projection. Laver les cellules avec 200 μL de tampon Perm. Centrifugez et agissez la plaque.
- Lavez les cellules deux fois avec 200 μL de CyFACS. Centrifugez et agissez la plaque.
- Dissoudre la lyosphère intracellulaire du panneau NK dans 50 μL de tampon Perm. Préparez un cocktail d’anticorps intracellulaires pour les échantillons de PBMC si vous le souhaitez.
- Coloration de l’intercalateur d’ADN. Effectuez la coloration de l’intercalateur d’ADN comme décrit à l’étape 1.2.9.1. Incuber la plaque pendant une nuit à 4 °C.
REMARQUE : L’intercalateur se lie à l’acide nucléique cellulaire et est utilisé pour identifier les cellules nucléées en cytométrie de masse. Les plaques peuvent être conservées recouvertes d’un film de paraffine jusqu’à une semaine à 4 °C. - Avant d’analyser les échantillons sur CyTOF, laver les cellules comme décrit aux étapes 1.2.9.3 et 1.2.9.4.
- Exécutez des échantillons sur CyTOF.
Résultats
Les anticorps ont été conjugués à des isotopes métalliques à l’aide de trousses d’étiquetage disponibles dans le commerce, conformément aux instructions du fabricant. Les clones d’anticorps ont été validés par cytométrie en flux et cytométrie de masse avant d’être utilisés dans ce panel. Une première liste de clones a été sélectionnée en fonction de l’examen de la littérature et de la disponibilité des anticorps. Les niveaux d’expression de certains ligands pour les récepteurs des cellules NK sont faibles ou indétectables sur les PBMC sains. Par conséquent, la coloration positive de certains anticorps a été validée par la coloration de cellules PBMC saines, de cellules K562 de leucémie myéloïde chronique, de cellules NALM6 de leucémie lymphoblastique aiguë ou de cellules B de leucémie lymphoblastique aiguë 697 (figure supplémentaire 1). Les clones sélectionnés pour le panel de cellules NK qui n’ont pas produit une coloration adéquate ou qui étaient trop coûteux ont été substitués à d’autres, comme détaillé dans le tableau supplémentaire 1 et illustré dans la figure supplémentaire 2.
L’appariement des isotopes métalliques avec les anticorps de ces panels a été effectué à l’aide des principes décrits par Takahashi et coll.16. Les marqueurs de lignée étaient d’intensité moyenne à élevée. Par conséquent, ils étaient principalement conjugués à des masses de sensibilité faible et moyenne, laissant des masses de sensibilité élevées disponibles pour la conjugaison aux anticorps contre des marqueurs plus faiblement exprimés. Un logiciel de conception de panel accessible au public a été utilisé pour détecter les problèmes de sensibilité à l’abondance (M ± 1 purge) ou d’oxydation (M + 16 purge), et les paires anticorps-métal ont été réattribuées en conséquence. De plus, plusieurs marqueurs ont été conjugués sur différents métaux avec des différences minimes notées sur l’intensité du signal (tableau supplémentaire 2 et figure supplémentaire 3). Les appariements anticorps-métal et les informations sur les clones pour les panels NK et ligand sont répertoriés dans le tableau 1 et le tableau 2 respectivement.
Des anticorps conjugués internes ont été titrés sur des PBMC à cinq titres différents : 0,625, 1,25, 2,5, 5 et 10 μg/mL. Le titre d’anticorps le plus faible, qui a entraîné l’intensité du signal la plus élevée et la meilleure séparation entre les populations positives et négatives, a été sélectionné sur la base d’une évaluation visuelle. Les titrages des panels NK et ligand sont illustrés respectivement à la figure 1 et à la figure 2. Pour certains marqueurs, une distinction claire entre les populations positives et négatives n’a pas été identifiée, en raison du fait que le marqueur est faiblement exprimé ou universellement positif. Afin de déterminer la dilution de travail la plus précise pour ces anticorps, les titres ont été évalués sur plusieurs types de cellules (PBMC, lymphocytes T, lymphocytes B ou cellules NK), ou sur des lignées cellulaires, afin de permettre l’identification des populations cellulaires positives et négatives (figure supplémentaire 4). L’indice de coloration (IS) pour chaque marqueur n’a pas été calculé car cette mesure n’est pas applicable aux données CyTOF17,18.
Les panneaux décrits ici ont été conçus pour être compatibles avec les exemples de codes-barres. Il existe plusieurs méthodes de codage à barres disponibles pour CyTOF. Les plus couramment utilisés sont un kit à base de palladium disponible dans le commerce, qui nécessite une fixation avant le codage à barres, et la méthode de codage à barres basée sur le CD45 décrite par Mei et al.15, qui permet le codage à barres de cellules vivantes. Pour évaluer quelle méthode de codage à barres répondait le mieux à nos besoins, nous avons testé la stabilité de la coloration des marqueurs de cellules NK après fixation dans une première version du panel de cellules NK (Figure supplémentaire 5). Nous avons constaté que l’expression d’une majorité de marqueurs de cellules NK était affectée par la fixation. Par conséquent, nous avons décidé d’utiliser une méthode de codage à barres modifiée basée sur CD45 sur des cellules vivantes15. Cette méthode de codage à barres utilise 102, 104, 106et 108, et diffère de la méthode trois sur six décrite à l’origine par Mei et coll., qui utilisait 104, 106, 108, 110, 113In et 115In. Les canaux d’indium n’ont pas été inclus dans notre système de codage à barres, car ils interféraient avec le signal de 115In-CD3. 110Le n’a pas été inclus, car il interférait avec le signal du Qdot HLA-DR et du Qdot CD19 dans les panels de cellules NK et de ligands, respectivement.
Bien que nous recommandions la purification des cellules NK avant la coloration, le panel de cellules NK est conçu pour permettre le phénotypage des cellules NK à partir de PBMC entiers. Un exemple de notre stratégie de contrôle des cellules NK est illustré à la figure 3A en utilisant des PBMC provenant d’un donneur sain. La coloration et les portes de chacun des marqueurs NK sont illustrées sur des cellules NK saines et isolées sur la figure 3B. Le panel de ligands est conçu pour détecter l’expression des ligands des cellules NK sur des PBMC entiers. La figure 4A illustre la stratégie de contrôle utilisée pour identifier les lymphocytes T CD4+ , les lymphocytes T CD8+ , les cellules NK, les monocytes et les lymphocytes B CD19+ dans les PBMC d’un donneur sain. Des exemples de coloration représentatifs pour chaque ligand sont présentés à la figure 4B à l’aide de PBMC de patients atteints de dengue aiguë et de personnes infectées par le VIH dont la charge virologique a été supprimée.
Pour assurer la stabilité du panneau dans le temps, notre protocole comprend deux options possibles : la lyophilisation par une société tierce en billes à usage unique ou la congélation d’aliquotes préfabriquées à -80 °C. Pour ce protocole, le panel NK a été lyophilisé et le panel de ligands a été congelé. Les deux méthodes ont été validées avant d’utiliser chaque panel sur des échantillons cliniques.
Nous avons produit plus de 700 réactions du panel NK à partir d’un mélange maître en effectuant plusieurs conjugaisons de chaque anticorps dans le panel. Après validation et titrage de chaque anticorps conjugué, les anticorps ont été combinés en un mélange maître, filtrés à travers une unité de filtre à seringue stérile de 0,1 μm et envoyés à une société tierce pour lyophilisation. Deux ensembles de lyosphères à coloration unique ont été fabriqués, l’un pour la coloration de surface et l’autre pour la coloration intracellulaire. Les anticorps non conjugués en interne (HLA-DR et CD16) n’ont pas pu être ajoutés à la lyosphère, en raison de la présence d’un stabilisateur d’anticorps, qui interfère avec le processus de lyophilisation. Ces anticorps sont ajoutés au panel le jour de la coloration. Une comparaison entre les colorations obtenues avant et après lyophilisation est présentée à la figure supplémentaire 6. Le clone de l’anticorps LILRB1 initialement utilisé dans les lyosphères n’a pas produit une coloration suffisamment forte (tableau supplémentaire 1 et figure supplémentaire 2). Un meilleur clone a ensuite été identifié, conjugué et ajouté au panel le jour de la coloration (tableau 1). Il a été noté que l’anticorps polyclonal KIR2DS2 utilisé dans les lyopshères produisait une coloration non spécifique après lyophilisation et nous ne recommandons pas son utilisation pour des analyses ultérieures (tableau supplémentaire 1 et figure supplémentaire 2). La plupart des colorations intracellulaires ont légèrement augmenté en intensité après la lyophilisation (Figure supplémentaire 6).
Avant de stocker le mélange de ligand à -80 °C, nous avons testé deux conditions de stockage différentes. Nous avons préparé un mélange maître plus petit de ce panneau et stocké les aliquotes à -80 °C et dans de l’azote liquide pendant environ deux mois. Au bout de deux mois, nous avons coloré des PBMC entiers avec les aliquotes congelées. Nous avons comparé la coloration à celle des PBMC du même donneur coloré avec le panel fraîchement préparé (figure supplémentaire 7). Nous avons constaté que le stockage à -80 °C et dans l’azote liquide ne modifie pas l’intensité du signal pour la plupart des marqueurs. En effet, l’intensité du signal anti-pan HLA classe I, anti-CD7, anti-CD4, anti-HLA-Bw4, anti-CD14, anti-CD11b et anti-LFA-3 est plus élevée lors de la congélation, en particulier dans le cas d’échantillons stockés à -80 °C. Nous n’avons pas pu déterminer si les intensités des signaux anti-LLT-1, anti-Nectine-1, anti-MICA/B, anti-DR4/5, anti-ULBP-1,2,5,6, anti-Nectine-2, anti-CD155 et anti-B7-H6 étaient affectées par la congélation, en raison du fait que les PBMC sains n’expriment pas des niveaux élevés de ces marqueurs. Cependant, la validation de ces marqueurs sur des lignées cellulaires (Figure supplémentaire 1) a été effectuée à l’aide d’anticorps conjugués stockés à -80 °C. Par conséquent, nous étions convaincus que le gel n’entraînait pas de perte significative de signal. L’intensité du signal a diminué lors de la congélation pour cinq marqueurs : anti-CD8, anti-ICAM-1, anti-CCR2, anti-CD33 et anti-CD56. Cependant, dans tous ces cas, la séparation nette entre les populations positives et négatives est restée. Étant donné que les anticorps conjugués aux métaux ne sont pas stables à 4 °C pendant de longues périodes, la congélation était nécessaire pour préserver la stabilité du panneau à long terme, et malgré une diminution de l’intensité de la coloration dans un sous-ensemble de marqueurs, nous avons pu conserver une séparation de coloration suffisante. Il est important de noter que la perte d’intensité du signal d’anti-CD8, d’anti-CCR2 et d’anti-CD56 était plus importante dans les échantillons stockés dans de l’azote liquide que dans ceux stockés à -80 °C. Sur la base de ces données, nous avons décidé de stocker le panneau à -80 °C.
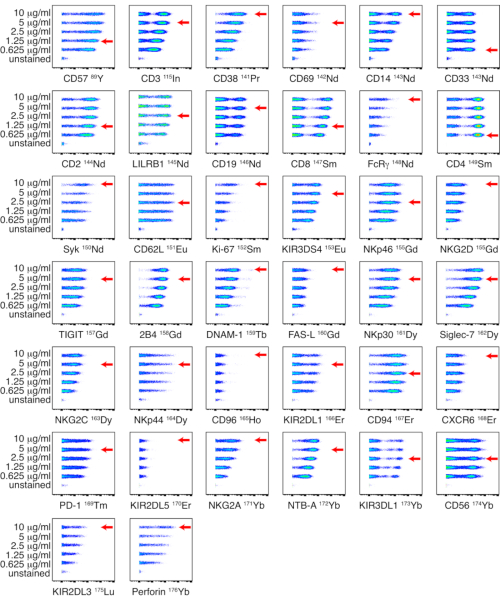
Figure 1 : Titrage des conjugués anticorps-métal conjugués en interne pour le panel NK. Des titrages d’anticorps conjugués internes ont été effectués sur des PBMC provenant d’un donneur sain à l’aide de cinq concentrations différentes : 0,625, 1,25, 2,5, 5 et 10 μg/mL. Les titres anti-CD3, anti-CD14, anti-CD33, anti-CD19, anti--1 et anti-CD56 ont été déterminés par contrôle sur des cellules vivantes. Les titres anti-CD4 et anti-CD8 ont été déterminés par gate sur les lymphocytes T. Les titres des anticorps restants ont été déterminés par contrôle sur les cellules NK. Étant donné que NKp44 n’est pas exprimé sur les cellules NK au repos, les titres ont été déterminés sur les PBMC stimulés par l’IL-2 et présentés sur les cellules NK. Les flèches rouges indiquent le titre sélectionné pour chaque anticorps. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Titrage des anticorps du panel de ligands conjugués en interne. Des titrages d’anticorps conjugués internes ont été effectués sur des PBMC provenant d’un donneur sain à l’aide de cinq concentrations différentes : 0,625, 1,25, 2,5, 5 et 10 μg/mL. Les titres anti-HLA-DR, anti-ICAM-1, anti-CCR2, anti-CD14, anti-CD11b et anti-LFA-3 ont été déterminés par déclenchement sur des cellules CD3-CD7-. Les titres anti-CD3, anti-pan HLA classe I, anti-CD7, anti-CD48, anti-LLT-1, anti-HLA-C,E, anti-HLA-E, anti-FasR, anti-Nectine-1, anti-MICA/MICB, anti-DR4/DR5, anti-ULBP-1,2,5,6, anti-Nectine-2, anti-CD155, anti-HLA-Bw4, anti-HLA-Bw6, anti-CD33, anti-CD56 et anti-B7-H6 ont été déterminés par contrôle sur des cellules vivantes. Les titres anti-CD4 et anti-CD8 ont été déterminés par déclenchement sur des cellules CD3+. Les flèches rouges indiquent le titre sélectionné pour chaque anticorps. Veuillez cliquer ici pour voir une version agrandie de cette figure.
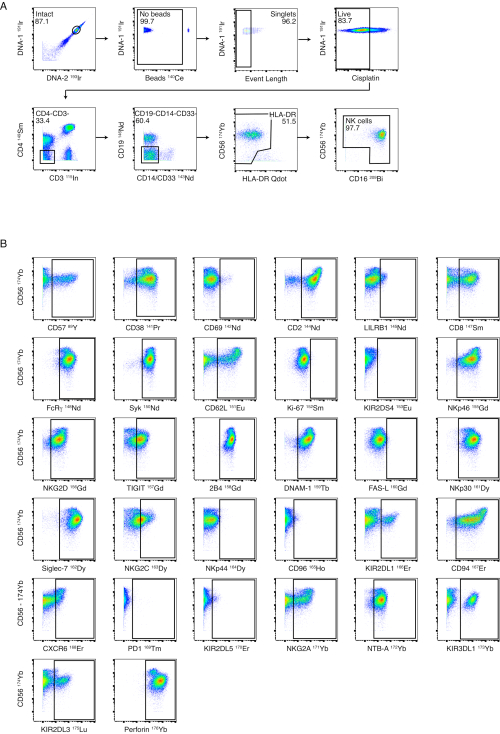
Figure 3 : Stratégie et performance du contrôle du panel NK. (A) Le déclenchement négatif en série de PBMC entiers vers des cellules NK est montré chez un donneur sain. Les portes intactes, à billes et à longueur d’événement garantissent une fermeture réussie aux cellules individuelles. La coloration au cisplatine a été réalisée sous forme de coloration vivante/morte. Les lymphocytes T et B ont été exclus à l’aide des CD3 et CD19. Les monocytes ont été exclus par un déclenchement négatif sur CD4 et CD14/CD33 et par un autre déclenchement négatif sur les cellulesbrillantes CD56-/HLA-DR. CD56 et CD16 ont été utilisés pour identifier différents sous-ensembles de cellules NK (CD56bright, CD56dim et CD56-). (B) Exemples d’expression des récepteurs des cellules NK sur les cellules NK d’un donneur sain purifié par isolement par billes magnétiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Déclenchement et performance du panel de ligands. (A) Déclenchement des principaux sous-ensembles de cellules immunitaires à partir de PBMC dérivés d’un donneur sain après normalisation, retrait des billes d’étalonnage et décodage à barres. (B) Expression des ligands des récepteurs des cellules NK ainsi que de plusieurs marqueurs myéloïdes sur les PBMC vivants. La coloration de tous les ligands sauf la Nectine-1 et la B7-H6 est mise en évidence sur les PBMC de patients atteints de dengue aiguë. La coloration pour la Nectine-1 et la B7-H6 est mise en évidence sur les PBMC de personnes infectées par le VIH dont la charge virale a été supprimée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Vérification des clones d’anticorps pour le panel de ligands sur des ligands cellulaires. Les anticorps dirigés contre les ligands des récepteurs des cellules NK qui sont exprimés à de faibles niveaux sur les PBMC sains ont été validés par coloration de lignées cellulaires. Les cellules de la leucémie myéloïde chronique K562 ont été colorées avec des anti-ICAM-1, anti-MICA/MICB, anti-DR4/DR5, anti-ULBP-1,2,5,6, anti-Nectine-2, anti-CD155 et anti-B7-H6. Les cellules NALM6 de la leucémie lymphoblastique aiguë ont été colorées avec l’anti-LLT-1 et les cellules de la leucémie lymphoblastique aiguë 697 ont été colorées avec l’anti-Nectin-1. Les diagrammes à points et les histogrammes montrant la coloration sur les PBMC sains sont en bleu. Les diagrammes à points et les histogrammes montrant la coloration sur la lignée cellulaire respective sont en rouge. Le pourcentage de cellules de la lignée cellulaire respective qui sont positives pour un marqueur donné est fourni. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Validation des clones d’anticorps. (A) Différents clones d’anticorps ont été testés chez des donneurs sains afin d’identifier le clone présentant la meilleure spécificité. 2B4, CXCR6, KIR2DS4, NKG2A et TIGIT sont affichés sur les cellules NK. CD56 et LILRB1 sont représentés sur des cellules vivantes. (B) Le clone de l’anticorps KIR2DS2 a montré une coloration non spécifique après lyophilisation. Un exemple de coloration sur le même donneur est fourni avant et après la lyophilisation. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Optimisation des paires anticorps/métal. La coloration des PBMC de donneurs sains est montrée. (A) Paires anticorps/métal testées pour le panneau. (B) Paires anticorps/métal utilisées dans le panneau. LILRB1 et PD1 sont représentés sur des cellules vivantes. Tous les autres marqueurs sont affichés sur les cellules NK. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4 : Titrage de marqueurs de cellules NK faiblement exprimés et généralement positifs. Les titres d’anticorps dirigés contre les marqueurs des cellules NK qui n’ont pas montré une population positive et négative claire ont été évalués à la fois sur les cellules NK (rouge) et sur les cellules B (bleues) ou les PBMC (gris) de donneurs sains. Les flèches indiquent le titre sélectionné pour chaque anticorps. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5 : Optimisation du protocole de codage à barres. La stabilité de l’épitope a été testée avant et après la fixation avec 2 % de paraformaldéhyde sur des PBMC provenant d’un donneur sain. (A) La coloration des CD3, CD14 et CD56 était similaire avant la fixation (rouge) et après la fixation (bleu), la coloration des CD4 et CD16 était significativement affectée par la fixation. (B) De nombreux marqueurs de cellules NK ont été affectés par la fixation, notamment CD2, CD38, KIR3DL2, CD62L, KIR2DS4, NKp46, NKG2C, NKp30, NKG2D, KIR3DL1, TIGIT, KIR2DL1, KIR2DL3 et NTB-A. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 6 : Confirmation de la stabilité du panel après lyophilisation. La stabilité des conjugués d’anticorps internes a été confirmée par la coloration des PBMC du même donneur de banque de sang avant la lyophilisation (en bleu) et après la lyophilisation (en rouge). Des colorations anti-CD3, anti-CD14, anti-CD33, anti-CD19, anti--1, anti-CD56 sont observées sur des cellules vivantes. Les anticorps anti-CD4 et anti-CD8 sont indiqués sur les cellules CD3+ . Les titres des anticorps restants sont indiqués sur les cellules NK, contrôlées selon le schéma de contrôle illustré à la figure 1. Notamment, l’anti-KIR2DS2 coloré de manière non spécifique après lyophilisation et n’a donc pas été utilisé pour des analyses ultérieures. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 7 : Confirmation de la stabilité du ligand à -80 °C. La stabilité des conjugués d’anticorps internes a été confirmée par la coloration de PBMC sains provenant du même donneur avec un mélange maître fraîchement préparé ainsi que le même mélange après stockage à -80°C ou dans de l’azote liquide. La coloration anti-HLA-DR, anti-ICAM-1 et anti-LFA-3 est mise en évidence sur les cellules CD3-CD7-. La coloration anti-CD3, anti-pan HLA classe I, anti-CD7, anti-CD48, anti-LLT-1, anti-HLA-E, anti-FasR, anti-Nectine-1, anti-MICA/B, anti-DR4/5, anti-ULBP-1,2,5,6, anti-Nectine-2, anti-CD155, anti-HLA-Bw4, anti-HLA-Bw6 et anti-B7-H6 est illustrée sur des cellules vivantes. Une coloration anti-CD8 et anti-CD4 est mise en évidence sur les cellules CD3+. Les colorations anti-HLA-C,E, anti-CCR2, anti-CD11b et anti-CD33 sont observées sur les cellules CD3-CD7-CD14+. La coloration anti-CD14 est mise en évidence sur les cellules CD3-CD7-CD33+ et la coloration anti-CD56 sur les cellules CD3-CD7+CD14-HLA-DR. Les histogrammes montrant la coloration avec le panneau fraîchement préparé sont en rouge. Les histogrammes montrant des taches avec le panneau après un stockage à -80°C pendant environ deux mois sont en bleu. Les histogrammes montrant une coloration au panel après stockage dans de l’azote liquide (LN2) pendant environ deux mois sont en vert. Les échantillons ont été colorés et analysés à des jours différents. Les fichiers ont été normalisés et les perles ont été retirées à l’aide du package premessa. Veuillez cliquer ici pour télécharger ce fichier.
| Spécificité | Clone | Isotope | But | Surface/Intracellulaire |
| CD57 | HCD57 | 89Y | Maturité/Mémoire | Surface |
| CD45 | HI30 | 102, 104, 106, 108 | Codes à barres | Surface |
| HLA-DR | Tü36 | Qdot 655 (112 cd-114cd) | Activation/Lignage | Surface |
| CD3 | UCHT | 115Dans | Lignée des lymphocytes T | Surface |
| CD38 | HIT2 | 141Pr | Marqueur d’activation | Surface |
| CD69 | FN50 | 142Nd | Marqueur d’activation | Surface |
| CD33 | WM53 | 143Nd | Lignée des monocytes | Surface |
| CD14 | M5E5 | 143Nd | Lignée myéloïde | Surface |
| CD2 | RPA-2.10 | 144Nd | Activation/Maturité | Surface |
| LILRB1 | MAB20172 | 145Nd | Récepteur inhibiteur | Surface |
| CD19 | HIB19 | 146Nd | Lignée des lymphocytes B | Surface |
| CD8 | SK1 | 147Sm | Lignées de lymphocytes T et activation/maturité des lymphocytes NK | Surface |
| FcRg | polyclonal | 148Nd | Maturité/Adaptatif | intracellulaire |
| CD4 | SK3 | 149Sm | Lignée des lymphocytes T | Surface |
| Syk | 4D10.2 | 150Nd | Signalisation | intracellulaire |
| CD62L | DREG-56 | 151UE | Activation | Surface |
| Ki-67 | Ki-67 | 152Sm | Prolifération | intracellulaire |
| KIR2DS4 | 179315 | 153UE | Récepteur d’activation | Surface |
| NKp46 | 9.00E+02 | 155Gd | Récepteur d’activation | Surface |
| NKG2D | 1D11 | 156Gd | Récepteur d’activation | Surface |
| TIGIT | 741182 | 157Gd | Récepteur inhibiteur | Surface |
| 2B4 | C1.7 | 158Gd | Récepteur d’activation | Surface |
| L’ADN 1 | DX11 | 159Tuberculose | Récepteur d’activation | Surface |
| SAF-L | NOK-1 | 160Gd | Apoptose | Surface |
| NKp30 | P30-15 | 161Dy | Récepteur d’activation | Surface |
| Siglec-7 | S7.7 | 162Dy | Récepteur inhibiteur | Surface |
| NKG2C | 134522 | 163Dy | Maturité/Mémoire | Surface |
| NKp44 | P44-8 (en anglais) | 164Dy | Récepteur d’activation | Surface |
| CD96 | NK92.39 | 165HO | Corécepteur NKG2 | Surface |
| KIR2DL1/KIR2DS5 | 143211 | 166Er | Récepteur inhibiteur | Surface |
| CD94 | DX22 | 167Er | Récepteur d’activation | Surface |
| CXCR6 | Référence K041E5 | 168Er | Mémoire | Surface |
| PD1 | EH12.2H7 | 169Tm | Récepteur inhibiteur | Surface |
| KIR2DL5 | UP-R1 | 170Er | Récepteur inhibiteur | Surface |
| NKG2A | 131411 | 171Yb | Récepteur inhibiteur | Surface |
| NTB-A | NT-7 | 172Tuberculose | Récepteur d’activation | Surface |
| KIR3DL1 | DX-9 | 173Yb | Récepteur inhibiteur | Surface |
| CD56 | NCAM16.2 | 174Yb | Lignée cellulaire NK | Surface |
| KIR2DL3 | 180701 | 175Lu | Récepteur inhibiteur | Surface |
| Perforine | B-D48 | 176Yb | Protéine cytolytique | intracellulaire |
| ADN-1/ADN-2 | NA | 191IR/193IR | Cellules nucléées | Surface |
| Cisplatine | NA | 194Pt/195pt | Viabilité | Surface |
| CD16 | 3G8 | 209Bi | Récepteur FcgRIII | Surface |
Tableau 1 : Panneau NK. Les marqueurs sont ordonnés en fonction de la masse isotopique du métal auquel ils ont été conjugués. 191Ir/193Ir est l’abondance naturelle de l’intercalateur d’acide nucléique. 194Pt/195Pt est l’abondance naturelle du cisplatine.
| Spécificité | Clone | Isotope | But | Surface/Intracellulaire |
| HLA-DR | L243 | 89Y | Cellules présentatrices d’antigène, marqueur d’activation | Surface |
| CD45 | HI30 | 102, 104, 106, 108 | Codes à barres | Surface |
| CD19 | SJ25-C1 | Qdot 655 (112 cd-114cd) | Lignage | Surface |
| CD3 | UUCHT1 | 115Dans | Lignage | Surface |
| Pan HLA classe I | W6/32 | 143Nd | Ligands KIR | Surface |
| CD7 | CD7-6B7 (en anglais) | 144Nd | Lignage | Surface |
| CD8 | SK1 | 145Nd | Lignage | Surface |
| CD48 | BJ40 | 146Nd | Ligand 2B4 | Surface |
| ICAM-1 | HA58 | 148Nd | Ligand LFA-1 | Surface |
| LLT-1 | 402659 | 149Sm | Ligand CD161 | Surface |
| CD4 | OKT4 | 151UE | Lignage | Surface |
| HLA-C,E | DT9 | 153UE | Ligands KIR | Surface |
| CCR2 | K036C2 | 154Sm | Marqueur fonctionnel monocytaire | Surface |
| HLA-E | 3D12 | 155Gd | Ligand NKG2A/CD94 et NKG2C/CD94 | Surface |
| Fas (CD95) | DX2 | 156Gd | Récepteur FasL | Surface |
| Nectine-1 | R1.302 | 157Gd | Ligand CD96 | Surface |
| MICA/B | 159227/236511 | 158Gd | Ligands NKG2D | Surface |
| DR4/5 | DJR1/DJR2-2 | 159Tuberculose | Récepteurs TRAIL | Surface |
| ULBP-1/2,5,6 | 170818/165903 | 161Dy | Ligands NKG2D | Surface |
| Nectine-2 | TX31 | 164Dy | Ligand DNAM-1, TIGIT et CD96 | Surface |
| CD155 | SKII.4 | 165HO | Ligand DNAM-1, TIGIT et CD96 | Surface |
| HLA-BW4 | REA274 | 166Er | KIR3DL1 ligand | Surface |
| HLA-BW6 | REA143 | 168Er | Allèle nul KIR | Surface |
| CD14 | M5E2 | 169Tm | Lignage | Surface |
| CD11b | ICRF44 | 170Er | Lignage | Surface |
| ZPH-3 | TS2/9 | 171Yb | Ligand CD2 | Surface |
| CD33 | WM53 | 172Yb | Lignage | Surface |
| CD56 | NCAM16.2 | 174Yb | Lignage | Surface |
| B7-H6 | 875001 | 176Yb | Ligand NKp30 | Surface |
| ADN-1/ADN-2 | NA | 191IR/193IR | Cellules nucléées | Surface |
| Cisplatine | NA | 194Pt/195pt | Viabilité | Surface |
| CD16 | 3G8 | 209Bi | Lignage | Surface |
Tableau 2 : Panneau de ligands. Les marqueurs sont ordonnés en fonction de la masse isotopique du métal auquel ils ont été conjugués. 191Ir/193Ir est l’abondance naturelle de l’intercalateur d’acide nucléique. 194Pt/195Pt est l’abondance naturelle du cisplatine.
| Spécificité | Clone | Vendeur | Numéro de catalogue | Surface/ Intracellulaire | Notes |
| 2B4 | 2-69 | BD Biosciences | 550814 | Surface | Nouveau clone validé avec une coloration améliorée |
| CD56 | N901 | Beckman Coulter | 6602705 | Surface | Nouveau clone validé avec une coloration améliorée |
| CXCR6 | 56811 | Systèmes de R&D | MAB699 | Surface | Nouveau clone validé à moindre coût |
| KIR2DS2 | polyclonal | Abcam | AB175486 | Surface | Coloration non spécifique constatée après lyophilisation - non utilisée pour les analyses |
| KIR2DS4 | FES172 | Beckman Coulter | N° A60796 | Surface | Nouveau clone validé avec une coloration améliorée/un coût réduit |
| LILRB1 | GHI/75 | Biolégende | 333702 | Surface | Nouveau clone validé avec une coloration améliorée |
| NKG2A | Z199 | Beckman Coulter | IM2750 | Surface | Nouveau clone validé à moindre coût |
| TIGIT | MBSA43 | Thermo Fisher Scientific | 16-9500-82 | Surface | Nouveau clone validé |
Tableau supplémentaire 1 : Anticorps pour le panel de cellules NK qui ont été testés, mais qui n’ont pas été utilisés.
| Isotope | Spécificité | Clone | Vendeur | Numéro de catalogue | Surface/ Intracellulaire |
| 143Nd | NKG2C | 134522 | Systèmes de R&D | MAB1381 | Surface |
| 145Nd | CD38 | HIT2 | Biolégende | 303502 | Surface |
| 146Nd | CD8 | SK1 | Biolégende | 344702 | Surface |
| 149Sm | CD2 | RPA-2.10 | Biolégende | 300202 | Surface |
| 151UE | Siglec-7 | S7.7 | Biolégende | 347702 | Surface |
| 154Sm | LILRB1 | 292319 | Systèmes de R&D | MAB20172 | Surface |
| 163Dy | KIR3DL1 | DX-9 | BD biosciences | 555964 | Surface |
| 168Er | CD62L | DREG-56 | Biolégende | 304802 | Surface |
| 171Yb | PD1 | EH12.2H7 | Biolégende | 329902 | Surface |
| 176Yb | CD69 | EH12.2H7 | Biolégende | 329902 | Surface |
Tableau supplémentaire 2 : Anticorps pour le panel de cellules NK qui ont été testés avec une appariement anticorps/métal différente.
Discussion
Nous décrivons ici la conception et l’application de deux panels CyTOF complémentaires visant à profiler le répertoire récepteur-ligand des cellules NK. Ce protocole comprend plusieurs étapes essentielles à l’obtention de données de qualité. CyTOF utilise des ions de métaux lourds, plutôt que des fluorochromes, comme sondes de marquage pour les anticorps19. Cette technologie est donc soumise à des signaux potentiellement contaminants provenant des métaux environnementaux20. Les sources potentielles d’impuretés métalliques comprennent le savon à vaisselle de laboratoire (baryum) et les tampons de laboratoire (mercure, plomb, étain). Pour cette raison, il est conseillé de préparer tous les tampons avec de l’eau ultra-pure et de stocker tous les réactifs dans des récipients en plastique ou en verre qui n’ont jamais été lavés avec du savon. Une autre étape critique de ce protocole est la coloration de viabilité, qui utilise une méthode à base de cisplatine telle que décrite par Fienberg et al.21. Cette méthode comprend une étape d’incubation d’une minute, au cours de laquelle le cisplatine marque préférentiellement les cellules non viables. La coloration au cisplatine doit être effectuée en l’absence de FBS. Par conséquent, les cellules doivent être soigneusement lavées avec du CyPBS avant la coloration. De plus, pour éviter la coloration hors cible des cellules viables, la coloration au cisplatine doit être éteinte avec du FBS après précisément une minute. Ce protocole a été optimisé pour une récupération cellulaire maximale et des performances de coloration. Par conséquent, les étapes de fixation et de perméabilisation sont également importantes. Plusieurs réactifs de fixation et de perméabilisation sont compatibles avec le CyTOF. Cependant, nous avons constaté que la fixation avec 2 % de PFA, suivie d’une perméabilisation avec un tampon de perméabilisation spécifique détaillé dans la Table des matériaux, entraînait une récupération cellulaire maximale. Comme il s’agit d’une méthode de perméabilisation transitoire, une coloration intracellulaire doit être effectuée dans le tampon de perméabilisation pour assurer une pénétrance adéquate des anticorps. Les cellules doivent également être soigneusement lavées avec le tampon de perméabilisation après la coloration intracellulaire pour éliminer les anticorps non liés.
Ce protocole permet plusieurs modifications possibles. Les panneaux CyTOF détaillés ici peuvent être personnalisés pour inclure des marqueurs supplémentaires ou remplacer ceux existants. En particulier, le panneau de ligand a été conçu avec plusieurs canaux ouverts pour permettre une flexibilité dans la conception du panneau. Toute modification ou ajout au panneau peut nécessiter un dépannage supplémentaire. En particulier, toute paire anticorps/isotope métallique doit être validée de manière approfondie, comme décrit ci-dessus, afin d’éviter tout problème de débordement du signal vers les canaux existants. Ces panneaux ont également été conçus pour être compatibles avec les codes-barres d’échantillons. Le codage à barres réduit la possibilité d’effets de lot et de transfert d’échantillon à échantillon, tout en minimisant la consommation deréactifs 22. Bien que le codage à barres entraîne généralement une amélioration globale de la qualité des données, cette étape n’est pas nécessaire pour l’acquisition de données CyTOF de bonne qualité et peut être entièrement ignorée. De même, bien que nous recommandions la purification des cellules NK avant la coloration, le panel de cellules NK est compatible avec le phénotypage des cellules NK à partir de PBMC entiers.
Cette méthode présente certaines limites. En raison de la nature intrinsèquement faible du CyTOF, cette méthode ne convient pas aux échantillons à faible nombre de cellules. Il est peu probable que de tels échantillons produisent des données de qualité suffisante pour l’analyse. De plus, étant donné que ces panneaux ont été spécifiquement conçus pour interroger les interactions entre les cellules NK et les cellules cibles, leur capacité à évaluer les interactions entre d’autres types de cellules, telles que les lymphocytes T CD8+ et les cellules myéloïdes, sont limitées. De même, ces panels ont été conçus pour l’immunophénotypage direct ex vivo et n’ont pas été testés ou validés pour une utilisation dans des conditions d’activation, telles que la stimulation par cytokines. De plus, bien que ces panels couvrent une liste complète de récepteurs et de ligands de cellules NK, ils ne sont pas entièrement exhaustifs, et plusieurs marqueurs potentiellement importants n’ont pas été inclus en raison de contraintes d’espace. Certains de ces marqueurs incluent, sans s’y limiter, KLRG1, CRACC, TIM-3, LAIR-1 dans le panel NK et-L1 dans le panel de ligands. Enfin, cette méthode n’est pas adaptée à une utilisation dans des échantillons fixés, étant donné que la majorité des épitopes des marqueurs de cellules NK sont affectés par fixation.
Le protocole décrit ici présente des avantages significatifs par rapport à d’autres méthodes. D’autres groupes ont décrit des panels de cytométrie en flux destinés à l’étude des cellules NK3, 4, 5, 6, 7, 8, 23. Par rapport à la cytométrie en flux, l’utilisation de CyTOF élimine les problèmes liés à la compensation des fluorophores, permettant la détection simultanée d’un grand nombre de marqueurs. Bien que d’autres aient également développé des panels CyTOF pour étudier les cellules NK9, 10, 11, 12, 13, 14, nous décrivons ici l’utilisation de deux panels CyTOF complémentaires, qui interrogent l’expression des récepteurs des cellules NK et de leurs ligands, fournissant ainsi une image plus détaillée de la fonction des cellules NK.
Notre groupe a utilisé ce protocole et l’un ou les deux pour caractériser la réponse des cellules NK humaines chez des donneurs sains et dans divers contextes pathologiques, y compris l’infection par le VIH et l’infection par le virus de la dengue 24,25,26,27,28. Bien qu’ils aient été conçus dans le but d’étudier les infections virales, ces panels se prêtent à l’étude des cellules NK dans d’autres conditions compte tenu de l’haleine des protéines qu’ils recouvrent. En fait, notre groupe a également utilisé ces panels pour caractériser le répertoire récepteur-ligand des cellules NK chez les patients immunodéficients et atteints de sclérose en plaques 25,27,29,30, ainsi que chez les souris humanisées31,32. En tant que tel, l’utilisation de ces panneaux peut être étendue à d’autres contextes. Par exemple, de nombreux récepteurs des cellules NK et ligands apparentés impliqués dans le cadre du cancer sont inclus dans ces panels, ce qui en fait d’excellents outils pour les études futures sur le rôle des cellules NK dans la réponse antitumorale. Plus largement, notre protocole de production et de stockage en série de panels CyTOF ainsi que de traitement parallèle d’échantillons peut être appliqué à l’exécution et à l’application de n’importe quel panel CyTOF.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs tiennent à remercier tous les membres actuels et anciens du Blish Laboratory qui ont contribué à ce panel. Merci au Groupe d’essais cliniques sur le sida et à l’équipe ACTG A5321, ainsi qu’à la Dre Sandra López-Vergès et à Davis Beltrán de l’Institut Gorgas Memorial d’études sur la santé pour la conservation des échantillons. Enfin, merci à Michael Leipold, Holden Maecker et au Centre de surveillance immunitaire humaine de Stanford pour l’utilisation de leurs machines Helios. Ce travail a été soutenu par NIH U19AI057229, NIH R21 AI135287, NIH R21 AI130532, NIH DP1 DA046089 et Burroughs Wellcome Fund Investigators in the Pathogenesis of Infectious Diseases #1016687 to CB, NIH Ruth L. Kirschstein Institutional National Research Service Award T32 AI007502, TL1 TR001084 et NIH/NIAID K08 AI138640 à EV, National Science Foundation Graduate Research Fellowship DGE-1656518 to JM et NIH training grant T32-AI-007290 (PI Olivia Martinez). L’étude de l’ACTG a reçu une subvention de AI-68634 (Centre de gestion des statistiques et des données), UM1-A1-26617, AI-131798 et AI-68636 (ACTG). CB est chercheur à la faculté Tashia et John Morgridge en médecine translationnelle pédiatrique de l’Institut de recherche sur la santé maternelle et infantile de Stanford et chercheur au Chan Zuckerberg Biohub.
matériels
| Name | Company | Catalog Number | Comments |
| 89Y | Sigma-Aldrich | 204919 | |
| 102-Palladium nitrate | Trace Sciences International | Special Order | |
| 104-Palladium nitrate | Trace Sciences International | Special Order | |
| 106-Palladium nitrate | Trace Sciences International | Special Order | |
| 108-Palladium nitrate | Trace Sciences International | Special Order | |
| 115In | Trace Sciences International | Special Order | |
| 141Pr | Fluidigm | 201141A | |
| 142Nd | Fluidigm | 201142A | |
| 143Nd | Fluidigm | 201143A | |
| 144Nd | Fluidigm | 201144A | |
| 145Nd | Fluidigm | 201145A | |
| 146Nd | Fluidigm | 201146A | |
| 147Sm | Fluidigm | 201147A | |
| 148Nd | Fluidigm | 201148A | |
| 149Sm | Fluidigm | 201149A | |
| 150Nd | Fluidigm | 201150A | |
| 151Eu | Fluidigm | 201151A | |
| 152Sm | Fluidigm | 201152A | |
| 153Eu | Fluidigm | 201153A | |
| 154Sm | Fluidigm | 201154A | |
| 155Gd | Fluidigm | 201155A | |
| 156Gd | Fluidigm | 201156A | |
| 157Gd | Trace Sciences International | N/A | |
| 158Gd | Fluidigm | 201158A | |
| 159Tb | Fluidigm | 201159A | |
| 160Gd | Fluidigm | 201160A | |
| 161Dy | Fluidigm | 201161A | |
| 162Dy | Fluidigm | 201162A | |
| 163Dy | Fluidigm | 201163A | |
| 164Dy | Fluidigm | 201164A | |
| 165Ho | Fluidigm | 201165A | |
| 166Er | Fluidigm | 201166A | |
| 167Er | Fluidigm | 201167A | |
| 168Er | Fluidigm | 201168A | |
| 169Tm | Fluidigm | 201169A | |
| 170Er | Fluidigm | 201170A | |
| 171Yb | Fluidigm | 201171A | |
| 172Yb | Fluidigm | 201172A | |
| 173Yb | Fluidigm | 201173A | |
| 174Yb | Fluidigm | 201174A | |
| 175Lu | Fluidigm | 201175A | |
| 176Yb | Fluidigm | 201176A | |
| 209Bi anti-CD16 | Fluidigm | 3209002B | Clone 3G8. Used at a 1:50 dilution. |
| 697 cells | Creative Bioarray | CSC-C0217 | |
| Amicon Ultra Centrifugal Filter Units 0.5 with Ultracel-30 Membrane, 30 kDa | Millipore | UFC503096 | |
| Anhydrous acetonitrile | Fisher Scientific | BP1165-50 | |
| anti-2B4 | Biolegend | 329502 | Clone C1.7. |
| anti-B7-H6 | R&D Systems | MAB7144 | Clone 875001. |
| anti-CCR2 | Biolegend | 357202 | Clone K036C2. |
| anti-CD2 | Biolegend | 300202 | Clone RPA-2.10. |
| anti-CD3 | Biolegend | 300402 | Clone UCHT1. |
| anti-CD4 | Biolegend | 317402 | Clone OKT4. |
| anti-CD4 | Biolegend | 344602 | Clone SK3. |
| anti-CD7 | Biolegend | 343102 | Clone CD7-6B7. |
| anti-CD8 | Biolegend | 344702 | Clone SK1. |
| anti-CD11b | Biolegend | 301302 | Clone ICRF44. |
| anti-CD14 | Biolegend | 301802 | Clone M5E2. |
| anti-CD19 | Biolegend | 302202 | Clone HIB19. |
| anti-CD33 | Biolegend | 303402 | Clone WM53. |
| anti-CD38 | Biolegend | 303502 | Clone HIT2. |
| anti-CD48 | Biolegend | 336702 | Clone BJ40. |
| anti-CD56 | BD Pharmingen | 559043 | Clone NCAM16.2. |
| anti-CD57 | Biolegend | 322302 | Clone HCD57. |
| anti-CD62L | Biolegend | 304802 | Clone DREG-56. |
| anti-CD69 | Biolegend | 310902 | Clone FN50. |
| anti-CD94 | Biolegend | 305502 | Clone DX22. |
| anti-CD95 | Biolegend | 305602 | Clone DX2. |
| anti-CD155 | Biolegend | 337602 | Clone SKII.4. |
| anti-CXCR6 | Biolegend | 356002 | Clone K041E5. |
| anti-DNAM-1 | BD Biosciences | 559787 | Clone DX11. |
| anti-DR4 | Biolegend | 307202 | Clone DJR1. |
| anti-DR5 | Biolegend | 307302 | Clone DJR2-2. |
| anti-FAS-L | Biolegend | 306402 | Clone NOK-1. |
| anti-FcRg | Millipore | 06-727 | Polyclonal antibody. |
| anti-HLA-C,E | Millipore | MABF233 | Clone DT9. |
| anti-HLA-Bw4 | Miltenyi Biotec | Special Order | Clone REA274. |
| anti-HLA-Bw6 | Miltenyi Biotec | 130-124-530 | Clone REA143. |
| anti-HLA-DR | Biolegend | 307602 | Clone L243. |
| anti-HLA-E | Biolegend | 342602 | Clone 3D12. |
| anti-ICAM-1 | Biolegend | 353102 | Clone HA58. |
| anti-Ki-67 | Biolegend | 350502 | Clone Ki-67. |
| anti-KIR2DL1/KIR2DS5 | R&D Systems | MAB1844 | Clone 143211. |
| anti-KIR2DL3 | R&D Systems | MAB2014 | Clone 180701. |
| anti-KIR2DL5 | Miltenyi Biotec | 130-096-200 | Clone UP-R1. |
| anti-KIR2DS4 | R&D Systems | MAB1847 | Clone 179315. |
| anti-KIR3DL1 | BD Biosciences | 555964 | Clone DX-9. |
| anti-LFA-3 | Biolegend | 330902 | Clone TS2/9. |
| anti-LILRB1 | R&D Systems | 292319 | Clone MAB20172. |
| anti-LLT-1 | R&D Systems | AF3480 | Clone 402659. |
| anti-MICA | R&D Systems | MAB1300-100 | Clone 159227. |
| anti-MICB | R&D Systems | MAB1599-100 | Clone 236511. |
| anti-Nectin-1 | Biolegend | 340402 | Clone R1.302. |
| anti-Nectin-2 | Biolegend | 337402 | Clone TX31. |
| anti-NKG2A | R&D Systems | MAB1059 | Clone 131411. |
| anti-NKG2C | R&D Systems | MAB1381 | Clone 134522. |
| anti-NKG2D | Biolegend | 320802 | Clone 1D11. |
| anti-NKp30 | Biolegend | 325202 | Clone P30-15. |
| anti-NKp44 | Biolegend | 325102 | Clone P44-8. |
| anti-NKp46 | Biolegend | 331902 | Clone 9E2. |
| anti-NTB-A | Biolegend | 317202 | Clone NT-7. |
| anti-Pan HLA class I | Biolegend | 311402 | Clone W6/32. |
| anti-PD1 | Biolegend | 329902 | Clone EH12.2H7. |
| anti-Perforin | Abcam | ab47225 | Clone B-D48. |
| anti-Siglec-7 | Biolegend | 347702 | Clone S7.7. |
| anti-Syk | Biolegend | 644302 | Clone 4D10.2. |
| anti-TACTILE | Biolegend | 338402 | Clone NK92.39. |
| anti-TIGIT | R&D Systems | MAB7898 | Clone 741182. |
| anti-ULBP-1 | R&D Systems | MAB1380-100 | Clone 170818. |
| anti-ULBP-2, 5, 6 | R&D Systems | MAB1298-100 | Clone 165903. |
| Antibody Stabilizer | Candor Bioscience | 131 050 | |
| Benzonase Nuclease | Millipore | 70664 | |
| Bond-Breaker TCEP Solution | Thermo Fisher Scientific | 77720 | |
| Bovine Serum Albumin solution | Sigma-Aldrich | A9576 | |
| Calcium chloride dihydrate (CaCl2+2H2O) | Sigma-Aldrich | 223506-25G | |
| Cis-Platinum(II)diamine dichloride (cisplatin) | Enzo Life Sciences | ALX-400-040-M250 | A 100 mM stock solution was prepared in DMSO and divided into 25 µL aliquots. Used at a 25 µM dilution for live/dead stain. Signal appears in 194Pt and 195Pt channels. |
| DMSO | Sigma-Aldrich | D2650 | |
| eBioscience Permeabilization Buffer | Thermo Fisher Scientific | 00-8333-56 | |
| EDTA (0.5 M) | Hoefer | GR123-100 | A double-concentrated HEPES buffer with EDTA was made according to the following recipe: 1.3 g NaCl (Thermo Fisher Scientific), 27 mg CaCl2+2H2O (Sigma-Aldrich), 23 mg MgCl2 (Sigma-Aldrich), 83.6 mg KH2PO4 (Thermo Fisher Scientific), 4 mL of 1M HEPES (Thermo Fisher Scientific), 2 mL of 0.5M EDTA (Hoefer, Holliston, MA, USA), and 100mL H2O. The pH of this double-concentrated HEPES buffer was adjusted to a pH of 7.3 using 1M HCl and 1M NaOH. |
| EQ Four Element Calibration Beads | Fluidigm | 201078 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | N/A | |
| Helios mass cytometer | Fluidigm | N/A | |
| HEPES (1M) | Thermo Fisher Scientific | 15630080 | |
| HyClone Antibiotic/Antimycotic Solution (Pen/Strep/Fungiezone) solution | Fisher Scientific | SV3007901 | |
| Iridium - 191Ir/193Ir intercalator | DVS Sciences (Fluidigm) | 201192B | Used at a 1:10000 dilution. |
| Isothiocyanobenzyl-EDTA (ITCB-EDTA) | Dojindo Molecular Technologies, Inc. | M030-10 | Diluted to 1.25 mg/mL in anhydrous acetonitrile. |
| K562 cells | American Type Culture Collection (ATCC) | ATCC CCL-243 | |
| L-Glutamine (200 mM) | Thermo Fisher Scientific | SH30034 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 208337-100G | |
| Maxpar X8 Antibody Labeling Kits | Fluidigm | N/A | No catalog number as kits come with metals. |
| Millex-VV Syringe Filter Unit, 0.1 µm | Millipore | SLVV033RS | |
| Milli-Q Advantage A10 Water Purification System | Millipore | Z00Q0V0WW | |
| MS Columns | Miltenyi Biotec | ||
| NALM6 cells | American Type Culture Collection (ATCC) | ATCC CRL-3273 | |
| Nanosep Centrifugal Devices with Omega Membrane 3K | Pall Corporation | OD003C35 | |
| NK Cell Isolation Kit, human | Miltenyi Biotec | 130-092-657 | |
| Paraformaldehyde (16%) | Electron Microscopy Sciences | 15710 | |
| PBS | Thermo Fisher Scientific | 10010023 | |
| Potassium Phosphate Monobasic (KH2PO4) | Fisher Scientific | MP021954531 | |
| Qdot 655 anti-CD19 | Thermo Fisher Scientific | Q10179 | Clone SJ25-C1. Used at a 1:50 dilution. Signal appears in 112Cd-114Cd channels. |
| Qdot 655 anti-HLA-DR | Thermo Fisher Scientific | Q22158 | Clone Tü36. Used at a 1:200 dilution. |
| Rockland PBS | Rockland Immunochemicals, Inc. | MB-008 | Used to make CyPBS (10X Rockland PBS diluted to 1X in Milli-Q water) and CyFACS buffers (10X Rockland PBS diluted to 1X in Milli-Q water with 0.1% BSA and 0.05% sodium azide). Buffers were sterile-filtered through a 0.22 µM filter and sotred at 4°C in Stericup bottles. |
| RPMI 1640 | Thermo Fisher Scientific | 21870092 | |
| Sodium azide (NaN3) | Sigma-Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stericup Quick Release-GP Sterile Vacuum Filtration System | Millipore Sigma | S2GPU10RE | |
| Tuning solution | Fluidigm | 201072 | |
| Washing solution | Fluidigm | 201070 |
Références
- Shimasaki, N., Jain, A., Campana, D. NK cells for cancer immunotherapy. Nature Reviews. Drug Discovery. 19 (3), 200-218 (2020).
- Vivier, E., Tomasello, E., Baratin, M., Walzer, T., Ugolini, S. Functions of natural killer cells. Nature Immunology. 9 (5), 503-510 (2008).
- Eller, M. A., Currier, J. R. OMIP-007: phenotypic analysis of human natural killer cells. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 81 (6), 447-449 (2012).
- Mahnke, Y. D., Beddall, M. H., Roederer, M. OMIP-029: Human NK-cell phenotypization. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 87 (11), 986-988 (2015).
- Hammer, Q., Romagnani, C. OMIP-039: Detection and analysis of human adaptive NKG2C + natural killer cells : Detection of Human Adaptive NKG2C + NK Cells. Cytometry. 91 (10), 997-1000 (2017).
- Liechti, T., Roederer, M. OMIP-058: 30-Parameter flow cytometry panel to characterize iNKT, NK, unconventional and conventional T cells. Cytometry. 95 (9), 946-951 (2019).
- Béziat, V., et al. NK cell responses to cytomegalovirus infection lead to stable imprints in the human KIR repertoire and involve activating KIRs. Blood. 121 (14), 2678-2688 (2013).
- Pfefferle, A., et al. Intra-lineage plasticity and functional reprogramming maintain natural killer cell repertoire diversity. Cell Reports. 29 (8), 2284-2294 (2019).
- Barcenilla, H., Åkerman, L., Pihl, M., Ludvigsson, J., Casas, R. Mass cytometry identifies distinct subsets of regulatory T cells and Natural killer cells associated with high risk for Type 1 diabetes. Frontiers in Immunology. 10, 982(2019).
- Kurioka, A., et al. CD161 defines a functionally distinct subset of pro-inflammatory Natural killer cells. Frontiers in Immunology. 9, 486(2018).
- Romee, R., et al. Cytokine-induced memory-like natural killer cells exhibit enhanced responses against myeloid leukemia. Science Translational Medicine. 8 (357), (2016).
- Shinko, D., et al. Mass cytometry reveals a sustained reduction in CD16+ Natural killer cells following chemotherapy in colorectal cancer patients. Frontiers in Immunology. 10, 2584(2019).
- Pohlmeyer, C. W., et al. Identification of NK cell subpopulations that differentiate HIV-infected subject cohorts with diverse levels of virus control. Journal of Virology. 93 (7), 01790(2019).
- Palgen, J. -L., et al. NK cell immune responses differ after prime and boost vaccination. Journal of Leukocyte Biology. 105 (5), 1055-1073 (2019).
- Mei, H. E., Leipold, M. D., Schulz, A. R. Barcoding of live human peripheral blood mononuclear cells for multiplexed mass cytometry. The Journal of Immunology. 194 (4), 2022-2031 (2015).
- Takahashi, C., et al. Mass cytometry panel optimization through the designed distribution of signal interference. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 91 (1), 39-47 (2017).
- Baumgart, S., Peddinghaus, A., Schulte-Wrede, U., Mei, H. E., Grützkau, A. OMIP-034: Comprehensive immune phenotyping of human peripheral leukocytes by mass cytometry for monitoring immunomodulatory therapies. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 91 (1), 34-38 (2017).
- Leipold, M. D. Another step on the path to mass cytometry standardization. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 87 (5), 380-382 (2015).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Leipold, M. D., Newell, E. W., Maecker, H. T. Multiparameter phenotyping of human PBMCs using mass cytometry. Methods in Molecular Biology. 1343, 81-95 (2015).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 81 (6), 467-475 (2012).
- Zivanovic, N., Jacobs, A., Bodenmiller, B. A practical guide to multiplexed mass cytometry. Current Topics in Microbiology and Immunology. 377, 95-109 (2014).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150 (3), 248-264 (2017).
- Wilk, A. J., et al. Charge-altering releasable transporters enable specific phenotypic manipulation of resting primary natural killer cells. BioRxiv. , 970491(2020).
- Vendrame, E., et al. TIGIT is upregulated by HIV-1 infection and marks a highly functional adaptive and mature subset of natural killer cells. AIDS. 34 (6), 801-813 (2020).
- Zhao, N. Q., et al. Natural killer cell phenotype is altered in HIV-exposed seronegative women. PloS One. 15 (9), 0238347(2020).
- McKechnie, J. L., et al. HLA upregulation during dengue virus infection suppresses the natural killer cell response. Frontiers in Cellular and Infection Microbiology. 9, 268(2019).
- McKechnie, J. L., et al. Mass cytometry analysis of the NK cell receptor-ligand repertoire reveals unique differences between dengue-infected children and adults. ImmunoHorizons. 4 (10), 634-647 (2020).
- Ranganath, T., et al. Characterization of the impact of daclizumab beta on circulating natural killer cells by mass cytometry. Frontiers in immunology. 11, 714(2020).
- Fernandez, I. Z., et al. A novel human IL2RB mutation results in T and NK cell--driven immune dysregulation. The Journal of Experimental Medicine. 216 (6), 1255-1267 (2019).
- Herndler-Brandstetter, D., et al. Humanized mouse model supports development, function, and tissue residency of human natural killer cells. Proceedings of the National Academy of Sciences. 114 (45), 9626-9634 (2017).
- Nikzad, R., et al. Human natural killer cells mediate adaptive immunity to viral antigens. Science Immunology. 4 (35), (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon