Method Article
Triagem de alto rendimento de compostos químicos para elucidar seus efeitos na persistência bacteriana
Neste Artigo
Resumo
Neste artigo de método, apresentamos uma estratégia de triagem de alto rendimento para identificar compostos químicos, como osmolitos, que têm um impacto significativo na persistência bacteriana.
Resumo
Persisters bacterianos são definidos como uma pequena subpopulação de variantes fenotípicas com a capacidade de tolerar altas concentrações de antibióticos. São uma importante preocupação para a saúde, pois têm sido associadas a infecções crônicas recorrentes. Embora a dinâmica estocástica e determinística dos mecanismos relacionados ao estresse seja conhecida por desempenhar um papel significativo na persistência, os mecanismos subjacentes à mudança fenotípica para/do estado de persistência não são completamente compreendidos. Embora os fatores de persistência desencadeados por sinais ambientais (por exemplo, esgotamento das fontes de carbono, nitrogênio e oxigênio) tenham sido extensivamente estudados, os impactos dos osmólises sobre a persistência ainda estão para ser determinados. Usando microarrays (ou seja, 96 placas de poço contendo vários produtos químicos), projetamos uma abordagem para elucidar os efeitos de vários osmólises na persistência de Escherichia coli de forma de alto rendimento. Essa abordagem é transformadora, pois pode ser prontamente adaptada para outras matrizes de triagem, como painéis de drogas e bibliotecas de genes knockout.
Introdução
Culturas bacterianas contêm uma pequena subpopulação de células persisteras que são temporariamente tolerantes a níveis extraordinariamente altos de antibióticos. As células persisteras são geneticamente idênticas aos seus parentes sensíveis a antibióticos, e sua sobrevivência tem sido atribuída à inibição transitória do crescimento1. Células persisteres foram descobertas pela primeira vez por Gladys Hobby2, mas o termo foi usado pela primeira vez por Joseph Bigger quando ele as identificou nas culturas Staphylococcus pyogenes tratadas com penicilina3. Um estudo seminal publicado por Balaban et al.4 descobriu dois tipos de persistência: variantes tipo I que são formadas principalmente por passagem através da fase estacionária, e variantes tipo II que são geradas continuamente durante o crescimento exponencial. Persisters são detectados por ensaios de sobrevivência clonogênico, nos quais amostras de cultura são colhidas em vários intervalos durante tratamentos com antibióticos, lavadas e banhadas em um meio de crescimento típico para contar as células sobreviventes que podem colonizar na ausência de antibióticos. A existência de persisters em uma cultura celular é avaliada por uma curva de morte bifásica4,5 onde a decadência exponencial inicial indica a morte de células sensíveis a antibióticos. No entanto, a tendência de morte diminui com o tempo, eventualmente levando a uma região platô que representa as células persisteres sobreviventes.
As células persisteres têm sido associadas a várias doenças como tuberculose6,fibrose cística7,candidíase8 e infecções do trato urinário9. Quase todos os microrganismos testados até agora foram encontrados para gerar fenótipos persisteres, incluindo mycobacterium tuberculosis6, Staphylococcus aureus10, Pseudomonas aeruginosa7 e Candida albicans8. Estudos recentes também fornecem evidências do surgimento de mutantes multidroga resistentes a subpopulaçõespersistentes 11,12. Esforços substanciais neste campo revelaram que os mecanismos de persistência são altamente complexos e diversos; tanto fatores estocásticos quanto determinísticos associados à resposta SOS13,14, espécies reativas de oxigênio (ROS)15,sistemas toxina/antitoxina (TA)16,autofagia ou auto-digestão17 e resposta rigorosa relacionada ao PPGPP18 são conhecidos por facilitar a formação de persister.
Apesar dos progressos significativos na compreensão do fenótipo persistência, os efeitos dos osmólises sobre a persistência bacteriana não foram totalmente compreendidos. Uma vez que a manutenção da pressão osmótica ideal é uma necessidade para o crescimento das células, o bom funcionamento e a sobrevivência, um estudo aprofundado dos osmólises poderia levar a potenciais alvos para estratégias anti-persistência. Embora a triagem laboriosa e de alto rendimento seja uma abordagem muito eficaz para identificar metabólitos e outros produtos químicos que desempenham um papel crucial no fenótipo de persistência19,20. Neste trabalho, discutiremos nosso método publicado19, onde usamos microarrays, ou seja, 96 placas de poço contendo vários osmólises (por exemplo, cloreto de sódio, ureia, nitrito de sódio, nitrato de sódio, cloreto de potássio), para identificar osmólitos que influenciam significativamente a persistência de E. coli.
Protocolo
1. Preparação de estoques médios de crescimento, solução de deloxacina e estoques de células E. coli
- Meio regular luria-bertani (LB): Adicione 10 g/L de triptona, 10 g/L de cloreto de sódio (NaCl) e 5 g/L de extrato de levedura em água desionizada (DI). Esterilize o meio autoclavando.
- Placas de ágar LB: Adicione 10 g/L de triptona, 10 g/L de NaCl, 5 g/L de extrato de levedura e 15 g/L de ágar em água DI e esterilize o meio por autoclavagem. Na temperatura desejada (~55 °C), despeje ~30 mL de ágar médio em placas quadradas (10 x 10 cm). Seque as placas e armazene a 4 °C.
- Meio LB modificado: Adicione 10 g/L de triptona e 5 g/L de extrato de levedura em água DI. Esterilize o meio autoclavando.
- Meio LB modificado incluindo um osmólise: Misture 2x meio LB modificado com uma solução de osmólitos de 2x em volumes iguais. Para preparar 2x médio LB modificado, adicione 20 g/L de triptona e 10 g/L de extrato de levedura em água DI e esterilize por autoclavagem. Para preparar a solução de osmólise 2x, dissolva o osmólise de interesse (quantidade 2x) em água DI e, em seguida, o filtro esterilize a solução.
NOTA: O osmólito de interesse e suas concentrações pode ser determinado a partir dos ensaios de triagem de Microarray do Fenótipo (ver Protocolo 4). No entanto, o osmólito testado e a concentração 2x associada podem ser ajustados dependendo da natureza da pesquisa. Por exemplo, para estudar o impacto do cloreto de sódio de 1 mM, uma solução de osmólito 2x seria uma solução de cloreto de sódio de 2 mM. - Solução de estoque de ofloxacina (5 mg/mL): Adicione 5 mg de sal de ofloxacina (OFX) em 1 mL de água DI. Adicione 10 μL de hidróxido de sódio de 10 M para aumentar a solubilidade do OFX na água e, em seguida, o filtro esterilizar a solução. Prepare as alíquotas e armazene a -20 °C.
NOTA: A ofloxacina é um antibiótico de quinolone que tem sido amplamente utilizado para células bacterianas em crescimento e não crescimento14,21. A concentração inibitória mínima (MIC) de OFX para células E. coli MG1655 está dentro da faixa de 0,039-0,078 μg/mL19,20. Observe também que outros antibióticos como ampicillina e kanamicina são comumente usados em pesquisas per persister. A escolha dos antibióticos depende da natureza do estudo. - E. coli Estoques de células MG1655: Inoculam 2 mL de meio LB normal com uma única colônia em um tubo de ensaio de 14 mL (snap-capped) e cultural as células em um agitador orbital a 250 rpm e 37 °C. Quando as células chegarem à fase estacionária, misture 500 μL da cultura celular com 500 μL de 50% de glicerol (estéril) em um frasco criogênico e armazene a -80 °C.
2. Propagação de células para eliminar persisters pré-existentes
- Para preparar uma cultura durante a noite, raspe uma pequena quantidade de células de um estoque de células congeladas com uma ponta de pipeta estéril (não descongele o estoque de células glicerol), e inocular as células em 2 mL de meio LB modificado em um tubo de ensaio com tampa de snap de 14 mL. Cultue as células em um agitador orbital a 250 rpm e 37 °C por 12 h.
- Primeira propagação
- Após 12 h, transfira 250 μL de cultura durante a noite para 25 mL de meio LB modificado fresco em um frasco de 250 mL perplexo coberto com uma folha de alumínio estéril.
- Cresça a cultura celular em um agitador orbital a 250 rpm e 37 °C até que as células atinjam a fase exponencial média (OD600 = 0,5).
- Meça a densidade óptica em 600 nm (OD600) usando um leitor de microplacas a cada 30 minutos.
- Segunda propagação
- Em OD600 = 0,5, dilui 250 μL de cultura celular do primeiro frasco em 25 mL de meio LB modificado fresco em um frasco de 250 mL perplexo.
- Cresça a segunda cultura celular em um agitador orbital a 250 rpm e 37 °C até OD600=0,5.
- Meça a densidade óptica a cada 30 minutos.
NOTA: Uma cultura noturna pode ter uma quantidade significativa de células persistência4,5,22. O método de ciclo de diluição/crescimento5 descrito acima pode ser usado para eliminar esses persistências pré-existentes antes de transferir as células para microarrays. Sua eliminação pode ser validada quantificando níveis persistência de culturas noturnas com o ensaio descrito no Protocolo 3. Este método já foi validado em um estudo anterior19.
3. Validando a eliminação de células persisteres pré-existentes
- Após a segunda propagação (ver passo 2.3), diluir 250 μL da cultura celular (OD600 = 0,5) em 25 mL de meio LB modificado fresco em um frasco de 250 mL perplexo.
NOTA: Para controles, diluir 250 μL de cultura durante a noite (ver passo 2.1) em 25 mL de meio LB modificado fresco em um frasco de 250 mL perplexo. Antes de transferir as células para o frasco, a densidade celular da cultura overnight deve ser ajustada em meio LB modificado fresco para obter OD600 = 0,5. - Adicione 25 μL de solução de estoque OFX (5 mg/mL) na suspensão celular e agite o frasco suavemente para tornar a cultura do ensaio homogênea. A concentração final de OFX é de 5 μg/mL na cultura de ensaio.
- Incubar a cultura do ensaio em um agitador orbital a 250 rpm e 37 °C.
- A cada hora durante o tratamento (incluindo 0h, o ponto de tempo antes de adicionar OFX na cultura de ensaio), transfira 1 mL da cultura de ensaio do frasco para um tubo de microcentrifuuge de 1,5 mL.
- Centrifugar a cultura de ensaio no tubo de microcentrifuuge a 17.000 x g por 3 min.
- Remova cuidadosamente 950 μL do supernatante sem perturbar a pelota celular.
- Adicione 950 μL de solução salina tamponada com fosfato (PBS) no tubo de microcentrifuuge.
- Repita as etapas de lavagem 3,5-3,7 por 3x no total até que a concentração de antibióticos esteja abaixo da concentração inibitória mínima (MIC).
- Após a lavagem final, resuspenja a pelota celular em 100 μL de solução PBS, resultando em uma amostra concentrada de 10x.
- Tome 10 μL da suspensão da célula e dilua-se seis vezes em 90 μL de solução PBS usando uma placa inferior bem redonda de 96.
- Local 10 μL de suspensões de células diluídas em placas de ágar frescas sem antibióticos. Para aumentar o limite de detecção, aplaque os 90 μL restantes de suspensão celular em uma placa de ágar fresca.
- Incubar as placas de ágar a 37 °C por 16 h e, em seguida, contar as unidades formadoras de colônias (UFC). Conta as taxas de diluição enquanto calcula o número total de UFC em 1 mL de cultura de ensaio. As curvas de morte são geradas pelo plotagem dos valores da UFC logarítmica em relação à duração do tratamento com antibióticos.
NOTA: Um tratamento OFX de 6 h é suficiente para obter uma curva de morte bifásica para células E. coli 19,20. O nível persistente de células propagadas (medida pela CFU em 6h) deve ser significativamente menor do que o da cultura da noite para o dia. Os procedimentos descritos nos Protocolos 2 e 3 podem ser realizados com LB regular, dependendo do desenho da pesquisa.
4. Triagens de placas de microarray
- Preparando culturas de microarray-cell
- Transfira 250 μL de células de fase exponencial para 25 mL de meio LB modificado fresco em um tubo de centrífuga de 50 mL. Misture suavemente a suspensão da célula para torná-la homogênea.
NOTA: As células de fase exponencial (OD600 = 0,5) são obtidas a partir da segunda etapa de propagação (ver passo 2.3). - Transfira a suspensão da célula diluída para um reservatório estéril de 50 mL.
- Utilizando uma pipeta multicanal, transfira 150 μL da suspensão celular para cada poço de microarray, ou seja, uma placa de 96 poços contendo vários osmólises.
NOTA: Poços que não possuem osmólises servem como controles. - Cubra a microarray com uma membrana de vedação permeável a gás.
- Incubar a placa em um agitador orbital a 37 °C e 250 rpm por 24 h.
NOTA: Nestes experimentos, foram utilizadas placas comercialmente disponíveis, como Phenotype Microarrays (PM-9 e PM-10) que incluem uma ampla gama de osmólises, tampões de pH e outros produtos químicos em estado seco em várias concentrações. Essas microarrays estão em formatos de placas de meia área 96. Os volumes de cultura devem ser ajustados dependendo do tipo de placas que estão sendo utilizadas. Microarrays também podem ser gerados manualmente (veja abaixo).
- Transfira 250 μL de células de fase exponencial para 25 mL de meio LB modificado fresco em um tubo de centrífuga de 50 mL. Misture suavemente a suspensão da célula para torná-la homogênea.
- Preparação manual de placas de microarray
- Transfira 75 μL de soluções de osmólise 2x para os poços de uma placa de meio-campo 96.
- Transfira 500 μL de células de fase exponencial para 25 mL de meio LB modificado de 2x em um tubo centrífuga de 50 mL. Misture suavemente a suspensão da célula para torná-la homogênea.
NOTA: A taxa de inoculação foi ajustada para ser consistente com o protocolo de triagem de microarray descrito em 4.1. - Adicione 75 μL da suspensão da célula em cada poço da placa de meio-campo 96 bem contendo soluções de osmólito 2x.
- Incubar a placa em um agitador orbital a 37 °C e 250 rpm por 24 h.
- Preparando placas de ensaio de periáscrito
- Prepare 25 mL de meio LB modificado contendo 5 μg/mL de OFX em um tubo de centrífuga de 50 mL e transfira este meio para um reservatório estéril.
- Transfira 190 μL de meio LB modificado com OFX do reservatório para cada poço de uma placa de poço de fundo plano genérico 96 (placa de ensaio persistente) usando uma pipeta multicanal.
- Remova a microarray do agitador (após a colheita por 24 h) e transfira 10 μL de culturas celulares da microarray para os poços da placa de ensaio persister, contendo meio LB modificado com OFX.
- Pegue 10 μL de suspensões celulares da placa de ensaio periculoso e dilua-se em série três vezes em 290 μL de solução PBS usando uma placa de poço de fundo redondo 96 e uma pipeta multicanal.
- Após a diluição seriada, local 10 μL de todas as suspensões celulares diluídas em série em placas de ágar fresco sem antibióticos usando uma pipeta multicanal.
- Incubar a placa de ensaio persister (preparada na etapa 4.3.3) em um agitador orbital a 37 °C e 250 rpm por 6h depois de cobrir a placa com uma membrana de vedação permeável a gás.
- Após a incubação de 6 h em um agitador, retire a placa de ensaio persister e repita os passos 4.3.4-4.3.5.
- Incubar as placas de ágar por 16 h a 37 °C e, em seguida, contar CFUs. Os níveis de UFC antes e 6 h após o tratamento antibiótico permitem calcular a fração persistência em cada poço. A CFU conta antes do tratamento OFX também ajudar a avaliar os efeitos dos osmólises e na viabilidade de E. coli.
5. Validação das condições identificadas
- Transfira 250 μL das células de fase exponencial da Etapa 2.3 para 25 mL de meio LB modificado fresco contendo o osmólise identificado a partir da triagem de microarray (ver Protocolo 4).
- Incubar o frasco em um agitador orbital a 250 rpm e 37 °C por 24 h.
- Após 24 h, remova o frasco do agitador e transfira 250 μL da cultura celular para 25 mL de meio LB modificado fresco em um frasco de 250 mL perplexo.
- Adicione 25 μL de solução de estoque OFX (5 mg/mL) na suspensão celular e agite o frasco suavemente para tornar a cultura do ensaio homogênea. Incubar o frasco em um shaker a 37 °C e 250 rpm.
- A cada hora durante o tratamento, transfira 1 mL da cultura de ensaio do frasco para um tubo de microcentrífuga de 1,5 mL.
- Centrifugar a cultura de ensaio no tubo de microcentrifuuge a 17.000 x g por 3 min.
- Remova 950 μL de supernascente e adicione 950 μL de PBS.
- Repita as etapas de lavagem 5.6 e 5.7 para 3x.
- Após a lavagem final, resuspenja a pelota de célula em 100 μL de solução PBS.
- Tome 10 μL da suspensão da célula e diluir em 6x em 90 μL de solução PBS usando uma placa inferior bem redonda de 96.
- Local 10 μL das suspensões celulares diluídas em uma placa de ágar fresca sem antibióticos. Para aumentar o limite de detecção, aplaque os 90 μL restantes de suspensão celular em uma placa de ágar fresca.
- Incubar a placa de ágar a 37 °C por 16 h e, em seguida, contar CFUs.
Resultados
A Figura 1 descreve nosso protocolo experimental. Os experimentos do ciclo de diluição/crescimento (ver Protocolo 2) foram adaptados de um estudo conduzido por Keren et al.5 para eliminar os persisters originários das culturas noturnas. Figura 2A é uma imagem representativa das placas de ágar usadas para determinar os níveis de CFU de culturas celulares antes e depois do tratamento OFX. Nesses experimentos, as células foram cultivadas em meio LB modificado com osmólises em placas de 96 poços de meia área, conforme descrito na etapa 4.2. Depois de incubar a placa em um agitador orbital por 24 h, o ensaio persister foi realizado usando uma placa de fundo plana genérica 96 bem (ver passo 4.3). Os osmólises e a concentração aqui testada foram escolhidos com base em nosso estudo anterior19, onde realizamos as etapas 4.1 e 4.3 utilizando a placa PM-9 que inclui vários osmólitos em diferentes concentrações (cloreto de sódio, cloreto de potássio, sulfato de sódio, etileno glicol, formato de sódio, ureia, lactato de sódio, fosfato de sódio, benzoato de sódio, sulfato de amônio, nitrito de sódio e nitrato de sódio). A primeira coluna da Figura 2A mostra as contagens de CFU do grupo controle. A segunda coluna representa uma condição em que as células foram cultivadas em nitrato de sódio de 100 mM; essa condição foi previamente encontrada para aumentar ligeiramente os níveis de persistência19. A terceira coluna representa uma condição em que as células foram cultivadas em nitrito de sódio de 60 mM, e essa condição foi previamente encontrada para diminuir significativamente os níveis de persistência em comparação com os controles19. Figura 2B é uma representação gráfica dos dados da UFC obtidos a partir das placas de ágar. A Figura 2C mostra as frações per persistidoras das culturas celulares testadas em 96 placas de poço. Para calcular as frações, as contagens persisteres foram normalizadas para a contagem celular obtida antes dos tratamentos antibióticos. A Figura 3 mostra as curvas de morte bifásica e as frações pervasoras, respectivamente, para as culturas de ensaio realizadas em frascos perplexos. Nesses experimentos, as células foram cultivadas pela primeira vez em 25 mL de meio LB modificado com os osmolitos indicados em frascos perplexos de 250 mL por 24 h, e então as células foram transferidas para frascos de ensaio pervaso para enumeração persistente, conforme descrito no Protocolo 5.
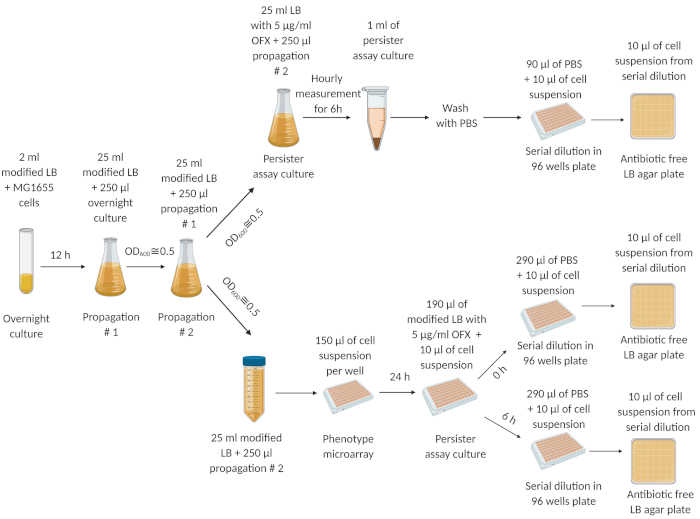
Figura 1: O procedimento experimental. As células de um estoque de células congeladas foram cultivadas durante a noite (12 h) em meio LB fresco. Às 12 horas, a cultura da noite foi diluída (1:100) em 25 mL de meio LB modificado e cultivada atéOD 600=0,5. Este passo de propagação foi repetido duas vezes. Após a etapa final de propagação, as células de fase exponencial (em OD600=0,5) foram diluídas (1:100) em meio LB modificado fresco em um frasco de 250 mL perplexo e um tubo centrífuga de 50 mL, respectivamente. A suspensão celular no frasco de 250 mL foi tratada com 5 μg/mL de OFX para quantificar os persisters. A suspensão celular no tubo centrífuga de 50 mL foi transferida para microarrays e incubada por 24 h em um agitador orbital a 250 rpm e 37 °C. As células das microarrays foram então transferidas para placas de ensaio persister para quantificar persisters. Esta figura foi criada usando biorender.com. Este valor foi modificado da nossa publicação anterior19. Clique aqui para ver uma versão maior desta figura.

Figura 2: Experimentos de microarray. (A) As células foram cultivadas pela primeira vez em meio LB modificado com ou sem osmólises indicados em uma placa de meio-campo 96 por 24 h. As células foram então transferidas para uma placa de fundo plano genérico 96 e tratadas com 5 μg/mL de OFX por 6 h. Antes e depois do tratamento de 6h, as células foram diluídas em série na PBS, banhadas em placas de ágar e incubadas a 37 °C por 16 h. Cada condição tem 8 réplicas técnicas. (B) Foram realizadas medições da UFC para avaliar os efeitos dos osmólises na viabilidade e persistência celular, respectivamente. A linha reta indica o limite de detecção (600 UFC). (C) O gráfico representa as frações persisteras das culturas celulares, calculadas tomando a razão da CFU conta após e antes do tratamento OFX. A fração persistência da cultura celular que tem nitrito de sódio de 60 mM não foi calculada, pois seu nível de persistência está abaixo do limite de detecção. Cada ponto de dados foi denotado pelo valor médio ± desvio padrão, calculado a partir de 8 réplicas técnicas. Clique aqui para ver uma versão maior desta figura.
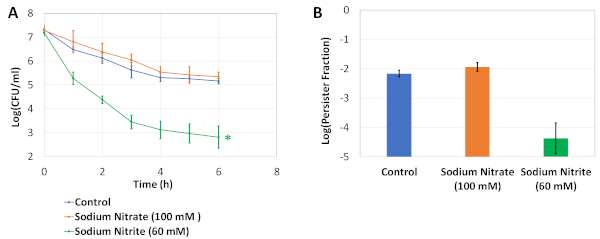
Figura 3: Experimentos de validação. (A) As células de fase exponencial foram transferidas para 25 mL de meio LB modificado fresco com osmólises indicados em frascos perplexos de 250 mL e cultivadas por 24 h. Em seguida, as células foram diluídas (1:100) em frascos perplexos de 250 mL contendo 25 mL de meio LB modificado e tratados com 5 μg/mL OFX por 6 h. As contagens de UFC nas culturas de ensaio foram monitoradas de hora em hora para gerar curvas de morte bifásica. * indica a condição que afeta significativamente os níveis de persistência ofx em comparação com controles sem osmólitos (t-teste de variação desigual de duas caudas, p<0,05). (B) O gráfico representa as frações persisteras das culturas. Cada ponto de dados foi denotado pelo valor médio ± desvio padrão, calculado a partir de 3 réplicas biológicas. Este valor foi modificado da nossa publicação anterior19. Clique aqui para ver uma versão maior desta figura.
Discussão
O alto ensaio de persistência de rendimento descrito aqui foi desenvolvido para elucidar os efeitos de vários produtos químicos na persistência de E. coli. Além das placas comerciais da PM, as microarrays podem ser construídas manualmente conforme descrito na etapa 4.2. Além disso, o protocolo aqui apresentado é flexível e pode ser usado para tela de outras microarrays, como painéis de drogas e bibliotecas celulares, que estão em 96 formatos de placas de poço. As condições experimentais, incluindo a fase de crescimento, taxa de inoculação e média, podem ser ajustadas para testar essas bibliotecas. Por exemplo, se alguém quiser telar uma biblioteca de celular, como a coleção de nocautes Keio E. coli 23,eles podem primeiro transferir as cepas da biblioteca para 96 placas de poços que incluem mídia fresca, usando uma pipeta multicanal. Uma vez que as células atingem a fase de crescimento desejada (por exemplo, fase exponencial ou estacionária), as células são então transferidas para placas de ensaio persistente, conforme descrito na etapa 4.3 para enumerar os níveis persisteres das cepas de nocaute. Da mesma forma, essa estratégia pode ser usada para exibir a coleção E. coli Promoter 24 (uma biblioteca de cepas de repórteres fluorescentes em 96 formatos de placas de poço) para identificar promotores que são ativados por antibióticos. Esses promotores podem ser facilmente detectados medindo os sinais de fluorescência de cepas em placas de ensaio persistente durante o tratamento com antibióticos.
Em nossos experimentos, usamos uma LB modificada (sem NaCl) para evitar qualquer efeito adicional que pudesse surgir da NaCl, considerando que as microarrays já possuem vários osmólitos. Embora o NaCl em meio LB regular seja conhecido por ser bom para preservar a integridade da membrana das células, foi relatado que o NaCl na faixa de 0-1% tem um efeito mínimo no crescimento celular25. Além disso, os resultados da coloração do iodeto propidium (PI) e dos ensaios dos persisters em nosso estudo anterior mostraram que a ausência de NaCl não afeta significativamente a integridade da membrana e a persistência das células E. coli 19. Essa modificação foi feita especificamente para abordar as preocupações em nosso estudo e pode ser alterada com base na natureza da pesquisa.
Também adaptamos um método desenvolvido por Keren et al.5 para eliminar persisters pré-existentes em nossas culturas celulares antes da inoculação nas microarrays. Os persisters tipo I são conhecidos por serem gerados durante a fase estacionária; portanto, a inoculação direta das células de culturas noturnas em placas de microarray transferiria um número significativo de persisters, o que pode dificultar os efeitos dos osmólises. Com os experimentos do ciclo de diluição/crescimento (ver Protocolo 2), conseguimos reduzir significativamente esses persisters pré-existentes decorrentes das culturas noturnas19. Notamos que temos cultivado as células em placas de microarray por 24 horas para poder contar todos os persisters, incluindo variantes tipo I que são formadas durante a fase estacionária na presença de osmólises. No entanto, essas técnicas adaptadas podem sempre ser modificadas dependendo da natureza do estudo.
Tratamos as células em microarrays com 5 μg/mL OFX para enumerar os persisters. Uma vez que o procedimento de lavagem para remover o antibiótico de 96 amostras seria muito trabalhoso, as células após o tratamento foram diluídas em série na PBS sem lavar (ver passo 4.2). Este procedimento diluiu a concentração OFX mais de 30 vezes. As suspensões celulares diluídas foram então banhadas em placas de ágar sem antibióticos onde o OFX foi ainda mais disperso. Nossos estudos preliminares onde repetimos esses experimentos com e sem lavagem verificaram que este método de diluição serial não afetou os níveis de persistência19.
Durante a tubulação multicanal em 96 placas de poço, deve-se misturar cuidadosamente as suspensões celulares para manter uma distribuição celular homogênea. Para isso, dependendo do volume de trabalho, seria benéfico notar o número mínimo de pipetamento necessário para tornar a solução homogênea. Para isso, realizamos um simples experimento de controle onde monitoramos a pipetação (para cima e para baixo) dispersando uma molécula de corante em PBS e meio nas condições aqui estudadas. Além disso, se trabalharmos com ambientes mais complexos, como tampões de pH, sugerimos medir o pH da cultura celular antes e durante a incubação em um shaker, pois a cultura celular tende a ser muito alcalina (pH ≈ 8) ao longo do tempo. Isso pode dar uma ideia da qualidade, bem como da faixa de pH do buffer utilizado. Além disso, para obter CFUs contáveis em placas de ágar a partir dos ensaios de alto rendimento, certas condições, como a taxa de inoculação celular, idade das culturas e parâmetros de diluição serial (volume PBS, taxas de diluição e o número de etapas de diluição, etc.) devem ser otimizadas antes de testar as microarrays. Finalmente, os resultados obtidos a partir das microarrays devem ser validados ainda mais em frascos, pois o volume da cultura, área de superfície e aeração em uma placa de poço 96 poderia ter efeitos adicionais sobre os resultados observados.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Gostaríamos de agradecer aos membros do Laboratório Orman por suas valiosas contribuições durante este estudo. Este estudo foi financiado pelo prêmio nih/NIAID K22AI125468 de transição de carreira e uma bolsa de startup da Universidade de Houston.
Materiais
| Name | Company | Catalog Number | Comments |
| 14-ml test tube | Fisher Scientific | 14-959-1B | |
| E. coli strain MG1655 | Princeton University | Obtained from Brynildsen lab | |
| Flat-bottom 96-well plate | USA Scientific | 5665-5161 | |
| Gas permeable sealing membrane | VWR | 102097-058 | Sterilized by gamma irradiation and free of cytotoxins |
| Half-area flat-bottom 96-well plate | VWR | 82050-062 | |
| LB agar | Fisher Scientific | BP1425-2 | Molecular genetics grade |
| Ofloxacin salt | VWR | 103466-232 | HPLC ≥97.5 |
| Phenotype microarray (PM-9 and PM-10) | Biolog | N/A | PM-9 and PM-10 plates contained various osmolytes and buffers respectively |
| Round-bottom 96-well plate | USA Scientific | 5665-0161 | |
| Sodium chloride | Fisher Scientific | S271-500 | Certified ACS grade |
| Sodium nitrate | Fisher Scientific | AC424345000 | ACS reagent grade |
| Sodium nitrite | Fisher Scientific | AAA186680B | 98% purity |
| Square petri dish | Fisher Scientific | FB0875711A | |
| Tryptone | Fisher Scientific | BP1421-500 | Molecular genetics grade |
| Varioskan lux multi mode microplate reader | Thermo Fisher Scientific | VLBL00D0 | Used for optical density measurement at 600 nm |
| Yeast extract | Fisher Scientific | BP1422-100 | Molecular genetics grade |
Referências
- Lewis, K. Persister cells, dormancy and infectious disease. Nature Reviews Microbiology. 5 (1), 48-56 (2007).
- Hobby, G. L., Meyer, K., Chaffee, E. Observations on the Mechanism of Action of Penicillin. Experimental Biology and Medicine. 50 (2), 281-285 (1942).
- Bigger, J. Treatment of staphylococcal infections with penicillin by intermittent sterilisation. The Lancet. 244 (6320), 497-500 (1944).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Keren, I., Kaldalu, N., Spoering, A., Wang, Y., Lewis, K. Persister cells and tolerance to antimicrobials. FEMS Microbiology Letters. 230 (1), 13-18 (2004).
- Keren, I., Minami, S., Rubin, E., Lewis, K. Characterization and transcriptome analysis of mycobacterium tuberculosis persisters. mBio. 2 (3), (2011).
- Mulcahy, L. R., Burns, J. L., Lory, S., Lewis, K. Emergence of Pseudomonas aeruginosa Strains Producing High Levels of Persister Cells in Patients with Cystic Fibrosis. Journal of Bacteriology. 192 (23), 6191-6199 (2010).
- LaFleur, M. D., Kumamoto, C. A., Lewis, K. Candida albicans biofilms produce antifungal-tolerant persister cells. Antimicrobial Agents and Chemotherapy. 50 (11), 3839-3846 (2006).
- Allison, K. R., Brynildsen, M. P., Collins, J. J. Metabolite-enabled eradication of bacterial persisters by aminoglycosides. Nature. 473 (7346), 216-220 (2011).
- Lechner, S., Lewis, K., Bertram, R. Staphylococcus aureus persisters tolerant to bactericidal antibiotics. Journal of Molecular Microbiology and Biotechnology. 22 (4), 235-244 (2012).
- Barrett, T. C., Mok, W. W. K., Murawski, A. M., Brynildsen, M. P. Enhanced antibiotic resistance development from fluoroquinolone persisters after a single exposure to antibiotic. Nature Communications. 10 (1), 1177 (2019).
- Windels, E. M., et al. Bacterial persistence promotes the evolution of antibiotic resistance by increasing survival and mutation rates. ISME Journal. 13 (5), 1239-1251 (2019).
- Dörr, T., Lewis, K., Vulić, M. SOS response induces persistence to fluoroquinolones in Escherichia coli. PLoS Genetics. 5 (12), 1000760 (2009).
- Völzing, K. G., Brynildsen, M. P. Stationary-phase persisters to ofloxacin sustain DNA damage and require repair systems only during recovery. mBio. 6 (5), (2015).
- Grant, S. S., Kaufmann, B. B., Chand, N. S., Haseley, N., Hung, D. T. Eradication of bacterial persisters with antibiotic-generated hydroxyl radicals. Proceedings of the National Academy of Sciences. 109 (30), 12147-12152 (2012).
- Gerdes, K., Maisonneuve, E. Bacterial Persistence and Toxin-Antitoxin Loci. Annual Review of Microbiology. 66 (1), 103-123 (2012).
- Orman, M. A., Brynildsen, M. P. Inhibition of stationary phase respiration impairs persister formation in E. coli. Nature Communications. 6 (1), 7983 (2015).
- Korch, S. B., Henderson, T. A., Hill, T. M. Characterization of the hipA7 allele of Escherichia coli and evidence that high persistence is governed by (p)ppGpp synthesis. Molecular Microbiology. 50 (4), 1199-1213 (2003).
- Karki, P., Mohiuddin, S. G., Kavousi, P., Orman, M. A. Investigating the effects of osmolytes and environmental ph on bacterial persisters. Antimicrobial Agents and Chemotherapy. 64 (5), 02393 (2020).
- Mohiuddin, S. G., Hoang, T., Saba, A., Karki, P., Orman, M. A. Identifying Metabolic Inhibitors to Reduce Bacterial Persistence. Frontiers in Microbiology. 11, 472 (2020).
- Brooun, A., Liu, S., Lewis, K. A dose-response study of antibiotic resistance in Pseudomonas aeruginosa biofilms. Antimicrobial Agents and Chemotherapy. 44 (3), 640-646 (2000).
- Luidalepp, H., Jõers, A., Kaldalu, N., Tenson, T. Age of inoculum strongly influences persister frequency and can mask effects of mutations implicated in altered persistence. Journal of Bacteriology. 193 (14), 3598-3605 (2011).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: The Keio collection. Molecular Systems Biology. 2, 0008 (2006).
- Zaslaver, A., et al. A comprehensive library of fluorescent transcriptional reporters for Escherichia coli. Nature Methods. 3 (8), 623-628 (2006).
- Hajmeer, M., Ceylan, E., Marsden, J. L., Fung, D. Y. C. Impact of sodium chloride on Escherichia coli O157:H7 and Staphylococcus aureus analysed using transmission electron microscopy. Food Microbiology. 23 (5), 446-452 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados