Method Article
Quantificação da agregação de proteínas insolúveis em Caenorhabditis elegans durante o envelhecimento com um novo fluxo de trabalho de aquisição independente de dados
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este novo fluxo de trabalho extrai e isola com eficiência proteínas insolúveis em SDS (insolublome) de Caenorhabditis elegans com material de partida mínimo para análise proteômica diferencial quantitativa. O protocolo usa uma análise abrangente de espectrometria de massa de aquisição independente de dados para quantificar o insolublome e análise bioinformática para obter insights biológicos sobre os mecanismos e patologias do envelhecimento.
Resumo
Nós e outros mostramos que o processo de envelhecimento resulta em um acúmulo de proteínas insolúveis em todo o proteoma. Derrubar genes que codificam as proteínas insolúveis em mais de 40% do tempo resulta em uma extensão da vida útil em C. elegans, sugerindo que muitas dessas proteínas são determinantes importantes do processo de envelhecimento. O isolamento e a identificação quantitativa dessas proteínas insolúveis são cruciais para entender os principais processos biológicos que ocorrem durante o envelhecimento. Aqui, apresentamos um protocolo modificado e aprimorado que detalha como extrair e isolar as proteínas insolúveis em SDS (insolublome) de C. elegans de forma mais eficiente para agilizar os fluxos de trabalho de espectrometria de massa por meio de uma nova análise proteômica quantitativa sem marcadores. Este protocolo aprimorado utiliza um sonicador altamente eficiente para lise de vermes que aumenta muito a eficiência da extração de proteínas e nos permite usar significativamente menos material de partida (aproximadamente 3.000 vermes) do que em protocolos anteriores (normalmente usando pelo menos 40.000 vermes). A análise proteômica quantitativa subsequente do insolubloma foi realizada usando aquisição dependente de dados (DDA) para descoberta e identificação de proteínas e aquisição independente de dados (DIA) para quantificação abrangente e mais precisa de proteínas. A análise bioinformática de proteínas quantificadas fornece potenciais candidatos que podem ser facilmente acompanhados por outros métodos moleculares em C. elegans. Com esse fluxo de trabalho, identificamos rotineiramente mais de 1000 proteínas e quantificamos mais de 500 proteínas. Este novo protocolo permite uma triagem eficiente de compostos com C. elegans. Aqui, validamos e aplicamos este protocolo aprimorado à cepa C . elegans N2-Bristol do tipo selvagem e confirmamos que os vermes N2 do dia 10 com idade apresentaram maior acúmulo do insolubloma do que os vermes jovens do dia 2.
Introdução
A homeostase proteica diminui progressivamente com o envelhecimento e resulta em aumento da agregação proteica 1,2,3. A agregação de proteínas está associada a várias doenças neurodegenerativas, incluindo doença de Alzheimer, doença de Parkinson, doença de Huntington e esclerose lateral amiotrófica4. O envelhecimento é considerado o principal fator de risco para o aparecimento de doenças neurodegenerativas associadas à agregação proteica. Proteínas propensas a formar agregados insolúveis estão frequentemente associadas à toxicidade celular e disfunção tecidual, o que pode acelerar ainda mais a agregação de outras proteínas 5,6,7. Alternativamente, agregados de proteínas insolúveis podem ativar mecanismos de defesa celular para remover as formas oligoméricas tóxicas da proteína do sistema. A eliminação de genes selecionados que codificam proteínas insolúveis modula a vida útil de Caenorhabditis elegans (C. elegans) no contexto de doenças relacionadas à idade e envelhecimento normal 5,8,9. Assim, estudar os mecanismos celulares e moleculares da agregação de proteínas é crucial para entender o envelhecimento e, em última análise, pode levar a abordagens para o tratamento de doenças neurodegenerativas.
O nematóide C. elegans tornou-se um dos organismos modelo mais amplamente utilizados para estudar a agregação de proteínas no envelhecimento e doenças relacionadas à idade devido às suas características únicas, como uma vida útil relativamente curta (cerca de 2 semanas), facilidade de cultivo e manipulação genética.
A capacidade de extrair e caracterizar proteínas insolúveis desempenhou um papel crítico na determinação das alterações relacionadas à idade associadas à agregação de proteínas em modelos de C. elegans. Para investigar a contribuição da agregação de proteínas para os processos normais de envelhecimento, nós5 e outros2 extraímos previamente e digerimos proteoliticamente o insolubloma de C. elegans jovens versus idosos, quimicamente marcados usando reagentes iTRAQ ('marcação isobárica para quantificação relativa e absoluta') e, em seguida, quantificados usando métodos baseados em MS. Usando um método de marcação isobárica e 120 mg de vermes úmidos (cerca de 40.000 vermes), conseguimos obter profundidade e cobertura significativas de insolubloma proteico5. A análise quantitativa demonstrou que 203 das 1200 proteínas identificadas foram significativamente enriquecidas no insolubloma de C. elegans envelhecidos em comparação com frações insolublomas semelhantes de vermes jovens5. Independentemente, David et al. também utilizaram um fluxo de trabalho iTRAQ LC-MS/MS para examinar alterações em agregados de proteínas com o envelhecimento normal2. Começando com cerca de 300 mg de vermes, eles identificaram ~ 1000 proteínas insolúveis usando duas réplicas biológicas e determinaram que ~ 700 de cerca de 1000 proteínas se acumularam 1,5 vezes ou mais com a idade em comparaçãocom os vermes jovens. No geral, esses resultados independentes indicam que a insolubilidade e agregação generalizada de proteínas são uma parte inerente do envelhecimento normal e podem afetar tanto a expectativa de vida quanto a incidência de doenças neurodegenerativas 2,5.
O estudo do insolublome nos permitiu determinar como as influências ambientais podem acelerar ou desacelerar o processo de envelhecimento. Klang et al. estabeleceram fluxos de trabalho proteômicos sem marcação em C. elegans para investigar o papel da metallostasia na longevidade10. Neste estudo, pelo menos 40.000 vermes foram usados para extrair o insolubloma10. Os dados mostraram que os níveis de ferro, cobre, cálcio e manganês aumentam com o envelhecimento e que alimentar os vermes com uma dieta com ferro elevado acelerou significativamente o acúmulo de proteínas insolúveis relacionado à idade10. Usando o mesmo fluxo de trabalho para examinar os efeitos da vitamina D no insolúvel de C. elegans, 38 proteínas foram quantificadas em vermes jovens (Dia 2) e 721 proteínas em vermes idosos (Dia 8). A alimentação com vitamina D reduziu significativamente a insolúvel de vermes idosos de 721 para 371 proteínas11. Investigações posteriores revelaram que a alimentação com vitamina D suprimiu a insolubilidade proteica com a idade, promoveu a homeostase proteica e prolongou a vida útil em vermes do tipo selvagem C . elegans N211. Assim, estudar o insolubloma pode ajudar a identificar novos moduladores do envelhecimento e doenças relacionadas à idade.
Embora o estudo do insolubloma tenha sido inestimável no progresso da compreensão do processo de envelhecimento, ele foi prejudicado pela necessidade de coletar grandes quantidades de material de amostra inicial. Groh et al. introduziram recentemente um fluxo de trabalho de quantificação proteômica sem marcação para estudar as mudanças inerentes à agregação de proteínas em C. elegans com o envelhecimento; no entanto, exigiu grandes quantidades de material de partida (350 mg de minhocas terrestres)12. No presente relatório, estabelecemos um novo protocolo de extração e isolamento aprimorado (Figura 1). O uso do sonicador altamente eficiente durante a lise do sem-fim melhorou significativamente a eficiência da extração e, subsequentemente, reduziu a quantidade de material de partida necessária, de 40.000 para 3.000 sem-fim. A combinação deste novo protocolo de isolamento de insolublome com um fluxo de trabalho de espectrometria de massa de aquisição independente de dados (DIA) sem marcação melhorou significativamente a profundidade e a cobertura da proteína. O protocolo aqui apresentado é econômico e facilmente modificado para permitir a realização de análises de insolublomas em outros sistemas modelo.
Protocolo
NOTA: Para uma melhor compreensão do procedimento experimental, consulte a Figura 1 para obter um esquema do fluxo de trabalho.
1. Cultura de massa de C. elegans de envelhecimento sincronizado
- Preparação da placa
- Despeje 30 mL de ágar NGM com ou sem 50 mM de fluorodesoxiuridina (FUdR) em cada placa de 90 mm e deixe as placas secarem por 2 dias em capelas de cultura.
- Na véspera da semeadura da placa de ágar NGM, inocular uma alíquota de 50 μL de estoque de glicerol congelado da cepa bacteriana OP50 em 1 L de meio LB pré-autoclavado em um balão de 2 L.
- Cultivar as bactérias a 37 °C numa incubadora agitável a 250 rpm durante 16 h.
- Após 16 h, centrifugue as culturas bacterianas a uma velocidade de 5.000 x g por 10 min.
- Descarte o sobrenadante e ressuspenda completamente o pellet bacteriano em 40 mL de solução S-basal autoclavada (contendo 50 mM K3PO4, 100 mM NaCl em água) para gerar a cultura de E. coli OP50 concentrada 25x.
- Dispense 2 mL da cultura bacteriana concentrada 25x em cada placa de ágar NGM e espalhe uniformemente para que a cultura bacteriana cubra toda a superfície do ágar.
- Remova as tampas das placas em uma coifa de cultura bacteriana por 2–3 h até que as placas estejam devidamente secas.
NOTA: Certifique-se de distribuir uniformemente as bactérias girando as placas a cada 15 minutos. Isso é importante, pois as bactérias tendem a se concentrar em um lado da placa, resultando em regiões com baixa densidade bacteriana. - Depois que as placas estiverem secas, feche as tampas e deixe-as em temperatura ambiente por 48 h para garantir a secagem completa da fonte de alimento OP50 E. coli .
NOTA: As placas podem ser preparadas com 2 semanas de antecedência e armazenadas a 4 °C antes do uso.
- Manutenção e preparação de coortes sincronizadas de C. elegans
- Para obter a entrada necessária de lisado de proteína (1,0–1,5 mg) para isolamento da fração insolúvel de SDS, use aproximadamente 3.000 vermes sincronizados por amostra.
- Transfira ~ 100 vermes adultos grávidos do tipo selvagem N2 para uma placa semeada por E. coli NGM 25x OP50 e mantenha os vermes a 20 ° C por ~ 72 h até que a placa esteja cheia de vermes adultos grávidas.
- Pipetar ~ 10 mL de solução S-basal em cada placa e coletar todos os vermes em um tubo de 15 mL.
NOTA: É importante usar pontas de baixa retenção ou pipetas Pasteur durante esta e todas as etapas futuras envolvendo a coleta de minhocas para evitar qualquer perda de minhocas devido à fixação às superfícies das pontas. - Girar a amostra a 520 x g durante 30 s. Remova o sobrenadante após girar. Para eliminar qualquer E. coli OP50, adicione 10 mL de solução S-basal ao pellet e gire novamente, seguido pela remoção do sobrenadante. Repita esta etapa.
- Adicione 10 mL de solução clareadora de hipoclorito de sódio (contendo 0,5 M KOH e 0,48% de hipoclorito de sódio em água) ao pellet de minhoca em um tubo cônico de 15 mL e vórtice vigorosamente por 2 min em temperatura ambiente.
- Centrifugue a 520 x g por 30 s à temperatura ambiente para centrifugar quaisquer corpos de vermes e ovos incompletamente dissolvidos. Em seguida, remova o sobrenadante.
- Repita as etapas 1.2.5 e 1.2.6 para garantir a remoção completa de todos os corpos de vermes e a liberação de ovos (view tubo sob microscópio para garantir).
- Lave o pellet de ovo pelo menos 4 vezes com 10 mL de solução S-basal para garantir a remoção completa da solução de alvejante.
NOTA: A remoção completa do alvejante é importante, pois o alvejante restante pode dificultar a eclosão dos ovos. - Deixar os ovos ressuspensos em 5 ml de meio basal S num tubo de 15 ml e colocá-los no rotor a 20 °C durante 24 h para permitir a eclosão dos ovos.
- Após 24 h, tome 10 μL de solução de larvas S-basais para contar o número de vermes nela. Utilizar pelo menos três alíquotas separadas de 10 μL de solução de larvas S-basais para a replicação da contagem de vermes. Calcule a média das três concentrações larvais. Com base na concentração média, dilua a solução de vermes para obter ~ 3.000 larvas por 200 μL de solução.
NOTA: Certifique-se de agitar o tubo suavemente invertendo, pois as larvas tendem a se assentar se não forem perturbadas e isso pode afetar a contagem de larvas. - Misture bem e adicione 200 μL de solução de larvas S-basais em cada uma das placas semeadas por bactérias NGM de 90 mm.
NOTA: Adicione gotas de solução de larvas S-basais em toda a placa para distribuição uniforme das larvas para evitar a depleção bacteriana de certos pontos. - Deixar secar a solução basal S e, em seguida, colocar as placas invertidas numa incubadora a 20 °C durante 48 h.
- Após 48 h, recolher os vermes da placa em solução S-basal e transferi-los para uma nova placa semeada com NGM contendo 50 mM de FUDR e incubar as placas a 20 °C.
NOTA: É importante transferir vermes no início do estágio L4 para garantir a eficiência do FUDR. - Colete amostras de adultos jovens do dia 2 após 48 h, adicionando 10 mL de solução S-basal na placa e transferindo a solução para um tubo de 15 mL. Permita que os vermes se assentem por gravidade e remova a solução basal S depois que ela se tornar clara.
- Lave o pellet de minhoca 2-3x em 5 mL de solução S-basal para remover qualquer bactéria aderida. Remova o máximo possível de solução S-basal e congele o pellet de minhoca (em tubos de 15 mL) em um banho de gelo seco/etanol. As amostras congeladas são armazenadas a -80 °C.
- Transfira os vermes de amostra para uma nova placa NGM OP50 E. coli contendo FUDR a cada dia alternado até o dia 10. Colete e congele os vermes conforme descrito nas etapas 1.2.14 e 1.2.15.
NOTA: Comece com um número maior de vermes para amostras do dia 10 para contabilizar qualquer perda no número de vermes devido à transferência e escavação.
2. Extração da fração insolúvel de SDS de vermes
- Descongele o pellet de minhoca congelado no gelo na presença de 900 μL de coquetel de tampão de lise de minhoca gelada contendo 20 mM de base Tris, pH 7,4, 100 mM de NaCl, 1 mM de MgCl2 e inibidor de protease livre de EDTA. Amostras de vórtice resumidamente para garantir a ressuspensão completa do pellet de minhoca no tampão de lise.
- Coloque os tubos de 15 mL no banho de sonicação e ajuste para 10 ciclos de sonicação (30 s ligado e 30 s desligado para cada ciclo) em alta intensidade. Repita por até cinco ciclos (certifique-se de que os vermes estejam totalmente lisados).
- Gire o lisado a 3.000 x g por 4 min na câmara fria. Após a centrifugação, descarte o pellet contendo quaisquer detritos de vermes e transfira cuidadosamente o sobrenadante para tubos de centrífuga de 1,5 mL pré-resfriados no gelo.
- Quantificar a concentração de proteínas utilizando o ensaio BCA. Transfira alíquotas de lisado, normalmente contendo cerca de 1 mg de proteína, para tubos de centrífuga de 1,5 mL pré-resfriados frescos.
- Centrifugue o lisado de proteína por 15 min a 20.000 x g na câmara fria.
- Sem perturbar o pellet, transfira o sobrenadante para um novo tubo de centrífuga de 1,5 mL e guarde-o como fração de proteína solúvel em água.
- Lave o pellet da etapa anterior em 500 μL de tampão de lise de verme contendo 1% de SDS à temperatura ambiente e centrifugue a 20.000 x g por 15 min em temperatura ambiente. Retirar o sobrenadante e conservar como fracção solúvel em SDS. Repita esta etapa de lavagem duas vezes para remover qualquer fração solúvel em SDS. O pellet restante após a terceira rodada de lavagem é definido como a fração de proteína insolúvel em SDS a 1%.
- Ressuspenda o pellet de proteína insolúvel SDS em 60 μL de ácido fórmico a 70% e vore vigorosamente para dissolver as proteínas. Repita o vórtice vigoroso quantas vezes forem necessárias para dissolver o pellet.
- Sonicar o pellet durante 30 min num banho-maria por ultrassónico à temperatura ambiente.
NOTA: O pellet insolúvel SDS é normalmente difícil de dissolver no tampão. No entanto, no final, todo o pellet deve ser dissolvido nesta etapa. - Seque as amostras em um concentrador a vácuo por 1 h para remover completamente a solução de ácido fórmico.
- Adicione 40 μL de 1x tampão de gel de amostra LDS ao pellet seco e aqueça a amostra a 95 °C por 10 min. Um breve vórtice e gire as amostras. Carregue 13 μL em um gel NUPAGE Bis-Tris 4-12% e execute o gel. Manchar o gel com uma coloração de proteína fluorescente para obter imagens. Guarde o restante da amostra para análise de espectrometria de massa (MS).
- Carregue a amostra restante em um gel Bis-Tris de 4-12% e execute por cerca de 20 minutos para digestão em gel MS.
3. Digestão em gel com protease de tripsina para isolar proteínas para análise de MS
- Prepare as seguintes soluções (frescas): 25 mM NH4HCO3 (pH 7–8), 25 mM NH4HCO3 em acetonitrila a 50% (ACN, pH 7–8) e ACN a 50% em ácido fórmico a 5%.
- Corte cada fatia de gel em pedaços pequenos (normalmente <1 mm2) e coloque-os em tubos siliconizados de 0,65 mL.
- Adicione cerca de 100 μL (ou o suficiente para cobrir) de uma solução de 25 mM NH4HCO3/50% ACN e vórtice em temperatura ambiente por 10 min. Extrair o sobrenadante e transferir para um tubo separado (a eliminar). Repita esta etapa mais duas vezes.
- Seque completamente os pedaços de gel em um concentrador a vácuo (~20 min).
- Prepare soluções frescas e adicione ~ 100 μL (ou o suficiente para cobrir) de 10 mM DTT em 25 mM NH4HCO3 aos pedaços de gel secos (use NH4HCO3 recém-preparado). Vórtice e gire brevemente. Deixar a reação prosseguir a 56 °C durante 1 h e agitar a 1400 rpm num misturador.
- Remova o sobrenadante e adicione 100 μL de iodoacetamida (IAA) 55 mM em 25 mM NH4HCO3 às peças de gel. Vórtice e gire brevemente. Permita que a reação prossiga no escuro por 45 min em RT.
- Remova o sobrenadante e descarte. Lave os pedaços de gel adicionando ~ 100 μL de 25mM NH4HCO3 e vórtice por 10 min. Gire brevemente e remova o sobrenadante, descartando-o.
- Desidrate os pedaços de gel adicionando ~ 100 μL (ou o suficiente para cobrir) de 25 mM NH4HCO3 em 50% ACN aos pedaços de gel e vórtice por 10 min. Em seguida, gire brevemente para remover e descartar o sobrenadante. Repita esta etapa duas ou até uma terceira vez se os pedaços de gel não estiverem muito secos, conforme indicado pelo tamanho menor dos pedaços de gel e pela cor branca turva.
- Seque completamente os pedaços de gel em um concentrador a vácuo (~20 min).
- Inicialmente, adicione 15 μL de solução de tripsina (250 ng de tripsina) a cada amostra e, em seguida, adicione 25 mM NH4HCO3 suficiente para cobrir os pedaços de gel (~ 100 μL). Vórtice por 10 min. Em seguida, gire brevemente e incube a 4 °C na câmara fria por 30 min sem misturar.
- Adicione 25 mM NH4HCO3 conforme necessário para cobrir completamente os pedaços de gel. Gire e incube a 37 ° C durante a noite por 16–20 h a 1400 rpm em um misturador.
- No dia seguinte, faça um vórtice breve e gire a digestão. Adicione cerca de 100 μL de água de grau HPLC, gire, parafilme o tubo e sonice continuamente por 10 min. Gire brevemente após a sonicação.
- Transfira a solução de digestão, representando a extração aquosa, para um tubo siliconizado limpo de 0,65 mL.
- Adicione ~ 100 μL de 50% de ACN / 5% de ácido fórmico aos pedaços de gel (o suficiente para cobri-los), vórtice 10 min à temperatura ambiente, gire brevemente e colete a solução e repita uma vez. Agrupe todas as soluções contendo os peptídeos extraídos desta etapa e da etapa 3.13 anterior em um único tubo.
- Vórtice as digestões extraídas. Seque os peptídeos completamente em um concentrador a vácuo (~ 2 h).
- Adicione 30 μL de ácido fórmico a 0,2% para ressuspender os peptídeos em um misturador na câmara fria por 10 min.
- Gire as amostras a 1850 x g em temperatura ambiente por 5 min. Aspire a solução peptídica e passe para um novo tubo siliconizado limpo de 0,65 mL. Em seguida, dessalinize a solução de peptídeos usando pontas de dessalinização C18 (veja abaixo).
4. Dessalinização de peptídeos digeridos com ponta de dessalinização C18
- Ajuste a pipeta para 10 μL e coloque a ponta de dessalinização C18. Molhe a ponta de dessalinização pipetando 10 μL de 100% de ACN e depois descartando-a. Repita esta etapa 2x.
- Lave a ponta de dessalinização C18 pipetando 10 μL de 50% de ACN, 49,8% de água e 0,2% de FA e depois descartando-a. Repita esta etapa 2x.
- Equilibre a ponta de dessalinização C18 pipetando 10 μL de 0,2% de FA em água e depois descartando-a. Repita esta etapa 2x.
- Defina a pipeta para 10 μL e carregue os peptídeos da solução para a resina pipetando os peptídeos digeridos através da resina para cima e para baixo 15x.
NOTA: A ação repetida garantirá a ligação de todos os peptídeos à resina das pontas de dessalinização. - Dessalinize os peptídeos digeridos ligados à resina pipetando 10 μL de 0,2% de FA em água e depois descartando-os. Repita esta etapa 4x para concluir a etapa de dessalinização.
- Em um novo tubo, eluir os peptídeos com 10 μL de 50% de ACN, 49,8% de água e 0,2% de FA pipetando para cima e para baixo 10x. Repita esta etapa uma vez para eluir o digerir da proteína pela segunda vez no mesmo tubo.
- Seque completamente os peptídeos dessalinizados em um concentrador a vácuo (~20 min).
- Ressuspenda peptídeos em 15 μL de 0,2% FA + 1 μL de peptídeos iRT (tempo de retenção indexado). Voxtex por 10 min, centrifugue a 12.000 x g por 2 min e, em seguida, transfira para um frasco de amostrador automático para análise de MS (veja abaixo).
5. Análise por espectrometria de massa de peptídeos digeridos usando DDA e DIA
NOTA: As amostras podem ser analisadas usando os métodos DDA ou DIA LC-MS/MS. Neste estudo, as amostras foram analisadas usando um sistema de HPLC nano-LC 2D acoplado a um espectrômetro de massa de alta resolução.
- Utilizar um sistema de HPLC combinado com um sistema de HPLC baseado em chip directamente ligado a um espectrómetro de massa de tempo de voo quadrupolo (podem também ser utilizadas outras configurações e sistemas LC-MS).
- Analise amostras usando HPLC-ESI-MS/MS de fase reversa.
- Construa o protocolo de cromatografia para carregar a amostra de peptídeos em um chip de pré-coluna C18. Lave e dessalinize os peptídeos carregados com solvente de carga (ácido fórmico a 0,1%) por 10 min a uma taxa de fluxo de 2 μL / min.
- Transferir os peptídeos para uma coluna analítica C18 e eluir a um caudal de 300 nL/min com um gradiente de 3 horas utilizando a fase móvel A (acetonitrila a 2%, ácido fórmico a 0,1%) e B (acetonitrila a 98%, ácido fórmico a 0,1%). A primeira etapa de eluição consiste em um gradiente linear de 5% B a 35% B durante um período de 80 min.
- Aumente a fase móvel B para 80% em 5 min e, em seguida, mantenha a fase móvel B em 80% por 8 min antes de mudar para 5% B para reequilibrar a coluna por 25 min.
- Configure um método de instrumento MS para DDA e defina os parâmetros do instrumento da seguinte forma.
- Use os seguintes parâmetros para o Experimento 1: varredura de íons precursores MS1 de m / z 400–1500 (tempo de acumulação de 250 ms); definir o limite de intensidade para acionar varreduras de MS/MS para íons de estados de carga 2–5 a 200 contagens; defina a exclusão dinâmica de íons precursores para 60 s).
- Use os seguintes parâmetros para o Experimento 2: Varreduras de íons de produtos MS/MS com uma faixa de varredura MS2 de m/z 100–1500 (tempo de acumulação de 100 ms por cada uma das 30 varreduras de íons de produtos por ciclo); defina a propagação da energia de colisão para CES=5 e marque 'modo de varredura de íons de produto de alta sensibilidade'.
NOTA: O método de aquisição DDA é usado para construir bibliotecas espectrais conforme descrito na etapa 6.3 (veja abaixo). Aqui, ele adquirirá espectros MS / MS para os 30 íons precursores mais abundantes após cada varredura MS1 por ciclo; O tempo total do ciclo é de cerca de 3,3 s.
- Obtenha o DIA. Configure um método de instrumento MS para o DIA e defina os parâmetros instrumentais da seguinte forma.
- Use os seguintes parâmetros para o Experimento 1: varredura de íons precursores MS1 de m / z 400-1250 (tempo de acumulação de 250 ms).
- Use os seguintes parâmetros para o Experimento 2: varreduras de íons de produtos MS/MS para 64 segmentos SWATH variáveis com uma faixa de varredura MS2 de m/z 100-1500 (tempo de acumulação de 45 ms por cada uma das 64 varreduras de íons de produtos por ciclo); defina a propagação da energia de colisão para CES=10, marque 'modo de varredura de íons de produto de alta sensibilidade'.
NOTA: Use o método de aquisição DIA/SWATH de janela de 64 variáveis, conforme descrito por Schilling et al.13 para realizar a quantificação sem marcação com um tempo de ciclo total de cerca de 3,2 s. Para aquisição de DDA, em vez de usar o quadrupolo Q1 para transmitir o íon precursor de uma faixa de massa estreita para a célula de colisão, uma faixa de largura de janela variável (por exemplo, m / z 5-90) é usada para passar incrementalmente sobre a faixa m / z completa (m / z 400-1250) com 64 segmentos SWATH, tempo de acumulação de 45 ms para cada segmento, resultando em um tempo de ciclo de 3,2 segundos, que inclui uma varredura MS1 com tempo de acúmulo de 250 ms. A largura da janela variável é ajustada de acordo com a complexidade da corrente iônica MS1 típica observada dentro de uma determinada faixa m/z usando um algoritmo SCIEX 'calculadora de janela variável'13 (janelas mais estreitas são aplicadas a faixas m/z 'ocupadas', janelas mais largas são aplicadas a faixas m/z com poucos íons precursores eluidores). Em outras plataformas MS, estratégias alternativas de seleção de janela DIA também podem ser implementadas.
6. Análise dos dados
NOTA: Certas configurações de análise de dados devem ser adaptadas a condições experimentais específicas. Por exemplo, o banco de dados de proteínas (arquivo fasta) selecionado dependerá da espécie a partir da qual a amostra foi preparada (neste protocolo usamos C. elegans).
- Use um mecanismo de pesquisa de banco de dados MS para analisar as aquisições de DDA e identificar proteínas. Gere um método de mecanismo de pesquisa darabase da seguinte maneira:
- Para Parâmetros de descrição de amostra, selecione Identificação; em Tipo de amostra, selecione Ácido iodoacético; em Alquilação de cisteína, selecione Tripsina; em Digestão (assumindo clivagem C-terminal em lisina e arginina), selecione o nome do espectrômetro de massa em Instrumento; e selecione Caenorhabditis elegans em Espécies.
- Para parâmetros de Processamento Específicos, verifique Modificações biológicas; em ID Focus, selecione SwissProt; em Banco de dados, marque ID completo; em Esforço de pesquisa, selecione 0,05 (10%) em Limite de proteína detectado; e marque Executar análise de taxa de descoberta falsa em Qualidade de resultados. Salve o método do mecanismo de pesquisa e envie os arquivos brutos do MS para o mecanismo de pesquisa do banco de dados para processamento, usando o método gerado.
NOTA: Em um processo iterativo, todas as varreduras MS e MS/MS foram recalibradas automaticamente pelo mecanismo de pesquisa com base nas anotações e resultados iniciais.
- Clique em Exportar Resumo de Peptídeos após a conclusão da pesquisa e filtre todos os resultados de identificação de peptídeos pelo limite de confiança de 99 no Excel (taxa de descoberta falsa (FDR) = 1%).
- Construa bibliotecas espectrais MS/MS para processamento adicional do arquivo de dados brutos do DIA e para quantificação adicional de dados relativos.
- Abra o software de análise quantitativa DIA. Selecione a guia Biblioteca e, em seguida, (na parte inferior da página) clique em Gerar Biblioteca Espectral a partir do "Mecanismo de Pesquisa de Banco de Dados" e abra um relatório FDR "Mecanismo de Pesquisa de Banco de Dados" (o arquivo de planilha *FDR.xlsx) que foi gerado automaticamente como parte do processo de pesquisa de banco de dados de arquivo de dados brutos DDA.
- Clique em Avançar, selecione o Esquema de Configurações da Biblioteca e clique em Avançar. Selecione Uniprot_Caenorhabditis elegans _proteome como banco de dados e clique em Concluir, a biblioteca espectral será gerada.
- Use o software de análise quantitativa DIA para analisar as aquisições de DIA para quantificação abrangente de proteínas relativas. O pipeline de análise de dados DIA é descrito a seguir.
- Para analisar e quantificar peptídeos, abra o software de análise quantitativa DIA para configurar um esquema de análise usando o esquema de modelo. O esquema do modelo está disponível no software clicando em Configurações, depois em Análise DIA e, finalmente, em Configurações de fábrica BGS.
- Configure os parâmetros no esquema de modelo da seguinte forma:
1) em identificação, selecione localização PTM (corte de probabilidade = 0,75).
2) em quantificação, selecione Quantidade do Grupo Principal como Quantidade de peptídeos de Soma, configure o Grupo Principal N como Máx. 7 e Mín 1, selecione Quantidade de Grupo Menor como Quantidade precursora de Soma e configure o Grupo Menor N como Máx. 10 e Mín 1, e selecione Filtragem de Dados como Qvalue esparso e não selecione Normalização de execução cruzada.
- Realize a análise de quantificação relativa.
- Selecione a guia Pipeline , clique em Configurar uma análise DIA a partir do arquivo, abra os arquivos brutos do MS DIA de interesse (por exemplo: amostra e controle), clique em Atribuir biblioteca espectral, selecione a biblioteca que foi gerada na versão 6.3, clique em Carregar e, por último, clique em Avançar.
- Selecione o esquema de análise configurado na versão 6.5.1 e clique em Avançar. Selecione o arquivo fasta do banco de dados apropriado (neste protocolo: Uniprot_Caenorhabditis elegans_proteome) e clique em Avançar. Defina a configuração da condição, atribua as diferentes condições de acordo com as amostras e clique em Avançar.
- Revise a visão geral da análise (resumo da configuração do experimento) e navegue até o Diretório de Saída e clique em Concluir. Por fim, clique em Executar Pipeline para executar a análise quantitativa sem rótulos.
NOTA: Os módulos estatísticos no 'Software de Análise Quantitativa DIA' executam automaticamente a análise FDR, geram mapas de calor e gráficos de vulcões comparando as diferentes condições, geram listas de peptídeos e proteínas identificados e quantificados e fornecem valores Q junto com mudanças relativas de dobra comparando diferentes condições. Aqui, a configuração das condições de amostra incluiu vermes envelhecidos do Dia 10 com duas réplicas biológicas e vermes jovens do Dia 2 com duas réplicas biológicas como controle.
Resultados
Os métodos tradicionais de lise de vermes têm várias desvantagens. Por exemplo, os métodos de sonicação baseados em sonda e batedor de esferas produzem calor excessivo, permitindo o contato da ponta ou esferas de metal diretamente com as amostras, resultando em recuperações variáveis de proteínas e desnaturação de proteínas. A moagem de nitrogênio líquido seguida de sonicação em tampão de lise pode ser demorada e requer um grande número de vermes. Devido às limitações dos métodos tradicionais de lise de vermes, os fluxos de trabalho anteriores de MS, como os métodos de rotulagem iTRAQ ou métodos sem rótulo que têm sido historicamente usados no sistema modelo de C. elegans para obter informações quantitativas sobre o insolublome, exigem grande entrada de material de partida (pelo menos 40.000 vermes). É necessário um trabalho trabalhoso de cultura de minhocas para obter esse número de minhocas. Além disso, os métodos de rotulagem exigem rótulos químicos isobáricos caros. Os métodos de quantificação sem rótulo são econômicos e têm métodos de preparação e rotulagem de amostras mais fáceis e diretos, mas exigem um número significativamente grande de vermes para atingir cobertura suficiente de análise de MS.
O sonicador que usamos aumenta muito a eficiência e a reprodutibilidade da lise do verme ao lisar várias amostras de vermes simultaneamente em um sonicador de banho-maria com temperatura controlada sem contaminação cruzada14, reduzindo significativamente a quantidade de material inicial do sem-fim necessário. Combinando o método de sonicação altamente eficiente e a abordagem quantitativa de MS sem marcação DIA, fomos capazes de quantificar de forma robusta a insolúbula de vermes idosos e jovens usando ~ 3.000 vermes. Aqui testamos e validamos a eficiência do protocolo e comparamos a insolúvel de vermes idosos e jovens de uma cepa de vermes do tipo selvagem, N2-Bristol C. elegans. Aplicamos este protocolo para extrair e isolar o insolubloma de ~ 3.000 C. elegans N2 envelhecidos e jovens (duas réplicas biológicas para cada condição), seguido de análise de MS com um espectrômetro de massa de tempo de voo quadrupolo ou outros sistemas de MS usando uma combinação de aquisição dependente de dados (DDA) e aquisições independentes de dados (DIA / SWATH) para identificação e quantificação de proteínas. As proteínas insolúveis foram analisadas primeiro em um gel de gradiente Bis-Tris 4-12% para determinar a quantidade de proteína em cada amostra de insolubloma. Conforme demonstrado na Figura 2, a amostra de insolubloma de vermes envelhecidos N2 (pistas 2 e 3, experimentos de replicação biológica) tem significativamente mais proteína do que amostras de vermes jovens N2 (pistas 1 e 4, experimentos de replicação biológica).
Após a digestão em gel, os perfis proteicos do insolubloma foram analisados por HPLC-MS. Usando esse fluxo de trabalho, geralmente podemos identificar 1000-1500 proteínas e quantificar 500-1.000 proteínas da fração insolúvel SDS com alta reprodutibilidade (dados não publicados). Aqui, fomos capazes de quantificar 989 proteínas do insolúvel de N2-Bristol C. elegans analisando os dados do DIA e removendo a redundância: 768 proteínas foram significativamente enriquecidas e 27 proteínas foram significativamente diminuídas no insolúvel do verme N2 envelhecido (Dia 10) em comparação com o jovem (dia 2) usando uma mudança de dobra de pelo menos 1,5 e um valor Q inferior a 0,01 (Figura 3A). Como visto no gráfico do histograma (Figura 3B), a mudança de dobra de proteínas significativamente alteradas mostra uma distribuição normal. Os vermes envelhecidos demonstraram ser significativamente enriquecidos para o insolubloma: A maior mudança observada mostrou que a abundância relativa de proteínas no insolubloma foi 592 vezes maior nos vermes velhos versus jovens; e para 32 proteínas, a abundância relativa de proteínas no insolubloma foi >250 vezes maior nos vermes velhos versus jovens, indicando mudanças dramáticas no insolubloma com a idade.
Depois de extrair a lista de proteínas insolúveis que são significativamente aumentadas nos vermes envelhecidos e identificadas pela base de vermes (WS271), a via KEGG e a análise da Ontologia Genética (GO) foram realizadas para determinar as vias que são enriquecidas no insolublome envelhecido para obter informações biológicas sobre como elas se relacionam com o envelhecimento. A análise da via KEGG das proteínas identificadas neste estudo mostra o enriquecimento de várias vias envolvendo ribossomos, mitocôndrias, proteassoma e spliceossomo (Figura 4A). A análise da ontologia genética mostra que o insolubloma de vermes idosos compreende muitas proteínas em categorias específicas, incluindo mitocôndrica, de desenvolvimento, determinantes da expectativa de vida adulta e proteínas ribossômicas (Figura 4B e Tabela Suplementar 1A). Em seguida, comparamos a lista de proteínas identificadas neste estudo com trabalhos publicados anteriormente de David et al.2 e Mark et al.11, conforme demonstrado em diagramas de Venn (Figura 5, A, 5B). A comparação mostrou sobreposição significativa das proteínas identificadas 394/721 e 444/721 com o estudo de David et al (Figura 5A) e Mark et al. (Figura 5B), respectivamente. As vias biológicas reveladas pela análise KEGG do insolubloma deste estudo também foram identificadas no passado, validando assim nossa metodologia (Tabela Suplementar 1B). A identificação dessas vias e proteínas sugere que elas podem servir como candidatas para investigações biológicas adicionais em relação à sua função no contexto do envelhecimento.
Em resumo, o uso do método de sonicação eficiente permite a lise de várias amostras de vermes ao mesmo tempo em um ambiente com temperaturas bem controladas e contaminação cruzada reduzida para obter alta cobertura de proteína com significativamente menos material inicial do verme. A combinação do método de sonicação eficiente com um fluxo de trabalho de quantificação de proteínas sem marcação DIA forneceu resultados confiáveis e reprodutíveis para a quantificação de proteínas de vermes insolúveis.

Figura 1. Fluxo de trabalho experimental do protocolo. C. elegans foram cultivados e coletados em dias diferentes. Após a lise do verme com um sonicador, a fração de proteína insolúvel em SDS a 1% (insolublome) foi extraída e isolada do lisado. O insolubloma foi então digerido por meio de digestão de tripsina em gel e quantificado por espectrometria de massa DIA, seguida de análise bioinformática. Clique aqui para ver uma versão maior desta figura.
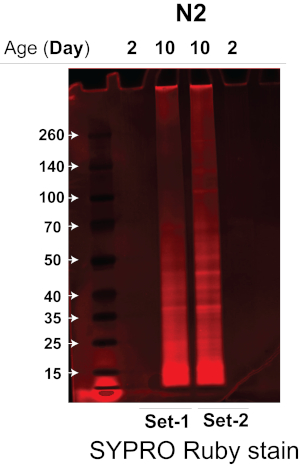
Figura 2. O gel SDS-PAGE do insolublome isolou vermes jovens versus velhos da cepa N2-Bristol. Os insolublemas de vermes jovens versus velhos da linhagem N2-Bristol foram analisados por SDS-PAGE para determinar a quantidade de proteína presente. O gel SDS-PAGE foi corado com uma coloração de proteína fluorescente para visualizar bandas de proteínas. Pistas 1 e 4: Insolublome de dois experimentos de replicação biológica de vermes N2 jovens (Dia 2). Pistas 2 e 3: Insolúvel de dois experimentos de replicação biológica de vermes N2 envelhecidos (Dia 10). Clique aqui para ver uma versão maior desta figura.

Figura 3. Candidatos a proteínas identificados como apresentando alteração significativa no insolubloma envelhecido versus jovem e suas distribuições de mudança de dobra. (A) Gráfico de vulcão para quantificação do insolúbulo de vermes N2-Bristol envelhecidos versus jovens. Os candidatos com uma alteração de dobra absoluta >=1,5 e valor Q <0,01 são mostrados como pontos vermelhos. (B) Gráfico de histograma para distribuição de mudança de dobra de proteínas insolúveis SDS significativamente enriquecidas em amostras de vermes envelhecidos versus jovens. Clique aqui para ver uma versão maior desta figura.

Figura 4. Via KEGG e análise de Ontologia Gênica (GO). (A) Análise da via KEGG do insolubloma do dia 10 organizado de acordo com o valor de p com a via altamente significativa mostrada na parte superior. (B) A análise da Ontologia Genética mostra que o insolubloma de vermes idosos é enriquecido para muitas proteínas em categorias específicas, incluindo proteínas mitocondriais, de desenvolvimento, determinantes da vida adulta e ribossomais. A exibição de gráfico de dispersão visualiza os termos GO em um "espaço semântico" onde os termos mais semelhantes são posicionados mais próximos. A cor da bolha reflete o valor de p obtido na análise STRING, enquanto seu tamanho reflete a generalidade do termo GO no banco de dados UniProt-GOA. Clique aqui para ver uma versão maior desta figura.

Figura 5. Sobreposição de proteína insolubloma identificada no insolubloma do dia 10 comparando este estudo com (A) David et al.2 e (B) Mark et al.11 estudos. Clique aqui para ver uma versão maior desta figura.
Tabela Suplementar 1 (relacionada à Figura 4 e Figura 5). (A) Ontologia gênica (processo biológico) analisada com o banco de dados STRING. (B) Lista detalhada de proteínas e vias KEGG identificadas neste estudo com códigos de cores que descrevem sua sobreposição com o trabalho publicado. Clique aqui para baixar este arquivo.
Discussão
Neste protocolo, relatamos um método aprimorado de preparo de amostras para a extração de proteínas insolúveis de C. elegans. Ao substituir a lise tradicional de vermes (por exemplo, sonicação por sonda ou técnicas de batedor de esferas) pelo sonicador eficiente, aumentamos o rendimento da extração de proteínas insolúveis e reduzimos o número de vermes necessários para a análise de MS sem marcação de 40.000 vermes para 3.000 vermes. Um mecanismo de pesquisa de banco de dados foi usado para identificação de proteínas a partir de dados DDA e a biblioteca espectral de C. elegans foi construída usando o 'DIA Quantitative Analysis Software' e os resultados de pesquisa de banco de dados DDA correspondentes (importando relatórios de arquivo FDR gerados pelo mecanismo de pesquisa de banco de dados). A quantificação relativa dos dados de insolubroma de C. elegans envelhecidos e jovens foi realizada usando o 'DIA Quantitative Analysis Software' para processar o novo conjunto de dados DIA e a biblioteca espectral gerada.
Várias etapas são críticas no protocolo. A curta vida útil de C. elegans o torna um sistema ideal para estudar o envelhecimento, em comparação com outros eucariotos, como células de mamíferos, mas é crucial isolar uma população homogênea de vermes ao estudar fenômenos relacionados ao envelhecimento. O FUDR foi usado neste protocolo para obter vermes de envelhecimento sincronizados. É importante transferir vermes no início de seu estágio L4 para a placa semeada com NGM contendo FUDR para garantir sua eficácia. Durante a lise do sem-fim usando o sonicador eficiente, a temperatura do banho-maria deve ser ajustada em 4 °C e a sonicação em 30 s ON e 30 s OFF para evitar o superaquecimento das amostras. Após a primeira rodada de sonicação por 10 ciclos (10 min), é importante verificar ao microscópio para garantir que todos os vermes foram lisados com eficiência. Caso contrário, são necessários mais ciclos de sonicação. Durante o processo de digestão em gel, cada fatia de gel deve ser cortada em pedaços do tamanho adequado (<1 mm2) - se muito pequenos, eles podem ser perdidos no processo de preparação da amostra e, se muito grandes, a digestão pode ser inadequada.
A necessidade de muito menos material de partida reduz significativamente o trabalho trabalhoso associado à cultura de minhocas para obter amostras para análises de insolubolos. No entanto, a extração e o isolamento da fração de proteína insolúvel em SDS a 1% envolvem várias etapas de lavagem e o manuseio cuidadoso da amostra é necessário para evitar a perda da amostra e garantir resultados reprodutíveis. A quantidade de material gerado para análise de MS é suficiente para ~ 3 injeções para análise subsequente de DDA e DIA, mas não para economizar para experimentos futuros. Além disso, apesar de seus efeitos potencialmente confundidores15, usamos a menor concentração possível de FUdR para esterilizar vermes durante o processo de envelhecimento. Estudos futuros podem contornar o uso de FUdR usando mutantes estéreis ou transferindo e coletando manualmente vermes.
O uso do sonicador altamente eficiente para lise de vermes permite a extração eficiente do insolubloma, permitindo boa cobertura de proteínas e análise DIA MS sem marcação econômica para quantificar o insolubloma usando um número muito reduzido de vermes. Reduz significativamente a carga de trabalho, permitindo a triagem de mais condições por experimento. Além disso, o fluxo de trabalho MS DIA sem rótulo é econômico e fornece profundidade e cobertura de proteína em níveis comparáveis aos métodos de rotulagem, incluindo iTRAQ, TMT ou SILAC. O modelo de C. elegans é um sistema de triagem rápida para pesquisa de envelhecimento. O fluxo de trabalho pode ser facilmente modificado e aplicado para estudar o envelhecimento e a pesquisa de doenças relacionadas à idade neste e em outros organismos. Por exemplo, em estudos em andamento, estamos aplicando esse fluxo de trabalho para investigar perfis proteicos de insolubloma e proteostase em vários modelos de C. elegans da doença de Alzheimer (DA), incluindo vermes Abeta, tau e Abeta / tau duplos com ou sem diferentes intervenções medicamentosas para futura triagem de drogas de alto rendimento.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi apoiado por uma bolsa de instrumentação compartilhada do NIH para um sistema TripleTOF (1S10 OD016281, Buck Institute), bolsa do NIH, RF1 AG057358 (GJL, JKA) e U01AG045844 de concessão do NIH (GJL). XX é apoiado por uma bolsa de pós-doutorado T32 (bolsa NIH 5T32AG000266, PI: Judith Campisi e Lisa Ellerby). MC é apoiado por uma bolsa de pós-doutorado da Fundação Larry L. Hillblom.
Materiais
| Name | Company | Catalog Number | Comments |
| Strains used | |||
| Esherichia coli OP50 | Caenorhabditis Genetics Center (CGC) | ||
| N2 (Bristol) | Caenorhabditis Genetics Center (CGC) | ||
| Buffer/Solution | |||
| NGM (Nematode Growth Media) | Recipe: 3 g/L NaCl, 23 g/L agar; 2.5 g/L peptone; 1 mM CaCl2, 5 mg/L cholesterol, 1 mM MgSO4, 25 mM KH2PO4 | ||
| S-basal solution | Recipe: 5.85 g/L NaCl, 1g/L K2HPO4, 6 g/L KH2PO4, H2O to 1 L | ||
| Sodium hypochlorite bleach solution | Recipe: Mix 0.5 mL 5 N NaOH with 1 ml Sodium hypochlorite (5%) and make volume to 5 mL with H20. | ||
| Material/ Equipment | |||
| Agar | Difco Granulated Agar, BD Biosciences | 90000-782 | |
| Bioruptor Plus sonication device | Diagenode, USA | B01020001 | |
| Cholesterol | Sigma | c8503 | |
| 2'-deoxy-5-fluorouridine | VWR | TCD2235 | |
| Glycerol | Millipore Sigma | 356350-1000ML | |
| LB broth, Miller | Millipore Sigma | 60801-450 | |
| Sodium dodecyl sulfate (SDS? | Sigma | L4509-250G | |
| Sodium chloride | Sigma | 59888 | |
| M880 Ultrasonic bath, 117 V, holds 5.5 gallons | VWR, USA | 89375-458 | |
| Magnesium sulphate | Sigma | M506 | |
| Magnesium chloride | Sigma | 208337 | |
| NGM agar plate | VWR Disposable Petri Dishes | 25384-342 | |
| NuPAGE LDS Sample Buffer (4X) | Thermo Fisher Scientific | NP0007 | |
| NuPAGE protein gels, 4-12% | Invitrogen | NP 0335BOX | |
| Protease inhibiotr cocktail (PIC) | Roche | 11836170001 | |
| Pierce BCA Assay | Thermo Fisher Scientific | 23225 | |
| Sodium hypochlorite 5% | VWR | JT9416-1 | |
| SYPRO Ruby Protein Gel Stain | Thermo Fisher Scientific | S12000 | |
| MS Section | |||
| Acetonitrile, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 36XL66 | |
| Agilent Zorbax 300Extend C18 column | Agilent Technologies Inc., Santa Clara, CA, USA | 770995-902 | |
| Ammonium bicarbonate | Sigma Aldrich, St. Louis, MO, USA | 9830 (1 kg) | |
| Dithiothreitol (DTT) | Sigma Aldrich, St. Louis, MO, USA | D9779-5G | |
| Eppendorf Thermomixer Compact | Eppendorf AG, Hamburg, Germany | T1317-1EA | |
| Formic acid | Sigma Aldrich, St. Louis, MO, USA | F0507-500ML | |
| Indexed retention time (iRT) normalization peptide standard | Biognosys AG, Schlieren, Zurich, Switzerland | Ki-3002-2 | |
| Iodoacetamide (IAA) | Sigma Aldrich, St. Louis, MO, USA | I1149-25G | |
| Methanol, HPLC Grade | Honeywell International Inc., Charlotte, NC, USA | 34885 | |
| Nano cHiPLC Trap ChromXP C18-CL, 200 um x 6 mm, 3 um, 120A. (pre-column chip) (200 um x 6 mm ChromXP C18-CL chip, 3 um, 300 A) | Sciex LLC, Framingham, MA, USA | 804-00006 | |
| Nano cHiPLC ChromXP 75 um by 15cm, C18-CL, 3 um, 120 A (analytical column chip) | Sciex LLC, Framingham, MA, USA | 804-00001 | |
| Orthoganol quadrupole time-of-flight (QqTOF) TripleTOP 6600 mass spectrometer | Sciex LLC, Framingham, MA, USA | Per quote | |
| ProteinPilot 5.0 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Savant SPD131DDA Speedvac Concentrator | Thermo Fisher Scientific, Waltham, MA, USA | SPD131DDA-115 | |
| Sequencing-grade lyophilized trypsin | Life Technologies | 23225 | |
| Spectronaut | Biognosys AG, Schlieren, Zurich, Switzerland | Sw-3001 | |
| SWATH 2.0 plugin into PeakView 2.2 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Ultra Plus nano-LC 2D HPLC system | Sciex LLC, Eksigent Division, Framingham, MA, USA | Model # 845 | |
| Water, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 600-30-76 | |
| Waters 1525 binary HPLC pump system | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters 2487 Dual Wavelength UV detector | Waters Corp., Milford, MA, USA | WAT081110 | |
| Waters 717plus Autosampler | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters Fraction Collector III | Waters Corp., Milford, MA, USA | 186001878 |
Referências
- Walther, D. M., et al. Widespread Proteome Remodeling and Aggregation in Aging C. elegans. Cell. , 919-932 (2015).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Labbadia, J., Morimoto, R. I. The Biology of Proteostasis in Aging and Disease. Annual Review of Biochemistry. (1), 435-464 (2013).
- Ross, C. A., Poirier, M. A. Protein aggregation and neurodegenerative disease. Nature Medicine. , 10-17 (2004).
- Reis-Rodrigues, P., et al. Proteomic analysis of age-dependent changes in protein solubility identifies genes that modulate lifespan. Aging Cell. 11 (1), 120-127 (2012).
- Ayyadevara, S., et al. Proteins that accumulate with age in human skeletal-muscle aggregates contribute to declines in muscle mass and function in Caenorhabditis elegans. Aging. 8 (12), 3486-3497 (2016).
- Huang, C., et al. Intrinsically aggregation-prone proteins form amyloid-like aggregates and contribute to tissue aging in Caenorhabditis elegans. eLife. 8, (2019).
- Morimoto, R. I. Proteotoxic stress and inducible chaperone networks in neurodegenerative disease and aging. Genes and Development. 22 (11), 1427-1438 (2008).
- Morimoto, R. I., Cuervo, A. M. Protein homeostasis and aging: Taking care of proteins from the cradle to the grave. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 64 (2), 167-170 (2009).
- Klang, I. M., et al. Iron promotes protein insolubility and aging in C. elegans. Aging. 6 (11), 975-991 (2014).
- Mark, K. A., et al. Vitamin D Promotes Protein Homeostasis and Longevity via the Stress Response Pathway Genes skn-1, ire-1, and xbp-1. Cell Reports. 17 (5), 1227-1237 (2016).
- Groh, N., et al. Methods to study changes in inherent protein aggregation with age in caenorhabditis elegans. Journal of Visualized Experiments. (129), 1-12 (2017).
- Schilling, B., Gibson, B. W., Hunter, C. L. Generation of High-Quality SWATH((R)) Acquisition Data for Label-free Quantitative Proteomics Studies Using TripleTOF((R)) Mass Spectrometers. Proteomics: Methods and Protocols, Methods in Molecular Biology. 1550, 223-233 (2017).
- Walther, D. M., et al. Widespread proteome remodeling and aggregation in aging C. elegans. Cell. 161 (4), 919-932 (2015).
- Angeli, S., et al. A DNA synthesis inhibitor is protective against proteotoxic stressors via modulation of fertility pathways in Caenorhabditis elegans. Aging. 5 (10), 759-769 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados