Method Article
Cuantificación de la agregación de proteínas insolubles en Caenorhabditis elegans durante el envejecimiento con un novedoso flujo de trabajo de adquisición independiente de datos
* Estos autores han contribuido por igual
En este artículo
Resumen
Este novedoso flujo de trabajo extrae y aísla de manera eficiente proteínas insolubles en SDS (insolubloma) de Caenorhabditis elegans con un material de partida mínimo para el análisis proteómico diferencial cuantitativo. El protocolo utiliza un análisis integral de espectrometría de masas de adquisición independiente de datos para cuantificar el insolubioma y un análisis bioinformático para obtener información biológica sobre los mecanismos y patologías del envejecimiento.
Resumen
Nosotros y otros hemos demostrado que el proceso de envejecimiento da como resultado una acumulación de proteínas insolubles en todo el proteoma. La eliminación de los genes que codifican las proteínas insolubles más del 40% de las veces da como resultado una extensión de la vida útil en C. elegans, lo que sugiere que muchas de estas proteínas son determinantes clave del proceso de envejecimiento. El aislamiento y la identificación cuantitativa de estas proteínas insolubles son cruciales para comprender los procesos biológicos clave que ocurren durante el envejecimiento. Aquí, presentamos un protocolo modificado y mejorado que detalla cómo extraer y aislar las proteínas insolubles en SDS (insolublome) de C. elegans de manera más eficiente para optimizar los flujos de trabajo de espectrometría de masas a través de un novedoso análisis proteómico cuantitativo sin etiquetas. Este protocolo mejorado utiliza un sonicador altamente eficiente para la lisis de lombrices que aumenta en gran medida la eficiencia para la extracción de proteínas y nos permite utilizar significativamente menos material de partida (aproximadamente 3.000 lombrices) que en los protocolos anteriores (normalmente utilizando al menos 40.000 gusanos). El análisis proteómico cuantitativo posterior del insolubioma se realizó utilizando la adquisición dependiente de datos (DDA) para el descubrimiento e identificación de proteínas y la adquisición independiente de datos (DIA) para una cuantificación completa y más precisa de proteínas. El análisis bioinformático de proteínas cuantificadas proporciona candidatos potenciales que pueden ser fácilmente seguidos con otros métodos moleculares en C. elegans. Con este flujo de trabajo, identificamos rutinariamente más de 1000 proteínas y cuantificamos más de 500 proteínas. Este nuevo protocolo permite un cribado eficiente de compuestos con C. elegans. Aquí, validamos y aplicamos este protocolo mejorado a la cepa N2-Bristol de C. elegans de tipo silvestre y confirmamos que los gusanos N2 envejecidos del día 10 mostraron una mayor acumulación del insoluloma que los gusanos jóvenes del día 2.
Introducción
La homeostasis de las proteínas disminuye progresivamente con el envejecimiento y da lugar a un aumento de la agregación de proteínas 1,2,3. La agregación de proteínas se asocia con varias enfermedades neurodegenerativas, como la enfermedad de Alzheimer, la enfermedad de Parkinson, la enfermedad de Huntington y la esclerosis lateral amiotrófica. El envejecimiento se considera uno de los principales factores de riesgo para la aparición de trastornos neurodegenerativos asociados a la agregación de proteínas. Las proteínas que son propensas a formar agregados insolubles a menudo están relacionadas con toxicidad celular y disfunción tisular, lo que podría acelerar aún más la agregación de otras proteínas 5,6,7. Alternativamente, los agregados de proteínas insolubles pueden activar los mecanismos de defensa celular para eliminar las formas oligoméricas tóxicas de la proteína del sistema. La eliminación de genes seleccionados que codifican proteínas insolubles modula la esperanza de vida de Caenorhabditis elegans (C. elegans) en el contexto de enfermedades relacionadas con la edad y el envejecimiento normal 5,8,9. Por lo tanto, el estudio de los mecanismos celulares y moleculares de la agregación de proteínas es crucial para comprender el envejecimiento y, en última instancia, puede conducir a enfoques para el tratamiento de enfermedades neurodegenerativas.
El nematodo C. elegans se ha convertido en uno de los organismos modelo más utilizados para estudiar la agregación de proteínas en el envejecimiento y las enfermedades relacionadas con la edad debido a sus características únicas, como una vida útil relativamente corta (alrededor de 2 semanas), la facilidad de cultivo y la manipulación genética.
La capacidad de extraer y caracterizar proteínas insolubles ha desempeñado un papel fundamental en la determinación de los cambios relacionados con la edad asociados con la agregación de proteínas en modelos de C. elegans. Para investigar la contribución de la agregación de proteínas a los procesos normales de envejecimiento,nosotros 5 y otros2 extrajeron previamente y digirieron proteolíticamente el insoluloma de C. elegans jóvenes frente a envejecidos, marcados químicamente utilizando reactivos iTRAQ ('marcaje isobárico para cuantificación relativa y absoluta'), y luego cuantificados utilizando métodos basados en MS. Utilizando un método de marcaje isobárico y 120 mg de lombrices húmedas (alrededor de 40.000 lombrices), pudimos obtener una profundidad y cobertura significativas de insolubioma proteico5. El análisis cuantitativo demostró que 203 de las 1200 proteínas identificadas estaban significativamente enriquecidas en el insolubioma de C. elegans envejecido en comparación con fracciones similares de insolubloma de gusanos jóvenes5. De forma independiente, David et al. también utilizaron un flujo de trabajo iTRAQ LC-MS/MS para examinar las alteraciones en los agregados de proteínas con el envejecimiento normal2. Comenzando con aproximadamente 300 mg de gusanos, identificaron ~ 1000 proteínas insolubles utilizando dos réplicas biológicas y determinaron que ~ 700 de aproximadamente 1000 proteínas se acumularon 1,5 veces o más con la edad en comparacióncon los gusanos jóvenes. En general, estos resultados independientes indican que la insolubilidad y la agregación generalizadas de proteínas son una parte inherente del envejecimiento normal y pueden afectar tanto a la esperanza de vida como a la incidencia de enfermedades neurodegenerativas 2,5.
El estudio del insoluloma nos ha permitido determinar cómo las influencias ambientales pueden acelerar o desacelerar el proceso de envejecimiento. Klang et al. establecieron flujos de trabajo proteómicos sin marcadores en C. elegans para investigar el papel de la metallostasis en la longevidad10. En este estudio, se utilizaron al menos 40.000 gusanos para extraer el insolublome10. Los datos mostraron que los niveles de hierro, cobre, calcio y manganeso aumentan con el envejecimiento y que alimentar a los gusanos con una dieta con hierro elevado aceleró significativamente la acumulación de proteínasinsolubles relacionada con la edad. Utilizando el mismo flujo de trabajo para examinar los efectos de la vitamina D en el insolubioma de C. elegans, se cuantificaron 38 proteínas en gusanos jóvenes (día 2) y 721 proteínas en gusanos envejecidos (día 8). La alimentación con vitamina D redujo significativamente el insoluloma de los gusanos envejecidos de 721 a 371 proteínas11. Investigaciones posteriores revelaron que la alimentación con vitamina D suprimía la insolubilidad de las proteínas con la edad, promovía la homeostasis de las proteínas y prolongaba la vida útil de los gusanos de tipo salvaje C. elegans N211. Por lo tanto, el estudio del insolubioma puede ayudar a identificar nuevos moduladores del envejecimiento y las enfermedades relacionadas con la edad.
Si bien el estudio del insolubioma ha sido invaluable para avanzar en la comprensión del proceso de envejecimiento, se ha visto obstaculizado por el requisito de recolectar grandes cantidades de material de muestra inicial. Groh et al. introdujeron recientemente un flujo de trabajo de cuantificación proteómica sin marcadores para estudiar los cambios inherentes a la agregación de proteínas en C. elegans con el envejecimiento; sin embargo, requirió grandes cantidades de material de partida (350 mg de lombrices molidas)12. En el presente informe, establecimos un nuevo protocolo de extracción y aislamiento mejorado (Figura 1). El uso del sonicador de alta eficiencia durante la lisis de gusanos mejoró significativamente la eficiencia de extracción y, posteriormente, redujo la cantidad de material de partida necesario, de 40.000 a 3.000 gusanos. La combinación de este novedoso protocolo de aislamiento de insolubloma con un flujo de trabajo de espectrometría de masas de adquisición independiente de datos (DIA) sin etiquetas mejoró significativamente la profundidad y la cobertura de las proteínas. El protocolo presentado aquí es rentable y fácilmente modificable para permitir la realización de análisis de insolubloma en otros sistemas modelo.
Protocolo
NOTA: Para una mejor comprensión del procedimiento experimental, consulte la Figura 1 para obtener un esquema del flujo de trabajo.
1. Cultivo masivo de C. elegans de envejecimiento sincronizado
- Preparación de la placa
- Vierta 30 mL de agar NGM con o sin 50 mM de fluorodesoxiuridina (FUdR) en cada placa de 90 mm y deje que las placas se sequen durante 2 días en campanas de cultivo.
- El día antes de sembrar la placa de agar NGM, inocular una alícuota de 50 μL de caldo de glicerol congelado de la cepa bacteriana OP50 en 1 L de medio LB preesterilizado en clave en un matraz de 2 L.
- Cultive las bacterias a 37 °C en una incubadora agitadora a 250 rpm durante 16 h.
- Después de 16 h, centrifugar los cultivos bacterianos a una velocidad de 5.000 x g durante 10 min.
- Deseche el sobrenadante y vuelva a suspender completamente el pellet bacteriano en 40 mL de solución S-basal esterilizada en autoclave (que contiene 50 mM K3PO4, 100 mM NaCl en agua) para generar el cultivo de E. coli OP50 concentrado 25x.
- Dispensar 2 mL del cultivo bacteriano concentrado 25x en cada placa de agar NGM y distribuir uniformemente para que el cultivo bacteriano cubra toda la superficie del agar.
- Retire las tapas de las placas en una campana de cultivo bacteriano durante 2-3 h hasta que las placas se sequen correctamente.
NOTA: Asegúrese de distribuir uniformemente las bacterias agitando las placas cada 15 minutos. Esto es importante ya que las bacterias tienden a concentrarse en un lado de la placa, lo que da como resultado regiones con baja densidad bacteriana. - Después de que las placas se sequen, cierre las tapas y deje las placas a temperatura ambiente durante 48 h para garantizar el secado completo de la fuente de alimento OP50 E. coli .
NOTA: Las placas pueden prepararse con 2 semanas de antelación y almacenarse a 4 °C antes de su uso.
- Mantenimiento y preparación de cohortes sincronizadas de C. elegans
- Para obtener el aporte requerido de lisado de proteínas (1,0-1,5 mg) para el aislamiento de la fracción insoluble en SDS, utilice aproximadamente 3.000 gusanos sincronizados por muestra.
- Transfiera ~100 gusanos adultos grávidos de tipo salvaje N2 a una placa NGM 25x OP50 con semillas de E. coli y mantenga los gusanos a 20 °C durante ~72 h hasta que la placa esté llena de gusanos adultos grávidos.
- Pipetee ~ 10 mL de solución S-basal en cada placa y recoja todos los gusanos en un tubo de 15 mL.
NOTA: Es importante utilizar puntas de baja retención o pipetas Pasteur durante este y todos los pasos futuros que impliquen la recolección de lombrices para evitar cualquier pérdida de gusanos debido a la fijación a las superficies de las puntas. - Centrifugar la muestra a 520 x g durante 30 s. Retire el sobrenadante después de girar. Para eliminar cualquier E. coli OP50, agregue 10 mL de solución S-basal al pellet y vuelva a centrifugar, seguido de la eliminación del sobrenadante. Repita este paso.
- Agregue 10 mL de solución blanqueadora de hipoclorito de sodio (que contiene 0,5 M de KOH y 0,48% de hipoclorito de sodio en agua) al pellet de gusano en un tubo cónico de 15 mL y vórtice vigorosamente durante 2 min a temperatura ambiente.
- Centrifugar a 520 x g durante 30 s a temperatura ambiente para centrifugar los cuerpos de gusanos y huevos disueltos incompletamente. A continuación, retire el sobrenadante.
- Repita los pasos 1.2.5 y 1.2.6 para asegurar la eliminación completa de todos los cuerpos de los gusanos y la liberación de los huevos (vea el tubo bajo el microscopio para asegurarse).
- Lave el pellet de huevo al menos 4 veces con 10 mL de solución S-basal para asegurar la eliminación completa de la solución de lejía.
NOTA: La eliminación completa de la lejía es importante, ya que la lejía restante puede dificultar la eclosión de los huevos. - Deje los huevos resuspendidos en 5 mL de medio basal S en un tubo de 15 mL y colóquelos en el rotor a 20 °C durante 24 h para permitir que los huevos eclosionen.
- Después de 24 h, tome 10 μL de solución de larvas S-basales para contar el número de lombrices que contiene. Utilice al menos tres alícuotas separadas de 10 μL de solución de larvas S-basales para replicar el recuento de gusanos. Calcular el promedio de las tres concentraciones de larvas. Según la concentración promedio, diluya la solución de lombriz para obtener ~ 3,000 larvas por 200 μL de solución.
NOTA: Asegúrese de agitar el tubo suavemente invirtiéndolo, ya que las larvas tienden a asentarse si no se les molesta y eso puede afectar el recuento de larvas. - Mezcle bien y agregue 200 μL de solución de larvas S-basales en cada una de las placas de 90 mm sembradas con bacterias NGM.
NOTA: Agregue gotas de solución de larvas S-basales en todo el plato para una distribución uniforme de las larvas y evitar el agotamiento bacteriano de ciertos puntos. - Deje secar la solución S-basal y luego coloque las placas invertidas en una incubadora a 20 °C durante 48 h.
- Después de 48 h, recoja los gusanos de la placa en una solución S-basal y transfiéralos a una placa fresca sembrada con NGM que contenga 50 mM de FUDR e incube placas a 20 °C.
NOTA: Es importante transferir los gusanos al principio de la etapa L4 para garantizar la eficiencia del FUDR. - Recoja muestras de adultos jóvenes del día 2 después de 48 h agregando 10 mL de solución basal S en la placa y transfiriendo la solución a un tubo de 15 mL. Permita que los gusanos se asienten por gravedad y elimine la solución basal S después de que se aclare.
- Lave el gránulo de lombriz 2-3 veces en 5 mL de solución S-basal para eliminar cualquier bacteria adherida. Retire la mayor cantidad posible de solución basal de S y congele el pellet de gusano (en tubos de 15 ml) en un baño de hielo seco/etanol. Las muestras congeladas se almacenan a -80 °C.
- Transfiera los gusanos de muestra a una placa nueva de E. coli NGM OP50 que contenga FUDR cada dos días hasta el día 10. Recoja y congele gusanos como se describe en los pasos 1.2.14 y 1.2.15.
NOTA: Comience con un número mayor de gusanos para las muestras del día 10 para tener en cuenta cualquier pérdida en el número de gusanos debido a la transferencia y la excavación.
2. Extracción de la fracción insoluble en SDS de los gusanos
- Descongele el pellet de gusano congelado en hielo en presencia de 900 μL de cóctel tampón de lisis de lombriz helado que contiene 20 mM de base Tris, pH 7,4, 100 mM de NaCl, 1 mM de MgCl2 e inhibidor de la proteasa sin EDTA. Muestras de vórtice breves para asegurar la resuspensión completa de la peleta de gusano en el tampón de lisis.
- Coloque los tubos de 15 ml en el baño de sonicación y configúrelos durante 10 ciclos de sonicación (30 s encendidos y 30 s apagados para cada ciclo) a alta intensidad. Repita hasta cinco ciclos (asegúrese de que los gusanos estén completamente lisados).
- Centrifugar el lisado a 3.000 x g durante 4 min en la cámara frigorífica. Después de la centrifugación, deseche el pellet que contenga cualquier residuo de gusano y transfiera con cuidado el sobrenadante a tubos de centrífuga nuevos preenfriados de 1,5 ml con hielo.
- Cuantificar la concentración de proteínas mediante el ensayo BCA. Transfiera las alícuotas de lisado, que normalmente contienen alrededor de 1 mg de proteína, a tubos de centrífuga de 1,5 ml recién enfriados y preenfriados.
- Centrifugar el lisado de proteínas durante 15 min a 20.000 x g en la cámara frigorífica.
- Sin alterar el pellet, transfiera el sobrenadante a un tubo de centrífuga nuevo de 1,5 ml y guárdelo como fracción de proteína soluble en agua.
- Lave el pellet del paso anterior en 500 μL de tampón de lisa de lombriz que contenga un 1% de SDS a temperatura ambiente y centrifugue a 20.000 x g durante 15 min a temperatura ambiente. Retire el sobrenadante y guárdelo como fracción soluble en SDS. Repita este paso de lavado dos veces para eliminar cualquier fracción soluble en SDS. El pellet restante después de la tercera ronda de lavado se define como la fracción de proteína insoluble en SDS del 1%.
- Vuelva a suspender el pellet de proteína insoluble en SDS en 60 μL de ácido fórmico al 70% y haga vórtice vigorosamente para disolver las proteínas. Repita el vórtice vigoroso tantas veces como sea necesario para disolver el pellet.
- Sonicar el pellet durante 30 min en un baño de agua ultrasónico a temperatura ambiente.
NOTA: El pellet insoluble en SDS suele ser difícil de disolver en el tampón. Sin embargo, al final todo el pellet debe disolverse en este paso. - Seque las muestras en un concentrador de vacío durante 1 h para eliminar completamente la solución de ácido fórmico.
- Añada 40 μL de 1x tampón de gel de muestra LDS al pellet seco y caliente la muestra a 95 °C durante 10 min. Realice un vórtice breve y gire las muestras. Cargue 13 μL en un gel NUPAGE Bis-Tris al 4-12% y ejecute el gel. Tiñe el gel con una tinción de proteína fluorescente para obtener imágenes. Guarde el resto de la muestra para el análisis de espectrometría de masas (MS).
- Cargue la muestra restante en un gel Bis-Tris al 4-12% y deje funcionar durante unos 20 minutos para la digestión en gel de MS.
3. Digestión en gel con tripsina proteasa para aislar proteínas para el análisis de EM
- Prepare las siguientes soluciones (frescas): 25 mM de NH4HCO3 (pH 7-8), 25 mM de NH4HCO3 en acetonitrilo al 50% (ACN, pH 7-8) y 50% de ACN en ácido fórmico al 5%.
- Corta cada rebanada de gel en trozos pequeños (normalmente <1 mm2) y colócalos en tubos siliconados de 0,65 mL.
- Agregue aproximadamente 100 μL (o lo suficiente para cubrir) de una solución de NH4HCO3/50% ACN de 25 mM y vórtice a temperatura ambiente durante 10 min. Extraiga el sobrenadante y transfiéralo a un tubo separado (para desechar). Repita este paso dos veces más.
- Seque completamente las piezas de gel en un concentrador de vacío (~20 min).
- Prepare soluciones frescas y agregue ~ 100 μL (o lo suficiente para cubrir) de 10 mM de DTT en 25 mM de NH4HCO3 a las piezas de gel secas (use NH4HCO3 recién preparado). Vórtice y gire brevemente. Deje que la reacción continúe a 56 °C durante 1 h y agitando a 1400 rpm en un mezclador.
- Retirar el sobrenadante y añadir 100 μL de 55 mM de yodoacetamida (IAA) en 25 mM de NH4HCO3 a las piezas de gel. Vórtice y gire brevemente. Permita que la reacción continúe en la oscuridad durante 45 minutos en RT.
- Retire el sobrenadante y deséchelo. Lave las piezas de gel agregando ~ 100 μL de 25 mM NH4HCO3 y agite el vórtice durante 10 min. Gire brevemente y retire el sobrenadante, desechando este último.
- Deshidrate las piezas de gel añadiendo ~100 μL (o lo suficiente para cubrir) de 25 mM de NH4HCO3 en 50% ACN a las piezas de gel y al vórtice durante 10 min. A continuación, gire brevemente para retirar y desechar el sobrenadante. Repita este paso dos veces o incluso una tercera vez si las piezas de gel no están completamente secas, como lo indica el tamaño más pequeño de las piezas de gel y el color blanco turbio.
- Seque completamente las piezas de gel en un concentrador de vacío (~20 min).
- Inicialmente agregue 15 μL de solución de tripsina (250 ng de tripsina) a cada muestra, y luego agregue suficientes 25 mM de NH4HCO3 para cubrir las piezas de gel (~ 100 μL). Vórtice durante 10 min. A continuación, centrifugar brevemente e incubar a 4 °C en la cámara frigorífica durante 30 minutos sin mezclar.
- Agregue 25 mM NH4HCO3 según sea necesario para cubrir completamente las piezas de gel. Centrifugar e incubar a 37 °C durante la noche durante 16-20 h a 1400 rpm en una mezcladora.
- Al día siguiente, haga un breve vórtice y gire la digestión. Agregue aproximadamente 100 μL de agua de grado HPLC, centrifuga, parafilm el tubo y sonique continuamente durante 10 minutos. Gira brevemente después de la sonicación.
- Transfiera la solución de digestión, que representa la extracción acuosa, a un tubo siliconado limpio de 0,65 mL.
- Agregue ~ 100 μL de 50% ACN / 5% de ácido fórmico a las piezas de gel (suficiente para cubrirlas), vórtice 10 min a temperatura ambiente, gire brevemente y recoja la solución, y repita una vez. Agrupe todas las soluciones que contengan los péptidos extraídos de este paso y del paso anterior 3.13 en un solo tubo.
- Vórtice las digestiones extraídas. Seque los péptidos completamente en un concentrador de vacío (~2 h).
- Añadir 30 μL de ácido fórmico al 0,2% para resuspender los péptidos en una batidora en la cámara frigorífica durante 10 min.
- Centrifugar las muestras a 1850 x g a temperatura ambiente durante 5 min. Aspire la solución peptídica y muévala a un nuevo tubo siliconado limpio de 0,65 ml. A continuación, desala la solución de péptidos utilizando puntas desalinizantes C18 (ver más abajo).
4. Desalinización de péptidos digeridos con punta desalinizante C18
- Ajuste la pipeta a 10 μL y coloque la punta desalinizadora C18. Humedece la punta desalinizante pipeteando 10 μL de 100% ACN y luego deséchela. Repita este paso 2 veces.
- Lave la punta desalinizante C18 pipeteando 10 μL de 50 % de ACN, 49,8 % de agua y 0,2 % de FA y luego deséchela. Repita este paso 2 veces.
- Equilibre la punta desalinizante de C18 pipeteando 10 μL de 0,2% de FA en agua y luego desechándolo. Repita este paso 2 veces.
- Ajuste la pipeta a 10 μL y cargue los péptidos de la solución a la resina pipeteando los péptidos digeridos a través de la resina hacia arriba y hacia abajo 15 veces.
NOTA: La acción repetida asegurará la unión de todos los péptidos a la resina de las puntas desalinizadoras. - Desala los péptidos digeridos unidos a la resina pipeteando 10 μL de AG al 0,2% en agua y luego desechándolos. Repita este paso 4 veces para completar el paso de desalinización.
- En un tubo nuevo, eluya los péptidos con 10 μL de 50 % de ACN, 49,8 % de agua y 0,2 % de FA pipeteando 10 veces hacia arriba y hacia abajo. Repita este paso una vez para eluir la digestión de proteínas por segunda vez en el mismo tubo.
- Seque completamente los péptidos desalados en un concentrador de vacío (~20 min).
- Resuspender péptidos en 15 μL de 0,2% de FA + 1 μL de péptidos iRT (tiempo de retención indexado). Voxtex durante 10 minutos, centrifugar a 12.000 x g durante 2 minutos y, a continuación, transferirlo a un vial de muestreador automático para el análisis de MS (ver más abajo).
5. Análisis por espectrometría de masas de péptidos digeridos mediante DDA y DIA
NOTA: Las muestras se pueden analizar utilizando los métodos DDA o DIA LC-MS/MS. En este estudio, las muestras se analizaron utilizando un sistema de HPLC nano-LC 2D acoplado a un espectrómetro de masas de alta resolución.
- Utilice un sistema de HPLC combinado con un sistema de HPLC basado en chip conectado directamente a un espectrómetro de masas de tiempo de vuelo cuadrupolar (también se pueden utilizar otras configuraciones y sistemas de LC-MS).
- Analice muestras utilizando HPLC-ESI-MS/MS de fase reversa.
- Construya el protocolo de cromatografía para cargar la muestra de péptidos en un chip de precolumna C18. Lavar y desalar los péptidos cargados con disolvente de carga (ácido fórmico al 0,1%) durante 10 min a un caudal de 2 μL/min.
- Transfiera los péptidos a un chip de columna analítica C18 y eluya a un caudal de 300 nL/min con un gradiente de 3 horas utilizando la fase móvil A (2% de acetonitrilo, 0,1% de ácido fórmico) y B (98% de acetonitrilo, 0,1% de ácido fórmico). La primera etapa de elución consiste en un gradiente lineal de 5% B a 35% B durante un período de 80 min.
- Aumente la fase móvil B al 80 % durante 5 minutos y, a continuación, mantenga la fase móvil B al 80 % durante 8 minutos antes de cambiar al 5 % B para volver a equilibrar la columna durante 25 minutos.
- Configure un método de instrumento MS para DDA y defina los parámetros del instrumento de la siguiente manera.
- Utilice los siguientes parámetros para el escaneo de iones precursores del Experimento 1:MS1 de m/z 400–1500 (tiempo de acumulación de 250 ms); establecer el umbral de intensidad para activar escaneos MS/MS para iones de estados de carga de 2 a 5 a 200 recuentos; Establezca la exclusión dinámica de iones precursores en 60 s).
- Utilice los siguientes parámetros para el Experimento 2: Escaneos de iones de productos MS/MS con un rango de escaneo de MS2 de m/z 100–1500 (tiempo de acumulación de 100 ms por cada uno de los 30 escaneos de iones de productos por ciclo); establezca la propagación de energía de colisión en CES = 5 y marque 'modo de escaneo de iones de productos de alta sensibilidad'.
NOTA: El método de adquisición DDA se utiliza para construir bibliotecas espectrales como se describe en el paso 6.3 (véase más abajo). Aquí, adquirirá espectros MS/MS para los 30 iones precursores más abundantes después de cada escaneo MS1 por ciclo; El tiempo total del ciclo es de unos 3,3 s.
- Configure un método de instrumento MS para DIA y defina los parámetros instrumentales de la siguiente manera.
- Utilice los siguientes parámetros para el Experimento 1: Escaneo de iones precursores MS1 de m/z 400-1250 (tiempo de acumulación de 250 ms).
- Utilice los siguientes parámetros para el Experimento 2: Escaneos de iones de productos MS/MS para 64 segmentos SWATH variables con un rango de escaneo MS2 de m/z 100-1500 (tiempo de acumulación de 45 mseg por cada uno de los 64 escaneos de iones de productos por ciclo); establezca la dispersión de energía de colisión en CES = 10, marque la marca 'modo de escaneo de iones de producto de alta sensibilidad'.
NOTA: Utilice el método de adquisición DIA/SWATH de ventana de 64 variables descrito por Schilling et al.13 para realizar una cuantificación sin etiquetas con un tiempo de ciclo total de aproximadamente 3,2 s. Para la adquisición de DDA, en lugar de utilizar el cuadrupolo Q1 para transmitir el ion precursor de un rango de masa estrecho a la celda de colisión, se utiliza un rango de ancho de ventana variable (por ejemplo, m/z 5–90) para pasar incrementalmente sobre el rango m/z completo (m/z 400–1250) con 64 segmentos SWATH, 45 ms de tiempo de acumulación para cada segmento, lo que da como resultado un tiempo de ciclo de 3,2 segundos, que incluye un escaneo MS1 con un tiempo de acumulación de 250 ms. El ancho de ventana variable se ajusta de acuerdo con la complejidad de la corriente de iones MS1 típica observada dentro de un cierto rango m/z utilizando un algoritmo SCIEX 'calculadora de ventana variable'13 (las ventanas más estrechas se aplican a los rangos m/z 'ocupados', las ventanas más anchas se aplican a los rangos m/z con pocos iones precursores eluyentes). En otras plataformas de MS, también se pueden implementar estrategias alternativas de selección de ventanas DIA.
6. Análisis de datos
NOTA: Ciertas configuraciones de análisis de datos deben adaptarse a condiciones experimentales específicas. Por ejemplo, la base de datos de proteínas (fichero fasta) seleccionada dependerá de la especie a partir de la cual se preparó la muestra (en este protocolo utilizamos C. elegans).
- Utilice un motor de búsqueda de base de datos de EM para analizar las adquisiciones de DDA e identificar proteínas. Genere un método de motor de búsqueda darabase de la siguiente manera:
- En Parámetros de descripción de muestra, seleccione Identificación; en Tipo de muestra, seleccione Ácido yodoacético; en Alquilación de cisteína, seleccione Tripsina; en Digestión (suponiendo la escisión C-terminal en lisina y arginina), seleccione el nombre del espectrómetro de masas en Instrumento; y seleccione Caenorhabditis elegans en Especie.
- Para los parámetros de procesamiento específicos, marque Modificaciones biológicas; en ID Focus, seleccione SwissProt; en Base de datos, marque ID completo; en Esfuerzo de búsqueda, seleccione 0,05 (10 %) en Umbral de proteína detectada; y marque Ejecutar análisis de tasa de detección falsa en Calidad de resultados. Guarde el método del motor de búsqueda y envíe los archivos sin procesar de MS al motor de búsqueda de la base de datos para su procesamiento, utilizando el método generado.
NOTA: En un proceso iterativo, el motor de búsqueda recalibró automáticamente todos los escaneos MS y MS/MS en función de las anotaciones y los resultados iniciales.
- Haga clic en Exportar resumen de péptidos al finalizar la búsqueda y filtre todos los resultados de identificación de péptidos por el umbral de confianza de 99 en Excel (tasa de descubrimiento falso (FDR) = 1%).
- Cree bibliotecas espectrales MS/MS para el procesamiento posterior del archivo de datos sin procesar DIA y para una mayor cuantificación de datos relativos.
- Abra el software de análisis cuantitativo DIA. Seleccione la pestaña Biblioteca y luego (en la parte inferior de la página) haga clic en Generar biblioteca espectral desde "Motor de búsqueda de base de datos" y abra un informe FDR de "Motor de búsqueda de base de datos" (el archivo de hoja de cálculo *FDR.xlsx) que se generó automáticamente como parte del proceso de búsqueda de la base de datos de archivos de datos sin procesar de DDA.
- Haga clic en Siguiente, seleccione el esquema de configuración de biblioteca y haga clic en Siguiente. Seleccione Uniprot_Caenorhabditis elegans _proteome como base de datos y luego haga clic en Finalizar, se generará la biblioteca espectral.
- Utilice el software de análisis cuantitativo DIA para analizar las adquisiciones de DIA para una cuantificación completa de proteínas relativas. La canalización de análisis de datos de DIA se describe a continuación.
- Para analizar y cuantificar péptidos, abra el software de análisis cuantitativo DIA para configurar un esquema de análisis mediante el esquema de plantilla. El esquema de plantilla está disponible en el software haciendo clic en Configuración, luego en Análisis DIA y, por último, en Configuración de fábrica de BGS.
- Configure los parámetros en el esquema de plantilla de la siguiente manera:
1) en identificación , seleccione Localización PTM (límite de probabilidad = 0,75).
2) en cuantificación, seleccione Cantidad de grupo principal como Cantidad de péptidos de suma, configure el grupo principal N como máximo 7 y mínimo 1, seleccione Cantidad de grupo menor como cantidad precursora de suma y configure el grupo menor N como máximo 10 y mínimo 1, y seleccione Filtrado de datos como Qvalue disperso y no seleccione Normalización de ejecución cruzada.
- Realizar análisis de cuantificación relativa.
- Seleccione la pestaña Canalización , haga clic en Configurar un análisis DIA a partir de archivo, abra los archivos sin procesar de MS DIA de interés (por ejemplo: muestra y control), haga clic en Asignar biblioteca espectral, seleccione la biblioteca que se generó en 6.3, haga clic en Cargar y, por último, haga clic en Siguiente.
- Seleccione el esquema de análisis configurado en 6.5.1 y haga clic en Siguiente. Seleccione el archivo fasta de la base de datos apropiado (en este protocolo: Uniprot_Caenorhabditis elegans_proteome) y haga clic en Siguiente. Defina la configuración de la condición y asigne las diferentes condiciones de acuerdo con las muestras y haga clic en Siguiente.
- Revise la descripción general del análisis (resumen de la configuración del experimento), vaya al directorio de salida y haga clic en Finalizar. Por último, haga clic en Ejecutar canalización para realizar el análisis cuantitativo sin etiquetas.
NOTA: Los módulos estadísticos del 'Software de análisis cuantitativo DIA' realizan automáticamente análisis FDR, generan mapas de calor y gráficos de volcanes comparando las diferentes condiciones, generan listas de péptidos y proteínas identificados y cuantificados y proporcionan valores Q junto con cambios de plegamiento relativos que comparan diferentes condiciones. Aquí, las condiciones de la muestra incluyeron gusanos de día 10 con dos réplicas biológicas y gusanos jóvenes del día 2 con dos réplicas biológicas como control.
Resultados
Los métodos tradicionales de lisis de lombrices tienen varias desventajas. Por ejemplo, los métodos de sonicación y batido de perlas basados en sondas producen un calor excesivo al permitir el contacto de la punta o las perlas metálicas directamente con las muestras, lo que da lugar a recuperaciones de proteínas variables y a la desnaturalización de las mismas. La molienda de nitrógeno líquido seguida de sonicación en tampón de lisis puede llevar mucho tiempo y requiere un gran número de gusanos. Debido a las limitaciones de los métodos tradicionales de lisis de gusanos, los flujos de trabajo de MS anteriores, como los métodos de etiquetado iTRAQ o los métodos sin etiquetas que se han utilizado históricamente en el sistema modelo de C. elegans para obtener información cuantitativa sobre el insolubloma, requieren una gran entrada de material de partida (al menos 40.000 gusanos). Se requiere un laborioso trabajo de cultivo de lombrices para obtener esta cantidad de lombrices. Además, los métodos de etiquetado requieren costosos marcadores químicos isobáricos. Los métodos de cuantificación sin etiquetas son rentables y tienen métodos de preparación y etiquetado de muestras más fáciles y sencillos, pero requieren un número significativamente grande de gusanos para lograr una cobertura suficiente de análisis de MS.
El sonicador que hemos utilizado aumenta en gran medida la eficiencia y la reproducibilidad de la lisis de lombrices mediante la lisis simultánea de varias muestras de gusanos en un sonicador de baño de agua a temperatura controlada sin contaminación cruzada14, reduciendo así significativamente la cantidad de material de gusano de partida necesario. Combinando el método de sonicación altamente eficiente y el enfoque cuantitativo de MS sin marcadores DIA, pudimos cuantificar de forma robusta el insolubioma de gusanos viejos y jóvenes utilizando ~3.000 gusanos. Aquí probamos y validamos la eficiencia del protocolo y comparamos la insoluloma de gusanos viejos y jóvenes de una cepa de gusano de tipo salvaje, N2-Bristol C. elegans. Aplicamos este protocolo para extraer y aislar el insoluloma de ~3.000 C . elegans N2 jóvenes y envejecidos (dos réplicas biológicas para cada condición), seguido de un análisis de MS con un espectrómetro de masas de tiempo de vuelo cuadrupolar u otros sistemas de MS utilizando una combinación de adquisición dependiente de datos (DDA) y adquisiciones independientes de datos (DIA / SWATH) para la identificación y cuantificación de proteínas. Las proteínas insolubles se analizaron primero en un gel Bis-Tris con un gradiente de 4-12% para determinar la cantidad de proteína en cada muestra de insolubloma. Como se demuestra en la Figura 2, la muestra de insolubioma de gusanos envejecidos N2 (carriles 2 y 3, experimentos de replicación biológica) tiene significativamente más proteína que las muestras de los gusanos jóvenes N2 (carriles 1 y 4, experimentos de replicación biológica).
Después de la digestión en gel, los perfiles proteicos del insolubioma se analizaron mediante HPLC-MS. Con este flujo de trabajo, generalmente podemos identificar entre 1000 y 1500 proteínas y cuantificar entre 500 y 1.000 proteínas a partir de la fracción insoluble en SDS con alta reproducibilidad (datos no publicados). Aquí pudimos cuantificar 989 proteínas del insolubioma de N2-Bristol C. elegans mediante el análisis de los datos de DIA y la eliminación de la redundancia: 768 proteínas se enriquecieron significativamente y 27 proteínas se redujeron significativamente en el insoluloma del gusano N2 envejecido (día 10) en comparación con el joven (día 2) utilizando un cambio de pliegue de al menos 1,5 y un valor Q de menos de 0,01 (Figura 3A). Como se ve en el gráfico del histograma (Figura 3B), el cambio de pliegue de las proteínas significativamente alteradas muestra una distribución normal. Se demostró que los gusanos envejecidos estaban significativamente enriquecidos para el insolubloma: el mayor cambio observado mostró que la abundancia relativa de proteínas en el insolubloma era 592 veces mayor en los gusanos viejos que en los jóvenes; Y para 32 proteínas, la abundancia relativa de proteínas en el insolubioma fue >250 veces mayor en los gusanos viejos que en los jóvenes, lo que indica cambios dramáticos en el insolubioma con la edad.
Después de extraer la lista de proteínas insolubles que están significativamente aumentadas en los gusanos envejecidos e identificadas por la base de gusanos (WS271), se realizó un análisis de la vía KEGG y de la ontología génica (GO) para determinar las vías que se enriquecen en el insoluloma envejecido para obtener información biológica sobre cómo se relacionan con el envejecimiento. El análisis de la vía KEGG de las proteínas identificadas en este estudio muestra el enriquecimiento de varias vías que involucran ribosomas, mitocondrias, proteasoma y espliceosoma (Figura 4A). El análisis de la ontología génica muestra que el insolubioma de los gusanos envejecidos comprende muchas proteínas en categorías particulares, incluidas las proteínas mitocondriales, de desarrollo, determinantes de la vida adulta y ribosómicas (Figura 4B y Tabla suplementaria 1A). A continuación, comparamos la lista de proteínas identificadas en este estudio con el trabajo publicado previamente por David et al.2 y Mark et al.11, como se demuestra en los diagramas de Venn (Figura 5A,5B). La comparación mostró una superposición significativa de las proteínas identificadas 394/721 y 444/721 con el estudio de David et al (Figura 5A) y Mark et al. (Figura 5B), respectivamente. Las vías biológicas reveladas por el análisis KEGG de insoluloma de este estudio también se han identificado en el pasado, validando así nuestra metodología (Tabla suplementaria 1B). La identificación de estas vías y proteínas sugiere que pueden servir como candidatos para futuras investigaciones biológicas con respecto a su función en el contexto del envejecimiento.
En resumen, el uso del eficiente método de sonicación permite la lisis de múltiples muestras de lombrices al mismo tiempo en un entorno con temperaturas bien controladas y una contaminación cruzada reducida para lograr una alta cobertura de proteínas con un número significativamente menor de material de lombriz de partida. La combinación del eficiente método de sonicación con un flujo de trabajo de cuantificación de proteínas sin etiquetas DIA ha proporcionado resultados fiables y reproducibles para la cuantificación de proteínas insolubles de gusano.

Figura 1. Flujo de trabajo experimental del protocolo. C. elegans se cultivó y colectó en diferentes días. Después de la lisis de lombrices con un sonicador, se extrajo y aisló del lisado la fracción de proteína insoluble en SDS al 1% (insolubloma). A continuación, el insolubioma se digierió mediante digestión con tripsina en gel y se cuantificó mediante espectrometría de masas DIA, seguido de un análisis bioinformático. Haga clic aquí para ver una versión más grande de esta figura.
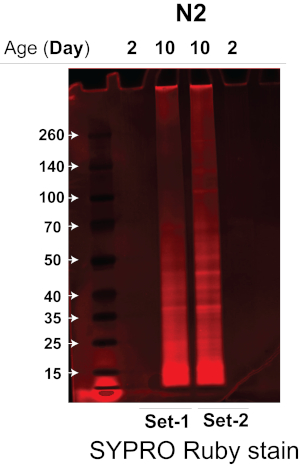
Figura 2. SDS-PAGE gel del insolubioma aislado de gusanos jóvenes frente a viejos de la cepa N2-Bristol. Los insolublomas de los gusanos jóvenes frente a los viejos de la cepa N2-Bristol se analizaron mediante SDS-PAGE para determinar la cantidad de proteína presente. El gel SDS-PAGE se tiñó con una tinción de proteína fluorescente para visualizar las bandas de proteínas. Carriles 1 y 4: Insolubioma de dos experimentos de réplicas biológicas de gusanos N2 jóvenes (Día 2). Carriles 2 y 3: Insolubioma de dos experimentos biológicos replicados de gusanos N2 envejecidos (Día 10). Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Se identificó que las proteínas candidatas mostraban una alteración significativa en el insolubioma envejecido frente al joven y sus distribuciones de cambio de pliegue. (A) Diagrama volcánico para la cuantificación de la insoluloma de gusanos N2-Bristol envejecidos frente a gusanos N2 jóvenes. Los candidatos con un cambio de pliegue absoluto >=1,5 y un valor Q <0,01 se muestran como puntos rojos. (B) Gráfico de histograma para la distribución de cambio de pliegue de proteínas insolubles en SDS significativamente enriquecidas en muestras de gusanos envejecidos frente a jóvenes. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Análisis de la vía KEGG y de la ontología génica (GO). (A) Análisis de la vía KEGG del insolubioma del día 10 ordenado según el valor p con la vía altamente significativa mostrada en la parte superior. (B) El análisis de ontología génica muestra que el insoluloma de los gusanos envejecidos está enriquecido para muchas proteínas en categorías particulares, incluidas las proteínas mitocondriales, de desarrollo, determinantes de la vida adulta y ribosómicas. La vista de diagrama de dispersión visualiza los términos GO en un "espacio semántico" donde los términos más similares se colocan más juntos. El color de la burbuja refleja el valor p obtenido en el análisis STRING, mientras que su tamaño refleja la generalidad del término GO en la base de datos UniProt-GOA. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5. Se identificó la superposición de proteínas del insolubloma en el día 10 comparando este estudio con los estudios (A) David et al.2 y (B) Mark et al.11 . Haga clic aquí para ver una versión más grande de esta figura.
Tabla complementaria 1 (relacionada con la Figura 4 y la Figura 5).(A) Ontología génica (proceso biológico) analizada con la base de datos STRING. (B) Lista detallada de proteínas y vías KEGG identificadas en este estudio con códigos de colores que representan su superposición con el trabajo publicado. Haga clic aquí para descargar este archivo.
Discusión
En este protocolo, reportamos un método mejorado de preparación de muestras para la extracción de proteínas insolubles de C. elegans. Al sustituir la lisis tradicional de los gusanos (por ejemplo, la sonicación con sonda o las técnicas de batido de perlas) por el eficiente sonicador, aumentamos el rendimiento de la extracción de proteínas insolubles y redujimos el número de gusanos necesarios para el análisis de EM sin marcadores de 40.000 a 3.000 gusanos. Se utilizó un motor de búsqueda de base de datos para la identificación de proteínas a partir de datos de DDA y la biblioteca espectral de C. elegans se construyó utilizando el 'DIA Quantitative Analysis Software' y los resultados de búsqueda de la base de datos DDA correspondientes (importando informes de archivos FDR generados por el motor de búsqueda de la base de datos). La cuantificación relativa de los datos de insoluloma de C. elegans envejecidos y jóvenes se llevó a cabo utilizando el 'Software de análisis cuantitativo DIA' para procesar el nuevo conjunto de datos DIA y la biblioteca espectral generada.
Varios pasos son críticos en el protocolo. La corta vida útil de C. elegans lo convierte en un sistema ideal para estudiar el envejecimiento, en comparación con otros eucariotas como las células de mamíferos, pero es crucial aislar una población homogénea de gusanos cuando se estudian los fenómenos relacionados con el envejecimiento. En este protocolo se utilizó FUDR para obtener gusanos envejecidos sincronizados. Es importante transferir los gusanos al principio de su etapa L4 a la placa sembrada con NGM que contiene FUDR para garantizar su efectividad. Durante la lisis de lombriz con el eficiente sonicador, la temperatura del baño de agua debe ajustarse a 4 °C y la sonicación a 30 s ON y 30 s OFF para evitar el sobrecalentamiento de las muestras. Después de la primera ronda de sonicación durante 10 ciclos (10 min), es importante comprobar bajo el microscopio para asegurarse de que todos los gusanos se han lisado de forma eficiente. De lo contrario, se necesitan más ciclos de sonicación. Durante el proceso de digestión en gel, cada rebanada de gel debe cortarse en trozos del tamaño adecuado (<1 mm2), si son demasiado pequeños, pueden perderse en el proceso de preparación de la muestra, y si son demasiado grandes, la digestión puede ser inadecuada.
La necesidad de mucho menos material de partida reduce significativamente el laborioso trabajo asociado con el cultivo de gusanos para obtener muestras para análisis de insoluboloma. Sin embargo, la extracción y el aislamiento de la fracción de proteína insoluble en SDS al 1% implica múltiples pasos de lavado y se requiere un manejo cuidadoso de la muestra para evitar la pérdida de la muestra y garantizar resultados reproducibles. La cantidad de material generado para el análisis de MS es suficiente para ~ 3 inyecciones para el análisis posterior de DDA y DIA, pero no para guardar para futuros experimentos. Además, a pesar de sus efectos potencialmente confusos15, utilizamos la concentración más baja posible de FUdR para esterilizar gusanos durante el proceso de envejecimiento. Es posible que estudios futuros eludan el uso de FUdR mediante el uso de mutantes estériles o mediante la transferencia y recolección manual de gusanos.
El uso del sonicador altamente eficiente para la lisis de lombrices permite una extracción eficiente del insolubloma, lo que permite una buena cobertura de proteínas y un análisis rentable de DIA MS sin etiquetas para cuantificar el insolubioma utilizando un número muy reducido de gusanos. Reduce significativamente la carga de trabajo, lo que permite la detección de más condiciones por experimento. Además, el flujo de trabajo MS DIA sin etiquetas es rentable y proporciona una profundidad y cobertura de proteínas a niveles comparables a los métodos de etiquetado, incluidos iTRAQ, TMT o SILAC. El modelo de C. elegans es un sistema de cribado rápido para la investigación del envejecimiento. El flujo de trabajo se puede modificar y aplicar fácilmente para estudiar el envejecimiento y la investigación de enfermedades relacionadas con la edad en este y otros organismos. Por ejemplo, en estudios en curso, estamos aplicando este flujo de trabajo para investigar los perfiles de proteínas de insolubloma y proteostasis en varios modelos de C . elegans con enfermedad de Alzheimer (EA), incluidos los gusanos Abeta, tau y Abeta/tau duales con o sin diferentes intervenciones farmacológicas para el futuro cribado de fármacos de alto rendimiento.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por una subvención de instrumentación compartida de los NIH para un sistema TripleTOF (1S10 OD016281, Buck Institute), una subvención de los NIH, RF1 AG057358 (GJL, JKA) y una subvención de los NIH U01AG045844 (GJL). XX cuenta con el apoyo de una beca postdoctoral T32 (beca NIH 5T32AG000266, PI: Judith Campisi y Lisa Ellerby). MC cuenta con el apoyo de una beca postdoctoral de la Fundación Larry L. Hillblom.
Materiales
| Name | Company | Catalog Number | Comments |
| Strains used | |||
| Esherichia coli OP50 | Caenorhabditis Genetics Center (CGC) | ||
| N2 (Bristol) | Caenorhabditis Genetics Center (CGC) | ||
| Buffer/Solution | |||
| NGM (Nematode Growth Media) | Recipe: 3 g/L NaCl, 23 g/L agar; 2.5 g/L peptone; 1 mM CaCl2, 5 mg/L cholesterol, 1 mM MgSO4, 25 mM KH2PO4 | ||
| S-basal solution | Recipe: 5.85 g/L NaCl, 1g/L K2HPO4, 6 g/L KH2PO4, H2O to 1 L | ||
| Sodium hypochlorite bleach solution | Recipe: Mix 0.5 mL 5 N NaOH with 1 ml Sodium hypochlorite (5%) and make volume to 5 mL with H20. | ||
| Material/ Equipment | |||
| Agar | Difco Granulated Agar, BD Biosciences | 90000-782 | |
| Bioruptor Plus sonication device | Diagenode, USA | B01020001 | |
| Cholesterol | Sigma | c8503 | |
| 2'-deoxy-5-fluorouridine | VWR | TCD2235 | |
| Glycerol | Millipore Sigma | 356350-1000ML | |
| LB broth, Miller | Millipore Sigma | 60801-450 | |
| Sodium dodecyl sulfate (SDS? | Sigma | L4509-250G | |
| Sodium chloride | Sigma | 59888 | |
| M880 Ultrasonic bath, 117 V, holds 5.5 gallons | VWR, USA | 89375-458 | |
| Magnesium sulphate | Sigma | M506 | |
| Magnesium chloride | Sigma | 208337 | |
| NGM agar plate | VWR Disposable Petri Dishes | 25384-342 | |
| NuPAGE LDS Sample Buffer (4X) | Thermo Fisher Scientific | NP0007 | |
| NuPAGE protein gels, 4-12% | Invitrogen | NP 0335BOX | |
| Protease inhibiotr cocktail (PIC) | Roche | 11836170001 | |
| Pierce BCA Assay | Thermo Fisher Scientific | 23225 | |
| Sodium hypochlorite 5% | VWR | JT9416-1 | |
| SYPRO Ruby Protein Gel Stain | Thermo Fisher Scientific | S12000 | |
| MS Section | |||
| Acetonitrile, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 36XL66 | |
| Agilent Zorbax 300Extend C18 column | Agilent Technologies Inc., Santa Clara, CA, USA | 770995-902 | |
| Ammonium bicarbonate | Sigma Aldrich, St. Louis, MO, USA | 9830 (1 kg) | |
| Dithiothreitol (DTT) | Sigma Aldrich, St. Louis, MO, USA | D9779-5G | |
| Eppendorf Thermomixer Compact | Eppendorf AG, Hamburg, Germany | T1317-1EA | |
| Formic acid | Sigma Aldrich, St. Louis, MO, USA | F0507-500ML | |
| Indexed retention time (iRT) normalization peptide standard | Biognosys AG, Schlieren, Zurich, Switzerland | Ki-3002-2 | |
| Iodoacetamide (IAA) | Sigma Aldrich, St. Louis, MO, USA | I1149-25G | |
| Methanol, HPLC Grade | Honeywell International Inc., Charlotte, NC, USA | 34885 | |
| Nano cHiPLC Trap ChromXP C18-CL, 200 um x 6 mm, 3 um, 120A. (pre-column chip) (200 um x 6 mm ChromXP C18-CL chip, 3 um, 300 A) | Sciex LLC, Framingham, MA, USA | 804-00006 | |
| Nano cHiPLC ChromXP 75 um by 15cm, C18-CL, 3 um, 120 A (analytical column chip) | Sciex LLC, Framingham, MA, USA | 804-00001 | |
| Orthoganol quadrupole time-of-flight (QqTOF) TripleTOP 6600 mass spectrometer | Sciex LLC, Framingham, MA, USA | Per quote | |
| ProteinPilot 5.0 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Savant SPD131DDA Speedvac Concentrator | Thermo Fisher Scientific, Waltham, MA, USA | SPD131DDA-115 | |
| Sequencing-grade lyophilized trypsin | Life Technologies | 23225 | |
| Spectronaut | Biognosys AG, Schlieren, Zurich, Switzerland | Sw-3001 | |
| SWATH 2.0 plugin into PeakView 2.2 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Ultra Plus nano-LC 2D HPLC system | Sciex LLC, Eksigent Division, Framingham, MA, USA | Model # 845 | |
| Water, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 600-30-76 | |
| Waters 1525 binary HPLC pump system | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters 2487 Dual Wavelength UV detector | Waters Corp., Milford, MA, USA | WAT081110 | |
| Waters 717plus Autosampler | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters Fraction Collector III | Waters Corp., Milford, MA, USA | 186001878 |
Referencias
- Walther, D. M., et al. Widespread Proteome Remodeling and Aggregation in Aging C. elegans. Cell. , 919-932 (2015).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Labbadia, J., Morimoto, R. I. The Biology of Proteostasis in Aging and Disease. Annual Review of Biochemistry. (1), 435-464 (2013).
- Ross, C. A., Poirier, M. A. Protein aggregation and neurodegenerative disease. Nature Medicine. , 10-17 (2004).
- Reis-Rodrigues, P., et al. Proteomic analysis of age-dependent changes in protein solubility identifies genes that modulate lifespan. Aging Cell. 11 (1), 120-127 (2012).
- Ayyadevara, S., et al. Proteins that accumulate with age in human skeletal-muscle aggregates contribute to declines in muscle mass and function in Caenorhabditis elegans. Aging. 8 (12), 3486-3497 (2016).
- Huang, C., et al. Intrinsically aggregation-prone proteins form amyloid-like aggregates and contribute to tissue aging in Caenorhabditis elegans. eLife. 8, (2019).
- Morimoto, R. I. Proteotoxic stress and inducible chaperone networks in neurodegenerative disease and aging. Genes and Development. 22 (11), 1427-1438 (2008).
- Morimoto, R. I., Cuervo, A. M. Protein homeostasis and aging: Taking care of proteins from the cradle to the grave. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 64 (2), 167-170 (2009).
- Klang, I. M., et al. Iron promotes protein insolubility and aging in C. elegans. Aging. 6 (11), 975-991 (2014).
- Mark, K. A., et al. Vitamin D Promotes Protein Homeostasis and Longevity via the Stress Response Pathway Genes skn-1, ire-1, and xbp-1. Cell Reports. 17 (5), 1227-1237 (2016).
- Groh, N., et al. Methods to study changes in inherent protein aggregation with age in caenorhabditis elegans. Journal of Visualized Experiments. (129), 1-12 (2017).
- Schilling, B., Gibson, B. W., Hunter, C. L. Generation of High-Quality SWATH((R)) Acquisition Data for Label-free Quantitative Proteomics Studies Using TripleTOF((R)) Mass Spectrometers. Proteomics: Methods and Protocols, Methods in Molecular Biology. 1550, 223-233 (2017).
- Walther, D. M., et al. Widespread proteome remodeling and aggregation in aging C. elegans. Cell. 161 (4), 919-932 (2015).
- Angeli, S., et al. A DNA synthesis inhibitor is protective against proteotoxic stressors via modulation of fertility pathways in Caenorhabditis elegans. Aging. 5 (10), 759-769 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados